Method Article
3D多色DNAFISH工具研究人类原细胞核结构
摘要
3D 多色 DNA FISH 是一种工具,用于可视化 3D 保留核内的多个基因组位点,明确定义其在单个细胞级别的核空间内的相互交互和定位。在这里,描述了一个分步协议,用于广泛的人类原细胞。
摘要
细胞生物学的一个主要问题是核空间内的基因组组织,以及染色质结构如何影响基因表达、细胞分化和分化等过程。研究基因组的3D架构的许多方法可以分为两个互补的类别:基于染色体构象捕获的技术(C技术)和成像。前者基于捕获固定细胞群中的染色体构象和近端DNA相互作用,而后者基于DNA荧光原位杂交(FISH)的3D保存核,允许当代可视化单个细胞水平(多色)中的多个位点,检查其在细胞核内的相互作用和分布(3D多色DNA FISH)。3D多色DNAFISH技术只对几个预定位点进行可视化,无法对核结构进行全面分析。然而,鉴于其结果的鲁棒性,3D多色DNA FISH与3D显微镜和图像重建相结合是验证基于C技术的结果和明确研究特定位点的位置和组织的可能方法在单个单元格级别。在这里,我们提出了一个适合广泛的人类原细胞的3D多色DNAFISH的一步一步方法,并讨论了获得成功和翔实的3D多色所需的所有实际行动、关键步骤、3D成像概念和数据分析不同生物背景下的DNAFISH。
引言
较高的真核细胞需要系统地压缩和压缩大量的遗传信息在一分钟的3D空间的细胞核1,2,3,4。今天,我们知道基因组在空间上按隔间和拓扑相关域5排序,DNA折叠的多级在不同基因组区域之间产生接触,可能涉及染色质环形成6,7。染色质的3D动态循环可以影响许多不同的生物过程,如转录8、9、分化和发育10、11、DNA修复12、13,而其扰动涉及各种疾病14、15、16和发育缺陷15、17、18。
已经开发出许多方法来研究3D基因组组织。染色体构象捕获技术(C技术,3C,4C,5C,高C和衍生物)已经开发,以研究基因组组织在固定细胞3,4,19,20。这些方法基于在物理接近的基因组位点之间捕获接触频率的能力。C-技术,根据其复杂性,捕捉全球3D基因组组织和细胞群的3,4,19,20的核拓扑。然而,3D交互在时间和空间上是动态的,由多路复用相互作用组成的单个细胞之间的高度可变,并且广泛是异质的21,22。
3D 多色DNA 荧光原位杂交 (FISH) 是一种允许在单个细胞级别上可视化特定基因组位点的技术,能够以与 C 技术互补的方式直接研究 3D 核架构。它表示当前用于明确验证 C 结果的技术。3D 多色 DNA FISH 使用荧光标记探针,与兴趣的基因组位点互补。使用不同的荧光镜和合适的显微镜设备,使核空间23、24内多个目标的当代可视化。近年来,FISH与显微镜技术的进步相结合,获得了高分辨率25、26或CRISPR-Cas方法的精细结构可视化,用于在活成像27、28中实现核酸可视化。尽管广泛采用,3D 多色DNA FISH 方法在许多实验室中仍然被认为是困难的,因为所使用的生物材料必须加以调整。
在这里,我们为适用于各种人类原细胞的3D多色DNAFISH(从细胞/探针制备到数据分析)提供全面协议,实现多个基因组位点的可视化并保留核的3D结构。为了研究核结构,必须保留核的三维结构。因此,与其他现有协议29,30,31相比,我们避免使用酒精梯度和酒精中可能影响染色质结构32的盖玻片。该方法根据保存的3DDNAFISH协议24,33改编,适用于广泛的人类原细胞,包括分离的体外或体外培养。不同的核形态和细胞学特征(例如,不同程度的核压实、细胞骨架丰度)有渗透和脱蛋白参数34。这些参数通常在其他协议24,33中描述,但没有提供不同细胞类型中程序的明确区分。此外,我们开发了一个名为NuCL_D(3D中的核接触定位器)16的特定工具,为数据分析提供了原理,从而以自动方式改进不同位点之间的3D接近及其核拓扑分布。
研究方案
1. DNA探针制备和标签程序,带尼克翻译
- 净化和清洁细菌人工染色体(BACs),质粒或PCR产品与特定的试剂盒(材料表),重新悬浮在ddH2O,通过电泳检查在胶质和定量。
- 根据表1,在50μL的最终体积中混合所有试剂,对步骤1.1的1.5⁄2 μgDNA执行尼克翻译。
注:直接贴标签的探头可以改善信噪比。通过 nick 翻译,用于生产带有各种 Alexa 氟铬的间接和直接标签的探头的套件已上市。 - 根据起始DNA材料的长度,在16°C的热混合器中孵育nick翻译混合物一段时间:PCR产物(每个PCR产品池为2,000bp),BAC DNA和质粒长达4小时。
- 检查在步骤 1.3 中由电泳在 2.2% 甘蔗凝胶上产生的探头的大小。
注: 最佳探头尺寸为 <200 bp (图 1A)。- 如果DNA消化不够,在反应中加入5U的DNA聚合酶I和0.05U的DNase I,并在16°C下孵育1⁄2小时,然后重新检查。用 0.5 mM EDTA(最终浓度)停止反应。将探头储存在-20°C。
- 对于每个DNA FISH实验,根据产生探针的起始DNA材料沉淀以下数量的探针:200纳克从nick翻译PCR产品,100纳克从nick翻译BACs,或300 ng从尼克翻译质粒。加入ddH2O至150μL、20 μg未标记鲑鱼精子DNA、3.5微克物种特异性Cot-1 DNA、3卷100%EtOH和1/10体积3M醋酸钠pH 5.2。在-80°C下沉淀1小时。
- 在4°C下以最大速度离心1小时,并丢弃上清液。用 70% EtOH 洗涤颗粒两次。
- 将颗粒重新悬浮在 2 μL 的 100% 形式酰胺 pH 7.0 中,在 40°C 下摇动 30 分钟(可能需要几个小时),然后加入相当于 4x 盐碱-柠酸钠 (SSC)/20% 的硫酸盐。
- 通过将 175.3 g 的 NaCl 和 88.2 g 柠酸钠混合在 1 L H2O. 高压灭菌器的最终体积中制备 20x SSC,过滤并制备等分。
注:对于多色DNA FISH,不同的探针可以沉淀在一起,但可以相互肛角的探针除外。在这些特定情况下,分别处理它们,沉淀并重新悬浮在步骤1.5_1.7中提及的试剂的探针中,相对于探针的数量。只有在以形式酰胺重新悬浮后,才能将探头集中。如果使用大于 10 mm 或小于 10 mm 的玻璃盖玻片,则向上/向下缩放步骤 1.5×1.7 中使用的试剂的体积,与滑动尺寸成比例。杂交探头可在-20°C下长期(最多两个月)储存。
- 通过将 175.3 g 的 NaCl 和 88.2 g 柠酸钠混合在 1 L H2O. 高压灭菌器的最终体积中制备 20x SSC,过滤并制备等分。
2. 细胞固定、预处理和渗透
注:渗透和脱蛋白通道是关键步骤。试剂的反应时间和浓度在很大程度上取决于细胞类型、细胞质丰度和核形态。
- 人类原发性T淋巴细胞的细胞固定、预处理和渗透
注:此方案适用于小细胞,小细胞核和小细胞质量。- 使用玻璃盖玻片(10 毫米,厚度 1.5H)。
- 用 ddH2O 清洗玻璃,然后用 70% EtOH 洗涤,然后晾干。对于 24 孔板的每个孔,使用一个玻璃杯。
- 直接在玻璃上加入200 μL 0.1%的聚L-莱辛(w/v)(材料表),形成落差。请注意,由于跌落必须留在玻璃表面,且不接触井2~5分钟。小心地将跌落排除在外,让玻璃干燥30分钟。
- 再执行步骤 2.1.3 两次。
- 将200 μL的悬浮细胞(2 x 106/mL,PBS悬浮在外体原发性T淋巴细胞)直接放在玻璃上,让细胞在室温(RT)下播种30分钟。
- 快速移除掉落。加入新鲜制作的4%PFA(在PBS/0.1%TWEEN 20中制备,pH 7.0,过滤)10分钟,并在RT固定种子细胞。
- 在 RT.渗透与 0.5% TPBS 在 RT 上洗涤 3 次,每次 5 分钟,使用 0.05% Triton X-100/PBS (TPBS)在 RT 上洗涤 10 分钟。
- 在250μL的PBS/孔中加入2.5 μL的RNase鸡尾酒(材料表),在37°C下加入1小时,以执行RNase治疗。
- 在PBS中冲洗,加入20%甘油/PBS,并在4°C下孵育过夜(ON)。
注: 此步骤的范围可以从 1 小时到 ON。幻灯片可在4°C至7天内保存在20%甘油/PBS中。 - 在干冰(15~30s)上冷冻,在RT处逐渐解冻,浸泡在20%的甘油/PBS中。再重复步骤 2.1.9 三次。
- 在 0.5% TPBS 中洗涤 5 分钟,在 RT.Wash 中,在 0.05% TPBS 中清洗,在 RT 中两次 5 分钟。在 0.1 N HCl 中孵育 12 分钟,在 RT 中冲洗 2x SSC。
- 在RT处孵育50%形式酰胺pH 7.0/2x SSC ON。
注:可以优化孵育时间并最终减少孵育时间。幻灯片可保持50%的甲酰胺/2x SSC数天。
- 人类原发性细胞细胞的细胞固定、预处理和渗透
注:此方案适用于具有大量细胞质的大细胞。- 在生长培养基(Dulbeco的改性鹰培养基(DMEM),20%胎儿牛血清(FBS),25纳克/mL纤维细胞生长因子(FGF),10纳克/mL表皮生长因子(EGF),10微克/mL人胰岛素,24孔板的盖玻片玻璃上直接生长人类原发性肌细胞。 1x谷氨酰胺,1x青霉素/链霉素),至少24小时(达到汇合的50-70%)。
注:为了便于粘附,明胶或胶原蛋白或多L-赖辛可用于在播种前涂覆盖玻片。 - 在PBS的2⁄3变化中冲洗细胞。加入新鲜制作的4%PFA,并在RT处固定细胞10分钟。
- 在 RT.渗透 0.5% TPBS 中,在 RT 中每洗 3 分钟 3 分钟,在 RT 中清洗 10 分钟。
- 在250μL的PBS/孔中加入2.5 μL的RNase鸡尾酒(材料表),在37°C下加入1小时,以执行RNase治疗。
- 在PBS中冲洗,加入20%甘油/PBS,并在RT孵育。
注: 此步骤的范围可以从 1 小时到 ON。幻灯片可在4°C至7天内保存在20%甘油/PBS中。 - 在干冰(15~30s)上冷冻,在RT处逐渐解冻,浸泡在20%的甘油/PBS中。重复此步骤三次。
- 在 PBS 中洗涤三次,每次 10 分钟。在 0.1 N HCl 中孵育 5 分钟,在 2x SSC 中冲洗。
- 在RT处孵育50%形式酰胺pH 7.0/2x SSC ON。
注:孵育时间可以优化并最终减少。幻灯片可保持50%的甲酰胺/2x SSC数天。 - 在 2x SSC 中平衡幻灯片(以 50% 形式酰胺/2x SSC) 进行 2 分钟。然后,在 PBS 中平衡 3 分钟。
- 治疗0.01 N HCl/0.0025%的pepsin从几秒钟到5分钟,取决于细胞类型。在此步骤中,在光学显微镜下观察细胞,并在核脱离细胞质后立即停止反应(步骤2.2.11),同时保持其结构完整(例如,核糖核苷酸仍然可见且完好无损)。
- 在 50 mM MgCl2/PBS 中,洗涤两次,每次 5 分钟,使胰蛋白蛋白失去活性。
- 在 PBS 中,在 1% PFA/PBS 中进行 1% 的后修复 1 分钟,洗涤 5 分钟。在 2x SSC 中每洗两次 5 分钟,然后加入 50% 的甲酰胺/2x SSC 至少 30 分钟。
- 在生长培养基(Dulbeco的改性鹰培养基(DMEM),20%胎儿牛血清(FBS),25纳克/mL纤维细胞生长因子(FGF),10纳克/mL表皮生长因子(EGF),10微克/mL人胰岛素,24孔板的盖玻片玻璃上直接生长人类原发性肌细胞。 1x谷氨酰胺,1x青霉素/链霉素),至少24小时(达到汇合的50-70%)。
3. 3D 多色DNA FISH 杂交
- 在80°C下使探头变性5分钟,然后迅速放到冰上。
- 将杂交探头加载到干净的显微镜幻灯片上。在杂交探针的掉落物上将带细胞倒置的盖玻片转动。
- 用橡胶水泥密封盖玻片。让橡胶水泥完全干燥。然后将幻灯片放在加热块上,在 75°C 下变性 4 分钟。
注:变性的时间和变性温度可以增强,高达80°C。 - 在漂浮在水浴中的金属盒中,在37°C开的金属箱中杂交。
注:为了提高信号噪声比,杂交温度可高达42°C。 - 剥去橡胶水泥,将滑片浸入 2x SCC 中,剥去玻璃盖玻片并将其转移到 6 孔板中的 2x SSC。
- 在 2x SSC 中洗涤三次,每次在 37°C 下 5 分钟,在孵化器摇床中以 90 rpm 的速度摇动。在 60°C 下,在 0.1x SSC 中洗涤 3 次,每次 5 分钟,在孵化器摇床中以 90 rpm 的速度摇动。在 4x SSC/0.2% TWEEN 20 中短暂冲洗。
4. 3D多色DNAFISH检测
注: 对于直接标记的探测器,请跳过步骤 4.1 和 4.2。
- 在 4x SSC/0.2% TWEEN 20/4% 牛血清白蛋白 (BSA) 中,在 24 孔板中 37°C 下块 20 分钟,在培养箱摇床中以 20 rpm 的速度摇动。
- 在37°C的黑暗和潮湿室中,用4xSSC/0.2%TWEEN 20/4%BSA稀释4xSSC/0.2%TWEEN 20/4%BSA,用适当浓度的抗二氧基星(1:150)和/或链球菌素(1:1,000)孵育。
- 在 4x SSC/0.2% TWEEN 20 中,在 37°C 下每次洗涤 5 分钟,在培养箱摇床中的 6 孔板中以 90 rpm 的速度摇动。
- 在 PBS 中平衡,在 24 孔板中,在 RT 中,在 2% 甲醛/PBS 中进行后固定 2 分钟。
注:直接标记的探头不需要固定后。 - 在 PBS 中短暂洗涤 5 次。1 纳克/mL DAPI (4,6-二酰胺-2-苯林多尔)/PBS 在 RT 上 5 分钟染色。
- 在 PBS 中短暂洗涤 5 次。安装防褪色溶液 (材料表)。
5. 3D 多色 DNA FISH 显微镜和分析
- 使用显微镜系统获取 3D 图像。
注:此处使用连续截面之间轴向距离为0.2±0.25 μm的宽场显微镜(图1B)。 - 使用不同的软件和工具(此处为 NuCL_D 或 3D 核触点定位器)分析 3D 图像堆栈。
注:开发工具NuCL_D是为了自动分析荧光细胞图像z堆栈中的3D多色DNAFISH。NuCL_D 在 3D 中从细胞图像堆栈中重建核,并检测和本地化荧光 3D 点。它测量原子核中斑点的相对定位(例如,与原子核和/或核的质心的距离)以及每个原子核和点间距离的最大半径。在 Cortesi 等人16中,对所使用的工具和算法进行了全面描述。 - 分析 NuCL_D 检索的数据(例如,指定基因组位点与核质心和接触频率之间的 3D 距离)。
注:对于指定基因组位点和核质心之间的3D距离,将每个原子核最大半径上的距离标准化,并将这些数据表示为与核质心的规范化距离的频率分布。对于远距离相互作用研究,接触频率应在10~20%21的范围内,阈值相互距离可能变化,可以放在2μm35左右。- 要执行统计分析,请分析每个生物复制大约 100 个核。将指定基因组位点之间的 3D 距离表示为低于所选阈值的阈值之间的距离的累积频率分布,并使用t检验来评估分布差异的重要性。此外,计算所选阈值值之间的相互作用的核阳性百分比,并使用 Fisher 的精确测试来评估百分比差异的重要性。
结果
本文中描述的三维多色DNA FISH方法允许当代可视化保存的3D核内的不同基因组位点(图1B)。该协议允许测量等位基因和不同基因组位点之间的距离,以评估其空间接近度,并评估其在核空间内的位置(例如,离心源或核外围的位点距离)16。但是,有许多关键步骤必须针对所使用的每种单元格类型进行准确和专门设置;强烈建议特别注意以下步骤,以成功 3D 多色 DNA FISH。
对于 DNA 探针制备,检查探针尺寸是否为 <200 bp (图 1A)。这种尺寸确保了3D多色DNA FISH的成功过程(图1B)。尼克翻译产生的次优DNAFISH探针可以部分消化(图2A)或过度消化(图2B)。在部分消化的探针中,由于探针无法进入核并适当杂交到互补基因组位点,该程序在细胞中将没有信号。过度消化的探头将导致非特异性信号,因为杂交中失去特异性,并因此增加背景。与图3B中的最佳消化探头相比,图3A显示了过度消化探头的代表性示例。
对于脱蛋白和消化,根据细胞类型遵循这些步骤。特别是,要考虑到核尺寸和细胞质丰度。对于人类原发前体分离的T淋巴细胞和细胞与小,高度压缩的核和低丰富的细胞质,HCl脱蛋白是至关重要的。使用 0.1 N HCl 进行 5 分钟的治疗不足以进行 DNA FISH 可视化。建议对N HCl进行12分钟的处理,以促进核对DNA探针的可及性,并维护核完整性(图4A)。细胞质的消化不需要采取良好的信号的DNA FISH (图4B).
对于具有大核和丰富细胞质的人类原发性异菌细胞和细胞,消化化步骤是根本的。细胞骨架的短小和次优的肽化会阻碍探针进入核(图4C),在缺乏DNAFISH信号的情况下结束。然而,如果细胞过度消化,核将不会保持完整(图4D),失去其3D结构。图4E提供了一个成功的3D多色DNAFISH的例子。
在杂交过程中,准确密封盖玻片;否则,探头将分散并干燥。必须迅速执行脱氧核糖核酸和杂交步骤,使探针和基因组DNA不会重新发合。可延长变性的持续时间。

图1:代表性DNA探针和3D多色DNAFISH。(A) 尼克翻译的最佳大小的DNA探针运行在2.2%的甘蔗凝胶(通道1,2),50 bp标记(M)。(B) 代表性3D多色DNAFISH核,使用探图绘制到人类原发性表细胞中的3q11.2区域(绿色)、10q26.3区域(红色)和8q24.13区域(洋红色)。核与 DAPI(蓝色)进行反染。63 倍放大倍率。比例尺 = 5 μm。请点击此处查看此图的较大版本。

图2:未优化消化的DNAFISH探针示例。(A) 未消化 (通道 1, 2) 或部分消化 (通道 3) 刻线翻译 DNA 探针运行在 2.2% agarose 凝胶, 2log 标记 (M).(B) 过度消化的nick翻译DNA探针运行在2.2%的糖凝胶,2log标记(M)。请点击此处查看此图的较大版本。
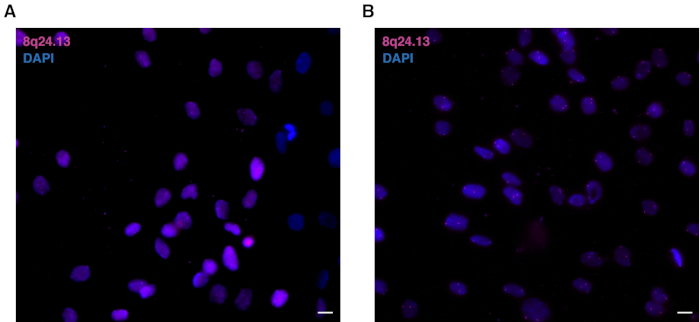
图3:使用次优或最优DNAFISH探针比较3D多色DNAFISH。(A) 代表性的3D多色DNAFISH核使用过度消化的探针映射到8q24.13区域(洋红色)在人类原发性表细胞。核与 DAPI(蓝色)进行反染。63 倍放大倍率。比例尺 = 10 μm. (B) 代表性 3D 多色 DNA FISH 核,使用最佳消化探针映射至人类原发性表细胞中的 8q24.13 区域 (洋红色)。核与 DAPI(蓝色)进行反染。63 倍放大倍率。比例尺 = 10 μm。请点击此处查看此图的较大版本。
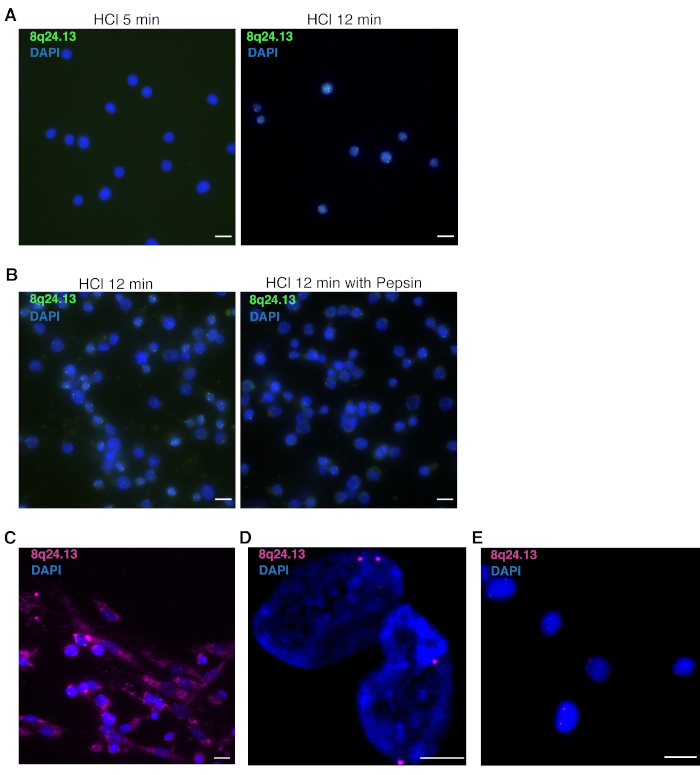
图4:3D多色DNAFISH结果下次优脱蛋白和消化化步骤的可能结果。(A) 代表3D多色DNAFISH核人类原发性T淋巴细胞处理5分钟(左)或12分钟(右)与0.1 N HCl,使用探针映射到8q24.13区域(绿色)。核与 DAPI(蓝色)进行反染。100 倍放大倍数。刻度条 = 10 μm . (B) 人类原发性 T 淋巴细胞的代表性 3D 多色 DNA FISH 核处理 12 分钟,使用 0.1 N HCl(左)或与 0.01 N HCl/0.0025% pepsin 结合 2 分钟(右),使用探针映射至 8q24.13 区域(绿色)。核与 DAPI(蓝色)进行反染。100 倍放大倍数。刻度条 = 10 μm. (C) 代表性 3D 多色 DNA FISH 核人类原发性表细胞的酶核处理短和次优的肽,使用探针映射到 8q24.13 区域 (洋红色)。核与 DAPI(蓝色)进行反染。63 倍放大倍率。刻度条 = 10 μm. (D) 代表性 3D 多色 DNA FISH 核人类原发性表细胞的酶细胞处理与长期消化化步骤,使用探针映射到 8q24.13 区域 (洋红色)。核与 DAPI(蓝色)进行反染。63 倍放大倍率。刻度条 = 5 μm . (E) 代表性 3D 多色 DNA FISH 核人类原发性肌细胞的处理与最佳 HCl/pepsin 条件使用探针映射到 8q24.13 区域 (洋红色).核与 DAPI(蓝色)进行反染。63 倍放大倍率。比例尺 = 25 μm.请点击此处查看此图的较大版本。
| 尼克翻译试剂 | 初始浓度 | 最终浓度 |
| dNTP (C-G-A) | 0.5 mM | 0.05 mM |
| dTTP | 0.1 mM | 0.01 mM |
| 生物锡/挖掘/Cy3 dUTP | 1 mM | 0.02 mM |
| Tris HCl pH 7.8 | 1米 | 50 mM |
| MgCl2 | 100 mM | 5 mM |
| β-梅卡托乙醇 | 100 mM | 10 mM |
| Bsa | 100 纳克/μL | 10 纳克/μL |
| DNA波尔I | 10 U/μL | 0.1 U/μL |
| DNase I | 1 U/μL | 0.002 U/μL |
| 脱氧核糖核酸 2 μg | Ⅹ | Ⅹ |
| ddH2O | 高达 50 μL |
表1:尼克翻译。表描述了所有的试剂,其浓度和建议的时间尼克翻译反应。
讨论
目前的方法描述了一个分步协议,以在广泛的人类原细胞上执行3D多色DNAFISH。虽然DNA FISH是一项广泛使用的技术,但保存下来的3D相间核上的3D多色DNAFISH在许多实验室中仍然难以执行,这主要是因为使用23、24的样品的特性。
探针刻翻译是成功3D多色DNAFISH的基本步骤;许多不同的基板(BAC、fosmid、质粒、PCR产物)可用于这种反应,反应和酶浓度的时序可根据基板的长度进行相应调整。适当的探头消化是根本(图1),因为非最优探头(图2)不会产生信号或非特异性信号(图3A)。渗透、脱蛋白和消化步骤是关键通道,它们强烈依赖于所使用的细胞类型。小核和低细胞骨架丰度的细胞,如外体分离的T淋巴细胞,需要脱蛋白,经过长期的0.1 N HCl治疗。此外,在PBS中,Triton X-100的百分比较高,可以帮助探针进入这些细胞的细胞核。相反,体外培养的人类原发性细胞细胞具有较大的核,细胞骨架含量高,需要消化带胰蛋白素的细胞结构。这些一般角色可以应用于广泛的细胞范围,最终根据特定的细胞特征组合不同的步骤。
强烈建议使用新鲜制备的生物材料、新鲜溶液(特别是带洗涤剂的溶液)和荧光试剂:在pH 7.0处过滤PFA;在pH 7.0时,对20x SSC进行高压和过滤;在pH 7.0处过滤的成型酰胺;无核酸酶水;和经过修改的 UTP 的一次性等分。使用 20% 甘油/PBS 或 50% 形式酰胺/2x SSC 进行长期孵育可促进杂交。HCl和/或胰蛋白蛋白治疗可进一步增加。杂交时间、探针量、浓度和培养时间均可进一步调整,以提高信噪比。
3D 多色 DNA FISH 是 C 技术的补充工具,是验证基于 C 的结果的标准方法。如果与3D显微镜和分析相结合,3D多色DNA FISH可以监测基因组位点及其在单细胞层核空间内的拓扑分布之间的接近。3D 多色 DNA FISH 可与其他方法(如 RNA FISH 和免疫荧光)进一步集成,全面了解基因组位点、RNA(信使RNA或调节性非编码RNA)和广泛范围之间的动力学和相互作用蛋白质,提供了一个独特的机会,可视化的核结构,并研究表观遗传机制,使细胞身份。
尽管FISH技术取得了巨大改进,具有超分辨率25,26,活细胞成像27,28,36,单分子检测37,和当代可视化多个目标与寡核苷酸阵列,如寡核苷酸阵列37,38与3D高通量方法39,技术的局限性仍然是预先确定的基因组位点的离散数量,可以可视化。这妨碍了对核结构的广泛分析。几项研究最近描述了连续的杂交方法,以解决单细胞的基因组组织问题,如条形码DNA FISH 40、41、42、43。还需要进一步努力,将3D多色DNAFISH的单细胞特性与基因组宽的特征结合,以用成像技术广泛可视化核结构异质性,因为一次可以测试的位点数量将会增加。
披露声明
作者没有什么可透露的。
致谢
作者感谢INGM成像设施(意大利米兰的"罗密欧·恩弗尼齐"(INGM)的技术援助,特别是C.Cordiglieri在3D多色DNAFISH图像中提供协助。收购。这项工作得到了以下对B.B.的赠款的支持:EPIGEN意大利旗舰计划、法国性心肌病协会(AFM-Telethon,第18754年赠款)和意大利卫生部的吉奥瓦尼·赖斯尔卡托里(GR-2011-02349383)。这项工作得到了以下对F.M.的资助:丰达齐奥内·卡里普洛(班多·乔瓦尼,赠款nr 2018-0321)。
材料
| Name | Company | Catalog Number | Comments |
| 24-well plates | Thermo Fisher Scientific | 142475 | |
| 6-well plates | Thermo Fisher Scientific | 140675 | |
| Anti-Digoxigenin 488 | DBA | DI7488 | |
| b-Mercaptoethanol | Sigma | M3148 | |
| bFGF | PeproTech | 100-18B | |
| Biotin 11 d-UTP | Thermo Fisher Scientific | R0081 | |
| BSA (bovine serum albumine) | Sigma | A7030 | |
| Coverlsips | Marienfeld | 117500 | |
| CY3 d-UTP | GE Healthcare | PA53022 | |
| DAPI (4,6-diamidino-2-phenylindole) | Thermo Fisher Scientific | D21490 | |
| Deoxyribonucleic acids single strand from salmon testes | Sigma | D7656 | |
| Dextran sulfate (powder) | Santa Cruz | sc-203917A | |
| Digoxigenin 11 d-UTP | Roche | 11093088910 | |
| DMEM | Thermo Fisher Scientific | 21969-035 500mL | |
| DNA polymerase I | Thermo Fisher Scientific | 18010-017 | |
| DNase I | Sigma | AMPD1 | |
| dNTPs (C-G-A-T) | Euroclone | BL0423A/C/G | |
| EGF | Sigma | E9644.2MG | |
| Ethanol | Sigma | 02860-1L | |
| FBS Hyclone | Thermo Fisher Scientific | SH30109 | |
| Formaldehyde solution | Sigma | F8775-25mL | |
| Formamide | Sigma | F9037 | |
| Glutammine | Thermo Fisher Scientific | 25030-024 100mL | |
| Glycerol | Sigma | G5516-100mL | |
| Glycogen | Thermo Fisher Scientific | AM9510 | |
| HCl | Sigma | 30721 | |
| Human Cot-1 DNA | Thermo Fisher Scientific | 15279-001 | |
| Insulin Human | Sigma | I9278-5 mL | |
| MgCl2 | Sigma | 63069 | |
| NaAc (Sodium Acetate, pH 5.2, 3 M) | Sigma | S2889 | |
| NaCl | Sigma | S9888 | |
| Paraformaldehyde | Sigma | 158127-25G | |
| PBS (phosphate-buffered saline) | Sigma | P4417 | |
| Pennycillin/Streptavidin | Thermo Fisher Scientific | 15070-063 100mL | |
| Pepsin | Biorad | P6887 | |
| PhasePrep BAC DNA Kit | Sigma | NA0100-1KT | |
| Poly-L-lysine solution | Sigma | P8920 | |
| ProLong Diamond Antifade Mountant | Thermo Fisher Scientific | P36970 | |
| PureLink Quick Gel Extraction & PCR Purification Combo Kit | Thermo Fisher Scientific | K220001 | |
| PureLink Quick Plasmid Miniprep Kit | Thermo Fisher Scientific | K210010 | |
| RNAse cocktail | Thermo Fisher Scientific | AM2288 | |
| Rubbercement | Bostik | ||
| Slides | VWR | 631-0114 | |
| Streptavidina Alexa fluor 647 | Thermo Fisher Scientific | S21374 | |
| Tri-Sodium Citrate | Sigma | 1110379026 | |
| Tris-HCl | Sigma | T3253-500g | |
| Triton X-100 | Sigma | T8787-250mL | |
| TWEEN 20 | Sigma | P9416-100mL |
参考文献
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nature Reviews: Genetics. 2 (4), 292-301 (2001).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Schmitt, A. D., Hu, M., Ren, B. Genome-wide mapping and analysis of chromosome architecture. Nature Reviews: Molecular Cell Biology. 17 (12), 743-755 (2016).
- Bonev, B., Cavalli, G. Organization and function of the 3D genome. Nature Reviews: Genetics. 17 (12), 772 (2016).
- van Steensel, B., Furlong, E. E. M. The role of transcription in shaping the spatial organization of the genome. Nature Reviews: Molecular Cell Biology. 20 (6), 327-337 (2019).
- Rajarajan, P., Gil, S. E., Brennand, K. J., Akbarian, S. Spatial genome organization and cognition. Nature Reviews: Neuroscience. 17 (11), 681-691 (2016).
- Pombo, A., Dillon, N. Three-dimensional genome architecture: players and mechanisms. Nature Reviews: Molecular Cell Biology. 16 (4), 245-257 (2015).
- Fanucchi, S., Shibayama, Y., Burd, S., Weinberg, M. S., Mhlanga, M. M. Chromosomal contact permits transcription between coregulated genes. Cell. 155 (3), 606-620 (2013).
- Therizols, P., et al. Chromatin decondensation is sufficient to alter nuclear organization in embryonic stem cells. Science. 346 (6214), 1238-1242 (2014).
- Gonzalez-Sandoval, A., et al. Perinuclear Anchoring of H3K9-Methylated Chromatin Stabilizes Induced Cell Fate in C. elegans Embryos. Cell. 163 (6), 1333-1347 (2015).
- Hubner, B., et al. Remodeling of nuclear landscapes during human myelopoietic cell differentiation maintains co-aligned active and inactive nuclear compartments. Epigenetics Chromatin. 8, 47 (2015).
- Aymard, F., et al. Genome-wide mapping of long-range contacts unveils clustering of DNA double-strand breaks at damaged active genes. Nature Structural & Molecular Biology. 24 (4), 353-361 (2017).
- Sellou, H., et al. The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage. Molecular Biology of the Cell. 27 (24), 3791-3799 (2016).
- Krijger, P. H., de Laat, W. Regulation of disease-associated gene expression in the 3D genome. Nature Reviews: Molecular Cell Biology. 17 (12), 771-782 (2016).
- Lupianez, D. G., Spielmann, M., Mundlos, S. Breaking TADs: How Alterations of Chromatin Domains Result in Disease. Trends in Genetics. 32 (4), 225-237 (2016).
- Cortesi, A., et al. 4q-D4Z4 chromatin architecture regulates the transcription of muscle atrophic genes in facioscapulohumeral muscular dystrophy. Genome Research. 29 (6), 883-895 (2019).
- Woltering, J. M., Noordermeer, D., Leleu, M., Duboule, D. Conservation and divergence of regulatory strategies at Hox Loci and the origin of tetrapod digits. PLoS Biology. 12 (1), 1001773 (2014).
- Woltering, J. M., Duboule, D. Tetrapod axial evolution and developmental constraints; Empirical underpinning by a mouse model. Mechanisms of Development. 138, 64-72 (2015).
- de Laat, W., Dekker, J. 3C-based technologies to study the shape of the genome. Methods. 58 (3), 189-191 (2012).
- Denker, A., de Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Finn, E. H., et al. Extensive Heterogeneity and Intrinsic Variation in Spatial Genome Organization. Cell. 176 (6), 1502-1515 (2019).
- Zheng, M., et al. Multiplex chromatin interactions with single-molecule precision. Nature. 566 (7745), 558-562 (2019).
- Solovei, I. Fluorescence in situ hybridization (FISH) on tissue cryosections. Methods in Molecular Biology. 659, 71-82 (2010).
- Cremer, M., et al. Multicolor 3D fluorescence in situ hybridization for imaging interphase chromosomes. Methods in Molecular Biology. 463, 205-239 (2008).
- Legant, W. R., et al. High-density three-dimensional localization microscopy across large volumes. Nature Methods. 13 (4), 359-365 (2016).
- Jungmann, R., et al. Quantitative super-resolution imaging with qPAINT. Nature Methods. 13 (5), 439-442 (2016).
- Chen, B., et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell. 155 (7), 1479-1491 (2013).
- Nelles, D. A., et al. Programmable RNA Tracking in Live Cells with CRISPR/Cas9. Cell. 165 (2), 488-496 (2016).
- Byron, M., Hall, L. L., Lawrence, J. B. A multifaceted FISH approach to study endogenous RNAs and DNAs in native nuclear and cell structures. Current Protocol of Human Genetics. , 15 (2013).
- Chaumeil, J., Augui, S., Chow, J. C., Heard, E. Combined immunofluorescence, RNA fluorescent in situ hybridization, and DNA fluorescent in situ hybridization to study chromatin changes, transcriptional activity, nuclear organization, and X-chromosome inactivation. Methods in Molecular Biology. 463, 297-308 (2008).
- Takizawa, T., Gudla, P. R., Guo, L., Lockett, S., Misteli, T. Allele-specific nuclear positioning of the monoallelically expressed astrocyte marker GFAP. Genes & Development. 22 (4), 489-498 (2008).
- Kraus, F., et al. Quantitative 3D structured illumination microscopy of nuclear structures. Nature Protocols. 12 (5), 1011-1028 (2017).
- Solovei, I., Cremer, M. 3D-FISH on cultured cells combined with immunostaining. Methods in Molecular Biology. 659, 117-126 (2010).
- Skinner, B. M., Johnson, E. E. Nuclear morphologies: their diversity and functional relevance. Chromosoma. 126 (2), 195-212 (2017).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Research. 25 (4), 582-597 (2015).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nature Methods. 9 (7), 743-748 (2012).
- Beliveau, B. J., et al. Single-molecule super-resolution imaging of chromosomes and in situ haplotype visualization using Oligopaint FISH probes. Nature Communication. 6, 7147 (2015).
- Beliveau, B. J., et al. Versatile design and synthesis platform for visualizing genomes with Oligopaint FISH probes. Proceedings of the National Academy of Sciences. 109 (52), 21301-21306 (2012).
- Samacoits, A., et al. A computational framework to study sub-cellular RNA localization. Nature Communication. 9 (1), 4584 (2018).
- Cardozo Gizzi, A. M., et al. Microscopy-Based Chromosome Conformation Capture Enables Simultaneous Visualization of Genome Organization and Transcription in Intact Organisms. Molecular Cell. 74 (1), 212-222 (2019).
- Mateo, L. J., et al. Visualizing DNA folding and RNA in embryos at single-cell resolution. Nature. 568 (7750), 49-54 (2019).
- Nir, G., et al. Walking along chromosomes with super-resolution imaging, contact maps, and integrative modeling. PLoS Genetics. 14 (12), 1007872 (2018).
- Bintu, B., et al. Super-resolution chromatin tracing reveals domains and cooperative interactions in single cells. Science. 362 (6413), (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。