Method Article
Herramienta 3D Multicolor DNA FISH para estudiar la arquitectura nuclear en células primarias humanas
En este artículo
Resumen
3D multicolor DNA FISH representa una herramienta para visualizar múltiples loci genómicos dentro de núcleos conservados 3D, definiendo inequívocamente sus interacciones recíprocas y localización dentro del espacio nuclear a un solo nivel de célula. Aquí, se describe un protocolo paso a paso para un amplio espectro de células primarias humanas.
Resumen
Una de las principales preguntas en la biología celular es la organización genómica dentro del espacio nuclear y cómo la arquitectura de la cromatina puede influir en procesos como la expresión génica, la identidad celular y la diferenciación. Muchos enfoques desarrollados para estudiar la arquitectura 3D del genoma se pueden dividir en dos categorías complementarias: tecnologías basadas en la captura de conformación cromosómica (tecnologías C) e imágenes. Mientras que el primero se basa en la captura de la conformación cromosómica y las interacciones proximales del ADN en una población de células fijas, la segunda, basada en la hibridación in situ por fluorescencia del ADN (FISH) en núcleos conservados en 3D, permite la visualización contemporánea de múltiples loci a un solo nivel de célula (multicolor), examinando sus interacciones y distribución dentro del núcleo (3D multicolor DNA FISH). La técnica del ADN multicolor 3D FISH tiene una limitación de visualizar sólo unos pocos loci predeterminados, no permitiendo un análisis exhaustivo de la arquitectura nuclear. Sin embargo, dada la solidez de sus resultados, 3D multicolor DNA FISH en combinación con la microscopía 3D y la reconstrucción de imágenes es un método posible para validar los resultados basados en la tecnología C y para estudiar inequívocamente la posición y organización de loci específicos a un solo nivel de célula. Aquí, proponemos un método paso a paso de 3D multicolor DNA FISH adecuado para una amplia gama de células primarias humanas y discutimos todas las acciones prácticas, pasos cruciales, nociones de imagen 3D y análisis de datos necesarios para obtener un multicolor 3D exitoso e informativo DNA FISH dentro de diferentes contextos biológicos.
Introducción
Los eucariotas más altos necesitan condensar y compactar sistemáticamente una enorme cantidad de información genética en el espacio 3D minuto del núcleo1,2,3,4. Hoy en día, sabemos que el genoma está ordenado espacialmente en compartimentos y dominios topológicamente asociados5 y que los múltiples niveles de plegado de ADN generan contactos entre diferentes regiones genómicas que pueden implicar la formación de bucles de cromatina6,7. El bucle dinámico 3D de la cromatina puede influir en muchos procesos biológicos diferentes como la transcripción8,9,la diferenciación y desarrollo10,11, la reparación del ADN12,13, mientras que sus perturbaciones están implicadas en diversas enfermedades14,15,16 y defectos de desarrollo15,17,18.
Se han desarrollado muchos enfoques para estudiar la organización del genoma 3D. Se han desarrollado tecnologías basadas en la captura de conformación cromosómica (tecnologías C, 3C, 4C, 5C, Hi-C y derivados) para estudiar la organización del genoma en células fijas3,4,19,20. Estos enfoques se basan en la capacidad de capturar las frecuencias de contacto entre loci genómicos en proximidad física. Las tecnologías C, dependiendo de su complejidad, capturan la organización mundial del genoma 3D y la topología nuclear de una población celular3,4,19,20. Sin embargo, las interacciones 3D son dinámicas en el tiempo y el espacio, altamente variables entre las células individuales que consisten en interacciones multiplex, y son ampliamente heterogéneas21,22.
La hibridación in situ de la fluorescencia del ADN multicolor 3D (FISH) es una técnica que permite la visualización de loci genólicos específicos a un nivel de célula única, permitiendo la investigación directa de la arquitectura nuclear 3D de manera complementaria a las tecnologías C. Representa una tecnología utilizada actualmente para validar inequívocamente los resultados C. 3D multicolor DNA FISH utiliza sondas etiquetadas fluorescentmente complementarias a los loci genómicos de los intereses. El uso de diferentes fluoróforos y equipos de microscopía adecuados permiten la visualización contemporánea de múltiples objetivos dentro del espacio nuclear23,24. En los últimos años, FISH se ha combinado con los avances tecnológicos en microscopía para obtener la visualización de estructuras de escala fina a alta resolución25,26 o con enfoques CRISPR-Cas para la visualización de los ácidos nucleicos en imágenes en vivo27,28. A pesar de la amplia adopción, el enfoque 3D multicolor DNA FISH todavía se considera difícil en muchos laboratorios porque el material biológico utilizado debe adaptarse.
Aquí, proporcionamos un protocolo integral para el ADN 3D multicolor FISH (desde la preparación de células/sondas hasta el análisis de datos) aplicable a una amplia gama de células primarias humanas, permitiendo la visualización de múltiples loci genómicos y preservando la estructura 3D de los núcleos. Para estudiar la arquitectura nuclear, se debe preservar la estructura 3D de los núcleos. Por esta razón, contrastando con otros protocolos existentes29,30,31,evitamos el uso de un gradiente alcohólico y el almacenamiento de los cubreobjetos en alcohol que puede afectar a la estructura de la cromatina32. El método se adapta a partir de los protocolos FISH de ADN 3D conservados24,33 para aplicarse a una amplia gama de células primarias humanas, tanto aisladas ex vivo o cultivadas in vitro. Existen parámetros de permeabilización y desproteinización para diferentes morfologías nucleares y características citológicas (por ejemplo, diferentes grados de compactación nuclear, abundancia de citoesqueleto)34. Estos parámetros se describen generalmente en otros protocolos24,33, sin proporcionar una clara discriminación del procedimiento dentro de diferentes tipos de células. Además, desarrollamos una herramienta específica llamada NuCL-D (localizador de contactos nucleares en 3D)16,proporcionando principios para el análisis de datos que mejorarán la proximidad 3D entre diferentes loci y su distribución topológica nuclear dentro del espacio nuclear de forma automatizada.
Protocolo
1. Procedimientos de preparación y etiquetado de la sonda de ADN con traducción de nick
- Purificar y limpiar cromosomas artificiales bacterianos (BAC), plásmidos o productos PCR con kits específicos(Tabla de materiales),resuspender en ddH2O, comprobar por electroforesis en un gel de agarosa y cuantificar.
- Realice la traducción de nicks en 1,5 x 2 g de ADN a partir del paso 1.1 en un volumen final de 50 l mezclando todos los reactivos en un tubo de ADN de unión baja de 0,5 ml de acuerdo con la Tabla 1.
NOTA: Las sondas etiquetadas directamente pueden mejorar la relación señal-ruido. Los kits para producir sondas etiquetadas indirectamente y directamente con varios fluorocromos Alexa por traducción de nick están disponibles comercialmente. - Incubar la mezcla de traducción de nick en un mezclador térmico a 16oC durante un tiempo dependiendo de la longitud del material de ADN inicial: 45 min para productos PCR (un grupo de productos PCR de 2.000 bp cada uno) y hasta 4 h para ADN BAC y plásmidos.
- Compruebe el tamaño de las sondas producidas en el paso 1.3 por electroforesis en un gel de agarosa del 2,2%.
NOTA: El tamaño óptimo de la sonda es <200 bp(Figura 1A).- Si el ADN no se digiere lo suficiente, añadir 5 U de ADN polimerasa I y 0,05 U de DNase I a la reacción e incubar durante 1 x 2 h a 16 oC y posteriormente volver a comprobar. Detener la reacción con 0,5 mM EDTA (concentración final). Almacene las sondas a -20 oC.
- Para cada experimento DNA FISH, precipita las siguientes cantidades de sondas dependiendo del material de ADN inicial a partir del cual se producen las sondas: 200 ng de productos de PCR traducidos por Nick, 100 ng de BACtraducidos nick, o 300 ng de plásmido traducido nick. Añadir ddH2O hasta 150 ol, 20 g de ADN de espermatozoides de salmón sin etiquetar, 3,5 g de ADN de cot-1 específico de la especie, 3 volúmenes de 100% EtOH y 1/10 volumen de acetato de sodio de 3 M pH 5.2. Precipitar a -80oC durante 1 h.
- Centrifugar a una velocidad máxima de 1 h a 4oC y desechar el sobrenadante. Lave el pellet dos veces con 70% EtOH.
- Resuspender el pellet en 2 sL de 100% de formamida pH 7.0, agitar a 40 oC durante 30 minutos (puede tomar hasta unas horas) y luego añadir un volumen igual de 4 x citrato de sodio salino (SSC) / 20% sulfato de dextran.
- Preparar 20x SSC mezclando 175,3 g de NaCl y 88,2 g de citrato de sodio en un volumen final de 1 L de H2O. Autoclave, filtrar y preparar alícuotas.
NOTA: Para el ADN multicolor FISH, las diferentes sondas se pueden precipitar juntas excepto para sondas que pueden recocido entre sí. En estos casos específicos, tratarlos por separado, precipitar y resuspender las sondas que dividen los reactivos mencionados en los pasos 1.5-1.7 con respecto al número de sondas. Agrupa las sondas sólo después de la resuspensión en formamida. Si se utilizan revestimientos de vidrio superiores a 10 mm o menos de 10 mm, escale/reduzca verticalmente el volumen de los reactivos utilizados en los pasos 1,5 a 1,7 proporcionalmente al tamaño de la diapositiva. Las sondas de hibridación se pueden almacenar a -20 oC durante un largo período de tiempo (hasta dos meses).
- Preparar 20x SSC mezclando 175,3 g de NaCl y 88,2 g de citrato de sodio en un volumen final de 1 L de H2O. Autoclave, filtrar y preparar alícuotas.
2. Fijación celular, pretratamiento y permeabilización
NOTA: Los pasajes de permeabilización y desproteinización son pasos cruciales. El tiempo de reacción y concentración de los reactivos depende en gran su nombre del tipo de célula, la abundancia del citoplasma y la morfología nuclear.
- Fijación celular, pretratamiento y permeabilización para linfocitos T primarios humanos
NOTA: Este protocolo es adecuado para células pequeñas, con núcleos pequeños y una baja cantidad de citoplasma.- Utilice tapas de vidrio (10 mm, espesor No. 1.5H).
- Lavar el vaso con ddH2O, luego con 70% EtOH y dejar que se sequen. Utilice un vaso para cada pocal de un plato de 24 pocillos.
- Añadir 200 s de poli-L-lisina al 0,1% (p/v)(Tabla de materiales)directamente en el vidrio para formar una gota. Preste atención ya que la gota debe permanecer en la superficie de vidrio sin tocar el pozo durante 2 x 5 minutos. Deje que el vidrio se seque durante 30 minutos.
- Realice el paso 2.1.3 dos veces más.
- Poner 200 l de células de suspensión (2 x 106/ml, suspensión de PBS de linfocitos T primarios ex vivo) directamente en el vidrio, permitir que las células siembren a temperatura ambiente (RT) durante 30 min.
- Retire rápidamente la gota. Añadir PFA 4% recién hecho (preparado en PBS/0.1% TWEEN 20, pH 7.0, filtrado) durante 10 min y fijar las células sembradas en RT.
- Lavar tres veces durante 5 min cada una con 0.05% Triton X-100/PBS (TPBS) en RT. Permeabilize con 0.5% TPBS para 10 min en RT.
- Realizar el tratamiento de RNase añadiendo 2,5 ml de cóctel de RNase(Tabla de materiales)en 250 ml de PBS/bien en una placa de 24 pocillos durante 1 h a 37 oC.
- Enjuagar en PBS, añadir un 20% de glicerol/PBS e incubar durante la noche (ON) a 4oC.
NOTA: Este paso puede variar de 1 h a ON. Las diapositivas se pueden mantener en un 20% de glicerol/PBS a 4 oC hasta 7 días. - Congele en hielo seco (15-30 s), descongele gradualmente a RT y empape en 20% de glicerol/PBS. Repita el paso 2.1.9 otras tres veces.
- Lavar en 0,5% TPBS durante 5 min a RT. Lavar en 0,05% TPBS, dos veces durante 5 min a RT. Incubar en 0,1 N HCl durante 12 min en RT. Enjuagar en 2x SSC.
- Incubar en 50% de formamida pH 7.0/2x SSC ON en RT.
NOTA: El tiempo de incubación se puede optimizar y eventualmente reducir. Las diapositivas se pueden mantener en un 50% de formamida/2x SSC durante varios días.
- Fijación celular, pretratamiento y permeabilización para mioblastos primarios humanos
NOTA: Este protocolo es adecuado para células grandes, con una gran cantidad de citoplasma.- Cultivar mioblastos primarios humanos directamente en las gafas de encubrimiento en placas de 24 pocillos en medio de crecimiento (medio águila modificado de Dulbecco (DMEM), suero bovino fetal del 20% (FBS), factor de crecimiento de fibroblastos de 25 ng/ml (FGF), factor de crecimiento epidérmico de 10 ng/ml (EGF), insulina humana de 10 g/ml, 1x glutamina, 1x penicilina/estreptomicina) durante al menos 24 h (alcanzando 50-70% de confluencia).
NOTA: Para facilitar la adhesión, la gelatina o el colágeno o la poli-L-lisina se pueden utilizar para recubrir el cubreobjetos antes de la sembrada. - Enjuague las células en 2 x 3 cambios de PBS. Agregue el 4% de PFA recién hecho y fije las células durante 10 minutos en RT.
- Lavar tres veces durante 3 min cada una en 0.01% TPBS en RT. Permeabilizar en 0.5% TPBS para 10 min en RT.
- Realizar el tratamiento de RNase añadiendo 2,5 ml de cóctel de RNase(Tabla de materiales)en 250 ml de PBS/bien en una placa de 24 pocillos durante 1 h a 37 oC.
- Enjuague en PBS, agregue 20% de glicerol/PBS e incubar ON en RT.
NOTA: Este paso puede variar de 1 h a ON. Las diapositivas se pueden mantener en un 20% de glicerol/PBS a 4 oC hasta 7 días. - Congele en hielo seco (15-30 s), descongele gradualmente a RT y empape en 20% de glicerol/PBS. Repita este paso tres veces.
- Lavar tres veces durante 10 minutos cada uno en PBS. Incubar en 0,1 N HCl durante 5 min a RT. Enjuague en 2x SSC.
- Incubar en 50% de formamida pH 7.0/2x SSC ON en RT.
NOTA: El tiempo de incubación se puede optimizar y eventualmente reducir. Las diapositivas se pueden mantener en un 50% de formamida/2x SSC durante varios días. - Diapositivas de equilibrio (mantenidas en 50% de formamida/2x SSC) en 2x SSC durante 2 min. A continuación, equilibrar en PBS durante 3 min.
- Tratar con 0.01 N HCl/0.0025% pepsina desde unos segundos hasta 5 min, dependiendo del tipo de celda. Durante este paso, observe las células bajo un microscopio óptico y detenga la reacción (paso 2.2.11) tan pronto como los núcleos estén libres del citoplasma, manteniendo su estructura intacta (por ejemplo, los nucleolisiguen visibles e intactos).
- Inactivar la pepsina lavando dos veces durante 5 min cada uno en 50 mM MgCl2/PBS.
- Post-fijo en 1% PFA/PBS durante 1 min. Lavar durante 5 min en PBS. Lavar dos veces durante 5 min cada uno en 2x SSC, y luego añadir 50% de formamida/2x SSC durante al menos 30 min.
- Cultivar mioblastos primarios humanos directamente en las gafas de encubrimiento en placas de 24 pocillos en medio de crecimiento (medio águila modificado de Dulbecco (DMEM), suero bovino fetal del 20% (FBS), factor de crecimiento de fibroblastos de 25 ng/ml (FGF), factor de crecimiento epidérmico de 10 ng/ml (EGF), insulina humana de 10 g/ml, 1x glutamina, 1x penicilina/estreptomicina) durante al menos 24 h (alcanzando 50-70% de confluencia).
3. Hibridación 3D multicolor DNA FISH
- Desnaturalizar las sondas a 80 oC durante 5 minutos, y luego poner rápidamente en hielo.
- Cargue las sondas de hibridación en una diapositiva limpia del microscopio. Gire el cubreobjetos con las células al revés en la gota de las sondas de hibridación.
- Selle la cubierta con cemento de goma. Deje que el cemento de goma se seque por completo. A continuación, coloque los portaobjetos en un bloque de calefacción y desnaturaliza a 75 oC durante 4 min.
NOTA: Se puede aumentar el tiempo de desnaturalización y la temperatura de la desnaturalización, hasta 80 oC. - Hibrida a 37oC ON en una caja metálica flotando en un baño de agua.
NOTA: Para mejorar la relación señal-ruido, la temperatura de hibridación puede llegar hasta 42 oC. - Pelar el cemento de goma, sumergir los portaobjetos en 2x SCC, quitar el cubreobjetos de vidrio y transferirlo a 2x SSC en placa de 6 pocillos.
- Lavar en 2x SSC tres veces durante 5 min cada uno a 37oC, agitando a 90 rpm en una coctelera de incubadora. Lavar en 0,1x SSC tres veces durante 5 min cada una a 60 oC, agitando a 90 rpm en una coctelera de incubadora. Enjuague brevemente en 4x SSC/0.2% TWEEN 20.
4. Detección de FISH de ADN multicolor 3D
NOTA: Para sondas etiquetadas directamente, omita los pasos 4.1 y 4.2.
- Bloquear en 4x SSC/0.2% TWEEN 20/4% albúmina sérica bovina (BSA) durante 20 min a 37oC en una placa de 24 pocillos, agitando a 20 rpm en una coctelera de incubadora.
- Incubar con la concentración adecuada de antidigoxigenina (1:150) y/o estreptavidina (1:1.000)(Tabla de materiales)diluida en 4x SSC/0.2% TWEEN 20/4% BSA durante 35 min en una cámara oscura y húmeda a 37oC.
- Lavar en 4x SSC/0.2% TWEEN 20 tres veces durante 5 min cada uno a 37oC, agitando a 90 rpm en una placa de 6 pocillos en una coctelera de incubadora.
- Equilibrar en PBS y post-fijar en 2% formaldehído/PBS durante 2 min a RT en una placa de 24 pocillos.
NOTA: Las sondas etiquetadas directamente no necesitan post-fijación. - Lavar 5 veces brevemente en PBS. Mancha con 1 ng/mL DAPI (4,6-diamidino-2-fenilindole)/PBS durante 5 min en RT.
- Lavar 5 veces brevemente en PBS. Montaje con solución antidescolora(Tabla de materiales).
5. Microscopía y análisis 3D multicolor DNA FISH
- Adquiera imágenes 3D con un sistema de microscopio.
NOTA: Aquí se utiliza un microscopio de campo ancho con una distancia axial de 0,2 x 0,25 m entre secciones consecutivas(Figura 1B). - Analice las pilas de imágenes 3D utilizando diferentes software y herramientas (aquí, NuCL-D o Localizador de Contactos Nucleares en 3D).
NOTA: La herramienta NuCL-D se ha desarrollado con el fin de analizar automáticamente 3D ADN multicolor FISH en pilas z de imagen de célula de fluorescencia. NuCL-D reconstruye los núcleos a partir de pilas de imágenes celulares en 3D, así como detecta y localiza puntos 3D fluorescentes. Mide el posicionamiento relativo de las manchas en el núcleo (por ejemplo, la distancia desde el centroide de los núcleos y/o la periferia de los núcleos) y el radio máximo para cada núcleo y las distancias entre puntos. La herramienta y el algoritmo utilizado están completamente descritos en Cortesi et al.16. - Analizar los datos (por ejemplo, distancias 3D entre los loci genómicos especificados y el centroide nuclear y las frecuencias de contacto) recuperados por NuCL-D.
NOTA: Para las distancias 3D entre los loci genómicos especificados y el centroide nuclear, normalice las distancias en el radio máximo para cada núcleo y represente estos datos como distribuciones de frecuencia de distancias normalizadas desde el centroide nuclear. Para los estudios de interacciones de largo alcance, se supone que las frecuencias de contacto están en el rango de 10 a 20%21 con un umbral de interdistancia que puede variar y se puede poner alrededor de 2 m35.- Para realizar análisis estadísticos, analice aproximadamente 100 núcleos por réplica biológica. Representar distancias 3D entre loci genómicos especificados como distribuciones de frecuencia acumulativa de distancias que están por debajo del umbral entre distancias seleccionado y utilizar una prueba tpara evaluar la importancia de las diferencias en las distribuciones. Además, calcule el porcentaje de núcleos positivos para las interacciones que están por debajo del umbral de interdistancia seleccionado, y utilice la prueba exacta de Fisher para evaluar la importancia de las diferencias en los porcentajes.
Resultados
El método de ADN multicolor 3D FISH descrito en este artículo permite la visualización contemporánea de diferentes loci genómicos dentro de los núcleos 3D conservados(Figura 1B). Este protocolo permite medir las distancias entre los alelos y los diferentes loci genómicos para evaluar su proximidad espacial, y evaluar su ubicación dentro del espacio nuclear (por ejemplo, la distancia de loci desde el centroide o la periferia de los núcleos)16. Sin embargo, hay muchos pasos cruciales que deben configurarse de forma precisa y específica para cada tipo de celda utilizado; es muy recomendable prestar especial atención a los siguientes pasos para el éxito del ADN 3D multicolor FISH.
Para la preparación de la sonda de ADN, compruebe que el tamaño de la sonda es <200 bp(Figura 1A). Este tamaño asegura un procedimiento exitoso de ADN 3D multicolor FISH(Figura 1B). Las sondas FISH de ADN subóptimas producidas por la traducción de nick pueden ser digeridas parcialmente(Figura 2A)o en exceso(Figura 2B). Con sondas parcialmente digeridas, el procedimiento no tendrá señal en las células, debido a la incapacidad de la sonda para entrar en los núcleos e hibridar adecuadamente a los loci genómicos complementarios. Las sondas excesivamente digeridas darán lugar a una señal inespecífica, debido a una pérdida de especificidad en la hibridación y un consecuente aumento del fondo. En la Figura 3A se muestra un ejemplo representativo de sondas sobre digeridas en comparación con una sonda digerida óptima en la Figura 3B.
Para la desproteinización y la pepsinización, siga estos pasos según el tipo de célula. En particular, tenga en cuenta el tamaño nuclear y la abundancia de citoplasma. Para los linfocitos T aislados ex vivo primarios humanos y células con núcleos pequeños y altamente compactados y citoplasma de bajo abundante abundante, la desproteinización de HCl es crucial. El tratamiento con 0,1 N HCl durante 5 min no es suficiente para la visualización de DNA FISH. 0.1 Se recomienda el tratamiento con HCl durante 12 minutos para promover la accesibilidad de los núcleos a las sondas de ADN y preservar la integridad nuclear(Figura 4A). No es necesaria la digestión pepsina del citoplasma para obtener una buena señal de DNA FISH(Figura 4B).
Para los mioblastos primarios humanos y las células que tienen núcleos grandes y citoplasma abundante, el paso de pepsinización es fundamental. Una pepsinización corta y subóptima del citoesqueleto obstaculizará la entrada de la sonda en los núcleos(Figura 4C),terminando en ausencia de una señal DNA FISH. Sin embargo, si las células están demasiado pepsinizadas, los núcleos no permanecerán intactos(Figura 4D),perdiendo su estructura 3D. En la Figura 4Ese proporciona un ejemplo de ADN multicolor 3D exitoso FISH.
Durante la hibridación, selle el cubreobjetos con precisión; de lo contrario, la sonda se dispersará y se secará. Las etapas de desnaturalización e hibridación deben realizarse rápidamente de modo que la sonda y el ADN genómico no vuelvan a ser recocidos. La duración de la desnaturalización se puede aumentar.

Figura 1: Sondas de ADN representativas y FISH de ADN multicolor 3D. (A) Nick tradujo sondas de ADN de tamaño óptimo en un gel de agarosa del 2,2% (carril 1, 2), marcador de 50 bp (M). (B) Representativo núcleo de ADN multicolor FISH 3D utilizando sondas mapeadas a 3q11.2 región (verde), 10q26.3 región (rojo) y 8q24.13 región (magenta) en mioblastos primarios humanos. Los núcleos están contrarrestados con DAPI (azul). Aumento de 63x. Barra de escala de 5 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Ejemplos de sondas FISH de ADN no digeridas de forma óptima. (A) No digeridas (carril 1, 2) o parcialmente digeridas (carril 3) sondas de ADN traducidas con nick funcionan con un gel de agarosa del 2,2%, marcador de 2log (M). (B) Sobre nick digerido sondas de ADN traducidas funcionan con un 2.2% gel de agarosa, marcador de 2log (M). Haga clic aquí para ver una versión más grande de esta figura.
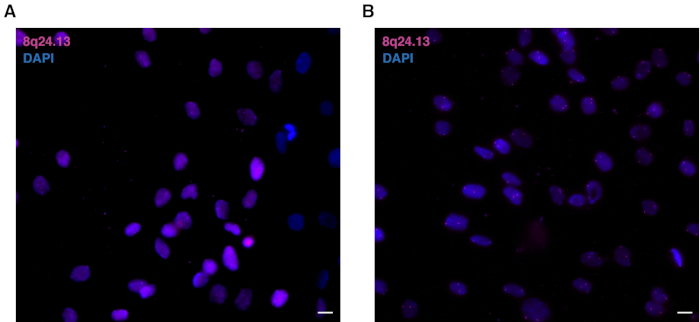
Figura 3: Comparación de ADN 3D multicolor FISH utilizando sondas FISH de ADN subóptimas u óptimas. (A) Núcleos de FISH de ADN multicolor 3D representativos utilizando mapeo de sonda sobre digerido a la región 8q24.13 (magenta) en mioblastos primarios humanos. Los núcleos están contrarrestados con DAPI (azul). Aumento de 63x. Barra de escala a 10 m.(B) Núcleos de FISH de ADN multicolor 3D representativos utilizando mapeo de sonda digerido de forma óptima a la región 8q24.13 (magenta) en mioblastos primarios humanos. Los núcleos están contrarrestados con DAPI (azul). Aumento de 63x. Barra de escala a 10 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.
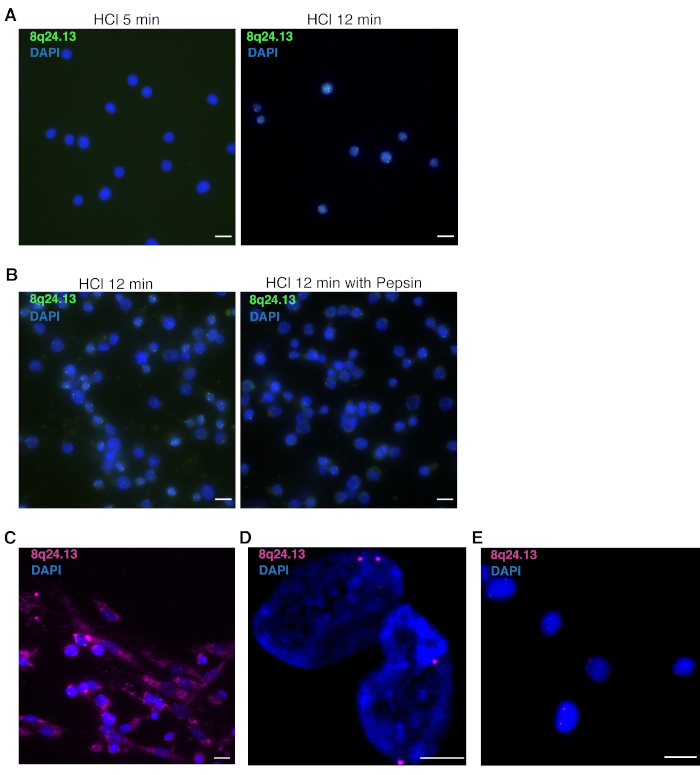
Figura 4: Posibles resultados de pasos de desproteinización y pepsinización subóptimos en los resultados 3D multicolor DNA FISH. (A) Núcleos de FISH de ADN multicolor 3D representativo de linfocitos T primarios humanos tratados durante 5 min (izquierda) o 12 min (derecha) con 0,1 N HCl, utilizando mapeo de sonda a la región 8q24.13 (verde). Los núcleos están contrarrestados con DAPI (azul). Aumento de 100x. Barra de escala a 10 m.(B) Núcleos de FISH de ADN multicolor 3D representativos de linfocitos T primarios humanos tratados durante 12 min con 0,1 N HCl (izquierda) o acoplados con 0,01 N HCl/0.0025% pepsina durante 2 min (derecha), utilizando mapeo de sonda a la región 8q24.13 (verde). Los núcleos están contrarrestados con DAPI (azul). Aumento de 100x. Barra de escala a 10 m.(C) Núcleos fish de ADN multicolor 3D representativos de mioblastos primarios humanos tratados con pepsinización corta y subóptima, utilizando la asignación de sondas a la región 8q24.13 (magenta). Los núcleos están contrarrestados con DAPI (azul). Aumento de 63x. Barra de escala a 10 m.(D) Núcleos de FISH de ADN multicolor 3D representativos de mioblastos primarios humanos tratados con paso de pepsinización prolongada, utilizando la asignación de sondas a la región 8q24.13 (magenta). Los núcleos están contrarrestados con DAPI (azul). Aumento de 63x. Barra de escala a 5 m.(E) núcleos FISH de ADN multicolor 3D representativos de mioblastos primarios humanos tratados con condiciones óptimas de HCl/pepsin utilizando la asignación de sondas a la región 8q24.13 (magenta). Los núcleos están contrarrestados con DAPI (azul). Aumento de 63x. Barra de escala de 25 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.
| Reactivos de traducción de Nick | Concentración inicial | Concentración final |
| dNWP (C-G-A) | 0,5 mM | 0,05 mM |
| dTTP | 0,1 mM | 0,01 mM |
| Biotina/Dig/Cy3 dUTP | 1 mM | 0,02 mM |
| Tris HCl pH 7.8 | 1 M | 50 mM |
| MgCl2 | 100 mM | 5 mM |
| -mercaptoetanol | 100 mM | 10 mM |
| Bsa | 100 ng/L | 10 ng/L |
| DNA Pol I | 10 U/L | 0,1 U/L |
| DNase I | 1 U/L | 0.002 U/L |
| ADN 2 g | X | X |
| ddH2O | Hasta 50 sL |
Tabla 1: Traducción de Nick. Tabla que describe todos los reactivos, su concentración y el momento sugerido para la reacción de traducción de nick.
Discusión
El método actual describe un protocolo paso a paso para realizar ADN FISH multicolor 3D en una amplia gama de células primarias humanas. Aunque DNA FISH es una tecnología de uso amplio, el ADN 3D multicolor FISH en núcleos interfásicos 3D conservados sigue siendo difícil de realizar en muchos laboratorios, principalmente debido a las características de las muestras utilizadas23,24.
La traducción de nick de sonda es un paso fundamental para el éxito 3D multicolor DNA FISH; muchos sustratos diferentes (BAC, fosmid, plásmido, productos PCR) se pueden utilizar para esta reacción, y el momento de la reacción y la concentración enzimática se puede ajustar en consecuencia con respecto a la longitud del sustrato. Una digestión adecuada de la sonda es fundamental(Figura 1),ya que las sondas no óptimas(Figura 2) no darán lugar a ninguna señal o señal inespecífica(Figura 3A). Los pasos de permeabilización, desproteinización y pepsinización son pasajes cruciales que dependen fuertemente del tipo de célula utilizado. Las células con núcleos pequeños y abundancia baja de citoesqueleto, como linfocitos T aislados ex vivo, requieren desproteinización con un tratamiento prolongado de 0,1 N HCl. Además, los lavados en PBS con porcentajes más altos de Triton X-100 pueden ayudar a la entrada de la sonda en los núcleos de estas células. Por el contrario, los mioblastos primarios humanos cultivados in vitro que presentan núcleos más grandes, con un alto contenido de citoesqueleto, necesitan digestión de las estructuras citosólicas con pepsina. Estos roles generales se pueden aplicar a una amplia gama de células, combinando finalmente los diferentes pasos dependiendo de las características celulares específicas.
Se recomienda encarecidamente el uso de material biológico recién preparado, soluciones frescas (en particular soluciones con detergente) y reactivos fluorescentes: PFA filtrado a pH 7.0; autoclave y filtrado 20x SSC a pH 7.0; formación filtrada a pH 7.0; agua libre de nucleasas; y alícuotas desechables de UTP modificado. La incubación prolongada con 20% de glicerol/PBS, o el 50% de formamida/2x SSC puede facilitar la hibridación. El tratamiento con HCl y/o pepsina puede aumentar aún más. El tiempo de hibridación, la cantidad de sondas, la concentración y el momento de incubación de antidigoxigenina y estreptavidina se pueden ajustar aún más para mejorar la relación señal-ruido.
3D multicolor DNA FISH representa una herramienta complementaria a las tecnologías C, el método estándar para validar los resultados basados en C. Si se combina con la microscopía 3D y el análisis, 3D multicolor DNA FISH puede monitorear la proximidad entre los loci genómicos y su distribución topológica dentro del espacio nuclear a nivel de una sola célula. 3D multicolor DNA FISH se puede integrar aún más con otras metodologías como el ARN FISH y la inmunofluorescencia para una visión general completa de la dinámica y las interacciones entre loci genómico, ARN (ARN mensajero o ARN reglamentario no codificante) y una amplia gama de proteínas, proporcionando una oportunidad única para visualizar la estructura nuclear e investigar los mecanismos epigenéticos que subtienden la identidad celular.
A pesar de la enorme mejora de las tecnologías FISH con super resolución25,26, imágenes de células vivas27,28,36, detección de molécula única37, y la visualización contemporánea de múltiples dianas con matrices de oligonucleótidos como Oligopaint37,38 con enfoques 3D de alto rendimiento39, una limitación de la tecnología sigue siendo el número discreto de loci genómicos predeterminados que pueden visualizar. Esto evita un amplio análisis de la arquitectura nuclear. Varios estudios han descrito recientemente métodos secuenciales de hibridación para abordar la organización del genoma en células individuales como el código de barras DNA FISH40,41,42,43. Se necesitarán más esfuerzos para combinar la naturaleza de una sola célula del ADN multicolor 3D FISH a las características amplias del genoma para visualizar ampliamente la heterogeneidad de la arquitectura nuclear con las tecnologías de imagen, ya que el número de loci que se pueden probar a la vez aumentará.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores reconocen la asistencia técnica del INGM Imaging Facility (Istituto Nazionale di Genetica Molecolare "Romeo ed Enrica Invernizzi" (INGM), Milán, Italia), en particular C. Cordiglieri, para la asistencia durante las imágenes 3D multicolor DNA FISH Adquisición. Este trabajo ha sido apoyado por las siguientes subvenciones a B.B.: el programa insignia italiano EPIGEN, la Asociación Francesa contre les Myopathies (AFM-Telethon, grant nr 18754) y Giovani Ricercatori, Ministerio de Salud de Italia (GR-2011-02349383). Este trabajo ha sido apoyado por la siguiente subvención a F.M.: Fondazione Cariplo (Bando Giovani, grant nr 2018-0321).
Materiales
| Name | Company | Catalog Number | Comments |
| 24-well plates | Thermo Fisher Scientific | 142475 | |
| 6-well plates | Thermo Fisher Scientific | 140675 | |
| Anti-Digoxigenin 488 | DBA | DI7488 | |
| b-Mercaptoethanol | Sigma | M3148 | |
| bFGF | PeproTech | 100-18B | |
| Biotin 11 d-UTP | Thermo Fisher Scientific | R0081 | |
| BSA (bovine serum albumine) | Sigma | A7030 | |
| Coverlsips | Marienfeld | 117500 | |
| CY3 d-UTP | GE Healthcare | PA53022 | |
| DAPI (4,6-diamidino-2-phenylindole) | Thermo Fisher Scientific | D21490 | |
| Deoxyribonucleic acids single strand from salmon testes | Sigma | D7656 | |
| Dextran sulfate (powder) | Santa Cruz | sc-203917A | |
| Digoxigenin 11 d-UTP | Roche | 11093088910 | |
| DMEM | Thermo Fisher Scientific | 21969-035 500mL | |
| DNA polymerase I | Thermo Fisher Scientific | 18010-017 | |
| DNase I | Sigma | AMPD1 | |
| dNTPs (C-G-A-T) | Euroclone | BL0423A/C/G | |
| EGF | Sigma | E9644.2MG | |
| Ethanol | Sigma | 02860-1L | |
| FBS Hyclone | Thermo Fisher Scientific | SH30109 | |
| Formaldehyde solution | Sigma | F8775-25mL | |
| Formamide | Sigma | F9037 | |
| Glutammine | Thermo Fisher Scientific | 25030-024 100mL | |
| Glycerol | Sigma | G5516-100mL | |
| Glycogen | Thermo Fisher Scientific | AM9510 | |
| HCl | Sigma | 30721 | |
| Human Cot-1 DNA | Thermo Fisher Scientific | 15279-001 | |
| Insulin Human | Sigma | I9278-5 mL | |
| MgCl2 | Sigma | 63069 | |
| NaAc (Sodium Acetate, pH 5.2, 3 M) | Sigma | S2889 | |
| NaCl | Sigma | S9888 | |
| Paraformaldehyde | Sigma | 158127-25G | |
| PBS (phosphate-buffered saline) | Sigma | P4417 | |
| Pennycillin/Streptavidin | Thermo Fisher Scientific | 15070-063 100mL | |
| Pepsin | Biorad | P6887 | |
| PhasePrep BAC DNA Kit | Sigma | NA0100-1KT | |
| Poly-L-lysine solution | Sigma | P8920 | |
| ProLong Diamond Antifade Mountant | Thermo Fisher Scientific | P36970 | |
| PureLink Quick Gel Extraction & PCR Purification Combo Kit | Thermo Fisher Scientific | K220001 | |
| PureLink Quick Plasmid Miniprep Kit | Thermo Fisher Scientific | K210010 | |
| RNAse cocktail | Thermo Fisher Scientific | AM2288 | |
| Rubbercement | Bostik | ||
| Slides | VWR | 631-0114 | |
| Streptavidina Alexa fluor 647 | Thermo Fisher Scientific | S21374 | |
| Tri-Sodium Citrate | Sigma | 1110379026 | |
| Tris-HCl | Sigma | T3253-500g | |
| Triton X-100 | Sigma | T8787-250mL | |
| TWEEN 20 | Sigma | P9416-100mL |
Referencias
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nature Reviews: Genetics. 2 (4), 292-301 (2001).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Schmitt, A. D., Hu, M., Ren, B. Genome-wide mapping and analysis of chromosome architecture. Nature Reviews: Molecular Cell Biology. 17 (12), 743-755 (2016).
- Bonev, B., Cavalli, G. Organization and function of the 3D genome. Nature Reviews: Genetics. 17 (12), 772 (2016).
- van Steensel, B., Furlong, E. E. M. The role of transcription in shaping the spatial organization of the genome. Nature Reviews: Molecular Cell Biology. 20 (6), 327-337 (2019).
- Rajarajan, P., Gil, S. E., Brennand, K. J., Akbarian, S. Spatial genome organization and cognition. Nature Reviews: Neuroscience. 17 (11), 681-691 (2016).
- Pombo, A., Dillon, N. Three-dimensional genome architecture: players and mechanisms. Nature Reviews: Molecular Cell Biology. 16 (4), 245-257 (2015).
- Fanucchi, S., Shibayama, Y., Burd, S., Weinberg, M. S., Mhlanga, M. M. Chromosomal contact permits transcription between coregulated genes. Cell. 155 (3), 606-620 (2013).
- Therizols, P., et al. Chromatin decondensation is sufficient to alter nuclear organization in embryonic stem cells. Science. 346 (6214), 1238-1242 (2014).
- Gonzalez-Sandoval, A., et al. Perinuclear Anchoring of H3K9-Methylated Chromatin Stabilizes Induced Cell Fate in C. elegans Embryos. Cell. 163 (6), 1333-1347 (2015).
- Hubner, B., et al. Remodeling of nuclear landscapes during human myelopoietic cell differentiation maintains co-aligned active and inactive nuclear compartments. Epigenetics Chromatin. 8, 47 (2015).
- Aymard, F., et al. Genome-wide mapping of long-range contacts unveils clustering of DNA double-strand breaks at damaged active genes. Nature Structural & Molecular Biology. 24 (4), 353-361 (2017).
- Sellou, H., et al. The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage. Molecular Biology of the Cell. 27 (24), 3791-3799 (2016).
- Krijger, P. H., de Laat, W. Regulation of disease-associated gene expression in the 3D genome. Nature Reviews: Molecular Cell Biology. 17 (12), 771-782 (2016).
- Lupianez, D. G., Spielmann, M., Mundlos, S. Breaking TADs: How Alterations of Chromatin Domains Result in Disease. Trends in Genetics. 32 (4), 225-237 (2016).
- Cortesi, A., et al. 4q-D4Z4 chromatin architecture regulates the transcription of muscle atrophic genes in facioscapulohumeral muscular dystrophy. Genome Research. 29 (6), 883-895 (2019).
- Woltering, J. M., Noordermeer, D., Leleu, M., Duboule, D. Conservation and divergence of regulatory strategies at Hox Loci and the origin of tetrapod digits. PLoS Biology. 12 (1), 1001773 (2014).
- Woltering, J. M., Duboule, D. Tetrapod axial evolution and developmental constraints; Empirical underpinning by a mouse model. Mechanisms of Development. 138, 64-72 (2015).
- de Laat, W., Dekker, J. 3C-based technologies to study the shape of the genome. Methods. 58 (3), 189-191 (2012).
- Denker, A., de Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Finn, E. H., et al. Extensive Heterogeneity and Intrinsic Variation in Spatial Genome Organization. Cell. 176 (6), 1502-1515 (2019).
- Zheng, M., et al. Multiplex chromatin interactions with single-molecule precision. Nature. 566 (7745), 558-562 (2019).
- Solovei, I. Fluorescence in situ hybridization (FISH) on tissue cryosections. Methods in Molecular Biology. 659, 71-82 (2010).
- Cremer, M., et al. Multicolor 3D fluorescence in situ hybridization for imaging interphase chromosomes. Methods in Molecular Biology. 463, 205-239 (2008).
- Legant, W. R., et al. High-density three-dimensional localization microscopy across large volumes. Nature Methods. 13 (4), 359-365 (2016).
- Jungmann, R., et al. Quantitative super-resolution imaging with qPAINT. Nature Methods. 13 (5), 439-442 (2016).
- Chen, B., et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell. 155 (7), 1479-1491 (2013).
- Nelles, D. A., et al. Programmable RNA Tracking in Live Cells with CRISPR/Cas9. Cell. 165 (2), 488-496 (2016).
- Byron, M., Hall, L. L., Lawrence, J. B. A multifaceted FISH approach to study endogenous RNAs and DNAs in native nuclear and cell structures. Current Protocol of Human Genetics. , 15 (2013).
- Chaumeil, J., Augui, S., Chow, J. C., Heard, E. Combined immunofluorescence, RNA fluorescent in situ hybridization, and DNA fluorescent in situ hybridization to study chromatin changes, transcriptional activity, nuclear organization, and X-chromosome inactivation. Methods in Molecular Biology. 463, 297-308 (2008).
- Takizawa, T., Gudla, P. R., Guo, L., Lockett, S., Misteli, T. Allele-specific nuclear positioning of the monoallelically expressed astrocyte marker GFAP. Genes & Development. 22 (4), 489-498 (2008).
- Kraus, F., et al. Quantitative 3D structured illumination microscopy of nuclear structures. Nature Protocols. 12 (5), 1011-1028 (2017).
- Solovei, I., Cremer, M. 3D-FISH on cultured cells combined with immunostaining. Methods in Molecular Biology. 659, 117-126 (2010).
- Skinner, B. M., Johnson, E. E. Nuclear morphologies: their diversity and functional relevance. Chromosoma. 126 (2), 195-212 (2017).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Research. 25 (4), 582-597 (2015).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nature Methods. 9 (7), 743-748 (2012).
- Beliveau, B. J., et al. Single-molecule super-resolution imaging of chromosomes and in situ haplotype visualization using Oligopaint FISH probes. Nature Communication. 6, 7147 (2015).
- Beliveau, B. J., et al. Versatile design and synthesis platform for visualizing genomes with Oligopaint FISH probes. Proceedings of the National Academy of Sciences. 109 (52), 21301-21306 (2012).
- Samacoits, A., et al. A computational framework to study sub-cellular RNA localization. Nature Communication. 9 (1), 4584 (2018).
- Cardozo Gizzi, A. M., et al. Microscopy-Based Chromosome Conformation Capture Enables Simultaneous Visualization of Genome Organization and Transcription in Intact Organisms. Molecular Cell. 74 (1), 212-222 (2019).
- Mateo, L. J., et al. Visualizing DNA folding and RNA in embryos at single-cell resolution. Nature. 568 (7750), 49-54 (2019).
- Nir, G., et al. Walking along chromosomes with super-resolution imaging, contact maps, and integrative modeling. PLoS Genetics. 14 (12), 1007872 (2018).
- Bintu, B., et al. Super-resolution chromatin tracing reveals domains and cooperative interactions in single cells. Science. 362 (6413), (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados