Method Article
Análise multiplexada do sequenciamento de mRNA celular único de células embrionárias de camundongos
Neste Artigo
Resumo
Aqui apresentamos um método multiplexado de sequenciamento de mRNA de célula única para perfilar a expressão gênica em tecidos embrionários de camundongos. O método de sequenciamento de mRNA de célula única baseada em gotículas (scRNA-Seq) em combinação com estratégias multiplexing pode perfilar células únicas a partir de várias amostras simultaneamente, o que reduz significativamente os custos de reagente e minimiza os efeitos experimentais do lote.
Resumo
O sequenciamento de mRNA de célula única tem feito progressos significativos nos últimos anos e tornou-se uma ferramenta importante no campo da biologia do desenvolvimento. Ele tem sido usado com sucesso para identificar populações de células raras, descobrir novos genes marcador, e decodificar informações espaciais e de desenvolvimento temporal. O método de célula única também evoluiu da tecnologia Fluidigm C1 microfluídica para as soluções baseadas em gotículas nos últimos dois a três anos. Aqui usamos o coração como um exemplo para demonstrar como perfilar as células do tecido embrionário do rato usando o método scRNA-Seq baseado em gotículas. Além disso, integramos duas estratégias no fluxo de trabalho para perfilar várias amostras em um único experimento. Usando um dos métodos integrados, temos simultaneamente perfilado mais de 9.000 células de oito amostras de coração. Estes métodos serão valiosos para o campo da biologia do desenvolvimento, fornecendo uma maneira rentável de perfil simultaneamente células únicas de diferentes origens genéticas, estágios de desenvolvimento ou locais anatômicos.
Introdução
O perfil transcricional de cada célula varia entre as populações celulares durante o desenvolvimento embrionário. Embora a hibridação in situ molecular única possa ser usada para visualizar a expressão de um pequeno número de genes1,o sequenciamento de mRNA de célula única (scRNA-Seq) fornece uma abordagem imparcial para ilustrar padrões de expressão em todo o genoma de genes em células únicas. Depois que foi publicado pela primeira vez em 20092,scRNA-Seq foi aplicado para estudar vários tecidos em vários estágios de desenvolvimento nos últimos anos3,4,5. Além disso, como o atlas de células humanas lançou seus projetos focados no desenvolvimento recentemente, mais dados de células únicas de tecidos embrionários humanos devem ser gerados em um futuro próximo.
O coração como o primeiro órgão a desenvolver desempenha um papel crítico no desenvolvimento embrionário. O coração consiste em tipos múltiplos da pilha e o desenvolvimento de cada tipo da pilha é temporal e espacial firmemente regulado. Ao longo dos últimos anos, a origem e linhagem celular de células cardíacas em estágios iniciais de desenvolvimento têm sido caracterizadas6, que fornecem uma tremenda ferramenta de navegação útil para a compreensão da patogênese congênita da doença cardíaca, bem como para o desenvolvimento de métodos tecnologicamente mais avançados para estimular a regeneração cardiomiócito7.
O scRNA-Seq passou por uma rápida expansão nos últimos anos8,9,10. Com os métodos recém-desenvolvidos, o projeto e a análise de experimentos unicelulares tornaram-se mais alcançáveis11,12,13,14. O método apresentado aqui é um procedimento comercial baseado nas soluções de gotículas (ver Tabela de Materiais)15,16. Este método apresenta a captura de células e conjuntos de contas de código de barras únicas em uma gota de emulsão de água de óleo controle de um sistema de controlador microfluídico. A taxa de carregamento celular nas gotículas é extremamente baixa, de modo que a maioria das emulsões de gotículas contenha apenas uma célula17. O projeto engenhoso do procedimento vem da separação da única pilha em emulsões da gotícula que ocorrem simultaneamente com código de barras, que permite a análise paralela de pilhas individuais usando RNA-Seq em uma população heterogeneous.
A incorporação de estratégias multiplexing é uma das adições importantes ao fluxo de trabalho tradicional de célulaúnica13,14. Esta adição é muito útil no descarte de dobradores celulares, reduzindo os custos experimentais e eliminando os efeitos do lote18,19. Uma estratégia de código de barras baseada em lipídios e uma estratégia de código de barras baseada em anticorpos (ver Tabela de Materiais)são os dois métodos multiplexing usados principalmente. Códigos de barras específicos são usados para rotular cada amostra em ambos os métodos, e as amostras rotuladas são então misturadas para captura de célulaúnica, preparação da biblioteca e sequenciamento. Posteriormente, os dados de sequenciamento agrupados podem ser separados analisando as sequências de código de barras(Figura 1)19. No entanto, existem diferenças significativas entre os dois métodos. A estratégia de código de barras baseada em lipídios é baseada em oligonucleotídeos modificados por lipídios, que não foram encontrados para ter qualquer preferência do tipo celular. Enquanto a estratégia de código de barras baseada em anticorpos só pode detectar as células que expressam as proteínas de antígeno19,20. Além disso, leva cerca de 10 min para manchar os lipídios, mas 40 min para manchar os anticorpos (Figura 1). Além disso, os oligonucleotídeos modificados por lipídios são mais baratos do que os oligonucleotídeos conjugados com anticorpos, mas não comercialmente disponíveis no momento da escrita deste artigo. Finalmente, a estratégia baseada em lipídios pode multiplex 96 amostras em um experimento, mas a estratégia baseada em anticorpos atualmente só pode multiplex 12 amostras.
O número de células recomendado para multiplex em um único experimento deve ser inferior a 2,5 x 104, caso contrário, levará a uma alta porcentagem de dobradores celulares e potencial contaminação por mRNA ambiente. Através das estratégias multiplexing, o custo de captura de célulaúnica, geração de cDNA e preparação da biblioteca para várias amostras será reduzido ao custo de uma amostra, mas o custo de sequenciamento permanecerá o mesmo.
Protocolo
O procedimento animal está de acordo com o Comitê Institucional de Cuidados e Uso de Animais da Universidade de Pittsburgh (IACUC).
1. Dissecção embrionária do coração do rato e preparação da suspensão da única pilha
NOTA: Esta etapa pode levar algumas horas, dependendo do número de embriões para dissecar.
- Para adquirir corações embrionários E18.5, eutanásia um rato CD1 grávida por administração de CO2. Use uma navalha para remover o cabelo indesejado na área abdominal e desinfetar a pele com 70% de etanol.
- Corte a pele do abdômen usando tesoura esterilizada e disseque cuidadosamente os embriões para fora e rapidamente colocá-los em soro de soro de soro frio tampão de fosfato (PBS) no gelo.
- Isolar corações de cada embrião individual cuidadosamente em um prato de 10 cm cheio de PBS frio um microscópio estereoscópico usando fórceps e tesouras.
NOTA: Mantenha as quatro câmaras intactas, dissecando o pulmão com o coração juntos e não pegar diretamente / puxar o coração com instrumentos cirúrgicos. - Transfira ~ 10 corações em um novo prato de 10 cm cheio de PBS frio e micro-dissecar os corações no átrio esquerdo, átrio direito, ventrículo esquerdo e ventrículo direito.
NOTA: Isto é suposto produzir mais de 1 x 106 pilhas de cada amostra. Recomendamos começar com pelo menos 1 x 105 células por amostra. - Transfira cada um dos 4 tecidos de câmara para um tubo de 1,5 mL e pique-os em pedaços usando uma tesoura. Centrífuga a 300 x g por 3 min para coletar os tecidos.
- Depois de aspirar o supernatant, adicione 1 mL de 0,25% Trypsin / EDTA para cada tubo e incubar em um banho de água 37 ° C por 10 min. Pipet para cima e para baixo suavemente 7-8 vezes usando uma pipeta P1000.
- Se o estágio embrionário for mais velho do que E11.5, adicione 1 mL de 10 mg/mL collagenase A/B mistura e incubar em 37 °C para 10-20 min. Suavemente pipet para cima e para baixo até que a maioria das células são dissociadas.
- Transfira as células para um tubo de 15 mL e adicione a solução de sal equilibrado (HBSS) de 8 mL Hank para diluir as enzimas. Desça as células a 300 x g por 5 min. Suspenda as células em 1 mL de PBS e transfira-as para um tubo de 1,5 mL. Filtrar as células através de um filtro de células de 40 μm.
- Pegue 15 μL de volume de cada amostra e misture com a mesma quantidade de 0,04% de trypan azul. Carregue isso em uma câmara de contagem de células e conte as células em um contador de células.
NOTA: Para gerar resultados de alta qualidade, a viabilidade celular é recomendada para ser superior a 95%.
2. Célula única multiplexing códigos de barras
NOTA: Esta etapa leva pelo menos 40 min, que varia com base no número de amostras processadas. Uma área de bancada limpa tratada com solução de descontaminação rnase é necessária para etapas de pré-amplificação (passo 2,11 a 3,11), e uma área de bancada limpa separada é necessária para as etapas pós-amplificação (as etapas após 3,11).
-
Procedimento de código de barras baseado em lipídios (procedimento opcional 1)
- Com base na concentração celular, mantenha menos de 5 x 105 células por amostra. Certifique-se de que a suspensão celular está livre de detritos e agregados celulares.
- Prepare 2 μM âncora / solução de estoque de código de barras e 2 μM co-âncora solução de estoque para cada amostra(Tabela 1).
NOTA: A âncora e co-âncora foram gentilmente presenteados pelo laboratório Dr. Zev J. Gartner. Para sintetizar esses oligonucleotídeos modificados por lipídios, as sequências de DNA foram conjugadas com ácido graxo em suporte sólido e purificadas pela cromatografia líquida de alto desempenho (HPLC)18,19. - Lave as células duas vezes com PBS e recolher as células em 300 x g para 5 min. Suspender as células em 180 μL de PBS.
- Adicione 20 μL de âncora / solução de estoque de código de barras e pipeta para cima e para baixo suavemente para misturar. Incubar no gelo por 5 min.
- Adicione 20 μL de solução de estoque co-âncora e pipeta para cima e para baixo suavemente para misturar, em seguida, incubar no gelo por mais 5 min.
- Adicione 1 mL de PBS frio com 1% BSA e centrífuga a 300 x g por 5 min a 4 °C. Lave pelo menos mais 2 vezes com gelo frio 1% BSA em PBS.
- Combine todas as amostras e filtre através de 40 filtros de células μm. Conte as células e mantenha a suspensão celular no gelo para usar na seção 3.
-
Procedimento de código de barras baseado em anticorpos (procedimento opcional 2)
- Centrífuga 1 x 106-2 x 106 células para cada amostra (a partir do passo 1.8) a 300 x g por 5 min e suspendê-los em 100 μL de buffer de coloração (Tabela 1) em 1,5 mL tubos de ligação baixa.
- Adicione 10 μL Fc bloqueando reagente e incubado por 10 min a 4 °C.
- Prepare anticorpos (ver Tabela de Materiais)por centrífuga a 14.000 x g por 10 min a 2-8 °C.
- Adicione 1 μg de cada anticorpo oligo-conjugado a 50 μL do amortecedor da coloração da pilha para fazer a solução da mancha do anticorpo20. Adicione uma solução de coloração de anticorpos a cada tubo de amostra. Incubar por 30 min a 4 °C.
- Lave as células 3 vezes com 1 mL de PBS, gire por 5 min a 350 x g a 4 °C.
- Piscina todas as amostras em proporções desejadas em 1 mL de tampão de coloração, girar para 5 min em 350 x g em 4 °C.
- Resuspenda as células em PBS em concentração apropriada (até 1.500 células/μL) e filtra as células através de um filtro de células de 40 μm. Imediatamente proceder para o próximo passo.
3. Geração de gotículas e transcrição reversa mRNA
NOTA: Este passo leva cerca de 90 min para uma reação multiplexed.
- Equilibre as contas de gel (ver Mesa de Materiais)à temperatura ambiente por 30 min. Retire os reagentes do kit de emulsão (GEMs) (ver Table of Materials)e mantê-los em sua temperatura indicada.
- Monte o chip B em um suporte de chip (ver Tabela de Materiais).
- Dispensar 75 μL de 50% de solução de glicerol para os poços não utilizados na linha 1; 40 μL na linha 2; 280 μL na linha 3. Não adicione glicerol em qualquer poço de recuperação na linha superior do chip.
- Prepare a mistura mestre no gelo de acordo com a tabela 1. Adicione o volume apropriado de suspensão celular e água livre de nuclease para misturar mestre de acordo com uma tabela de calculadora de volume de suspensão celular17 e delicadamente pipeta a mistura. Dispense 75 μL da mistura da pilha no centro inferior da amostra bem na fileira 1 sem introduzir bolhas.
- Vortex as contas de gel para 30 s usando um adaptador vortex e lentamente dispensar 40 μL de contas de gel para o centro inferior da conta de gel bem na linha 2 sem introduzir bolhas.
NOTA: É fundamental esperar por 30 s entre a adição de células e contas de gel para evitar a falha de molhar. - Para o poço de óleo partitioning na linha 3, dispensar 280 μL de óleo partitioning através da parede lateral do poço.
NOTA: Carregar menos de 270 μL de óleo partitioning levará à geração de GEMs anormais. - Anexar a junta no chip, não pressione para baixo sobre a junta e mantê-lo horizontal para evitar molhar a junta.
- Carregue o chip montado com a junta no controlador de cromo e execute o programa de célula única B de cromo (ver Tabela de Materiais),imediatamente siga para a próxima etapa quando o programa for concluído.
- Tire o chip e descarte a junta. Dobre a tampa de volta para expor poços em 45°, verifique o nível líquido para se certificar de que não há tamancos estão presentes.
- Lentamente aspirar 100 μL de GEMs dos pontos mais baixos do poço de recuperação e verificar a uniformidade dos GEMs. Dispense GEMs em um novo tubo de reação em cadeia de polimerase (PCR) no gelo com as pontas da pipeta contra a parede lateral do tubo.
NOTA: Se o excesso de camada aquosa é observado, sugere-se para repreparar as amostras. Importante, tire uma foto da mistura quando os GEMs ainda estão nas pontas da pipeta. Esta imagem pode dizer se há uma falha de molhar, GEMs parcialmente emulsionados e tamancos reagentes. A fotografia também pode ser usada como prova para obter reembolso da empresa de reagentes com reagentes de substituição e chips. - Coloque o tubo em um cycler térmico e realizar o procedimento de transcrição reversa(Tabela 2).
NOTA: Pare aqui ou siga para o próximo passo. O produto PCR pode ser armazenado a -20 °C por até uma semana.
4. amplificação cDNA
NOTA: Este passo leva cerca de 150 min.
- Limpeza de transcrição reversa de célula única pós
- Retire os reagentes de amplificação de cDNA do kit GEMs (ver Tabela de Materiais)e mantê-los em sua temperatura indicada.
- Adicione 125 μL de agente de recuperação à amostra à temperatura ambiente para adquirir uma mistura bifásica. Nenhum líquido opaco deve ser observado e evitar pipetting ou vortexing a mistura.
- Depois de esperar por 60 s, retire lentamente 125 μL de agente de recuperação do fundo do tubo.
- Vortex as contas magnéticas (ver Tabela de Materiais)completamente para 30 s e usá-lo imediatamente para preparar a mistura de limpeza de contas(Tabela 1). Os reagentes devem ser adicionados sequencialmente como listado.
- Vortex a mistura de limpeza de contas e adicionar 200 μL à amostra. Pipeta a mistura 10 vezes, em seguida, incubar-lo por 10 min à temperatura ambiente.
- Adicione os reagentes sequencialmente como listados na Tabela 1 para preparar a solução de elução e elute o cDNA da seguinte forma.
- Coloque as amostras no ímã (alta posição) (ver Tabela de Materiais)até que a solução se limpe, em seguida, retire o supernatant.
- Adicione 200 μL de 80% de etanol à pelota. Espere por 30 s, em seguida, retire o etanol.
- Repita o passo 4.1.6.2 por mais 2 vezes. Centrífuga brevemente e coloque no ímã (posição baixa). Retire cuidadosamente o etanol restante e o ar seco por menos de 2 min.
NOTA: NÃO exceda a secagem do ar após 2 min, caso contrário, a eficiência de elução diminuirá. - Retire a amostra do ímã. Adicione 35,5 μL de solução de elução e pipeta para misturar 15 vezes. Incubar 2 min à temperatura ambiente.
- Coloque a amostra no ímã (alta posição) até que a solução se limpe. Transfira 35 μL da amostra para uma nova tira de tubo.
NOTA: Este procedimento de purificação também é utilizado nas etapas 4.2.1.6, 4.2.2.3, 5.8, 6.1.10 e 6.2.6. Preste atenção à concentração de contas magnéticas e ao volume de água tampão/ultrapura e b usada para elute as amostras a cada passo.
- Quantificar o tamanho, concentração e integridade do CDNA eluted usando um instrumento automatizado de eletroforese21 (ver Tabela de Materiais)(Figura 2).
- Amplificação do CDNA
-
amplificação cDNA usando a estratégia de código de barras baseada em lipídios (procedimento opcional 1)
- Prepare a mistura de reação de amplificação(Tabela 1)no gelo.
- Adicione a mistura de reação de amplificação a 35 μL de amostras de cDNA (do passo 4.1.6.7). Pipeta a mistura, e centrífuga brevemente. Incubar a mistura em um cycler térmico que segue o procedimento da amplificação do cDNA(tabela 2).
- Após o vórtice completamente, adicione 120 μL de reagente seleto e 100 μL de água ultrapura a 100 μL de amostra para adquirir uma concentração de 0,6 x de reagente seleto (ver Tabela de Materiais). Pipeta a mistura por 15 vezes.
- Incubar para 5 min à temperatura ambiente e, em seguida, coloque a amostra em ímã até que as soluções se tornem claras.
NOTA: CDNA endógeno na fração de contas e multiplexing cDNA de código de barras está no supernatant. - Transfira o supernatant em um tubo de ligação baixa de 1,5 mL para multiplexing construção da biblioteca cDNA de código de código de código de código na etapa 6.1.
- Limpe o cDNA endógeno seguindo as etapas em 4.1.6 e elute-as com 40 μL do amortecedor de EB.
- Executar 1 μL de amostra de cDNA purificada em um instrumento de eletroforese automatizado (ver Tabela de Materiais)para analisar/quantificar o cDNA.
- Aliquot 10 μL de cDNA em um novo tubo PCR para a construção de bibliotecas endógenas.
NOTA: Pare aqui ou siga para o próximo passo. A amostra restante pode ser armazenada em -20 °C por até 4 semanas para gerar bibliotecas adicionais, se necessário.
-
amplificação de cDNA na estratégia de código de barras baseada em anticorpos (procedimento opcional 2)
- Adicione 2 pmol de HTO e ADT primer aditivo e 15 μL de primers cDNA para 50 μL de mistura de reação de amplificação (Tabela 1) e realizar amplificação cDNA com o procedimento de amplificação cDNA(Tabela 2).
- Use 0,6 x reagente seleto para separar cDNA endógeno (fração de contas) e multiplexing código de barras cDNA (em supernatant). Lembre-se de salvar o supernatant para realizar a construção da biblioteca de código de barras no passo 6.2.
- Purificar e elute a transcrição endógena cDNA seguindo as etapas em 4.1.6, executar o controle de qualidade (QC) das bibliotecas e alíquota 10 μL em um tubo novo do PCR para a construção endogenous da biblioteca.
-
amplificação cDNA usando a estratégia de código de barras baseada em lipídios (procedimento opcional 1)
5. Preparação da Biblioteca transcrição endógena
NOTA: Este passo leva cerca de 120 min.
- Mantenha os reagentes de construção da biblioteca de expressão gênica do kit da biblioteca (ver Tabela de Materiais)em sua temperatura indicada, respectivamente.
- Prepare a mistura de fragmentação(Tabela 1)sobre gelo, pipeta para misturar e centrífuga brevemente.
- Adicione 25 μL EB buffer para a amostra de cDNA purificada de 10 μL (do passo 4.2.1.8 ou 4.2.2.3) e adicione a mistura de fragmentação recém-preparada de 15 μL à amostra, pipeta a mistura 15 vezes no gelo e centrífuga brevemente.
- Transfira a amostra para um pedalador térmico pré-resfriado e inicie o programa pcr para fragmentação, reparo final e adesão(Tabela 2).
- Vortex o reagente seleto para suspender contas magnéticas e usar sucessivamente 0.6x e 0.8x reagents seletos para fazer uma seleção dobro-face do tamanho de acordo com o guia17,22. Use 50 μL de buffer EB para elute o DNA.
- Prepare a mistura de ligadura adaptativa (Tabela 1),em seguida, pipette a mistura completamente e centrífuga brevemente.
- Adicione 50 μL da mistura da ligadura do adaptador a 50 μL da amostra, a mistura da pipeta outra vez por 15 vezes e a centrífuga momentaneamente. Realizar a ligadura adaptativa de acordo com o protocolo em um cycler térmico(Tabela 2).
- Use 0,8x reagente seleto para purificar o produto de ligadura e elute a amostra purificada com 30 μL de buffer EB (ver passo 4.1.6).
- Prepare a mistura de PCR do índice de amostra(Tabela 1)e adicione 60 μL à amostra purificada. Adicione 10 μL de índice de amostra à amostra, mistura de pipeta para cima e para baixo por 5 vezes e centrífuga brevemente, incubar em um cycler térmico seguindo o protocolo do índice de amostra(Tabela 2).
NOTA: Pare aqui ou siga para o próximo passo. Se for usado mais de um poço, escolha um índice de amostra específico (ver Tabela de Materiais)para cada poço. Lembre-se de registrar o ID do índice usado para cada poço e garantir nenhuma sobreposição em uma execução de sequenciamento multiplexado. - Sucessivamente usar 0,6x e 0,8x selecionar reagentes para fazer uma seleção de tamanho duplo-face para adquirir 35 μL de dna biblioteca celular endógena purificada17.
- QC a biblioteca celular endógena antes do sequenciamento (Figura 2).
6. Preparação de bibliotecas de cDNA multiplexing sample barcode
NOTA: Este passo leva pelo menos 120 min.
-
Exemplo de geração de biblioteca de código de barras para a estratégia de multiplexing baseada em lipídios (procedimento opcional 1)
- Adicione 520 μL de reagente seleto e 360 μL de isopropanol para provar cDNA de código de barras da etapa 4.2.1.5 para obter uma concentração de reagente selecionada de 3,2x. Pipeta a mistura 10 vezes e incubar à temperatura ambiente por 5 min.
- Coloque o tubo em um rack magnético e aguarde a solução para limpar. Em seguida, descartar o supernatant.
- Use 500 μL de 80% de etanol para lavar contas duas vezes em um ímã e esperar por 30 s após cada lavagem.
- Brevemente centrífuga as contas e coloque em um ímã. Retire o etanol restante com uma micropipette P10 e deixe contas por 2 min.
- Retire o tubo do rack ímã e resuspender contas em 50 μL de buffer EB. Pipet cima e para baixo para misturar completamente. Incubar à temperatura ambiente por 2 min.
- Devolva o tubo para um ímã e aguarde a solução para limpar. Transfira o supernatant (cDNA de código de barras de amostra) para um novo tubo PCR. Tenha cuidado para não transferir contas.
- Quantificar a concentração de código de barras de amostra cDNA23.
- Prepare a mistura da biblioteca de código de barras lipídica(Tabela 1),adicione 3,5 ng de cDNA de código de barras purificado (do passo 6.1.6) e água livre de nuclease para um volume total de 50 μl.
- Mantenha-o em um cycler térmico seguindo a biblioteca de código de barras pcr baseada em lipídios(Tabela 2).
- Use 1.6x reagente selecionado para purificar o produto PCR e elute o DNA com 25 μL de buffer EB (ver passo 4.1.6).
- Quantificar a concentração da biblioteca usando um método de análise de DNA de alta sensibilidade24 da diluição inicial de 1:5 (Figura 2).
-
Exemplo de geração de biblioteca de código de barras para a estratégia de multiplexing baseada em anticorpos (procedimento opcional 2)
- Adicione o volume de reação adicional de 1,4 x de reagente selecionado ao supernatant contendo códigos de barras de amostra adquiridos do passo 4.2.2.3 para obter uma relação de reagente selecionada 2x.
- Lave as contas com 80% de etanol seguindo os passos em 4,1,6 e elute o cDNA de código de barras com água ultrapura.
- Executar o protocolo de seleção com reagente 2x selecionar pela segunda vez e elute usando água ultrapura.
- Prepare a mistura da biblioteca de código de barras de anticorpos(Tabela 1),e adicione 45 μL de cDNA de código de barras purificado da última etapa.
- Incubar em um cycler térmico seguindo a biblioteca de código de barras de anticorpos PCR(Tabela 2)20.
- Use 1.6x reagente selecionado para purificar o produto PCR e elute a amostra purificada com 30 μL de água ultrapura (ver passo 4.1.6).
7. Sequenciamento da Biblioteca
NOTA: Várias plataformas de sequenciamento de próxima geração, como HiSeq 4000 e NovaSeq, podem ser usadas para sequenciar as bibliotecas de transcrição endógenas e as bibliotecas de código de barras multiplexing.
- Use uma plataforma de sequenciamento de próxima geração de escolha para sequenciar as bibliotecas de transcrição endógenas e bibliotecas multiplexing de código de barras.
- Diluir as bibliotecas de acordo com as recomendações de um especialista em uma empresa de sequenciamento ou instalação de sequenciamento. O mínimo 20.000 leituras por a pilha é recomendada para a biblioteca endógena do transcript e 3000 leituras para as bibliotecas do código de barras.
8. Análise de dados
NOTA: Desmultiplex os dados de sequenciamento usando o recurso baseado em nuvem BaseSpace ou executando o pacote bcl2fastq em um servidor UNIX.
-
Análise de dados de transcriptoma endógeno
- Com os dados fastq gerados a partir de software demultiplexing, executar "mkfastq" no pipeline de análise de dados comercialmente disponíveis (ver Tabela de Materiais)para demultiplex mais cada código de barras GEMs.
- Executar "contagem" para realizar o alinhamento, filtragem, contagem de código de barras e contagem umi.
- Opcionalmente, execute "agressor" para agregar várias faixas de sequenciamento de um único experimento.
- Use "navegador de células" (ver Tabela de Materiais)para visualizar dados, células cluster, identificar genes expressos diferencialmente e gerar parcelas de mapa de calor tSNE ou expressão gênica.
- Opcionalmente, use uma plataforma baseada em R bem conservada25 (ver Tabela de Materiais)para normalizar e dimensionar dados, identificar genes expressos diferencialmente e gerar parcelas tSNE/UMAP e mapas de calor de expressão gênica (Figura 3).
-
Multiplexing análise de dados de código de barras
-
Análise dos dados da estratégia de código de barras baseada em lipídios
- Use o pipeline de análise de dados comercialmente disponível ou o pacote dedemultiplex R(https://github.com/chris-mcginnis-ucsf/MULTI-seq)para converter os arquivos FASTQ de código de barras de amostra em uma matriz de contagem umi de código de barras de amostra.
- Carregue a matriz de contagem umi de código de barras juntamente com dados de transcriptoma endógenos para uma plataforma baseada em R (ver Tabela de Materiais)para análise de integração (Figura 3).
-
Análise dos dados da estratégia de código de barras baseada em anticorpos
- Use a "contagem" do pipeline de análise de dados comercialmente disponível para mapear os códigos de barras, fornecendo o arquivo CSV da biblioteca e o arquivo de referência de referência de hashtag saque.
- Carregue a matriz unificada de código de barras de recursos de saída, que contém contagens de expressão gênica juntamente com as contagens de código de barras de recurso para cada código de barras celular, para uma plataforma baseada em R para análise a jusante.
-
Análise dos dados da estratégia de código de barras baseada em lipídios
Resultados
Neste estudo, usamos o coração embrionário do camundongo como um exemplo para exibir como o sequenciamento multiplexado de mRNA de célula única foi realizado para processar as diferentes amostras de partes separadas de um órgão simultaneamente. Os corações do rato E18.5 CD1 foram isolados e dissecados no átrio esquerdo (LA), átrio direito (RA), ventrículo esquerdo (LV) e ventrículo direito (RV). As células atrial e ventricular foram então remetidos de forma independente usando um procedimento de código de barras baseado em lipídios e misturados antes da geração gems e transcrição reversa. A visão geral esquemática é mostrada na Figura 1. Nós quantificado a concentração de cDNA antes da construção da biblioteca (Figura 2A). Uma das distinções na realização de scRNA-Seq multiplexado do scRNA-Seq padrão é que a biblioteca cDNA endógena e a biblioteca de cDNA de código de barras foram adquiridas separadamente após a amplificação e purificação do cDNA (Passo 4.2.1 e 4.2.2.2). As duas bibliotecas também foram qualificadas em nosso experimento(Figura 2B,C). O sequenciamento de última geração e a análise de dados foram realizados seguidos pela construção de bibliotecas e qc.
Usamos a plataforma HiSeqX para sequenciar ambas as bibliotecas na mesma faixa de sequenciamento. Com os dados de sequenciamento, primeiro separamos os dados de transcrição endógenos e os dados de código de barras usando o programa BaseSpace. Em seguida, analisamos a expressão de código de barras em cada célula e encontramos 8 grupos de células individuais que expressam exclusivamente um tipo de código de barras, representando células de 8 amostras diferentes(Figura 3A). Além disso, também descobrimos que algumas células não expressam nenhum código de barras, que definimos como células negativas, e algumas células expressam dois códigos de barras diferentes, que representam dobradas(Figura 3B). Em resumo, descobrimos que cerca de 70% das células são monotões, 25% das células são negativas e 5% das células são dobradas.
Com as células singlet, podemos realizar análises a jusante para entender a heterogeneidade celular e regulamentos moleculares. As análises potenciais podem ser a anotação do tipo celular (Figura 4A),identificação de tipo de células novas/raras (Figura 4B),análise comparativa da zona anatômica (Figura 4C),e análise de via de ontologia gênica, como separações de fase de ciclo celular (Figura 4D).

Figura 1: Fluxo de trabalho de sequenciamento de mRNA de célula única multiplexado. Os corações embrionários do dia 18.5 do estágio foram analisados usando um procedimento multiplexed do sequenciamento da única pilha. RT = transcrição reversa. Clique aqui para ver uma versão maior deste número.

Figura 2: Resultados representativos do QC em etapas diferentes. (A)ANÁLISE QC do cDNA da etapa 4.1.7. O tamanho do fragmento alvo é de 200 a 9000 bp. (B) Biblioteca de código de barras e(C)biblioteca de código de barras foram analisados com um instrumento de eletroforese automatizado. O tamanho do fragmento alvo para a biblioteca endógena é de 300-600 bp, e o tamanho do DNA da biblioteca de código de barras é de cerca de 172 bp. Por favor, clique aqui para ver uma versão maior deste número.
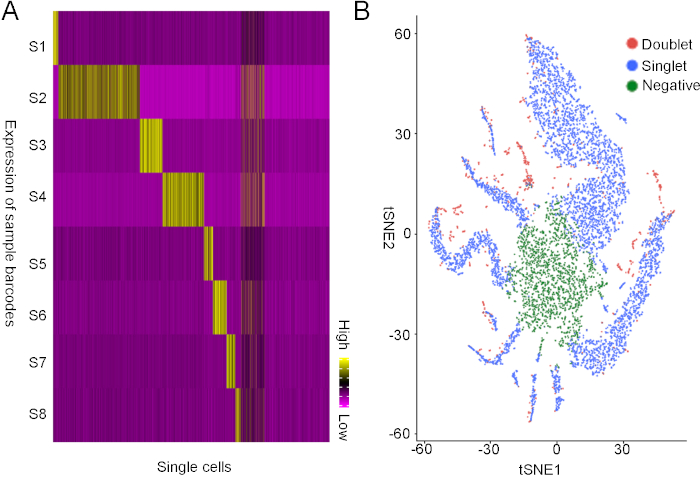
Figura 3: Demultiplexing os dados de sequenciamento da estratégia de código de barras baseada em lipídios. (A)Análise não supervisionada da expressão do código de barras. O eixo X representa células únicas, e o eixo y representa códigos de barras. Cada uma das 8 populações de células únicas foi identificada para expressar exclusivamente um dos 8 códigos de barras. Observe que algumas células expressam mais de um código de barras, e algumas células não expressam nenhum código de barras. (B) t-SNE enredo das células singlet, células duplas, e células negativas. Clique aqui para ver uma versão maior deste número.

Figura 4: Análise avançada de dados transcricionais de células únicas. (A-D) Dados de células únicas podem ser analisados de diferentes maneiras para entender a heterogeneidade celular e as vias moleculares. Listamos várias aplicações aqui como exemplos. Células únicas foram carregadas em um pacote R para identificar tipos de células (A), populações de células raras (B),zonas anatômicas celulares(C),e fases do ciclo celular (D). Clique aqui para ver uma versão maior deste número.
| Nome da mistura | Composição |
| Mistura de colagem | 10 mg/mL colagem A e 10 mg/mL colagem B, dissolvido em HBSS ++ com 40% FBS. |
| 2 μM Anchor/Barcode stock solution 2 μM Anchor/Barcode stock solution 2 μM Anchor/Barcode stock solution 2 μ | Misture 50 μM âncora e 10 μM de código de barras em 1:1 proporção molar em PBS (sem FBS ou BSA) para um volume total de 25 μL. |
| 2 μM Co-Anchor solução de ações | Diluir 1 μL 50 μM Co-Anchor com 24 μL PBS (sem FBS ou BSA). |
| Tampão de coloração | PBS contendo 2% BSA, 0,01% Tween 20 |
| Mistura mestre | 20 μL RT Reagent, 3.1 μL Oligo, 2 μL Reducing Agent B, 8.3 μL RT Enzyme C. |
| Mistura da limpeza dos grânulos | 182 μL Cleanup Buffer, 8 μL Selection Reagent, 5 μL Reducing Agent B, 5 μL Nuclease-free Water. |
| Mistura de reação de amplificação | 1 μL de 10 μM Lipid-etiquetado primer aditivo, 15 μL cDNA primer, 50 μL Amp Mix |
| Solução de lubrificação | 98 μL Buffer EB, 1 μL 10% Tween 20, 1 μL Reducing Agent B. |
| Mistura de fragmentação | 5 μL Fragmentação Buffer, 10 μL Fragmentação Enzima. |
| Mistura de ligadura de adaptador | 20 μL Ligação Buffer, 10 μL DNA Ligase, 20 μL Adaptador Oligos. |
| Mistura pcr do índice de amostra | 50 μL Amp Mix, 10 μL SI Primer |
| Mistura da biblioteca de código de barras lipídico | 26,25 μL de 2× Hot Start mix mestre, 2,5 μL de 10 μM RPIX primer, 2,5 μL de 10 μM TruSeq Universal Adapter primer (ver tabela de materiais) |
| Mistura da biblioteca do código de barras do anticorpo | 50 μL de 2× Hot Start mix mestre, 2,5 μL de 10 μM RPIX primer, 2,5 μL de 10 μM P5-smart-pcr híbrido oligo |
Tabela 1: As misturas de reagente usadas no protocolo.
| Procedimento de incubação | Temperatura(1) | Tempo |
| Incubação GEM-RT | Temperatura da tampa 53 °C | |
| Passo 1 | 53 °C | 45 min |
| Passo 2 | 85 °C | 5 min |
| Passo 3 | 4 °C | Segurar |
| 10x Genômica cDNA Amplificação | Temperatura da tampa 105 °C | |
| Passo 1 | 98 °C | 3 min |
| Passo 2 | 98 °C | 15 s |
| Passo 3 | 63 °C | 20 s |
| Passo 4 | 72 °C | 1 min |
| Passo 5 | Repita os passos 2 a 4 para 12 ciclos no total (2) | |
| Passo 6 | 72 °C | 1 min |
| Passo 7 | 4 °C | Segurar |
| Construção da biblioteca | Temperatura da tampa 65 °C | |
| Bloco pré-legal | 4 °C | Segurar |
| Fragmentação | 32 °C | 5 min |
| Reparação final e a-rejeito | 65 °C | 30 min |
| Segurar | 4 °C | Segurar |
| Ligação do adaptador | Temperatura da tampa 30 °C | |
| Passo 1 | 20 °C | 15 min |
| Passo 2 | 4 °C | Segurar |
| Índice de amostra PCR | Temperatura da tampa 105 °C | |
| Passo 1 | 98 °C | 45 s |
| Passo 2 | 98 °C | 20 s |
| Passo 3 | 54 °C | 30 s |
| Passo 4 | 72 °C | 20 s |
| Passo 5 | Repita os passos 2 a 4 para 12 ciclos no total (3) | |
| Passo 6 | 72 °C | 1 min |
| Passo 7 | 4 °C | Segurar |
| Pcr da biblioteca de código de barras lipídico | ||
| Passo 1 | 95 °C | 5 min |
| Passo 2 | 98 °C | 15 s |
| Passo 3 | 60 °C | 30 s |
| Passo 4 | 72 °C | 30 s |
| Passo 5 | Repita os passos 2 a 4 para 10 ciclos no total (4) | |
| Passo 6 | 72 °C | 1 min |
| Passo 7 | 4 °C | Segurar |
| Biblioteca de código de barras de anticorpos PCR | ||
| Passo 1 | 95 °C | 3 min |
| Passo 2 | 95 °C | 20 s |
| Passo 3 | 60 °C | 30 s |
| Passo 4 | 72 °C | 20 s |
| Passo 5 | Repita os passos 2 a 4 para 8 ciclos no total (5) | |
| Passo 6 | 72 °C | 5 min |
| Passo 7 | 4 °C | Segurar |
Tabela 2: O procedimento de incubação utilizado no protocolo. (1) Preste atenção à temperatura da tampa diferente usada em cada procedimento. (2) Definir números totais de ciclo de acordo com a carga celular: 13 ciclos para carga celular <500; 12 ciclos para 500-6.000 carga celular; 11 ciclos para carga celular de 6.000 celulares. (3) Definir números totais de ciclo de acordo com a entrada cDNA: 14-16 ciclos para 1-25 ng cDNA; 12-14 ciclos para 25-150 ng cDNA; 10-12 ciclos para 150-500 ng cDNA; 8-10 ciclos para 500-1.000 ng cDNA; 6-8 ciclos para 1000-1500 ng cDNA. (4) Definir números totais do ciclo de acordo com a entrada cDNA: 8-12 ciclos. (5) Definir números totais do ciclo de acordo com a entrada cDNA: 6-10 ciclos.
| Lipídios baseados em código de barras Oligonucleotídeos | |
| Âncora LMO | 5'-TGGAATTCTCGTGCCAAGGGTAACGATCCAGCTGTCACT-Lipid-3' |
| LMO co-âncora | 5'-Lipid-AGTGACAGCTGGATCGTTAC-3' |
| Código de Barras Oligo | 5'-CCTTGGCACCCGAGAATTCCANNNNNNNNNA30-3' |
| Primer aditivo de código de barras lipídico | 5'-CTTGGCACCCGAGAATTCC-3' |
| RPIX Primer | 5'-CAAGCAGAAGACGGCATACGAGATnNNNNNNGTGACTGGAGTTCC TTGGCACCCGAGAATTCCA-3' TTGGCACCCGAGAATTCCA-3' |
| Primer adaptador universal | 5'-AATGATACGGACCACCACCTCTACACTCTTCCCTACACGAC GCTCTTCCGATCT-3' GCTCTTCCGATCT-3' |
| Anticorpobaseado em código de barras Oligonucleotídeos | |
| Oligo de código de barras do anticorpo | 5'-GTGACTGGAGTTTCAGACGTGTGCTCTTCCGATCTNNNNNNNNNNNNNNN NNBAaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa*a*a-3' |
| Primer aditivo de HTO | 5'-GTGACTGGAGTTTCAGACGTGTGCTC-3' |
| Primer aditivo ADT | 5'-CCTTGGCACCCGAGAATTCC-3' |
| P5-smart-pcr híbrido oligo | 5'-AATGATACGGACCACCACCTCTACACGCCTGTCCGCGGAA GCAGTGGTATCAACGCAGAGT*A*C-3' |
Tabela 3: Sequências de oligonucleotídeos utilizadas neste protocolo. N = Código de barras ou sequência de índice; * = Phosphorothioate bond * = Phosphorothioate bond
Discussão
Neste estudo, demonstramos um protocolo para analisar perfis de transcrição de células únicas. Também fornecemos dois métodos opcionais para amostras multiplex no fluxo de trabalho scRNA-Seq. Ambos os métodos provaram ser viáveis em vários laboratórios e forneceram soluções para executar um experimento de célula única sem efeito de custo e lote18,26.
Há alguns passos que devem ser seguidos cuidadosamente ao passar pelo protocolo. Uma suspensão de célula única ideal deve ter >90% das células viáveis e a densidade celular também deve estar dentro de um intervalo específico27. É fundamental obter uma boa qualidade das células para minimizar a presença de agregados celulares, detritos e fibras. Os agregados celulares têm impacto negativo no multiplexing da amostra e têm um risco potencial para entupir a máquina geradora de gotículas17. De um modo geral, um filtro de células de 30-40 μm é ideal para remover grandes grupos e detritos, preservando as amostras de células porque a maioria das células diminuirá abaixo de 30 μm após a dissociação. Os núcleos unicelulares são recomendados para serem usantes se o diâmetro celular for maior que 30 μm. Em estágios embrionários iniciais, o tamanho da célula para todos os tipos de células de camundongodeve ser menor que 30 μm. No entanto, em estágios posteriores, os cardiomiócitos no coração, neurônios no cérebro, células musculares nos membros, e algumas células de gordura podem ter um tamanho celular maior do que 30 μm. O tamanho da célula deve ser medido para esses tipos de células antes de iniciar os experimentos de célulaúnica.
As estratégias multiplexing fornecem uma maneira de analisar simultaneamente um grande número de amostras de forma econômica. Além disso, ao traçar o perfil de várias amostras juntas, podemos evitar significativamente os efeitos do lote e identificar dobradores celulares. Estas vantagens serão muito atrativas ao único campo de pilha. No entanto, existem alguns fatores que podem limitar seu uso. Como mais células são multiplexed em um único experimento, a relação de dobradura celular também irá aumentar. Embora esses dobradores possam ser identificados e removidos analisando os dados multiplexing de código de barras, isso levará a um grande desperdício de leituras de sequenciamento. Além disso, à medida que mais células são agrupadas, as células são mais fáceis de quebrar e causar um aumento do mRNA ambiente, que será capturado em gotículas com células e interferir com a sensibilidade à detecção. Esperamos que uma maior otimização do pipeline experimental de análise de fluxo de trabalho ou bioinformática resolva esses dois problemas em um futuro próximo.
Divulgações
Os autores não têm conflitos de interesse a serem divulgados.
Agradecimentos
Agradecemos a David M. Patterson e Christopher S. McGinnis do laboratório Dr. Zev J. Gartner por seu suprimento gentil dos reagentes de código de barras baseados em lipídios e sugestões sobre as etapas experimentais e análise de dados. Este trabalho foi fundado pelos Institutos Nacionais de Saúde (HL13347202).
Materiais
| Name | Company | Catalog Number | Comments |
| 10% Tween-20 | Bio-Rad | 1610781 | |
| 10x Chip Holder | 10x Genomics | 120252 330019 | |
| 10x Chromium Controller | 10x Genomics | 120223 | |
| 10x Magnetic Separator | 10x Genomics | 120250 230003 | |
| 10x Vortex Adapter | 10x Genomics | 330002, 120251 | |
| 10x Vortex Clip | 10x Genomics | 120253 230002 | |
| 4200 TapeStation System | Agilent | G2991AA | |
| Agilent High Sensitivity DNA Kit | Agilent | 5067-4626 | University of Pittsburgh Health Sciences Sequencing Core |
| Barcode Oligo | Integrated DNA Technologies | Single-stranded DNA | 25 nmol |
| Buffer EB | Qiagen | 19086 | |
| CD1 mice | Chales River | Strain Code 022 | ordered pregnant mice |
| Centrifuge 5424R | Appendorf | 2231000214 | |
| Chromium Chip B Single Cell Kit, 48 rxns | 10x Genomics | 1000073 | Store at ambient temperature |
| Chromium i7 Multiplex Kit, 96 rxns | 10x Genomics | 120262 | Store at -20 °C |
| Chromium Single Cell 3' GEM Kit v3,4 rxns | 10x Genomics | 1000094 | Store at -20 °C |
| Chromium Single Cell 3' Library Kit v3 | 10x Genomics | 1000095 | Store at -20 °C |
| Chromium Single Cell 3' v3 Gel Beads | 10x Genomics | 2000059 | Store at -80 °C |
| Collagenase A | Sigma/Millipore | 10103578001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| Collagenase B | Sigma/Millipore | 11088807001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| D1000 ScreenTape | Agilent | 5067-5582 | University of Pittsburgh Health Sciences Sequencing Core |
| DNA LoBind Tube Microcentrifuge Tube, 1.5 mL | Eppendorf | 022431021 | |
| DNA LoBind Tube Microcentrifuge Tube, 2.0 mL | Eppendorf | 022431048 | |
| Dynabeads MyOne SILANE | 10x Genomics | 2000048 | Store at 4 °C, used in Beads Cleanup Mix (Table 1) |
| DynaMag-2 Magnet | Theromo Scientific | 12321D | |
| Ethanol, Pure (200 Proof, anhydrous) | Sigma | E7023-500mL | |
| Falcon 15mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-70C | |
| Falcon 50mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-49A | |
| Fetal Bovine Serum, qualified, United States | Fisher Scientific | 26140079 | Store at -20 °C |
| Finnpipette F1 Multichannel Pipettes, 10-100μl | Theromo Scientific | 4661020N | |
| Finnpipette F1 Multichannel Pipettes, 1-10μl | Theromo Scientific | 4661000N | |
| Flowmi Cell Strainer | Sigma | BAH136800040 | Porosity 40 μm, for 1000 uL Pipette Tips, pack of 50 each |
| Glycerin (Glycerol), 50% (v/v) | Ricca Chemical Company | 3290-32 | |
| HBSS, no calcium, no magnesium | Thermo Fisher Scientific | 14170112 | |
| Human TruStain FcX (Fc Receptor Blocking Solution) | BioLegend | 422301 | Add 5 µl of Human TruStain FcX per million cells in 100 µl staining volume |
| Isopropanol (IPA) | Fisher Scientific | A464-4 | |
| Kapa HiFi HotStart ReadyMix (2X) | Fisher Scientific | NC0295239 | Store at -20 °C, used in Lipid-tagged barcode library mix (Table 1) |
| Lipid Barcode Primer (Multi-seq Primer) | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
| Low TE Buffer (10 mM Tris-HCl pH 8.0, 0.1 mM EDTA) | Thermo Fisher Scientific | 12090-015 | |
| MasterCycler Pro | Eppendorf | 950W | |
| Nuclease-Free Water (Ambion) | Thermo Fisher Scientific | AM9937 | |
| PCR Tubes 0.2 ml 8-tube strips | Eppendorf | 951010022 | |
| Phosphate-Buffered Saline (PBS) 1X without calcium & magnesium | Corning Cellgro | 21-040-CV | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin (alternative to Thermo Fisher product) | Sigma-Aldrich | SRE0036 | |
| Pipet 4-pack (0.1–2.5μL, 0.5-10μL, 10–100μL, 100–1,000μL variable-volume pipettes | Fisher Scientific | 05-403-151 | |
| Selection reagent (SPRIselect Reagent Kit) | Beckman Coulter | B23318 (60ml) | |
| Template Switch Oligo | 10x Genomics | 3000228 | Store at -20 °C, used in Master Mix (Table 1) |
| The antibody based barcoding strategy is also known as Cell Hashing | |||
| The cell browser is Loup Cell Browser | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/visualization/latest/what-is-loupe-cell-browser | |
| The commercial available analysis pipline in step 8.1 is Cell Ranger | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/what-is-cell-ranger | |
| The lipid based barcoding strategy is also known as MULTI-seq | |||
| The well maintained R platform is Seurat V3 | satijalab | https://satijalab.org/seurat/ | |
| TipOne RPT 0.1-10/20 ul XL ultra low retention filter pipet tip | USA Scientific | 1180-3710 | |
| TipOne RPT 1000 ul XL ultra low retention filter pipet tip | USA Scientific | 1182-1730 | |
| TipOne RPT 200 ul ultra low retention filter pipet tip | USA Scientific | 1180-8710 | |
| TotalSeq-A0301 anti-mouse Hashtag 1 Antibody | BioLegend | 155801 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 2 Antibody | BioLegend | 155803 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 3 Antibody | BioLegend | 155805 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TrueSeq RPI primer | Integrated DNA Technologies | Single-stranded DNA | 100 nmol, used in Lipid-tagged barcode library mix (Table 1) |
| Trypan Blue Solution, 0.4% | Fisher Scientific | 15250061 | |
| Trypsin-EDTA (0.25%), phenol red | Fisher Scientific | 25200-056 | |
| Universal I5 | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
Referências
- Raj, A., Van Den Bogaard, P., Rifkin, S. A., Van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877(2008).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6 (5), 377(2009).
- Li, G., Plonowska, K., Kuppusamy, R., Sturzu, A., Wu, S. M. Identification of cardiovascular lineage descendants at single-cell resolution. Development. 142 (5), 846-857 (2015).
- DeLaughter, D. M., et al. Single-cell resolution of temporal gene expression during heart development. Developmental Cell. 39 (4), 480-490 (2016).
- Li, G., et al. Single cell expression analysis reveals anatomical and cell cycle-dependent transcriptional shifts during heart development. Development. 146 (12), dev173476(2019).
- Meilhac, S. M., Buckingham, M. E. The deployment of cell lineages that form the mammalian heart. Nature Reviews Cardiology. 1, (2018).
- Liu, Z., et al. Single-cell transcriptomics reconstructs fate conversion from fibroblast to cardiomyocyte. Nature. 551 (7678), 100(2017).
- Lafzi, A., Moutinho, C., Picelli, S., Heyn, H. Tutorial: guidelines for the experimental design of single-cell RNA sequencing studies. Nature Protocols. 1, (2018).
- The Tabula Muris Consortium. Single-cell transcriptomics of 20 mouse organs creates a Tabula Muris. Nature. 562 (7727), 367(2018).
- Gawad, C., Koh, W., Quake, S. R. Single-cell genome sequencing: current state of the science. Nature Reviews Genetics. 17 (3), 175(2016).
- Grün, D., van Oudenaarden, A. Design and analysis of single-cell sequencing experiments. Cell. 163 (4), 799-810 (2015).
- Ziegenhain, C., et al. Comparative analysis of single-cell RNA sequencing methods. Molecular cell. 65 (4), 631-643 (2017).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17 (1), 77(2016).
- Islam, S., et al. Characterization of the single-cell transcriptional landscape by highly multiplex RNA-seq. Genome Research. 21 (7), 1160-1167 (2011).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161 (5), 1202-1214 (2015).
- Klein, A. M., et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 161 (5), 1187-1201 (2015).
- Library Prep -Single Cell Gene Expression -Official 10x Genomics Support. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/library-prep (2018).
- McGinnis, C. S., et al. MULTI-seq: sample multiplexing for single-cell RNA sequencing using lipid-tagged indices. Nature Methods. 1, (2019).
- Stoeckius, M., et al. Cell hashing with barcoded antibodies enables multiplexing and doublet detection for single cell genomics. Genome Biology. 19 (1), 224(2018).
- Chan, M. M., et al. Molecular recording of mammalian embryogenesis. Nature. 570 (7759), 77-82 (2019).
- Agilent 4200 TapeStation System. , Agilent. https://www.agilent.com/cs/library/datasheets/public/5991-6029EN.pdf (2019).
- SPRIselect User Guide. , Beckman Coulter. https://research.fhcrc.org/content/dam/stripe/hahn/methods/mol_biol/SPRIselect%20User%20Guide.pdf (2012).
- Qubit 4 Fluorometer User Guide. , Thermo Fisher Scientific. https://assets.thermofisher.com/TFS-Assets/LSG/manuals/MAN0017209_Qubit_4_Fluorometer_UG.pdf (2018).
- Agilent High Sensitivity DNA Kit Guide. , Agilent. https://www.agilent.com/cs/library/usermanuals/public/High%20Sensitivity_DNA_KG.pdf (2016).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411(2018).
- Weber, R. J., Liang, S. I., Selden, N. S., Desai, T. A., Gartner, Z. J. Efficient targeting of fatty-acid modified oligonucleotides to live cell membranes through stepwise assembly. Biomacromolecules. 15 (12), 4621-4626 (2014).
- Single Cell Protocols Cell Preparation Guide. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/sample-prep (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados