Method Article
Analisi del sequenziamento dell'mRNA a cella singola multiplexed delle cellule embrionali del topo
In questo articolo
Riepilogo
Qui abbiamo presentato un metodo di sequenziamento dell'mRNA a singola cellula multiplexed per profilare l'espressione genica nei tessuti embrionali del topo. Il metodo di sequenziamento dell'mRNA a singola cellula (scRNA-Seq) basato su droplet in combinazione con strategie di multiplexing può profilare singole cellule da più campioni contemporaneamente, riducendo significativamente i costi del reagente e riducendo al minimo gli effetti batch sperimentali.
Abstract
Il sequenziamento di mRNA a singola cellula ha compiuto progressi significativi negli ultimi anni ed è diventato uno strumento importante nel campo della biologia dello sviluppo. È stato utilizzato con successo per identificare popolazioni di cellule rare, scoprire nuovi geni marcatori e decodificare informazioni spaziali e temporali sullo sviluppo. Il metodo a singola cellula si è evoluto anche dalla tecnologia Fluidigm C1 basata microfluidica alle soluzioni a base di gocciolamento negli ultimi due o tre anni. Qui abbiamo usato il cuore come esempio per dimostrare come profilare le cellule del tessuto embrionale del topo usando il metodo scRNA-Seq basato su gocciolamento. Inoltre, abbiamo integrato due strategie nel flusso di lavoro per profilare più campioni in un unico esperimento. Utilizzando uno dei metodi integrati, abbiamo contemporaneamente profilato più di 9.000 cellule da otto campioni di cuore. Questi metodi saranno preziosi per il campo della biologia dello sviluppo fornendo un modo conveniente per profilare contemporaneamente singole cellule provenienti da diversi background genetici, stadi di sviluppo o posizioni anatomiche.
Introduzione
Il profilo trascrizionale di ogni singola cellula varia tra le popolazioni cellulari durante lo sviluppo embrionale. Sebbene l'ibridazione molecolare singola in situ possa essere utilizzata per visualizzare l'espressione di un piccolo numero di geni1, il sequenziamento dell'mRNA a singola cellula (scRNA-Seq) fornisce un approccio imparziale per illustrare i modelli di espressione dei geni a livello di genoma nei singoli geni. Dopo che è stato pubblicato per la prima volta nel 20092, scRNA-Seq è stato applicato per studiare più tessuti in più fasi di sviluppo negli ultimi anni3,4,5. Inoltre, poiché l'atlante delle cellule umane ha recentemente lanciato i suoi progetti incentrati sullo sviluppo, si prevede che nel prossimo futuro saranno generati più dati di cellule singole provenienti da tessuti embrionali umani.
Il cuore come primo organo a svilupparsi svolge un ruolo critico nello sviluppo embrionale. Il cuore è costituito da più tipi di cellule e lo sviluppo di ogni tipo di cellula è strettamente regolato temporalmente e spazialemente. Negli ultimi anni, l'origine e il lignaggio cellulare delle cellule cardiache nelle fasi iniziali dello sviluppo sono state caratterizzate6, che forniscono un enorme strumento di navigazione utile per comprendere la patogenesi congenita delle malattie cardiache, nonché per sviluppare metodi più tecnologicamente avanzati per stimolare la rigenerazione cardiomiocita7.
Lo scRNA-Seq ha subito una rapida espansione negli ultimi anni8,9,10. Con i metodi appena sviluppati, la progettazione e l'analisi di esperimenti a cella singola è diventato più realizzabile11,12,13,14. Il metodo qui presentato è una procedura commerciale basata sulle soluzioni di gocciolina (vedi Tabella dei materiali)15,16. Questo metodo è dotato di catturare celle e insiemi di perline codificate in modo univoco in una goccia di emulsione olio-acqua sotto il controllo di un sistema di controllo microfluidico. La velocità di caricamento delle celle nelle goccioline è estremamente bassa in modo che la maggior parte delle emulsioni delle goccioline contengano una sola cella17. L'ingegnoso design della procedura deriva dalla separazione di una singola cellula in emulsioni di goccioline che si verificano contemporaneamente con la codifica a barre, che consente l'analisi parallela di singole cellule utilizzando RNA-Seq su una popolazione eterogenea.
L'incorporazione di strategie di multiplexing è una delle aggiunte importanti al tradizionale flusso di lavoro a cella singola13,14. Questa aggiunta è molto utile per eliminare i doppietti cellulari, ridurre i costi sperimentali ed eliminare gli effetti di lotto18,19. Una strategia di codifica a barre basata su lipidi e una strategia di codifica a barre basata su anticorpi (vedi Tabella dei materiali) sono i due metodi di multiplexing per lo più utilizzati. I codici a barre specifici vengono utilizzati per etichettare ogni campione in entrambi i metodi e i campioni etichettati vengono quindi miscelati per l'acquisizione di una singola cella, la preparazione della libreria e la sequenza. Successivamente, i dati di sequenza in pool possono essere separati analizzando le sequenze di codici a barre (Figura 1)19. Tuttavia, esistono differenze significative tra i due metodi. La strategia di codifica a barre basata su lipidi si basa su oligonucleotidi modificati ai lipidi, che non è stato trovato per avere preferenze di tipo di cellula. Mentre la strategia di codifica a barre basata su anticorpi può rilevare solo le cellule che esprimono le proteine dell'antigene19,20. Inoltre, ci vogliono circa 10 min per macchiare i lipidi ma 40 min per macchiare gli anticorpi (Figura 1). Inoltre, gli oligonucleotidi modificati dai lipidi sono più economici degli oligonucleotidi coniugati da anticorpi, ma non disponibili in commercio al momento della scrittura di questo articolo. Infine, la strategia basata sui lipidi può multiplex 96 campioni in un esperimento, ma la strategia basata su anticorpi attualmente può essere solo multiplex 12 campioni.
Il numero di cellule raccomandato al multiplex in un singolo esperimento dovrebbe essere inferiore a 2,5 x 104,altrimenti porterà a un'alta percentuale di doppietti cellulari e a una potenziale contaminazione da mRNA ambientali. Attraverso le strategie di multiplexing, il costo dell'acquisizione di una singola cella, della generazione di cDNA e della preparazione della libreria per più campioni sarà ridotto al costo di un campione, ma il costo di sequenziamento rimarrà lo stesso.
Protocollo
La procedura animale è in accordo con l'Università di Pittsburgh Institutional Animal Care and Use Committee (IACUC).
1. Dissezione del cuore embrionale del topo e preparazione della sospensione a cella singola
NOTA: Questo passaggio potrebbe richiedere alcune ore a seconda del numero di embrioni da sezionare.
- Per acquisire cuori embrionali E18.5, eutanasia un topo CD1 incinta dalla somministrazione di CO2. Utilizzare un rasoio per rimuovere i capelli indesiderati nella zona addominale e disinfettare la pelle con il 70% di etanolo.
- Tagliare la pelle dell'addome utilizzando le forbici sterilizzate e sezionare attentamente gli embrioni e metterli rapidamente in salina (PBS) con tamponadi di fosfato freddo (PBS) sul ghiaccio.
- Isolare i cuori da ogni singolo embrione con attenzione in un piatto di 10 cm riempito con PBS freddo sotto un microscopio stereoscopico utilizzando pinze e forbici.
NOTA: Mantenere intatte le quattro camere sezionando il polmone con il cuore e NON prendere/tirare direttamente il cuore con strumenti chirurgici. - Trasferisci 10 cuori in un nuovo piatto di 10 cm pieno di PBS freddo e micro-dissenetta i cuori in atrio sinistro, atrio destro, ventricolo sinistro e ventricolo destro.
NOTA: Si presuppone che questo produca più di 1 x 106 celle da ogni campione. Si consiglia di iniziare con almeno 1 x 105 celle per campione. - Trasferire ciascuno dei tessuti a 4 camere in un tubo da 1,5 mL e tagliarli a pezzi con le forbici. Centrifuga a 300 x g per 3 min per raccogliere i tessuti.
- Dopo aver aspirato il supernatante, aggiungere 1 mL di 0,25% Trippsin/EDTA ad ogni tubo e incubare in un bagno d'acqua 37 C per 10 minuti. Pipet su e giù delicatamente 7-8 volte utilizzando una pipetta P1000.
- Se lo stadio embrionale è più vecchio di E11.5, aggiungere 1 mL di 10 mg/mL di miscela A/B collagenae mg/mL e incubare a 37 gradi centigradi per 10-20 min. Delicatamente pipet su e giù fino a quando la maggior parte delle cellule sono dissociate.
- Trasferire le cellule in un tubo da 15 mL e aggiungere la soluzione di sale bilanciato (HBSS) di 8 mL per diluire gli enzimi. Girare verso il basso le celle a 300 x g per 5 min. Sospendere le cellule in 1 mL di PBS e trasferirle in un tubo da 1,5 mL. Filtrare le cellule attraverso un colino cellulare da 40 m.
- Prendere 15 l. di volume da ogni campione e mescolare con la stessa quantità di 0,04% di blu trypan. Caricare questo su una camera di conteggio delle celle e contare le celle in un contatore di celle.
NOTA: per generare risultati di alta qualità, si consiglia di ottenere una redditività cellulare superiore al 95%.
2. Codice a barre multiplexing a cella singola
NOTA: Questo passaggio richiede almeno 40 min che varia in base al numero di campioni elaborati. Per le fasi di pre-amplificazione (passaggio da 2.11 a 3.11) è necessaria un'area di banco pulita e un'area di panca pulita separata per le fasi di post-amplificazione (i passaggi successivi alla 3.11).
-
Procedura di codifica a barre basata su lipidi (procedura opzionale 1)
- In base alla concentrazione cellulare, mantenere meno di 5 x 105 cellule per campione. Assicurarsi che la sospensione cellulare sia priva di detriti e aggregati cellulari.
- Preparare 2 soluzioni di stock di ancoraggio/codice a barre m e una soluzione di stock di co-ancoraper di 2 M per ogni campione (Tabella 1).
NOTA: L'ancora e il co-ancorasono stati gentilmente dotati dal laboratorio del Dr. J. Gartner. Per sintetizzare queste oligonucleotidi modificate dai lipidi, le sequenze di DNA sono state coniugate con acido grasso su un supporto solido e purificate dalla cromatografia liquida ad alte prestazioni in fase invertita (HPLC)18,19. - Lavare le cellule due volte con PBS e raccogliere le cellule a 300 x g per 5 min. Sospendi le celle in 180 - L di PBS.
- Aggiungere 20 l di ancora/codice a barre e pipetta su e giù delicatamente per mescolare. Incubare sul ghiaccio per 5 min.
- Aggiungere 20 -L di co-anchor stock solution e pipetta su e giù delicatamente per mescolare, quindi incubare sul ghiaccio per altri 5 minuti.
- Aggiungere 1 mL di PBS freddo con 1% di BSA e centrifugare a 300 x g per 5 min a 4 gradi centigradi. Lavare almeno altre 2 volte con ghiaccio freddo 1% BSA in PBS.
- Unire tutti i campioni insieme e filtrare attraverso 40 ceppi cellulari m. Contare le celle e mantenere la sospensione cellulare sul ghiaccio da utilizzare nella sezione 3.
-
Procedura di codifica a barre basata su anticorpi (procedura opzionale 2)
- Centrifuga 1 x 106-2 x 106 celle per ogni campione (dal punto 1,8) a 300 x g per 5 min e sospenderle in 100 l of coloring buffer (Tabella 1) in tubi a basso legame di 1,5 mL.
- Aggiungete 10 di ragnoagenti per l'FC e incubate per 10 min a 4 gradi centigradi.
- Preparare gli anticorpi (vedi Tabella dei materiali)centrifugando a 14.000 x g per 10 min a 2-8 gradi centigradi.
- Aggiungete 1 g di ogni anticorpo coniugato all'oligo a 50 gradi di tampone di colorazione cellulare per creare la soluzione di colorazione degli anticorpi20. Aggiungere una soluzione di colorazione anticorpale ad ogni tubo campione. Incubare per 30 min a 4 gradi centigradi.
- Lavare le cellule 3 volte con 1 mL di PBS, ruotare per 5 min a 350 x g a 4 gradi centigradi.
- Raggruppare tutti i campioni alle proporzioni desiderate in 1 mL di buffer di colorazione, girare per 5 min a 350 x g a 4 gradi centigradi.
- Risospendere le cellule in PBS a una concentrazione appropriata (fino a 1.500 cellule/L) e filtrare le cellule attraverso un colino cellulare di 40 m. Procedere immediatamente al passaggio successivo.
3. Generazione di gocciolini e trascrizione inversa dell'mRNA
NOTA: Questo passaggio richiede circa 90 min per una reazione multiplexata.
- E' l'ACquienza (vedi Tabella dei materiali)a temperatura ambiente per 30 min. Estrarre i reagenti dal kit di perline di gel in emulsione (DGM) (vedere Tabella dei materiali) e tenerli alla loro temperatura indicata.
- Assemblare il chip B in un supporto di truciolo (vedere Tabella dei materiali).
- Distribuisce 75 ldi del 50% di lecerolo nei pozzi inutilizzati nella fila 1; 40 -L nella fila 2; 280 -L nella riga 3. Non aggiungere glicerolo in eventuali pozzi di recupero sulla fila superiore del chip.
- Preparare il master mix sul ghiaccio secondo la Tabella 1. Aggiungere il volume appropriato di sospensione cellulare e acqua priva di nuclesino per master mix secondo una tabella calcolatrice volume sospensione cella17 e pipette delicatamente il mix. Distribuisci 75 l di miscela cellulare nel centro inferiore del campione bene nella fila 1 senza introdurre bolle.
- Vorticare le perline di gel per 30 s utilizzando un adattatore vortice e lentamente erogare 40 gradi di perline di gel nel centro inferiore del tallone di gel ben nella fila 2 senza introdurre bolle.
NOTA: È fondamentale attendere 30 s tra l'aggiunta di cellule e perline di gel per evitare il fallimento dell'umidità. - Per il partizionamento bene dell'olio nella riga 3, erogare 280 l di partizionamento dell'olio attraverso la parete laterale del pozzo.
NOTA: il caricamento di meno di 270 unità di partizionamento dell'olio porterà alla generazione anomala di GePM. - Fissare la guarnizione sul chip, non premere verso il basso sulla guarnizione e tenerlo orizzontale per evitare di bagnare la guarnizione.
- Caricare il chip assemblato con la guarnizione nel controller cromo ed eseguire il programma a cella singola di cromo B (vedere Tabella dei materiali), procedere immediatamente al passaggio successivo al termine del programma.
- Estrarre il chip e scartare la guarnizione. Piegare il coperchio indietro per esporre i pozze a 45 gradi, controllare il livello del liquido per assicurarsi che non siano presenti zoccoli.
- Aspirano lentamente 100 L di GEM dai punti più bassi del pozzo di recupero e controllano l'uniformità dei GEM. Distribuisci i GEM in un nuovo tubo di reazione a catena di polimerasi (PCR) sul ghiaccio con le punte pipette contro la parete laterale del tubo.
NOTA: se si osserva un livello acquoso in eccesso, si consiglia di preparare nuovamente i campioni. È importante sottolineare che scattare una foto della miscela quando i GEM sono ancora nelle punte pipette. Questa immagine può capire se c'è un guasto di bagnatura, GEM parzialmente emulsionati e intasamenti reagenti. La fotografia può anche essere utilizzata come prova per ottenere il rimborso dalla società di reagenti con reagenti e chip sostitutivi. - Mettere il tubo in un ciclore termico ed eseguire la procedura di trascrizione inversa (Tabella 2).
NOTA: Fermatevi qui o procedete al passaggio successivo. Il prodotto PCR può essere immagazzinato a -20 gradi centigradi per un massimo di una settimana.
4. Amplificazione cDNA
NOTA: Questo passaggio richiede circa 150 min.
- Pulizia della trascrizione inversa di una singola cella
- Estrarre i reagenti di amplificazione cDNA dal kit GEM (vedere Tabella dei materiali)e tenerli alla temperatura indicata.
- Aggiungere 125 l di agente di recupero al campione a temperatura ambiente per acquisire una miscela bifasica. Nessun liquido opaco deve essere osservato ed evitare pipettaggio o vortice la miscela.
- Dopo aver atteso 60 s, rimuovere lentamente 125 l dell'agente di recupero dal fondo del tubo.
- Vorticare le perline magnetiche (vedi Tabella dei materiali) accuratamente per 30 s e immediatamente usarlo per preparare la miscela di pulizia delle perline ( Tabella1). I reagenti devono essere aggiunti in sequenza come elencato.
- Vorticare la miscela di pulizia delle perline e aggiungere 200 l al campione. Pipette la miscela 10 volte poi incubare per 10 min a temperatura ambiente.
- Aggiungere i reagenti in sequenza come indicato nella Tabella 1 per preparare la soluzione di eluizione ed eluire il cDNA come segue.
- Posizionare i campioni sul magnete (alta posizione) (vedere Tabella dei materiali) fino a quando la soluzione non cancella, quindi rimuovere il super-natante.
- Aggiungere al pellet 200 l dell'80%. Attendere 30 s, quindi rimuovere l'etanolo.
- Ripetere il passaggio 4.1.6.2 per altre 2 volte. Centrifuga brevemente e posizionare sul magnete (posizione bassa). Rimuovere con cura l'etanolo rimanente e l'aria asciutta per meno di 2 min.
NOTA: NON superare l'essiccazione dell'aria oltre i 2 min, altrimenti l'efficienza di eluizione diminuirà. - Rimuovere il campione dal magnete. Aggiungere 35,5 litri di soluzione di eluizione e pipetta per mescolare 15 volte. Incubare 2 min a temperatura ambiente.
- Posizionare il campione sul magnete (posizione elevata) fino a quando la soluzione non si schiarisce. Trasferire 35 -L del campione su una nuova striscia di tubi.
NOTA: questa procedura di purificazione viene utilizzata anche nei passaggi 4.2.1.6, 4.2.2.3, 5.8, 6.1.10 e 6.2.6. Prestare attenzione alla concentrazione di perline magnetiche e al volume di acqua tampone/acqua ultrapura EB utilizzata per eluire i campioni ad ogni passo.
- Quantificare le dimensioni, la concentrazione e l'integrità del cDNA eluito utilizzando uno strumento automatizzato di elettroforesi21 (vedi Tabella dei materiali)(Figura 2).
- Amplificazione del cDNA
-
amplificazione cDNA utilizzando la strategia di codifica a barre basata su lipidi (procedura opzionale 1)
- Preparare sulla ghiaccio la miscela di reazione all'amplificazione (Tabella 1).
- Aggiungere la miscela di reazione di amplificazione a 35 gradi centigradi di campioni di cDNA (dal punto 4.1.6.7). Pipette il mix, e centrifugare brevemente. Incubare la miscela in un ciclista termico seguendo la procedura di amplificazione cDNA(tabella 2).
- Dopo aver vortice accuratamente, aggiungere 120 l di reagente selezionato e 100 L di acqua ultrapura a 100 l di campione per acquisire una concentrazione di 0,6x di reagente selezionato (vedere Tabella dei materiali). Pipette la miscela per 15 volte.
- Incubare per 5 min a temperatura ambiente e quindi posizionare il campione sul magnete fino a quando le soluzioni diventano chiare.
NOTA: CDNA endogeno nella frazione di perline e cDNA con codice a barre multiplexing è nel supernatante. - Trasferire il supernatante in un tubo di legame basso da 1,5 mL per la costruzione della libreria cDNA con codice a barre multiplexing al punto 6.1.
- Pulire il cDNA endogeno seguendo i passaggi in 4.1.6 ed eluirli con 40 gradi di buffer EB.
- Eseguire 1 l di campione di cDNA purificato su uno strumento di elettroforesi automatizzato (vedi Tabella dei materiali) per analizzare/quantificare il cDNA.
- Aliquota 10 L di cDNA in un nuovo tubo PCR per la costruzione endogena della biblioteca.
NOTA: Fermatevi qui o procedete al passaggio successivo. Il campione rimanente può essere conservato in -20 gradi centigradi per un massimo di 4 settimane per generare librerie aggiuntive, se necessario.
-
amplificazione cDNA nella strategia di codifica a barre basata su anticorpi (procedura opzionale 2)
- Aggiungete 2 pmol di HTO e ADT additive primer e 15 - L di primer cDNA a 50 gradi di miscela di reazione di amplificazione (Tabella 1) ed eseguire l'amplificazione cDNA con la procedura di amplificazione cDNA (Tabella 2).
- Utilizzare 0.6x select reagent to separate endogeno cDNA (perline fraction) e multiplexing codice a barre cDNA (in supernatant). Ricordarsi di salvare il supernatante per eseguire la costruzione della libreria di codice a barre nel passaggio 6.2.
- Purificare ed elusire il cDNA trascrizione endogeno seguendo i passaggi in 4.1.6, eseguire il controllo di qualità (QC) delle librerie aliquote e 10 L in un nuovo tubo PCR per la costruzione endogena della biblioteca.
-
amplificazione cDNA utilizzando la strategia di codifica a barre basata su lipidi (procedura opzionale 1)
5. Preparazione della libreria di trascrizioni endogeni
NOTA: Questo passaggio richiede circa 120 min.
- Mantenere i reagenti di costruzione della biblioteca di espressione genica dal kit di biblioteca (vedere Tabella dei materiali) rispettivamente alla loro temperatura indicata.
- Preparare la miscela di frammentazione (Tabella 1) su ghiaccio, pipetta per mescolare e centrifugare brevemente.
- Aggiungere il buffer EB da 25 L al campione di cDNA purificato da 10 l (dal passo 4.2.1.8 o 4.2.2.3) e quindi aggiungere brevemente la miscela di frammentazione di 15 gradi, pipettare la miscela 15 volte sul ghiaccio e centrifugare brevemente.
- Trasferire il campione in un ciclore termico pre-raffreddato e avviare il programma PCR per la frammentazione, la riparazione finale e la coda A (Tabella 2).
- Vortex il reagente selezionare per sospendere perline magnetiche e successivamente utilizzare 0.6x e 0.8x selezionare reagenti per effettuare una selezione di dimensioni fronte-retro secondo la guida utente17,22. Utilizzare 50 l di tampone EB per eluire il DNA.
- Preparare la miscela di legatura dell'adattatore (Tabella 1), quindi pipettare accuratamente la miscela e centrifugare brevemente.
- Aggiungete 50 - L di miscela di ligazione dell'adattatore a 50 gradi di campione, mescolate di nuovo pipetta per 15 volte e centrifugate brevemente. Eseguire la legatura dell'adattatore in base al protocollo in un ciclore termico (Tabella 2).
- Utilizzare 0,8x selezionare il reagente per purificare il prodotto di legatura ed eluire il campione purificato con 30 l di buffer EB (vedere il passaggio 4.1.6).
- Preparare l'indice del campione miscela PCR (tabella 1) e aggiungere al campione purificato 60 . Aggiungete al campione 10 - L di indice del campione, pipette mescolano su e giù per 5 volte e centrifugano brevemente, incubare in un ciclore termico seguendo il protocollo dell'indice campione (Tabella 2).
NOTA: Fermatevi qui o procedete al passaggio successivo. Se viene utilizzato più pozzi, scegliere un indice di esempio specifico (vedere Tabella dei materiali) per ogni bene. Ricordarsi di registrare l'ID indice utilizzato per ogni bene e non assicurarsi alcuna sovrapposizione in un'esecuzione di sequenziamento multiplex. - Successivamente utilizzare reagenti di selezione 0,6x e 0,8x per effettuare una selezione a doppio lato delle dimensioni per acquisire 35 - L di libreria cellulare endogena purificata DNA17.
- QC la libreria cellulare endogena prima del sequenziamento (Figura 2).
6. Preparazione delle librerie cDNA del codice a barre di esempio multiplexing
NOTA: questo passaggio richiede almeno 120 min.
-
Generazione di libreria di codici a barre di esempio per la strategia di multiplexing basata su lipidi (procedura opzionale 1)
- Aggiungere 520 l di reagente selezionato e 360 l di isopropanol per campionare il cDNA del codice a barre dal punto 4.2.1.5 per ottenere una concentrazione di reagente a selezione 3,2x. Pipette la miscela 10 volte e incubare a temperatura ambiente per 5 min.
- Posizionare il tubo su un rack magnetico e attendere che la soluzione si schiarisca. Poi scartare il super-natante.
- Utilizzare 500 l dell'80% di etanolo per lavare le perline due volte su un magnete e attendere 30 s dopo ogni lavaggio.
- Centrifugare brevemente le perline e posizionare su un magnete. Rimuovere l'etanolo rimanente con una micropipetta P10 e lasciare perline per 2 min.
- Rimuovere il tubo dal rack magnete e respendere le perline in 50 . Pipet su e giù per mescolare accuratamente. Incubare a temperatura ambiente per 2 min.
- Riportare il tubo su un magnete e attendere che la soluzione si schiarisca. Trasferire il supernatante (codice a barre campione cDNA) in un nuovo tubo PCR. Fare attenzione a non trasferire le perline.
- Quantificare la concentrazione del codice a barre campione cDNA23.
- Preparare la miscela della libreria di codici a barre lipidi (Tabella 1),aggiungere 3,5 ng di cDNA con codice a barre purificato (dal punto 6.1.6) e acqua priva di nuclea per un volume totale di 50 gradi centigradi.
- Conservarlo in un termociclore seguendo la libreria di codici a barre a base di lipidi PCR (Tabella 2).
- Utilizzare 1,6x selezionare il reagente per purificare il prodotto PCR ed eluire il DNA con 25 l di buffer EB (vedere il passaggio 4.1.6).
- Quantificare la concentrazione della biblioteca utilizzando un metodo di analisi del DNA ad alta sensibilità24 dalla diluizione iniziale di 1:5 (Figura 2).
-
Generazione di libreria di codici a barre di esempio per la strategia di multiplexing basata su anticorpi (procedura opzionale 2)
- Aggiungere ulteriore volume di reazione 1.4x del reagente selezionato al super-natante contenente codici a barre campione acquisiti dal passaggio 4.2.2.3 per ottenere un rapporto di reagente selezionato 2x.
- Lavare le perline con l'80% di etanolo seguendo i passaggi in 4.1.6 ed eluire il cDNA con codice a barre con acqua ultrapura.
- Eseguire il protocollo di selezione con 2x select reagente per la seconda volta ed elusio utilizzando acqua ultrapura.
- Preparare la miscela della libreria di codici a barre dell'anticorpo (Tabella 1) e aggiungere 45 l di cDNA con codice a barre purificato dall'ultimo passaggio.
- Incubare in un cilindro termico seguendo la libreria di codici a barre anticorpali PCR (Tabella 2)20.
- Utilizzare 1,6x selezionare il reagente per purificare il prodotto PCR ed eluire il campione purificato con 30 gradi di acqua ultrapura (vedere il passaggio 4.1.6).
7. Sequenziamento delle librerie
NOTA: è possibile utilizzare più piattaforme di sequenziamento di nuova generazione come HiSeq 4000 e NovaSeq per sequenziare le librerie di trascrizioni endogene e le librerie di codici a barre multiplexing.
- Utilizzare una piattaforma di sequenziamento di nuova generazione scelta per sequenziare le librerie di trascrizioni endogene e le librerie di codici a barre multiplexing.
- Diluire le biblioteche in base alle raccomandazioni di un esperto in una società di sequenziamento o di un impianto di sequenziamento. Per la libreria di trascrizioni endogena e 3000 per le librerie di codici a barre sono consigliate minime 20.000 letture per cella e 3000 letture.
8. Analisi dei dati
NOTA: de-multiplex i dati di sequenziamento utilizzando la risorsa basata su cloud BaseSpace o eseguendo il pacchetto bcl2fastq su un server UNIX.
-
Analisi endogena dei dati del trascrittoma
- Con i dati fastq generati dal software di demultiplexing, eseguire "mkfastq" sulla pipeline di analisi dei dati disponibile in commercio (vedere Tabella dei materiali) per demultiplex ulteriormente ogni codice a barre GEM.
- Eseguire "count" per eseguire l'allineamento, il filtro, il conteggio dei codici a barre e il conteggio UMI.
- Facoltativamente, eseguire "aggr" per aggregare più corsie di sequenza da un singolo esperimento.
- Utilizzare il "browser cellulare" (vedere La tabella dei materiali) per visualizzare dati, raggruppare le cellule, identificare geni espressi in modo differenziale e generare grafici della mappa termica tSNE o dell'espressione genica.
- Facoltativamente, utilizzare una piattaforma basata su R ben gestita25 (vedere Tabella dei materiali) per normalizzare e scalare i dati, identificare geni espressi in modo differenziale e generare grafici tSNE/UMAP e mappe di calore dell'espressione genica ( Figura3).
-
Analisi dei dati dei codici a barre multiplexing
-
Analisi dei dati della strategia di codifica a barre basata su lipidi
- Utilizzare la pipeline di analisi dei dati disponibile in commercio o il pacchetto deMULTIplex R (https://github.com/chris-mcginnis-ucsf/MULTI-seq) per convertire i file FASTQ del codice a barre di esempio in una matrice di conteggio UMI del codice a barre di esempio.
- Caricare la matrice di conteggio del codice a barre UMI con dati transcriptome endogeni in una piattaforma basata su R (vedere Tabella dei materiali) per l'analisi dell'integrazione ( Figura3).
-
Analisi dei dati della strategia di codifica a barre basata su anticorpi
- Utilizzare "count" dalla pipeline di analisi dei dati disponibile in commercio per mappare i codici a barre fornendo il file CSV della libreria e il file CSV di riferimento della funzione hashtag.
- Caricare la matrice di codici a barre delle feature unificate di output, che contiene i conteggi delle espressioni geniche insieme ai conteggi dei codici a barre delle entità geografiche per ogni codice a barre della cella, in una piattaforma basata su R per l'analisi a valle.
-
Analisi dei dati della strategia di codifica a barre basata su lipidi
Risultati
In questo studio, abbiamo usato il cuore embrionale del topo come esempio per mostrare come è stato eseguito il sequenziamento di mRNA a singola cellula multiplexed per elaborare simultaneamente i diversi campioni da parti separate di un organo. I cuori del topo E18.5 CD1 sono stati isolati e sezionati nell'atrio sinistro (LA), nell'atrio destro (RA), nel ventricolo sinistro (LV) e nel ventricolo destro (RV). Le cellule atriale e ventricolalare sono state poi codificate a barre in modo indipendente utilizzando una procedura di codifica a barre a base di lipidi e mescolate insieme prima della generazione e della trascrizione inversa dei GEM. La panoramica dello schema è illustrata nella Figura 1. Abbiamo quantificato la concentrazione di cDNA prima della costruzione della biblioteca (Figura 2A). Una delle distinzioni nell'esecuzione di scRNA-Seq multiplexed dallo standard scRNA-Seq è che la libreria cDNA endogena e la libreria cDNA del codice a barre di esempio sono state acquisite separatamente dopo l'amplificazione e la purificazione del cDNA (Passaggio 4.2.1 e 4.2.2.2). Le due librerie sono state qualificate anche nell'esperimento (Figura 2B,C). Il sequenziamento di nuova generazione e l'analisi dei dati sono stati eseguiti, seguiti dalla costruzione della libreria e dal QC.
Abbiamo usato la piattaforma HiSeqX per sequenziare entrambe le librerie nella stessa corsia di sequenziamento. Con i dati di sequenziamento, abbiamo prima separato i dati endogeni della trascrizione e i dati dei codici a barre utilizzando il programma BaseSpace. Poi abbiamo analizzato l'espressione del codice a barre in ogni singola cella e abbiamo trovato 8 gruppi di celle singole che esprimono in modo univoco un tipo di codice a barre, che rappresenta celle da 8 campioni diversi (Figura 3A). Inoltre, abbiamo anche scoperto che alcune celle non esprimono alcun codice a barre, che abbiamo definito come celle negative, e alcune celle esprimono due diversi codici a barre, che rappresentano doppie(Figura 3B). In sintesi, abbiamo scoperto che circa il 70% delle cellule sono singlet, il 25% delle cellule sono negative e il 5% delle cellule sono doppietti.
Con le cellule singlet, possiamo eseguire ulteriori analisi a valle per comprendere l'eterogeneità cellulare e le normative molecolari. Le analisi potenziali possono essere l'annotazione di tipo cella (Figura 4A), l'identificazione del tipo di cella novel/rara (Figura 4B), l'analisi comparativa della zona anatomica (Figura 4C) e l'analisi del percorso di ontologia genica, ad esempio le separazioni delle fasi del ciclo cellulare (Figura 4D).

Figura 1: Flusso di lavoro di sequenziamento dell'mRNA a singola cellula multiplexed. I cuori di stadio 18.5 giorno embrionale sono stati analizzati utilizzando una procedura di sequenziamento a singola cellula a base di gocciolamento multiplexed. F - trascrizione inversa. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: risultati del QC rappresentativo in diversi passaggi. (A) Analisi QC del cDNA dal punto 4.1.7. La dimensione del frammento di destinazione è da 200 a 9000 bp. (B) libreria endogena e (C) libreria di codici a barre sono stati analizzati con uno strumento di elettroforesi automatizzato. La dimensione del frammento di destinazione per la libreria endogena è 300-600 bp, e la dimensione del DNA della libreria di codici a barre è di circa 172 bp.
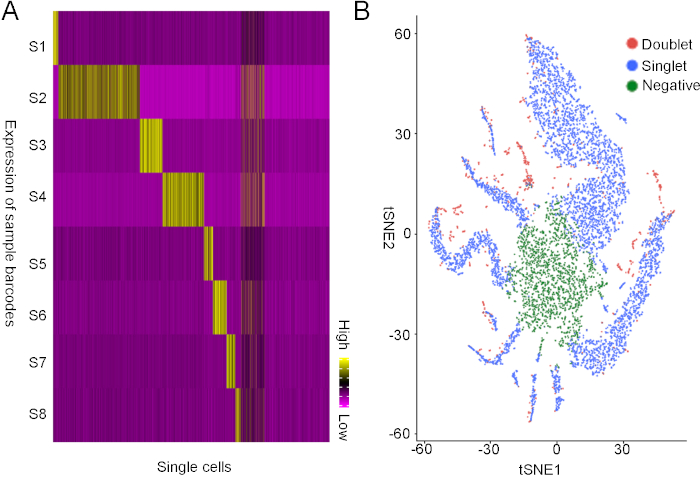
Figura 3: Demultiplexing dei dati di sequenza dalla strategia di codifica a barre basata su lipidi. (A) Analisi senza supervisione dell'espressione del codice a barre. L'asse X rappresenta singole celle, quando l'asse y rappresenta i codici a barre. Ognuna delle 8 popolazioni di cellule singole è stata identificata per esprimere in modo univoco uno degli 8 codici a barre. Si noti che alcune celle esprimono più di un codice a barre e alcune celle non esprimono alcun codice a barre. (B) t-SNE stampa delle celle singlet, delle celle doppie e delle celle negative. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Analisi avanzata dei dati trascrizionali a cella singola. (A-D) I dati a cella singola possono essere analizzati in modi diversi per comprendere l'eterogeneità cellulare e i percorsi molecolari. Abbiamo elencato diverse applicazioni qui come esempi. Sono state caricate singole celle in un pacchetto R per identificare i tipi di cellule (A), le popolazioni di cellule rare(B),le zone anatomiche delle cellule (C) e le fasi del ciclo cellulare (D). Fare clic qui per visualizzare una versione più grande di questa figura.
| Nome miscela | Composizione |
| Miscela di collagenasi | 10 mg/mL collagenase A e 10 mg/mL collagenasi B, disciolto in HBSS, con 40% FBS. |
| 2 soluzione di stock di ancoraggio/codice a barre M | Mescolare 50 - ancoraggio M e 10 m filo di codice a barre in rapporto 1:1 molare in PBS (senza FBS o BSA) per un volume totale di 25 . |
| 2 soluzione di co-ancorare | Diluire 1 -L 50 M Co-Anchor con 24 PBS (senza FBS o BSA). |
| Buffer di colorazione | PBS contenente il 2% di BSA, 0,01% Tween 20 |
| Miscela Master | 20 -L Reagente, 3,1 Oligo L, 2 -L- Agente di riduzione B, 8,3 L RT Enzima C. |
| Miscela di pulizia perline | 182 - Buffer di pulitura ll, 8 - Reagente di selezione L, 5 - Agente di riduzione del ll B, 5 -L di acqua senza Nucleasi. |
| Miscela di reazione all'amplificazione | 1 luna di 10 M di primer additivo con etichettatura a itudino, 15 cDNA primer, 50 -L Amp Mix |
| Soluzione di eluzione | 98 Buffer EB, 1 L 10% Tween 20, 1 -L-L-Agente Diriduzione B. |
| Miscela di frammentazione | Buffer di frammentazione 5 L, 10 Enzima di frammentazione ll. |
| Miscela di ligazione dell'adattatore | Buffer di ligazione di 20 luna, 10 ligasi di DNA, 20 Oligos adattatore. |
| Esempio di indice miscela PCR | 50 -L Amp Mix, 10 - L SI Primer |
| Miscela della libreria di codici a barre lipidi | 26,25 L di 2 o più Hot Start master mix, 2,5 l of 10 M RPIX primer, 2,5 l di 10 M -TruSeq Universal Adapter primer (vedi tabella dei materiali) |
| Miscela della libreria di codici a barre dell'anticorpo | 50 l di 2 o più Hot Start master mix, 2,5 litri di 10 M Di OPix primer, 2,5 l di 10 x M P5-smart-pcr oligo ibrido |
Tabella 1: Le miscele di reagenti utilizzate nel protocollo.
| Procedura di incubazione | Temperatura(1) | Tempo |
| Incubazione GEM-RT | Temperatura del coperchio 53 gradi centigradi | |
| Fase 1 | 53 gradi centigradi | 45 min |
| Fase 2 | 85 gradi centigradi | 5 min |
| Fase 3 | 4 gradi centigradi | Tenere |
| 10x Genomica cDNA Amplificazione | Temperatura del coperchio 105 gradi centigradi | |
| Fase 1 | 98 gradi centigradi | 3 min |
| Fase 2 | 98 gradi centigradi | 15 s |
| Fase 3 | 63 gradi centigradi | 20 s |
| Fase 4 | 72 gradi centigradi | 1 min |
| Fase 5 | Ripetere i passaggi da 2 a 4 per 12 cicli in totale (2) | |
| Fase 6 | 72 gradi centigradi | 1 min |
| Fase 7 | 4 gradi centigradi | Tenere |
| Costruzione della biblioteca | Temperatura del coperchio 65 gradi centigradi | |
| Blocco pre-cool | 4 gradi centigradi | Tenere |
| Frammentazione | 32 gradi centigradi | 5 min |
| Riparazione finale e coda A | 65 gradi centigradi | 30 min |
| Tenere | 4 gradi centigradi | Tenere |
| Legatura dell'adattatore | Temperatura del coperchio 30 gradi centigradi | |
| Fase 1 | 20 gradi centigradi | 15 min |
| Fase 2 | 4 gradi centigradi | Tenere |
| Indice di esempio PCR | Temperatura del coperchio 105 gradi centigradi | |
| Fase 1 | 98 gradi centigradi | 45 s |
| Fase 2 | 98 gradi centigradi | 20 s |
| Fase 3 | 54 gradi centigradi | 30 s |
| Fase 4 | 72 gradi centigradi | 20 s |
| Fase 5 | Ripetere i passaggi da 2 a 4 per 12 cicli in totale (3) | |
| Fase 6 | 72 gradi centigradi | 1 min |
| Fase 7 | 4 gradi centigradi | Tenere |
| Libreria di codici a barre Lipid PCR | ||
| Fase 1 | 95 gradi centigradi | 5 min |
| Fase 2 | 98 gradi centigradi | 15 s |
| Fase 3 | 60 gradi centigradi | 30 s |
| Fase 4 | 72 gradi centigradi | 30 s |
| Fase 5 | Ripetere i passaggi da 2 a 4 per 10 cicli in totale (4) | |
| Fase 6 | 72 gradi centigradi | 1 min |
| Fase 7 | 4 gradi centigradi | Tenere |
| Libreria di codici a barre anticorpali PCR | ||
| Fase 1 | 95 gradi centigradi | 3 min |
| Fase 2 | 95 gradi centigradi | 20 s |
| Fase 3 | 60 gradi centigradi | 30 s |
| Fase 4 | 72 gradi centigradi | 20 s |
| Fase 5 | Ripetere i passaggi da 2 a 4 per 8 cicli in totale (5) | |
| Fase 6 | 72 gradi centigradi | 5 min |
| Fase 7 | 4 gradi centigradi | Tenere |
Tabella 2: La procedura di incubazione utilizzata nel protocollo. (1) Prestare attenzione alle diverse temperature del coperchio utilizzate in ogni procedura. (2) Impostare i numeri di ciclo totale in base al carico della cella: 13 cicli per il carico di celle <500; 12 cicli per 500-6.000 carico cellulare; 11 cicli per >6.000 carico cellulare. (3) Impostare i numeri di ciclo totali in base all'input cDNA: 14-16 cicli per 1-25 ng cDNA; 12-14 cicli per 25-150 ng cDNA; 10-12 cicli per 150-500 ng cDNA; 8-10 cicli per 500-1,000 ng cDNA; 6-8 cicli per 1000-1500 ng cDNA. (4) Impostare i numeri di ciclo totali in base all'ingresso cDNA: 8-12 cicli. 5 Impostare i numeri di ciclo totali in base all'ingresso cDNA: 6-10 cicli.
| Codice a barre a base di lipidi Oligonucleotides | |
| Ancora LMO | 5'-TGGAATTCTCGGGGCCAGGGTAACGATCTCTCTCTCACT-Lipid-3' |
| Co-Ancora LMO | 5'-Lipid-AGTGACAGCTGGATCGTTAC-3' |
| Codice a barre Oligo | 5'-CCTTGGCACCCGAGAATTCCANNNNNNNNNNA30-3' |
| Primer additivo per codifica a barre per lipidi | 5'-CTTGGCACCCGAGAATTCC-3' |
| RPIX Primer | 5'-CAAGCAGAAGAGGCAGAGAGAGAGAGAGAGAGAGAGA TTGGCACCACCCGAGAATTCCA-3' |
| Primer adattatore universale | 5'-AATGATACGGCGACCACCBAATCTCTCTCTCTCTCCTACACGAGAGA GCTCTTCCGATCTCT-3' |
| Anticorpi basati a codice a barre Oligonucleotidi | |
| Anticorpo barcodifica oligo | 5'-GTGACTGGAGTTCAGAGTGTGCTCTTCGATCTNNNNNNNNNN NNBAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA |
| Primer additivo HTO | 5'-GTGACTGGAGTTCAGAGTGCTC-3' |
| Primer additivo ADT | 5'-CCTTGGCACCCGAGAATTCC-3' |
| Oligo ibrido P5-smart-pcr | 5'-AATGATACGGCGACCACCGAGATCTACACGCCTCTCTGGA GCAGTGGTATCAACGCAGAGT |
Tabella 3: Sequenze Oligonucleotide utilizzate in questo protocollo. N - Codice a barre o sequenza di indice; - Obbligazione fosforo
Discussione
In questo studio, abbiamo dimostrato un protocollo per analizzare i profili trascrizionali a singola cellula. Abbiamo anche fornito due metodi opzionali ai campioni multiplex nel flusso di lavoro scRNA-Seq. Entrambi i metodi si sono dimostrati fattibili in vari laboratori e hanno fornito soluzioni per eseguire un esperimento a cella singola conveniente e privo di effetti in batch18,26.
Ci sono alcuni passaggi che dovrebbero essere seguiti con attenzione quando si passa attraverso il protocollo. Una sospensione a cella singola ideale dovrebbe avere >90% di cellule vitali e la densità delle celle dovrebbe anche essere all'interno di un intervallo specifico27. È fondamentale per ottenere una buona qualità delle cellule per ridurre al minimo la presenza di aggregati cellulari, detriti e fibre. Gli aggregati cellulari hanno un impatto negativo sul multiplexing del campione e hanno un potenziale rischio di intasare la macchina di generazione di gocciolini17. In generale, un colino cellulare di 30-40 m è ideale per rimuovere grandi ciuffi e detriti, preservando i campioni di cellule, perché la maggior parte delle cellule si ridurrà al di sotto dei 30 m dopo la dissociazione. Si raccomanda invece di utilizzare nuclei a cella singola se il diametro della cellula è maggiore di 30 m. Nelle prime fasi embrionali, la dimensione delle cellule per tutti i tipi di cellule del topo deve essere inferiore a 30 m. Tuttavia, nelle fasi successive, i cardiomiociti nel cuore, i neuroni nel cervello, le cellule muscolari negli arti e alcune cellule adipose possono avere una dimensione cellulare superiore a 30 m. Le dimensioni delle cellule dovrebbero essere misurate per questi tipi di cellule prima di iniziare gli esperimenti a singola cellula.
Le strategie di multiplexing forniscono un modo per analizzare contemporaneamente un gran numero di campioni in modo conveniente. Inoltre, profilando più campioni insieme, possiamo evitare in modo significativo gli effetti batch e identificare i doppietto cellulari. Questi vantaggi saranno molto interessanti per il campo a cella singola. Tuttavia, ci sono alcuni fattori che possono limitare il loro utilizzo. Man mano che più cellule vengono multiplexate in un singolo esperimento, aumenterà anche il rapporto di doppietta cellulare. Anche se questi doppietti possono essere identificati e rimossi analizzando i dati del codice a barre multiplexing, si tradurrà in un grande spreco di letture di sequenziamento. Inoltre, man mano che più cellule sono raggruppate, le cellule sono più facili da rompere e causare un aumento dell'mRNA ambientale, che verrà catturato in goccioline con le cellule e interferirà con la sensibilità di rilevamento. Ci aspettiamo che un'ulteriore ottimizzazione del flusso di lavoro sperimentale o della pipeline di analisi bioinformatica risolverà questi due problemi nel prossimo futuro.
Divulgazioni
Gli autori non hanno conflitti di interesse da divulgare.
Riconoscimenti
Ringraziamo David M. Patterson e Christopher S. McGinnis del laboratorio Dr. .ev J. Gartner per la loro fornitura gentile di reagenti di codifica a barre a base di lipidi e suggerimenti sui passaggi sperimentali e l'analisi dei dati. Questo lavoro è stato fondato dai National Institutes of Health (HL13347202).
Materiali
| Name | Company | Catalog Number | Comments |
| 10% Tween-20 | Bio-Rad | 1610781 | |
| 10x Chip Holder | 10x Genomics | 120252 330019 | |
| 10x Chromium Controller | 10x Genomics | 120223 | |
| 10x Magnetic Separator | 10x Genomics | 120250 230003 | |
| 10x Vortex Adapter | 10x Genomics | 330002, 120251 | |
| 10x Vortex Clip | 10x Genomics | 120253 230002 | |
| 4200 TapeStation System | Agilent | G2991AA | |
| Agilent High Sensitivity DNA Kit | Agilent | 5067-4626 | University of Pittsburgh Health Sciences Sequencing Core |
| Barcode Oligo | Integrated DNA Technologies | Single-stranded DNA | 25 nmol |
| Buffer EB | Qiagen | 19086 | |
| CD1 mice | Chales River | Strain Code 022 | ordered pregnant mice |
| Centrifuge 5424R | Appendorf | 2231000214 | |
| Chromium Chip B Single Cell Kit, 48 rxns | 10x Genomics | 1000073 | Store at ambient temperature |
| Chromium i7 Multiplex Kit, 96 rxns | 10x Genomics | 120262 | Store at -20 °C |
| Chromium Single Cell 3' GEM Kit v3,4 rxns | 10x Genomics | 1000094 | Store at -20 °C |
| Chromium Single Cell 3' Library Kit v3 | 10x Genomics | 1000095 | Store at -20 °C |
| Chromium Single Cell 3' v3 Gel Beads | 10x Genomics | 2000059 | Store at -80 °C |
| Collagenase A | Sigma/Millipore | 10103578001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| Collagenase B | Sigma/Millipore | 11088807001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| D1000 ScreenTape | Agilent | 5067-5582 | University of Pittsburgh Health Sciences Sequencing Core |
| DNA LoBind Tube Microcentrifuge Tube, 1.5 mL | Eppendorf | 022431021 | |
| DNA LoBind Tube Microcentrifuge Tube, 2.0 mL | Eppendorf | 022431048 | |
| Dynabeads MyOne SILANE | 10x Genomics | 2000048 | Store at 4 °C, used in Beads Cleanup Mix (Table 1) |
| DynaMag-2 Magnet | Theromo Scientific | 12321D | |
| Ethanol, Pure (200 Proof, anhydrous) | Sigma | E7023-500mL | |
| Falcon 15mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-70C | |
| Falcon 50mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-49A | |
| Fetal Bovine Serum, qualified, United States | Fisher Scientific | 26140079 | Store at -20 °C |
| Finnpipette F1 Multichannel Pipettes, 10-100μl | Theromo Scientific | 4661020N | |
| Finnpipette F1 Multichannel Pipettes, 1-10μl | Theromo Scientific | 4661000N | |
| Flowmi Cell Strainer | Sigma | BAH136800040 | Porosity 40 μm, for 1000 uL Pipette Tips, pack of 50 each |
| Glycerin (Glycerol), 50% (v/v) | Ricca Chemical Company | 3290-32 | |
| HBSS, no calcium, no magnesium | Thermo Fisher Scientific | 14170112 | |
| Human TruStain FcX (Fc Receptor Blocking Solution) | BioLegend | 422301 | Add 5 µl of Human TruStain FcX per million cells in 100 µl staining volume |
| Isopropanol (IPA) | Fisher Scientific | A464-4 | |
| Kapa HiFi HotStart ReadyMix (2X) | Fisher Scientific | NC0295239 | Store at -20 °C, used in Lipid-tagged barcode library mix (Table 1) |
| Lipid Barcode Primer (Multi-seq Primer) | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
| Low TE Buffer (10 mM Tris-HCl pH 8.0, 0.1 mM EDTA) | Thermo Fisher Scientific | 12090-015 | |
| MasterCycler Pro | Eppendorf | 950W | |
| Nuclease-Free Water (Ambion) | Thermo Fisher Scientific | AM9937 | |
| PCR Tubes 0.2 ml 8-tube strips | Eppendorf | 951010022 | |
| Phosphate-Buffered Saline (PBS) 1X without calcium & magnesium | Corning Cellgro | 21-040-CV | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin (alternative to Thermo Fisher product) | Sigma-Aldrich | SRE0036 | |
| Pipet 4-pack (0.1–2.5μL, 0.5-10μL, 10–100μL, 100–1,000μL variable-volume pipettes | Fisher Scientific | 05-403-151 | |
| Selection reagent (SPRIselect Reagent Kit) | Beckman Coulter | B23318 (60ml) | |
| Template Switch Oligo | 10x Genomics | 3000228 | Store at -20 °C, used in Master Mix (Table 1) |
| The antibody based barcoding strategy is also known as Cell Hashing | |||
| The cell browser is Loup Cell Browser | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/visualization/latest/what-is-loupe-cell-browser | |
| The commercial available analysis pipline in step 8.1 is Cell Ranger | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/what-is-cell-ranger | |
| The lipid based barcoding strategy is also known as MULTI-seq | |||
| The well maintained R platform is Seurat V3 | satijalab | https://satijalab.org/seurat/ | |
| TipOne RPT 0.1-10/20 ul XL ultra low retention filter pipet tip | USA Scientific | 1180-3710 | |
| TipOne RPT 1000 ul XL ultra low retention filter pipet tip | USA Scientific | 1182-1730 | |
| TipOne RPT 200 ul ultra low retention filter pipet tip | USA Scientific | 1180-8710 | |
| TotalSeq-A0301 anti-mouse Hashtag 1 Antibody | BioLegend | 155801 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 2 Antibody | BioLegend | 155803 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 3 Antibody | BioLegend | 155805 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TrueSeq RPI primer | Integrated DNA Technologies | Single-stranded DNA | 100 nmol, used in Lipid-tagged barcode library mix (Table 1) |
| Trypan Blue Solution, 0.4% | Fisher Scientific | 15250061 | |
| Trypsin-EDTA (0.25%), phenol red | Fisher Scientific | 25200-056 | |
| Universal I5 | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
Riferimenti
- Raj, A., Van Den Bogaard, P., Rifkin, S. A., Van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877(2008).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6 (5), 377(2009).
- Li, G., Plonowska, K., Kuppusamy, R., Sturzu, A., Wu, S. M. Identification of cardiovascular lineage descendants at single-cell resolution. Development. 142 (5), 846-857 (2015).
- DeLaughter, D. M., et al. Single-cell resolution of temporal gene expression during heart development. Developmental Cell. 39 (4), 480-490 (2016).
- Li, G., et al. Single cell expression analysis reveals anatomical and cell cycle-dependent transcriptional shifts during heart development. Development. 146 (12), dev173476(2019).
- Meilhac, S. M., Buckingham, M. E. The deployment of cell lineages that form the mammalian heart. Nature Reviews Cardiology. 1, (2018).
- Liu, Z., et al. Single-cell transcriptomics reconstructs fate conversion from fibroblast to cardiomyocyte. Nature. 551 (7678), 100(2017).
- Lafzi, A., Moutinho, C., Picelli, S., Heyn, H. Tutorial: guidelines for the experimental design of single-cell RNA sequencing studies. Nature Protocols. 1, (2018).
- The Tabula Muris Consortium. Single-cell transcriptomics of 20 mouse organs creates a Tabula Muris. Nature. 562 (7727), 367(2018).
- Gawad, C., Koh, W., Quake, S. R. Single-cell genome sequencing: current state of the science. Nature Reviews Genetics. 17 (3), 175(2016).
- Grün, D., van Oudenaarden, A. Design and analysis of single-cell sequencing experiments. Cell. 163 (4), 799-810 (2015).
- Ziegenhain, C., et al. Comparative analysis of single-cell RNA sequencing methods. Molecular cell. 65 (4), 631-643 (2017).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17 (1), 77(2016).
- Islam, S., et al. Characterization of the single-cell transcriptional landscape by highly multiplex RNA-seq. Genome Research. 21 (7), 1160-1167 (2011).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161 (5), 1202-1214 (2015).
- Klein, A. M., et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 161 (5), 1187-1201 (2015).
- Library Prep -Single Cell Gene Expression -Official 10x Genomics Support. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/library-prep (2018).
- McGinnis, C. S., et al. MULTI-seq: sample multiplexing for single-cell RNA sequencing using lipid-tagged indices. Nature Methods. 1, (2019).
- Stoeckius, M., et al. Cell hashing with barcoded antibodies enables multiplexing and doublet detection for single cell genomics. Genome Biology. 19 (1), 224(2018).
- Chan, M. M., et al. Molecular recording of mammalian embryogenesis. Nature. 570 (7759), 77-82 (2019).
- Agilent 4200 TapeStation System. , Agilent. https://www.agilent.com/cs/library/datasheets/public/5991-6029EN.pdf (2019).
- SPRIselect User Guide. , Beckman Coulter. https://research.fhcrc.org/content/dam/stripe/hahn/methods/mol_biol/SPRIselect%20User%20Guide.pdf (2012).
- Qubit 4 Fluorometer User Guide. , Thermo Fisher Scientific. https://assets.thermofisher.com/TFS-Assets/LSG/manuals/MAN0017209_Qubit_4_Fluorometer_UG.pdf (2018).
- Agilent High Sensitivity DNA Kit Guide. , Agilent. https://www.agilent.com/cs/library/usermanuals/public/High%20Sensitivity_DNA_KG.pdf (2016).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411(2018).
- Weber, R. J., Liang, S. I., Selden, N. S., Desai, T. A., Gartner, Z. J. Efficient targeting of fatty-acid modified oligonucleotides to live cell membranes through stepwise assembly. Biomacromolecules. 15 (12), 4621-4626 (2014).
- Single Cell Protocols Cell Preparation Guide. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/sample-prep (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon