Method Article
マウス胚細胞の多重化単細胞mRNAシーケンシング解析
要約
ここでは、マウス胚組織における遺伝子発現をプロファイルする多重化単一細胞mRNAシーケンシング法を提示した。多重化戦略と組み合わせた液滴ベースの単一細胞mRNAシーケンシング(scRNA-Seq)法は、複数のサンプルから単一細胞を同時にプロファイルできるため、試薬コストを大幅に削減し、実験的なバッチ効果を最小限に抑えることができます。
要約
単一細胞mRNAシーケンシングはここ数年で大きな進歩を遂げ、発生生物学の分野で重要なツールとなっています。希少細胞集団の同定、新規マーカー遺伝子の発見、空間的・時間的発達情報の解読に成功しています。単一細胞法は、マイクロ流体ベースのFluidigm C1技術から、過去2~3年で液滴ベースのソリューションへと進化しました。ここでは、液滴ベースのscRNA-Seq法を用いてマウス胚組織細胞をプロファイリングする方法を示す例として心臓を用いた。さらに、1 回の実験で複数のサンプルをプロファイリングするために、2 つの戦略をワークフローに統合しました。統合された方法の1つを使用して、我々は同時に8つの心臓サンプルから9,000以上の細胞をプロファイリングしました。これらの方法は、異なる遺伝的背景、発達段階、または解剖学的位置からの単一細胞を同時にプロファイルする費用対効果の高い方法を提供することにより、発生生物学分野にとって貴重です。
概要
各単一細胞の転写プロファイルは、胚発生中の細胞集団によって異なる。situハイブリダイゼーションにおける単一分子は、少数の遺伝子1の発現を可視化するために使用することができるが、単一細胞mRNAシーケンシング(scRNA-Seq)は、単一細胞における遺伝子のゲノムワイド発現パターンを例示する偏りのないアプローチを提供する。2009年2月に最初に出版された後、scRNA-Seqは、近年3、4、5の複数の発達段階で複数の組織を研究するために適用されている。また、ヒト細胞アトラスが最近発達に焦点を当てたプロジェクトを立ち上げているため、近い将来、ヒト胚組織からの単一細胞データの生成が期待されています。
最初に発達する臓器としての心臓は胚発生において重要な役割を果たす。心臓は複数の細胞型で構成され、各細胞型の発達は時間的および空間的に厳密に調節される。ここ数年、初期の発達段階における心臓細胞の起源と細胞系統は、先天性心疾患の病因を理解するための非常に有用なナビゲーションツールを提供する6を特徴とし、心筋細胞再生7を刺激するより技術的に高度な方法を開発する。
scRNA-Seqは、近年8、9、10で急速な拡大を遂げています。新たに開発された方法により、単一細胞実験の設計と解析がより達成可能になりました。ここで提示する方法は、液滴溶液に基づく商業手順である(材料表を参照)15、16。この方法は、マイクロ流体コントローラシステムの制御下にある油水エマルジョン液滴中の細胞および一意にバーコードされたビーズのセットを捕捉することを特徴とする。液滴への細胞負荷の速度は極めて低く、液滴エマルションの大部分は1つの細胞17のみを含む。この手順の独創的な設計は、バーコードングと同時に発生する液滴エマルジョンへの単一細胞分離から来ており、異種集団でRNA-Seqを使用して個々の細胞の並列分析を可能にします。
多重化戦略の組み込みは、従来の単一細胞ワークフロー13、14への重要な追加の1つである。この添加は、細胞ダブレットを廃棄し、実験コストを削減し、バッチ効果18、19を排除するのに非常に有用である。脂質ベースのバーコードング戦略と抗体ベースのバーコードング戦略(材料表参照)は、主に多重化法の2つである。特定のバーコードを使用して両方の方法で各サンプルにラベルを付け、ラベル付きサンプルを混合して単一セルのキャプチャ、ライブラリの準備、およびシーケンス処理を行います。その後、バーコードシーケンスを分析することによって、プールされたシーケンスデータを分離することができます (図 1)19.ただし、2 つの方法には大きな違いがあります。脂質ベースのバーコード戦略は、脂質修飾オリゴヌクレオチドに基づいており、細胞型の好みを有することが見つかっていない。抗体ベースのバーコードング戦略は、抗原タンパク質19、20を発現する細胞のみを検出できるが。また、脂質を染色するのに約10分かかりますが、抗体を染色するのに40分かかります(図1)。さらに、脂質修飾オリゴヌクレオチドは抗体共役オリゴヌクレオチドよりも安価であるが、本稿の執筆時点では市販されていない。最後に、脂質ベースの戦略は1回の実験で96サンプルを多重化できますが、抗体ベースの戦略は現在12サンプルしか多重化できません。
単一の実験で多重化する推奨細胞数は2.5 x 104より小さくする必要があり、そうでなければ、細胞ダブレットの高い割合と潜在的な周囲mRNA汚染につながります。多重化戦略により、複数のサンプルの単一セルキャプチャ、cDNA生成、ライブラリ調製のコストは1サンプルのコストに削減されますが、シーケンシングコストは変わりません。
プロトコル
動物の手順は、ピッツバーグ大学の動物ケアと使用委員会(IACUC)に従っています。
1. マウス胚性心臓解剖と単細胞懸濁製剤
メモ:この手順は、解剖する胚の数に応じて数時間かかる場合があります。
- E18.5胚性心臓を取得するには、CO2投与により妊娠中のCD1マウスを安楽死させる。カミソリを使って腹部の不要な髪を取り除き、70%のエタノールで皮膚を消毒します。
- 殺菌されたはさみを使って腹部の皮膚を切り取り、胚を慎重に解剖し、すぐに氷の上の冷たいリン酸緩衝生理食塩分(PBS)に入れます。
- 鉗子とはさみを使用して立体顕微鏡の下で冷たいPBSで満たされた10センチの皿で慎重に個々の胚から心臓を隔離する。
注:心臓で肺を解剖して4つのチャンバーをそのままにし、手術器具で心臓を直接キャッチ/プルしないでください。 - 約10の心臓を冷たいPBSで満たされた新しい10cmの皿に移し、心臓を左心房、右心房、左心室、右心室にマイクロ解剖する。
注: これは、各サンプルから 1 x 106 個を超えるセルを生成することを前提としています。サンプルあたり少なくとも 1 x 105セルから開始することをお勧めします。 - 4つのチャンバーティッシュをそれぞれ1.5mLチューブに移し、はさみを使って細かく刻みます。300 x gで遠心分離機を3分間採取し、組織を採取する。
- 上清を吸引した後、各チューブに0.25%トリプシン/EDTAの1mLを加え、P1000ピペットを使用して37°水浴で10分間上下にインキュベートします。
- 胚期がE11.5より古い場合は、10mg/mLコラゲターゼA/B混合物を1mL加え、37°Cで10〜20分間インキュベートします。
- 細胞を15mLチューブに移し、8 mLハンクのバランス塩溶液(HBSS)を加え、酵素を希釈します。細胞を300 x gで5分間スピンダウンし、細胞を1mLのPBSでサスペンドし、1.5 mLチューブに移します。40μmのセルストレーナーを使用してセルをフィルタリングします。
- 各サンプルから15μLのボリュームを取り、同じ量の0.04%のトリパンブルーと混合します。これをセルカウントチャンバーにロードし、セルカウンタ内のセルをカウントします。
注:高品質の結果を生成するには、細胞生存率を95%以上にすることをお勧めします。
2. 単一セル多重バーコード
注: この手順では、処理されたサンプルの数によって異なる少なくとも 40 分かかります。RNase除染液で処理されたクリーンベンチエリアは、増幅前のステップ(ステップ2.11~3.11)に必要であり、増幅後のステップ(3.11後のステップ)には別のクリーンベンチエリアが必要です。
-
脂質ベースのバーコード化手順(オプション手順1)
- 細胞濃度に基づいて、サンプルあたり5 x 105細胞未満に保ちます。細胞懸濁液に破片や細胞凝集体が含まれないようにします。
- 各サンプルに対して 2 μM アンカー/バーコード・ストック・ソリューションと 2 μM 共同アンカー・ストック・ソリューションを準備します (表 1)。
注:アンカーと共同アンカーは、ゼヴ・J・ガートナー博士の研究室から親切に贈られました。これらの脂質修飾オリゴヌクレオチドを合成するために、DNA配列を固体支持体上で脂肪酸と共役させ、逆相高速液体クロマトグラフィー(HPLC)18、19によって精製した。 - 細胞をPBSで2回洗浄し、300 x gで細胞を5分間回収します。
- 20°Lのアンカー/バーコードストック液とピペットを加え、軽く上下に混ぜます。氷の上で5分間インキュベートする。
- 20°Lのコアンカーストック液とピペットを加えて軽く上下に混ぜ、さらに5分間氷の上でインキュベートします。
- 1%BSAで冷たいPBSを1mL、300 x gで4°Cで5分間加えます。PBSで氷冷1%BSAで少なくとも2回洗う。
- すべてのサンプルを一緒に結合し、40μmの細胞ストレーナーを通してろ過する。細胞を数え、セクション3で使用するために氷上の細胞懸濁液を維持します。
-
抗体ベースのバーコード付きバーコード手順(オプションの手順2)
- 各サンプル(ステップ1.8から)の遠心分離機1x106x 106細胞を300 x gで5分間、1.5mLの低結合管に100μLの染色緩衝液(表1)で一時停止する。
- 10μL Fcブロッキング試薬を加え、4°Cで10分間インキュベートします。
- 抗体を調製し(材料表を参照)、2~8°Cで10分間14,000 x gで遠心分離して調製します。
- 細胞染色緩衝液の50μLに各オリゴ共役抗体1μgを加えて、抗体染色液20を作製する。各サンプルチューブに1つの抗体染色溶液を追加します。4 °Cで30分間インキュベートします。
- 細胞を1mLのPBSで3回洗浄し、4°Cで350xgで5分間回転させる。
- すべてのサンプルを1 mLの染色バッファーで所望の割合でプールし、4°Cで350 x gで5分間スピンする。
- 適切な濃度(最大1,500細胞/μL)でPBSの細胞を再サスペンドし、40μmの細胞ストレーナーを介して細胞を濾過します。すぐに次の手順に進みます。
3. 液滴生成とmRNA逆転写
注:このステップは、1つの多重化反応のために約90分かかります。
- ゲルビーズを室温まで30分間平衡化します( 材料表参照)。ゲルビーズインエマルジョン(GEM)キットから試薬を取り出し(材料表を参照)、記載された温度に保ちます。
- チップ B をチップホルダーに組み立てます (「材料表」を参照)。
- 50%グリセロール溶液の75°Lを行1の未使用の井戸に分配します。行 2 の 40 μL;行 3 で 280 μL。チップの一番上の行の回復井戸にグリセロールを追加しないでください。
- 表1に従って氷の上にマスターミックスを準備します。細胞懸濁量計算機表17に従ってマスターミックスに適切な量のセル懸濁液およびヌクレアーゼフリー水を加え、ミックスを穏やかにピペットする。気泡を導入することなく、サンプル井戸の底部中央に75°Lの細胞混合物を分配する。
- ボルテックスは、ボルテックスアダプタを使用して30sのゲルビーズをバーテックスし、気泡を導入することなく、ゲルビーズの下部中央に40°Lのゲルビーズをゆっくりと分配します。
メモ:濡れ障害を避けるために、細胞とゲルビーズを追加する間に30sを待つことが重要です。 - 3列目の仕切り油の場合は、井戸の側壁を通して280°Lの仕切り油を分配します。
注: 270 μL 未満の仕切りオイルをロードすると、MM の生成が異常になります。 - ガスケットをチップに取り付け、ガスケットを押し下げないようにして水平にしておき、ガスケットを濡らさないようにしてください。
- 組み立てられたチップをクロムコントローラにロードし、クロム単一セルBプログラムを実行します(材料表を参照)。
- チップを取り出し、ガスケットを捨てます。ふたを折って井戸を45°で露出させ、液体レベルを確認して詰まりがないことを確認します。
- 回復井戸の最も低い点からGEMの100°Lをゆっくりと吸引し、GEMの均一性を確認します。チューブの側壁に対するピペットチップを用いた氷上の新しいポリメラーゼ連鎖反応(PCR)チューブにGEMを分配します。
注:過剰な水層が観察された場合は、サンプルを再準備することをおおめします。重要なのは、GEM がまだピペットの先端にある場合に、混合物の写真を撮ります。この画像は、湿潤障害、部分的に乳化されたGEM、および試薬の詰まりがあるかどうかを示すことができます。写真は、交換用試薬やチップで試薬会社から払い戻しを得るための証拠としても使用できます。 - チューブをサーマルサイクラーに入れ、逆転写手順を実行します(表2)。
注: ここで停止するか、次の手順に進みます。PCR製品は-20°Cで最大1週間保存できます。
4. cDNA増幅
注: この手順には約 150 分かかります。
- 単一細胞逆転写のクリーンアップを投稿
- GEM キットから cDNA 増幅試薬を取り出し (材料表を参照)、指定された温度に保ちます。
- 125 μL の回収剤を室温でサンプルに加え、二橋混合物を得た。不透明な液体は観察されず、ピペットや渦を避けるべきです。
- 60sを待った後、チューブの底部から125μLの回収剤をゆっくりと取り外します。
- 磁気ビーズのボルテックス(材料表参照)は30s徹底的に使用し、すぐにビーズクリーンアップ混合物を調製するために使用します(表1)。試薬は、リストに従って順次添加する必要があります。
- ビーズをクリーンアップ混合物をボルテックスし、サンプルに200 μLを加えます。混合物を10回ピペットし、室温で10分間インキュベートする。
- 表1に示すように試薬を順次添加し、溶出液を調製し、次のようにcDNAを溶出する。
- 溶液がクリアされるまで、サンプルを磁石(高い位置)に置き(材料表を参照)、上清を取り除きます。
- ペレットに200μLの80%エタノールを加えます。30s待ってからエタノールを取り出します。
- 手順 4.1.6.2 をもう 2 回繰り返します。遠心分離機を短時間、磁石の上に置きます(低位置)。残りのエタノールと空気乾燥を2分未満に慎重に取り除きます。
注:2分を超えて空気乾燥を超えないと、それ以外の場合は溶出効率が低下します。 - 磁石からサンプルを取り出します。溶出液とピペットを35.5μL加え、15回混ぜます。室温で2分間インキュベートする。
- 溶液がクリアされるまで、サンプルを磁石(高い位置)に置きます。サンプルの35°Lを新しいチューブストリップに移します。
注: この精製手順は、手順 4.2.1.6、4.2.2.3、5.8、6.1.10、および 6.2.6 でも使用されます。各ステップでサンプルを溶出するために使用される磁気ビーズの濃度とEBバッファー/超純水の体積に注意してください。
- 自動電気泳動器具21を用いて溶出したcDNAの大きさ、濃度、完全性を定量化する(材料表参照)(図2)。
- cDNAの増幅
-
脂質ベースのバーコード化戦略を用いたcDNA増幅(オプション手順1)
- 氷上に増幅反応混合物(表1)を調製する。
- 増幅反応混合物を35μLのcDNAサンプルに加えます(ステップ4.1.6.7から)。ミックスをピペットし、遠心分離機を簡単に。cDNA増幅手順に従ってサーマルサイクラーで混合物をインキュベートする(表2)。
- 十分にボルテックスした後、120μLの選択試薬と100μLの超純水を100μLのサンプルに加えて、0.6倍の濃度の選択試薬を得た(材料表を参照)。混合物を15回ピペット。
- 室温で5分間インキュベートし、溶液が透明になるまで試料を磁石の上に置きます。
注:ビーズ画分および多重バーコード付きcDNA中の内因性cDNAは上清中である。 - ステップ6.1でバーコード化されたcDNAライブラリ構造を多重化するために、上清を1.5 mL低結合チューブに移します。
- 4.1.6の手順に従って内因性のcDNAを洗浄し、40 μLのEBバッファで溶出します。
- 自動化された電気泳動装置で精製されたcDNAサンプルを1μL実行し(材料表を参照)、cDNAを分析/定量します。
- 内因性ライブラリー構築のための新しいPCRチューブにcDNAのアリコート10°L。
注: ここで停止するか、次の手順に進みます。残りのサンプルは-20 °Cで最大4週間保存し、必要に応じて追加のライブラリを生成することができます。
-
抗体ベースのバーコード作成戦略におけるcDNA増幅(オプション手順2)
- HTOおよびADT添加プライマーの2pmolと15μLのcDNAプライマーを増幅反応混合物の50μLに加え(表1)、cDNA増幅手順を用いてcDNA増幅を行う(表2)。
- 内因性cDNA(ビーズ分画)と多重バーコードcDNA(上清)を分離するには、0.6x選択試薬を使用します。ステップ6.2でバーコードライブラリの構築を実行するために上清を保存することを忘れないでください。
- 4.1.6のステップに従って内因性転写物cDNAを精製および溶出し、ライブラリの品質管理(QC)を行い、アリコート10μLを内因性ライブラリー構築用の新しいPCRチューブに入れます。
-
脂質ベースのバーコード化戦略を用いたcDNA増幅(オプション手順1)
5. 内因性トランスクリプトライブラリの準備
注: この手順には約 120 分かかります。
- 遺伝子発現ライブラリー構築試薬は、ライブラリキットからそれぞれその示された温度に保ちます(材料表を参照)。
- 断片化混合物(表1)を氷上に調製し、ピペットを混合し、遠心分離する。
- 10 μL 精製 cDNA サンプル (ステップ 4.2.1.8 または 4.2.2.3 から) に 25 μL EB バッファーを追加し、新たに調製した 15 μL 断片化混合物をサンプルに追加し、混合物を氷上で 15 回ピペットし、遠心分離機を簡単に取り付けます。
- サンプルを予冷されたサーマルサイクラーに移し、断片化、エンドリペア、およびAテーリングのためのPCRプログラムを開始します(表2)。
- ボルテックスは、磁気ビーズを中断し、0.6xおよび0.8x選択試薬を連続的に使用し、ユーザガイド17、22に従って両面サイズ選択を行う。50 μL の EB バッファーを使用して DNA を溶出します。
- アダプターライゲーション混合物(表1)を調製し、次いで混合物を十分にピペットし、遠心分離機を簡潔に行う。
- 50 μLのアダプターライゲーション混合物を50°Lのサンプルに加え、ピペットを15回再び混合し、遠心分離機を短時間で加えます。サーマルサイクラーのプロトコルに従ってアダプタライゲーションを実行します(表2)。
- 0.8x選択試薬を使用してライゲーション生成物を精製し、30 μLのEB緩衝液で精製サンプルを溶出します(ステップ4.1.6を参照)。
- サンプルインデックスPCR混合物(表1)を調製し、精製されたサンプルに60μLを加えます。サンプルインデックスの10μLをサンプルに加え、ピペットを5回上下に混ぜ、遠心分離機を短時間、サンプルインデックスプロトコルに続くサーマルサイクラーでインキュベートする(表2)。
注: ここで停止するか、次の手順に進みます。複数のウェルを使用する場合は、ウェルごとに 1 つの特定のサンプル インデックス(「材料表」を参照)を選択します。各ウェルに使用されるインデックス ID を記録し、多重シーケンス実行で重複しないようにしてください。 - 0.6xおよび0.8x選択試薬を連続的に使用して、精製された内因性細胞ライブラリーDNADNA17の35μLを取得する両面サイズ選択を行う。
- シーケンシング前の内因性細胞ライブラリーをQCする(図2)。
6. 多重化サンプルバーコードcDNAライブラリの調製
注: この手順には少なくとも 120 分かかります。
-
脂質ベース多重化戦略のサンプルバーコードライブラリ生成(オプション手順1)
- ステップ4.2.1.5のサンプルバーコードcDNAに520μLの選択試薬と360 μLのイソプロパノールを加え、3.2倍の選択試薬濃度を得ます。混合物を10回ピペットし、室温で5分間インキュベートする。
- チューブを磁気ラックに置き、溶液がクリアするのを待ちます。その後、上清を廃棄します。
- 500 μLの80%エタノールを使用して、磁石でビーズを2回洗浄し、各洗浄後30s待ちます。
- ビーズを短く遠心分離し、磁石の上に置きます。残りのエタノールをP10マイクロピペットで取り出し、ビーズを2分間残します。
- マグネットラックからチューブを取り外し、50°LのEBバッファでビーズを再サスペンドします。徹底的に混合するために上下にパイプ。室温で2分間インキュベートする。
- チューブを磁石に戻し、溶液がクリアするのを待ちます。上清(サンプルバーコードcDNA)を新しいPCRチューブに移します。ビーズを移さないように注意してください。
- サンプルバーコードcDNA23の濃度を定量する。
- 脂質バーコードライブラリ混合物を調製し(表1)、精製されたバーコード付きcDNA(ステップ6.1.6から)とヌクレアーゼフリー水の3.5ngを加え、総体積50μlです。
- 脂質ベースのバーコードライブラリPCR(表2)に続くサーマルサイクラーに保管してください。
- 1.6x選択試薬を使用してPCR製品を精製し、25μLのEBバッファーでDNAを溶出します(ステップ4.1.6を参照)。
- 1:5の初期希釈から高感度DNA分析法24を用いてライブラリー濃度を定量する(図2)。
-
抗体ベースの多重化戦略のサンプルバーコードライブラリ生成(オプション手順2)
- ステップ4.2.2.3から取得したサンプルバーコードを含む上清に、選択試薬の1.4倍反応量を追加し、2倍の選択試薬比を得た。
- 4.1.6のステップに従って80%エタノールでビーズを洗浄し、バーコードされたcDNAを超純水で溶出します。
- 2x選択試薬で選択プロトコルを2回目に行い、超純水を使用して溶出します。
- 抗体バーコードライブラリー混合物(表1)を調製し、最後のステップから精製されたバーコード付きcDNAを45μL添加する。
- 抗体バーコードライブラリーPCR(表2)20に続くサーマルサイクラーでインキュベートする。
- 1.6x選択試薬を使用してPCR製品を精製し、30μLの超純水で精製サンプルを溶出します(ステップ4.1.6を参照)。
7. ライブラリの順序付け
注: HiSeq 4000 や NovaSeq などの複数の次世代シーケンス プラットフォームを使用して、内因性トランスクリプト ライブラリと多重バーコード ライブラリをシーケンスできます。
- 次世代シーケンスプラットフォームを使用して、内因性トランスクリプトライブラリと多重化バーコードライブラリをシーケンスします。
- シーケンシング会社またはシーケンシング施設の専門家からの推奨事項に従ってライブラリを希釈します。内因性トランスクリプト ライブラリには、セルあたり最低 20,000 回の読み取り、バーコード ライブラリでは 3000 回の読み取りが推奨されます。
8. データ分析
注: クラウドベースのリソース BaseSpace を使用するか、UNIX サーバーで bcl2fastq パッケージを実行して、シーケンス データを多重化解除します。
-
内因性トランスクリプトームデータ解析
- 多重化解除ソフトウェアから生成された fastq データを使用して、市販のデータ分析パイプラインで "mkfastq" を実行し(「mkfastq」を実行して、各 GEM バーコードをさらに多重化します)。
- "count" を実行して、配置、フィルター処理、バーコード カウント、および UMI カウントを実行します。
- 必要に応じて、"aggr" を実行して、1 つの実験から複数のシーケンス レーンを集約します。
- 「細胞ブラウザ」(材料表参照)を使用して、データを可視化し、クラスターセルを作成し、微分発現遺伝子を同定し、tSNEまたは遺伝子発現ヒートマッププロットを生成します。
- 必要に応じて、適切に管理された R ベースのプラットフォーム25 (材料表を参照) を使用して、データを正規化およびスケーリングし、微分的に発現された遺伝子を識別し、tSNE/UMAP プロットと遺伝子発現ヒートマップを生成します (図 3)。
-
多重バーコードデータ分析
-
脂質ベースのバーコード戦略からのデータ分析
- 市販のデータ分析パイプラインまたは deMULTIplex R パッケージ (https://github.com/chris-mcginnis-ucsf/MULTI-seq) を使用して、サンプル バーコード FASTQ ファイルをサンプルバーコード UMI カウント マトリックスに変換します。
- 統合分析のために、バーコード UMI カウント マトリックスを内因性トランスクリプトーム データと共に R ベースのプラットフォームに読み込みます(材料表を参照)。
-
抗体ベースのバーコード戦略からのデータ分析
- 市販のデータ分析パイプラインの「count」を使用して、ライブラリ CSV ファイルとハッシュタグ機能リファレンス CSV ファイルを提供してバーコードをマップします。
- 各セルバーコードの特徴バーコード数と共に遺伝子発現数を含む出力統合フィーチャバーコードマトリックスを、下流解析用の R ベースのプラットフォームにロードします。
-
脂質ベースのバーコード戦略からのデータ分析
結果
本研究では、マウス胚性心臓を例として、臓器の別々の部分から異なるサンプルを同時に処理するために多重化された単一細胞mRNAシーケンシングがどのように行われたかを示した。E18.5 CD1マウスの心臓を単離し、左心房(LA)、右心房(RA)、左心室(LV)および右心室(RV)に解剖した。心房細胞と心室細胞を脂質ベースのバーコード付きバーコード手順を用いて独立してバーコード化し、GEMの生成と逆転写の前に混合した。概略の概要を図 1に示します。ライブラリー構築前にcDNA濃度を定量した(図2A)。標準scRNA-Seqから多重化されたscRNA-Seqを行う場合の違いの1つは、内因性cDNAライブラリとサンプルバーコードcDNAライブラリがcDNA増幅および精製後に別々に取得されたことです(ステップ4.2.1および4.2.2.2)。2 つのライブラリも実験で修飾されました (図 2B,C)。次世代シーケンシングとデータ解析を行い、その後、ライブラリ構築とQCを行いました。
HiSeqX プラットフォームを使用して、両方のライブラリを同じシーケンスレーンでシーケンスしました。シーケンス データを使用して、まず BaseSpace プログラムを使用して内因性トランスクリプト データとバーコード データを分離しました。次に、各セルのバーコード式を分析し、1 種類のバーコードを一意に表す 8 つのグループのバーコードを見つけ、8 つの異なるサンプルのセルを表します (図 3A)。さらに、負のセルとして定義したバーコードを表さないセルや、ダブレットを表す 2 つの異なるバーコードを表すセルがあることもわかっていました (図 3B)。要約すると、細胞の約70%がシングルト、25%の細胞が陰性、5%が二重であることがわかりました。
シングルト細胞を使用すると、さらに下流の解析を行い、細胞の不均一性と分子規制を理解することができます。潜在的な分析は、細胞型注釈(図4A)、新規/希少細胞型同定(図4B)、解剖学的ゾーン比較解析(図4C)、および細胞周期相分離などの遺伝子オントロジー経路解析(図4D)であり得る。

図1:多重化単一細胞mRNAシーケンシングワークフロー胚の18.5段階の心臓は、多重液滴ベースの単一細胞シーケンシング手順を用いて分析した。RT = 逆転写。この図の大きなバージョンを表示するには、ここをクリックしてください。

図2:異なるステップでの代表的なQC結果。(A) ステップ4.1.7からのcDNAのQC分析標的断片サイズは200~9000bp.(B)内因性ライブラリーであり、(C)バーコードライブラリを自動電気泳動装置で分析した。内因性ライブラリのターゲットフラグメントサイズは300~600 bpで、バーコードライブラリのDNAサイズは約172bpです。
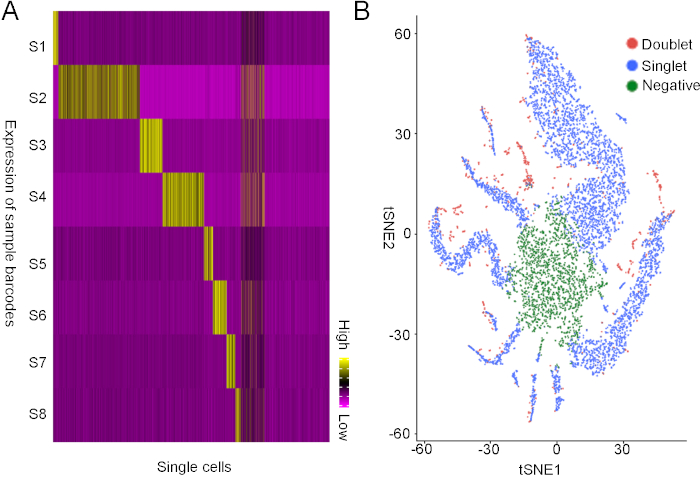
図3:脂質ベースのバーコード化戦略からのシーケンシングデータの分解。(A) バーコード式の教師なし分析。X 軸は単一のセルを表し、Y 軸はバーコードを表します。8個の単一セル集団のそれぞれは、8つのバーコードのうちの1つを一意に表現するために同定された。一部のセルは複数のバーコードを表し、一部のセルはバーコードを表しません。(B)単一細胞、ダブレット細胞、および負細胞のt-SNEプロット。この図の大きなバージョンを表示するには、ここをクリックしてください。

図4:単一細胞転写データの高度な分析(A-D)単一細胞データは、細胞の不均一性および分子経路を理解するために異なる方法で分析することができる。ここでは、いくつかのアプリケーションを例として挙べています。単一のセルを R パッケージに読み込んで、細胞の種類 (A)、希少な細胞集団(B)、細胞解剖学的ゾーン(C)、および細胞周期相 (D) を識別します。この図の大きなバージョンを表示するには、ここをクリックしてください。
| 混合名 | 組成 |
| コラゲラゲアゼ混合物 | 10 mg/mL コラゲアーゼ A および 10 mg/mL コラゲアーゼ B, 40% FBS で HBSS++ に溶解. |
| 2 μM アンカー/バーコードストックソリューション | 50 μM アンカーと 10 μM バーコードストランドを PBS (FBS または BSA なし) のモル比で混合し、合計体積を 25 μL にします。 |
| 2 μM コアンカーストックソリューション | 24 μL PBS (FBS または BSA なし) で 1 μL 50 μM コアンカーを希釈します。 |
| 染色バッファー | 2% BSA を含む PBS, 0.01% ツイーン 20 |
| マスター混合物 | 20 μL RT試薬、3.1 μL オリゴ、2 μL 還元剤 B、8.3 μL RT 酵素 C. |
| ビーズクリーンアップ混合物 | 182°Lクリーンアップバッファ、8μL選択試薬、5μL還元剤B、5°Lヌクレアーゼフリーウォーター。 |
| 増幅反応混合物 | 10 μM 脂質タグ付き添加プライマーの 1 μL、15 μL cDNA プライマー、50 μL アンプミックス |
| 溶出液 | 98°LバッファEB、1 μL 10%テンテン20、1°L還元剤B。 |
| 断片化混合物 | 5μL断片化緩衝液、10μL断片化酵素。 |
| アダプターライゲーション混合物 | 20°Lライゲーションバッファー、10 μL DNA リガーゼ、20 μL アダプタ オリゴ。 |
| サンプルインデックスPCR混合物 | 50°Lアンプミックス、10°L SIプライマー |
| 脂質バーコードライブラリー混合物 | 26.25°Lの2×ホットスタートマスターミックス、10μM RPIXプライマーの2.5°L、10μM TruSeqユニバーサルアダプタプライマーの2.5°L(材料表を参照) |
| 抗体バーコードライブラリー混合物 | 50°Lの2×ホットスタートマスターミックス、10μM RPIXプライマーの2.5°L、10μM P5-スマートpcrハイブリッドオリゴの2.5 μL |
表1:プロトコルで使用される試薬混合物。
| インキュベーション手順 | 温度(1) | 時間 |
| GEM-RT インキュベーション | 蓋温度 53 °C | |
| ステップ 1 | 53 °C | 45 分 |
| ステップ 2 | 85 °C | 5 分 |
| ステップ 3 | 4 °C | 保持 |
| 10xゲノミクスcDNA増幅 | ふた温度 105 °C | |
| ステップ 1 | 98 °C | 3 分 |
| ステップ 2 | 98 °C | 15 s |
| ステップ 3 | 63 °C | 20 s |
| ステップ 4 | 72 °C | 1 分 |
| ステップ 5 | 合計 12 サイクルに対して手順 2 ~ 4 を繰り返します (2) | |
| ステップ 6 | 72 °C | 1 分 |
| ステップ 7 | 4 °C | 保持 |
| 図書館建設 | 蓋温度 65°C | |
| プリクールブロック | 4 °C | 保持 |
| 断片化 | 32 °C | 5 分 |
| エンドリペアとAテーリング | 65 °C | 30 分 |
| 保持 | 4 °C | 保持 |
| アダプターライゲーション | 蓋温度 30°C | |
| ステップ 1 | 20 °C | 15 分 |
| ステップ 2 | 4 °C | 保持 |
| サンプルインデックスPCR | ふた温度 105 °C | |
| ステップ 1 | 98 °C | 45 s |
| ステップ 2 | 98 °C | 20 s |
| ステップ 3 | 54 °C | 30 s |
| ステップ 4 | 72 °C | 20 s |
| ステップ 5 | 合計 12 サイクルに対して手順 2 ~ 4 を繰り返します(3) | |
| ステップ 6 | 72 °C | 1 分 |
| ステップ 7 | 4 °C | 保持 |
| 脂質バーコードライブラリー PCR | ||
| ステップ 1 | 95 °C | 5 分 |
| ステップ 2 | 98 °C | 15 s |
| ステップ 3 | 60 °C | 30 s |
| ステップ 4 | 72 °C | 30 s |
| ステップ 5 | 合計 10 サイクルに対して手順 2 ~ 4 を繰り返します (4) | |
| ステップ 6 | 72 °C | 1 分 |
| ステップ 7 | 4 °C | 保持 |
| 抗体バーコードライブラリーPCR | ||
| ステップ 1 | 95 °C | 3 分 |
| ステップ 2 | 95 °C | 20 s |
| ステップ 3 | 60 °C | 30 s |
| ステップ 4 | 72 °C | 20 s |
| ステップ 5 | 合計で 8 サイクルの手順 2 ~ 4 を繰り返します(5) | |
| ステップ 6 | 72 °C | 5 分 |
| ステップ 7 | 4 °C | 保持 |
表 2: プロトコルで使用されるインキュベーション手順。(1) すべての手順で使用される異なる蓋温度に注意してください。(2) セル負荷に応じてサイクル総数を設定する: <500 セル負荷の 13 サイクル;500-6,000細胞負荷のための12周期;>6,000 セル荷重の 11 サイクル。(3) cDNA入力に従って合計サイクル番号を設定する: 1-25 ng cDNAの 14-16 サイクル;25-150 ng cDNAのための12-14サイクル;150-500 ng cDNAのための10-12サイクル;500-1,000 ng cDNAのための8-10サイクル;1000-1500 ng cDNAのための6-8周期。(4) cDNA入力に従って合計サイクル番号を設定する:8-12サイクル。(5) cDNA入力に従って合計サイクル番号を設定します:6〜10サイクル。
| 脂質ベースのバーコードオリゴヌクレオチド | |
| アンカー LMO | 5'-TGGAATCTCGGGTGTGTGTGTGTGTGTGTGTCTCTCTCTCTCTCTCTCTCTCT-脂質-3' |
| コアンカー LMO | 5'-脂質-アグガカGCGTGTGTタック-3' |
| バーコード・オリゴ | 5'-CCTGGCACCCGAGAATTCCANNNNNNA30-3' |
| 脂質バーコード添加プライマー | 5'-CTTGGCACCCGAGAATCC-3' |
| RPIXプライマー | 5'-CAAGCAGAAGACガッカガトンンGTGTガクトCC TTGGCACCCGAGATCCA-3' |
| ユニバーサルアダプタプライマー | 5'-AATGATACGGCGACCACCACCGAGATCTCTCTTCTTACACACACAC GCTCTCCGATCT-3' |
| 抗体ベースのバーコードオリゴヌクレオチド | |
| 抗体バーコードオリゴ | 5'-GTGACTGGGAGTTCAGACGTGTCTCTCTCTCTCTNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN NNBAAAAAAAAAAAAAAAAAAAAAAAAAAAAA*A*A-3' |
| HTO添加剤プライマー | 5'-GTGACTGGGGTGTGTGTGTGTGTGTTC-3' |
| ADT添加剤プライマー | 5'-CCTGGCACCCGAGAATCC-3' |
| P5-スマートpcrハイブリッドオリゴ | 5'-AATGATACGGCGACCACCGAGATCTACACGCCTCTCTCTGCGTGGGAA GCAGTGTGTCAACGCAGAGT*A*C-3' |
表3:このプロトコルで使用されるオリゴヌクレオチド配列。N = バーコードまたはインデックス シーケンス。* = ホスホロチオエート結合
ディスカッション
本研究では、単一細胞転写プロファイルを解析するプロトコルを実証した。また、scRNA-Seqワークフローでサンプルを多重化する2つのオプションの方法も提供しています。両方の方法は、様々なラボで実現可能であることが証明され、費用対効果が高く、バッチ効果のない単一細胞実験を実行するためのソリューションを提供した。
プロトコルを使用する際には、注意深く従う必要があるいくつかの手順があります。理想的な単一細胞懸濁液は、生存細胞の90%を持ち、細胞密度も特定の範囲27内である必要があります。細胞凝集体、破片、繊維の存在を最小限に抑えるためには、細胞の良質を得ることが重要です。細胞集合体は、サンプル多重化に悪影響を及ぼし、液滴発生機17を詰まらせる潜在的なリスクを有する。一般的に言えば、30~40μmのセルストレーナーは、解離後にほとんどの細胞が30μm以下に収縮するため、細胞サンプルを保存しながら大きな塊や破片を除去するのに最適です。細胞径が30μmを超える場合は、代わりに単一細胞核を使用することをお勧めします。初期の胚の段階では、すべての種類のマウス細胞の細胞サイズは30μm未満でなければなりません。しかし、後の段階では、心臓の心筋細胞、脳内のニューロン、四肢の筋細胞、および一部の脂肪細胞は30μmを超える細胞サイズを有し得る。
多重化戦略は、費用対効果の高い方法で多数のサンプルを同時に分析する方法を提供します。さらに、複数のサンプルを一緒にプロファイリングすることで、バッチ効果を大幅に回避し、セルダブレットを特定できます。これらの利点は、単一のセルフィールドに非常に魅力的になります。ただし、使用を制限する要因がいくつかあります。1 回の実験でより多くのセルが多重化されるため、セルのダブレット比も増加します。これらのダブレットは、多重バーコードデータを分析することによって識別および除去することができますが、シーケンス読み取りの大きな無駄につながります。さらに、より多くの細胞が一緒にプールされるにつれて、細胞が破壊しやすくなり、周囲のmRNAの増加を引き起こし、細胞と一緒に液滴に捕捉され、検出感度を妨げます。我々は、実験ワークフローまたはバイオインフォマティクス分析パイプラインのさらなる最適化が近い将来、これら2つの問題を解決することを期待している。
開示事項
著者たちは開示する利害の対立を持っていない。
謝辞
ゼブ・J・ガートナー博士のデビッド・M・パターソンとクリストファー・S・マクギニス博士のサンプル・J・ガートナー博士に感謝し、脂質ベースのバーコード試薬の供給と実験ステップとデータ分析に関する提案を行いました。この作品は国立衛生研究所(HL13347202)によって設立されました。
資料
| Name | Company | Catalog Number | Comments |
| 10% Tween-20 | Bio-Rad | 1610781 | |
| 10x Chip Holder | 10x Genomics | 120252 330019 | |
| 10x Chromium Controller | 10x Genomics | 120223 | |
| 10x Magnetic Separator | 10x Genomics | 120250 230003 | |
| 10x Vortex Adapter | 10x Genomics | 330002, 120251 | |
| 10x Vortex Clip | 10x Genomics | 120253 230002 | |
| 4200 TapeStation System | Agilent | G2991AA | |
| Agilent High Sensitivity DNA Kit | Agilent | 5067-4626 | University of Pittsburgh Health Sciences Sequencing Core |
| Barcode Oligo | Integrated DNA Technologies | Single-stranded DNA | 25 nmol |
| Buffer EB | Qiagen | 19086 | |
| CD1 mice | Chales River | Strain Code 022 | ordered pregnant mice |
| Centrifuge 5424R | Appendorf | 2231000214 | |
| Chromium Chip B Single Cell Kit, 48 rxns | 10x Genomics | 1000073 | Store at ambient temperature |
| Chromium i7 Multiplex Kit, 96 rxns | 10x Genomics | 120262 | Store at -20 °C |
| Chromium Single Cell 3' GEM Kit v3,4 rxns | 10x Genomics | 1000094 | Store at -20 °C |
| Chromium Single Cell 3' Library Kit v3 | 10x Genomics | 1000095 | Store at -20 °C |
| Chromium Single Cell 3' v3 Gel Beads | 10x Genomics | 2000059 | Store at -80 °C |
| Collagenase A | Sigma/Millipore | 10103578001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| Collagenase B | Sigma/Millipore | 11088807001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| D1000 ScreenTape | Agilent | 5067-5582 | University of Pittsburgh Health Sciences Sequencing Core |
| DNA LoBind Tube Microcentrifuge Tube, 1.5 mL | Eppendorf | 022431021 | |
| DNA LoBind Tube Microcentrifuge Tube, 2.0 mL | Eppendorf | 022431048 | |
| Dynabeads MyOne SILANE | 10x Genomics | 2000048 | Store at 4 °C, used in Beads Cleanup Mix (Table 1) |
| DynaMag-2 Magnet | Theromo Scientific | 12321D | |
| Ethanol, Pure (200 Proof, anhydrous) | Sigma | E7023-500mL | |
| Falcon 15mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-70C | |
| Falcon 50mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-49A | |
| Fetal Bovine Serum, qualified, United States | Fisher Scientific | 26140079 | Store at -20 °C |
| Finnpipette F1 Multichannel Pipettes, 10-100μl | Theromo Scientific | 4661020N | |
| Finnpipette F1 Multichannel Pipettes, 1-10μl | Theromo Scientific | 4661000N | |
| Flowmi Cell Strainer | Sigma | BAH136800040 | Porosity 40 μm, for 1000 uL Pipette Tips, pack of 50 each |
| Glycerin (Glycerol), 50% (v/v) | Ricca Chemical Company | 3290-32 | |
| HBSS, no calcium, no magnesium | Thermo Fisher Scientific | 14170112 | |
| Human TruStain FcX (Fc Receptor Blocking Solution) | BioLegend | 422301 | Add 5 µl of Human TruStain FcX per million cells in 100 µl staining volume |
| Isopropanol (IPA) | Fisher Scientific | A464-4 | |
| Kapa HiFi HotStart ReadyMix (2X) | Fisher Scientific | NC0295239 | Store at -20 °C, used in Lipid-tagged barcode library mix (Table 1) |
| Lipid Barcode Primer (Multi-seq Primer) | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
| Low TE Buffer (10 mM Tris-HCl pH 8.0, 0.1 mM EDTA) | Thermo Fisher Scientific | 12090-015 | |
| MasterCycler Pro | Eppendorf | 950W | |
| Nuclease-Free Water (Ambion) | Thermo Fisher Scientific | AM9937 | |
| PCR Tubes 0.2 ml 8-tube strips | Eppendorf | 951010022 | |
| Phosphate-Buffered Saline (PBS) 1X without calcium & magnesium | Corning Cellgro | 21-040-CV | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin (alternative to Thermo Fisher product) | Sigma-Aldrich | SRE0036 | |
| Pipet 4-pack (0.1–2.5μL, 0.5-10μL, 10–100μL, 100–1,000μL variable-volume pipettes | Fisher Scientific | 05-403-151 | |
| Selection reagent (SPRIselect Reagent Kit) | Beckman Coulter | B23318 (60ml) | |
| Template Switch Oligo | 10x Genomics | 3000228 | Store at -20 °C, used in Master Mix (Table 1) |
| The antibody based barcoding strategy is also known as Cell Hashing | |||
| The cell browser is Loup Cell Browser | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/visualization/latest/what-is-loupe-cell-browser | |
| The commercial available analysis pipline in step 8.1 is Cell Ranger | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/what-is-cell-ranger | |
| The lipid based barcoding strategy is also known as MULTI-seq | |||
| The well maintained R platform is Seurat V3 | satijalab | https://satijalab.org/seurat/ | |
| TipOne RPT 0.1-10/20 ul XL ultra low retention filter pipet tip | USA Scientific | 1180-3710 | |
| TipOne RPT 1000 ul XL ultra low retention filter pipet tip | USA Scientific | 1182-1730 | |
| TipOne RPT 200 ul ultra low retention filter pipet tip | USA Scientific | 1180-8710 | |
| TotalSeq-A0301 anti-mouse Hashtag 1 Antibody | BioLegend | 155801 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 2 Antibody | BioLegend | 155803 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 3 Antibody | BioLegend | 155805 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TrueSeq RPI primer | Integrated DNA Technologies | Single-stranded DNA | 100 nmol, used in Lipid-tagged barcode library mix (Table 1) |
| Trypan Blue Solution, 0.4% | Fisher Scientific | 15250061 | |
| Trypsin-EDTA (0.25%), phenol red | Fisher Scientific | 25200-056 | |
| Universal I5 | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
参考文献
- Raj, A., Van Den Bogaard, P., Rifkin, S. A., Van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877(2008).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6 (5), 377(2009).
- Li, G., Plonowska, K., Kuppusamy, R., Sturzu, A., Wu, S. M. Identification of cardiovascular lineage descendants at single-cell resolution. Development. 142 (5), 846-857 (2015).
- DeLaughter, D. M., et al. Single-cell resolution of temporal gene expression during heart development. Developmental Cell. 39 (4), 480-490 (2016).
- Li, G., et al. Single cell expression analysis reveals anatomical and cell cycle-dependent transcriptional shifts during heart development. Development. 146 (12), dev173476(2019).
- Meilhac, S. M., Buckingham, M. E. The deployment of cell lineages that form the mammalian heart. Nature Reviews Cardiology. 1, (2018).
- Liu, Z., et al. Single-cell transcriptomics reconstructs fate conversion from fibroblast to cardiomyocyte. Nature. 551 (7678), 100(2017).
- Lafzi, A., Moutinho, C., Picelli, S., Heyn, H. Tutorial: guidelines for the experimental design of single-cell RNA sequencing studies. Nature Protocols. 1, (2018).
- The Tabula Muris Consortium. Single-cell transcriptomics of 20 mouse organs creates a Tabula Muris. Nature. 562 (7727), 367(2018).
- Gawad, C., Koh, W., Quake, S. R. Single-cell genome sequencing: current state of the science. Nature Reviews Genetics. 17 (3), 175(2016).
- Grün, D., van Oudenaarden, A. Design and analysis of single-cell sequencing experiments. Cell. 163 (4), 799-810 (2015).
- Ziegenhain, C., et al. Comparative analysis of single-cell RNA sequencing methods. Molecular cell. 65 (4), 631-643 (2017).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17 (1), 77(2016).
- Islam, S., et al. Characterization of the single-cell transcriptional landscape by highly multiplex RNA-seq. Genome Research. 21 (7), 1160-1167 (2011).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161 (5), 1202-1214 (2015).
- Klein, A. M., et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 161 (5), 1187-1201 (2015).
- Library Prep -Single Cell Gene Expression -Official 10x Genomics Support. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/library-prep (2018).
- McGinnis, C. S., et al. MULTI-seq: sample multiplexing for single-cell RNA sequencing using lipid-tagged indices. Nature Methods. 1, (2019).
- Stoeckius, M., et al. Cell hashing with barcoded antibodies enables multiplexing and doublet detection for single cell genomics. Genome Biology. 19 (1), 224(2018).
- Chan, M. M., et al. Molecular recording of mammalian embryogenesis. Nature. 570 (7759), 77-82 (2019).
- Agilent 4200 TapeStation System. , Agilent. https://www.agilent.com/cs/library/datasheets/public/5991-6029EN.pdf (2019).
- SPRIselect User Guide. , Beckman Coulter. https://research.fhcrc.org/content/dam/stripe/hahn/methods/mol_biol/SPRIselect%20User%20Guide.pdf (2012).
- Qubit 4 Fluorometer User Guide. , Thermo Fisher Scientific. https://assets.thermofisher.com/TFS-Assets/LSG/manuals/MAN0017209_Qubit_4_Fluorometer_UG.pdf (2018).
- Agilent High Sensitivity DNA Kit Guide. , Agilent. https://www.agilent.com/cs/library/usermanuals/public/High%20Sensitivity_DNA_KG.pdf (2016).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411(2018).
- Weber, R. J., Liang, S. I., Selden, N. S., Desai, T. A., Gartner, Z. J. Efficient targeting of fatty-acid modified oligonucleotides to live cell membranes through stepwise assembly. Biomacromolecules. 15 (12), 4621-4626 (2014).
- Single Cell Protocols Cell Preparation Guide. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/sample-prep (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved