Method Article
Analyse du séquençage de l'ARNm à cellule unique multiplexée des cellules embryonnaires de souris
Dans cet article
Résumé
Ici nous avons présenté une méthode multiplexée de séquençage d'ARNm de cellules simples pour profiler l'expression de gène dans les tissus embryonnaires de souris. La méthode de séquençage de l'ARNm à cellule unique à base de gouttelettes (scRNA-Seq) combinée à des stratégies de multiplexage peut dresser le profil des cellules uniques à partir de plusieurs échantillons simultanément, ce qui réduit considérablement les coûts des réactifs et minimise les effets expérimentaux des lots.
Résumé
Le séquençage de l'ARNm à cellule unique a fait des progrès significatifs au cours des dernières années et est devenu un outil important dans le domaine de la biologie du développement. Il a été utilisé avec succès pour identifier les populations de cellules rares, découvrir de nouveaux gènes marqueurs et décoder l'information sur le développement spatial et temporel. La méthode à cellule unique a également évolué de la technologie Fluidigm C1 à base de microfluidique aux solutions à base de gouttelettes au cours des deux à trois dernières années. Ici, nous avons utilisé le cœur comme un exemple pour démontrer comment profiler les cellules de tissu embryonnaire de souris en utilisant la méthode scRNA-Seq basée sur les gouttelettes. En outre, nous avons intégré deux stratégies dans le flux de travail pour profiler plusieurs échantillons dans une seule expérience. En utilisant l'une des méthodes intégrées, nous avons simultanément profilé plus de 9 000 cellules à partir de huit échantillons de cœur. Ces méthodes seront précieuses pour le domaine de la biologie du développement en fournissant un moyen rentable de profiler simultanément des cellules individuelles provenant de différents milieux génétiques, stades de développement ou endroits anatomiques.
Introduction
Le profil transcriptionnel de chaque cellule varie selon les populations cellulaires au cours du développement embryonnaire. Bien que l'hybridation moléculaire in situ unique puisse être utilisée pour visualiser l'expression d'un petit nombre de gènes1, le séquençage de l'ARNm à cellule unique (scRNA-Seq) fournit une approche impartiale pour illustrer les modèles d'expression à l'échelle du génome des gènes dans les cellules individuelles. Après sa première publication en 20092, scRNA-Seq a été appliqué pour étudier plusieurs tissus à plusieurs stades de développement au cours des dernières années3,4,5. De plus, comme l'atlas des cellules humaines a lancé récemment ses projets axés sur le développement, on s'attend à ce que davantage de données sur les cellules individuelles provenant de tissus embryonnaires humains soient générées dans un proche avenir.
Le cœur en tant que premier organe à se développer joue un rôle critique dans le développement embryonnaire. Le cœur se compose de plusieurs types de cellules et le développement de chaque type de cellule est étroitement réglementé temporellement et spatialement. Au cours des dernières années, l'origine et la lignée cellulaire des cellules cardiaques aux premiers stades de développement ont été caractérisées6, qui fournissent un outil de navigation extrêmement utile pour comprendre la pathogénie congénitale des maladies cardiaques, ainsi que pour le développement de méthodes plus avancées technologiquement pour stimuler la régénération cardiomyocyte7.
Le scRNA-Seq a subi une expansion rapide ces dernières années8,9,10. Avec les méthodes nouvellement développées, la conception et l'analyse des expériences à cellule unique est devenue plus réalisable11,12,13,14. La méthode présentée ici est une procédure commerciale basée sur les solutions droplet (voir Tableau des Matériaux)15,16. Cette méthode consiste à capturer des cellules et des ensembles de perles codées à barres uniques dans une gouttelette d'émulsion d'huile-eau sous le contrôle d'un système de contrôleur microfluidique. Le taux de chargement cellulaire dans les gouttelettes est extrêmement faible de sorte que la majorité des émulsions de gouttelettes ne contiennent qu'une seule cellule17. La conception ingénieuse de la procédure provient de la séparation d'une seule cellule en émulsions de gouttelettes se produisant simultanément avec le codage à barres, qui permet l'analyse parallèle des cellules individuelles utilisant l'ARN-Seq sur une population hétérogène.
L'incorporation de stratégies de multiplexage est l'un des ajouts importants au flux de travail traditionnel à cellule unique13,14. Cet ajout est très utile dans le rejet des doublets cellulaires, la réduction des coûts expérimentaux, et l'élimination des effets de lot18,19. Une stratégie de codage à barres basée sur les lipides et une stratégie de codage à barres à base d'anticorps (voir Tableau des matériaux) sont les deux méthodes de multiplexage principalement utilisées. Des codes-barres spécifiques sont utilisés pour étiqueter chaque échantillon dans les deux méthodes, et les échantillons étiquetés sont ensuite mélangés pour la capture d'une seule cellule, la préparation de la bibliothèque et le séquençage. Par la suite, les données de séquençage mises en commun peuvent être séparées en analysant les séquences de codes à barres (Figure 1)19. Cependant, des différences significatives existent entre les deux méthodes. La stratégie de codage à barres à base de lipides est basée sur des oligonucléotides modifiés par les lipides, qui n'ont pas eu de préférences de type cellulaire. Alors que la stratégie de codage à barres à base d'anticorps ne peut détecter que les cellules exprimant les protéines d'antigène19,20. En outre, il faut environ 10 min pour tacher les lipides, mais 40 min pour tacher les anticorps (Figure 1). En outre, les oligonucléotides lipides modifiés sont moins chers que les oligonucléotides conjugués aux anticorps, mais ne sont pas disponibles dans le commerce au moment de la rédaction de cet article. Enfin, la stratégie à base de lipides peut multiplex96 échantillons dans une expérience, mais la stratégie basée sur les anticorps ne peut actuellement multiplex 12 échantillons.
Le nombre de cellules recommandée au multiplexe dans une seule expérience devrait être inférieur à 2,5 x 104, sinon, il conduira à un pourcentage élevé de doublets cellulaires et la contamination potentielle de l'ARNm ambiant. Grâce aux stratégies de multiplexage, le coût de la capture d'une seule cellule, de la production d'ADNc et de la préparation de plusieurs échantillons sera réduit au coût d'un échantillon, mais le coût de séquençage demeurera le même.
Protocole
La procédure animale est conforme au Comité institutionnel de soins et d'utilisation des animaux de l'Université de Pittsburgh (IACUC).
1. Dissection cardiaque embryonnaire de souris et préparation de suspension de cellules simples
REMARQUE : Cette étape peut prendre quelques heures en fonction du nombre d'embryons à disséquer.
- Pour acquérir des cœurs embryonnaires E18.5, euthanasiez une souris CD1 enceinte par administration de CO2. Utilisez un rasoir pour enlever les poils indésirables dans la région abdominale et désinfecter la peau avec 70% d'éthanol.
- Couper la peau de l'abdomen à l'aide de ciseaux stérilisés et disséquer soigneusement les embryons et les mettre rapidement dans la saline froide tamponnée par le phosphate (PBS) sur la glace.
- Isolez soigneusement les cœurs de chaque embryon dans un plat de 10 cm rempli de PBS froid sous un microscope stéréoscopique à l'aide de forceps et de ciseaux.
REMARQUE : Gardez les quatre chambres intactes en disséquant le poumon avec le cœur ensemble et NE pas attraper/tirer directement le cœur avec des instruments chirurgicaux. - Transférer 10 cœurs dans un nouveau plat de 10 cm rempli de PBS froid et micro-disséquer les cœurs dans l'oreillette gauche, l'oreillette droite, le ventricule gauche et le ventricule droit.
REMARQUE : On suppose que cela donne plus de 1 x 106 cellules de chaque échantillon. Nous vous recommandons de commencer avec au moins 1 x 105 cellules par échantillon. - Transférer chacun des 4 tissus de chambre dans un tube de 1,5 ml et les couper en morceaux à l'aide de ciseaux. Centrifugeuse à 300 x g pendant 3 min pour recueillir les tissus.
- Après avoir aspirant le supernatant, ajouter 1 ml de 0,25 % de trypsine/EDTA à chaque tube et incuber dans un bain d'eau de 37 oC pendant 10 min. Pipet de haut en bas en douceur 7-8 fois à l'aide d'une pipette P1000.
- Si le stade embryonnaire est plus ancien que E11.5, ajouter 1 ml de 10 mg/mL de collagène A/B mélange et incuber à 37 oC pendant 10-20 min. Doucement pipet vers le haut et vers le bas jusqu'à ce que la plupart des cellules sont dissociées.
- Transférer les cellules dans un tube de 15 ml et ajouter la solution de sel équilibrée (HBSS) de 8 ml de Hank pour diluer les enzymes. Faites tourner les cellules à 300 x g pendant 5 min. Suspendre les cellules dans 1 ml de PBS et les transférer dans un tube de 1,5 ml. Filtrer les cellules à travers une passoire cellulaire de 40 m.
- Prenez 15 ll de volume de chaque échantillon et mélangez avec la même quantité de 0,04 % de bleu trypan. Chargez ceci sur une chambre de comptage de cellules et comptez les cellules dans un compteur de cellules.
REMARQUE : Pour générer des résultats de haute qualité, il est recommandé de rendre la viabilité cellulaire supérieure à 95 %.
2. Barcoding multiplexing à cellule unique
REMARQUE : Cette étape prend au moins 40 min qui varie en fonction du nombre d'échantillons traités. Une aire de banc propre traitée avec une solution de décontamination RNase est nécessaire pour les étapes de pré-amplification (étape 2.11 à 3.11), et une zone de banc propre séparée est nécessaire pour les étapes de post-amplification (les étapes après 3.11).
-
Procédure de codage à barres à base de lipides (procédure facultative 1)
- Selon la concentration cellulaire, conservez moins de 5 x 105 cellules par échantillon. Assurez-vous que la suspension cellulaire est exempte de débris et d'agrégats cellulaires.
- Préparer une solution de stock d'ancre/code à barres de 2 M et une solution de co-ancrage pour chaque échantillon (tableau 1).
REMARQUE : L'ancre et le co-animateur ont été gentiment doués par le laboratoire Dr Zev J. Gartner. Pour synthétiser ces oligonucléotides modifiés par les lipides, des séquences d'ADN ont été conjuguées avec de l'acide gras sur un support solide et purifiées par une chromatographie liquide à haute performance en phase inversée (HPLC)18,19. - Laver les cellules deux fois avec du PBS et les recueillir à 300 x g pendant 5 min. Suspendre les cellules dans 180 'L de PBS.
- Ajouter 20 l de solution de stock d'ancre/code à barres et pipette de haut en bas doucement pour mélanger. Incuber sur glace pendant 5 min.
- Ajouter 20 l de co-ancre de la solution de bouillon et pipette de haut en bas doucement pour mélanger, puis incuber sur la glace pendant encore 5 min.
- Ajouter 1 ml de PBS froid avec 1% de BSA et centrifugeuse à 300 x g pendant 5 min à 4 oC. Laver au moins 2 fois de plus avec du froid glacé 1% BSA en PBS.
- Combiner tous les échantillons ensemble et filtrer à travers des passoires à cellules de 40 m. Comptez les cellules et maintenez la suspension cellulaire sur la glace à utiliser dans la section 3.
-
Procédure de codage à barres à base d'anticorps (procédure facultative 2)
- Centrifugeuse 1 x 106-2 x 106 cellules pour chaque échantillon (à partir de l'étape 1,8) à 300 x g pendant 5 min et les suspendre dans 100 'L de tampon de coloration (tableau 1) dans des tubes de liaison bas de 1,5 mL.
- Ajouter 10 réagents de blocage Fc ll et couver pendant 10 min à 4 oC.
- Préparer les anticorps (voir Tableau des matériaux)en centrifugeant à 14 000 x g pendant 10 min à 2-8 oC.
- Ajouter 1 g de chaque anticorps conjugué à l'oligo à 50 l de tampon de coloration cellulaire pour faire une solution de coloration desanticorps 20. Ajoutez une solution de coloration d'anticorps à chaque tube d'échantillon. Incuber pendant 30 min à 4 oC.
- Laver les cellules 3 fois avec 1 ml de PBS, faire tourner pendant 5 min à 350 x g à 4 oC.
- Mettre en commun tous les échantillons aux proportions désirées dans 1 ml de tampon de coloration, faire tourner pendant 5 min à 350 x g à 4 oC.
- Resuspendre les cellules en PBS à concentration appropriée (jusqu'à 1 500 cellules/L) et filtrer les cellules à travers une passoire cellulaire de 40 m. Passez immédiatement à l'étape suivante.
3. La génération de gouttelettes et la transcription inversée d'ARnm
REMARQUE: Cette étape prend environ 90 min pour une réaction multiplexée.
- Équilibrez les perles de gel (voir Tableau des matériaux)à température ambiante pendant 30 min. Sortez les réactifs du kit de perles de gel en émulsion (GEMs) (voir Tableau des matériaux)et gardez-les à leur température indiquée.
- Assembler la puce B dans un support de puce (voir Tableau des matériaux).
- Distribuer 75 l de solution de glycérol à 50 % dans les puits inutilisés de la rangée 1; 40 l dans la rangée 2; 280 l dans la rangée 3. N'ajoutez pas de glycérol dans les puits de récupération de la rangée supérieure de la puce.
- Préparer le mélange maître sur glace selon le tableau 1. Ajoutez le volume approprié de suspension de cellules et de l'eau nucléane-libre pour maîtriser le mélange selon une table de calculatrice de volume de suspension de cellules17 et pipette doucement le mélange. Distribuer 75 l de mélange cellulaire dans le centre inférieur de l'échantillon bien dans la rangée 1 sans introduire de bulles.
- Vortex les perles de gel pour 30 s à l'aide d'un adaptateur vortex et lentement distribuer 40 L de perles de gel dans le centre inférieur de la perle de gel bien dans la rangée 2 sans introduire de bulles.
REMARQUE: Il est essentiel d'attendre 30 s entre l'ajout de cellules et de perles de gel pour éviter l'échec de mouillage. - Pour le puits de cloisonnement dans la rangée 3, distribuer 280 l d'huile de partitionnement à travers la paroi latérale du puits.
REMARQUE : Le chargement de moins de 270 L d'huile de partitionnement entraînera une génération anormale de GEM. - Fixez le joint sur la puce, ne pressez pas sur le joint et gardez-le à l'horizontale pour éviter de mouiller le joint.
- Chargez la puce assemblée avec le joint dans le contrôleur de chrome et exécutez le programme de cellule unique B de chrome (voir tableau des matériaux),passez immédiatement à l'étape suivante lorsque le programme se termine.
- Sortez la puce et jetez le joint. Repliez le couvercle pour exposer les puits à 45 degrés, vérifiez le niveau de liquide pour vous assurer qu'il n'y a pas de sabots.
- Aspirez lentement 100 L de GEM des points les plus bas du puits de récupération et vérifiez l'uniformité des GEM. Distribuez les GEM dans un nouveau tube de réaction en chaîne de polymérase (PCR) sur la glace avec les pointes de pipette contre la paroi latérale du tube.
REMARQUE : Si l'on observe une couche aqueuse excessive, il est suggéré de répréparer les échantillons. Surtout, prenez une photo du mélange lorsque les GEM sont encore dans les pointes de pipette. Cette image peut indiquer s'il y a une défaillance de mouillage, des GEM partiellement émulsifiés, et des sabots de réactif. La photographie peut également être utilisée comme preuve pour obtenir le remboursement de la compagnie de réactif avec des réactifs et des puces de remplacement. - Mettre le tube dans un cycleur thermique et effectuer la procédure de transcription inverse (tableau 2).
REMARQUE : Arrêtez-vous ici ou passez à l'étape suivante. Le produit PCR peut être stocké à -20 oC pendant une semaine.
4. amplification de cDNA
REMARQUE: Cette étape prend environ 150 min.
- Nettoyage de la transcription inverse de cellule unique
- Sortez les réactifs d'amplification de l'ADNc du kit GEMs (voir Tableau des matériaux)et gardez-les à leur température indiquée.
- Ajouter 125 l'agent de récupération à l'échantillon à température ambiante pour acquérir un mélange biphasique. Aucun liquide opaque ne doit être observé et éviter la pipetage ou le vortexing du mélange.
- Après avoir attendu 60 s, retirez lentement 125 L d'agent de récupération du fond du tube.
- Vortex les perles magnétiques (voir Tableau des matériaux) à fond pour 30 s et l'utiliser immédiatement pour préparer le mélange de nettoyage des perles (tableau 1). Les réactifs doivent être ajoutés de façon séquentielle comme indiqué.
- Vortex les perles de nettoyage mélange et ajouter 200 L à l'échantillon. Pipette le mélange 10 fois puis l'incuber pendant 10 min à température ambiante.
- Ajouter les réactifs de façon séquentielle comme indiqué dans le tableau 1 pour préparer la solution d'élution et l'élinguer comme suit.
- Placez les échantillons sur l'aimant (position élevée) (voir Tableau des matériaux)jusqu'à ce que la solution se dégage, puis retirez le supernatant.
- Ajouter 200 l d'éthanol à 80 % au granule. Attendez 30 s, puis retirez l'éthanol.
- Répétez l'étape 4.1.6.2 pour un autre 2 fois. Centrifugeuse brièvement et placer sur l'aimant (position basse). Retirer soigneusement le reste de l'éthanol et sécher à l'air pendant moins de 2 minutes.
REMARQUE : NE PAS dépasser le séchage à l'air au-delà de 2 min, sinon l'efficacité de l'élution diminuera. - Retirez l'échantillon de l'aimant. Ajouter 35,5 L de solution d'élution et de pipette pour mélanger 15 fois. Incuber 2 min à température ambiante.
- Placez l'échantillon sur l'aimant (position élevée) jusqu'à ce que la solution se dégage. Transférer 35 ll de l'échantillon sur une nouvelle bande de tube.
REMARQUE : Cette procédure de purification est également utilisée dans les étapes 4.2.1.6, 4.2.2.3, 5.8, 6.1.10, et 6.2.6. Faites attention à la concentration de perles magnétiques et au volume d'eau tampon/ultrapure EB utilisée pour éliuter les échantillons à chaque étape.
- Quantifier la taille, la concentration et l'intégrité de l'ADNc élugé à l'aide d'un instrument automatisé d'électrophorèse21 (voir tableau des matériaux) (Figure 2).
- Amplification de l'ADNc
-
amplification de l'ADNc à l'aide de la stratégie de codage à barres à base de lipides (procédure facultative 1)
- Préparer le mélange de réaction d'amplification (tableau 1) sur la glace.
- Ajouter le mélange de réaction d'amplification à 35 'L d'échantillons d'ADNc (à partir de l'étape 4.1.6.7). Pipette le mélange, et centrifugeuse brièvement. Incuber le mélange dans un cycleur thermique suivant la procédure d'amplification de l'ADNc (tableau 2).
- Après le vortex, ajouter 120 l de réactif sélecteur et 100 l d'eau ultrapure à 100 l d'échantillon pour acquérir une concentration de 0,6 x de réactif sélecteur (voir Tableau des matériaux). Pipette le mélange pour 15 fois.
- Incuber 5 min à température ambiante, puis placer l'échantillon sur l'aimant jusqu'à ce que les solutions deviennent claires.
REMARQUE : L'ADNc endogène dans la fraction de perles et l'ADNc à code à barres multiplexing est dans le supernatant. - Transférer le supernatant dans un tube de liaison basse de 1,5 ml pour la construction de la bibliothèque de cDNA à code à barres à l'étape 6.1.
- Nettoyez l'ADNc endogène en suivant les étapes en 4.1.6 et élichez-les avec 40 l'adhérence EB.
- Exécuter 1 ldde d'échantillon purifié d'ADNc sur un instrument automatisé d'électrophoresis (voir Tableau des matériaux) pour analyser/quantifier l'ADNc.
- Aliquot 10 l de cDNA dans un nouveau tube PCR pour la construction de la bibliothèque endogène.
REMARQUE : Arrêtez-vous ici ou passez à l'étape suivante. L'échantillon restant peut être stocké dans -20 oC pendant jusqu'à 4 semaines pour générer des bibliothèques supplémentaires si nécessaire.
-
amplification de cDNA dans la stratégie de codage à barres basée sur l'anticorps (procédure facultative 2)
- Ajouter 2 pmol d'amorce additif HTO et ADT et 15 l d'amorces d'ADNc à 50 ll de mélange de réaction d'amplification (tableau 1) et effectuer l'amplification de l'ADNc avec la procédure d'amplification de l'ADNc (tableau 2).
- Utilisez 0.6x sélectionnez le réactif pour séparer l'ADNc endogène (fraction de perles) et le code à barres multiplexant cDNA (dans supernatant). N'oubliez pas d'enregistrer le supernatant pour effectuer la construction de la bibliothèque de codage à barres à l'étape 6.2.
- Purifez et élinchez la transcription endogène cDNA en suivant les étapes dans 4.1.6, effectuer le contrôle de la qualité (QC) des bibliothèques et aliquot 10 L dans un nouveau tube PCR pour la construction de bibliothèque endogène.
-
amplification de l'ADNc à l'aide de la stratégie de codage à barres à base de lipides (procédure facultative 1)
5. Préparation endogène de la bibliothèque de transcription
REMARQUE: Cette étape prend environ 120 min.
- Maintenir les réactifs de construction de la bibliothèque d'expression génique du kit de bibliothèque (voir tableau des matériaux) à leur température indiquée, respectivement.
- Préparer le mélange de fragmentation (tableau 1) sur glace, pipette à mélanger et centrifugeuse brièvement.
- Ajouter un tampon EB de 25 l l à l'échantillon d'ADNc purifié de 10 l (à partir de l'étape 4.2.1.8 ou 4.2.2.3) puis ajouter le mélange de fragmentation de 15 l nouvellement préparé à l'échantillon, pipette le mélange 15 fois sur la glace et centrifugeuse brièvement.
- Transférer l'échantillon dans un cycleur thermique pré-refroidi et lancer le programme PCR pour la fragmentation, la réparation de fin et le suivi A (tableau 2).
- Vortex le réactif de sélection pour suspendre les perles magnétiques et utiliser successivement 0.6x et 0.8x sélectionner les réactifs pour faire une sélection de taille double face selon le guide utilisateur17,22. Utilisez 50 ll de tampon EB pour élituer l'ADN.
- Préparer le mélange de ligature adaptateur (tableau 1), puis pipette le mélange à fond et centrifugeuse brièvement.
- Ajouter 50 ll de mélange de ligation adaptateur à 50 l d'échantillon, mélanger à nouveau la pipette pendant 15 fois et centrifugeuse brièvement. Effectuer la ligature de l'adaptateur selon le protocole dans un cycleur thermique (tableau 2).
- Utilisez 0.8x sélectionnez le réactif pour purifier le produit de ligature et élifier l'échantillon purifié avec 30 ll de tampon EB (voir l'étape 4.1.6).
- Préparer le mélange PCR de l'indice d'échantillon (tableau 1) et ajouter 60 l à l'échantillon purifié. Ajouter 10 l'index de l'échantillon à l'échantillon, mélanger les pipettes vers le haut et vers le bas pendant 5 fois et la centrifugeuse brièvement, incuber dans un cycleur thermique suivant le protocole d'indice d'échantillon (tableau 2).
REMARQUE : Arrêtez-vous ici ou passez à l'étape suivante. Si plus d'un puits est utilisé, choisissez un indice d'échantillon spécifique (voir Tableau des matériaux) pour chaque puits. N'oubliez pas d'enregistrer l'ID d'index utilisé pour chaque puits et de ne pas vous chevaucher dans une exécution de séquençage multiplexée. - Utilisez successivement 0.6x et 0.8x sélectionner des réactifs pour faire une sélection de taille redoublement pour acquérir 35 L de l'ADN de bibliothèque cellulaire endogène purifié17.
- QC la bibliothèque cellulaire endogène avant le séquençage (Figure 2).
6. Préparation des bibliothèques d'ADNC de code à barres de multiplexage
REMARQUE: Cette étape prend au moins 120 min.
-
Génération de bibliothèques de codes à barres pour la stratégie de multiplexage à base de lipides (procédure facultative 1)
- Ajouter 520 l de réactifs sélectionnés et 360 l d'isopropanol pour échantillonner l'adincadané du code-barres à partir de l'étape 4.2.1.5 pour obtenir une concentration de réactif sélecteur de 3,2x. Pipette le mélange 10 fois et couver à température ambiante pendant 5 min.
- Placez le tube sur un support magnétique et attendez que la solution se dégage. Ensuite, jetez le supernatant.
- Utilisez 500 l'éthanol de 80 % pour laver les perles deux fois sur un aimant et attendez 30 s après chaque lavage.
- Centrifuger brièvement les perles et les placer sur un aimant. Retirer le reste de l'éthanol à l'eau à l'œil d'une micropipette P10 et laisser les perles pendant 2 min.
- Retirez le tube de la grille d'aimant et suspendez les perles dans un tampon EB de 50 l. Pipet up and down pour bien mélanger. Incuber à température ambiante pendant 2 min.
- Remettre le tube à un aimant et attendre que la solution se dégage. Transférer le supernatant (exemple de code à barres cDNA) dans un nouveau tube PCR. Veillez à ne pas transférer de perles.
- Quantifier la concentration du code-barres de l'échantillon cDNA23.
- Préparer le mélange de bibliothèque de codes à barres lipidiques (tableau 1), ajouter 3,5 ng d'ADNc purifié à code à barres (à partir de l'étape 6.1.6) et de l'eau sans nucléane pour un volume total de 50 'l.
- Gardez-le dans un cycleur thermique suivant la bibliothèque de codes à barres à base de lipides PCR (tableau 2).
- Utilisez 1.6x sélectionnez le réactif pour purifier le produit PCR et élifier l'ADN avec 25 ll de tampon EB (voir l'étape 4.1.6).
- Quantifier la concentration de la bibliothèque à l'aide d'une méthode d'analyse de l'ADN à haute sensibilité24 à partir de la dilution initiale de 1:5 (figure 2).
-
Génération de bibliothèques de codes à barres pour la stratégie de multiplexage basée sur les anticorps (procédure facultative 2)
- Ajoutez un volume de réaction supplémentaire de 1,4x de réactif sélectionné au supernatant contenant des codes-barres d'échantillon acquis à partir de l'étape 4.2.2.3 pour obtenir un rapport de réactif 2x sélectionné.
- Laver les perles avec 80% d'éthanol en suivant les étapes en 4.1.6 et éliper le cDNA à code à barres avec de l'eau ultrapure.
- Effectuer le protocole de sélection avec 2x sélectionner réactif pour une deuxième fois et elute à l'aide d'eau ultrapure.
- Préparer le mélange de bibliothèque de codes à barres d'anticorps (tableau 1), et ajouter 45 l d'ADNc purifié à code à barres à partir de la dernière étape.
- Incuber dans un cycleur thermique suivant la bibliothèque de codes-barres d'anticorps PCR (tableau 2)20.
- Utilisez 1,6x pour purifier le produit PCR et élifier l'échantillon purifié avec 30 ll d'eau ultrapure (voir l'étape 4.1.6).
7. Séquençage de la bibliothèque
REMARQUE : Plusieurs plates-formes de séquençage de nouvelle génération telles que HiSeq 4000 et NovaSeq peuvent être utilisées pour séquencer les bibliothèques de transcription endogènes et les bibliothèques de codes à barres multiplexantes.
- Utilisez une plate-forme de séquençage de nouvelle génération de choix pour séquencer les bibliothèques de transcription endogènes et les bibliothèques de codes à barres multiplexantes.
- Diluer les bibliothèques selon les recommandations d'un expert d'une entreprise de séquençage ou d'un établissement de séquençage. Un minimum de 20 000 lectures par cellule est recommandé pour la bibliothèque de transcriptions endogènes et 3 000 lectures pour les bibliothèques de codes à barres.
8. Analyse des données
REMARQUE : De-multiplex les données de séquençage à l'aide de la ressource basée sur le cloud BaseSpace ou en exécutant le paquet bcl2fastq sur un serveur UNIX.
-
Analyse endogène des données de transcriptome
- Avec les données fastq générées à partir d'un logiciel de multiplexage, exécutez "mkfastq" sur le pipeline d'analyse de données disponible dans le commerce (voir Tableau des matériaux) pour démultiplexer chaque code à barres GEMs.
- Exécutez « compter » pour effectuer l'alignement, le filtrage, le comptage des codes à barres et le comptage UMI.
- En option, exécutez «aggr» pour agréger plusieurs voies de séquençage à partir d'une seule expérience.
- Utilisez le « navigateur cellulaire » (voir tableau des matériaux) pour visualiser les données, regrouper les cellules, identifier les gènes exprimés différemment et générer des parcelles de cartes thermiques tSNE ou d'expression génique.
- En option, utilisez une plate-forme R25 bien entretenue (voir Tableau des matériaux) pour normaliser et mettre à l'échelle les données, identifier les gènes exprimés différemment et générer des parcelles tSNE/UMAP et des cartes thermiques d'expression génique (figure 3).
-
Analyse multiplexée des données de code à barres
-
Analyse des données issues de la stratégie de codage à barres à base de lipides
- Utilisez le pipeline d'analyse de données disponible dans le commerce ou le paquet deMULTIplex R (https://github.com/chris-mcginnis-ucsf/MULTI-seq) pour convertir l'exemple de codes à barres fichiers FASTQ en une matrice de comptage UMI de code-barres.
- Chargez la matrice de comptage UMI de code à barres ainsi que les données endogènes de transcriptome à une plate-forme R -(voir tableau des matériaux) pour l'analyse d'intégration (Figure 3).
-
Analyse des données issues de la stratégie de codage à barres basée sur les anticorps
- Utilisez le « compte » du pipeline d'analyse de données disponible dans le commerce pour cartographier les codes-barres en fournissant le fichier CSV de la bibliothèque et le fichier de référence CSV.
- Chargez la matrice de code-barres de fonctionnalité unifiée de sortie, qui contient des comptes d'expression génique à côté des comptes de code à barres de fonctionnalité pour chaque code-barres de cellule, à une plate-forme R-basée pour l'analyse en aval.
-
Analyse des données issues de la stratégie de codage à barres à base de lipides
Résultats
Dans cette étude, nous avons utilisé le cœur embryonnaire de souris comme exemple pour montrer comment le séquençage multiplexé d'ARNm à cellule unique a été exécuté pour traiter les différents échantillons des parties séparées d'un organe simultanément. Les coeurs de souris CD1 d'E18.5 ont été isolés et disséqués dans l'oreillette gauche (LA), l'oreillette droite (RA), le ventricule gauche (LV) et le ventricule droit (RV). Les cellules auriculaires et ventriculaires ont ensuite été codées indépendamment à l'aide d'une procédure de codage à barres à base de lipides et mélangées ensemble avant la génération et la transcription inversée des GEM. L'aperçu schématique est indiqué dans la figure 1. Nous avons quantifié la concentration d'ADNc avant la construction de la bibliothèque (Figure 2A). Une des distinctions dans l'exécution de scRNA-Seq multiplexé de la norme scRNA-Seq est que la bibliothèque endogène de cDNA et la bibliothèque de cDNA de code à barres d'échantillon ont été acquises séparément après amplification et purification de cDNA (étape 4.2.1 et 4.2.2.2). Les deux bibliothèques ont également été qualifiées dans notre expérience (Figure 2B,C). Le séquençage de la prochaine génération et l'analyse des données ont été effectués, suivis de la construction de la bibliothèque et du QC.
Nous avons utilisé la plate-forme HiSeqX pour séquencer les deux bibliothèques dans la même voie de séquençage. Avec les données de séquençage, nous avons d'abord séparé les données de transcription endogènes et les données de code à barres à l'aide du programme BaseSpace. Ensuite, nous avons analysé l'expression du code à barres dans chaque cellule unique et trouvé 8 groupes de cellules uniques qui expriment uniquement un type de code à barres, représentant des cellules de 8 échantillons différents (Figure 3A). En outre, nous avons également constaté que certaines cellules n'expriment aucun code-barres, que nous avons défini comme des cellules négatives, et certaines cellules expriment deux codes-barres différents, qui représentent des doublets (Figure 3B). En résumé, nous avons constaté qu'environ 70% des cellules sont des singlets, 25% des cellules sont négatives et 5% des cellules sont des doublets.
Avec les cellules singlet, nous pouvons effectuer d'autres analyses en aval pour comprendre l'hétérogénéité cellulaire et les règlements moléculaires. Les analyses potentielles peuvent être l'annotation de type cellulaire (figure 4A),l'identification nouvelle/rare de type de cellule (figure 4B), l'analyse comparative de zone anatomique (figure 4C), et l'analyse de voie d'ontologie génique telle que les séparations de phase de cycle cellulaire (figure 4D).

Figure 1 : Flux de travail de séquençage du séquençage de l'ARNm à cellule unique multiplexée. Les coeurs embryonnaires de jour 18.5 d'étape ont été analysés utilisant une procédure multiplexed de séquençage simple de cellule de gouttelette. RT - transcription inversée. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Résultats du QC représentatif à différentes étapes. (A) Analyse QC de l'ADNc à partir de l'étape 4.1.7. La taille du fragment cible est de 200 à 9000 bp. (B) Bibliothèque endogène et (C) bibliothèque de codes à barres ont été analysés avec un instrument automatisé d'électrophorèse. La taille du fragment cible pour la bibliothèque endogène est de 300-600 pb, et la taille de l'ADN de la bibliothèque de codes à barres est d'environ 172 bp. S'il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
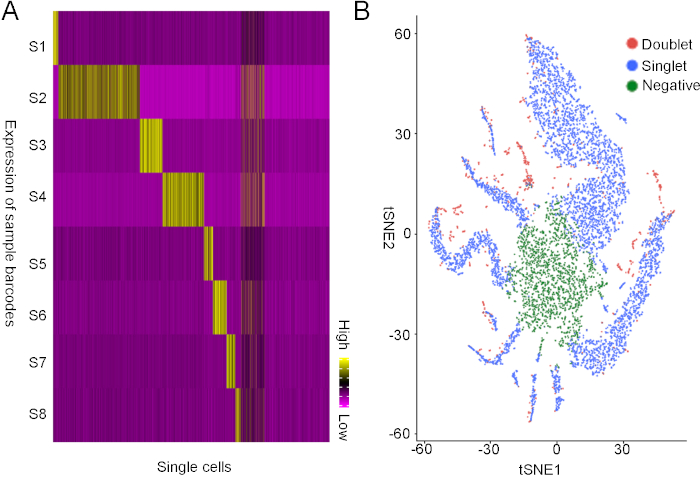
Figure 3 : Multiplexixer les données de séquençage de la stratégie de codage à barres basée sur les lipides. (A) Analyse non supervisée de l'expression du code-barres. L'axe X représente les cellules simples, et l'axe y représente les codes-barres. Chacune des 8 populations de cellules individuelles a été identifiée pour exprimer de façon unique l'un des 8 codes-barres. Notez que certaines cellules expriment plus d'un code-barres, et certaines cellules n'expriment pas de codes-barres. (B) t-SNE parcelle des cellules singlet, les cellules de doublet, et les cellules négatives. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Analyse avancée des données transcriptionnelles d'une seule cellule. (A-D) Les données à cellule unique peuvent être analysées de différentes façons pour comprendre l'hétérogénéité cellulaire et les voies moléculaires. Nous avons énuméré plusieurs applications ici comme exemples. Les cellules simples ont été chargées dans un paquet R pour identifier les types de cellules (A), les populations de cellules rares (B), les zones anatomiques cellulaires (C), et les phases du cycle cellulaire (D). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| Nom du mélange | Composition |
| Mélange de collagène | 10 mg/mL de collagène A et 10 mg/mL de collagène B, dissous dans HBSSMD avec 40 % de FBS. |
| 2 M Solution de stock Anchor/Barcode | Mélanger l'ancre de 50 M et le brin de code à barres de 10 M m en 1:1 dans le PBS (sans FBS ou BSA) pour un volume total de 25 l. |
| 2 M Co-Anchor solution d'actions | Diluer 1 L 50 M Co-Anchor avec 24 L PBS (sans FBS ou BSA). |
| Tampon de coloration | PBS contenant 2% BSA, 0.01% Tween 20 |
| Mélange de maître | 20 L RT Reagent, 3,1 Oligo, 2 L Agent de réduction B, 8,3 L RT Enzyme C. |
| Mélange de nettoyage de perles | 182 'L Cleanup Buffer, 8 'L Selection Reagent, 5 'L Reducing Agent B, 5 'L Nuclease-free Water. |
| Mélange de réaction d'amplification | 1 L de 10 apprêt additif sénéende marqué par les lipides, 15 apprêt à l'adoxyde cDNA L, 50 'L Amp Mix |
| Solution d'élution | 98 L Buffer EB, 1 'L 10% Tween 20, 1 'L Reducing Agent B. |
| Mélange de fragmentation | 5 tampon de fragmentation ll, 10 'L Enzymatique de fragmentation. |
| Mélange de ligation adaptateur | 20 l Ligation Buffer, 10 'L DNA Ligase, 20 'L Adaptor Oligos. |
| Mélange PCR d'indice d'échantillon | 50 l Amp Mix, 10 'L SI Primer |
| Mélange de bibliothèque de code à barres de lipide | 26,25 l de mix maître Hot Start de 2 degrés, 2,5 l de 10 apprêt RPIX M, 2,5 L de 10 M TruSeq Universal Adaptateur amorce (voir tableau des matériaux) |
| Mélange de bibliothèque de code-barres d'anticorps | 50 l de mix maître Hot Start, 2,5 l d'amorce RPIX de 10 M, 2,5 L de 10 oligo hybride p5-pcr |
Tableau 1 : Les mélanges de réactifs utilisés dans le protocole.
| Procédure d'incubation | Température(1) | Temps |
| GEM-RT Incubation | Température du couvercle 53 oC | |
| Étape 1 | 53 oC | 45 min |
| Étape 2 | 85 oC | 5 min |
| Étape 3 | 4 oC | Tenir |
| 10x Amplification de l'ADNC génomique | Température du couvercle 105 oC | |
| Étape 1 | 98 oC | 3 min |
| Étape 2 | 98 oC | 15 s |
| Étape 3 | 63 oC | 20 s |
| Étape 4 | 72 oC | 1 min |
| Étape 5 | Répéter les étapes 2 à 4 pour 12 cycles au total (2) | |
| Étape 6 | 72 oC | 1 min |
| Étape 7 | 4 oC | Tenir |
| Construction de la bibliothèque | Température du couvercle 65 oC | |
| Bloc pré-cool | 4 oC | Tenir |
| Fragmentation | 32 oC | 5 min |
| Réparation de fin et A-tailing | 65 oC | 30 min |
| Tenir | 4 oC | Tenir |
| Ligation d'adaptateur | Température du couvercle à 30 oC | |
| Étape 1 | 20 oC | 15 min |
| Étape 2 | 4 oC | Tenir |
| Exemple d'index PCR | Température du couvercle 105 oC | |
| Étape 1 | 98 oC | 45 s |
| Étape 2 | 98 oC | 20 s |
| Étape 3 | 54 oC | 30 s |
| Étape 4 | 72 oC | 20 s |
| Étape 5 | Répéter les étapes 2 à 4 pour 12 cycles au total (3) | |
| Étape 6 | 72 oC | 1 min |
| Étape 7 | 4 oC | Tenir |
| Bibliothèque de codes à barres lipidiques PCR | ||
| Étape 1 | 95 oC | 5 min |
| Étape 2 | 98 oC | 15 s |
| Étape 3 | 60 oC | 30 s |
| Étape 4 | 72 oC | 30 s |
| Étape 5 | Répéter les étapes 2 à 4 pour 10 cycles au total (4) | |
| Étape 6 | 72 oC | 1 min |
| Étape 7 | 4 oC | Tenir |
| Bibliothèque de codes à barres d'anticorps PCR | ||
| Étape 1 | 95 oC | 3 min |
| Étape 2 | 95 oC | 20 s |
| Étape 3 | 60 oC | 30 s |
| Étape 4 | 72 oC | 20 s |
| Étape 5 | Répéter les étapes 2 à 4 pour 8 cycles au total (5) | |
| Étape 6 | 72 oC | 5 min |
| Étape 7 | 4 oC | Tenir |
Tableau 2 : Procédure d'incubation utilisée dans le protocole. (1) Faites attention à la température différente du couvercle utilisée dans chaque procédure. (2) Définir les nombres totaux de cycle en fonction de la charge cellulaire : 13 cycles pour la charge de cellules de 500 lt; 12 cycles pour une charge de 500 à 6 000 cellules; 11 cycles pour la charge cellulaire de 6 000 euros. (3) Définir les nombres totaux de cycle selon l'entrée de cDNA : 14-16 cycles pour 1-25 ng cDNA ; 12-14 cycles pour 25-150 ng cDNA; 10-12 cycles pour 150-500 ng cDNA; 8-10 cycles pour 500-1000 ng cDNA; 6-8 cycles pour 1000-1500 ng cDNA. (4) Définir les nombres totaux de cycle en fonction de l'entrée de l'ADNc : 8-12 cycles. (5) Définir les nombres totaux de cycle en fonction de l'entrée de l'ADNc : 6-10 cycles.
| Code à barres à base de lipides Oligonucleotides | |
| Ancre LMO | 5'-TGGAATTCCTGgGTGCCAGGGTAACGATCCAGCTGTCACT-Lipid-3' |
| Co-Anchor LMO | 5'-Lipid-AGTGACAGCTGGATCGTTAC-3' |
| Code-barres Oligo | 5'-CCTTGGCACCCGAGATCCANNNNNNNNNNNNNNA30-3' |
| Lipid barcoding Additive Primer | 5'-CTTGGCACCCGAGAATTCC-3' |
| RPIX Amorce | 5'-CAAGCAGAAGAAGACGGCATACGAGATNNNNNNNGTGACTGGAGTTCC TTGGCACCCGAGAATTCCA-3' |
| Apprêt adaptateur universel | 5'-AATGATACGGCGACCACCGAGATCTACACTTTTCCCTACACGAC GCTCTTCCGATCT-3' |
| Oligonucléotides à barres à base d'anticorps | |
| Oligo de codage à barres d'anticorps | 5'-GTGACTGGAGTCAGACGTGTGCTTTCcGATCTNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN NNBAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA-A-3' |
| Amorce additif HTO | 5'-GTGACTGGAGTCAGACGTGTGCTC-3' |
| ADT additif ADT Aprimeur | 5'-CCTTGGCACCCGAGAATTCC-3' |
| Oligo hybride P5-smart-pcr | 5'-AATGATACGGCGACCACCGAGATCTACACGCCTGTCCGCGGAA GCAGTGGTATCAACGCAGAGT-A-C-3' |
Tableau 3 : Séquences d'oligonucléotide utilisées dans ce protocole. N - Code à barres ou séquence d'index; - Lien phosphorothioate
Discussion
Dans cette étude, nous avons démontré un protocole pour analyser des profils transcriptionnels d'une seule cellule. Nous avons également fourni deux méthodes facultatives aux échantillons de multiplexe dans le flux de travail scRNA-Seq. Les deux méthodes se sont avérées réalisables dans divers laboratoires et ont fourni des solutions pour exécuter une expérience de cellule unique rentable et sans effet de lot18,26.
Il y a quelques étapes qui doivent être suivies avec soin lors de l'adoption du protocole. Une suspension idéale de cellule unique devrait avoir 'gt;90% des cellules viables et la densité cellulaire devrait également être dans une gamme spécifique27. Il est essentiel d'obtenir une bonne qualité des cellules pour minimiser la présence d'agrégats cellulaires, de débris et de fibres. Les agrégats cellulaires ont un impact négatif sur le multiplexage de l'échantillon et ont un risque potentiel d'obstruer la machine génératrice de gouttelettes17. D'une manière générale, une passoire à cellules de 30 à 40 m est idéale pour enlever les gros touffes et les débris tout en préservant les échantillons cellulaires, car la plupart des cellules rétréciront en dessous de 30 m après la dissociation. Il est recommandé d'utiliser des noyaux à cellule unique si le diamètre de la cellule est supérieur à 30 m. Au début de l'embryon, la taille des cellules pour tous les types de cellules de souris devrait être inférieure à 30 m. Cependant, à des stades ultérieurs, les cardiomyocytes dans le cœur, les neurones dans le cerveau, les cellules musculaires dans les membres, et certaines cellules adipeuses peuvent avoir une taille cellulaire supérieure à 30 m. La taille des cellules devrait être mesurée pour ces types de cellules avant de commencer les expériences à cellule unique.
Les stratégies de multiplexage permettent d'analyser simultanément un grand nombre d'échantillons de manière rentable. En outre, en profilant plusieurs échantillons ensemble, nous pouvons éviter de manière significative les effets de lot et identifier les doublets cellulaires. Ces avantages seront très attrayants pour le champ de cellules uniques. Cependant, certains facteurs peuvent limiter leur utilisation. Comme plus de cellules sont multiplexées dans une seule expérience, le rapport de doublet de cellules augmentera également. Bien que ces doublets puissent être identifiés et supprimés en analysant les données de code à barres multiplexants, cela conduira à un grand gaspillage de lectures de séquençage. En outre, comme plus de cellules sont regroupées, les cellules sont plus faciles à briser et provoquer une augmentation de l'ARNm ambiant, qui sera capturé dans des gouttelettes avec des cellules et interférer avec la sensibilité de détection. Nous nous attendons à ce qu'une optimisation plus poussée du flux de travail expérimental ou du pipeline d'analyse bioinformatique résoudra ces deux problèmes dans un proche avenir.
Déclarations de divulgation
Les auteurs n'ont aucun conflit d'intérêts à divulguer.
Remerciements
Nous remercions David M. Patterson et Christopher S. McGinnis du laboratoire Dr Zev J. Gartner pour leur aimable approvisionnement en réactifs à barres à base de lipides et leurs suggestions sur les étapes expérimentales et l'analyse des données. Ce travail a été fondé par les National Institutes of Health (HL13347202).
matériels
| Name | Company | Catalog Number | Comments |
| 10% Tween-20 | Bio-Rad | 1610781 | |
| 10x Chip Holder | 10x Genomics | 120252 330019 | |
| 10x Chromium Controller | 10x Genomics | 120223 | |
| 10x Magnetic Separator | 10x Genomics | 120250 230003 | |
| 10x Vortex Adapter | 10x Genomics | 330002, 120251 | |
| 10x Vortex Clip | 10x Genomics | 120253 230002 | |
| 4200 TapeStation System | Agilent | G2991AA | |
| Agilent High Sensitivity DNA Kit | Agilent | 5067-4626 | University of Pittsburgh Health Sciences Sequencing Core |
| Barcode Oligo | Integrated DNA Technologies | Single-stranded DNA | 25 nmol |
| Buffer EB | Qiagen | 19086 | |
| CD1 mice | Chales River | Strain Code 022 | ordered pregnant mice |
| Centrifuge 5424R | Appendorf | 2231000214 | |
| Chromium Chip B Single Cell Kit, 48 rxns | 10x Genomics | 1000073 | Store at ambient temperature |
| Chromium i7 Multiplex Kit, 96 rxns | 10x Genomics | 120262 | Store at -20 °C |
| Chromium Single Cell 3' GEM Kit v3,4 rxns | 10x Genomics | 1000094 | Store at -20 °C |
| Chromium Single Cell 3' Library Kit v3 | 10x Genomics | 1000095 | Store at -20 °C |
| Chromium Single Cell 3' v3 Gel Beads | 10x Genomics | 2000059 | Store at -80 °C |
| Collagenase A | Sigma/Millipore | 10103578001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| Collagenase B | Sigma/Millipore | 11088807001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| D1000 ScreenTape | Agilent | 5067-5582 | University of Pittsburgh Health Sciences Sequencing Core |
| DNA LoBind Tube Microcentrifuge Tube, 1.5 mL | Eppendorf | 022431021 | |
| DNA LoBind Tube Microcentrifuge Tube, 2.0 mL | Eppendorf | 022431048 | |
| Dynabeads MyOne SILANE | 10x Genomics | 2000048 | Store at 4 °C, used in Beads Cleanup Mix (Table 1) |
| DynaMag-2 Magnet | Theromo Scientific | 12321D | |
| Ethanol, Pure (200 Proof, anhydrous) | Sigma | E7023-500mL | |
| Falcon 15mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-70C | |
| Falcon 50mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-49A | |
| Fetal Bovine Serum, qualified, United States | Fisher Scientific | 26140079 | Store at -20 °C |
| Finnpipette F1 Multichannel Pipettes, 10-100μl | Theromo Scientific | 4661020N | |
| Finnpipette F1 Multichannel Pipettes, 1-10μl | Theromo Scientific | 4661000N | |
| Flowmi Cell Strainer | Sigma | BAH136800040 | Porosity 40 μm, for 1000 uL Pipette Tips, pack of 50 each |
| Glycerin (Glycerol), 50% (v/v) | Ricca Chemical Company | 3290-32 | |
| HBSS, no calcium, no magnesium | Thermo Fisher Scientific | 14170112 | |
| Human TruStain FcX (Fc Receptor Blocking Solution) | BioLegend | 422301 | Add 5 µl of Human TruStain FcX per million cells in 100 µl staining volume |
| Isopropanol (IPA) | Fisher Scientific | A464-4 | |
| Kapa HiFi HotStart ReadyMix (2X) | Fisher Scientific | NC0295239 | Store at -20 °C, used in Lipid-tagged barcode library mix (Table 1) |
| Lipid Barcode Primer (Multi-seq Primer) | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
| Low TE Buffer (10 mM Tris-HCl pH 8.0, 0.1 mM EDTA) | Thermo Fisher Scientific | 12090-015 | |
| MasterCycler Pro | Eppendorf | 950W | |
| Nuclease-Free Water (Ambion) | Thermo Fisher Scientific | AM9937 | |
| PCR Tubes 0.2 ml 8-tube strips | Eppendorf | 951010022 | |
| Phosphate-Buffered Saline (PBS) 1X without calcium & magnesium | Corning Cellgro | 21-040-CV | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin (alternative to Thermo Fisher product) | Sigma-Aldrich | SRE0036 | |
| Pipet 4-pack (0.1–2.5μL, 0.5-10μL, 10–100μL, 100–1,000μL variable-volume pipettes | Fisher Scientific | 05-403-151 | |
| Selection reagent (SPRIselect Reagent Kit) | Beckman Coulter | B23318 (60ml) | |
| Template Switch Oligo | 10x Genomics | 3000228 | Store at -20 °C, used in Master Mix (Table 1) |
| The antibody based barcoding strategy is also known as Cell Hashing | |||
| The cell browser is Loup Cell Browser | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/visualization/latest/what-is-loupe-cell-browser | |
| The commercial available analysis pipline in step 8.1 is Cell Ranger | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/what-is-cell-ranger | |
| The lipid based barcoding strategy is also known as MULTI-seq | |||
| The well maintained R platform is Seurat V3 | satijalab | https://satijalab.org/seurat/ | |
| TipOne RPT 0.1-10/20 ul XL ultra low retention filter pipet tip | USA Scientific | 1180-3710 | |
| TipOne RPT 1000 ul XL ultra low retention filter pipet tip | USA Scientific | 1182-1730 | |
| TipOne RPT 200 ul ultra low retention filter pipet tip | USA Scientific | 1180-8710 | |
| TotalSeq-A0301 anti-mouse Hashtag 1 Antibody | BioLegend | 155801 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 2 Antibody | BioLegend | 155803 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 3 Antibody | BioLegend | 155805 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TrueSeq RPI primer | Integrated DNA Technologies | Single-stranded DNA | 100 nmol, used in Lipid-tagged barcode library mix (Table 1) |
| Trypan Blue Solution, 0.4% | Fisher Scientific | 15250061 | |
| Trypsin-EDTA (0.25%), phenol red | Fisher Scientific | 25200-056 | |
| Universal I5 | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
Références
- Raj, A., Van Den Bogaard, P., Rifkin, S. A., Van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877(2008).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6 (5), 377(2009).
- Li, G., Plonowska, K., Kuppusamy, R., Sturzu, A., Wu, S. M. Identification of cardiovascular lineage descendants at single-cell resolution. Development. 142 (5), 846-857 (2015).
- DeLaughter, D. M., et al. Single-cell resolution of temporal gene expression during heart development. Developmental Cell. 39 (4), 480-490 (2016).
- Li, G., et al. Single cell expression analysis reveals anatomical and cell cycle-dependent transcriptional shifts during heart development. Development. 146 (12), dev173476(2019).
- Meilhac, S. M., Buckingham, M. E. The deployment of cell lineages that form the mammalian heart. Nature Reviews Cardiology. 1, (2018).
- Liu, Z., et al. Single-cell transcriptomics reconstructs fate conversion from fibroblast to cardiomyocyte. Nature. 551 (7678), 100(2017).
- Lafzi, A., Moutinho, C., Picelli, S., Heyn, H. Tutorial: guidelines for the experimental design of single-cell RNA sequencing studies. Nature Protocols. 1, (2018).
- The Tabula Muris Consortium. Single-cell transcriptomics of 20 mouse organs creates a Tabula Muris. Nature. 562 (7727), 367(2018).
- Gawad, C., Koh, W., Quake, S. R. Single-cell genome sequencing: current state of the science. Nature Reviews Genetics. 17 (3), 175(2016).
- Grün, D., van Oudenaarden, A. Design and analysis of single-cell sequencing experiments. Cell. 163 (4), 799-810 (2015).
- Ziegenhain, C., et al. Comparative analysis of single-cell RNA sequencing methods. Molecular cell. 65 (4), 631-643 (2017).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17 (1), 77(2016).
- Islam, S., et al. Characterization of the single-cell transcriptional landscape by highly multiplex RNA-seq. Genome Research. 21 (7), 1160-1167 (2011).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161 (5), 1202-1214 (2015).
- Klein, A. M., et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 161 (5), 1187-1201 (2015).
- Library Prep -Single Cell Gene Expression -Official 10x Genomics Support. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/library-prep (2018).
- McGinnis, C. S., et al. MULTI-seq: sample multiplexing for single-cell RNA sequencing using lipid-tagged indices. Nature Methods. 1, (2019).
- Stoeckius, M., et al. Cell hashing with barcoded antibodies enables multiplexing and doublet detection for single cell genomics. Genome Biology. 19 (1), 224(2018).
- Chan, M. M., et al. Molecular recording of mammalian embryogenesis. Nature. 570 (7759), 77-82 (2019).
- Agilent 4200 TapeStation System. , Agilent. https://www.agilent.com/cs/library/datasheets/public/5991-6029EN.pdf (2019).
- SPRIselect User Guide. , Beckman Coulter. https://research.fhcrc.org/content/dam/stripe/hahn/methods/mol_biol/SPRIselect%20User%20Guide.pdf (2012).
- Qubit 4 Fluorometer User Guide. , Thermo Fisher Scientific. https://assets.thermofisher.com/TFS-Assets/LSG/manuals/MAN0017209_Qubit_4_Fluorometer_UG.pdf (2018).
- Agilent High Sensitivity DNA Kit Guide. , Agilent. https://www.agilent.com/cs/library/usermanuals/public/High%20Sensitivity_DNA_KG.pdf (2016).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411(2018).
- Weber, R. J., Liang, S. I., Selden, N. S., Desai, T. A., Gartner, Z. J. Efficient targeting of fatty-acid modified oligonucleotides to live cell membranes through stepwise assembly. Biomacromolecules. 15 (12), 4621-4626 (2014).
- Single Cell Protocols Cell Preparation Guide. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/sample-prep (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon