Method Article
Multiplexed Single Cell mRNA Sequencing Analysis of Mouse Embryonic Cells
En este artículo
Resumen
Aquí presentamos un método de secuenciación de ARNm de una sola célula multiplexado para perfilar la expresión génica en tejidos embrionarios de ratón. El método de secuenciación de mRNA de una sola célula basada en gotas (scRNA-Seq) en combinación con estrategias de multiplexación puede perfilar celdas individuales de varias muestras simultáneamente, lo que reduce significativamente los costos de reactivos y minimiza los efectos experimentales por lotes.
Resumen
La secuenciación de ARNm de una sola célula ha hecho progresos significativos en los últimos años y se ha convertido en una herramienta importante en el campo de la biología del desarrollo. Se ha utilizado con éxito para identificar poblaciones de células raras, descubrir nuevos genes marcadores y decodificar información espacial y temporal del desarrollo. El método de una sola célula también ha evolucionado de la tecnología Fluidigm C1 basada en microfluidos a las soluciones basadas en gotas en los últimos dos o tres años. Aquí usamos el corazón como ejemplo para demostrar cómo perfilar las células del tejido embrionario del ratón usando el método scRNA-Seq basado en gotas. Además, hemos integrado dos estrategias en el flujo de trabajo para generar perfiles de varias muestras en un solo experimento. Utilizando uno de los métodos integrados, hemos perfilado simultáneamente más de 9.000 células de ocho muestras de corazón. Estos métodos serán valiosos para el campo de la biología del desarrollo al proporcionar una manera rentable de perfilar simultáneamente células individuales de diferentes orígenes genéticos, etapas de desarrollo o ubicaciones anatómicas.
Introducción
El perfil transcripcional de cada célula individual varía entre las poblaciones celulares durante el desarrollo embrionario. Aunque la hibridación molecular in situ única se puede utilizar para visualizar la expresión de un pequeño número de genes1, la secuenciación de ARNm de una sola célula (scRNA-Seq) proporciona un enfoque imparcial para ilustrar los patrones de expresión de genes en todo el genoma en células individuales. Después de su publicación por primera vez en 20092, scRNA-Seq se ha aplicado para estudiar múltiples tejidos en múltiples etapas de desarrollo en los últimos años3,4,5. Además, a medida que el atlas de células humanas ha lanzado recientemente sus proyectos centrados en el desarrollo, se espera que en un futuro próximo se generen más datos de células únicas de tejidos embrionarios humanos.
El corazón como el primer órgano en desarrollarse desempeña un papel crítico en el desarrollo embrionario. El corazón consta de varios tipos de células y el desarrollo de cada tipo de célula está estrechamente regulado temporal y espacialmente. En los últimos años, el origen y el linaje celular de las células cardíacas en las primeras etapas del desarrollo se han caracterizado6,que proporcionan una tremenda herramienta de navegación útil para la comprensión de la patogénesis congénita de enfermedades cardíacas, así como para el desarrollo de métodos más tecnológicamente avanzados para estimular la regeneración de cardiomiocitos7.
El scRNA-Seq ha experimentado una rápida expansión en los últimos años8,9,10. Con los métodos recién desarrollados, el diseño y análisis de experimentos de una sola célula se ha vuelto más alcanzable11,12,13,14. El método presentado aquí es un procedimiento comercial basado en las soluciones de gotas (ver Tabla de Materiales)15,16. Este método cuenta con la captura de células y conjuntos de cuentas con código de barras única en una gota de emulsión aceite-agua bajo el control de un sistema de controlador microfluídico. La velocidad de carga de celdas en las gotas es extremadamente baja, por lo que la mayoría de las emulsiones de gotas contienen solo una celda17. El ingenioso diseño del procedimiento proviene de la separación de una sola célula en emulsiones de gotas que ocurren simultáneamente con la codificación de barras, lo que permite el análisis paralelo de células individuales utilizando ARN-Seq en una población heterogénea.
La incorporación de estrategias de multiplexación es una de las adiciones importantes al flujo de trabajo de una sola célula tradicional13,14. Esta adición es muy útil para desechar los dobletes celulares, reducir los costos experimentales y eliminar los efectos por lotes18,19. Una estrategia de codificación de barras basada en lípidos y una estrategia de codificación de barras basada en anticuerpos (ver Tabla de Materiales)son los dos métodos de multiplexación utilizados principalmente. Los códigos de barras específicos se utilizan para etiquetar cada muestra en ambos métodos, y las muestras etiquetadas se mezclan para la captura de una sola celda, la preparación de la biblioteca y la secuenciación. Después, los datos de secuenciación agrupados se pueden separar analizando las secuencias de códigos de barras (Figura 1)19. Sin embargo, existen diferencias significativas entre los dos métodos. La estrategia de codificación de barras basada en lípidos se basa en oligonucleótidos modificados con lípidos, que no se ha encontrado que tenga ninguna preferencia de tipo de célula. Mientras que la estrategia de codificación de barras basada en anticuerpos sólo puede detectar las células que expresan las proteínas antígeno19,20. Además, se tarda unos 10 minutos en manchar los lípidos, pero 40 minutos para manchar los anticuerpos(Figura 1). Además, los oligonucleótidos modificados con lípidos son más baratos que los oligonucleótidos conjugados con anticuerpos, pero no están disponibles comercialmente en el momento de escribir este artículo. Por último, la estrategia basada en lípidos puede multiplexar 96 muestras en un solo experimento, pero la estrategia basada en anticuerpos actualmente solo puede multiplexar 12 muestras.
El número de celda recomendado para multiplexar en un solo experimento debe ser inferior a 2,5 x 104, de lo contrario, conducirá a un alto porcentaje de dobletes celulares y una posible contaminación del ARNm ambiental. A través de las estrategias de multiplexación, el costo de captura de una sola célula, generación de ADNc y preparación de bibliotecas para múltiples muestras se reducirá al costo de una muestra, pero el costo de secuenciación seguirá siendo el mismo.
Protocolo
El procedimiento animal está de acuerdo con el Comité Institucional de Cuidado y Uso de Animales de la Universidad de Pittsburgh (IACUC).
1. Disección embrionaria del corazón del ratón y preparación de la suspensión de una sola célula
NOTA: Este paso podría tardar unas horas dependiendo del número de embriones para diseccionar.
- Para adquirir corazones embrionarios E18.5, eutanasia un ratón CD1 embarazada por administración de CO2. Utilice una maquinilla de afeitar para eliminar el vello no deseado en la zona abdominal y desinfectar la piel con 70% de etanol.
- Corta la piel del abdomen usando tijeras esterilizadas y disecciona cuidadosamente los embriones y ponlos rápidamente en solución salina fría con fosfato (PBS) en hielo.
- Aísle los corazones de cada embrión individual cuidadosamente en un plato de 10 cm lleno de PBS frío bajo un microscopio estereoscópico usando fórceps y tijeras.
NOTA: Mantenga las cuatro cámaras intactas diseciendo el pulmón con el corazón y NO coja/tire directamente del corazón con instrumentos quirúrgicos. - Transfiera 10 corazones en un nuevo plato de 10 cm lleno de PBS frío y microdiseque los corazones en la aurícula izquierda, la aurícula derecha, el ventrículo izquierdo y el ventrículo derecho.
NOTA: Se supone que esto produce más de 1 x 106 celdas de cada muestra. Recomendamos comenzar con al menos 1 x 105 celdas por muestra. - Transfiera cada uno de los 4 tejidos de la cámara a un tubo de 1,5 ml y córtelos en trozos usando tijeras. Centrífuga a 300 x g durante 3 min para recoger los tejidos.
- Después de aspirar el sobrenadante, añadir 1 ml de 0,25% de trippsina/EDTA a cada tubo e incubar en un baño de agua de 37 oC durante 10 minutos.
- Si la etapa embrionaria es más antigua que E11.5, agregue 1 ml de mezcla a/B de colagenasa de 10 mg/ml e incubaa a 37 oC durante 10-20 minutos.
- Transfiera las células a un tubo de 15 ml y agregue la solución salina equilibrada (HBSS) de Hank de 8 ml para diluir las enzimas. Gire las células a 300 x g durante 5 min. Suspenda las celdas en 1 ml de PBS y transfiéralas a un tubo de 1,5 ml. Filtrar las células a través de un colador de células de 40 m.
- Tomar 15 ml de volumen de cada muestra y mezclar con la misma cantidad de 0,04% trypan azul. Cargue esto en una cámara de conteo de celdas y cuente las celdas en un contador de celdas.
NOTA: Para generar resultados de alta calidad, se recomienda que la viabilidad celular sea superior al 95%.
2. Codificación de barras de multiplexación de células únicas
NOTA: Este paso tarda al menos 40 minutos, lo que varía en función del número de muestras procesadas. Se requiere un área de banco limpia tratada con solución de descontaminación RNase para los pasos de preamplificación (pasos 2.11 a 3.11), y se requiere un área de banco limpia separada para los pasos posteriores a la amplificación (los pasos después de 3.11).
-
Procedimiento de codificación de barras basado en lípidos (procedimiento opcional 1)
- Basado en la concentración celular, mantenga menos de 5 x 105 células por muestra. Asegúrese de que la suspensión celular esté libre de escombros y agregados celulares.
- Preparar una solución de stock de anclaje/código de barras de 2 M y una solución de stock de co-ancla de 2 M para cada muestra(Tabla 1).
NOTA: El ancla y el co-ancla fueron amablemente regalados por el laboratorio del Dr. Zev J. Gartner. Para sintetizar estos oligonucleótidos modificados con lípidos, las secuencias de ADN se conjugaron con ácido graso sobre un soporte sólido y se purificaron mediante cromatografía líquida de alto rendimiento (HPLC)18,19. - Lave las células dos veces con PBS y recoja las células a 300 x g durante 5 min. Suspenda las células en 180 s de PBS.
- Agregue 20 sl de solución de stock de anclaje/código de barras y pipetee hacia arriba y hacia abajo suavemente para mezclar. Incubar sobre hielo durante 5 min.
- Añadir 20 ml de solución de material de co-ancla y pipetear hacia arriba y hacia abajo suavemente para mezclar, luego incubar sobre hielo durante otros 5 minutos.
- Añadir 1 ml de PBS frío con 1% de BSA y centrifugar a 300 x g durante 5 min a 4oC. Lavar al menos 2 veces más con hielo frío 1% BSA en PBS.
- Combine todas las muestras y filtre a través de coladores de células de 40 m. Cuente las células y mantenga la suspensión celular en hielo para usaren en la sección 3.
-
Procedimiento de codificación de barras basado en anticuerpos (procedimiento opcional 2)
- Centrifugar 1 x 106-2 x 106 células para cada muestra (del paso 1.8) a 300 x g durante 5 min y suspenderlas en 100 l de tampón de tinción(Tabla 1) en tubos de unión baja de 1,5 ml.
- Añadir el reactivo de bloqueo de 10 S C e incubar durante 10 min a 4 oC.
- Preparar anticuerpos (ver Tabla de Materiales)centrifugando a 14.000 x g durante 10 min a 2-8oC.
- Añadir 1 g de cada anticuerpo oligoconjugado a 50 ml de tampón de tinción de células para hacer la solución de tinción de anticuerpos20. Agregue una solución de tinción de anticuerpos a cada tubo de muestra. Incubar durante 30 min a 4oC.
- Lavar las células 3 veces con 1 ml de PBS, girar durante 5 min a 350 x g a 4 oC.
- Apool todas las muestras en las proporciones deseadas en 1 ml de tampón de tinción, gire durante 5 min a 350 x g a 4 oC.
- Resuspenda las células en PBS a la concentración adecuada (hasta 1.500 células/L) y filtre las células a través de un colador de células de 40 m. Proceda inmediatamente al siguiente paso.
3. Generación de gotas y transcripción inversa de ARNm
NOTA: Este paso tarda unos 90 minutos para una reacción multiplexada.
- Equilibrar las perlas de gel (ver Tabla de Materiales)a temperatura ambiente durante 30 min. Saque los reactivos del kit de perlas de gel en emulsión (GEM) (ver Tabla de Materiales)y manténgalos a la temperatura indicada.
- Montar el chip B en un soporte de viruta (consulte Tabla de materiales).
- Dispensar 75 l de solución de glicerol al 50% en los pozos no utilizados en la fila 1; 40 l en la fila 2; 280 l en la fila 3. No agregue glicerol en ningún pozo de recuperación en la fila superior del chip.
- Prepare la mezcla maestra sobre hielo de acuerdo con la Tabla 1. Agregue el volumen adecuado de suspensión celular y agua libre de nucleasas para mezclar principalmente de acuerdo con una tabla17 de la calculadora de volumen de suspensión celular y pipetee suavemente la mezcla. Dispensar 75 l de mezcla celular en el centro inferior de la muestra bien en la fila 1 sin introducir burbujas.
- Vortex las perlas de gel para 30 s utilizando un adaptador de vórtice y dispensar lentamente 40 l de perlas de gel en el centro inferior del cordón de gel bien en la fila 2 sin introducir burbujas.
NOTA: Es fundamental esperar 30 s entre la adición de células y perlas de gel para evitar la falla de humectación. - Para el pozo de partición de aceite en la fila 3, dispensar 280 l de aceite de partición a través de la pared lateral del pozo.
NOTA: La carga de menos de 270 l de aceite de partición dará lugar a una generación anormal de GEM. - Coloque la junta en el chip, no presione la junta y manténgala horizontal para evitar mojar la junta.
- Cargue el chip montado con la junta en el controlador de cromo y ejecute el programa de cromo de una sola célula B (consulte Tabla de materiales),proceda inmediatamente al siguiente paso cuando el programa se complete.
- Saque el chip y deseche la junta. Doblar la tapa hacia atrás para exponer los pozos a 45o, comprobar el nivel de líquido para asegurarse de que no haya obstrucciones.
- Aspira lentamente 100 oL de GEM desde los puntos más bajos de la recuperación y comprueba la uniformidad de los GEM. Dispensar GEM en un nuevo tubo de reacción en cadena de polimerasa (PCR) en hielo con las puntas de la pipeta contra la pared lateral del tubo.
NOTA: Si se observa un exceso de capa acuosa, se recomienda repreparar las muestras. Es importante destacar que tome una foto de la mezcla cuando los FMA todavía estén en las puntas de las pipetas. Esta imagen puede indicar si hay un fallo de humectación, GEM parcialmente emulsionados y obstrucciones de reactivos. La fotografía también se puede utilizar como evidencia para obtener el reembolso de la empresa de reactivos con reactivos de reemplazo y virutas. - Coloque el tubo en un ciclor térmico y realice el procedimiento de transcripción inversa(Tabla 2).
NOTA: Deténgase aquí o continúe con el siguiente paso. El producto PCR se puede almacenar a -20 oC durante un máximo de una semana.
4. Amplificación del ADNc
NOTA: Este paso tarda unos 150 minutos.
- Limpieza de transcripción inversa de una sola celda
- Saque los reactivos de amplificación de ADNc del kit GEMs (ver Tabla de Materiales)y manténgalos a la temperatura indicada.
- Añadir 125 ml de agente de recuperación a la muestra a temperatura ambiente para adquirir una mezcla bifásica. No se debe observar ningún líquido opaco y evitar pipetear o vórtice la mezcla.
- Después de esperar 60 s, retire lentamente 125 s de agente de recuperación de la parte inferior del tubo.
- Vortex las perlas magnéticas (ver Tabla de Materiales) a fondo durante 30 s y utilizarlo inmediatamente para preparar la mezcla de limpieza de perlas (Tabla 1). Los reactivos deben agregarse secuencialmente como se indica.
- Vortex la mezcla de limpieza de perlas y añadir 200 l a la muestra. Pipetear la mezcla 10 veces y luego incubarla durante 10 minutos a temperatura ambiente.
- Agregue los reactivos secuencialmente como se enumeran en la Tabla 1 para preparar la solución de elución y eluir el ADNc de la siguiente manera.
- Coloque las muestras en el imán (posición alta) (ver Tabla de materiales)hasta que la solución se despeje y, a continuación, retire el sobrenadante.
- Añadir 200 l de etanol al 80% al pellet. Espere 30 s y, a continuación, retire el etanol.
- Repita el paso 4.1.6.2 para otras 2 veces. Centrífuga brevemente y colóquela en el imán (posición baja). Retire cuidadosamente el etanol restante y seque al aire durante menos de 2 minutos.
NOTA: NO exceda el secado por aire más allá de 2 minutos, de lo contrario la eficiencia de elución disminuirá. - Retire la muestra del imán. Añadir 35,5 ml de solución de elución y pipeta para mezclar 15 veces. Incubar 2 min a temperatura ambiente.
- Coloque la muestra sobre el imán (posición alta) hasta que la solución se despeje. Transfiera 35 l de la muestra a una nueva tira de tubo.
NOTA: Este procedimiento de purificación también se utiliza en los pasos 4.2.1.6, 4.2.2.3, 5.8, 6.1.10 y 6.2.6. Preste atención a la concentración de perlas magnéticas y al volumen de tamón EB/agua ultrapura utilizada para eluir las muestras en cada paso.
- Cuantifique el tamaño, la concentración y la integridad del ADNc eludado utilizando un instrumento automatizado de electroforesis21 (ver Tabla de Materiales) (Figura 2).
- Amplificación del ADNc
-
amplificación de ADNc mediante la estrategia de codificación de barras basada en lípidos (procedimiento opcional 1)
- Preparar la mezcla de reacción de amplificación (Tabla 1) sobre hielo.
- Añadir la mezcla de reacción de amplificación a 35 ml de muestras de ADNc (a partir del paso 4.1.6.7). Pipetear la mezcla, y centrifugar brevemente. Incubar la mezcla en un ciclor térmico siguiendo el procedimiento de amplificación del ADNc (Tabla 2).
- Después de vórtice a fondo, añadir 120 l de reactivo selecto y 100 l de agua ultrapura a 100 l de muestra para adquirir una concentración de 0,6x de reactivo selecto (ver Tabla de materiales). Pipetear la mezcla 15 veces.
- Incubar durante 5 minutos a temperatura ambiente y luego colocar la muestra en el imán hasta que las soluciones se vuelvan claras.
NOTA: El ADNC endógeno en la fracción de perlas y la multiplexación múltiple de cDNA con códigos de barras está en el sobrenadante. - Transfiera el sobrenadante en un tubo de unión baja de 1,5 ml para multiplexar la construcción de la biblioteca cDNA con códigos de barras en el paso 6.1.
- Limpie el ADNC endógeno siguiendo los pasos descritos en 4.1.6 y eluyéquelos con 40 ml de tampón DE EB.
- Ejecutar 1 l de muestra de ADNc purificado en un instrumento de electroforesis automatizado (ver Tabla de Materiales)para analizar/cuantificar el ADNc.
- Aliquot 10 l de ADNc en un nuevo tubo pcR para la construcción endógena de la biblioteca.
NOTA: Deténgase aquí o continúe con el siguiente paso. El ejemplo restante se puede almacenar en -20 oC durante un máximo de 4 semanas para generar bibliotecas adicionales si es necesario.
-
amplificación de ADNc en la estrategia de codificación de barras basada en anticuerpos (procedimiento opcional 2)
- Añadir 2 pmol de imprimación aditiva HTO y ADT y 15 l de imprimaciones de ADNc a 50 l de mezcla de reacción de amplificación (Tabla 1) y realizar la amplificación del ADNc con el procedimiento de amplificación del ADNc(Tabla 2).
- Utilice el reactivo de selección 0.6x para separar el ADNC endógeno (fracción de cuentas) y la multiplexación del código de barras cDNA (en supernadante). Recuerde guardar el sobrenadante para realizar la construcción de la biblioteca de codificación de barras en el paso 6.2.
- Purificar y eluir la transcripción endógena cDNA siguiendo los pasos descritos en 4.1.6, realizar el control de calidad (QC) de las bibliotecas y alícuota 10 l en un nuevo tubo de PCR para la construcción de bibliotecas endógenas.
-
amplificación de ADNc mediante la estrategia de codificación de barras basada en lípidos (procedimiento opcional 1)
5. Preparación de la Biblioteca de Transcripción Endógena
NOTA: Este paso tarda unos 120 min.
- Mantenga los reactivos de construcción de la biblioteca de expresión génica del kit de la biblioteca (ver Tabla de materiales)a la temperatura indicada, respectivamente.
- Preparar brevemente la mezcla de fragmentación(Tabla 1) sobre hielo, pipetear para mezclar y centrifugar.
- Agregue el tampón EB de 25 l a la muestra de ADN cDNA purificado de 10 l (a partir del paso 4.2.1.8 o 4.2.2.3) y luego agregue la mezcla de fragmentación de 15 ol recién preparada a la muestra, pipetee la mezcla 15 veces sobre hielo y centrífuga brevemente.
- Transfiera la muestra a un ciclor térmico preenfriado e inicie el programa PCR para la fragmentación, la reparación final y la cola A(Tabla 2).
- Vortex el reactivo selecto para suspender perlas magnéticas y utilizar sucesivamente 0.6x y 0.8x seleccionar reactivos para hacer una selección de tamaño de doble cara de acuerdo con la guía del usuario17,22. Utilice 50 ml de tampón de EB para eluir el ADN.
- Preparar la mezcla de ligadura del adaptador(Tabla 1), luego pipetear la mezcla a fondo y centrifugar brevemente.
- Añadir 50 ml de mezcla de ligadura de adaptador a 50 ml de muestra, mezclar pipeta de nuevo durante 15 veces y centrifugar brevemente. Realice la ligadura del adaptador según el protocolo en un ciclor térmico (Tabla 2).
- Utilice un reactivo selecto 0.8x para purificar el producto de ligadura y eluir la muestra purificada con 30 ml de tampón de EB (ver paso 4.1.6).
- Preparar la mezcla de PCR de índice de muestra(Tabla 1) y añadir 60 l a la muestra purificada. Añadir 10 l de índice de muestra a la muestra, mezclar pipetas hacia arriba y hacia abajo durante 5 veces e centrifugar brevemente, incubar en un ciclor térmico siguiendo el protocolo de índice de muestra(Tabla 2).
NOTA: Deténgase aquí o continúe con el siguiente paso. Si se utiliza más de un pozo, elija un índice de muestra específico (consulte Tabla de materiales)para cada pozo. Recuerde registrar el ID de índice utilizado para cada pozo y asegúrese de que no se superponga en una ejecución de secuenciación multiplexada. - Utilice sucesivamente reactivos de selección de 0,6x y 0,8x para realizar una selección de tamaño de doble cara para adquirir 35 ml de ADN de biblioteca celular endógena purificada17.
- QC la biblioteca celular endógena antes de la secuenciación (Figura 2).
6. Preparación de la muestra de multiplexación Bibliotecas de código de barras cDNA cDNA
NOTA: Este paso tarda al menos 120 min.
-
Generación de biblioteca de códigos de barras de ejemplo para la estrategia de multiplexación basada en lípidos (procedimiento opcional 1)
- Añadir 520 l de reactivo selecto y 360 l de isopropanol para muestrear el código de barras cDNA del paso 4.2.1.5 para obtener una concentración de reactivo selecto de 3.2x. Pipetear la mezcla 10 veces e incubar a temperatura ambiente durante 5 min.
- Coloque el tubo en un bastidor magnético y espere a que la solución se despeje. A continuación, deseche el sobrenadante.
- Utilice 500 s de etanol al 80% para lavar las perlas dos veces en un imán y esperar 30 s después de cada lavado.
- Centrifugar brevemente las perlas y colocar las cuentas en un imán. Retire el etanol restante con un micropipeta P10 y deje las perlas durante 2 min.
- Retire el tubo del bastidor del imán y vuelva a suspender las perlas en 50 ml de tampón EB. Pipet a arriba y abajo para mezclar a fondo. Incubar a temperatura ambiente durante 2 min.
- Vuelva a colocar el tubo en un imán y espere a que la solución se despeje. Transfiera el sobrenadante (código de barras cDNA de muestra) a un nuevo tubo PCR. Tenga cuidado de no transferir ninguna cuenta.
- Cuantificar la concentración de código de barras de muestra cDNA23.
- Preparar la mezcla de la biblioteca de códigos de barras lipídicas(Tabla 1), añadir 3,5 ng de ADNC con código de barras purificado (del paso 6.1.6) y agua libre de nucleasas para un volumen total de 50 l.
- Guárdelo en un ciclor térmico siguiendo la biblioteca de códigos de barras a base de lípidos PCR (Tabla 2).
- Utilice el reactivo de selección 1.6x para purificar el producto PCR y eluir el ADN con 25 ml de tampón de EB (ver paso 4.1.6).
- Cuantificar la concentración de la biblioteca utilizando un método de análisis de ADN de alta sensibilidad24 a partir de la dilución inicial de 1:5(Figura 2).
-
Generación de biblioteca de códigos de barras de ejemplo para la estrategia de multiplexación basada en anticuerpos (procedimiento opcional 2)
- Agregue un volumen de reacción 1.4x adicional de reactivo selecto al sobrenadante que contiene códigos de barras de muestra adquiridos en el paso 4.2.2.3 para obtener una relación de reactivos de selección 2x.
- Lave las perlas con 80% de etanol siguiendo los pasos descritos en 4.1.6 y eluya el ADNc con códigos de barras con agua ultrapura.
- Realice el protocolo de selección con un reactivo selecto 2x por segunda vez y eluda con agua ultrapura.
- Prepare la mezcla de la biblioteca de códigos de barras de anticuerpos(Tabla 1) y añada 45 ml de ADNC con códigos de barras purificados desde el último paso.
- Incubar en un ciclor térmico siguiendo la biblioteca de códigos de barras de anticuerpos PCR (Tabla 2)20.
- Utilice el reactivo de selección 1.6x para purificar el producto PCR y eluir la muestra purificada con 30 ml de agua ultrapura (ver paso 4.1.6).
7. Secuenciación de bibliotecas
NOTA: Se pueden utilizar varias plataformas de secuenciación de próxima generación como HiSeq 4000 y NovaSeq para secuenciar las bibliotecas de transcripción endógenas y las bibliotecas de códigos de barras de multiplexación.
- Utilice una plataforma de secuenciación de próxima generación de su elección para secuenciar las bibliotecas de transcripción endógenas y las bibliotecas de códigos de barras de multiplexación.
- Diluir las bibliotecas de acuerdo con las recomendaciones de un experto en una empresa de secuenciación o una instalación de secuenciación. Se recomienda un mínimo de 20.000 lecturas por celda para la biblioteca de transcripciones endógenas y 3000 lecturas para las bibliotecas de códigos de barras.
8. Análisis de datos
NOTA: Desmultiplexar los datos de secuenciación mediante el recurso basado en la nube BaseSpace o ejecutando el paquete bcl2fastq en un servidor UNIX.
-
Análisis endógeno de datos de transcriptoma
- Con los datos fastq generados a partir del software de desmultiplexación, ejecute "mkfastq" en la canalización de análisis de datos disponible comercialmente (consulte Tabla de materiales)para demultiplexar aún más cada código de barras DEM.
- Ejecute "count" para realizar la alineación, el filtrado, el recuento de códigos de barras y el recuento de UMI.
- Opcionalmente, ejecute "aggr" para agregar varios carriles de secuenciación de un solo experimento.
- Utilice "navegador celular" (consulte Tabla de materiales)para visualizar datos, células de racimo, identificar genes expresados diferencialmente y generar gráficas de mapa de calor tSNE o de expresión génica.
- Opcionalmente, utilice una plataforma basada en R25 bien mantenida (ver Tabla de materiales)para normalizar y escalar datos, identificar genes expresados diferencialmente y generar gráficas tSNE/UMAP y mapas de calor de expresión génica(Figura 3).
-
Multiplexación del análisis de datos de códigos de barras
-
Análisis de los datos de la estrategia de codificación de barras basada en lípidos
- Utilice la canalización de análisis de datos disponible comercialmente o el paquete deMULTIplex R(https://github.com/chris-mcginnis-ucsf/MULTI-seq) para convertir los archivos FASTQ del código de barras de muestra en una matriz de recuento UMI de código de barras de ejemplo.
- Cargue la matriz de recuento UMI del código de barras junto con los datos de transcriptoma endógenos en una plataforma basada en R (consulte Tabla de materiales)para el análisis de integración (Figura 3).
-
Análisis de los datos de la estrategia de codificación de barras basada en anticuerpos
- Utilice "count" de la canalización de análisis de datos disponible comercialmente para asignar los códigos de barras proporcionando el archivo CSV de la biblioteca y el archivo CSV de referencia de la característica de hashtag.
- Cargue la matriz de código de barras de entidadunificada de salida, que contiene recuentos de expresiones génicas junto con recuentos de códigos de barras de entidades para cada código de barras de celda, en una plataforma basada en R para el análisis posterior.
-
Análisis de los datos de la estrategia de codificación de barras basada en lípidos
Resultados
En este estudio, utilizamos el corazón embrionario de ratón como ejemplo para exhibir cómo se realizó la secuenciación de ARNm de una sola célula multiplexada para procesar las diferentes muestras de partes separadas de un órgano simultáneamente. Los corazones del ratón E18.5 CD1 fueron aislados y diseccionados en la aurícula izquierda (LA), la aurícula derecha (RA), el ventrículo izquierdo (LV) y el ventrículo derecho (RV). Las células auriculares y ventriculares fueron descodificadoras de forma independiente mediante un procedimiento de codificación de barras basado en lípidos y mezcladas antes de la generación de GEM y transcripción inversa. La descripción general esquemática se muestra en la Figura 1. Cuantificamos la concentración de ADNc antes de la construcción de la biblioteca(Figura 2A). Una de las distinciones en la realización de scRNA-Seq multiplexado desde el scRNA-Seq estándar es que la biblioteca endógena de ADNc y la biblioteca de ADNc de código de barras de muestra se adquirieron por separado después de la amplificación y purificación del ADNc (Paso 4.2.1 y 4.2.2.2). Las dos bibliotecas también fueron calificadas en nuestro experimento(Figura 2B,C). La secuenciación de próxima generación y el análisis de datos se realizaron seguidos de la construcción de bibliotecas y el control de calidad.
Usamos la plataforma HiSeqX para secuenciar ambas bibliotecas en el mismo carril de secuenciación. Con los datos de secuenciación, primero separamos los datos de transcripción endógenos y los datos de código de barras utilizando el programa BaseSpace. Luego analizamos la expresión de código de barras en cada celda y encontramos 8 grupos de celdas individuales que expresan de forma única un tipo de código de barras, representando celdas de 8 muestras diferentes(Figura 3A). Además, también encontramos que algunas celdas no expresan ningún código de barras, que definimos como celdas negativas, y algunas celdas expresan dos códigos de barras diferentes, que representan dobletes(Figura 3B). En resumen, encontramos que alrededor del 70% de las células son singletes, el 25% de las células son negativas y el 5% de las células son dobletes.
Con las células singlete, podemos realizar más análisis aguas abajo para entender la heterogeneidad celular y las regulaciones moleculares. Los análisis potenciales pueden ser la anotación de tipo celular(Figura 4A),la identificación del tipo de célula novedosa/rara (Figura 4B),el análisis comparativo de la zona anatómica(Figura 4C)y el análisis de la vía ontológica genética, como las separaciones de fase del ciclo celular(Figura 4D).

Figura 1: Flujo de trabajo de secuenciación de mRNA de una sola célula multiplexado. Los corazones embrionarios de la etapa 18.5 del día se analizaron utilizando un procedimiento de secuenciación de una sola célula basado en gotas multiplexadas. RT - Transcripción inversa. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Resultados representativos de control de calidad en diferentes pasos. (A) Análisis de QC del ADNc del paso 4.1.7. El tamaño del fragmento objetivo es de 200 a 9000 bp. (B) Biblioteca endógena y (C) biblioteca de códigos de barras se analizaron con un instrumento de electroforesis automatizada. El tamaño del fragmento de destino para la biblioteca endógena es de 300-600 bp, y el tamaño de ADN de la biblioteca de códigos de barras es de alrededor de 172 bp. Por favor, haga clic aquí para ver una versión más grande de esta figura.
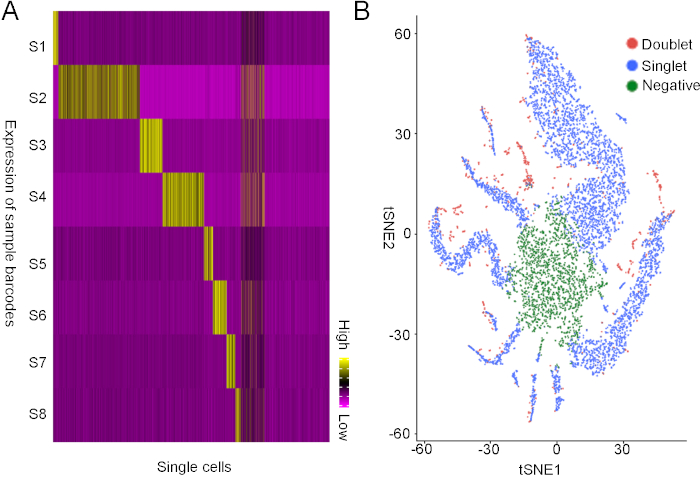
Figura 3: Demultiplexación de los datos de secuenciación de la estrategia de codificación de barras basada en lípidos. (A) Análisis no supervisado de la expresión del código de barras. El eje X representa celdas individuales y el eje Y representa códigos de barras. Cada una de las 8 poblaciones de células individuales fue identificada para expresar de manera única uno de los 8 códigos de barras. Tenga en cuenta que algunas celdas expresan más de un código de barras, y algunas celdas no expresan ningún código de barras. (B) gráfica t-SNE de las celdas singlet, celdas de doblete y celdas negativas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Análisis avanzado de datos transcripcionales de una sola célula. (A-D) Los datos de una sola célula se pueden analizar de diferentes maneras para entender la heterogeneidad celular y las vías moleculares. Hemos enumerado varias aplicaciones aquí como ejemplos. Las celdas individuales se cargaron en un paquete R para identificar los tipos de celda (A), las poblaciones de células raras (B), las zonas anatómicas de células(C)y las fases del ciclo celular (D). Haga clic aquí para ver una versión más grande de esta figura.
| Nombre de la mezcla | Composición |
| Mezcla de colagenasa | 10 mg/ml de colagenasa A y 10 mg/ml de colagenasa B, disuelta en HBSS++ con 40% FBS. |
| Solución de stock de 2 M Anchor/Barcode | Mezclar el anclaje de 50 m y la hebra de código de barras de 10 m en una relación molar de 1:1 en PBS (sin FBS o BSA) para un volumen total de 25 l. |
| Solución de stock de co-ancla de 2 M | Diluir el co-ancla de 1 L 50 m con 24 PBS de L (sin FBS o BSA). |
| Tampón de tinción | PBS que contiene 2% De BSA, 0.01% Tween 20 |
| Mezcla maestra | Reactivo RT de 20 l, Oligo de 3,1 l, Agente reductor B de 2 l, Enzima C RT de 8,3 l. |
| Mezcla de limpieza de perlas | Búfer de limpieza de 182 L, reactivo de selección de 8 l, agente reductor B de 5 l, agua libre de nucleasas de 5 l. |
| Mezcla de reacción de amplificación | 1 L de imprimación aditiva etiquetada con lípidos de 10 m, imprimación de ADNado de 15 l, mezcla de 50 amperios |
| Solución elución | 98 L Tampón EB, 1 L 10% Tween 20, 1 -L Agente Reductor B. |
| Fragmentación Mezcla | Búfer de fragmentación de 5 l, enzima de fragmentación de 10 l. |
| Mezcla de ligadura del adaptador | Tampón de ligadura de 20 l, ligadosa de ADN de 10 l, comisos de adaptador de 20 l. |
| Ejemplo de mezcla de PCR de índice | Mezcla de 50 Amp, 10 S SI Primer |
| Mezcla de biblioteca de código sólido | 26,25 ml de mezcla maestra de arranque en caliente de 2o, 2,5 ml de imprimación RPIX de 10 m, 2,5 ml de imprimación de adaptador universal TruSeq de 10 m (ver tabla de materiales) |
| Mezcla de biblioteca de códigos de barras de anticuerpos | 50 ml de mezcla maestra de inicio en caliente de 2o, 2,5 ml de imprimación RPIX de 10 m, 2,5 ml de oligo híbrido p5-inteligente-pcr de 10 m |
Tabla 1: Las mezclas de reactivos utilizadas en el protocolo.
| Procedimiento de incubación | Temperatura(1) | hora |
| Gem-RT Incubación | Temperatura de la tapa 53 oC | |
| Paso 1 | 53 oC | 45 min |
| Paso 2 | 85 oC | 5 min |
| Paso 3 | 4 oC | Mantener |
| 10x Amplificación de ADNómica cDNA | Temperatura de la tapa 105 oC | |
| Paso 1 | 98 oC | 3 min |
| Paso 2 | 98 oC | 15 s |
| Paso 3 | 63 oC | 20 s |
| Paso 4 | 72 oC | 1 min |
| Paso 5 | Repita los pasos 2 a 4 para 12 ciclos en total (2) | |
| Paso 6 | 72 oC | 1 min |
| Paso 7 | 4 oC | Mantener |
| Construcción de bibliotecas | Temperatura de la tapa 65 oC | |
| Bloque pre-cool | 4 oC | Mantener |
| Fragmentación | 32 oC | 5 min |
| Reparación final y cola A | 65 oC | 30 min |
| Mantener | 4 oC | Mantener |
| Ligadura del adaptador | Temperatura de la tapa 30 oC | |
| Paso 1 | 20 oC | 15 min |
| Paso 2 | 4 oC | Mantener |
| Ejemplo de índice PCR | Temperatura de la tapa 105 oC | |
| Paso 1 | 98 oC | 45 s |
| Paso 2 | 98 oC | 20 s |
| Paso 3 | 54 oC | 30 s |
| Paso 4 | 72 oC | 20 s |
| Paso 5 | Repita los pasos 2 a 4 para 12 ciclos en total (3) | |
| Paso 6 | 72 oC | 1 min |
| Paso 7 | 4 oC | Mantener |
| Biblioteca de códigos de barras de lípidos PCR | ||
| Paso 1 | 95 oC | 5 min |
| Paso 2 | 98 oC | 15 s |
| Paso 3 | 60 oC | 30 s |
| Paso 4 | 72 oC | 30 s |
| Paso 5 | Repita los pasos 2 a 4 para 10 ciclos en total (4) | |
| Paso 6 | 72 oC | 1 min |
| Paso 7 | 4 oC | Mantener |
| Biblioteca de códigos de barras de anticuerpos PCR | ||
| Paso 1 | 95 oC | 3 min |
| Paso 2 | 95 oC | 20 s |
| Paso 3 | 60 oC | 30 s |
| Paso 4 | 72 oC | 20 s |
| Paso 5 | Repita los pasos 2 a 4 para 8 ciclos en total (5) | |
| Paso 6 | 72 oC | 5 min |
| Paso 7 | 4 oC | Mantener |
Tabla 2: El procedimiento de incubación utilizado en el protocolo. (1) Preste atención a la temperatura de la tapa utilizada en cada procedimiento. (2) Establecer números de ciclo total de acuerdo con la carga de la célula: 13 ciclos para <500 carga de celda; 12 ciclos para 500-6,000 carga de celda; 11 ciclos para >6,000 carga de celdas. (3) Ajuste los números de ciclo total de acuerdo con la entrada de ADNc: 14-16 ciclos para 1-25 ng cDNA; 12-14 ciclos para 25-150 ng ADNc; 10-12 ciclos para 150-500 ng adNC; 8-10 ciclos para 500-1,000 ng cDNA; 6-8 ciclos para 1000-1500 ng adNM. (4) Ajuste los números de ciclo total de acuerdo con la entrada de ADNc: 8-12 ciclos. (5) Ajuste los números de ciclo total de acuerdo con la entrada de ADNc: 6-10 ciclos.
| Oligonucnucleótidos de codificación de barras a base de lípidos | |
| Anclaje LMO | 5'-TGGAATTCGGGCCAAGGGTAACGATCCAGCTGTCACT-Lípido-3' |
| LMO de co-ancla | 5'-Lípido-AGTGACAGCTGGATCGTTAC-3' |
| Barcode Oligo | 5'-CCTTGGCACCCGAGAATTCCANNNNNNNNNA30-3' |
| Codificación de barras de lípidos Additive Primer | 5'-CTTGGCACCCGAGAATTCC-3' |
| RPIX Primer | 5'-CAAGCAGAAGACGGCATACGAGATNNNNNNNNNNGTGGACTGGAGTAGTTCC TTGGCACCCGAGAATTCCA-3' |
| Primer adaptador universal | 5'-AATGATACGGCGACCACCGAGATCTACCTCTTCCCTACACGAC GCTCTTCCGATCT-3' |
| Oligonucleótidos de codificación de anticuerpos basados en anticuerpos | |
| Oli de codificación de barras de anticuerpos | 5'-GTGACTAGTTCAGACGTGCTCTTCCGATCTNNNNNNNNNNNNNNNNNNNNNNNNNNNNN NNBAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA*A*A-3' |
| Aditivo HTO Primer | 5'-GTGACTGGAGTTCAGACGTGCTC-3' |
| Aditivo ADT Primer | 5'-CCTTGGCACCCGAGAATTCC-3' |
| P5-smart-pcr híbrido oligo | 5'-AATGATACGGCGACCACCGAGATCTCTCTCTCCGCGGAA GCAGTGGTATCAACGCAGAGT*A*C-3' |
Tabla 3: Secuencias de oligonucleótidos utilizadas en este protocolo. N - Secuencia de código de barras o índice; * Enlace de fosfotioato
Discusión
En este estudio, hemos demostrado un protocolo para analizar perfiles transcripcionales de una sola célula. También hemos proporcionado dos métodos opcionales para multiplexar muestras en el flujo de trabajo scRNA-Seq. Ambos métodos han demostrado ser factibles en varios laboratorios y han proporcionado soluciones para ejecutar un experimento de una sola célula rentable y libre de efectos por lotes18,26.
Hay algunos pasos que deben seguirse cuidadosamente al pasar por el protocolo. Una suspensión de una sola célula ideal debe tener >90% de las células viables y la densidad celular también debe estar dentro de un rango específico27. Es fundamental obtener una buena calidad de las células para minimizar la presencia de agregados celulares, desechos y fibras. Los agregados celulares tienen un impacto negativo en la multiplexación de muestras y tienen un riesgo potencial de obstruir la máquina generadora de gotas17. En términos generales, un colador de células de 30-40 m es ideal para eliminar grandes grumos y escombros, preservando al mismo tiempo las muestras celulares, ya que la mayoría de las células se encogerán por debajo de 30 m después de la disociación. Se recomienda utilizar núcleos de una sola célula si el diámetro de la célula es mayor que 30 m. En las primeras etapas embrionarias, el tamaño de la célula para todos los tipos de células de ratón debe ser menor que 30 m. Sin embargo, en etapas posteriores, los cardiomiocitos en el corazón, las neuronas en el cerebro, las células musculares en las extremidades, y algunas células de grasa pueden tener un tamaño de célula mayor de 30 m. Tamaño de la célula debe medirse para estos tipos de células antes de comenzar los experimentos de una sola célula.
Las estrategias de multiplexación proporcionan una manera de analizar simultáneamente un gran número de muestras de una manera rentable. Además, al generar perfiles de varias muestras juntas, podemos evitar significativamente los efectos por lotes e identificar los dobletes de celda. Estas ventajas serán muy atractivas para el campo de una sola celda. Sin embargo, hay algunos factores que pueden limitar su uso. A medida que más celdas se multiplexan en un solo experimento, la relación de doblete de celda también aumentará. Aunque esos dobletes se pueden identificar y eliminar mediante el análisis de los datos del código de barras de multiplexación, esto conducirá a un gran desperdicio de lecturas de secuenciación. Además, a medida que más celdas se agrupan, las celdas son más fáciles de romper y causan un aumento del ARNm ambiental, que se capturará en gotas con las células e interferirá con la sensibilidad de detección. Esperamos que una mayor optimización del flujo de trabajo experimental o de la canalización de análisis bioinformático resuelva estos dos problemas en un futuro próximo.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos a David M. Patterson y Christopher S. McGinnis del laboratorio Dr. Zev J. Gartner por su amable suministro de los reactivos de codificación de barras basados en lípidos y sugerencias sobre los pasos experimentales y el análisis de datos. Este trabajo fue fundado por los Institutos Nacionales de Salud (HL13347202).
Materiales
| Name | Company | Catalog Number | Comments |
| 10% Tween-20 | Bio-Rad | 1610781 | |
| 10x Chip Holder | 10x Genomics | 120252 330019 | |
| 10x Chromium Controller | 10x Genomics | 120223 | |
| 10x Magnetic Separator | 10x Genomics | 120250 230003 | |
| 10x Vortex Adapter | 10x Genomics | 330002, 120251 | |
| 10x Vortex Clip | 10x Genomics | 120253 230002 | |
| 4200 TapeStation System | Agilent | G2991AA | |
| Agilent High Sensitivity DNA Kit | Agilent | 5067-4626 | University of Pittsburgh Health Sciences Sequencing Core |
| Barcode Oligo | Integrated DNA Technologies | Single-stranded DNA | 25 nmol |
| Buffer EB | Qiagen | 19086 | |
| CD1 mice | Chales River | Strain Code 022 | ordered pregnant mice |
| Centrifuge 5424R | Appendorf | 2231000214 | |
| Chromium Chip B Single Cell Kit, 48 rxns | 10x Genomics | 1000073 | Store at ambient temperature |
| Chromium i7 Multiplex Kit, 96 rxns | 10x Genomics | 120262 | Store at -20 °C |
| Chromium Single Cell 3' GEM Kit v3,4 rxns | 10x Genomics | 1000094 | Store at -20 °C |
| Chromium Single Cell 3' Library Kit v3 | 10x Genomics | 1000095 | Store at -20 °C |
| Chromium Single Cell 3' v3 Gel Beads | 10x Genomics | 2000059 | Store at -80 °C |
| Collagenase A | Sigma/Millipore | 10103578001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| Collagenase B | Sigma/Millipore | 11088807001 | Store powder at 4 °C, store at -20 °C after it dissolves |
| D1000 ScreenTape | Agilent | 5067-5582 | University of Pittsburgh Health Sciences Sequencing Core |
| DNA LoBind Tube Microcentrifuge Tube, 1.5 mL | Eppendorf | 022431021 | |
| DNA LoBind Tube Microcentrifuge Tube, 2.0 mL | Eppendorf | 022431048 | |
| Dynabeads MyOne SILANE | 10x Genomics | 2000048 | Store at 4 °C, used in Beads Cleanup Mix (Table 1) |
| DynaMag-2 Magnet | Theromo Scientific | 12321D | |
| Ethanol, Pure (200 Proof, anhydrous) | Sigma | E7023-500mL | |
| Falcon 15mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-70C | |
| Falcon 50mL High Clarity PP Centrifuge Tube | Corning Cellgro | 14-959-49A | |
| Fetal Bovine Serum, qualified, United States | Fisher Scientific | 26140079 | Store at -20 °C |
| Finnpipette F1 Multichannel Pipettes, 10-100μl | Theromo Scientific | 4661020N | |
| Finnpipette F1 Multichannel Pipettes, 1-10μl | Theromo Scientific | 4661000N | |
| Flowmi Cell Strainer | Sigma | BAH136800040 | Porosity 40 μm, for 1000 uL Pipette Tips, pack of 50 each |
| Glycerin (Glycerol), 50% (v/v) | Ricca Chemical Company | 3290-32 | |
| HBSS, no calcium, no magnesium | Thermo Fisher Scientific | 14170112 | |
| Human TruStain FcX (Fc Receptor Blocking Solution) | BioLegend | 422301 | Add 5 µl of Human TruStain FcX per million cells in 100 µl staining volume |
| Isopropanol (IPA) | Fisher Scientific | A464-4 | |
| Kapa HiFi HotStart ReadyMix (2X) | Fisher Scientific | NC0295239 | Store at -20 °C, used in Lipid-tagged barcode library mix (Table 1) |
| Lipid Barcode Primer (Multi-seq Primer) | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
| Low TE Buffer (10 mM Tris-HCl pH 8.0, 0.1 mM EDTA) | Thermo Fisher Scientific | 12090-015 | |
| MasterCycler Pro | Eppendorf | 950W | |
| Nuclease-Free Water (Ambion) | Thermo Fisher Scientific | AM9937 | |
| PCR Tubes 0.2 ml 8-tube strips | Eppendorf | 951010022 | |
| Phosphate-Buffered Saline (PBS) 1X without calcium & magnesium | Corning Cellgro | 21-040-CV | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin (alternative to Thermo Fisher product) | Sigma-Aldrich | SRE0036 | |
| Pipet 4-pack (0.1–2.5μL, 0.5-10μL, 10–100μL, 100–1,000μL variable-volume pipettes | Fisher Scientific | 05-403-151 | |
| Selection reagent (SPRIselect Reagent Kit) | Beckman Coulter | B23318 (60ml) | |
| Template Switch Oligo | 10x Genomics | 3000228 | Store at -20 °C, used in Master Mix (Table 1) |
| The antibody based barcoding strategy is also known as Cell Hashing | |||
| The cell browser is Loup Cell Browser | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/visualization/latest/what-is-loupe-cell-browser | |
| The commercial available analysis pipline in step 8.1 is Cell Ranger | 10x Genomics | https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/what-is-cell-ranger | |
| The lipid based barcoding strategy is also known as MULTI-seq | |||
| The well maintained R platform is Seurat V3 | satijalab | https://satijalab.org/seurat/ | |
| TipOne RPT 0.1-10/20 ul XL ultra low retention filter pipet tip | USA Scientific | 1180-3710 | |
| TipOne RPT 1000 ul XL ultra low retention filter pipet tip | USA Scientific | 1182-1730 | |
| TipOne RPT 200 ul ultra low retention filter pipet tip | USA Scientific | 1180-8710 | |
| TotalSeq-A0301 anti-mouse Hashtag 1 Antibody | BioLegend | 155801 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 2 Antibody | BioLegend | 155803 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TotalSeq-A0302 anti-mouse Hashtag 3 Antibody | BioLegend | 155805 | 0.1 - 1.0 µg of antibody in 100 µl of staining buffer for every 1 million cells |
| TrueSeq RPI primer | Integrated DNA Technologies | Single-stranded DNA | 100 nmol, used in Lipid-tagged barcode library mix (Table 1) |
| Trypan Blue Solution, 0.4% | Fisher Scientific | 15250061 | |
| Trypsin-EDTA (0.25%), phenol red | Fisher Scientific | 25200-056 | |
| Universal I5 | Integrated DNA Technologies | Single-stranded DNA | 100 nmol |
Referencias
- Raj, A., Van Den Bogaard, P., Rifkin, S. A., Van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877(2008).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6 (5), 377(2009).
- Li, G., Plonowska, K., Kuppusamy, R., Sturzu, A., Wu, S. M. Identification of cardiovascular lineage descendants at single-cell resolution. Development. 142 (5), 846-857 (2015).
- DeLaughter, D. M., et al. Single-cell resolution of temporal gene expression during heart development. Developmental Cell. 39 (4), 480-490 (2016).
- Li, G., et al. Single cell expression analysis reveals anatomical and cell cycle-dependent transcriptional shifts during heart development. Development. 146 (12), dev173476(2019).
- Meilhac, S. M., Buckingham, M. E. The deployment of cell lineages that form the mammalian heart. Nature Reviews Cardiology. 1, (2018).
- Liu, Z., et al. Single-cell transcriptomics reconstructs fate conversion from fibroblast to cardiomyocyte. Nature. 551 (7678), 100(2017).
- Lafzi, A., Moutinho, C., Picelli, S., Heyn, H. Tutorial: guidelines for the experimental design of single-cell RNA sequencing studies. Nature Protocols. 1, (2018).
- The Tabula Muris Consortium. Single-cell transcriptomics of 20 mouse organs creates a Tabula Muris. Nature. 562 (7727), 367(2018).
- Gawad, C., Koh, W., Quake, S. R. Single-cell genome sequencing: current state of the science. Nature Reviews Genetics. 17 (3), 175(2016).
- Grün, D., van Oudenaarden, A. Design and analysis of single-cell sequencing experiments. Cell. 163 (4), 799-810 (2015).
- Ziegenhain, C., et al. Comparative analysis of single-cell RNA sequencing methods. Molecular cell. 65 (4), 631-643 (2017).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17 (1), 77(2016).
- Islam, S., et al. Characterization of the single-cell transcriptional landscape by highly multiplex RNA-seq. Genome Research. 21 (7), 1160-1167 (2011).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161 (5), 1202-1214 (2015).
- Klein, A. M., et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 161 (5), 1187-1201 (2015).
- Library Prep -Single Cell Gene Expression -Official 10x Genomics Support. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/library-prep (2018).
- McGinnis, C. S., et al. MULTI-seq: sample multiplexing for single-cell RNA sequencing using lipid-tagged indices. Nature Methods. 1, (2019).
- Stoeckius, M., et al. Cell hashing with barcoded antibodies enables multiplexing and doublet detection for single cell genomics. Genome Biology. 19 (1), 224(2018).
- Chan, M. M., et al. Molecular recording of mammalian embryogenesis. Nature. 570 (7759), 77-82 (2019).
- Agilent 4200 TapeStation System. , Agilent. https://www.agilent.com/cs/library/datasheets/public/5991-6029EN.pdf (2019).
- SPRIselect User Guide. , Beckman Coulter. https://research.fhcrc.org/content/dam/stripe/hahn/methods/mol_biol/SPRIselect%20User%20Guide.pdf (2012).
- Qubit 4 Fluorometer User Guide. , Thermo Fisher Scientific. https://assets.thermofisher.com/TFS-Assets/LSG/manuals/MAN0017209_Qubit_4_Fluorometer_UG.pdf (2018).
- Agilent High Sensitivity DNA Kit Guide. , Agilent. https://www.agilent.com/cs/library/usermanuals/public/High%20Sensitivity_DNA_KG.pdf (2016).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411(2018).
- Weber, R. J., Liang, S. I., Selden, N. S., Desai, T. A., Gartner, Z. J. Efficient targeting of fatty-acid modified oligonucleotides to live cell membranes through stepwise assembly. Biomacromolecules. 15 (12), 4621-4626 (2014).
- Single Cell Protocols Cell Preparation Guide. , 10x Genomics. https://support.10xgenomics.com/single-cell-gene-expression/sample-prep (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados