Method Article
Teste anticorpo-livre para a análise da atividade do methyltransferase do RNA
Neste Artigo
Resumo
Aqui, um ensaio in vitro anticorpo-livre para a análise direta da atividade do metiltransferase no RNA transcrito sintético ou in vitro é descrito.
Resumo
Há mais de 100 modificações quimicamente distintas do RNA, dois terços dos quais consistem em metilações. O interesse nas modificações do RNA, e especialmente nos methylations, reemergiu devido aos papéis importantes desempenharam pelas enzimas que escrevem e os apagam em processos biológicos relevantes à doença e ao cancro. Aqui, um ensaio in vitro sensível para a análise exata da atividade do escritor do metilação do RNA em RNAs transcritos sintéticos ou in vitro é fornecido. Este ensaio usa um formulário tritiado de S-adenosyl-Methionine, tendo por resultado a rotulagem direta do RNA misturado com trítio. A baixa energia da radiação trítio torna o método seguro, e métodos pré-existentes de amplificação de sinal de trítio, tornam possível quantificar e visualizar o RNA metilado sem o uso de anticorpos, que são comumente propensos a artefatos. Quando este método for escrito para o methylation do RNA, poucos ajustes fazem-no aplicável ao estudo de outras modificações do RNA que podem radioativamente ser etiquetadas, tais como a acetilação do RNA com a coenzima A do acetil 14C. globalmente, este ensaio permite avaliar rapidamente o RNA condições de metilação, inibição com inibidores da molécula pequena, ou o efeito do RNA ou mutantes enzimáticos, e fornece uma ferramenta poderosa para validar e expandir os resultados obtidos nas células.
Introdução
O DNA, o RNA e as proteínas estão sujeitos a modificações que regulam firmemente a expressão gênica1. Entre essas modificações, ocorrem metilações em todos os três biopolímeros. Os methylations do ADN e da proteína foram estudados muito bem durante as últimas três décadas. Ao contrário, o interesse no metilação do RNA foi reacendeu somente recentemente à luz dos papéis importantes que as proteínas que escrevem, apagam ou ligam o methylations do RNA jogam no desenvolvimento e na doença2. Além do que funções mais conhecidas nas RNAs abundantes ribossomal e da transferência, as vias do metilação do RNA regulam a estabilidade específica3,4do RNA do Mensageiro, emenda5 e a tradução6,7, Mirna que processa8,9 e transcricional que pausam e liberam10,11.
Aqui, um método simples e robusto para a verificação in vitro da atividade de RNA metiltransferase no cenário de um laboratório de biologia molecular é relatado (resumido na Figura 1). Muitos estudos avaliam a atividade de um metiltransferase do RNA através do dot-borrão com um anticorpo de encontro à modificação do RNA do interesse. Entretanto, Dot-blot não verifica a integridade do RNA em cima da incubação com o methyltransferase do RNA. Isto é importante porque mesmo as contaminações menores de proteínas de recombinação com nucleases podem conduzir à degradação parcial do RNA e aos resultados confundimento. Além disso, mesmo os anticorpos altamente específicos da modificação do RNA podem reconhecer RNAs não modificado com seqüências ou estruturas específicas. O ensaio in vitro de RNA metiltransferase aqui relatado aproveita o fato de que a S-adenosil metionina pode ser tritiada no doador do grupo metil (Figura 1), permitindo que o RNA metilado seja detectado com precisão sem o uso de anticorpos . As instruções são fornecidas para in vitro a transcrição e a purificação de uma transcrição do interesse e do teste da metilação da transcrição dita pela enzima do interesse. Este método é flexível e robusto, e pode ser ajustado de acordo com as necessidades de um determinado projeto. Por exemplo, in vitro transcritos e purificados RNAs, quimicamente sintetizadas RNAs, mas também RNAs celulares podem ser usados. Este ensaio fornece informações quantitativas a forma de contagens de cintilação, bem como informações qualitativas, mostrando onde exatamente o RNA metilado é executado em um gel. Isto pode fornecer a introspecção original na função de um methyltransferase do RNA, particular ao usar RNAs celulares como um substrato, porque fornece um método para observar diretamente o tamanho do RNA ou dos RNAs que são alvejados para o methylation.
Protocolo
1. a transcrição in vitro e a purificação do gel do RNA alvo
- Clone a seqüência de interesse em plasmíos contendo T7 e/ou SP6 promotores usando técnicas de clonagem molecular estabelecidas12 ou kits.
- Linearização do modelo de DNA para transcrição in vitro
- Amplificar a seqüência de interesse por PCR do plasmídeo com primers projetados para incluir a região promotora T7 através da pastilha, + 20-30 BP upstream e downstream como descrito anteriormente13.
- Alternativamente, digerir 5 μg do plasmídeo utilizando um local de corte da enzima de restrição (RE) disponível a jusante da pastilha de acordo com as instruções fornecidas pelo fornecedor do RE.
Nota: Lembre-se de verificar se a pastilha não foi cortada pelo RE selecionado. A seleção do RE usada nesta etapa pode afetar dramaticamente o rendimento da transcrição. Preferencialmente, linearizar com uma extremidade sem corte ou 5 '-overhanging produzindo o re para evitar a comutação do modelo da polimerase do RNA T7 na costa oposta14do ADN. Tente RE diferente se os rendimentos iniciais forem insuficientes.
- Purify o ADN resultante usando o jogo para a extração e a limpeza do gel do Agarose do ADN. Elute o DNA com 30 μL de água.
- Misture bem por vortexing, Pipet 1,5 μL e medir a concentração de DNA com um Espectrofotômetro de microespectrômetro.
- Use 250 ng do ADN para verific a qualidade e o tamanho do ADN purified em uma electroforese do gel de agarose de 1% no amortecedor de 1x TBE migrado para 1 h em 4 V/cm.
- Descongelar os reagentes congelados do kit de transcrição in vitro. Coloque o T7 RNA polymerase Mix no gelo, e os outros reagentes em um pulverizador em temperatura ambiente. Imediatamente após o descongelar completo, gire rapidamente os tubos rNTP para 5 s para coletar a solução na parte inferior dos tubos e coloc no gelo. Mantenha o buffer de reação 10x à temperatura ambiente para evitar a precipitação.
- Pipet em um tubo de 1,5 mL os seguintes componentes da reação da transcrição de 20 μL na temperatura ambiente na ordem indicada: 6 μL da água, 2 μL (0,1-1 μg) do molde linear do ADN, 2 μL da solução do ATP de 75 milímetros, 2 μL da solução de 75 mM GTP , 2 μL de solução CTP de 75 mM, 2 μL de solução UTP de 75 mM, 2 μL de tampão de reacção 10x e 2 μL de mistura de RNA polimerase T7.
Nota: para o modelo de DNA gerado por PCR, use 100-200 ng DNA; para o modelo do ADN gerado pela digestão da enzima da limitação de um plasmídeo, use ~ 1 μg. recombinante ativo sua6-TAGGED T7 RNA polimerase pode ser purificada em E. coli15. - Misture o tubo completamente. A rotação rápida para 5 s para coletar a solução da reação na parte inferior do tubo, a seguir incubar em 37 ° c para 2-4 h.
Nota: cada transcrição vai se comportar de forma diferente, dependendo do modelo de DNA, seu comprimento, sua seqüência ou estrutura. Otimize as condições de transcrição in vitro testando diferentes tempos de incubação até 6 h; diferentes concentrações de modelo de DNA, T7 RNA polimerase, ou NTP; ou adição de MgCl suplementar2 ao que está presente na mistura de RNA polimerase T7. - No final do período de incubação, adicionar 1 μL de DNase I por 20 μL de reacção e incubar a 37 ° c durante 15 min.
Nota: a reacção de transcrição pode ser temporariamente mantida no gelo até que o gel de poliacrilamida esteja pronto. - Proceder com a purificação por gel de poliacrilamida.
Nota: desde que o RNA é sensível à contaminação do pH e do RNase, use o equipamento dedicado RNA e os reagentes RNase-livres. - Determinar a percentagem de poliacrilamida para o gel, dependendo do tamanho da transcrição de juros: 3,5% para 100-2000 NT (XC: 460 NT; BB: 100 NT), 5% para 80-500 NT (XC: 260 NT; BB: 65 NT), 8% para 60-400 NT (XC: 160 NT; BB: 45 NT), 12% para 40-200 NT (XC: 70 NT; BB: 20 NT), 15% para 25-150 NT (XC: 60 NT; BB: 15 NT), e 20% para 6-100 NT (XC: 45 NT; BB: 12 NT).
- Prepare a ureia desnaturando o gel de poliacrilamida combinando os seguintes reagentes em um tubo cônico de 50 mL: 9,6 g de ureia de grau molecular, 2 mL de 10x TBE, x mL de acrilamida a 40%: mistura de bis-acrilamida (29:1) e água até a marca de 20 mL no tubo , onde x = 20 mL/(40%/percentagem de gel%).
Cuidado: os géis de polyacrylamide contêm frequentemente o acrilamida un-polimerizado que é um material tóxico que possa produzir um perigo quando introduzido ao ambiente. Descarte os géis de poliacrilamida através do programa de resíduos químicos da instituição. - Microondas para 15 s a 30% de potência. Coloque no pulverizador por 10 min à temperatura ambiente ou até que a ureia tenha sido completamente dissolvida.
- Enquanto a mistura de gel está girando, recuperar uma gaveta de gel (18-bem, 1 mm de espessura; 13,3 x 8,7 cm (W x L)), retire o pente e posicione a gaveta para fundição de gel.
- Gire rapidamente o tubo de 50 mL para 5 s para coletar a solução do gel na parte inferior do tubo. Adicionar 125 μL de solução de persulfato de amónio a 10% (APS). Coloque no pulverizador para ~ 1 min. Quick spin novamente por 5 s.
- Adicione 25 μL de tetrametilenodiamina (TEMED) e misture cuidadosamente pipetando para cima e para baixo 5 vezes com uma pipeta de 25 mL evitando bolhas.
- Pipeta no molde do gel e insira com cuidado o pente do gel que evita bolhas.
- Aperte o selo adicionando um grande grampo da pasta sobre a parte superior da gaveta.
- Permita que o gel polimerize por 1 h.
- Uma vez que o gel solidified, retire o grampo da pasta. Insira o cartucho na caixa de eletroforese. Adicione o amortecedor de 1x TBE nos reservatórios superiores e inferiores.
- Recupere a reacção de transcrição. Adicione água até 100 μL e, em seguida, adicione 100 μL de tampão de carregamento de gel 2x. Prepare a escada misturando a quantidade recomendada com água a 10 μL e 10 μL de tampão de carregamento do gel 2x a um tubo separado de 1,5 mL.
- Incubar a escada e a reacção de transcrição in vitro no termomisturador a 70 ° c durante 15 min.
- Enquanto as amostras estão desnaturando a 70 ° c, Retire cuidadosamente o pente, limpe os poços pipetando para cima e para baixo cada poço com uma pipeta P1000, e imediatamente Pre-Run por 10 min em 100 V.
- Uma vez que as amostras tenham completado a sua desnaturação de 15 min a 70 ° c, retire os tubos do termomisturador e coloque imediatamente no gelo.
- Limpe cada poço do gel novamente com a pipeta P1000. Carregar 20 μl da escada no primeiro poço à esquerda, 20 μL da amostra de RNA em 10 poços separados, e 20 μL de tampão de carregamento do gel 1x nos poços não utilizados.
- Execute o gel em 100 V para 60-240 min, dependendo do% poliacrilamida do gel.
Nota: os corantes no tampão de carregamento do gel serão separados em duas bandas à medida que atravessam o gel, uma faixa azul de bromofenol azul (BB) migrante lenta e uma banda ciano de xylene cyanol (XC) de migração rápida. A migração aproximada destas bandas em diferentes% de poliacrilamida géis é bem conhecida (ver passo 1,11) e pode ser usada para estimar a migração de RNA no gel. - Uma vez que o gel foi parado, Retire cuidadosamente o gel da gaveta.
- Coloc o gel em uma caixa limpa que contem uma solução de 50 mL do amortecedor de 1x TBE com 50 μL da mancha do gel do ácido nucleico e incubar por 5 minutos em um balancim para manchar o RNA.
- Tome um antes e após a imagem da excisão da faixa do gel no sistema da imagem latente do gel, preferivelmente usando a luz azul em vez da transiluminação UV.
- Extirpar cada faixa de interesse usando uma ponta descartável nuclease-livre do corte do gel. Depois de cada poço, transfira a ponta contendo a fatia de gel para um tubo de 1,5 mL e gire brevemente para recolher a fatia de gel. Repetir até que todas as bandas tenham sido recolhidas no mesmo tubo de 1,5 mL.
- Uma vez recolhidas todas as fatias de gel, adicione 100 μL de água sem nuclease ou tampão TE ao tubo de 1,5 mL. Conservar a 4 ° c para ~ 48 h. Isto permite que o RNA saia das fatias do gel na solução.
- Depois de 48 h, Pipet a água ou tampão TE para um tubo de 1,5 mL fresco. Elimine as restantes fatias de gel. Purify o RNA através do jogo da limpeza como segue.
- Equilibrar a coluna de giro à temperatura ambiente durante pelo menos 30 min.
- Para o 100 μL de solução de RNA no novo tubo de 1,5 mL da etapa 1,32, adicione 350 μL de tampão RLT e misture bem por 2 min em um Nutator. Gire por 1 s em 200 x g para coletar a solução na parte inferior do tubo.
- Adicione 675 μL de 100% de EtOH e misture bem durante 2 min no Nutator. Gire por 1 s em 200 x g e prossiga imediatamente para o próximo passo.
- Transferir 565 μL da mistura para a coluna de centrifugação e girar durante 1 min a 15.000 x g. Esvazie o tubo de recolha por aspiração.
- Repita a etapa anterior com a segunda metade da amostra.
- Adicione 500 μL de tampão de RPE à coluna e gire por 1 minuto em 15.000 x g. Esvazie o tubo de recolha por aspiração.
- Adicionar 750 μL de 80% de etanol à coluna e girar por 1 min a 15.000 x g. Esvazie o tubo de recolha por aspiração.
- Coloque a coluna em um novo tubo de coleta de 2 mL com a tampa aberta e gire em 15.000 x g por 5 min.
- Transfira a coluna para um novo tubo de 1,5 mL.
- Adicione 17 μL de água no centro da coluna e gire em 15.000 x g por 1 min para elute. Elute novamente usando mais 17 μL de água. O volume total recuperado deve ser de 32 μL.
- Misture bem por vortexing, Pipet 1,5 μL e meça a concentração do RNA usando um Espectrofotômetro de microespectrômetro.
- Verific a qualidade da purificação do RNA pelo gel de desnaturação do poliacrilamida da ureia como nas etapas 1.11-1.29.
2. ensaio in vitro de RNA metiltransferase
- Configurar o ensaio de 100 μL de RNA metiltransferase num tubo de 1,5 mL no gelo da seguinte forma: 23 μL de água, 10 μL de TBS 10x (500 mM de Tris-HCl, pH 7,5; 1,5 M NaCl), 2 μL de 0, 5 M de EDTA, 5 μL de 100 mM DTT , 40 μL de 50% de glicerol, 4 μL de 58 μM 3H-Sam, 5 μL de cocktail inibidor da protease 20x, 1 μL de RNaseOUT (opcional), 5 ΜL de RNA e 5 μL de metiltransferase.
Cuidado: o material tritiado radioativo é perigoso e deve somente ser segurado ao desgastar luvas, um revestimento do laboratório e todo o outro PPE necessário. Todas as pontas e tubos de pipeta em contato com material radioativo são considerados resíduos radioativos sólidos. Elimine todos os resíduos radioactivos sólidos e líquidos de acordo com o protocolo de resíduos radioactivos aprovados pelo laboratório.
Nota: inclua amostras de controle sem o metiltransferase e sem o RNA. As concentrações do reagente podem exigir a optimização e/ou a inclusão de sais divalentes do cation, tais como MgCl2, ou Sam sem rótulo. A escala óptima da concentração final do RNA é de 50 nanômetro a 1 μm, quando a concentração do metiltransferase for de 25 nanômetro a 300 nanômetro. - Misture cuidadosamente o tubo. Girar 5 s em 200 x g para recolher a solução na parte inferior do tubo. Incubar o tubo (s) a 37 ° c durante 2 h.
- Limpe a reação usando a purificação da coluna como na etapa 1,32.
Cuidado: tenha muito cuidado para dispor adequadamente de quaisquer materiais radioactivos, especialmente durante as lavagens de coluna (pipeta para fora em vez de aspirantes a resíduos de tubos de recolha). - Realizar contagem de cintilação líquida
- Configure o rack de contagem de cintilação com um frasco por amostra, um frasco para injetáveis para medição de fundo e um frasco para injetáveis para o teste de furto. Encha os frascos com 5 mL de solução de contagem de cintilação.
- Adicione 10 μL de cada amostra de RNA radioativo eluído em 1 frasco para injetáveis e aperte a tampa e misture suavemente.
- Prepare os frascos de teste de furto. Esfregue cuidadosamente cotonetes em todas as superfícies e equipamentos utilizados durante o protocolo. Adicionar cotonetes aos frascos preenchidos com 5 mL de solução de cintilação e apertar a tampa.
- Execute as amostras no contador de cintilação da seguinte maneira. Abra a capa contrária, insira o rack na máquina e feche o capô. Selecione contar único rack. Selecione selecionar programade usuário. Selecione ou crie um programa que mede trítio (3H) para 60 s. hit Count rack. Repita a contagem de cintilação três vezes.
Nota: o equipamento irá medir a contagem de cintilação de cada amostra e saída para a tela e uma impressão. O protocolo pode ser pausado aqui se desejado. As amostras de RNA remanescentes da etapa 2,3 devem ser congeladas em-80 ° c para uso posterior.
- Prossiga com o autoradiograma
- Prepare e Pre-Run uréia desnaturando gel de poliacrilamida como nas etapas 1.11-1.20.
- Pipete 20 μL de material de RNA radioactivo para um novo tubo de 1,5 mL contendo 20 μL de tampão de carregamento de gel 2x. Misture bem. Prepare a escada como na etapa 1,21. Incubar as amostras a 70 ° c durante 15 min.
- Lave os poços do gel mais uma vez imediatamente antes da carga da amostra. Carregar 20 μl da escada preparada, 20 μL das amostras e 20 μL de tampão de carga do gel 1x nas vias restantes. Executar o gel em 100 V por 60-180 min, dependendo da percentagem de poliacrilamida.
- Uma vez que o gel termine de funcionar, retire o gel da gaveta e coloque-o numa caixa contendo 50 ml de tampão TBE 1x com 5 μL de mancha de gel de ácido nucleico ultrassensível.
- Incubar por 5 min no balancim para manchar o RNA.
- Tome com cuidado o gel fora da caixa e coloc o no transiluminador UV do sistema da imagem latente do gel com os poços acima e a escada na esquerda.
- Concentre a câmera no gel, gire sobre a luz UV e tome então uma imagem do gel expondo de 50 ms a 1 s dependendo da intensidade do sinal.
- Desative a exposição UV e salve a imagem como arquivo TIFF.
- Coloque o gel de volta na caixa. Remover TBE. Fixar o gel com 50 mL de solução de fixação (50% de metanol, 10% de ácido acético, 40% de água ultraleve) durante 30 min à temperatura ambiente num balancim.
- Mova suavemente o gel novamente para uma caixa preta fresca contendo 25 mL da solução de melhoramento da autoradiografia. Na ausência de uma caixa preta, cubra a caixa com a folha de alumínio para proteger a solução da luz.
- Incubar durante 30 min à temperatura ambiente no balancim.
- Levante suavemente o gel e coloque-o virado para baixo sobre uma folha de plástico com os poços para cima e a escada do lado direito. Coloque duas folhas de papel de cromatografia na parte de trás do gel. Vire suavemente a pilha inteira.
- Pré-aqueça o secador do gel a 80 ° c. Afaste a tampa plástica do secador de gel. Insira o envoltório, o gel e a pilha de papel da cromatografia a tampa plástica e mova a tampa plástica de volta para baixo para criar um selo.
- Seque por 1 h a 80 ° c no secador de gel.
- Desligue o secador de gel e retire suavemente a pilha. Retire o envoltório e o segundo papel de cromatografia. Tape o papel restante da cromatografia com o gel secado em uma gaveta do corresponde.
- Adicione 1 folha de filme autoradiografia na sala escura.
- Coloque a gaveta a-80 ° c e desenvolva a película após 1 h a 4 semanas dependendo da intensidade do sinal, que pode ser julgada com base nas contagens de cintilação previamente medidas: 1-4 semanas para 250-1000 CPM, 24 h a 1 semana para 1000-10000 CPM e 1 h a 24 h para > 10000 CP M.
- Uma vez que o filme é desenvolvido, coloque o filme em cima da gaveta e cuidadosamente marcar com um marcador de laboratório as 4 bordas do gel, cada poço, e a posição dos corantes XC e BB.
- Digitalizar o filme em 300 ou 600 pixels por polegada de resolução e salvar a imagem como arquivo TIFF.
Resultados
Reacção de transcrição in vitro
Figura 2 A representa uma corrida típica de uma reação de transcrição in vitro com o RNA polimerase T7 do 7Sk snRNA, que é um RNA relativamente curto (331 NT) e altamente estruturado. Como mostrado nessa imagem crua, existem várias bandas indesejadas, tanto mais curtas quanto mais longas que 7SK, provavelmente resultantes de eventos aleatórios de iniciação ou terminação transcripcional. Devido a isso, a purificação do gel após a reação de transcrição in vitro é importante para obter uma amostra de RNA limpa, como mostrado na Figura 2B. Neste ponto, o RNA de interesse pode ser identificado por sua localização em relação à escada e purificada pela purificação do gel.
Como mencionado anteriormente, pode ser necessário otimizar o comprimento da reação de transcrição antes da etapa de purificação do gel. As reações de transcrição que correm por muito tempo podem resultar em quantidades extremamente elevadas de produção de RNA, tornando difícil a identificação a jusante da transcrição correta. Também é importante verificar a identidade do transcrito purificado por transcrição reversa e PCR quantitativo (RTqPCR) usando primers específicos.
Ensaio in vitro de RNA metiltransferase
A Figura 3 mostra um resultado representativo de um ensaio de RNA metiltransferase descrito no protocolo usando o limite inferior de nossas concentrações recomendadas de RNA e proteína. Este ensaio permite os resultados quantitativos das contagens de cintilação, bem como os resultados qualitativos do autoradiograma. Aqui, mepce, um metiltransferase do RNA sabido para metilato 7sk10, foi mostrado para poder igualmente metilato U6. Além disso, como mostrado recentemente para 7SK16, a ligação de histona H4 a MePCE também inibe a metilação U6. Pudemos observar isso tanto na contagem de cintilação (Figura 3C) quanto no autoradiograma (Figura 3B), mostrando a robustez deste protocolo.
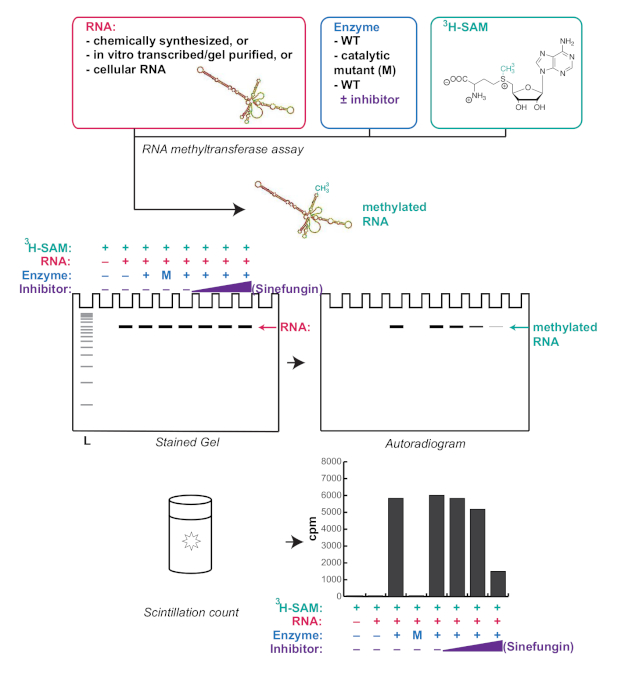
Figura 1 . Respresentação esquemática de um fluxo de trabalho típico do ensaio do metiltransferase do RNA e resultados esperados. O sinefungin é um inibidor competitivo da metiltransferase. L: escada; WT: tipo selvagem; M: mutante catalítico; CPM: contagens por min. por favor clique aqui para ver uma versão maior desta figura.
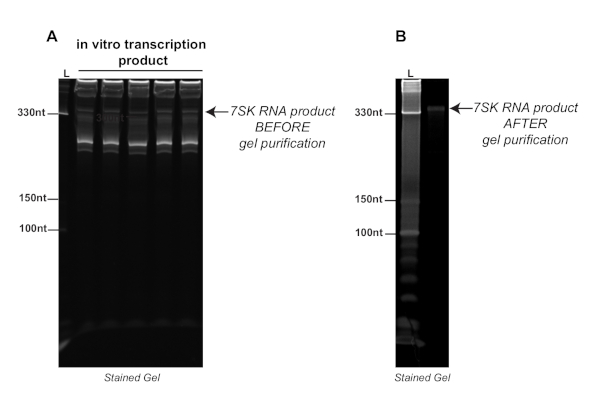
Figura 2 . Experimento representativo mostrando o produto da transcrição in vitro antes (A) e após (B) purificação em um gel de desnaturação de ureia-poliacrilamida 8% corado com coloração de ácido nucleico. As setas apontam para a transcrição 7SK antes e após a purificação do gel. L: escada. Por favor clique aqui para ver uma versão maior desta figura.

Figura 3 . Ensaio in vitro de metil-transferase de RNA realizado com MePCE contra U6. Ensaio in vitro de metiltransferase utilizando GST-MePCE recombinante (25 nm), 3H-Sam radioativo como doador do grupo metilo e transcrito in vitro U6 RNA (50 nm) como substrato. O autoradiograma foi exposto por 2 semanas, a fim de detectar a atividade residual (250 CPM) na amostra + histona H4. Por favor clique aqui para ver uma versão maior desta figura.
Discussão
Aqui, um método simples e robusto para a verificação in vitro da atividade do metiltransferase do RNA para transcritos específicos é relatado. O ensaio aproveita o fato de que a metionina S-adenosil pode ser tritiada no doador do grupo metil (Figura 1), permitindo que o RNA metilado seja detectado com precisão sem o uso de anticorpos. No entanto, é importante notar que este ensaio não pode indicar qual resíduo ou grupo químico é metilado pela enzima. Para identificar ou verificar se um resíduo específico é metilado, outros métodos como a análise mutacional, o bloco de transcrição reversa ou a análise de espectrometria de massa do substrato de RNA podem ser usados em conjunto com o ensaio de RNA metiltransferase.
A escolha de RNAs e seu modo de síntese depende do tamanho do RNA. RNAs específicas de tamanhos entre 18 e 120 NT pode ser convenientemente personalizado-sintetizado a partir de muitos fornecedores de ácidos nucleicos de renome. No entanto, mais comumente, RNAs específicas de vários tamanhos são transcritas in vitro. Os modelos de DNA para transcrição in vitro podem ser gerados de várias maneiras e exigem que a sequência de interesse seja localizada a jusante de um promotor T7 ou SP6. Quando extremidades precisas de RNAs são importantes, o plasmídeo pRZ é recomendado17. A alta sensibilidade do ensaio (Figura 3) também permite substituir um RNA purificado com uma mistura de RNA celular, total ou RNAs de mensageiro, por exemplo. De fato, o gel e o autoradiograma fornecem um método para observar diretamente o tamanho do RNA (s) direcionado para a metilação.
As condições relatadas aqui na etapa 2,1 do protocolo são ideais para a família BIN3 de metiltransferases, que tem dois homólogos em humanos, mepce10 e BCDIN3D9. É importante ressaltar que as condições do ensaio precisam ser ajustadas à proteína específica e ao RNA de interesse. Por exemplo, mostrou-se que a presença de MgCl2 diminui a atividade de BCDIN3D18, entretanto, MgCl2 pode ser um coordenador importante da estrutura do RNA, e poderia assim constituir um componente importante do ensaio para o outro RNA metiltransferases.
A vantagem de usar um autoradiograma, que pode ser exposto por um longo período de tempo, é que ele pode permitir detectar atividade muito fraca que não é detectada no ensaio de cintilação. Geralmente isso indica que a proteína é uma metiltransferase, mas que uma ou mais das condições de reação ou reagentes precisam ser otimizados: enzima; substrato, cofactor, condições tampão, etc9. Por exemplo, a purificação enzimática pode precisar ser melhorada para remover ou alterar a posição da tag. Também pode ser que o RNA usado como um substrato precisa ser dobrado corretamente ou para interagir com outra proteína ou fator de RNA a ser metilado pela enzima. Assim, a literatura pré-existente e/ou os próprios resultados nas pilhas precisam de ser revistos com cuidado para ajustar as condições do ensaio para a enzima e o RNA do interesse.
O ensaio também é extremamente flexível. Por exemplo, pode ser um passo precursor para outro tipo de ensaio, de tal forma que o RNA radioativamente metilado é utilizado em ensaios quantitativos e qualitativos de demetilase11, ou em ensaios de RNA Binding, permitindo Visualizar especificamente o comportamento do ARN metilado comparado com o não modificado. O ensaio também pode ser levemente ajustado para analisar a modificação de RNAs que utilizam coenzimas que podem ser rotuladas radioativamente, como a Acetil Coenzima a para o estudo da acetilação do RNA19,20.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores gostariam de agradecer ao Dr. Turja Kanti Debnath por sua ajuda com a ChemDraw. A pesquisa no laboratório de Xhemalçe é apoiada pelo departamento de defesa-programa de investigação médica dirigido Congressionally-prêmio da descoberta do cancro da mama (W81XWH-16-1-0352), concessão de NIH R01 GM127802 e fundos do start-up do Instituto de celular e Biologia molecular e faculdade de ciências naturais da Universidade do Texas em Austin, EUA.
Materiais
| Name | Company | Catalog Number | Comments |
| 10 bp DNA Ladder | Invitrogen | 10821-015 | 10 bp DNA Ladder kit. |
| 10% Ammonium Persulfate (APS) | N/A | N/A | For urea denaturing polyacrylamide gel (For 10 mL, dissolve 1g in 8 mL of milliQ water; adjust volume to 10 mL with milliQ water; filter the solution using a 10 mL syringe equiped with a 0.45 µm filter). |
| 10X TBE Buffer | N/A | N/A | For urea denaturing polyacrylamide gel (For 1L, add 108 g of Tris Base, 55 g of Boric Acid to a cylinder with a stir bar; add 800 mL of distilled water and let dissolve; add 40mL of 0.5 M Na2EDTA (pH 8.0); adjust volume to 1L with milliQ water; filter the solution using a 0.22µm filter). |
| 10X TBS | N/A | N/A | For 1L, add 60.5 g of Tris Base, 87.6 g of NaCl to a cylinder with a stir bar; add 800 mL of distilled water and let dissolve; adjust pH to 7.5 with concentrated HCl; adjust volume to 1L with milliQ water; filter the solution using a 0.22 µm filter. |
| Acrylamide: Bis-Acrylamide 29:1 (40% Solution/Electrophoresis), Fisher BioReagents | Fisher | BP1408-1 | For urea denaturing polyacrylamide gel. |
| ADENOSYL-L-METHIONINE, S-[METHYL-3H]; (SAM[3H]) | Perkin Elmer | NET155V250UC | For in vitro methylation of RNA; Concentration = 1.0 mCi/mL; Specific activity = 17.1 Ci/mmol; Molarity= (1.0 Ci/L)/(83.2 Ci/mmol) = 0.0584 mmol/L = 58.4 µM. Upon receipt of the frozen 3H-SAM tube, thaw it at 4°C, make 20 µL aliquots, and freeze them at -30°C. Never refreeze and reuse a partially used aliquot. |
| Amersham Hypercassette Autoradiography Cassettes | GE Healthcare | RPN11649 | For autoradiogram gel exposure. |
| Amersham Hyperfilm MP | GE Healthcare | 28906846 | For autoradiogram gel exposure. |
| Beckman Scintillation Counter | Beckman | LS6500 | For liquid scintillation count. |
| Biorad Mini Horizontal Electrophoresis System | Biorad | 1704466 | Mini Horizontal Electrophoresis System. |
| cOmplete Mini EDTA-free Protease Inhibitor Cocktail Tablets | Roche Applied Science | 4693159001 | For a 20X solution, dissolve 1 tablet in 0.525 mL of nuclease free water. |
| Criterion Cell | Biorad | 345-9902 | RNase free empty cassette for polyacrylamide gel. |
| Criterion empty Cassettes | Biorad | 1656001 | Vertical midi-format electrophoresis cell. |
| DeNovix DS-11 Microvolume Spectrophotometer | DeNovix | DS-11-S | Microvolume Spectrophotometer for measuring DNA and RNA concentration. |
| Ecoscint Original | National Diagnostics | LS-271 | For liquid scintillation count. |
| Fisherbrand 7mL HDPE Scintillation Vials | Fisher | 03-337-1 | For liquid scintillation count. |
| Fluoro-Hance-Quick Acting Autoradiography Enhancer | RPI CORP | 112600 | For autoradiogram gel pretreatment. |
| Gel dryer | Biorad | 1651745 | For drying gel. |
| Gel Loading Buffer II | Ambion | AM8547 | For loading RNA in denaturing polyacrylamide urea gel (composition: 95% Formamide, 18 mM EDTA, and 0.025% SDS, Xylene Cyanol, and Bromophenol Blue). |
| GeneCatcher disposable gel excision tips | Gel Company | NC9431993 | For removing bands from agarose and polyacrylamide gels. |
| Megascript Kit | Ambion | AM1333 | For in vitro transcription with T7 RNA polymerase. |
| Perfectwestern Extralarge Container | Genhunter Corporation | NC9226382 (clear)/ NC9965364 (black) | Gel staining box. |
| pRZ | Addgene | #27663 | Plasmid for producing in vitro transcripts with homogeneous ends |
| Qiagen RNeasy MinElute Cleanup | Qiagen | 74204 | For RNA clean-up, use modified protocol provided in the protocol. |
| QIAquick Gel Extraction Kit (50) | Qiagen | 28704 | Kit for gel extraction and clean up of dsDNA fragment used for in vitro transcription. |
| Saran Premium Plastic Wrap | Saran Wrap | Amazon | For drying gel. |
| SYBR Gold | Invitrogen | S11494 | Ultra sensitive nucleic acid gel stain. |
| SYBR Safe | Invitrogen | S33102 | Nucleic acid gel stain. |
| TE | Sigma | 93283-100ML | 10 mM Tris-HCl, 1 mM disodium EDTA, pH 8.0 |
| TEMED | Fisher | 110-18-9 | For urea denaturing polyacrylamide gel. |
| Thermomixer with SMARTBLOCK 24X 1.5mL TUBES | eppendorf | 5382000023/5361000038 | For temperature controlled incubation of 1.5 mL tubes. |
| TOPO TA Cloning Kit | life technologies | Kits for fast cloning of Taq polymerase–amplified PCR products into vectors containing T7 and/or SP6 promoters for in vitro RNA transcription. | |
| TURBO DNase (2 U⁄µL) | Ambion | AM2238 | For DNA removal from in vitro transcription reactions. |
| Urea | Sigma | 51456-500G | For urea denaturing polyacrylamide gel. |
| Whatman 3MM paper | GE Healthcare | 3030-154 | Chromatography paper for drying gel. |
Referências
- Xhemalce, B. From histones to RNA: role of methylation in cancer. Briefings in Functional Genomics. 12 (3), 244-253 (2013).
- Shelton, S. B., Reinsborough, C., Xhemalce, B. Who Watches the Watchmen: Roles of RNA Modifications in the RNA Interference Pathway. PLoS Genetics. 12 (7), 1006139 (2016).
- Mauer, J., et al. Reversible methylation of m(6)Am in the 5' cap controls mRNA stability. Nature. 541 (7637), 371-375 (2017).
- Wang, X., et al. N6-methyladenosine-dependent regulation of messenger RNA stability. Nature. 505 (7481), 117-120 (2014).
- Pendleton, K. E., et al. The U6 snRNA m(6)A Methyltransferase METTL16 Regulates SAM Synthetase Intron Retention. Cell. 169 (5), 824-835 (2017).
- Meyer, K. D., et al. 5' UTR m(6)A Promotes Cap-Independent Translation. Cell. 163 (4), 999-1010 (2015).
- Wang, X., et al. N(6)-methyladenosine Modulates Messenger RNA Translation Efficiency. Cell. 161 (6), 1388-1399 (2015).
- Alarcon, C. R., Lee, H., Goodarzi, H., Halberg, N., Tavazoie, S. F. N6-methyladenosine marks primary microRNAs for processing. Nature. 519 (7544), 482-485 (2015).
- Xhemalce, B., Robson, S. C., Kouzarides, T. Human RNA methyltransferase BCDIN3D regulates microRNA processing. Cell. 151 (2), 278-288 (2012).
- Jeronimo, C., et al. Systematic analysis of the protein interaction network for the human transcription machinery reveals the identity of the 7SK capping enzyme. Molecular Cell. 27 (2), 262-274 (2007).
- Liu, W., et al. Brd4 and JMJD6-associated anti-pause enhancers in regulation of transcriptional pause release. Cell. 155 (7), 1581-1595 (2013).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. Journal of Visual Experimentation. , (2019).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. Journal of Visual Experimentation. (63), e3998 (2012).
- Rong, M., Durbin, R. K., McAllister, W. T. Template strand switching by T7 RNA polymerase. Journal of Biological Chemistry. 273 (17), 10253-10260 (1998).
- Rio, D. C. Expression and purification of active recombinant T7 RNA polymerase from E. coli. Cold Spring Harb Protocols. 2013 (11), (2013).
- Shelton, S. B., et al. Crosstalk between the RNA Methylation and Histone-Binding Activities of MePCE Regulates P-TEFb Activation on Chromatin. Cell Reports. 22 (6), 1374-1383 (2018).
- Walker, S. C., Avis, J. M., Conn, G. L. General plasmids for producing RNA in vitro transcripts with homogeneous ends. Nucleic Acids Research. 31 (15), 82 (2003).
- Blazer, L. L., et al. A Suite of Biochemical Assays for Screening RNA Methyltransferase BCDIN3D. SLAS Discovery. 22 (1), 32-39 (2017).
- Arango, D., et al. Acetylation of Cytidine in mRNA Promotes Translation Efficiency. Cell. 175 (7), 1872-1886 (2018).
- Ito, S., et al. Human NAT10 is an ATP-dependent RNA acetyltransferase responsible for N4-acetylcytidine formation in 18 S ribosomal RNA (rRNA). Journal of Biological Chemistry. 289 (52), 35724-35730 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados