Method Article
Analyse d'activité sans anticorps pour l'analyse de l'activité de méthyltransferase d'ARN
Dans cet article
Résumé
Ici, un analyse in vitro sans anticorps pour l'analyse directe de l'activité de méthyltransferase sur l'ARN synthétique ou in vitro transcrit est décrit.
Résumé
Il y a plus de 100 modifications chimiquement distinctes de l'ARN, dont les deux tiers se composent de méthylations. L'intérêt pour les modifications de l'ARN, et en particulier les méthylations, est réapparu en raison des rôles importants joués par les enzymes qui les écrivent et les effacent dans les processus biologiques pertinents à la maladie et au cancer. Ici, un analyse in vitro sensible pour l'analyse précise de l'activité d'auteur de méthylation d'ARN sur les ARN transcrits synthétiques ou in vitro est fourni. Cet exemple utilise une forme tritiée de S-adenosyl-méthionine, résultant en l'étiquetage direct de l'ARN méthylé avec du tritium. La faible énergie du rayonnement tritium rend la méthode sûre, et les méthodes préexistantes d'amplification du signal de tritium, permettent de quantifier et de visualiser l'ARN méthylé sans l'utilisation d'anticorps, qui sont généralement sujettes aux artefacts. Bien que cette méthode soit écrite pour la méthylation de l'ARN, peu de réglages le rendent applicable à l'étude d'autres modifications de l'ARN qui peuvent être étiquetées radioactivement, comme l'acétylation d'ARN avec 14C coenzyme acétyle A. Dans l'ensemble, cet essai permet d'évaluer rapidement l'ARN conditions de méthylation, l'inhibition avec les inhibiteurs de petites molécules, ou l'effet de l'ARN ou des mutants enzymatiques, et fournit un outil puissant pour valider et élargir les résultats obtenus dans les cellules.
Introduction
L'ADN, l'ARN et les protéines sont sujets à des modifications qui régulent étroitement l'expression des gènes1. Parmi ces modifications, les méthylations se produisent sur les trois biopolymères. L'ADN et les protéines méthylations ont été très bien étudiés au cours des trois dernières décennies. En revanche, l'intérêt pour la méthylation de l'ARN n'a été relancé que récemment à la lumière des rôles importants que les protéines qui écrivent, effacent ou lient les méthylations de l'ARN jouent dans le développement et la maladie2. En plus de fonctions mieux connues dans l'abondante ribosomal et le transfert RNAs, voies de méthylation ARN réguler la stabilité spécifique de l'ARN messager3,4, épissage5 et traduction6,7, traitement miRNA8,9 et pause transcriptionnelle et la libération10,11.
Ici, une méthode simple et robuste pour la vérification in vitro de l'activité de méthyltransferase d'ARN dans le cadre d'un laboratoire de biologie moléculaire est rapportée (résumée dans la figure 1). Beaucoup d'études évaluent l'activité d'un méthyltransferase d'ARN par le point-blot avec un anticorps contre la modification d'ARN de l'intérêt. Cependant, point-blot ne vérifie pas l'intégrité de l'ARN lors de l'incubation avec le méthyltransferase d'ARN. Ceci est important parce que même les contaminations mineures des protéines recombinantes avec des nucléases peuvent conduire à une dégradation partielle de l'ARN et des résultats confondants. En outre, même les anticorps très spécifiques de modification d'ARN peuvent reconnaître les ARN non modifiés avec des séquences ou des structures spécifiques. L'analyse in vitro de méthyltransferase d'ARN rapportée ici tire parti du fait que la méthionine de S-adenosyl peut être tritiated sur le donneur de groupe méthyle (figure 1), permettant que l'ARN méthylé soit détecté avec précision sans l'utilisation des anticorps . Des instructions sont fournies pour la transcription in vitro et la purification d'une transcription d'intérêt et de l'essai de la méthylation de ladite transcription par l'enzyme d'intérêt. Cette méthode est flexible et robuste, et peut être ajustée en fonction des besoins de n'importe quel projet donné. Par exemple, les ARN transcrits et purifiés in vitro, les ARN synthétisés chimiquement, mais aussi les ARN cellulaires peuvent être utilisés. Cet essai fournit l'information quantitative sous forme de compte de scintillation, aussi bien que l'information qualitative en montrant où exactement l'ARN méthylé fonctionne sur un gel. Cela peut fournir un aperçu unique de la fonction d'un ARN méthyltransferase, en particulier lors de l'utilisation des ARN cellulaires comme substrat, car il fournit une méthode pour observer directement la taille de l'ARN ou des ARN qui sont ciblés pour la méthylation.
Protocole
1. Transcription in vitro et purification de gel de l'ARN cible
- Clonez la séquence d'intérêt en plasmides contenant des promoteurs T7 et/ou SP6 en utilisant des techniques de clonage moléculaire établies12 ou des kits.
- La linéarisation du modèle d'ADN pour la transcription in vitro
- Amplifiez la séquence d'intérêt par PCR du plasmide avec des amorces conçues pour inclure la région de promoteur T7 par l'insertion, 20-30 bp en amont et en aval comme décrit précédemment13.
- Vous pouvez également digérer 5 g de plasmide à l'aide d'un site de coupe en zymace (RE) disponible en aval de l'encart selon les instructions fournies par le fournisseur de l'ER.
REMARQUE : N'oubliez pas de vérifier que l'insert n'est pas coupé par l'ER sélectionné. La sélection de l'ER utilisée dans cette étape peut avoir une incidence considérable sur le rendement de la transcription. De préférence, linéarisez-vous avec une extrémité émoussée ou 5'-surplombant produisant RE pour éviter la commutation de modèle de la polymérase d'ARN T7 sur le brin d'ADN opposé14. Essayez des RE différents si les rendements initiaux sont insuffisants.
- Purifez l'ADN résultant à l'aide du kit pour l'extraction et le nettoyage du gel d'algrose d'ADN. Élichez l'ADN avec 30 l'eau.
- Bien mélanger par vortex, pipet 1,5 l et mesurer la concentration d'ADN à l'aide d'un spectrophotomètre à microvolume.
- Utilisez 250 ng d'ADN pour vérifier la qualité et la taille de l'ADN purifié sur une électrophorèse de gel d'agarose de 1% dans 1x tampon TBE migré pendant 1 h à 4 V/cm.
- Décongeler les réactifs congelés du kit de transcription in vitro. Placer le mélange de polymère d'ARN T7 sur la glace et les autres réactifs sur un nutator à température ambiante. Immédiatement après le dégel complet, faire tourner rapidement les tubes rNTP pendant 5 s pour recueillir la solution au fond des tubes et les placer sur la glace. Gardez le tampon de réaction 10x à température ambiante pour éviter les précipitations.
- Pipet dans un tube de 1,5 ml les composants suivants de la réaction de transcription de 20 l à température ambiante dans l'ordre indiqué : 6 l d'eau, 2 l (0,1 - 1 g) de modèle d'ADN linéaire, 2 l de 75 mM solution ATP, 2 l de 75 mM solution GTP , 2 l de 75 mM de solution CTP, 2 l de 75 mM de solution UTP, 2 l de 10x Reaction Buffer et 2 OL de T7 RNA Polymerase Mix.
REMARQUE : Pour le modèle d'ADN généré par PCR, utilisez 100 à 200 ng d'ADN; pour le modèle d'ADN généré par le digest d'enzyme de restrictiond'un plasmide, utilisez 1 g. Recombinant actif Sa polymère d'ARN T7 6-étiquetée peut être purifiée dans E. coli15. - Mélanger le tube à fond. Rotation rapide pour 5 s pour recueillir la solution de réaction au fond du tube, puis incuber à 37 oC pendant 2-4 h.
REMARQUE : Chaque transcription se comportera différemment selon le modèle d'ADN, sa longueur, sa séquence ou sa structure. Optimiser les conditions de transcription in vitro en testant différents temps d'incubation jusqu'à 6 h; différentes concentrations de modèle d'ADN, T7 RNA Polymerase, ou NTP; ou l'ajout de MgCl2 supplémentaire à ce qui est présent dans le mélange de polymère d'ARN T7. - À la fin de la période d'incubation, ajouter 1 l de DNase I par réaction de 20 L et incuber à 37 oC pendant 15 min.
REMARQUE : La réaction de transcription peut être temporairement maintenue sur la glace jusqu'à ce que le gel de polyacrylamide soit prêt. - Procéder à la purification par gel polyacrylamide.
REMARQUE : Puisque l'ARN est sensible au pH et à la contamination par la NAD, utilisez de l'équipement dédié à l'ARN et des réactifs exempts de RNase. - Déterminer le pourcentage de polyacrylamide pour le gel en fonction de la taille de la transcription d'intérêt: 3,5% pour 100-2000 nt (XC: 460 nt; BB: 100 nt), 5% pour 80-500 nt (XC: 260 nt; BB: 65 nt), 8% pour 60-400 nt (XC: 160 nt; BB: 45 nt), 12% pour 40-200 nt (XC: 70 nt; BB: 20 nt), 15% pour 25-150 nt (XC: 60 nt; BB: 15 nt), et 20% pour 6-100 nt (XC: 45 nt; BB: 12 nt).
- Préparer le gel polyacrylamide dénaturant l'urée en combinant les réactifs suivants dans un tube conique de 50 ml : 9,6 g d'urée moléculaire, 2 mL de 10x TBE, x mL de mélange acrylamide:bis-acrylamide (29:1) et de l'eau jusqu'à la marque de 20 mL sur le tube , où x -20 mL/(40%/gel pourcentage %).
CAUTION : Les gels de polyacrylamide contiennent souvent de l'acrylamide non polymérisé qui est un matériau toxique qui peut produire un danger lorsqu'il est introduit dans l'environnement. Éliminer les gels de polyacrylamide dans le cadre du programme de déchets chimiques de l'institution. - Micro-ondes pour 15 s à 30% de puissance. Placer sur le discitor pendant 10 min à température ambiante ou jusqu'à ce que l'urée soit complètement dissoute.
- Pendant que le mélange de gel est en rotation, récupérez une cassette en gel (18 puits, 1 mm d'épaisseur; 13,3 x 8,7 cm (W x L)), retirez le peigne et placez la cassette pour la coulée de gel.
- Tournez rapidement le tube de 50 ml pendant 5 s pour recueillir la solution de gel au fond du tube. Ajouter 125 L de 10% de solution de persulfate d'ammonium (APS). Placer sur le discauor pendant 1 min. Tourner à nouveau pendant 5 s.
- Ajouter 25 l de téramethylènediamine (TEMED) et mélanger soigneusement en pipetting de haut en bas 5 fois avec une pipette de 25 ml en évitant les bulles.
- Pipette dans la fonte de gel et insérer soigneusement le peigne de gel en évitant les bulles.
- Resserrer le joint en ajoutant un grand clip de liant sur le dessus de la cassette.
- Laisser le gel polymériser pendant 1 h.
- Une fois le gel solidifié, retirez le clip de liant. Insérez la cassette dans la boîte d'électrophoresis. Ajouter 1x tampon TBE dans les réservoirs supérieurs et inférieurs.
- Récupérer la réaction de transcription. Ajouter de l'eau jusqu'à 100 l, puis ajouter 100 l de 2x Gel Loading Buffer. Préparer l'échelle en mélangeant la quantité recommandée avec de l'eau à 10 l et 10 L de 2x Gel Loading Buffer à un tube séparé de 1,5 ml.
- Incuber à la fois l'échelle et la réaction de transcription in vitro dans le thermomélangeur à 70 oC pendant 15 min.
- Pendant que les échantillons dénaturent à 70 oC, retirez soigneusement le peigne, nettoyez les puits en faisant monter et descendre chaque puits à l'eau avec une pipette P1000, et immédiatement pré-exécuter pendant 10 min à 100 V.
- Une fois les échantillons terminés, leur dénaturation de 15 min à 70 oC, retirez les tubes du thermomélangeur et placez-les immédiatement sur la glace.
- Nettoyez chaque puits du gel à nouveau avec la pipette P1000. Chargez 20 l l de l'échelle sur le premier puits à gauche, 20 l de l'échantillon d'ARN sur 10 puits distincts, et 20 l de tampon de chargement de gel 1x sur les puits inutilisés.
- Exécuter le gel à 100 V pendant 60 -240 min, selon le % de polyacrylamide du gel.
REMARQUE : Les colorants du Gel Loading Buffer se sépareront en deux bandes lorsqu'elles traversent le gel, une bande bleue de Bromophenol Blue (BB) qui migre lentement et une bande cyande de Xylène Cyanol (XC) qui migre rapidement. La migration approximative de ces bandes dans différents gels de polyacrylamide en % est bien connue (voir l'étape 1.11) et peut être utilisée pour estimer la migration de l'ARN dans le gel. - Une fois le gel arrêté, retirer soigneusement le gel de la cassette.
- Placer le gel dans une boîte propre contenant une solution de 50 ml de tampon 1x TBE avec 50 l de tache de gel d'acide nucléique et incuber pendant 5 min sur une berceuse pour tacher l'ARN.
- Prenez une photo avant et après l'excision de bande du gel sur le système d'imagerie de gel, de préférence en utilisant la lumière bleue au lieu de la transillumination UV.
- Accise chaque bande d'intérêt à l'aide d'une pointe de coupe de gel jetable sans nauséabonde. Après chaque puits, transférer la pointe contenant la tranche de gel dans un tube de 1,5 ml et faire tourner brièvement pour recueillir la tranche de gel. Répéter l'opération jusqu'à ce que toutes les bandes aient été recueillies dans le même tube de 1,5 ml.
- Une fois que toutes les tranches de gel ont été récoltées, ajouter 100 l d'eau sans nauséabonde ou tampon TE au tube de 1,5 ml. Conserver à 4 oC pour 48 h. Cela permet à l'ARN de sortir les tranches de gel dans la solution.
- Après 48 h, pipet l'eau ou tampon TE à un tube frais de 1,5 ml. Disposer des tranches de gel restantes. Purifie l'ARN par l'intermédiaire du kit de nettoyage comme suit.
- Équilibrez la colonne de spin à température ambiante pendant au moins 30 min.
- À la solution de 100 oL d'ARN dans le nouveau tube de 1,5 ml à partir de l'étape 1,32, ajouter 350 l de tampon RLT et bien mélanger pendant 2 min sur un écrou. Tourner pendant 1 s à 200 x g pour recueillir la solution au fond du tube.
- Ajouter 675 'L de 100% EtOH et bien mélanger pendant 2 min sur le diateur. Faites tourner pendant 1 s à 200 x g et passez immédiatement à l'étape suivante.
- Transférer 565 l de l'mélange sur la colonne de spin et tourner pendant 1 min à 15 000 x g. Videz le tube de collecte par aspiration.
- Répétez l'étape précédente avec la deuxième moitié de l'échantillon.
- Ajouter 500 l de tampon RPE à la colonne et tourner pendant 1 min à 15 000 x g. Videz le tube de collecte par aspiration.
- Ajouter 750 l'éthanol de 80 % à la colonne et tourner pendant 1 min à 15 000 x g. Videz le tube de collecte par aspiration.
- Placez la colonne dans un nouveau tube de collecte de 2 ml avec le couvercle ouvert et tournez à 15 000 x g pendant 5 min.
- Transférer la colonne dans un nouveau tube de 1,5 ml.
- Ajouter 17 l'eau au centre de la colonne et tourner à 15 000 x g pendant 1 min pour s'élituer. Elute à nouveau en utilisant un autre 17 L d'eau. Le volume total récupéré doit être de 32 l.
- Bien mélanger par vortex, pipet 1,5 l et mesurer la concentration de l'ARN à l'aide d'un spectrophotomètre à microvolume.
- Vérifiez la qualité de la purification de l'ARN par l'urée dénaturant le gel polyacrylamide comme dans les étapes 1.11-1.29.
2. L'ARN in vitro de méthyltransferase
- Mettre en place l'essai de méthyltransferase de 100 l'ARN dans un tube de 1,5 ml sur la glace comme suit : 23 l d'eau, 10 L de 10x TBS (500 mM Tris-HCl, pH 7,5; 1,5 M NaCl), 2 'L de 0,05 M EDTA, 5 'L de 100 mM TNtT , 40 l de 50 % de glycérol, 4 l de 58 M 3H-SAM, 5 l de 20x Protease Inhibitor Cocktail, 1 L de RNaseOUT (facultatif), 5 l d'ARN et 5 l de Méthyltransferase.
CAUTION : Les matières tritiated radioactives sont dangereuses et ne doivent être manipulées que lorsqu'elles portent des gants, une blouse de laboratoire et tout autre EPI nécessaire. Tous les bouts de pipette et les tubes en contact avec des matières radioactives sont considérés comme des déchets radioactifs solides. Éliminer tous les déchets radioactifs solides et liquides selon le protocole approuvé par le laboratoire sur les déchets radioactifs.
REMARQUE : Inclure des échantillons témoins sans méthyltransferase et sans ARN. Les concentrations de réactifs peuvent nécessiter une optimisation et/ou l'inclusion de sels de cation divalents, tels que mgCl2, ou SAM non étiqueté. La plage optimale de concentration finale d'ARN est de 50 nM à 1 M, tandis que la concentration de méthyltransferase est de 25 nM à 300 nM. - Bien mélanger en faisant un vortex en tourbillonnant doucement le tube. Spin 5 s à 200 x g pour recueillir la solution au fond du tube. Incuber le tube (s) à 37 oC pendant 2 h.
- Nettoyez la réaction à l'aide de la purification de la colonne comme à l'étape 1.32.
CAUTION: Soyez très prudent pour éliminer correctement les matières radioactives, en particulier pendant les lavages de colonne (pipette au lieu d'aspirater les déchets des tubes de collecte). - Effectuer le nombre de scintillation liquide
- Configurez le support de comptage de scintillation avec une fiole par échantillon, une fiole pour la mesure de fond et une fiole pour le test de balayage. Remplissez les flacons de 5 ml de solution de comptage de scintillation.
- Ajouter 10 l de chaque échantillon d'ARN radioactif élucidé en 1 flacon, serrer le couvercle et mélanger délicatement.
- Préparer les flacons de test de balayage. Frotter soigneusement les cotons-tiges sur toutes les surfaces et l'équipement utilisés pendant le protocole. Ajouter des écouvillons aux flacons remplis de 5 ml de solution de scintillation et serrer le couvercle.
- Exécuter les échantillons sur le compteur de scintillation comme suit. Ouvrez le capot du comptoir, insérez la grille dans la machine et fermez le capot. Sélectionnez Count Single Rack. Sélectionnez Select User Program. Sélectionnez ou créez un programmequi mesure le tritium (3 H) pour 60 s. Hit Count Rack. Répétez le nombre de scintillation trois fois.
REMARQUE : L'équipement mesurera le nombre de scintillation de chaque échantillon et de la sortie à la fois à l'écran et à une impression. Protocole peut être mis en pause ici si désiré. Les échantillons d'ARN restants de l'étape 2.3 doivent être congelés à -80 oC pour une utilisation ultérieure.
- Procéder avec l'autoradiogramme
- Préparer et pré-exécuter l'urée dénaturant gel polyacrylamide comme dans les étapes 1.11-1.20.
- Pipette 20 l de matière d'ARN radioactif dans un nouveau tube de 1,5 ml contenant 20 l de tampon de chargement de gel 2x. Bien mélanger. Préparer l'échelle comme dans l'étape 1.21. Incuber les échantillons à 70 oC pendant 15 min.
- Laver les puits du gel une fois de plus immédiatement avant le chargement de l'échantillon. Chargez 20 l l de l'échelle préparée, 20 'L des échantillons, et 20 'L de 1x Gel Loading Buffer sur les voies restantes. Exécuter le gel à 100 V pendant 60-180 min, selon le pourcentage de polyacrylamide.
- Une fois que le gel finit de fonctionner, retirez le gel de la cassette et placez-le dans une boîte contenant 50 ml de tampon 1x TBE avec 5 L de tache de gel d'acide nucléique ultrasensible.
- Incuber pendant 5 min sur le rocker pour tacher l'ARN.
- Sortez soigneusement le gel de la boîte et placez-le sur le transilluminateur UV du système d'imagerie gel avec les puits vers le haut et l'échelle sur la gauche.
- Concentrez la caméra sur le gel, allumez la lumière UV, puis prenez une image du gel en exposant de 50 ms à 1 s selon l'intensité du signal.
- Éteignez l'exposition aux UV et enregistrez l'image sous forme de fichier Tiff.
- Remettre le gel dans la boîte. Supprimer TBE. Fixer le gel avec 50 ml de solution de fixation (50% de méthanol, 10% d'acide acétique, 40% d'eau ultra-pure) pendant 30 min à température ambiante sur un rocker.
- Déplacez doucement le gel à nouveau dans une boîte noire fraîche contenant 25 ml de la solution d'amélioration de l'autoradiographie. En l'absence d'une boîte noire, recouvrir la boîte de papier d'aluminium pour protéger la solution de la lumière.
- Incuber 30 min à température ambiante sur le rocker.
- Soulevez délicatement le gel et placez-le face cachée sur une feuille de pellicule plastique avec les puits vers le haut et l'échelle sur le côté droit. Déposer deux feuilles de papier chromatographique sur le dos du gel. Retournez doucement toute la pile.
- Préchauffer le séchoir à gel à 80 oC. Reculez le couvercle en plastique sur le séchoir à gel. Insérez l'enveloppe, le gel et la pile de papier chromatographique sous le couvercle en plastique et déplacez le couvercle en plastique vers le bas pour créer un joint.
- Sécher pendant 1 h à 80 oC dans le séchoir à gel.
- Éteignez le séchoir à gel et retirez délicatement la pile. Retirer l'enveloppe et le deuxième papier chromatographique. Tapez le reste du papier chromatographique avec le gel séché dans une cassette autoradiogramme.
- Ajouter 1 feuille de film d'autoradiographie dans la pièce sombre.
- Placez la cassette à -80 oC et développez le film après 1 h à 4 semaines en fonction de l'intensité du signal, qui peut être jugé en fonction des comptes de scintillation précédemment mesurés : 1-4 semaines pour 250-1 000 cpm, 24 h à 1 semaine pour 1 000 à 10 000 cpm et 1 h à 24 h pour le cp de l'année. m.
- Une fois le film développé, placez le film sur le dessus de la cassette et marquez soigneusement avec un marqueur de laboratoire les 4 bords du gel, chacun puits, et la position des colorants XC et BB.
- Scannez le film à 300 ou 600 pixels par pouce de résolution et enregistrez l'image comme fichier Tiff.
Résultats
Réaction de transcription in vitro
Figure 2 A représente une course typique d'une réaction de transcription in vitro avec la polymérase d'ARN T7 du snRNA 7SK, qui est un ARN relativement court (331 nt) et très structuré. Comme le montre cette image brute, il existe plusieurs bandes indésirables, à la fois plus courtes et plus longues que 7SK, résultant probablement d'événements aléatoires d'initiation transcriptionnelle ou de terminaison. Pour cette raison, la purification du gel après la réaction de transcription in vitro est importante pour obtenir un échantillon d'ARN propre comme le montre la figure 2B. À ce stade, l'ARN d'intérêt peut être identifié par son emplacement par rapport à l'échelle et purifié par la purification de gel.
Comme mentionné précédemment, il peut être nécessaire d'optimiser la longueur de la réaction de transcription avant l'étape de purification du gel. Les réactions de transcription qui duissent trop longtemps peuvent entraîner des quantités extrêmement élevées de production d'ARN, ce qui rend difficile l'identification en aval de la transcription correcte. Il est également important de vérifier l'identité de la transcription purifiée par la transcription inversée et le PCR quantitatif (RTqPCR) à l'aide d'amorces spécifiques.
L'ARN in vitro de méthyltransferase d'assay
La figure 3 montre un résultat représentatif d'un résultat de méthyltransferase d'ARN décrit dans le protocole utilisant la limite inférieure de nos concentrations recommandées d'ARN et de protéine. Cet essai permet à la fois des résultats quantitatifs des dénombrements de scintillation, ainsi que des résultats qualitatifs de l'autoradiogramme. Ici, MePCE, un méthyltransferase d'ARN connu pour méthylate 7SK10,s'est avéré capable de methylate également U6. En outre, comme récemment montré pour 7SK16, la liaison de l'histone H4 à MePCE inhibe également la méthylation U6. Nous avons pu l'observer à la fois dans le nombre de scintillation (figure 3C) et dans l'autoradiogramme (figure 3B),montrant la robustesse de ce protocole.
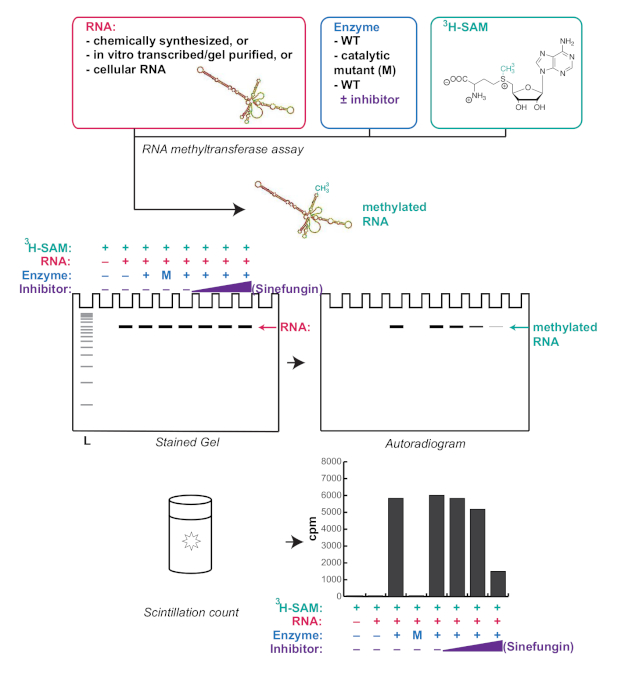
Figure 1 . Représentation schématique d'un flux de travail typique d'analyse de méthyltransferase d'ARN et des résultats attendus. Sinefungin est un inhibiteur de méthyltransferase compétitif. L: échelle; WT: type sauvage; M: mutant catalytique; cpm: compte par min. S'il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
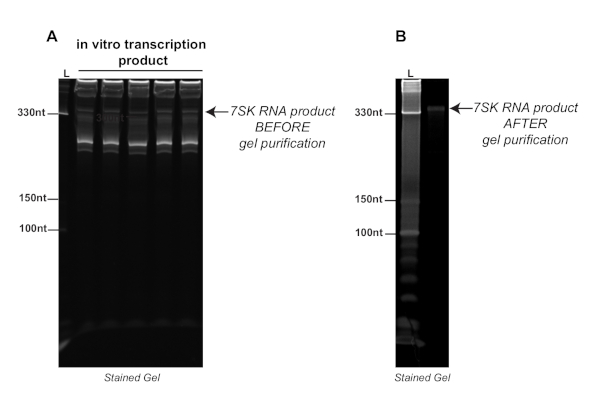
Figure 2 . Expérience représentative montrant le produit de la transcription in vitro avant (A) et après (B) la purification sur un gel dénaturant d'urée-polyacrylamide de 8% souillé avec la tache d'acide nucléique. Les flèches pointent vers la transcription 7SK avant et après la purification du gel. L: échelle. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 . L'ARN in vitro méthyl-transferase d'assay effectué avec MePCE contre U6. L'assay de méthyltransferase in vitro utilisant la TPS-MePCE recombinante (25 nM), 3SAM H-radioactive comme donneur de groupe méthyle et in vitro transcrit l'ARN U6 (50 nM) comme substrat. L'autoradiogramme a été exposé pendant 2 semaines afin de détecter l'activité résiduelle (250 cpm) dans l'échantillon de L'Histone H4. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Discussion
Ici, une méthode simple et robuste pour la vérification in vitro de l'activité de méthyltransferase d'ARN vers des transcriptions spécifiques est rapportée. L'analyse profite du fait que la méthionine de S-adenosyl peut être tritiaté sur le donneur de groupe méthyle (figure 1), permettant que l'ARN méthylé soit détecté avec précision sans l'utilisation d'anticorps. Cependant, il est important de noter que cet essai ne peut pas indiquer quel résidu ou groupe chimique est méthylé par l'enzyme. Pour identifier ou vérifier qu'un résidu spécifique est méthylé, d'autres méthodes telles que l'analyse mutationnelle, le bloc de transcription inversé ou l'analyse de spectrométrie de masse du substrat d'ARN peuvent être employées en conjonction avec l'essai de méthyltransferase d'ARN.
Le choix des ARN et leur mode de synthèse dépend de la taille de l'ARN. Les ARN spécifiques de tailles comprises entre 18 et 120 nt peuvent être commodément synthétisés sur mesure par de nombreux fournisseurs réputés d'acide nucléique. Cependant, le plus souvent, des ARN spécifiques de différentes tailles sont transcrits in vitro. Les modèles d'ADN pour la transcription in vitro peuvent être générés de diverses manières, et exigent que la séquence d'intérêt soit située en aval d'un promoteur T7 ou SP6. Lorsque les extrémités précises des ARN sont importantes, le plasmide pRZ est recommandé17. La sensibilité élevée de l'analyse (Figure 3) permet également de remplacer un ARN purifié par un mélange d'ARN cellulaire, soit total ou messager ARN par exemple. En effet, le gel et l'autoradiogramme fournissent une méthode pour observer directement la taille de l'ARN (s) ciblé pour la méthylation.
Les conditions signalées ici à l'étape 2.1 du protocole sont optimales pour la famille BIN3 de méthyltransferases, qui a deux homologs chez l'homme, MePCE10 et BCDIN3D9. Il est important de souligner que les conditions d'assay doivent être ajustées à la protéine et à l'ARN spécifiques d'intérêt. Par exemple, il a été démontré que la présence de MgCl2 diminue l'activité BCDIN3D18, cependant, MgCl2 peut être un coordonnateur important de la structure de l'ARN, et pourrait donc constituer une composante importante de l'analyse pour d'autres ARN Methyltransferases.
L'avantage d'utiliser un autoradiogramme, qui peut être exposé pendant une longue période de temps, est qu'il peut permettre de détecter une activité très faible qui n'est pas détectée dans l'analyse de scintillation. Habituellement, cela indique que la protéine est une méthyltransferase, mais qu'une ou plusieurs des conditions de réaction ou des réactifs doivent être optimisés: enzyme; substrat, cofactor, conditions tampon, etc.9. Par exemple, la purification enzymatique peut devoir être améliorée pour enlever ou modifier la position de l'étiquette. Il se peut également que l'ARN utilisé comme substrat doit être plié correctement ou d'interagir avec un autre facteur de protéines ou d'ARN pour être méthylé par l'enzyme. Ainsi, la littérature préexistante et/ou les propres résultats dans les cellules doivent être soigneusement examinés pour établir les conditions d'essai pour l'enzyme et l'ARN d'intérêt.
L'assay est également extrêmement flexible. Par exemple, il peut être une étape précurseur à un autre type d'essai, de sorte que l'ARN méthylé radioactif est utilisé dans les essais quantitatifs et qualitatifs de méthylase11, ou dans les essais de liaison d'ARN permettant de visualiser spécifiquement le comportement de la l'ARN méthylé comparé à l'ARN non modifié. L'essai peut également être légèrement ajusté pour analyser la modification des ARN qui utilisent des coenzymes qui peuvent être étiquetées radioactivement, telles que la coenzyme A d'acétyle pour l'étude de l'acétylation d'ARN19,20.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Les auteurs tient à remercier le Dr Turja Kanti Debnath pour son aide à ChemDraw. La recherche dans le laboratoire xhemalçe est soutenue par le ministère de la Défense - Congressionally Directed Medical Research Program - Breast Cancer Breakthrough Award (W81XWH-16-1-0352), NIH Grant R01 GM127802 et des fonds de démarrage de l'Institut de cellulaire et Molecular Biology et le College of Natural Sciences de l'Université du Texas à Austin, États-Unis.
matériels
| Name | Company | Catalog Number | Comments |
| 10 bp DNA Ladder | Invitrogen | 10821-015 | 10 bp DNA Ladder kit. |
| 10% Ammonium Persulfate (APS) | N/A | N/A | For urea denaturing polyacrylamide gel (For 10 mL, dissolve 1g in 8 mL of milliQ water; adjust volume to 10 mL with milliQ water; filter the solution using a 10 mL syringe equiped with a 0.45 µm filter). |
| 10X TBE Buffer | N/A | N/A | For urea denaturing polyacrylamide gel (For 1L, add 108 g of Tris Base, 55 g of Boric Acid to a cylinder with a stir bar; add 800 mL of distilled water and let dissolve; add 40mL of 0.5 M Na2EDTA (pH 8.0); adjust volume to 1L with milliQ water; filter the solution using a 0.22µm filter). |
| 10X TBS | N/A | N/A | For 1L, add 60.5 g of Tris Base, 87.6 g of NaCl to a cylinder with a stir bar; add 800 mL of distilled water and let dissolve; adjust pH to 7.5 with concentrated HCl; adjust volume to 1L with milliQ water; filter the solution using a 0.22 µm filter. |
| Acrylamide: Bis-Acrylamide 29:1 (40% Solution/Electrophoresis), Fisher BioReagents | Fisher | BP1408-1 | For urea denaturing polyacrylamide gel. |
| ADENOSYL-L-METHIONINE, S-[METHYL-3H]; (SAM[3H]) | Perkin Elmer | NET155V250UC | For in vitro methylation of RNA; Concentration = 1.0 mCi/mL; Specific activity = 17.1 Ci/mmol; Molarity= (1.0 Ci/L)/(83.2 Ci/mmol) = 0.0584 mmol/L = 58.4 µM. Upon receipt of the frozen 3H-SAM tube, thaw it at 4°C, make 20 µL aliquots, and freeze them at -30°C. Never refreeze and reuse a partially used aliquot. |
| Amersham Hypercassette Autoradiography Cassettes | GE Healthcare | RPN11649 | For autoradiogram gel exposure. |
| Amersham Hyperfilm MP | GE Healthcare | 28906846 | For autoradiogram gel exposure. |
| Beckman Scintillation Counter | Beckman | LS6500 | For liquid scintillation count. |
| Biorad Mini Horizontal Electrophoresis System | Biorad | 1704466 | Mini Horizontal Electrophoresis System. |
| cOmplete Mini EDTA-free Protease Inhibitor Cocktail Tablets | Roche Applied Science | 4693159001 | For a 20X solution, dissolve 1 tablet in 0.525 mL of nuclease free water. |
| Criterion Cell | Biorad | 345-9902 | RNase free empty cassette for polyacrylamide gel. |
| Criterion empty Cassettes | Biorad | 1656001 | Vertical midi-format electrophoresis cell. |
| DeNovix DS-11 Microvolume Spectrophotometer | DeNovix | DS-11-S | Microvolume Spectrophotometer for measuring DNA and RNA concentration. |
| Ecoscint Original | National Diagnostics | LS-271 | For liquid scintillation count. |
| Fisherbrand 7mL HDPE Scintillation Vials | Fisher | 03-337-1 | For liquid scintillation count. |
| Fluoro-Hance-Quick Acting Autoradiography Enhancer | RPI CORP | 112600 | For autoradiogram gel pretreatment. |
| Gel dryer | Biorad | 1651745 | For drying gel. |
| Gel Loading Buffer II | Ambion | AM8547 | For loading RNA in denaturing polyacrylamide urea gel (composition: 95% Formamide, 18 mM EDTA, and 0.025% SDS, Xylene Cyanol, and Bromophenol Blue). |
| GeneCatcher disposable gel excision tips | Gel Company | NC9431993 | For removing bands from agarose and polyacrylamide gels. |
| Megascript Kit | Ambion | AM1333 | For in vitro transcription with T7 RNA polymerase. |
| Perfectwestern Extralarge Container | Genhunter Corporation | NC9226382 (clear)/ NC9965364 (black) | Gel staining box. |
| pRZ | Addgene | #27663 | Plasmid for producing in vitro transcripts with homogeneous ends |
| Qiagen RNeasy MinElute Cleanup | Qiagen | 74204 | For RNA clean-up, use modified protocol provided in the protocol. |
| QIAquick Gel Extraction Kit (50) | Qiagen | 28704 | Kit for gel extraction and clean up of dsDNA fragment used for in vitro transcription. |
| Saran Premium Plastic Wrap | Saran Wrap | Amazon | For drying gel. |
| SYBR Gold | Invitrogen | S11494 | Ultra sensitive nucleic acid gel stain. |
| SYBR Safe | Invitrogen | S33102 | Nucleic acid gel stain. |
| TE | Sigma | 93283-100ML | 10 mM Tris-HCl, 1 mM disodium EDTA, pH 8.0 |
| TEMED | Fisher | 110-18-9 | For urea denaturing polyacrylamide gel. |
| Thermomixer with SMARTBLOCK 24X 1.5mL TUBES | eppendorf | 5382000023/5361000038 | For temperature controlled incubation of 1.5 mL tubes. |
| TOPO TA Cloning Kit | life technologies | Kits for fast cloning of Taq polymerase–amplified PCR products into vectors containing T7 and/or SP6 promoters for in vitro RNA transcription. | |
| TURBO DNase (2 U⁄µL) | Ambion | AM2238 | For DNA removal from in vitro transcription reactions. |
| Urea | Sigma | 51456-500G | For urea denaturing polyacrylamide gel. |
| Whatman 3MM paper | GE Healthcare | 3030-154 | Chromatography paper for drying gel. |
Références
- Xhemalce, B. From histones to RNA: role of methylation in cancer. Briefings in Functional Genomics. 12 (3), 244-253 (2013).
- Shelton, S. B., Reinsborough, C., Xhemalce, B. Who Watches the Watchmen: Roles of RNA Modifications in the RNA Interference Pathway. PLoS Genetics. 12 (7), 1006139 (2016).
- Mauer, J., et al. Reversible methylation of m(6)Am in the 5' cap controls mRNA stability. Nature. 541 (7637), 371-375 (2017).
- Wang, X., et al. N6-methyladenosine-dependent regulation of messenger RNA stability. Nature. 505 (7481), 117-120 (2014).
- Pendleton, K. E., et al. The U6 snRNA m(6)A Methyltransferase METTL16 Regulates SAM Synthetase Intron Retention. Cell. 169 (5), 824-835 (2017).
- Meyer, K. D., et al. 5' UTR m(6)A Promotes Cap-Independent Translation. Cell. 163 (4), 999-1010 (2015).
- Wang, X., et al. N(6)-methyladenosine Modulates Messenger RNA Translation Efficiency. Cell. 161 (6), 1388-1399 (2015).
- Alarcon, C. R., Lee, H., Goodarzi, H., Halberg, N., Tavazoie, S. F. N6-methyladenosine marks primary microRNAs for processing. Nature. 519 (7544), 482-485 (2015).
- Xhemalce, B., Robson, S. C., Kouzarides, T. Human RNA methyltransferase BCDIN3D regulates microRNA processing. Cell. 151 (2), 278-288 (2012).
- Jeronimo, C., et al. Systematic analysis of the protein interaction network for the human transcription machinery reveals the identity of the 7SK capping enzyme. Molecular Cell. 27 (2), 262-274 (2007).
- Liu, W., et al. Brd4 and JMJD6-associated anti-pause enhancers in regulation of transcriptional pause release. Cell. 155 (7), 1581-1595 (2013).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. Journal of Visual Experimentation. , (2019).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. Journal of Visual Experimentation. (63), e3998 (2012).
- Rong, M., Durbin, R. K., McAllister, W. T. Template strand switching by T7 RNA polymerase. Journal of Biological Chemistry. 273 (17), 10253-10260 (1998).
- Rio, D. C. Expression and purification of active recombinant T7 RNA polymerase from E. coli. Cold Spring Harb Protocols. 2013 (11), (2013).
- Shelton, S. B., et al. Crosstalk between the RNA Methylation and Histone-Binding Activities of MePCE Regulates P-TEFb Activation on Chromatin. Cell Reports. 22 (6), 1374-1383 (2018).
- Walker, S. C., Avis, J. M., Conn, G. L. General plasmids for producing RNA in vitro transcripts with homogeneous ends. Nucleic Acids Research. 31 (15), 82 (2003).
- Blazer, L. L., et al. A Suite of Biochemical Assays for Screening RNA Methyltransferase BCDIN3D. SLAS Discovery. 22 (1), 32-39 (2017).
- Arango, D., et al. Acetylation of Cytidine in mRNA Promotes Translation Efficiency. Cell. 175 (7), 1872-1886 (2018).
- Ito, S., et al. Human NAT10 is an ATP-dependent RNA acetyltransferase responsible for N4-acetylcytidine formation in 18 S ribosomal RNA (rRNA). Journal of Biological Chemistry. 289 (52), 35724-35730 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon