Method Article
Analisi anticorp-free per l'analisi dell'attività di RNA methyltransferase
In questo articolo
Riepilogo
Qui, viene descritto un saggio in vitro privo di anticorpi per l'analisi diretta dell'attività di metiltransferasi sull'RNA transscritto sintetico o in vitro.
Abstract
Ci sono più di 100 modifiche chimicamente distinte dell'RNA, due terzi delle quali consistono di metilazioni. L'interesse per le modifiche dell'RNA, e in particolare le metilazioni, è riemerso a causa dei ruoli importanti svolti dagli enzimi che li scrivono e li cancellano nei processi biologici rilevanti per la malattia e il cancro. Qui, viene fornito un saggio in vitro sensibile per un'analisi accurata dell'attività degli autori di metilazione dell'RNA sugli RNA trascritti sintetici o in vitro. Questo saggio utilizza una forma tritiata di S-adenosyl-methionine, con conseguente etichettatura diretta dell'RNA metilato con trizio. La bassa energia della radiazione di trizio rende il metodo sicuro, e i metodi preesistenti di amplificazione del segnale di trizio, consentono di quantificare e visualizzare l'RNA metilato senza l'uso di anticorpi, che sono comunemente inclini a manufatti. Mentre questo metodo è scritto per la metilazione dell'RNA, poche modifiche lo rendono applicabile allo studio di altre modifiche dell'RNA che possono essere etichettate radioattivamente, come l'acetilazione dell'RNA con coenzima acetil 14C A. Nel complesso, questo saggio consente di valutare rapidamente l'RNA condizioni di metilazione, inibizione con piccoli inibitori molecolari, o l'effetto di mutanti di RNA o enzimi, e fornisce un potente strumento per convalidare ed espandere i risultati ottenuti nelle cellule.
Introduzione
Il DNA, l'RNA e le proteine sono soggetti a modifiche che regolano strettamente l'espressione genica1. Tra queste modifiche, le metilazioni si verificano su tutti e tre i biopolimeri. Le metilazioni del DNA e delle proteine sono state studiate molto bene negli ultimi tre decenni. Al contrario, l'interesse per la metilazione dell'RNA è stato riacceso solo di recente alla luce dei ruoli importanti che le proteine che scrivono, cancellano o legano le metilazioni dell'RNA giocano nello sviluppo e nella malattia2. Oltre alle funzioni più conosciute negli ABbondanti RNA ribosomici e di trasferimento, le vie di metilazione dell'RNA regolano la stabilità specifica dell'RNA messaggero3,4, splicing5 e traduzione6,7, Elaborazione miRNA8,9 e pausa trascrizionale e rilascio10,11.
Qui, viene riportato un metodo semplice e robusto per la verifica in vitro dell'attività di metiltransferasi dell'RNA nell'impostazione di un laboratorio di biologia molecolare (riassunto nella Figura 1). Molti studi valutano l'attività di un RNA metiltransferase attraverso dot-blot con un anticorpo contro la modificazione dell'RNA di interesse. Tuttavia, dot-blot non verifica l'integrità dell'RNA in caso di incubazione con l'RNA metiltransferase. Questo è importante perché anche piccole contaminazioni di proteine ricombinanti con nucleasi possono portare a parziale degradazione dell'RNA e risultati confusi. Inoltre, anche gli anticorpi altamente specifici per la modificazione dell'RNA possono riconoscere gli RNA non modificati con sequenze o strutture specifiche. Il saggio di metiltransferasi in vitro di RNA qui riportato sfrutta il fatto che la methionina S-adenosyl può essere tritiata sul donatore di gruppo metilico (Figura 1), consentendo l'rilevazione accurata dell'RNA metilato senza l'uso di anticorpi . Vengono fornite istruzioni per la trascrizione in vitro e la purificazione di una trascrizione di interesse e di test della metilazione di tale trascrizione da parte dell'enzima di interesse. Questo metodo è flessibile e robusto e può essere regolato in base alle esigenze di un determinato progetto. Ad esempio, possono essere utilizzati RNA in vitro trascritti e purificati, RNA sintetizzati chimicamente, ma anche RNA cellulari. Questo saggio fornisce informazioni quantitative sotto forma di conteggi scintilliali, così come informazioni qualitative mostrando dove esattamente l'RNA metilato viene eseguito su un gel. Questo può fornire una visione unica della funzione di una metiltransferasi dell'RNA, in particolare quando si utilizzano RNA cellulari come substrato, in quanto fornisce un metodo per osservare direttamente le dimensioni dell'RNA o degli RNA che sono mirati alla metilazione.
Protocollo
1. Trascrizione in vitro e purificazione del gel dell'RNA bersaglio
- Clonare la sequenza di interesse in plasmidi contenenti promotori T7 e/o SP6 utilizzando tecniche di clonazione molecolare stabilite12 o kit.
- Linearizzazione del modello di DNA per la trascrizione in vitro
- Amplificare la sequenza di interesse da parte della PCR del plasmide con primer progettati per includere la regione Promotore T7 attraverso l'inserto, 20-30 bp a monte e a valle come descritto in precedenza13.
- In alternativa, digerire 5 g del plasmico utilizzando un enzima di restrizione (RE) sito di taglio disponibile a valle dell'inserto secondo le istruzioni fornite dal fornitore della RE.
NOTA: Ricordarsi di verificare che l'inserto non sia tagliato dal RE selezionato. La selezione del RE utilizzato in questo passaggio può influire notevolmente sulla resa della trascrizione. Preferibilmente, linearizzare con un'estremità smussata o 5'-overhanging producendo RE per evitare la commutazione del modello della polimerasi dell'RNA T7 sul filamento di DNA opposto14. Provare RE diverso se i rendimenti iniziali sono insufficienti.
- Purificare il DNA risultante utilizzando il kit per l'estrazione e la pulizia del gel di agarose del DNA. Elutare il DNA con 30 litri d'acqua.
- Mescolare bene vorticendo, pipet 1,5 l e misurare la concentrazione del DNA con uno spettrofotometro a microvolume.
- Utilizzare 250 ng di DNA per verificare la qualità e le dimensioni del DNA purificato su un 1% di elettroforesi gel di agarose in 1t TBE buffer migrato per 1 h a 4 V/cm.
- Scongelare i reagenti congelati del kit di trascrizione in vitro. Posizionare il Mix di polimerasi T7 RNA sul ghiaccio e gli altri reagenti su un nutatore a temperatura ambiente. Subito dopo lo scongelamento completo, girare velocemente i tubi rNTP per 5 s per raccogliere la soluzione sul fondo dei tubi e posizionare sul ghiaccio. Mantenere il buffer di reazione 10x a temperatura ambiente per evitare precipitazioni.
- Convogliare in un tubo da 1,5 mL i seguenti componenti della reazione di trascrizione di 20 .L a temperatura ambiente nell'ordine indicato: 6 L di acqua, 2 l (0,1 - 1 g) di modello di DNA lineare, 2 L di 75 mM soluzione ATP, 2 . , 2 - L di soluzione CTP 75 mM, 2 -L di soluzione UTP da 75 mM, 2 -L di 10x Reaction Buffer e 2 -L di T7 RNA Polymerase Mix.
NOTA: Per il modello di DNA generato dalla PCR, utilizzare 100 - 200 ng DNA; per il modello di DNA generato dalla digestione enzimatica di restrizione di un plasmide, utilizzare il ricombinante attivo da 1 opprimente Il suo polimerano t7 RNAa 6tag Può essere purificato in E. coli15. - Mescolare accuratamente il tubo. Rotazione rapida per 5 s per raccogliere la soluzione di reazione nella parte inferiore del tubo, quindi incubare a 37 gradi centigradi per 2-4 h.
NOTA: ogni trascrizione si comporterà in modo diverso a seconda del modello di DNA, della sua lunghezza, della sua sequenza o struttura. Ottimizzare le condizioni di trascrizione in vitro testando tempi di incubazione diversi fino a 6 h; diverse concentrazioni di modello di DNA, T7 RNA Polymerase, o NTP; o l'aggiunta di MgCl2 supplementare a ciò che è presente nel Mix di polimerasi t7 RNA. - Alla fine del periodo di incubazione, aggiungere 1 L di DNase I per una reazione a 20 gradi e incubare a 37 gradi centigradi per 15 min.
NOTA: La reazione di trascrizione può essere temporaneamente mantenuta sul ghiaccio fino a quando il gel di poliacrilammide non è pronto. - Procedere con la purificazione con gel poliacrilammide.
NOTA: poiché l'RNA è sensibile alla contaminazione da pH e RNase, utilizzare apparecchiature dedicate all'RNA e reagenti privi di RNase. - Determinare la percentuale di poliacrilammide per il gel a seconda delle dimensioni della trascrizione di interesse: 3,5% per 100-2000 nt (XC: 460 nt ; BB: 100 nt), 5% per 80-500 nt (XC: 260 nt ; BB: 65 nt), 8% per 60-400 nt (XC: 160 nt ; BB: 45 nt), 12% per 40-200 nt (XC: 70 nt ; BB: 20 nt), 15% per 25-150 nt (XC: 60 nt ; BB: 15 nt) e 20% per 6-100 nt (XC: 45 nt ; BB: 12 nt).
- Preparare il gel di poliacrilamide di demilazione dell'urea combinando i seguenti reagenti in un tubo conico da 50 mL: 9,6 g di urea di grado molecolare, 2 mL di 10x TBE, x mL del 40% di acrilammide 40%:bis-acrylamide mix (29:1) e acqua fino al marchio 20 mL sul tubo , dove x-20 mL/(40%/percentuale di gel%).
AVVISO: i gel di poliacrilammide spesso contengono acrilammide non polimerizzato, che è un materiale tossico che può produrre un pericolo quando introdotto nell'ambiente. Smaltire i gel di poliacrilammide attraverso il programma di rifiuti chimici dell'istituzione. - Microonde per 15 s al 30% di potenza. Mettere sul nutatore per 10 min a temperatura ambiente o fino a quando l'urea si è completamente sciolta.
- Mentre la miscela di gel sta ruotando, recuperare una cassetta di gel (18-bene, 1 mm di spessore; 13.3 x 8.7 cm (W x L)), rimuovere il pettine e posizionare la cassetta per la colata gel.
- Rotazione rapida del tubo da 50 mL per 5 s per raccogliere la soluzione gel nella parte inferiore del tubo. Aggiungere 125 L del 10% di soluzione persolfati di ammonio (APS). Posizionare sul nodo per 1 min.
- Aggiungere 25 -L di tetrametiletilenediamina (TEMED) e mescolare accuratamente pipetting su e giù 5 volte con una pipetta da 25 mL evitando le bolle.
- Pipette nel colata gel e inserire con attenzione il pettine gel evitando bolle.
- Stringere il sigillo aggiungendo una grande clip legante sopra la parte superiore della cassetta.
- Lasciare che il gel polimerizzi per 1 h.
- Una volta che il gel si è solidificato, rimuovere la clip legante. Inserire la cassetta nella scatola dell'elettroforesi. Aggiungere 1x tampone TBE nei serbatoi superiore e inferiore.
- Recuperare la reazione di trascrizione. Aggiungere acqua fino a 100 l, quindi aggiungere 100 l una di 2x Gel Loading Buffer. Preparare la scala mescolando la quantità raccomandata con acqua a 10 l e 10 -L di 2x Gel Loading Buffer in un tubo separato da 1,5 mL.
- Incubare sia la scala che la reazione di trascrizione in vitro nel termomixer a 70 gradi centigradi per 15 min.
- Mentre i campioni si denano a 70 gradi centigradi, rimuovere con cura il pettine, pulire i pozzetti pipettando su e giù ogni pozzo con una pipetta P1000, e immediatamente pre-esecuzione per 10 min a 100 V.
- Una volta che i campioni hanno completato la loro denaturazione di 15 min a 70 gradi centigradi, rimuovere i tubi dal termomixer e posizionare immediatamente sul ghiaccio.
- Pulire nuovamente ogni pozzetto del gel con la pipetta P1000. Caricare 20 l della scala sul primo pozzo a sinistra, 20 l del campione di RNA su 10 pozzi separati e 20 l of 1x Gel Loading Buffer sui pozze non utilizzati.
- Eseguire il gel a 100 V per 60 -240 min, a seconda della % poliacrilammide del gel.
NOTA: i coloranti nel Buffer di carico Gel si separeranno in due bande mentre attraversano il gel, una banda blu Bromophenol Blue (BB) lenta che migra e una banda cianica Xylene Cyanol (XC) che migra velocemente. La migrazione approssimativa di queste bande in diversi gel di poliacrilammide %è ben nota (vedi fase 1.11) e può essere utilizzata per stimare la migrazione dell'RNA nel gel. - Una volta che il gel è stato fermato, rimuovere con attenzione il gel dalla cassetta.
- Mettere il gel in una scatola pulita contenente una soluzione di 50 mL di 1x tampone TBE con 50 -L di macchia di gel acido nucleico e incubare per 5 min su un rocker per macchiare l'RNA.
- Scattare un'immagine prima e dopo l'escissione della banda del gel sul sistema di imaging gel, preferibilmente utilizzando la luce blu invece della traslazione UV.
- Ascisto ogni banda di interesse utilizzando una punta di taglio gel usa e getta senza nuclesino. Dopo ogni pozzo, trasferire la punta contenente la fetta di gel in un tubo da 1,5 mL e ruotare brevemente per raccogliere la fetta di gel. Ripetere fino a quando tutte le bande sono state raccolte nello stesso tubo da 1,5 mL.
- Una volta raccolte tutte le fette di gel, aggiungere 100 l of nuclease-free water o TE buffer al tubo da 1,5 mL. Conservare a 4 gradi centigradi per 48 ore. Ciò consente all'RNA di uscire dalle fette di gel nella soluzione.
- Dopo 48 h, convogliare l'acqua o il tampone DI TE in un tubo fresco da 1,5 mL. Smaltire le fette di gel rimanenti. Purificare l'RNA tramite kit di pulizia come segue.
- Equilibrate la colonna di spin a temperatura ambiente per almeno 30 min.
- Per la soluzione di 100 L di RNA nel nuovo tubo da 1,5 mL del passaggio 1,32, aggiungere 350 L di buffer RLT e mescolare bene per 2 min su un nutator. Girare per 1 s a 200 x g per raccogliere la soluzione nella parte inferiore del tubo.
- Aggiungere 675 luna del 100% EtOH e mescolare bene per 2 min sul nutatore. Girare per 1 s a 200 x g e procedere immediatamente al passaggio successivo.
- Trasferire 565 l dell'impasto sulla colonna di spin e girare per 1 min a 15.000 x g. Svuotare il tubo di raccolta per aspirazione.
- Ripetere il passaggio precedente con la seconda metà del campione.
- Aggiungere 500 l di buffer RPE alla colonna e ruotare per 1 min a 15.000 x g. Svuotare il tubo di raccolta per aspirazione.
- Aggiungere 750 l dell'80% di etanolo alla colonna e ruotare per 1 min a 15.000 x g. Svuotare il tubo di raccolta per aspirazione.
- Posizionare la colonna in un nuovo tubo di raccolta da 2 mL con il coperchio aperto e ruotare a 15.000 x g per 5 min.
- Trasferire la colonna in un nuovo tubo da 1,5 mL.
- Aggiungere 17 l'acqua al centro della colonna e ruotare a 15.000 x g per 1 min per eluire. Elutare di nuovo con altri 17 - L di acqua. Il volume totale recuperato deve essere di 32.
- Mescolare bene vorticendo, pipet 1,5 l e misurare la concentrazione dell'RNA utilizzando uno spettrofotometro a microvolume.
- Controllare la qualità della purificazione dell'RNA mediante gel di poliacrilammide ad urea denatura come nei passaggi 1.11-1.29.
2. Analisi di metiltransferasi in RNA in vitro
- Impostare il saggio di 100 ml di metiltransferase in un tubo da 1,5 mL sul ghiaccio come segue: 23 -L di acqua, 10 -L di 10x TBS (500 mM Tris-HCL, pH 7,5; 1,5 M NaCl), 2 : L di 0,05 M EDTA, 5 . , 40 -L del 50% glicerolo, 4 - L di 58 M 3H-SAM, 5 -L di 20x Protease Inhibitor Cocktail, 1 -L di RNaseOUT (opzionale), 5 l di RNA e 5 -L di Methyltransferase.
AVVISO: il materiale tritiato radioattivo è pericoloso e deve essere maneggiato solo indossando guanti, un camice da laboratorio e qualsiasi altro PPE necessario. Tutte le punte e i tubi delle pipette a contatto con il materiale radioattivo sono considerati scorie radioattive solide. Smaltire tutte le scorie radioattive solide e liquide secondo il protocollo di scorie radioattive approvato dal laboratorio.
NOTA: Includere campioni di controllo senza metiltransferasi e senza l'RNA. Le concentrazioni di reagente possono richiedere l'ottimizzazione e/o l'inclusione di sali di cation divalenti, come MgCl2 o SAM senza etichetta. L'intervallo ottimale di concentrazione finale dell'RNA è compreso tra 50 nM e 1m, mentre la concentrazione di metiltransferate è compresa tra 25 nM e 300 nM. - Mescolare accuratamente vorticendo delicatamente il tubo. Girare 5 s a 200 x g per raccogliere la soluzione nella parte inferiore del tubo. Incubare i tubi a 37 gradi centigradi per 2 h.
- Pulire la reazione utilizzando la purificazione della colonna come nel passaggio 1.32.
AVVISO: Fare molta attenzione a smaltire correttamente i materiali radioattivi, in particolare durante gli autoforni della colonna (pipette fuori invece di aspirare i rifiuti dai tubi di raccolta). - Eseguire il conteggio scintillio liquido
- Impostare il rack del conteggio scintillazione con una fiala per campione, una fiala per la misurazione dello sfondo e una fiala per il test di scorrimento. Riempire le fiale con 5 mL di soluzione di conteggio scintillio.
- Aggiungete 10 l di ogni campione di RNA radioattivo eluito in 1 fiala, e stringete il coperchio e mescolate delicatamente.
- Preparare le fiale di prova. Strofinare accuratamente tamponi di cotone su tutte le superfici e le attrezzature utilizzate durante il protocollo. Aggiungere tamponi alle fiale riempite con 5 mL di soluzione scintillante e stringere il coperchio.
- Eseguire gli esempi sul contatore di scintillazione come indicato di seguito. Aprire il cofano del contatore, inserire il rack nella macchina e chiudere il cofano. Selezionare Conteggio rack singolo . Selezionare Seleziona programma utente . Selezionare o creare un programma che misura il trizio (3H) per 60 s. Hit Count Rack. Ripetere il conteggio scintillio tre volte.
NOTA: l'apparecchiatura misurerà il conteggio scintillio di ogni campione ed emesso sia sullo schermo che su una stampa. Protocollo può essere messo in pausa qui, se lo si desidera. I campioni di RNA rimanenti a partire dalla fase 2.3 devono essere congelati a -80 gradi centigradi per un uso successivo.
- Procedere con l'autoradiogramma
- Preparare e pre-eseguire il gel di poliacrilammide di profilazione come nei passi 1.11-1.20.
- Pipette 20 -L di materiale di RNA radioattivo in un nuovo tubo da 1,5 mL contenente 20 -L di 2x Gel Loading Buffer. Mescolare bene. Preparare la scala come al punto 1.21. Incubare i campioni a 70 gradi centigradi per 15 min.
- Lavare i pozze del gel ancora una volta immediatamente prima del caricamento del campione. Caricare 20 l della scala preparata, 20 l dei campioni e 20 -L di 1x Gel Loading Buffer sulle corsie rimanenti. Eseguire il gel a 100 V per 60-180 min, a seconda della percentuale di poliacrilammide.
- Al termine della corsa del gel, togliere il gel dalla cassetta e metterlo in una scatola contenente 50 mL di 1x tampone TBE con 5 .L di macchia di gel acido nucleico ultrasensibile.
- Incubare per 5 min sul rocker per macchiare l'RNA.
- Estrarre con attenzione il gel dalla scatola e posizionarlo sul transilluminatore UV del sistema di imaging gel con i pozzi e la scala a sinistra.
- Mettere a fuoco la fotocamera sul gel, accendere la luce UV e quindi scattare un'immagine del gel esponendo da 50 ms a 1 s a seconda dell'intensità del segnale.
- Disattivare l'esposizione ai raggi UV e salvare l'immagine come file Tiff.
- Riposizionare il gel nella scatola. Rimuovere TBE. Fissare il gel con 50 mL di soluzione di fissaggio (50% di metanolo, 10% di acido acetico, 40% acqua ultra-pura) per 30 min a temperatura ambiente su un rocker.
- Spostare delicatamente il gel in una scatola nera fresca contenente 25 mL della soluzione di miglioramento dell'autoradiografia. In assenza di una scatola nera, coprire la scatola con un foglio di alluminio per proteggere la soluzione dalla luce.
- Incubare per 30 min a temperatura ambiente sul rocker.
- Sollevare delicatamente il gel e posizionarlo a faccia in giù su un foglio di involucro di plastica con i pozze e la scala sul lato destro. Posizionare due fogli di carta cromatografica sul retro del gel. Capovolgere delicatamente l'intera pila.
- Preriscaldare l'asciugatrice ad gel a 80 gradi centigradi. Spostare indietro il coperchio di plastica sull'asciugatrice ad appiglio. Inserire la pila di carta avvolgente, gel e cromatografia sotto il coperchio di plastica e spostare il coperchio di plastica verso il basso per creare un sigillo.
- Asciugare per 1 h a 80 gradi centigradi nell'asciugatrice ad acqua gel.
- Spegnere l'asciugatrice ad gel e rimuovere delicatamente la pila. Rimuovere l'avvolgimento e la seconda carta cromatografica. Nastro rimanente carta cromatografia con il gel essiccato in una cassetta di autoradiogramma.
- Aggiungi 1 foglio di pellicola di autoradiografia nella stanza buia.
- Posizionare la cassetta a -80 gradi centigradi e sviluppare il film dopo 1 h a 4 settimane a seconda dell'intensità del segnale, che può essere giudicato in base ai conteggi di scintillazione precedentemente misurati: 1-4 settimane per 250-1.000 cpm, 24 h a 1 settimana per 1.000-10.000 cpm e da 1 h a 24 h per >10.000 cp m.
- Una volta che il film è sviluppato, posizionare il film sulla parte superiore della cassetta e segnare con attenzione con un marcatore di laboratorio i 4 bordi del gel, ogni pozzo, e la posizione dei coloranti XC e BB.
- Eseguire la scansione della pellicola con una risoluzione di 300 o 600 pixel per pollice e salvare l'immagine come file Tiff.
Risultati
Reazione di trascrizione in vitro
Figura 2 A rappresenta una tipica esecuzione da una reazione di trascrizione in vitro con la polimerasi t7 RNA dello snRNA 7SK, che è relativamente breve (331 nt) e RNA altamente strutturato. Come mostrato in quell'immagine grezza, ci sono più bande indesiderate, sia più brevi che più lunghe di 7SK, probabilmente risultanti da eventi di inizio o terminazione trascrizionali casuali. Per questo motivo, la purificazione del gel dopo la reazione di trascrizione in vitro è importante per ottenere un campione di RNA pulito, come mostrato nella Figura 2B. A questo punto, l'RNA di interesse può essere identificato dalla sua posizione rispetto alla scala e purificato dalla purificazione del gel.
Come accennato in precedenza, potrebbe essere necessario ottimizzare la lunghezza della reazione di trascrizione prima della fase di purificazione del gel. Le reazioni di trascrizione che durano troppo a lungo possono portare a quantità estremamente elevate di produzione di RNA, rendendo difficile l'identificazione a valle della trascrizione corretta. È inoltre importante verificare l'identità della trascrizione purificata mediante la trascrizione inversa e la PCR quantitativa (RTqPCR) utilizzando primer specifici.
Analisi di metiltransferasi in vitro RNA
La figura 3 mostra un risultato rappresentativo di un'analisi della metiltransferasi dell'RNA descritta nel protocollo utilizzando il limite inferiore delle concentrazioni raccomandate di RNA e proteine. Questo test consente sia risultati quantitativi dai conteggi scintillativi, sia risultati qualitativi dall'autoradiogramma. Qui, MePCE, un metiltransferase dell'RNA noto al metillato 7SK10, ha dimostrato di essere in grado di metilare anche U6. Inoltre, come recentemente mostrato per 7SK16, il legame di istone H4 a MePCE inibisce anche la metilazione U6. Siamo stati in grado di osservare questo sia nel conteggio scintillazione (Figura 3C) e l'autoradiogramma (Figura 3B), mostrando la robustezza di questo protocollo.
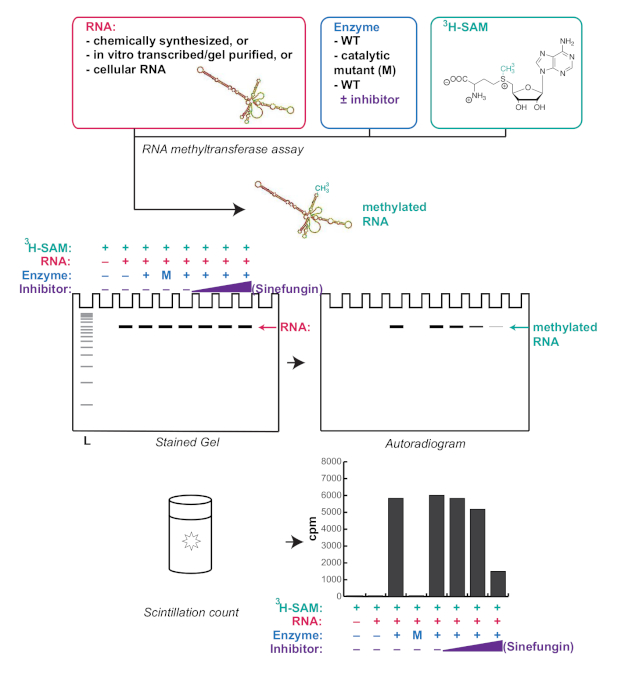
Figura 1 . Rappresentazione schematica di un tipico flusso di lavoro di saggio di metiltransferasi dell'RNA e dei risultati attesi. La sinefungina è un inibitore competitivo per il metiltransferasi. L: scala; WT: tipo selvaggio; M: mutante catalitico; cpm: conteggi per min. Fare clic qui per visualizzare una versione più grande di questa figura.
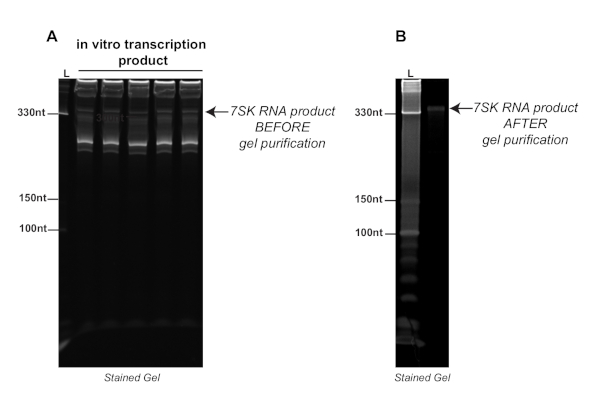
Figura 2 . Esperimento rappresentativo che mostra il prodotto della trascrizione in vitro prima (A) e dopo la purificazione (B) su una denatura di ure poliacrilammide 8% gel macchiato con macchia di acido nucleico. Le frecce indicano la trascrizione 7SK prima e dopo la purificazione del gel. L: scala. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3 . Analisi del trasferimento metilico in vitro eseguito con MePCE contro l'U6. Assaggio di metiltransferasi in vitro utilizzando GST-MePCE ricombinante (25 nM), 3SAM H-radioattivo come donatore di gruppo metilico e RNA U6 in vitro trascritto in vitro (50 nM) come substrato. L'autoradiogramma è stato esposto per 2 settimane al fine di rilevare l'attività residua (250 cpm) nel campione di Istone H4. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Qui, viene riportato un metodo semplice e robusto per la verifica in vitro dell'attività di metiltransferasi dell'RNA verso trascrizioni specifiche. Il saggio sfrutta il fatto che la methionina S-adenosil può essere tritiata sul donatore del gruppo metilico (Figura 1), consentendo di rilevare con precisione l'RNA metilato senza l'uso di anticorpi. Tuttavia, è importante notare che questo saggio non può indicare quale residuo o gruppo chimico è metilato dall'enzima. Per identificare o verificare che un residuo specifico sia metilato, altri metodi come l'analisi mutazionale, il blocco di trascrizione inversa o l'analisi della spettrometria di massa del substrato di RNA possono essere utilizzati in combinazione con il saggio di metiltransferasi dell'RNA.
La scelta degli RNA e la loro modalità di sintesi dipende dalle dimensioni dell'RNA. RNA specifici di dimensioni comprese tra 18 e 120 nt possono essere comodamente sintetizzati su misura da molti fornitori di acido nucleico di fama. Tuttavia, più comunemente, gli RNA specifici di varie dimensioni sono trascritti in vitro. I modelli di DNA per la trascrizione in vitro possono essere generati in vari modi e richiedono che la sequenza di interesse si trovi a valle di un promotore T7 o SP6. Quando le estremità precise degli RNA sono importanti, si consiglia di17plasmide il pR. L'elevata sensibilità dell'analisi (Figura 3) permette anche di sostituire un RNA purificato con una miscela di RNA cellulare, sia rHA totali che messaggeri, per esempio. Infatti, il gel e l'autoradiogramma forniscono un metodo per osservare direttamente le dimensioni degli RNA mirati per la metilazione.
Le condizioni qui riportate nel passaggio 2.1 del protocollo sono ottimali per la famiglia BIN3 di metiltransferasi, che ha due omologhi negli esseri umani, MePCE10 e BCDIN3D9. È importante sottolineare che le condizioni di saggio devono essere adattate alla proteina specifica e all'RNA di interesse. Ad esempio, è stato dimostrato che la presenza di MgCl2 riduce l'attività BCDIN3D18, tuttavia, MgCl2 può essere un importante coordinatore della struttura dell'RNA e potrebbe quindi costituire una componente importante del saggio per altri RNA Methyltransferases.
Il vantaggio di utilizzare un autoradiogramma, che può essere esposto per un lungo periodo di tempo, è che può consentire di rilevare attività molto deboli che non viene rilevata nel saggio di scintillazione. Di solito questo indica che la proteina è un metiltransferasi, ma che una o più delle condizioni di reazione o reagenti devono essere ottimizzati: enzima; substrato, cofattore, condizioni di buffer, ecc9. Ad esempio, potrebbe essere necessario migliorare la purificazione degli enzimi per rimuovere o modificare la posizione del tag. Può anche essere che l'RNA utilizzato come substrato deve essere piegato correttamente o per interagire con un altro fattore proteico o RNA per essere metilato dall'enzima. Pertanto, la letteratura preesistente e/o i propri risultati nelle cellule devono essere attentamente rivisti per impostare le condizioni di analisi per l'enzima e l'RNA di interesse.
Il saggio è anche estremamente flessibile. Ad esempio, può essere un passo precursore verso un altro tipo di saggio, in modo tale che l'RNA metilato radioattivo viene utilizzato nei saggi quantitativi e qualitativi demetillasi11, o nei saggi di legame dell'RNA che consentono di visualizzare specificamente il comportamento del RNA metilato rispetto a quello non modificato. Il saggio può anche essere leggermente regolato per analizzare la modifica degli RNA che utilizzano coenze che possono essere etichettati radioattivamente, come il coenzima acetil A per lo studio dell'acetilazione dell'RNA19,20.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare il Dr. Turja Kanti Debnath per il suo aiuto con ChemDraw. La ricerca nel laboratorio di Xhemalèe è supportata dal Department Of Defense - Congressionally Directed Medical Research Program - Breast Cancer Breakthrough Award (W81XWH-16-1-0352), NIH Grant R01 GM127802 e fondi di start-up dell'Istituto Biologia molecolare e College of Natural Sciences presso l'Università del Texas ad Austin, USA.
Materiali
| Name | Company | Catalog Number | Comments |
| 10 bp DNA Ladder | Invitrogen | 10821-015 | 10 bp DNA Ladder kit. |
| 10% Ammonium Persulfate (APS) | N/A | N/A | For urea denaturing polyacrylamide gel (For 10 mL, dissolve 1g in 8 mL of milliQ water; adjust volume to 10 mL with milliQ water; filter the solution using a 10 mL syringe equiped with a 0.45 µm filter). |
| 10X TBE Buffer | N/A | N/A | For urea denaturing polyacrylamide gel (For 1L, add 108 g of Tris Base, 55 g of Boric Acid to a cylinder with a stir bar; add 800 mL of distilled water and let dissolve; add 40mL of 0.5 M Na2EDTA (pH 8.0); adjust volume to 1L with milliQ water; filter the solution using a 0.22µm filter). |
| 10X TBS | N/A | N/A | For 1L, add 60.5 g of Tris Base, 87.6 g of NaCl to a cylinder with a stir bar; add 800 mL of distilled water and let dissolve; adjust pH to 7.5 with concentrated HCl; adjust volume to 1L with milliQ water; filter the solution using a 0.22 µm filter. |
| Acrylamide: Bis-Acrylamide 29:1 (40% Solution/Electrophoresis), Fisher BioReagents | Fisher | BP1408-1 | For urea denaturing polyacrylamide gel. |
| ADENOSYL-L-METHIONINE, S-[METHYL-3H]; (SAM[3H]) | Perkin Elmer | NET155V250UC | For in vitro methylation of RNA; Concentration = 1.0 mCi/mL; Specific activity = 17.1 Ci/mmol; Molarity= (1.0 Ci/L)/(83.2 Ci/mmol) = 0.0584 mmol/L = 58.4 µM. Upon receipt of the frozen 3H-SAM tube, thaw it at 4°C, make 20 µL aliquots, and freeze them at -30°C. Never refreeze and reuse a partially used aliquot. |
| Amersham Hypercassette Autoradiography Cassettes | GE Healthcare | RPN11649 | For autoradiogram gel exposure. |
| Amersham Hyperfilm MP | GE Healthcare | 28906846 | For autoradiogram gel exposure. |
| Beckman Scintillation Counter | Beckman | LS6500 | For liquid scintillation count. |
| Biorad Mini Horizontal Electrophoresis System | Biorad | 1704466 | Mini Horizontal Electrophoresis System. |
| cOmplete Mini EDTA-free Protease Inhibitor Cocktail Tablets | Roche Applied Science | 4693159001 | For a 20X solution, dissolve 1 tablet in 0.525 mL of nuclease free water. |
| Criterion Cell | Biorad | 345-9902 | RNase free empty cassette for polyacrylamide gel. |
| Criterion empty Cassettes | Biorad | 1656001 | Vertical midi-format electrophoresis cell. |
| DeNovix DS-11 Microvolume Spectrophotometer | DeNovix | DS-11-S | Microvolume Spectrophotometer for measuring DNA and RNA concentration. |
| Ecoscint Original | National Diagnostics | LS-271 | For liquid scintillation count. |
| Fisherbrand 7mL HDPE Scintillation Vials | Fisher | 03-337-1 | For liquid scintillation count. |
| Fluoro-Hance-Quick Acting Autoradiography Enhancer | RPI CORP | 112600 | For autoradiogram gel pretreatment. |
| Gel dryer | Biorad | 1651745 | For drying gel. |
| Gel Loading Buffer II | Ambion | AM8547 | For loading RNA in denaturing polyacrylamide urea gel (composition: 95% Formamide, 18 mM EDTA, and 0.025% SDS, Xylene Cyanol, and Bromophenol Blue). |
| GeneCatcher disposable gel excision tips | Gel Company | NC9431993 | For removing bands from agarose and polyacrylamide gels. |
| Megascript Kit | Ambion | AM1333 | For in vitro transcription with T7 RNA polymerase. |
| Perfectwestern Extralarge Container | Genhunter Corporation | NC9226382 (clear)/ NC9965364 (black) | Gel staining box. |
| pRZ | Addgene | #27663 | Plasmid for producing in vitro transcripts with homogeneous ends |
| Qiagen RNeasy MinElute Cleanup | Qiagen | 74204 | For RNA clean-up, use modified protocol provided in the protocol. |
| QIAquick Gel Extraction Kit (50) | Qiagen | 28704 | Kit for gel extraction and clean up of dsDNA fragment used for in vitro transcription. |
| Saran Premium Plastic Wrap | Saran Wrap | Amazon | For drying gel. |
| SYBR Gold | Invitrogen | S11494 | Ultra sensitive nucleic acid gel stain. |
| SYBR Safe | Invitrogen | S33102 | Nucleic acid gel stain. |
| TE | Sigma | 93283-100ML | 10 mM Tris-HCl, 1 mM disodium EDTA, pH 8.0 |
| TEMED | Fisher | 110-18-9 | For urea denaturing polyacrylamide gel. |
| Thermomixer with SMARTBLOCK 24X 1.5mL TUBES | eppendorf | 5382000023/5361000038 | For temperature controlled incubation of 1.5 mL tubes. |
| TOPO TA Cloning Kit | life technologies | Kits for fast cloning of Taq polymerase–amplified PCR products into vectors containing T7 and/or SP6 promoters for in vitro RNA transcription. | |
| TURBO DNase (2 U⁄µL) | Ambion | AM2238 | For DNA removal from in vitro transcription reactions. |
| Urea | Sigma | 51456-500G | For urea denaturing polyacrylamide gel. |
| Whatman 3MM paper | GE Healthcare | 3030-154 | Chromatography paper for drying gel. |
Riferimenti
- Xhemalce, B. From histones to RNA: role of methylation in cancer. Briefings in Functional Genomics. 12 (3), 244-253 (2013).
- Shelton, S. B., Reinsborough, C., Xhemalce, B. Who Watches the Watchmen: Roles of RNA Modifications in the RNA Interference Pathway. PLoS Genetics. 12 (7), 1006139 (2016).
- Mauer, J., et al. Reversible methylation of m(6)Am in the 5' cap controls mRNA stability. Nature. 541 (7637), 371-375 (2017).
- Wang, X., et al. N6-methyladenosine-dependent regulation of messenger RNA stability. Nature. 505 (7481), 117-120 (2014).
- Pendleton, K. E., et al. The U6 snRNA m(6)A Methyltransferase METTL16 Regulates SAM Synthetase Intron Retention. Cell. 169 (5), 824-835 (2017).
- Meyer, K. D., et al. 5' UTR m(6)A Promotes Cap-Independent Translation. Cell. 163 (4), 999-1010 (2015).
- Wang, X., et al. N(6)-methyladenosine Modulates Messenger RNA Translation Efficiency. Cell. 161 (6), 1388-1399 (2015).
- Alarcon, C. R., Lee, H., Goodarzi, H., Halberg, N., Tavazoie, S. F. N6-methyladenosine marks primary microRNAs for processing. Nature. 519 (7544), 482-485 (2015).
- Xhemalce, B., Robson, S. C., Kouzarides, T. Human RNA methyltransferase BCDIN3D regulates microRNA processing. Cell. 151 (2), 278-288 (2012).
- Jeronimo, C., et al. Systematic analysis of the protein interaction network for the human transcription machinery reveals the identity of the 7SK capping enzyme. Molecular Cell. 27 (2), 262-274 (2007).
- Liu, W., et al. Brd4 and JMJD6-associated anti-pause enhancers in regulation of transcriptional pause release. Cell. 155 (7), 1581-1595 (2013).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. Journal of Visual Experimentation. , (2019).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. Journal of Visual Experimentation. (63), e3998 (2012).
- Rong, M., Durbin, R. K., McAllister, W. T. Template strand switching by T7 RNA polymerase. Journal of Biological Chemistry. 273 (17), 10253-10260 (1998).
- Rio, D. C. Expression and purification of active recombinant T7 RNA polymerase from E. coli. Cold Spring Harb Protocols. 2013 (11), (2013).
- Shelton, S. B., et al. Crosstalk between the RNA Methylation and Histone-Binding Activities of MePCE Regulates P-TEFb Activation on Chromatin. Cell Reports. 22 (6), 1374-1383 (2018).
- Walker, S. C., Avis, J. M., Conn, G. L. General plasmids for producing RNA in vitro transcripts with homogeneous ends. Nucleic Acids Research. 31 (15), 82 (2003).
- Blazer, L. L., et al. A Suite of Biochemical Assays for Screening RNA Methyltransferase BCDIN3D. SLAS Discovery. 22 (1), 32-39 (2017).
- Arango, D., et al. Acetylation of Cytidine in mRNA Promotes Translation Efficiency. Cell. 175 (7), 1872-1886 (2018).
- Ito, S., et al. Human NAT10 is an ATP-dependent RNA acetyltransferase responsible for N4-acetylcytidine formation in 18 S ribosomal RNA (rRNA). Journal of Biological Chemistry. 289 (52), 35724-35730 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon