Method Article
Visualização fluorescente da Mango-Tagged RNA em géis de poliacrilamida através de um método de pós-coloração
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui nós apresentamos um método sensível, rápido, e discriminando da mancha do borne-gel aos RNAs da imagem etiquetados com os aptâmeros do RNA Mango mim, II, III, ou IV, usando géis nativos ou desnaturando do gel da electroforese do poliacrilamida (página). Depois de executar géis de página padrão, o RNA com a tag Mango pode ser facilmente manchado com TO1-biotina e depois analisado usando leitores de fluorescência comumente disponíveis.
Resumo
Os géis nativos e desnaturantes de poliacrilamida são rotineiramente utilizados para caracterizar a mobilidade complexa de ribonucleoproteína (RNP) e medir o tamanho do RNA, respectivamente. Como muitas técnicas da gel-imagem latente usam manchas não específicas ou as pontas de prova caras do fluoróforo, sensíveis, discriminando, e as metodologias econômicas da gel-imagem são altamente desejáveis. As seqüências do núcleo da manga do RNA são os motivos pequenos da seqüência (19 – 22 NT) que, quando fechados por uma haste arbitrária do RNA, podem ser simplesmente e acrescentado barata a um RNA do interesse. Estes Tag da manga ligam com a afinidade e a especificidade elevadas a um ligante thiazole-alaranjado do fluoróforo chamado to1-biotina, que se transforma milhares de vezes mais fluorescentes em cima da ligação. Aqui nós mostramos que Mango I, II, III, e IV pode ser usado especificamente para a imagem de RNA em géis com alta sensibilidade. Tão pouco quanto 62,5 fmol de RNA em géis nativos e 125 fmol de RNA em géis desnaturantes podem ser detectados por géis de imersão em um tampão de imagem contendo potássio e 20 nM TO1-biotina por 30 min. Nós demonstramos a especificidade do sistema Mango-etiquetado por imagem uma Mango-etiquetou o RNA bacteriano 6S no contexto de uma mistura complexa do RNA bacteriano total.
Introdução
Mango é um sistema de marcação de RNA constituído por um conjunto de quatro pequenos aptâmeros de RNA fluorescente que se ligam firmemente (ligação nanomolar) a derivados simples do thiazole-Orange (to1-biotina, Figura 1a)1,2,3 . Em cima da ligação, a fluorescência deste ligante é aumentada 1.000-a 4.000-dobre dependendo do aptamer específico. O brilho elevado do sistema da manga, que para a manga III excede aquele da proteína fluorescente verde aumentada (eGFP), combinada com a afinidade obrigatória do nanomolar dos aptamers da manga do RNA, permite que seja usado na imagem latente e na purificação do RNA complexos2,4.
As estruturas do raio X de Mango mim5, II6, e III7 foram determinadas à alta resolução, e todos os três aptâmeros utilizam um quadriplex do RNA para BIND to1-biotina (Figura 1b-D). Os núcleos compactos de todos os três aptâmeros são isolados da seqüência externa do RNA através dos motivos compactos do adaptador. Mango I e II ambos utilizam um adaptador de loop GNRA-like flexível para conectar seus núcleos de Mango a um duplex RNA arbitrário (Figura 1b, C). Em contraste, Mango III usa um motivo triplex rígido para conectar seu núcleo a uma hélice de RNA arbitrária (Figura 1D, resíduos roxos), enquanto a estrutura de Mango IV não é conhecida atualmente. Porque o núcleo ligand-obrigatório de cada um destes aptâmeros é separado da seqüência externa do RNA por estes adaptadores helicoidais, parece provável que podem tudo ser incorporados simplesmente em uma variedade de RNAs. O RNA regulamentar de 6s bacteriano (Mango i), componentes do spliceossoma do fermento (manga i), e o RNA humano de 5S, o RNA U6, e um scarna de C/D (Mango II e IV) foram marcados com sucesso nesta forma2,8, sugerindo que muitos os RNAs biológicos podem ser etiquetados usando o sistema do aptâmeros da manga do RNA.
A desnaturação e os géis nativos são comumente usados para estudar RNAs. Os géis denaturing são usados frequentemente para julgar o tamanho do RNA ou o processamento do RNA, mas tipicamente, no caso de um borrão do Norte, por exemplo, exija diversas etapas lentas e sequenciais a fim gerar uma imagem. Quando outros aptamers cromogênicos fluorogênicos do RNA, tais como o espinafre do RNA e os bróculos, forem usados com sucesso para a imagem latente do gel9, nenhum sistema cromogênicos fluorogênicos do aptâmeros até agora possui o brilho elevado e a afinidade do sistema da manga, fazendo lhe do interesse considerável para investigar as habilidades de imagem de gel de Mango. Neste estudo, nós perguntamos se o sistema da manga do RNA poderia simplesmente ser estendido à imagem latente do gel, porque os comprimentos de onda da excitação e da emissão de TO1-biotina (510 nanômetro e 535 nanômetro, respectivamente) são apropriados para a imagem latente no canal do eGFP comum à maioria de fluorescente Instrumentação de digitalização de gel.
O borne-gel que mancha o protocolo apresentado aqui fornece uma maneira rápida de detectar especificamente moléculas Mango-etiquetadas do RNA em géis nativos e desnaturando da electroforese do gel do poliacrilamida (página). Este método de coloração envolve géis de imersão em um tampão contendo potássio e TO1-biotina. Os aptâmeros da manga do RNA são G-quadruplex baseado e o potássio é exigido para estabilizar tais estruturas. Usando o RNA transcrito dos moldes mínimos do ADN da Mango-codificação (veja a seção do protocolo), nós podemos simplesmente detectar tão pouco quanto o fmol 62,5 do RNA em géis nativos e em 125 fmol do RNA em géis desnaturando, usando um protocolo de mancha direto. Em contraste com manchas de ácido nucleico inespecíficos comuns (ver tabela de materiais, referida SG de Hereon), podemos identificar claramente RNA com a tag Mango mesmo quando altas concentrações de RNA total não marcado estão presentes na amostra.
Protocolo
1. preparação dos reagentes
-
Solução de pós-coloração TO1-biotina (solução de coloração de gel)
- Faça 1 L de tampão fosfato de 1 M a pH 7,2 a 25 ° c adicionando 342 mL de 1 M de na2HPO4 e 158 ml de 1 m NaH2po4. Ajuste o pH para 7,2 a 50 mM adicionando a solução de fosfato apropriada. Estéril-filtre usando um filtro de 0,2 μm e armazene a solução no plasticware na temperatura ambiente.
- Prepare a solução de coloração de gel 5x (sem TO1-biotina) da seguinte forma. Faça uma solução de 1 L misturando 247,5 mL de ddH2O, 700 ml de 1 m de kcl, 50 ml de tampão fosfato de 1 m (pH 7,2) e 2,5 ml de Tween 20. Estéril-filtre usando um filtro de 0,2 μm e armazene a solução no plasticware. Ele pode ser armazenado em temperatura ambiente.
Nota: MgCl2 pode ser adicionado a esta solução, se necessário, pois pode potencialmente estabilizar os complexos de RNA. Isto terá um impacto modesto no sinal fluorescente2. - Compo uma solução de coloração do gel 1x usando a solução de coloração do gel 5x da etapa 1.1.2 e, imediatamente antes do uso, suplementá-la com o fluoróforo da to1-biotina (veja a tabela dos materiais) a uma concentração final de 20 nanômetro. Por exemplo, adicione 20 mL de solução de coloração de gel 5x a 80 mL de água desionizada para fazer 100 mL de solução de coloração de gel 1x e adicione 2 μL de um estoque de TO1-biotina de 1 mM (preparado em dimetilformamida).
Nota: o coeficiente de extinção do corante TO1-biotina a 500 nm é de 63.000 M-1· cm-1, medido na solução de coloração1.
-
PÁGINA de desnaturação (solução de carregamento de gel de desnaturação 2x e soluções A, B, C, persulfato de amônio e tetrametilethylenediamina)
- Prepare 50 mL de solução de carga de gel de desnaturação 2x misturando 40 mL de formamida, 0,5 mL de ácido etilenodiaminotetracético de 0,5 M (EDTA, pH 8,0) e 9,5 mL ddH2O. A solução pode ser armazenada à temperatura ambiente. Corantes de carga não são adicionados a esta solução, pois pode obscurecer a imagem fluorescente.
- Prepare 2x desnaturando gel de carregamento da tintura como segue. Ao purificar o RNA e os oligonucleotides do ADN, adicione 0,5 ml de 2,5% (w/v) de bromofenol azul (BB) e de 0,5 ml de 2,5% (w/v) de cyanol do do xileno (XC) à solução descrita em etapa 1.2.1, e adicione 8,5 ml de DDH2O em vez de 9,5 ml. A solução pode ser armazenada à temperatura ambiente.
- Preparar 100 mL de solução A pesando 200,2 g de ureia (peso da fórmula: 60, 6 g/mol) e adicioná-lo a 312,5 mL de 40% 19:1 acrilamida: N, n'-metilenebisacrilamida. Mexa-o com uma barra de agitação magnética na velocidade máxima até dissolver (cerca de 3 h) e compõem a solução para 500 mL usando ddH2o. Note que as concentrações finais são de 6,667 M de ureia e 25% 19:1 acrilamida: N, n'-metilenebisacrilamida. Conservar a 4 ° c em plasticware limpo.
- Prepare 1 L de solução B pesando 400,4 g de ureia e faça a solução para 1 L com ddH2o; agitar até que a ureia tenha dissolvido. A solução B pode ser mantida à temperatura ambiente.
- Prepare a solução C (10x Tris-borato-EDTA [TBE]) da seguinte forma. Prepare 4 L de 10x TBE pesando 432 g de base Tris e 220 g de ácido bórico, acrescentando 160 mL de 0,5 M de EDTA com um pH de 8 (filtrado), e fazendo a solução para 4 L com ddH2o. Um grande estoque deste tampão é feito porque é usado igualmente como o amortecedor running do gel.
- Prepare 10% de persulfato de amônio (APS) dissolvendo 1 g de APS em um volume total de 10 mL de ddH2O. armazene a 4 ° c.
- Manter tetrametilethylenediamine (TEMED) acessível. Guarde-o a 4 ° c juntamente com a solução APS.
-
PÁGINA nativa (2x solução de carregamento de gel nativa)
- Prepare 50 mL de solução de carregamento de gel nativo 2x misturando 25 mL de 100% de glicerol, 10 mL de solução de coloração de gel 5x e 15 mL de ddH2o para compensar a solução para 50 ml.
Nota: como na seção de desnaturação do gel, os corantes de carregamento podem ser adicionados a este estoque, mas sua adição pode potencialmente obscurecer o sinal fluorescente no gel e, assim, deve ser evitado.
- Prepare 50 mL de solução de carregamento de gel nativo 2x misturando 25 mL de 100% de glicerol, 10 mL de solução de coloração de gel 5x e 15 mL de ddH2o para compensar a solução para 50 ml.
2. preparação e carregamento de géis de desnaturação
- Prepare um gel de desnaturação com uma percentagem adequada seguindo a tabela 1. Considere o sistema específico da carcaça do gel; são comumente usados géis de 30 mL. A percentagem de gel adequada pode ser estimada utilizando a tabela 2: selecionar a percentagem de poliacrilamida onde os corantes BB e XC têm motilidade mais rápida e lenta, respetivamente, do que o RNA de interesse, para garantir a separação de banda alta no tamanho relevante Gama.
- Misture as soluções A, B e C de acordo com a tabela 1 e adicione APS e TEMED imediatamente antes de derramar o gel. Misture bem as soluções em plasticware limpos.
- Despeje a solução de gel em um aparelho de fundição de gel apropriado, depois de garantir que todos os componentes são escrupulosamente limpos. Levante um lado do aparelho para que o gel seja ligeiramente inclinado ao derramar, para evitar que as bolhas de ar se tornem presas dentro do próprio gel.
- Insira o pente desejado e deixe-o polimerizar (aproximadamente 30 min). Observe a polimerização olhando atentamente para uma mudança no índice de refração em torno dos poços de gel. Compo o tanque do gel usando a solução 1x C (1x TBE) e remova com cuidado o pente. Usando uma seringa, aspirar os poços imediatamente antes do carregamento da amostra.
- Prepare amostras de desnaturação da seguinte forma. Adicionar 2x desnaturando gel solução de carga para as amostras de RNA de interesse para fazer a solução de carregamento de gel de desnaturação 1x e calor-denature a 95 ° c por 5 min usando um termocicer ou banho de água.
- Antes de carregar as amostras, ar-esfrie-os diversos minutos até que estejam frescos ao toque. Carregue as amostras mergulando-as na parte inferior de cada poço, usando pontas do gel-carregamento.
Nota: as pontas redondas podem ser usadas para géis de 1 mm de espessura ou mais; Use pontas planas para géis mais finos. - Execute os géis desnaturantes à temperatura ambiente. Assegure-se de que a wattagem seja suficientemente baixa de modo que as placas de vidro do sistema do gel não rachem.
Nota: em nosso laboratório, 28 W para as placas de 20 cm x de 16 cm eram suficientes para esta finalidade.
3. preparação e carregamento de géis nativos
- Prepare um gel nativo com uma percentagem adequada referindo-se à tabela 3. Considere o sistema específico de fundição de gel, mas tenha em mente que os géis de 30 mL são comumente usados. Misture as soluções de 1x TBE, 40% 29:1 acrylamide: N, n'-methylenebisacrylamide, e glicerol de acordo com a tabela 3, e adicionar APS e TEMED imediatamente antes de derramar o gel. Proceda conforme descrito nas etapas 2,3 e 2,4.
- Prepare amostras nativas, adicionando 2x solução de carregamento de gel nativa para as amostras de RNA de interesse para fazer a solução 1x e permitir que ele incubar em temperatura ambiente para 100 min antes de executar o gel para garantir a dobra de RNA completo.
- Funcione o gel nativo em uma sala fria de 4 ° c, assegurando-se de que a wattagem seja suficientemente baixa para não aquecer o gel.
Nota: em nosso laboratório, 14 W para as placas de 20 cm x de 16 cm eram suficientes para esta finalidade.
4. preparação do RNA pelo run-off T7 transcrição
Nota: as sequências de DNA utilizadas para a transcrição de run-off10 de construções de manga de RNA foram encomendadas comercialmente. Neste método, os oligonucleotídeos do ADN que contêm o complemento reverso (RC) de ambos a seqüência a ser transcrita e o promotor T7 são hibridizados a uma seqüência superior da vertente do promotor T7 e transcritas então in vitro. Abaixo, para cada oligonucleotídeo, o RC da seqüência de núcleo Mango é mostrado em negrito e o RC da região do promotor T7 é mostrado em itálico. Os resíduos na fonte regular correspondem às regiões helicoidais complementares de outra maneira arbitrárias exigidas para permitir que o núcleo da manga dobre corretamente.
Mango I: GCA CGT ACT CTC CTC TCC GCA CCG TCC CTT cgt ACG TGC cta tag TGA GTC GTA TTA AAG
Mango II: GCA CGT ACT CTC CTC TTC CTC TCC TCT CCT cgt ACG TGC cta tag TGA GTC GTA TTA AAG
Mango III: GGC ACG TAC GAA tat ACC Aca TAC CAA TCC TTC cTT CGT ACG TGC cta tag TGA GTC GTA TTA AAG
Manga IV: GCA CGT ACT CGC CTC ATC CTC ACC Act CCC TCG GTA CGT CCG tat Agt gag TCG tat Taa AG
T7 Top Strand: CTT TAA TAC GAC TCA CTA TAG G
- Purificação do gel preparative de oligonucleotídeos do ADN
- Para uma síntese de DNA em escala de 0,2 μmol, Ressuspender o oligonucleotídeo deprotected DNA em 100 μL de ddH2o e 100 μl de corante de carregamento de desnaturação 2x (da etapa 1.2.2). Neste caso, incluindo corantes de carregamento de gel na solução de carga na mesma concentração que na etapa 1.2.2 é preferível.
- Purify o oligonucleotide do ADN usando um gel desnaturando da escala preparativa de 50 ml da porcentagem apropriada do poliacrilamida; Use a tabela 2 para escolher a percentagem de gel. Por exemplo, use um gel de 8% para uma seqüência de 50 NT. Idealmente, o azul do bromofenol deve funcionar mais rapidamente do que o oligonucleotide, e o cyanol do do xileno deve funcionar mais lento.
Nota: em nosso laboratório, tais géis foram fundidos usando espaçadores de fundição com 1,5 mm de espessura e um pente de carregamento de gel com poços de 2 – 2,5 cm de largura. - Carregar 100 μL de soluções de oligonucleotídeo de ADN preparadas no passo 4.1.2 por gel preparativo bem como descrito nos passos 2.4 – 2.7.
- Seque com cuidado a parte externa do gel, retire as placas de vidro, e cubra ambos os lados do gel no envoltório plástico. Coloque-o em uma tela de imagem fluorescente (placas de cromatografia de camada fina de alumínio impregnadas com fluoróforo são uma solução econômica) e usar um curto-comprimento de onda UV mão-realizada lâmpada para visualizar as bandas de DNA por sombreamento UV.
- Uma sombra nítida e bem definida deve ser observada se a síntese de DNA for de alta qualidade. Marque as bandas no envoltório de plástico, usando um marcador permanente.
Nota: Proteja a pele e os olhos da luz UV e mantenha as exposições curtas para evitar danificar a amostra de ácido nucleico. - Colocando o gel em uma placa de vidro limpa, cuidadosamente cortar as bandas marcadas e coloque cada fragmento de gel em 400 μL de 300 mM NaCl. Elute o DNA durante a noite, usando um rotator à temperatura ambiente.
- Recupere o eluente em um tubo de centrifugação limpo e adicione 2,5 equivalentes de etanol para preciporar o DNA. Vórtice bem e coloque o tubo a-20 ° c por 30 min.
- Pellet a amostra em uma centrífuga de bancada em 156.000 x g por 30 min a 4 ° c. Retire cuidadosamente o sobrenadante e ressuscitem o pellet em ddH2o. Use um espectrofotômetro para determinar com precisão a concentração de oligonucleotídeo de DNA. Armazene a amostra como um estoque de 10 μM para a conveniência em-20 ° c.
- Transcrição de run-off e purificação de gel de amostras de RNA
- Prepare o estoque do trifosfato do nucleosídeo 5x (NTP) misturando estoques líquidos de NTP compo de uma concentração final do trifosfato do guanosina de 40 milímetros (GTP, 11.400 M-1· cm-1), trifosfato do citidina de 25 milímetros (CTP, 7.600 M-1· cm-1), 25 milímetros trifosfato de adenosina (ATP, 5.000 M-1· cm-1) e trifosfato de URIDINA 10 mM (UTP, 10.000 M-1· cm-1); todos os coeficientes de extinção a 260 nm.
Nota: os estoques líquidos ou pulverizados podem ser obtidos comercialmente. Se preparar estoques primários de pó, ajuste cuidadosamente o pH para um pH final de 7,9, usando NaOH de 1 M. Aliquot o estoque em 1,5 mL de tubos de microcentrífuga e armazená-lo em-20 ° c. - Prepare 100 mL de estoque de tampão de transcrição de 10x T7 misturando 25,6 mL de Tris-HCl de 1 M, 14,4 mL de base Tris de 1 M, 26 mL de 1 M MgCl2, 10 ml de Triton X-100 de 10% e 0,637 g de spermidina. Compõem o estoque para um volume final de 100 mL, adicionando ddH2O.
Nota: um pH final de 7,9 da solução 1x deve ser confirmado usando um medidor de pH calibrado. O 10x deve ser estéril-filtrado e armazenado em-20 ° c em alíquotas apropriadamente feitas medida. - Realize a transcrição da seguinte forma.
- Adicione os seguintes reagentes para compensar uma concentração final de tampão de transcrição 1x T7 usando o estoque de buffer de transcrição de 10x T7 e DDH2o (da etapa 4.2.2), 1x ntps, 10 mm de ditiotreitol (DTT), seqüência de 1 μm T7 Top Strand (ver seção 4 Nota para o seqüência), 1 μM gel-purified a seqüência do ADN da manga (etapa 2,1), e a enzima do polymerase do RNA T7 (1 U/μL).
- Vortex, gire para baixo, e incubar a solução em 37 ° c para 2 h ou até que se torne nebuloso e um precipitar branco se forma na parte inferior do tubo. Uma transcrição de 50 μL deve resultar em 50 μL de RNA de ~ 50 μM após A purificação do gel. Adicione um volume igual de tintura de desnaturação 2x e armazene-o em-20 ° c até pronto para a purificação do gel.
- Gel-purify o RNA resultante como descrito na seção da purificação do gel dos oligonucleotídeos do ADN seguindo as etapas 4.1.2 \ u 20124.1.8, substituindo a amostra do RNA para o ADN.
Nota: o RNA é extremamente sensível à degradação de RNase, assim que assegure-se de que em todas as etapas, as luvas e um revestimento limpo do laboratório estejam desgastados em todas as vezes. Assegure-se de que todas as amostras estejam preparadas e armazenadas em plasticware de uso único para proteger contra a contaminação por RNase, e Lave cuidadosamente as placas de vidro com água e sabão quente antes do uso, enxague-as cuidadosamente com ddH2o e seque os copos com o assistência do etanol aplicado a partir de uma garrafa de aperto. - Utilizar 1 μL da amostra de RNA final e, utilizando um espectrofotômetro à base de gotas, determinar a absorvância a 260 nm. Usando um coeficiente de extinção determinado pelo método vizinho mais próximo, calcule a concentração de RNA e ajuste a concentração da amostra para 10 μM com ddH2O para conveniência. Guarde as amostras de RNA a-20 ° c.
- Prepare o estoque do trifosfato do nucleosídeo 5x (NTP) misturando estoques líquidos de NTP compo de uma concentração final do trifosfato do guanosina de 40 milímetros (GTP, 11.400 M-1· cm-1), trifosfato do citidina de 25 milímetros (CTP, 7.600 M-1· cm-1), 25 milímetros trifosfato de adenosina (ATP, 5.000 M-1· cm-1) e trifosfato de URIDINA 10 mM (UTP, 10.000 M-1· cm-1); todos os coeficientes de extinção a 260 nm.
- Escherichia coli extração de ácido nucleico bruto total
Observação: o seguinte protocolo é um exemplo. Este protocolo utiliza endogenamente expresso Mango I-Tagged 6S RNA de um plasmídeo em e. coli, e a indução deste plasmídeo é descrita em outro lugar em detalhe4.- Para os propósitos deste estudo, prepare 500 mL de células induzidas tanto para o pEcoli-RNA Mango quanto para os plasmídos pEcoli-T1 (as células são induzidas em um OD600Nm de 1). Pellet as células e armazená-los em-80 ° c antes de usar.
- Execute a extração do RNA como segue.
- Tome 0,5 mL da pelota induzida da pilha de pEcoli e adicione 500 μL do phenol equilibrado. Em seguida, vortex a amostra; a solução tornar-se-á branca leitosa. Centrifugue na velocidade máxima por 2 minutos para separar as camadas.
- Extraia a camada superior, que será ligeiramente amarela, e repita a etapa 4.3.2.1 adicionando um volume igual de phenol, centrifugação, e extraindo para um total de 5x (ou até a camada média, que é branco opaco, vai afastado e somente duas camadas desobstruídas são deixadas).
- Adicione o volume aquoso extraído de fenol a um volume igual de clorofórmio, vortex e, em seguida, centrifugue na velocidade máxima por 2 min.
- Extraia a camada aquosa e adicione NaCl a uma concentração final de 300 mM. Precipeie o ácido nucleico resultante pela adição de 2,5 equivalentes de etanol, vortex, spin down e precipitado a-20 ° c por pelo menos 30 min.
- Pellet em uma centrífuga de bancada em 15,6 x 1.000 x g a 4 ° c por 30 min. Retire cuidadosamente o sobrenadante e ressuscitem o pellet em 100 ΜL de DDH2o.
- Adicione uma etapa da digestão de DNase I a este procedimento, seguindo o protocolo referenciado11, para obter o RNA total.
Nota: Vortex vigorosamente até que o pellet é completamente dissolvido em ddH2o.
5. pós-gel de coloração
- Prepare a solução de coloração do gel 1x como por a receita na etapa 1.1.3 e adicione-a a um recipiente de vidro limpo que seja largamente bastante caber confortavelmente o gel.
Nota: os recipientes de vidro de borosilicate com tampas encaixando do ajuste servem a esta finalidade bem. - Adicione o suficiente 1x gel mancha solução para o recipiente de modo que o gel é completamente coberto com a solução e os chapinha líquido sobre a parte superior do gel quando ele é colocado sobre o rotor orbital.
Nota: o recipiente deve ser grande o suficiente para caber o gel para que ele possa se mover e ter o suficiente buffer no recipiente para cobrir totalmente o gel. - Uma vez que o gel nativo ou desnaturante terminou de funcionar (seção 2 ou 3, respectivamente), retire o gel do aparelho e corte seus poços. Pode ser útil remover um canto do gel para a orientação mais tarde na análise.
- Transfira cuidadosamente o gel para a solução de coloração de gel 1x preparada na etapa 5,2.
Nota: os géis são frágeis e propensos a quebrar por isso tenha cuidado ao transferir o gel. Mantenha a tampa no recipiente de coloração em todas as vezes de modo a não contaminar o gel. - Coloc o gel em um rotator orbital em uma velocidade de 100 RPM por 30 minutos na temperatura ambiente.
Nota: Certifique-se de que o gel não dobre de volta para si mesmo; caso contrário, o RNA pode difundir fora do gel e assim rotular uma parte diferente do gel.
6. Imaging Mango-Tagged RNAs em gel
- Decantar cuidadosamente o tampão de imagem. Enxague rapidamente o gel com água, assegurando restos líquidos suficientes para manter o gel ligeiramente móvel no recipiente.
- Transfira cuidadosamente o gel para a Imager. Transferência cuidadosamente pegando os lados do gel e colocando-o na bandeja de Imager; Alternativamente, o gel pode ser lentamente derramado sobre a bandeja. Certifique-se que não há bolhas debaixo do gel e que não há excesso de líquido o gel. Uma pipeta rolou sobre o gel pode ser útil remover todo o líquido adicional.
Nota: Use uma toalha de papel para absorver o excesso de fluido. - Tome uma imagem do gel, observando a fluorescência entre o comprimento de onda 510 nm da excitação e o comprimento de onda 535 nm da emissão (por exemplo, luz verde em 520 ajustes da fluorescência do comprimento de onda do nm no Imager). Certifique-se de que as definições estão totalmente otimizadas no instrumento e siga atentamente as instruções para o instrumento utilizado.
Resultados
Short Mango-Tagged RNAs foram preparados como descrito na seção de protocolo. Supondo-se que a fluorescência em condições de desnaturação seria mais difícil de observar devido à presença de ureia nos géis, estudamos pela primeira vez a resistência dos aptâmeros de manga à ureia, que atua como um ácido nucleico desnaturante. Verificou-se que os aptâmeros de manga são substancialmente resistentes à desnaturação até uma concentração de ureia de aproximadamente 1 M (Figura 2a). Antes de adicionar a solução de coloração de gel a um gel desnaturante, a concentração final de ureia no gel é de 6 M. adicionando solução de coloração suficiente para diminuir esta concentração para 1 M seria, portanto, ideal para garantir a fluorescência de manga completa para todos os quatro aptamers. Na prática, o protocolo de coloração alcançou menos do que este resultado inteiramente óptimo, mas este poderia simplesmente ser retificado se necessário usando mais solução de mancha ou pelo expediente simples de mudar a solução uma vez durante a mancha.
Uma vez que o Mango-Tagged RNA construções foram executados em um gel desnaturante, o tempo de coloração foi otimizado para a máxima fluorescência de gel, carregando três diferentes Mango III quantidades em um 8% gel de desnaturação (Figura 2b). Um curso do tempo revelou que após 5 minutos da imersão na solução de mancha do gel, A fluorescência era claramente visível. A fluorescência máxima do constructo Mango III foi obtida após 20 – 40 min de coloração, após o que as pequenas RNAs utilizadas neste estudo passaram a se difundir fora do gel, resultando em perda de sinal fluorescente (Figura 2b). Conseqüentemente, ambos os géis nativos e desnaturantes foram manchados por 30 minutos cada. As construções mais longas do RNA podiam facilmente tolerar uns tempos manchados mais longos porque seriam muito menos prováveis difundir fora do gel.
Alguns dos aptâmeros RNA Mango dobrado mais rapidamente do que outros. Cada um dos aptâmeros de manga utilizados neste estudo foi incubado em um tampão de gel suplementado com 1,5 M de ureia e 100 nm to1-biotina corante e analisado usando um fluorômetro. Mango I, II e III foram totalmente dobrados após 10 min, enquanto Mango IV tornou-se substancialmente dobrado somente após 40 min (Figura 3a). Na ausência de ureia, o dobramento foi muito mais rápido do que era esperado (Figura 3B). Para garantir que as amostras de gel nativas foram totalmente dobrado, nós amostras pré-incubadas para 100 min antes de executá-los em géis nativos. Praticamente, os dados na Figura 3 sugerem que este tempo poderia ser substancialmente reduzido, dependendo do aptâmeros Mango usado.
Uma vez que o protocolo foi otimizado para detectar a fluorescência do aptâmeros do RNA Mango, a sensibilidade do método de pós-coloração foi determinada para cada uma das variantes de Mango em ambos os géis nativos. Foram observadas bandas únicas correspondentes a RNAs bem dobradas para cada um dos quatro mangos em géis nativos (figura 4a). Em cima da diluição de série, tão pouco quanto 62,5 fmol de Mango II podiam ser observados, quando tão pouco quanto 125 fmol de Mango I, III, e IV foram visualizados facilmente. A quantificação de géis nativos foi log-linear em cerca de 1,5 ordens de magnitude, com Mango I, II e IV comportando-se de forma mais linear do que Mango III (Figura 4B).
Os resultados dos géis de desnaturação foram ligeiramente menos sensíveis do que os géis nativos, mas foram mais lineares. Tão pouco quanto 125 fmol de Mango II e III foram facilmente detectados (Figura 4C). Curiosamente, a quantificação (Figura 4D) indicou que os géis desnaturantes eram log-lineares ou duas ordens de magnitude. Nós supor que, em contraste com os géis nativos onde as dobras do RNA foram sujeitados talvez à desnaturação parcial durante o processo running do gel, a presença de Urea no gel desnaturando pôde fornecer uma maneira mais homogênea de dobrar os aptâmeros uma vez que são coloc em a solução de coloração TO1-biotina.
Tal como com todas as metodologias de coloração de gel, se o gel não for cuidadosamente transferido para o contentor, ou se a velocidade de rotação for demasiado elevada durante o período de coloração, o gel pode dobrar para si próprio (Figura 5a). Isto pode conduzir à amostra Mango-etiquetada do RNA que difunde de um lugar do gel a outra mas pode facilmente ser evitada na prática. Em géis nativos e, particularmente, para Mango IV, observamos que a dobradura incompleta pode se manifestar no aparecimento de múltiplas bandas presumivelmente correspondentes a conformações RNA parcialmente/misfolded (Figura 5b) resultantes de tempos de dobramento mais curtos. As edições de dobramento em géis nativos podem ser evitadas preincuating amostras do RNA apropriadamente como descrito previamente e executando géis nativos em uma sala fria. Em géis desnaturantes, onde o RNA se dobra in situ dentro do gel, o proteínas não era um problema significativo. Finalmente, na ausência de RNA, muito pouca fluorescência do fundo foi observada em um ou outro sistema do gel.
Em seguida, a especificidade do RNA Mango tag foi estudada por sobreexpressar o RNA regulamentar de 6S em bactérias. Este RNA foi previamente etiquetado usando Mango I (Figura 1e)4. As células bacterianas foram transformadas com o plasmídeo de pEcoli-RNA Mango (doravante plasmídeo M) ou o plasmídeo pEcoli-T1 como controle negativo (doravante, E plasmídeo). As pilhas transformadas foram crescidas no meio líquido do caldo lisogenia até que um OD600 de 1,0 estêve alcangado. As culturas foram então induzidas com 50 μM de isopropílico β-D-1-tiogalactopyranoside (IPTG) por 40 min. As células foram colhidas por centrifugação a 6.000 x g por 15 min. o RNA total foi extraído usando a extração de clorofórmio fenol-fenóis de pelotas de células12 , conforme descrito na seção protocolo. As amostras totais do RNA foram concentradas pela precipitação do ethanol e tratadas então com o DNase mim, seguindo o protocolo, para remover o ADN11. Antes de usar, o RNA foi concentrado pela precipitação do etanol.
O RNA bacteriano total foi executado em géis desnaturação de 8% (Figura 6) e manchado com o SG13 ou o to1-Biotin. Para coloração SG, 10 μL de 10, 000x SG foi adicionado a 100 mL de tampão de gel; caso contrário, o protocolo de coloração era idêntico ao utilizado para a TO1-biotina. Como esperado, a coloração SG forte foi observada para uma infinidade de RNAs, mas mais proeminente para ribossomal (rRNA) e transferência RNAs (tRNA) (Figura 6, painel esquerdo). Quando a coloração Mango-dependente (pistas de M) poderia ser considerada nestes géis SG-manchados, não poderiam ser identificadas excepcionalmente dado o teste padrão de mancha complexo observado usando esta mancha universal. Em contraste, os géis TO1-biotina destacaram bandas dependentes de manga como as bandas mais proeminentes. Somente as bandas de RNA ribossômicas são consideradas em competição com as bandas 6S Mango-Dependent. Uma série de bandas não especificamente manchadas também pode ser observada. No entanto, as bandas dependentes de Mango foram novamente dominantes, tendo apenas as bandas rRNA e tRNA como concorrentes fracamente competindo (Figura 6, painel direito).

Figura 1: sistema de aptâmeros de manga. (A) fluoróforo to1-biotina. (B) Mango I. (C) Mango II. (D) Mango III. Os painéis B–D mostram a estrutura secundária de cada apdomador. P1 é uma haste arbitrária. O GNRA-like haste-loop (aqui GAAA) encontrado em Mango I e II é mostrado em vermelho, o motivo triplex de Mango III é mostrado em roxo. (E) o RNA regulamentar 6s marcado com Mango I. por favor clique aqui para ver uma versão maior desta figura.
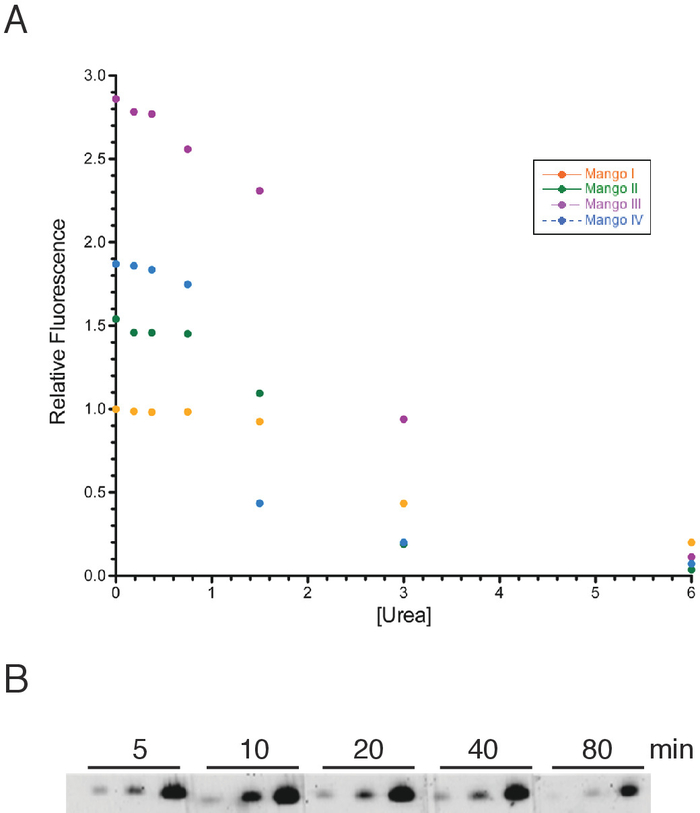
Figura 2: efeito da ureia na fluorescência do aptâmeros da manga e nos tempos de coloração ideais. (A) titulação de ureia utilizando 50 NM RNA Mango I (círculos alaranjados), Mango II (círculos verdes), Mango III (círculos roxos) e Mango IV (círculos azuis), juntamente com corante 100 nm to1-biotina e nas concentrações de ureia indicadas. As amostras foram incubadas por 40 min antes que a fluorescência fosse lida em um comprimento de onda da excitação de 510 nanômetro e em um comprimento de onda da emissão de 535 nanômetro. (B) o RNA da manga III foi carregado em um gel desnaturando de 8% e manchado com uma solução do gel que contem a tintura final do to1-biotina de 20 nanômetro. Para cada ponto de tempo indicado, 0, 64, 0,32 e 1,6 pmol de Mango III RNA foi usado da esquerda para a direita. A imagem do gel foi visualizada com um Imager da fluorescência usando um laser de 520 nanômetro e uma exposição de 10 minutos. Por favor clique aqui para ver uma versão maior desta figura.

Figura 3: tempos de dobra dos apdomadores de manga na presença e ausência de ureia. (A) na presença de 1,5 M de ureia e 100 nm to1-biotina, os cursos de tempo de fluorescência foram realizados com 50 nm de cada construção de RNA Mango (RNA Mango I: pontos alaranjados, Mango II: pontos verdes, Mango III: pontos roxos e Mango IV: pontos azuis). (B) idênticos ao painel a, excepto na ausência de ureia. Todos os cursos de tempo foram realizados à temperatura ambiente. Por favor clique aqui para ver uma versão maior desta figura.

Figura 4: imagens fluorescentes de página nativa e desnaturante com construções de RNA Mango. (A) um gel nativo de 8% com construções serialmente DILUÍDAS do RNA Mango. As faixas I, II, III e IV contêm, respectivamente, 8 quantidades finais de pmol de RNA Mango I, II, III e IV. Os painéis direito são duas diluições seriais duplas contendo 4 pmol, 2 pmol, 1 pmol, 0,5 pmol, 0,25 pmol, 0,125 pmol e 0, 625 pmol de qualquer Mango I, II, III ou IV, conforme indicado. Lane 12 não contém RNA. (B) quantificação de três repetições do gel nativo (desvio padrão da média mostrada para cada). (C) um gel desnaturação de 8% com as mesmas amostras carregadas como no painel a, à exceção do fato que a solução de carregamento do gel de desnaturação foi usada em vez da solução nativa do carregamento do gel. D) três repetições quantificadas de gel desnaturante (desvio-padrão da média indicada para cada). Todas as imagens do gel foram visualizadas um Imager do gel com um laser de 520 nanômetro e uma exposição de 10 minutos. Por favor clique aqui para ver uma versão maior desta figura.

Figura 5: géis suboptimal e dobradura incompleta de Mango IV em um gel nativo de 8%. (A) uma diluição seriada da espécie mostrada na Figura 4 para Mango II, mas mostrando o efeito da dobradura de gel durante o protocolo de coloração. (B) amostras nativas do gel da manga IV que não foram permitidas dobrar para o tempo bastante no amortecedor nativo antes do carregamento do gel exibem bandas duplas. Caso contrário, esses resultados são semelhantes aos resultados da Mango IV mostrados na figura 4a. Todas as imagens do gel foram visualizadas usando um Imager do gel com um laser de 520 nanômetro e uma exposição de 10 minutos. Por favor clique aqui para ver uma versão maior desta figura.

Figura 6: RNAs com a tag Mango pode ser detectada na presença de RNA total, usando a coloração de to1-biotina. 8% de géis de desnaturação foram carregados com 100 ng de RNA total e foram executados por 30 min. O gel esquerdo foi manchado com SG e o painel direito com TO1-biotina. Para ambos os painéis, as pistas rotuladas E foram carregadas com 100 ng de pEcoli-T1 (sem tag Mango) e as pistas rotuladas M foram carregadas com 100 ng de pEcoli-RNA Mango (6S RNA marcados com uma tag Mango I). TO1-as imagens do gel manchado biotina foram visualizadas usando um Imager com um laser de 520 nanômetro e uma exposição de 10 minutos. As imagens de gel com coloração SG foram visualizadas usando um gerador de gel usando um laser de 460 nm e uma exposição de 10 min. Por favor clique aqui para ver uma versão maior desta figura.
| Porcentagem | VOLUME DO GEL | |||
| 20 mL de | de 30 mL | 50 mL | ||
| 5 | Um | 4 | 6 | 10 |
| B | 14 | 21 | 35 | |
| C | 2 | 3 | 5 | |
| 6 | Um | 4,8 | 7,2 | 12 |
| B | 13,2 | 19,8 | 33 | |
| C | 2 | 3 | 5 | |
| 8 | Um | 6,4 | 9,6 | 16 |
| B | 11,6 | 17,4 | 29 | |
| C | 2 | 3 | 5 | |
| 10 | Um | 8 | 12 | 20 |
| B | 10 | 15 | 25 | |
| C | 2 | 3 | 5 | |
| 12 | Um | 9,6 | 14,4 | 24 |
| B | 8,4 | 12,6 | 21 | |
| C | 2 | 3 | 5 | |
| 15 | Um | 12 | 18 | 30 |
| B | 6 | 9 | 15 | |
| C | 2 | 3 | 5 | |
| 20 | Um | 16 | 24 | 40 |
| B | 2 | 3 | 5 | |
| C | 2 | 3 | 5 | |
| APS (μL) | 48 | 72 | 120 | |
| TEMED (μL) | 20 | 30 | 50 | |
Tabela 1: desnaturando a tabela da carcaça do gel da página. A = solução A, B = solução B, C = solução C.
| Gel de desnaturação% | BB (~ mobilidade NT) | XC (~ mobilidade em NT) |
| 5 | 35 | 130 |
| 6 | 26 | 106 |
| 8 | 19 | 70-80 |
| 10 | 12 | 55 |
| 20 | 8 | 28 |
| 23 | 5-6 |
Tabela 2: as mobilidades aproximadas do gel de corantes de carga de gel de bromofenol azul (BB) e xileno cianol (XC) em géis de desnaturação de poliacrilamida.
| Porcentagem | VOLUME DO GEL | |||
| 20 mL de | de 30 mL | 50 mL | ||
| 5 | 1X TBE | 16,5 | 24,75 | 41,25 |
| 40% 29:1 acrilamida: N, n'-methylenebisacrylamide | 2,5 | 3,75 | 6,25 | |
| Glicerol | 1 | 1,5 | 2,5 | |
| 6 | 1X TBE | 16 | 24 | 40 |
| 40% 29:1 acrilamida: N, n'-methylenebisacrylamide | 3 | 4,5 | 7,5 | |
| Glicerol | 1 | 1,5 | 2,5 | |
| 8 | 1X TBE | 15 | 22,5 | 37,5 |
| 40% 29:1 acrilamida: N, n'-methylenebisacrylamide | 4 | 6 | 10 | |
| Glicerol | 1 | 1,5 | 2,5 | |
| 10 | 1X TBE | 14 | 21 | 35 |
| 40% 29:1 acrilamida: N, n'-methylenebisacrylamide | 5 | 7,5 | 12,5 | |
| Glicerol | 1 | 1,5 | 2,5 | |
| 12 | 1X TBE | 13 | 19,5 | 32,5 |
| 40% 29:1 acrilamida: N, n'-methylenebisacrylamide | 6 | 9 | 15 | |
| Glicerol | 1 | 1,5 | 2,5 | |
| 15 | 1X TBE | 11,5 | 17,25 | 28,75 |
| 40% 29:1 acrilamida: N, n'-methylenebisacrylamide | 7,5 | 11,25 | 18,75 | |
| Glicerol | 1 | 1,5 | 2,5 | |
| 20 | 1X TBE | 9 | 13,5 | 22,5 |
| 40% 29:1 acrilamida: N, n'-methylenebisacrylamide | 10 | 15 | 25 | |
| Glicerol | 1 | 1,5 | 2,5 | |
| APS (μL) | 48 | 72 | 120 | |
| TEMED (μL) | 20 | 30 | 50 | |
Tabela 3: tabela de fundição de gel de página nativa.
Discussão
Uma vantagem significativa da tag fluorescente Mango é que uma única tag pode ser usada de várias maneiras. O brilho elevado e a afinidade destes aptâmeros fazem-nos úteis não somente para na visualização da pilha2 mas igualmente para in vitro o RNA ou a purificação4de RNP. Conseqüentemente, a imagem latente do gel estende a versatilidade da etiqueta da manga em uma maneira direta. A sensibilidade da imagem latente do gel da manga é ligeiramente menos do que aquela de um borrão do Norte14 mas pode facilmente detectar 60 – 120 fmol da amostra do RNA, sem precisar a transferência longa e tediosa da membrana e etapas de sondagem. Isso é comparável à eficiência de sondagem baseada em hibridação encontrada anteriormente para pequenas RNAs em gel15. Enquanto outras metodologias de aptâmeros fluorogênicas — particularmente, RNA espinafre — têm maior sensibilidade e especificidade9, nenhum atualmente tem simultaneamente o alto brilho e afinidade do sistema de aptâmeros Mango, que permite que uma única tag RNA seja usado para a imagem latente celular, purificação de RNP, e agora imagem latente do gel.
Há alguns passos críticos neste protocolo de coloração de gel. Ao trabalhar com soluções de RNA, as soluções devem ser filtradas estéreis e plasticware de uso único devem ser usados. Catuion, rodando géis nativos como complexos ou estruturas de RNA pode ser facilmente desnaturado se os níveis de potência para o gel são muito altos e resultam em aquecimento de gel. Assegure-se de que qualquer vidro usado esteja limpo e não contaminado com RNases. Além disso, sempre tenha cuidado ao transferir e pegar géis como eles são frágeis e pode ser propenso a ruptura.
A mancha TO1-biotina penetra géis rapidamente, mas os dados aqui apresentados também indicam que o dobramento de Mango IV, em particular, pode ser limitante de taxa (Figura 2 e Figura 3). Utilizando as condições indicadas na seção protocolo, observamos comportamento log-linear para todos os quatro aptâmeros em duas ordens de magnitude em géis de desnaturação, tornando o método útil para quantificação (Figura 4C, D). Desde que os aptâmeros pequenos da manga usaram-se neste estudo facilmente difunde fora da matriz do gel, nós esperamos que a quantificação melhore para construções mais longas do RNA.
A metodologia de imagem gel tag Mango demonstrada aqui é robusta e é esperada para ser capaz de ser simplesmente estendido em termos de sensibilidade e especificidade. Mango I, II, e III dobrar de forma confiável, enquanto Mango IV não. Embora não tenhamos explorado os protocolos de destulação, prevemos que tal abordagem poderia também simplesmente melhorar a especificidade. Embora além do escopo deste trabalho, a fluorescência e a marca de biotina conferidas ao RNA com a tag Mango ao usar o fluoróforo de TO1-biotina parece altamente provável para agilizar ainda mais a análise e a purificação do gel. Técnicas secundárias comercialmente disponíveis da biotina-rotulagem, por exemplo, prometem realçar mais os limites da deteção deste sistema simples da Mango-etiquetagem do RNA. Da mesma forma, parece provável que os complexos de proteína de RNA com marcas de manga nativas possam ser eluídos de um gel e recuperados usando grânulos magnéticos de estreptavidina para capturar o complexo de RNA eluída. Isto simplificaria ainda mais a purificação rotineira de complexos de RNAs e RNA biologicamente importantes pelo simples expediente de adicionar um tag Mango ao RNA de interesse.
Divulgações
A patente está pendente no sistema fluorogénico Mango.
Agradecimentos
Os autores agradecem Razvan Cojocaru e Amir Abdolahzadeh por sua assistência técnica e Lena Dolgosheina para revisar o manuscrito. Financiamento foi fornecido para este projeto por um Canadian natural Sciences and Engineering Research Council (NSERC) concessão de operação para P.J.U.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.8mm Thick Comb 14 Wells for 30 mL PAGE gels | LabRepCo | 11956042 | |
| 101-1000 µL tips | Fisher | 02-707-511 | |
| 20-200 µL low retention tips | Fisher Scientific | 02-717-143 | |
| Acrylamide:N,N'-methylenebisacrylamide (40% 19:1) | Bioreagents | BP1406-1 | Acute toxicity |
| Acrylamide:N,N'-methylenebisacrylamide (40% 29:1) | Fisher | BP1408-1 | Acute toxicity |
| Agar | Anachemia | 02116-380 | |
| Aluminium backed TLC plate | Sigma-Aldrich | 1164840001 | |
| Amersham Imager 600 | GE Healthcare Lifesciences | 29083461 | |
| Ammonium Persulfate | Biorad | 161-0700 | Harmful |
| BL21 cells | NEB | C2527H | |
| Boric Acid | ACP | B-2940 | |
| Bromophenol Blue sodium salt | Sigma | B8026-25G | |
| Chloloform | ACP | C3300 | |
| Dithiothreitol | Sigma Aldrich Alcohols | D0632-5G | |
| DNase I | ThermoFisher | EN0525 | |
| EDTA Disodium Salt | ACP | E-4320 | |
| Ethanol | Commerial | P016EAAN | |
| Flat Gel Loading tips | Costar | CS004854 | |
| Formamide 99% | Alfa Aesar | A11076 | |
| Gel apparatus set with spacers and combs | LabRepCo | 41077017 | |
| Glass Dish with Plastic lid | Pyrex | 1122963 | Should be large enough to fit your gel piece |
| Glycerol | Anachemia | 43567-540 | |
| HCl | Anachemia | 464140468 | |
| ImageQuanTL | GE Healthcare Lifesciences | 29000605 | |
| IPTG | Invitrogen | 15529-019 | |
| KCl | ACP | P-2940 | |
| MgCl2 | Caledron | 4903-01 | |
| MgSO4 | Sigma-Aldrich | M3409 | |
| NaCl | ACP | S-2830 | |
| NaOH | BDH | BDH9292 | |
| Orbital Rotator | Lab-Line | ||
| Phenol | Invitrogen | 15513-039 | |
| Round Gel Loading tips | Costar | CS004853 | |
| Sodium Phosphate dibasic | Caledron | 8120-1 | |
| Sodium Phosphate monobasic | Caledron | 8180-01 | |
| SYBRGold | ThermoFisher | S11494 | |
| T7 RNA Polymerase | ABM | E041 | |
| TEMED | Sigma-Aldrich | T7024-50 ml | |
| TO1-3PEG-Biotin Fluorophore | ABM | G955 | |
| Tris Base | Fisher | BP152-500 | |
| Tryptone | Fisher | BP1421-500 | |
| Tween-20 | Sigma | P9496-100 | |
| Urea | Fisher | U15-3 | |
| Xylene Cyanol | Sigma | X4126-10G | |
| Yeast Extract | Bioshop | YEX401.500 |
Referências
- Dolgosheina, E. V., et al. RNA Mango Aptamer-Fluorophore: A Bright, High-Affinity Complex for RNA Labeling and Tracking. ACS Chemical Biology. , (2014).
- Autour, A., et al. Fluorogenic RNA Mango aptamers for imaging small non-coding RNAs in mammalian cells. Nature Communications. 9, 656(2018).
- Dolgosheina, E. V., Unrau, P. J. Fluorophore-binding RNA aptamers and their applications: Fluorophore-binding RNA aptamers. Wiley Interdisciplinary Reviews: RNA. , (2016).
- Panchapakesan, S. S., et al. Ribonucleoprotein Purification and Characterization using RNA Mango. RNA. , 1592-1599 (2017).
- Trachman, R. J. III, et al. Structural basis for high-affinity fluorophore binding and activation by RNA Mango. Nature Chemical Biology. 13, 807(2017).
- Trachman, R. J., et al. Crystal Structures of the Mango-II RNA Aptamer Reveal Heterogeneous Fluorophore Binding and Guide Engineering of Variants with Improved Selectivity and Brightness. Biochemistry. 57, 3544-3548 (2018).
- Trachman, R., et al. Mango-III is a compact fluorogenic RNA aptamer of unusual structural complexity. Nature Chemical Biology. , in review (2018).
- Panchapakesan, S. S. S., Jeng, S. C. Y., Unrau, P. J. RNA complex purification using high-affinity fluorescent RNA aptamer tags. Annals of the New York Academy of Sciences. , (2015).
- Filonov, G. S., Kam, C. W., Song, W., Jaffrey, S. R. In-gel imaging of RNA processing using Broccoli reveals optimal aptamer expression strategies. Chemistry & Biology. 22, 649-660 (2015).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic Acids Research. 15, 8783-8798 (1987).
- A Typical DNase I Reaction Protocol (M0303). NEB. , Available from: https://www.neb.com/protocols/1/01/01/a-typical-dnase-i-reaction-protocol-m0303 (2018).
- Sambrook, J., Russell, D. W. Purification of Nucleic Acids by Extraction with Phenol:Chloroform. Cold Spring Harbor Protocols. 2006 (1), pdb.prot4455 (2006).
- Tuma, R. S., et al. Characterization of SYBR Gold Nucleic Acid Gel Stain: A Dye Optimized for Use with 300-nm Ultraviolet Transilluminators. Analytical Biochemistry. 268, 278-288 (1999).
- Streit, S., Michalski, C. W., Erkan, M., Kleeff, J., Northern Friess, H. Northern blot analysis for detection and quantification of RNA in pancreatic cancer cells and tissues. Nature Protocols. 4, 37-43 (2009).
- Ebhardt, H. A., Unrau, P. J. Characterizing multiple exogenous and endogenous small RNA populations in parallel with subfemtomolar sensitivity using a streptavidin gel-shift assay. RNA. 15, 724-731 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados