Method Article
ポスト染色法によるポリアクリルアミドゲル中のマンゴータグRNAの蛍光可視化
要約
ここでは、RNAマンゴーアプタマーI、II、III、またはIVでタグ付けされたRNAを画像化する敏感で迅速かつ判別的なポストゲル染色法を、天然または未消化ポリアクリルアミドゲル電気泳動(PAGE)ゲルを用いて提示する。標準的なPAGEゲルを実行した後、マンゴータグ付きRNAはTO1-Biotinで容易に染色し、一般的に利用可能な蛍光リーダーを使用して分析することができます。
要約
天然および変性ポリアクリルアミドゲルは、リボヌクレオプロテイン(RNP)複合体移動性を特徴付け、それぞれRNAサイズを測定するために日常的に使用されます。多くのゲルイメージング技術は、非特異的な染色や高価な蛍光色素プローブを使用するため、敏感、判別、および経済的なゲルイメージング方法論が非常に望ましい。RNAマンゴーコア配列は、任意のRNAステムによって閉じた場合、単にかつ安価に目的とするRNAに付加することができる小さな(19-22 nt)配列モチーフである。これらのマンゴータグは、TO1-Biotinと呼ばれるチアゾールオレンジ蛍光リガンドに高い親和性と特異性を結合し、結合時に数千倍の蛍光になります。ここで、マンゴーI、II、III、およびIVは、高感度のゲル中のRNAを特異的に画像化するために使用できることを示す。天然ゲル中のRNAの62.5 fmolと、入型ゲル中のRNAの125 fmolは、カリウムを含む画像バッファーにゲルを浸漬し、20nM TO1-Biotinを30分間検出することができる。マンゴータグ付き6S細菌RNAを、全細菌RNAの複雑な混合物のコンテキストでイメージングすることにより、マンゴータグ付きシステムの特異性を実証する。
概要
マンゴーは、チアゾールオレンジ(TO1-Biotin、図1A)1、2、3の単純な誘導体に密結合する4つの小さな蛍光RNAアプタマーのセットからなるRNAタグ付けシステムです。.結合すると、このリガンドの蛍光は、特定のアプタマーに応じて1,000〜4,000倍に増加する。マンゴーIIIの高輝度は、強化された緑色蛍光タンパク質(eGFP)を超えるマンゴー系の高輝度と、RNAマンゴーアプタマーのナノモル結合親和性と組み合わせることで、RNAのイメージングと精製の両方に使用することができます。複合体2,4.
マンゴーI5、II6、およびIII7のX線構造は高分解能に決定されており、3つのアプタマーはすべてRNA四分円を利用してTO1-Biotin(図1B-D)を結合する。すべての3つのアプタマーの密集した中心は密集したアダプターのモチーフによって外的なRNA配列から隔離される。マンゴーIとIIは、柔軟なGNRAのようなループアダプタを使用して、マンゴーコアを任意のRNAデュプレックスに接続します(図1B,C)。対照的に、マンゴーIIIは、そのコアを任意のRNAらせん(図1D、紫色の残渣)に接続するために剛性トリプレックスモチーフを使用していますが、マンゴーIVの構造は現在知られていません。これらのアプタマーのリガンド結合コアは、これらのヘリカルアダプターによって外部RNA配列から分離されるので、それらはすべて単に様々なRNAに組み込むことができる可能性が高い。細菌6S調節RNA(マンゴーI)、酵母スプリセオソーム(マンゴーI)の成分、およびヒト5S RNA、U6 RNA、およびC/D scaRNA(マンゴーIIおよびIV)はすべて、この方法でうまくタグ付けされている2、8、多くであることを示唆している生物学的RNAは、RNAマンゴーアプタマー系を使用してタグ付けすることができます。
RNAの研究には、未生および天然ゲルが一般的に使用されます。転移ゲルは、RNAサイズまたはRNA処理を判断するためによく使用されますが、典型的には、北部のブロットの場合、例えば、画像を生成するためにいくつかの遅い、逐次的なステップを必要とします。RNAホウレンソウやブロッコリーなどの他のRNAフルオロゲンアプタマーはゲルイメージング9に成功していますが、これまでフルオロゲンアプタマー系はマンゴー系の高輝度と親和性を持たないため、かなりの関心を持っています。マンゴーのゲルイメージング能力を調べるために。本研究では、TO1-Biotin(それぞれ510nmおよび535nm)の励起波長がほとんどの蛍光に共通するeGFPチャネルでのイメージングに適しているので、RNAマンゴーシステムを単にゲルイメージングに拡張できるかどうか疑問に思いました。ゲルスキャンの器械使用。
ここで提示されるポストゲル染色プロトコルは、マンゴータグ付きRNA分子をネイティブおよび脱帰性ポリアクリルアミドゲル電気泳動(PAGE)ゲルで特異的に検出する迅速な方法を提供します。この染色方法は、カリウムおよびTO1-Biotinを含む緩衝液にゲルを浸すことを含む。RNAマンゴーアプタマーはG-四重化基基であり、カリウムはそのような構造を安定化するために必要とされる。最小限のマンゴーコードDNAテンプレートから転写されたRNA(プロトコルセクションを参照)を使用して、単純な染色プロトコルを使用して、ネイティブゲル中のRNAの62.5 fmolと脱変性ゲル中のRNAの125 fmolをわずかしか検出できません。一般的な非特異的核酸染色とは対照的に(材料の表を参照)、サンプル中にタグ付けされていない全高濃度のRNAが存在する場合でも、マンゴータグ付きRNAを明確に同定することができます。
プロトコル
1. 試薬の調製
-
TO1-ビオチンポスト染色液(ゲル染色液)
- Na2 HPO4の1Mの342 mLと1M NaH2 PO4の158mLを加えて25°CでpH 7.2で1Mリン酸バッファーの1Lを作る。適切なリン酸溶液を加えて50mMでpHを7.2に調整します。0.2 μmフィルターを使用した滅菌フィルターを使用し、溶液を室温でプラスチック製品に保存します。
- 5倍ゲル染色液(TO1-Biotinを除く)を以下のように調製する。ddH2Oの247.5 mL、1M KClの700mL、1Mリン酸バッファーの50mL(pH 7.2)、および2.5mLのTween 20を混合して1L溶液を構成する。0.2 μmフィルターを使用した滅菌フィルターを使用し、溶液をプラスチック製品に保存します。室温で保存できます。
注:MgCl2は、RNA複合体を安定化させる可能性があるため、必要に応じてこのソリューションに添加することができます。これは、蛍光シグナル2に適度な影響を与えます。 - ステップ1.1.2から5倍ゲル染色液を使用して1xゲル染色溶液を構成し、使用直前にTO1-Biotin蛍油体(材料の表を参照)で20nMの最終濃度に補足します。例えば、脱イオン水の80mLに5倍ゲル染色液の20mLを加え、1xゲル染色液の100mLを作り、1mM TO1-Biotinストックの2μLを添加する(ジメチルホルミドで調製)。
注:500nmのTO1-ビオチン色素の絶滅係数は63,000M-1〜cm-1であり、染色液1で測定した。
-
経行性ページ(2x伝電ゲル負荷溶液および溶液A、B、C、ツル酸アンモニウム、テトラメチレチレンジアミアミン)
- 40mLのホルムアミド、0.5Mの0.5mLの0.5mLの0.5mL(EDTA、pH 8.0)、および9.5mL ddH2Oを混合して2倍の再帰性ゲル負荷溶液の50mLを調出す。溶液は室温で貯えることができる。この溶液には蛍光イメージングが不明瞭になる可能性があるため、読み込み染料は添加されません。
- 2x脱帰ゲル装填剤を以下のように調剤する。RNAおよびDNAオリゴヌクレオチドを精製する場合は、2.5%(w/v)ブロモフェノールブルー(BB)の0.5mLおよび0.5mLの2.5%(w/v)キシレンシアノール(XC)をステップ1.2.1に記載の溶液に加え、9.5MLの代わりにddH2Oの8.5mLを添加する。溶液は室温で貯えることができる。
- 尿素200.2g(式重量:60.06g/mol)を計量し、40%19:1アクリルアミドの312.5 mLに添加することにより、溶液Aの100 mLを調製する:N,N'-メチレンビサクリアミド。溶かされるまで最大速度(約3時間)で磁気攪拌バーで攪拌し、ddH2O.を使用して500 mLに溶液を構成することに注意してください 6.667 M尿素と25%19:1アクリルアミド:N,N'-メチレンビサクリアミド.清潔なプラスチック製品で4°Cに保管してください。
- 尿素の400.4gを計量して溶液Bの1Lを調べ、ddH2 Oで1Lに溶液を構成する。尿素が溶け出すまでかき混ぜる。溶液Bは室温で保つことができる。
- 溶液C(10xトリスボレート-EDTA[TBE])を以下のように調製する。トリスベースの432gと220gのホウ酸を計量し、0.5M EDTAの160mLをpH8(濾過)で加え、ddH2Oで4Lに溶液を作り上げ、10倍TBEの4Lを調製する。このバッファの大きなストックは、ゲル実行バッファとしても使用されるので作られます。
- 1gのAPSを4°CでddH2 O.ストアの総体積10mLに溶解することにより、10%のツル酸アンモニウム(APS)を調作する。
- テトラメチルチレンジアンミン(TEMED)を手元に置いてください。APS溶液と一緒に4°Cに保管してください。
-
ネイティブページ(2xネイティブゲルローディングソリューション)
- 100%グリセロールの25mL、5倍ゲル染色液の10mL、およびddH2Oの15mLを混合して2x天然ゲル負荷溶液の50 mLを調作し、50mLに溶液を構成する。
注:変色ゲルセクションと同様に、このストックにローディング染料を添加することができますが、その添加はゲル中の蛍光シグナルを不明瞭にする可能性があるため、避ける必要があります。
- 100%グリセロールの25mL、5倍ゲル染色液の10mL、およびddH2Oの15mLを混合して2x天然ゲル負荷溶液の50 mLを調作し、50mLに溶液を構成する。
2. 脱電ゲルの調製と装填
- 表1に従って、適切なパーセンテージで脱帰ゲルを調調します。特定のゲル鋳造システムを検討してください。30 mLゲルが一般的に使用されます。適切なゲルパーセンテージは、表2を使用して推定することができます: BB染料とXC染料が、目的のRNAよりも運動性が速く、より遅いポリアクリルアミド率を選択し、関連するサイズで高帯域分離を確保します。範囲。
- 表1に従って溶液A、B、Cを混合し、ゲルを注ぐ直前にAPSとTEMEDを加える。クリーンなプラスチック製品でよく溶液を混ぜます。
- すべての成分が綿密に洗浄されていることを確認した後、適切なゲル鋳造装置にゲル溶液を注ぎます。注ぐときにゲルがわずかに傾けるように装置の片側を持ち上げ、気泡がゲル自体の中に閉じ込められるのを避ける。
- 目的の櫛を挿入し、重合するためにそれを残します (約 30 分).ゲルウェルの周りの屈折率の変化を注意深く見て、重合を観察します。1x溶液C(1x TBE)を使用してゲルタンクを構成し、櫛を慎重に取り外します。注射器を使用して、サンプルロードの直前に井戸を吸引します。
- 次のように脱帰サンプルを調調します。目的のRNAサンプルに2x変性ゲルローディング液を添加し、サーモサイクラーまたは水浴を使用して95°Cで変性を1倍にし、95°Cで熱変性を作ります。
- サンプルをロードする前に、彼らはタッチに冷却されるまで、それらを数分冷やします。ゲルローディングのヒントを使用して、各ウェルの底にそれらを重ねてサンプルをロードします。
注:ラウンドチップは、1ミリメートル以上の厚さのゲルに使用することができます。薄いゲルには平らな先端を使用してください。 - 変電ゲルを室温で実行します。ゲルシステムのガラス板が割れないように、ワット数が十分に低いことを確認します。
注:私たちの研究室では、20センチメートルx 16センチメートルプレートのための28 Wは、この目的のために十分でした。
3. 天然ゲルの調製と装填
- 表 3を参照して、適切なパーセンテージでネイティブゲルを準備します。特定のゲル鋳造システムを検討してくださいが、30 mLゲルが一般的に使用されていることを心に留めておいてください。表3に従って1x TBE、40%29:1アクリルアミドの溶液を混合し、表3に従ってグリセロールを、ゲルを注ぐ直前にAPSとTEMEDを追加します。手順 2.3 および 2.4 に説明するように進みます。
- 目的のRNAサンプルに2xネイティブゲルローディング溶液を加えてネイティブサンプルを調べ、溶液を1倍にし、ゲルを実行する前に室温で100分間インキュベートして完全なRNA折りたたみを保証します。
- 4°Cのコールドルームでネイティブゲルを実行し、ゲルを加熱しないようにワット数が十分に低くなることを確認します。
注:私たちの研究室では、20センチメートルx 16センチメートルプレートのための14 Wは、この目的のために十分でした。
4. ランオフT7転写によるRNA調製
注:RNAマンゴー構造体のランオフ転写10に使用されるDNA配列は、商業的に発注された。この方法では、転写する配列とT7プロモーターの両方の逆補体(RC)を含むDNAオリゴヌクレオチドが、T7プロモーター上鎖配列にハイブリダイズされ、次いでインビトロで転写される。以下、オリゴヌクレオチド毎に、マンゴーコア配列のRCを太字で示し、T7プロモーター領域のRCを斜体で示す。通常のフォントの残渣は、マンゴーコアが適切に折り畳まれるのに必要な任意の補完ヘリカル領域に対応します。
マンゴーI:GCA CGT ACT CTC CTC TC GC GG CTG CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT CTT
マンゴーII:GCA CGT ACT CTC CTC CtC TCC TCT TCT TCT CC CC CC CC CT CT CT CT CT CT CT CT CT CT CT CT CT CT CT CT CT CT CT CT CT CT CT CGT GT TGC TGC CTA タグ TGA GTC GTA タッタ AAG
マンゴーIII:GGC ACG TAC GA A AT ACC ACA TAC CAA TCC TTC CTT CG TGC TGC CTA タグ TGA GTC GTA タッタ AAG
マンゴーIV:GCA CGT ACT CGC CTC CTC ACC アクト CCC TCG GTA GTA GCC TAT AGT AGT TCG TAT TAA AG
T7トップストランド:CTT TAA TAC GAC TCA CTA タグ G
- DNAオリゴヌクレオチドの予備ゲル精製
- 0.2 μmolスケールのDNA合成の場合、dH2 Oの100 μLおよび2x変性荷ロイヤの100 μL(ステップ1.2.2から)で脱保護DNAオリゴヌクレオチドを再中断します。この場合、ステップ1.2.2と同濃度の装填液中にゲル積載染料を含む方が好ましい。
- 適切なポリアクリルアミド率の50 mL予備スケール変性ゲルを用いてDNAオリゴヌクレオチドを精製する。表 2を使用してゲルのパーセンテージを選択します。たとえば、50 nt 配列に 8% のゲルを使用します。理想的には、ブロモフェノールブルーはオリゴヌクレオチドよりも速く走るべきであり、キシレンシアノールは遅く走るべきである。
注:私たちの研究室では、このようなゲルは、厚さ1.5ミリメートルの鋳造スペーサーと2〜2.5センチメートル幅の井戸を持つゲルローディング櫛を使用して鋳造されました。 - ステップ2.4-2.7で説明したように、工程4.1.2で調製したDNAオリゴヌクレオチド溶液の100 μLを調製します。
- 慎重にゲルの外側を乾燥させ、ガラス板を取り除き、ゲルの両面をラップで覆います。蛍光イメージングスクリーン(蛍光色素を含浸したアルミニウム支持層クロマトグラフィープレートは経済的な溶液)の上に置き、短波長のUVハンドヘルドランプを使用してUVシャドウイングによってDNAバンドを可視化します。
- DNA合成が高品質の場合は、鮮明で明確に定義された影を観察する必要があります。パーマネントマーカーを使用して、ラップにバンドをマークします。
注:紫外線から皮膚や目を保護し、核酸サンプルを損傷しないように露出を短く保ちます。 - きれいなガラス板の上にゲルを置き、マークされたバンドを慎重に切り取り、各ゲル断片を300 mM NaClの400 μLに置きます。室温で回転子を使用して、一晩DNAを溶出させます。
- きれいな遠心管で溶出剤を回収し、2.5相当のエタノールを加えてDNAを沈殿させます。渦はよく、30分間-20 °Cにチューブを置きます。
- 4°Cで30分間156,000 x gのベンチトップ遠心分離機でサンプルをペレットします。上清を慎重に取り除き、ddH2O. 分光光度計を使用してDNAオリゴヌクレオチド濃度を正確に決定します。-20 °C.で便宜上、サンプルを10μMのストックとして保管してください。
- RNAサンプルのランオフ転写とゲル精製
- 40mMグアノシン三リン酸(GTP、11,400M -1~cm-1)、25mMシチジン三リン酸(CTP、7,600M-1 cm-1)の最終濃度で構成される液体NTPストックを混合して5xヌクレオシド三リン酸(NTP)ストックを調製アデノシン三リン酸 (ATP, 5,000 M-1·cm-1),および 10 mM ウリジン三リン酸 (UTP, 10,000 M-1·cm-1);260 nmですべての絶滅係数。
注:液体または粉末状の株式は、商業的に得ることができます。粉末から一次在庫を調製する場合は、1M NaOHを使用して、pHを7.9の最終pHに慎重に調整します。1.5mLマイクロ遠心管でストックをアリコートし、-20 °Cに保存します。 - 1Mトリス-HClの25.6mL、1Mトリスベースの14.4mL、1M MgCl2の26mL、10%トリトンX-100の10mL、および0.637gの精子を混合して10x T7転写バッファーストックの100mLを調製する。ddH2O を追加して、100 mL の最終ボリュームにストックを構成します。
注:1x溶液の7.9の最終的なpHは、校正されたpHメーターを使用して確認する必要があります。10xは無菌濾過され、適切なサイズのアリコートで-20°Cで保存する必要があります。 - 転写は以下の通り行う。
- 10x T7転写バッファーストックとddH2O(ステップ4.2.2)、1x NtP、10 mMのジチオスレイトール(DTT)、1μM T7トップストランドシーケンスを使用して、1x T7転写バッファーの最終濃度を構成するために、以下の試薬を追加します(セクション4注を参照してください。配列は、1 μMゲル精製マンゴーDNA配列(ステップ2.1)、およびT7 RNAポリメラーゼ酵素(1U/μL)である。
- 渦は、スピンダウンし、2時間またはそれが曇りになるまで、またはチューブの底部に白い沈殿物の形態になるまで37 °Cで溶液をインキュベートします。50 μL 転写は、ゲル精製後に約50 μLの50 μL RNAをもたらす必要があります。2xの脱熟色色物の等しい容積を追加し、ゲル精製の準備ができるまで-20 °Cに保存します。
- DNAオリゴヌクレオチドゲル精製部に記載されている得られたRNAを、次のステップ4.1.2\u20124.1.8に従って精製し、DNAのRNA試料を置換する。
注:RNAはRNaseの劣化に非常に敏感なので、すべてのステップで手袋と清潔なラボコートが常に着用されていることを確認してください。すべてのサンプルがRNase汚染から保護するために単一使用のプラスチック製品で準備され、保管されていることを確認し、使用前に熱い石鹸と水でガラス板を注意深く洗い、ddH2Oで十分に洗い流し、ガラス製品を乾燥させます。スクイーズボトルから適用されたエタノールの援助。 - 最終的なRNAサンプルの1 μLを使用し、液滴ベースの分光光度計を使用して、260 nmでの吸光度を決定します。最も近い近傍法によって決定された絶滅係数を使用して、RNA濃度を計算し、便宜上ddH2Oでサンプル濃度を10μMに調整します。RNAサンプルを-20°Cに保存します。
- 40mMグアノシン三リン酸(GTP、11,400M -1~cm-1)、25mMシチジン三リン酸(CTP、7,600M-1 cm-1)の最終濃度で構成される液体NTPストックを混合して5xヌクレオシド三リン酸(NTP)ストックを調製アデノシン三リン酸 (ATP, 5,000 M-1·cm-1),および 10 mM ウリジン三リン酸 (UTP, 10,000 M-1·cm-1);260 nmですべての絶滅係数。
- エシェリヒア・コリ総粗核酸抽出
注: 次のプロトコルは例です。このプロトコルは、大腸菌中のプラスミドから内因性に発現したマンゴーIタグ付き6S RNAを使用し、このプラスミドからの誘導は詳細4に他の場所で説明される。- 本研究の目的のために、ペコli-RNAマンゴーとペコli-T1プラスミドの両方に対して500mLの誘導細胞を調製する(細胞は1のOD600nmで誘導される)。細胞をペレットにし、使用前に-80°Cで保存してください。
- 次のようにRNA抽出を行う。
- 誘導ペコリ細胞ペレットの0.5 mLを取り、平衡化フェノールの500 μLを追加します。次に、サンプルを渦に入れ、溶液は乳白色になります。層を分離するために2分間の最高速度で遠心分離。
- わずかに黄色になる最上層を抽出し、フェノールの同じ体積を追加して、遠心分離し、抽出を合計5倍(または不透明な白色の中間層が消えるまで、2つの明確な層だけが残っている)によってステップ4.3.2.1を繰り返します。
- フェノール抽出水量をクロロホルム、渦、および最大速度で2分間遠心分離機の等量に加えます。
- 水層を抽出し、300 mMの最終濃度にNaClを追加します。得られた核酸を、エタノール、渦、スピンダウン、-20°Cの2.5等価物を添加して少なくとも30分間沈殿させる。
- ベンチトップ遠心分離機のペレットを15.6 x 1,000 x gで4°Cで30分間慎重に取り出し、ddH2Oの100 μLでペレットを再ステージェントします。
- この手順にDNase I消化ステップを追加し、参照プロトコル11に従って、全RNAを得る。
注:ペレットがddH2 Oに完全に溶解するまで、渦が激しく溶解する。
5. ポストゲル染色
- ステップ1.1.3のレシピに応じた1xゲル染色液を準備し、ゲルに快適にフィットするのに十分な広さのあるきれいなガラス容器に追加します。
注:スナップフィット蓋が付いているホウケイ酸塩ガラス容器は、この目的によく役立ちます。 - 容器に十分な1xゲル染色液を加え、ゲルが溶液で完全に覆われ、液体が軌道回転子に置かれたときにゲルの上にスラッシュします。
注:容器はゲルを動かすのに十分な大きさでなければならず、容器内に十分なバッファーがゲルを完全に覆う必要があります。 - ネイティブゲルまたは変化ゲル(セクション2または3)が実行を終了したら、装置からゲルを取り出し、その井戸を切り取ります。解析の後半で向きのゲルの角を取り除くのに役立ちます。
- ステップ5.2で調製した1xゲル染色溶液にゲルを慎重に移します。
注:ゲルは壊れやすく、壊れやすいので、ゲルを移す際には注意が必要です。ゲルが汚染しないように、常に染色容器の蓋を保管してください。 - ゲルを100rpmの速度で、室温で30分間回転します。
注: ゲルがそれ自体に折り返されないようにしてください。それ以外の場合、RNAはゲルから拡散し、ゲルの別の部分にラベルを付けます。
6. ゲル中のマンゴータグ付きRNAのイメージング
- イメージング バッファを慎重にデカントします。ゲルを水で素早くすすいで、容器内でゲルをわずかに移動させ続けるのに十分な液体が残っていることを確認します。
- ゲルをイメージャーに慎重に移します。慎重にゲルの側面をピックアップし、イメージャートレイに置くことによって転送します。または、ゲルをトレイにゆっくりと注ぐことができます。ゲルの下に気泡がなく、ゲルの下に余分な液体がないことを確認してください。ゲルの上に転がされたピペットは、余分な液体を除去するのに有用でありうる。
注:余分な水分を浸すためにペーパータオルを使用してください。 - ゲル画像を撮り、励起波長510nmと発光波長535nmとの間の蛍光を観察する(例えば、イメージャー上の520nm波長蛍光設定における緑色光)。設定が計測器で完全に最適化されていることを確認し、使用する機器の指示に注意深く従ってください。
結果
短いマンゴータグ付きRNAは、プロトコルセクションで説明されているように調製されました。ゲル中の尿素の存在により、回帰条件での蛍光が最も観察しにくいと仮定し、まず核酸の脱窒剤として作用する尿素に対するマンゴーアプタマーの耐性を検討した。マンゴーアプタマーは、約1M(図2A)の尿素濃度までの脱彩に対して実質的に耐性があることがわかった。経常ゲルにゲル染色液を添加する前に、ゲル中の尿素の最終濃度は6Mであるこの濃度を1Mに減少させる十分な染色液を加えることは、したがって、4つのアプタマーすべてに対して完全なマンゴー蛍光を確保するのに最適であろう。実際には、染色プロトコルは、この完全に最適な結果よりも少ない達成したが、これは単に、より多くの染色溶液を使用するか、染色中に一度溶液を変更する単純な便宜によって、必要に応じて修正することができる。
マンゴータグ付きRNA構築物が変性ゲルに実行されると、染色時間は、3つの異なるマンゴーIII量を8%の変性ゲルにロードすることにより、最大ゲル蛍光のために最適化されました(図2B)。時間経過の上で、ゲル染色液に5分浸漬した後、蛍光がはっきりと見えることを明らかにした。マンゴーIII構築物の最大蛍光は、染色の20-40分後に得られ、その後、この研究で使用された小さなRNAがゲルから拡散し始め、蛍光シグナルの損失を引き起こした(図2B)。その結果、天然ゲルと脱原ゲルの両方をそれぞれ30分間染色した。より長いRNA構築物は、ゲルから拡散する可能性がはるかに低いので、より長い染色時間を容易に許容できる。
RNAマンゴーアプタマーのいくつかは、他のものよりも速く折りたたまれた。本研究で用いたマンゴーアプタマーのそれぞれを、1.5M尿素および100nM TO1-Biotin染料を補うゲルバッファーでインキュベートし、蛍光計を用いて分析した。マンゴーI、II、IIIは10分後に完全に折りたたまれたのに対し、マンゴーIVは40分後に実質的に折りたたまれた(図3A)。尿素が存在しない場合、折りたたみは予想通りはるかに速かった(図3B)。天然ゲルサンプルが完全に折りたたまれたことを確認するために、天然ゲルに実行する前に100分間サンプルをプリインキュベートしました。実際には、図3のデータは、使用するマンゴー アプタマーに応じて、この時間を大幅に削減できることを示唆しています。
プロトコルがRNAマンゴーアプタマー蛍光を検出するように最適化されると、ポスト染色法の感度は、両方の天然ゲルのマンゴー変異体ごとに決定された。よく折り畳まれたRNAに対応する単一バンドは、天然ゲル中の4つのマンゴーのそれぞれについて観察された(図4A)。連続希釈時には、マンゴーIIの62.5fmolがわずか62.5fmolで観察でき、マンゴーI、III、IVの125fmolは容易に可視化された。天然ゲルの定量化は、マンゴーIII(図4B)よりも直線的に振る舞うマンゴーI、II、IVで、約1.5桁の大きさで対数線形であった。
伝性ゲルの結果は、天然ゲルよりもわずかに感受性が低かったが、より線形であった。マンゴーIIとIIIの125fmolは、簡単に検出されました(図4C)。興味深いことに、定量化(図4D)は、非連続ゲルが対数線形または2桁の大きさであることを示した。我々は、RNAの折り目がゲル走行プロセス中に部分的な転向を受けたネイティブゲルとは対照的に、転移ゲル中の尿素の存在は、アプタマーを一度に折りたたむより均質な方法を提供するかもしれない、と仮定する。TO1-ビオチン染色溶液。
すべてのゲル染色方法と同様に、ゲルが慎重に容器に移されていない場合、または染色期間中に回転速度が高すぎると、ゲルがそれ自体に折り返してもよい(図5A)。これにより、マンゴータグ付きRNAサンプルがゲルの1つの場所から別の場所に拡散する可能性がありますが、実際には簡単に回避できます。ネイティブゲル、特にマンゴーIVでは、折りたたみ時間の短縮に起因する部分的/誤折されたRNA適合(図5B)に対応すると思われる複数のバンドの出現で不完全な折りたたみが現れることが観察されました。ネイティブゲルの折りたたみ問題は、前述のようにRNAサンプルを適切に事前に分け、コールドルームでネイティブゲルを実行することで回避できます。RNAがゲル内で折り畳まれる脱電性ゲルでは、誤折は大きな問題ではなかった。最後に、RNAがない場合、いずれのゲル系においてもバックグラウンド蛍光はほとんど認められなかった。
次に、RNAマンゴータグの特異性を、細菌中の6S調節RNAを過剰発現させることにより検討した。このRNAは以前、マンゴーI(図1E)4を使用してタグ付けされていた。細菌細胞は、ペコli-RNAマンゴープラスミド(以降Mプラスミド)またはペコli-T1プラスミドを陰性対照(以降Eプラスミド)で形質転換した。形質転換細胞は、1.0のOD600に達するまで液体リソジェニーブロス培地で増殖した。次いで、培養物を50μMイソプロピルβ-D-1-チオガラクトピラノシド(IPTG)で40分間誘導した。細胞を6,000xgで15分間遠心分離して採取し、プロトコルセクションに記載されているように細胞ペレット12からフェノールフェノールクロロホルム抽出を用いて抽出した。全RNA試料をエタノール沈殿により濃縮し、次いでDNase Iで処理し、プロトコルに従って、DNA11を除去した。使用する前に、RNAをエタノール沈殿により濃縮した。
全細菌RNAを8%変性ゲル(図6)に駆除し、SG13またはTO1-Biotinのいずれかで染色した。SG染色の場合、10,000x SGの10μLをゲルバッファーの100mLに添加した。それ以外の場合、染色プロトコルはTO1-Biotinに使用されるものと同じであった。予想通り、多数のRNAに対して強いSG染色が認められたが、リボソーム(rRNA)および転写RNA(tRNA)に対して最も顕著であった(図6、左パネル)。マンゴー依存染色(Mレーン)は、これらのSG染色ゲルで見ることができましたが、この普遍的な染色を用いて観察された複雑な染色パターンを考えると、一意に同定することはできませんでした。対照的に、TO1-Biotin染色ゲルは、マンゴー依存バンドを最も顕著なバンドとして強調した。リボソームRNAバンドのみが6Sマンゴー依存バンドと競合していると見られる。多くの非特異的に染色されたバンドも観察された。それにもかかわらず、マンゴー依存バンドは再び支配的であり、rRNAとtRNAバンドだけが弱い競争相手として競合している(図6、右パネル)。

図1:マンゴーアプタマーシステム。(A) TO1-ビオチン蛍動体(B) マンゴー I. (C) マンゴー II.(D) マンゴーIII。パネルB–Dは、各アプタマーの二次構造を示す。P1は任意の茎である。マンゴーIとIIで見つかったGNRAのようなステムループ(ここではGAAA)は赤で示され、マンゴーIIIのトリプレックスモチーフは紫色で示されています。(E) マンゴーIでタグ付けされた6S調節RNAは、こちらをクリックして、この図の大きなバージョンを表示してください。
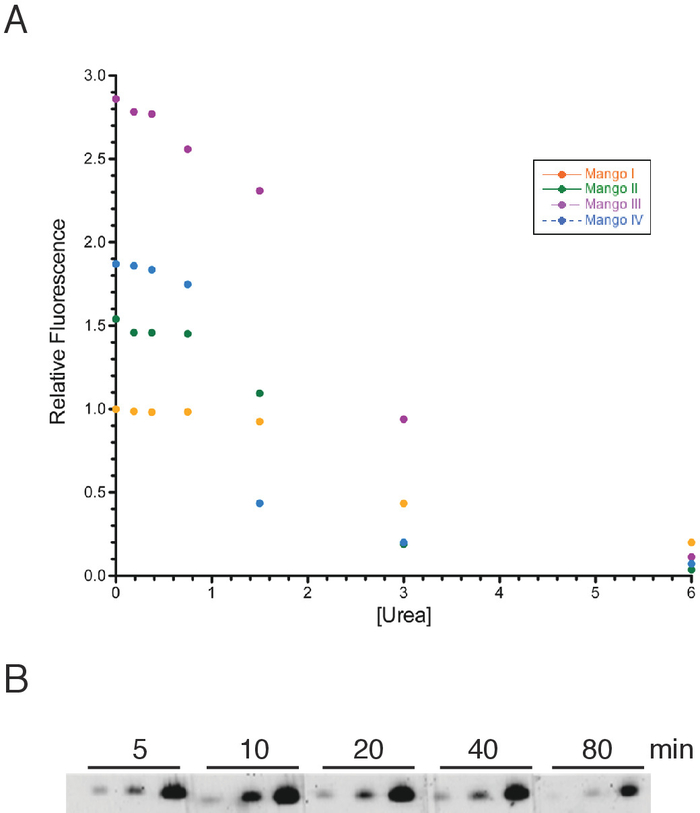
図2:マンゴーアプタマー蛍光および最適染色時間に対する尿素の影響(A) 50 nM RNAマンゴーI(オレンジ色の円)、マンゴーII(緑色の円)、マンゴーIII(紫色の円)、マンゴーIV(青い円)を用いて尿素滴滴定を行い、100nM TO1-Biotin染料および示された尿素濃度を用いて。サンプルを40分間インキュベートし、510nmの励起波長と535nmの発光波長で読み取った。(B)マンゴーIII RNAを8%変性ゲルに装填し、20nM最終TO1-ビオチン色素を含むゲル溶液で染色した。示された各時点について、マンゴーIII RNAの0.064、0.32、および1.6pmolを左から右に使用した。ゲル画像を520nmレーザーと10分露光を用いて蛍光イメージャーで可視化した。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図3:尿素の有無におけるマンゴーアプタマーの折りたたみ時間。(A) 1.5M尿素及び100nM TO1-Biotinの存在下で、各RNAマンゴー構築物(RNAマンゴーI:オレンジドット、マンゴーII:緑の点、マンゴーIII:紫のドット、マンゴーIV:青ドット)の50nMを用いて蛍光時間コースを実施した。(B) 尿素がない場合を除き、パネルAと同一である。 すべての時間コースは室温で行われました。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図4:RNAマンゴー構造を有する原発性および脱帰性PAGEの蛍光画像。(A) 連続的に希釈されたRNAマンゴー構造を持つ8%の天然ゲル。レーンI、II、III、およびIVはそれぞれ、それぞれ8pmolの最終量のRNAマンゴーI、II、III、およびIVを含む。右側のパネルは、4 pmol、2 pmol、1 pmol、0.5 pmol、0.25 pmol、0.125 pmol、および 0.0625 pmol のいずれかのマンゴー I、II、III、または IV を含む 2 つの連続希釈です。レーン12にはRNAが含じっていない。(B)天然ゲルの3回の反復の定量(それぞれに示す平均の標準偏差)。(C) パネルAと同じ試料を搭載した8%の回電ゲルは、天然ゲルローディング溶液の代わりに脱電ゲルローディング溶液が使用されたという事実を除いて。(D)脱退性ゲルの3つの定量複製(それぞれに示す平均の標準偏差)。全てのゲル画像を520nmレーザーと10分露光でゲルイメージャーを可視化した。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図5:8%天然ゲル中のマンゴーIVの最適でないゲルおよび不完全な折りたたみ。(A) マンゴーIIに対して図4に示す並例希釈であるが、染色プロトコル中のゲル折りたたみの効果を示す。(B)ゲル負荷前にネイティブバッファーで十分に長く折りたたむことが許されなかったマンゴーIV天然ゲル試料は、二重バンドを示す。それ以外の場合、これらの結果は図 4Aに示すマンゴー IV の結果に似ています。すべてのゲル画像は、520 nmレーザーと10分の露光を用いたゲルイメージャーを用いて可視化した。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図6:MANGOタグ付きRNAは、TO1-Biotin染色を用いて、全RNAの存在下で検出することができる。8%の回電ゲルを全RNAの100ngをロードし、30分間実行した。左ゲルをSGで染色し、右パネルをTO1-Biotinで染色した。どちらのパネルでも、Eというラベルの付いたレーンは100ngのペコリ-T1(マンゴータグなし)をロードし、Mというラベルの付いたレーンは100ngのペコリRNAマンゴー(6S RNAはマンゴーIタグでタグ付けされた)でロードされました。TO1-Biotin染色ゲル画像は、520nmレーザーと10分露光のイメージャーを用いて可視化した。SG染色ゲル画像を460nmレーザーと10分露光を用いてゲルイメージャーを用いて可視化した。この図のより大きなバージョンを表示するには、ここをクリックしてください。
| 割合 | ゲル容積 | |||
| 20 mL | 30 mL | 50 mL | ||
| 5パーセント | A | 4 | 6 | 10歳 |
| B | 14歳 | 21歳 | 35歳 | |
| C | 2 | 3 | 5 | |
| 6パーセント | A | 4.8年 | 7.2年 | 12歳 |
| B | 13.2年 | 19.8年 | 33歳 | |
| C | 2 | 3 | 5 | |
| 8パーセント | A | 6.4年 | 9.6年 | 16歳 |
| B | 11.6年 | 17.4年 | 29歳 | |
| C | 2 | 3 | 5 | |
| 10パーセント | A | 8 | 12歳 | 20歳 |
| B | 10歳 | 15歳 | 25名 | |
| C | 2 | 3 | 5 | |
| 12パーセント | A | 9.6年 | 14.4年 | 24歳 |
| B | 8.4年 | 12.6年 | 21歳 | |
| C | 2 | 3 | 5 | |
| 15パーセント | A | 12歳 | 18歳 | 30歳 |
| B | 6 | 9 | 15歳 | |
| C | 2 | 3 | 5 | |
| 20パーセント | A | 16歳 | 24歳 | 40歳 |
| B | 2 | 3 | 5 | |
| C | 2 | 3 | 5 | |
| APS (μL) | 48歳 | 72歳 | 120の | |
| テメッド (μL) | 20歳 | 30歳 | 50歳 | |
表1:ページゲル鋳造テーブルを脱行する。A = 解次 A、B = ソリューション B、C = ソリューション C。
| 脱電ゲル % | BB (~モビリティ nt) | XC (nt のモビリティ) |
| 5 | 35歳 | 130の |
| 6 | 26歳 | 106の |
| 8 | 19歳 | 70から80 |
| 10歳 | 12歳 | 55歳 |
| 20歳 | 8 | 28歳 |
| 23歳 | 5-6日 |
表2:ポリアクリルアミド脱窒ゲル中のブロモフェノールブルー(BB)及びキシレンシアノール(XC)ゲル装填染料のおおよそのゲル動員。
| 割合 | ゲル容積 | |||
| 20 mL | 30 mL | 50 mL | ||
| 5パーセント | 1X TBE | 16.5年 | 24.75年 | 41.25 |
| 40% 29:1 アクリルアミド:N,N'-メチレンビサクリラム化物 | 2.5年 | 3.75 | 6.25 | |
| グリセロール | 1 | 1.5年 | 2.5年 | |
| 6パーセント | 1X TBE | 16歳 | 24歳 | 40歳 |
| 40% 29:1 アクリルアミド:N,N'-メチレンビサクリラム化物 | 3 | 4.5年 | 7.5年 | |
| グリセロール | 1 | 1.5年 | 2.5年 | |
| 8パーセント | 1X TBE | 15歳 | 22.5年 | 37.5歳 |
| 40% 29:1 アクリルアミド:N,N'-メチレンビサクリラム化物 | 4 | 6 | 10歳 | |
| グリセロール | 1 | 1.5年 | 2.5年 | |
| 10パーセント | 1X TBE | 14歳 | 21歳 | 35歳 |
| 40% 29:1 アクリルアミド:N,N'-メチレンビサクリラム化物 | 5 | 7.5年 | 12.5年 | |
| グリセロール | 1 | 1.5年 | 2.5年 | |
| 12パーセント | 1X TBE | 13歳 | 19.5年 | 32.5歳 |
| 40% 29:1 アクリルアミド:N,N'-メチレンビサクリラム化物 | 6 | 9 | 15歳 | |
| グリセロール | 1 | 1.5年 | 2.5年 | |
| 15パーセント | 1X TBE | 11.5年 | 17.25年 | 28.75円 |
| 40% 29:1 アクリルアミド:N,N'-メチレンビサクリラム化物 | 7.5年 | 11.25年 | 18.75円 | |
| グリセロール | 1 | 1.5年 | 2.5年 | |
| 20パーセント | 1X TBE | 9 | 13.5年 | 22.5年 |
| 40% 29:1 アクリルアミド:N,N'-メチレンビサクリラム化物 | 10歳 | 15歳 | 25名 | |
| グリセロール | 1 | 1.5年 | 2.5年 | |
| APS (μL) | 48歳 | 72歳 | 120の | |
| テメッド (μL) | 20歳 | 30歳 | 50歳 | |
表 3: ネイティブ PAGE ゲル鋳造テーブル。
ディスカッション
マンゴー蛍光タグの大きな利点は、1つのタグが複数の方法で使用できることです。これらのアプタマーの高輝度と親和性は、細胞可視化2だけでなく、インビトロRNAまたはRNP精製4にも有用です。従って、ゲルイメージ投射はマンゴーのタグの多様性を簡単な方法で拡張する。マンゴーゲルイメージング感度は、ノーザンブロット14のそれよりもわずかに小さいですが、長くて退屈な膜移動およびプローブステップを必要とせずに、RNAサンプルの60-120 fmolを簡単に検出できます。これは、ゲル15の小さなRNAに対して以前に見つかったハイブリダイゼーションベースのプローブ効率に匹敵します。他の蛍光性アプタマー法論(特にRNAほうれん草)は、より大きな感度と特異性を有するが、現在、マンゴーアプタマー系の高輝度と親和性を同時に持つものは存在しない。細胞イメージング、RNP精製、および現在ゲルイメージングに使用されます。
このゲル染色プロトコルにはいくつかの重要な手順があります。RNAソリューションを使用する場合、溶液は無菌濾過され、単一使用のプラスチック製品を使用する必要があります。Catuionは、複合体またはRNA構造として天然ゲルを実行すると、ゲルの電力レベルが高すぎると容易に変性し、ゲル加熱をもたらす可能性があります。使用済みのガラス製品が清潔で、RNasesに汚染されていないことを確認します。また、ゲルは壊れやすく、破損しやすいため、ゲルを移動して拾うときは常に注意してください。
TO1-Biotin染色はゲルに急速に浸透しますが、ここで示すデータは、マンゴーIVの折りたたみが、特にレート制限でありうることも示しています(図2および図3)。プロトコルセクションで述べた条件を用いて、脱電ゲルの2桁以上の4つのアプタマーすべてに対して対数線形挙動を観察し、定量化に有用な方法を作った(図4C,D)。本研究で用いられる小さなマンゴーアプタマーはゲルマトリックスから容易に拡散するので、より長いRNA構築物に対して定量が改善することが期待されます。
ここで示すマンゴータグゲルイメージング方法論は堅牢であり、感度と特異性の面で単純に拡張できることが期待されています。マンゴーI、II、IIIは確実に折りたたまれ、マンゴーIVは折りたたまない。デステインプロトコルについては説明していませんが、このようなアプローチは単に特異性を向上させる可能性もあると予想しています。本研究の範囲を超えて、TO1-Biotin蛍光色素を用いた際にマンゴータグRNAに付与された蛍光およびビオチンタグは、ゲル分析および精製をさらに合理化する可能性が高いと考えられる。市販の二次ビオチン標識技術は、例えば、この単純なRNAマンゴータグ付けシステムの検出限界をさらに高めることを約束する。同様に、マンゴータグ付きRNAタンパク質複合体をゲルからタンパク質から取り出し、レンサプタビジン磁気ビーズを用いて回収し、ELUTED RNA複合体を捕捉できる可能性が高いと思われます。これにより、目的のRNAにマンゴータグを追加するという単純な便宜により、生物学的に重要なRNAおよびRNA複合体のルーチン精製がさらに簡素化されます。
開示事項
マンゴーフルオロゲン系に関する特許が出願中です。
謝辞
著者らは、ラズヴァン・コジョカルとアミール・アブドラザデの技術支援とレナ・ドルゴシーナに原稿を校正してくれたことに感謝している。このプロジェクトの資金は、カナダ自然科学工学研究協議会(NSERC)がP.J.Uに対する運営助成金によって提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 0.8mm Thick Comb 14 Wells for 30 mL PAGE gels | LabRepCo | 11956042 | |
| 101-1000 µL tips | Fisher | 02-707-511 | |
| 20-200 µL low retention tips | Fisher Scientific | 02-717-143 | |
| Acrylamide:N,N'-methylenebisacrylamide (40% 19:1) | Bioreagents | BP1406-1 | Acute toxicity |
| Acrylamide:N,N'-methylenebisacrylamide (40% 29:1) | Fisher | BP1408-1 | Acute toxicity |
| Agar | Anachemia | 02116-380 | |
| Aluminium backed TLC plate | Sigma-Aldrich | 1164840001 | |
| Amersham Imager 600 | GE Healthcare Lifesciences | 29083461 | |
| Ammonium Persulfate | Biorad | 161-0700 | Harmful |
| BL21 cells | NEB | C2527H | |
| Boric Acid | ACP | B-2940 | |
| Bromophenol Blue sodium salt | Sigma | B8026-25G | |
| Chloloform | ACP | C3300 | |
| Dithiothreitol | Sigma Aldrich Alcohols | D0632-5G | |
| DNase I | ThermoFisher | EN0525 | |
| EDTA Disodium Salt | ACP | E-4320 | |
| Ethanol | Commerial | P016EAAN | |
| Flat Gel Loading tips | Costar | CS004854 | |
| Formamide 99% | Alfa Aesar | A11076 | |
| Gel apparatus set with spacers and combs | LabRepCo | 41077017 | |
| Glass Dish with Plastic lid | Pyrex | 1122963 | Should be large enough to fit your gel piece |
| Glycerol | Anachemia | 43567-540 | |
| HCl | Anachemia | 464140468 | |
| ImageQuanTL | GE Healthcare Lifesciences | 29000605 | |
| IPTG | Invitrogen | 15529-019 | |
| KCl | ACP | P-2940 | |
| MgCl2 | Caledron | 4903-01 | |
| MgSO4 | Sigma-Aldrich | M3409 | |
| NaCl | ACP | S-2830 | |
| NaOH | BDH | BDH9292 | |
| Orbital Rotator | Lab-Line | ||
| Phenol | Invitrogen | 15513-039 | |
| Round Gel Loading tips | Costar | CS004853 | |
| Sodium Phosphate dibasic | Caledron | 8120-1 | |
| Sodium Phosphate monobasic | Caledron | 8180-01 | |
| SYBRGold | ThermoFisher | S11494 | |
| T7 RNA Polymerase | ABM | E041 | |
| TEMED | Sigma-Aldrich | T7024-50 ml | |
| TO1-3PEG-Biotin Fluorophore | ABM | G955 | |
| Tris Base | Fisher | BP152-500 | |
| Tryptone | Fisher | BP1421-500 | |
| Tween-20 | Sigma | P9496-100 | |
| Urea | Fisher | U15-3 | |
| Xylene Cyanol | Sigma | X4126-10G | |
| Yeast Extract | Bioshop | YEX401.500 |
参考文献
- Dolgosheina, E. V., et al. RNA Mango Aptamer-Fluorophore: A Bright, High-Affinity Complex for RNA Labeling and Tracking. ACS Chemical Biology. , (2014).
- Autour, A., et al. Fluorogenic RNA Mango aptamers for imaging small non-coding RNAs in mammalian cells. Nature Communications. 9, 656(2018).
- Dolgosheina, E. V., Unrau, P. J. Fluorophore-binding RNA aptamers and their applications: Fluorophore-binding RNA aptamers. Wiley Interdisciplinary Reviews: RNA. , (2016).
- Panchapakesan, S. S., et al. Ribonucleoprotein Purification and Characterization using RNA Mango. RNA. , 1592-1599 (2017).
- Trachman, R. J. III, et al. Structural basis for high-affinity fluorophore binding and activation by RNA Mango. Nature Chemical Biology. 13, 807(2017).
- Trachman, R. J., et al. Crystal Structures of the Mango-II RNA Aptamer Reveal Heterogeneous Fluorophore Binding and Guide Engineering of Variants with Improved Selectivity and Brightness. Biochemistry. 57, 3544-3548 (2018).
- Trachman, R., et al. Mango-III is a compact fluorogenic RNA aptamer of unusual structural complexity. Nature Chemical Biology. , in review (2018).
- Panchapakesan, S. S. S., Jeng, S. C. Y., Unrau, P. J. RNA complex purification using high-affinity fluorescent RNA aptamer tags. Annals of the New York Academy of Sciences. , (2015).
- Filonov, G. S., Kam, C. W., Song, W., Jaffrey, S. R. In-gel imaging of RNA processing using Broccoli reveals optimal aptamer expression strategies. Chemistry & Biology. 22, 649-660 (2015).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic Acids Research. 15, 8783-8798 (1987).
- A Typical DNase I Reaction Protocol (M0303). NEB. , Available from: https://www.neb.com/protocols/1/01/01/a-typical-dnase-i-reaction-protocol-m0303 (2018).
- Sambrook, J., Russell, D. W. Purification of Nucleic Acids by Extraction with Phenol:Chloroform. Cold Spring Harbor Protocols. 2006 (1), pdb.prot4455 (2006).
- Tuma, R. S., et al. Characterization of SYBR Gold Nucleic Acid Gel Stain: A Dye Optimized for Use with 300-nm Ultraviolet Transilluminators. Analytical Biochemistry. 268, 278-288 (1999).
- Streit, S., Michalski, C. W., Erkan, M., Kleeff, J., Northern Friess, H. Northern blot analysis for detection and quantification of RNA in pancreatic cancer cells and tissues. Nature Protocols. 4, 37-43 (2009).
- Ebhardt, H. A., Unrau, P. J. Characterizing multiple exogenous and endogenous small RNA populations in parallel with subfemtomolar sensitivity using a streptavidin gel-shift assay. RNA. 15, 724-731 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved