Method Article
Visualización fluorescente de ARN etiquetado con Mango en geles de poliacrilamida a través de un método de postmancha
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí presentamos un método de tinción post-gel sensible, rápido y discriminatorio para la imagen de ARN etiquetados con aptámeros de mango de ARN I, II, III o IV, utilizando geles de electroforesis de gel de poliacrilamida (PAGE) nativos o desnaturalizantes. Después de ejecutar geles PAGE estándar, el ARN con etiqueta Mango se puede teñir fácilmente con TO1-Biotin y luego analizarse utilizando lectores de fluorescencia comúnmente disponibles.
Resumen
Los geles nativos y desnaturalizantes de poliacrilamida se utilizan habitualmente para caracterizar la movilidad compleja de ribonucleoproteína (RNP) y medir el tamaño del ARN, respectivamente. Como muchas técnicas de imágenes de gel utilizan manchas inespecíficas o sondas de fluoróforo costosas, son altamente deseables metodologías sensibles, discriminatorias y económicas de imágenes de gel. Las secuencias de núcleo de Mango de ARN son pequeños motivos de secuencia (19-22 nt) que, cuando están cerrados por un tallo de ARN arbitrario, pueden ser simplemente y baratos anexados a un ARN de interés. Estas etiquetas de Mango se unen con alta afinidad y especificidad a un ligando de fluoróforo tiazol-naranja llamado TO1-Biotina, que se vuelve miles de veces más fluorescente al encuadernar. Aquí mostramos que Mango I, II, III y IV se puede utilizar para crear imágenes específicas de ARN en geles con alta sensibilidad. Tan solo 62,5 fmol de ARN en geles nativos y 125 fmol de ARN en geles desnaturalizantes se pueden detectar empapando geles en un tampón de imágenes que contiene potasio y 20 nM TO1-Biotina durante 30 min. Demostramos la especificidad del sistema etiquetado con Mango mediante la toma de imágenes de un ARN bacteriano 6S con etiqueta de Mango en el contexto de una mezcla compleja de ARN bacteriano total.
Introducción
Mango es un sistema de etiquetado de ARN que consiste en un conjunto de cuatro pequeños aptámeros fluorescentes de ARN que se unen firmemente (unión nanomolar) a derivados simples del tiazol-naranja (TO1-Biotina, Figura 1A)1,2,3 . Tras la unión, la fluorescencia de este ligando se incrementa de 1.000 a 4.000 veces dependiendo del aptámero específico. El alto brillo del sistema Mango, que para Mango III supera al de la proteína fluorescente verde mejorada (eGFP), combinada con la afinidad de unión nanomolar de los aptámeros de mango de ARN, permite su uso tanto en la imagen como en la purificación del ARN complejos2,4.
Las estructuras de rayosX de Mango I 5, II6y III7 se han determinado a alta resolución, y los tres aptámeros utilizan un ARN cuádruplex para unir TO1-Biotina (Figura1B–D). Los núcleos compactos de los tres aptámeros están aislados de la secuencia de ARN externa a través de motivos de adaptador compactos. Mango I y II utilizan un adaptador de bucle flexible similar a GNRA para conectar sus núcleos de mango a un dúplex de ARN arbitrario (Figura1B,C). Por el contrario, Mango III utiliza un motivo rígido triplex para conectar su núcleo a una hélice de ARN arbitraria (Figura1D,residuos púrpuras), mientras que la estructura de Mango IV no se conoce actualmente. Como el núcleo de unión de ligandos de cada uno de estos aptámeros está separado de la secuencia de ARN externa por estos adaptadores helicoidales, parece probable que todos puedan ser simplemente incorporados en una variedad de ARN. El ARN regulador 6S bacteriano (Mango I), componentes del espliceosoma de levadura (Mango I), y el ARN 5S humano, ARN U6, y un escalonado C/D (Mango II y IV) han sido etiquetados con éxito en esta moda2,8, lo que sugiere que muchos LOS ARN biológicos se pueden etiquetar utilizando el sistema aptamer de ARN Mango.
Los geles nativos y desnaturalizantes se utilizan comúnmente para estudiar los ARN. Los geles dedesnaturalización se utilizan a menudo para juzgar el tamaño del ARN o el procesamiento de ARN, pero normalmente, en el caso de una mancha del norte, por ejemplo, requieren varios pasos lentos y secuenciales para generar una imagen. Mientras que otros aptámeros fluorogénicos de ARN, como el ARN Spinach y el Brócoli, se han utilizado con éxito para imágenes en gel9,ningún sistema de aptámeros fluorogénicos hasta la fecha posee el alto brillo y la afinidad del sistema Mango, por lo que es de considerable interés para investigar las habilidades de rayos de gel de Mango. En este estudio, nos preguntamos si el sistema de mango de ARN podría simplemente extenderse a las imágenes de gel, ya que las longitudes de onda de excitación y emisión de TO1-Biotina (510 nm y 535 nm, respectivamente) son apropiadas para la toma de imágenes en el canal eGFP común a la mayoría de los fluorescentes instrumentación de escanado en gel.
El protocolo de tinción post-gel presentado aquí proporciona una manera rápida de detectar específicamente moléculas de ARN etiquetadas con Mango en geles de electroforesis de gel de poliacrilamida (PAGE) nativos y desnaturalizantes. Este método de tinción consiste en remojar geles en un tampón que contiene potasio y TO1-Biotina. Los aptámeros de mango de ARN son basados en G-quadruplex y se requiere potasio para estabilizar dichas estructuras. Utilizando el ARN transcrito a partir de plantillas mínimas de ADN de codificación de Mango (ver la sección de protocolo), simplemente podemos detectar tan poco como 62.5 fmol de ARN en geles nativos y 125 fmol de ARN en geles desnaturalizantes, utilizando un protocolo de tinción directa. A diferencia de las manchas comunes de ácido nucleico no específico (ver Tabla de Materiales,referida a SG a partir de aquí), podemos identificar claramente el ARN etiquetado con Mango incluso cuando hay altas concentraciones de ARN total sin etiquetar en la muestra.
Protocolo
1. Preparación de los reactivos
-
To1-Solución de postmancha de biotina (solución de tinción de gel)
- Hacer 1 L de tampón de fosfato de 1 M a pH 7,2 a 25 oC añadiendo 342 ml de 1 M de Na2HPO4 y 158 ml de 1 M NaH2PO4. Ajuste el pH a 7,2 a 50 mM añadiendo la solución de fosfato adecuada. Filtro estéril con un filtro de 0,2 m y almacene la solución en plásticos a temperatura ambiente.
- Prepare la solución de tinción de gel 5x (sin TO1-Biotina) de la siguiente manera. Componer una solución de 1 L mezclando 247,5 ml de ddH2O, 700 ml de 1 M KCl, 50 ml de tampón de fosfato de 1 M (pH 7,2) y 2,5 ml de Tween 20. Filtro estéril con un filtro de 0,2 m y almacene la solución en plástico. Se puede almacenar a temperatura ambiente.
NOTA: MgCl2 se puede añadir a esta solución si es necesario, ya que potencialmente puede estabilizar los complejos de ARN. Esto tendrá un impacto modesto en la señal fluorescente2. - Componer una solución de tinción de gel 1x utilizando la solución de tinción de gel 5x del paso 1.1.2 y, inmediatamente antes de su uso, complementarla con fluoróforo TO1-Biotina (ver Tabla de Materiales)a una concentración final de 20 nM. Por ejemplo, añadir 20 ml de solución de tinción de gel 5x a 80 ml de agua desionizada para hacer 100 ml de solución de tinción de gel 1x, y añadir 2 ml de un stock de 1 mM TO1-Biotina (preparado en dimetilformamida).
NOTA: El coeficiente de extinción para el tinte TO1-Biotina a 500 nm es de 63.000 M-1cm-1, medido en la solución de tinción1.
-
PAGINA Desnaturalante (2x solución de carga de gel desnaturalizante y soluciones A, B, C, persulfato de amonio y tetrametiletilendiamina)
- Preparar 50 ml de solución de carga de gel desnaturalizante 2veces mezclando 40 ml de formamida, 0,5 ml de ácido etilendiaminetetraacético de 0,5 M (EDTA, pH 8.0) y 9,5 ml de ddH2O. La solución se puede almacenar a temperatura ambiente. Los colorantes de carga no se agregan a esta solución, ya que puede oscurecer las imágenes fluorescentes.
- Prepare el tinte de carga de gel desnaturalizador 2x de la siguiente manera. Al purificar los arnlos y los oligonucleótidos de ADN, añada 0,5 ml de 2,5% (p/v) azul bromofenol (BB) y 0,5 ml de 2,5% (p/v) de xileno cianol (XC) a la solución descrita en el paso 1.2.1, y agregue 8,5 ml de ddH2O en lugar de 9,5 ml. La solución se puede almacenar a temperatura ambiente.
- Preparar 100 ml de solución A pesando 200,2 g de urea (peso de fórmula: 60,06 g/mol) y añadiéndola a 312,5 ml de 40% 19:1 acrilamida:N,N'-metilenhemenacrilamida. Revuelva con una barra de agitación magnética a máxima velocidad hasta que se disuelva (aproximadamente 3 h) y coma la solución a 500 ml utilizando ddH2O. Tenga en cuenta que las concentraciones finales son 6.667 M de urea y 25% 19:1 acrilamida:N,N'-metilenbisacrilamida. Conservar a 4oC en plásticos limpios.
- Preparar 1 L de la solución B pesando 400,4 g de urea y conforman la solución a 1 L con ddH2O; revuelva hasta que la urea se haya disuelto. La solución B se puede mantener a temperatura ambiente.
- Prepare la solución C (10x Tris-borate-EDTA [TBE]) de la siguiente manera. Preparar 4 L de 10x TBE pesando 432 g de base Tris y 220 g de ácido bórico, añadiendo 160 ml de 0,5 M EDTA con un pH de 8 (filtrado), y conformando la solución a 4 L con ddH2O. Un gran stock de este búfer se hace, ya que también se utiliza como tampón de ejecución de gel.
- Preparar el 10% de persulfato de amonio (APS) disolviendo 1 g de APS en un volumen total de 10 ml de ddH2O. Almacenar a 4 oC.
- Mantenga a mano la tetrametiletilemediamina (TEMED). Almacenar lo a 4 oC junto con la solución APS.
-
PAGE nativo (2x solución de carga de gel nativo)
- Preparar 50 ml de solución de carga de gel nativa 2x mezclando 25 ml de 100% glicerol, 10 ml de solución de tinción de gel 5x y 15 ml de ddH2O para componer la solución a 50 ml.
NOTA: Al igual que en la sección de gel dedesnaturalización, se pueden añadir colorantes de carga a este stock, pero su adición puede oscurecer potencialmente la señal fluorescente en el gel y, por lo tanto, debe evitarse.
- Preparar 50 ml de solución de carga de gel nativa 2x mezclando 25 ml de 100% glicerol, 10 ml de solución de tinción de gel 5x y 15 ml de ddH2O para componer la solución a 50 ml.
2. Preparación y carga de geles desnaturalizantes
- Preparar un gel desnaturalizante con un porcentaje adecuado siguiendo la Tabla 1. Considere el sistema específico de fundición de gel; Los geles de 30 ml se utilizan comúnmente. El porcentaje de gel adecuado se puede estimar utilizando la Tabla2: seleccione el porcentaje de poliacrilamida donde los colorantes BB y XC tengan motilidades más rápidas y lentas, respectivamente, que el ARN de interés, para garantizar la separación de bandas altas en el tamaño pertinente Gama.
- Mezcle las soluciones A, B y C según la Tabla 1 y agregue APS y TEMED inmediatamente antes de verter el gel. Mezcle bien las soluciones en plásticos limpios.
- Vierta la solución de gel en un aparato de fundición de gel adecuado, después de asegurarse de que todos los componentes estén escrupulosamente limpios. Levante un lado del aparato para que el gel se incline ligeramente al verter, para evitar que las burbujas de aire queden atrapadas dentro del gel en sí.
- Inserte el peine deseado y déjelo polimerizar (aproximadamente 30 min). Observe la polimerización mirando de cerca un cambio en el índice de refracción alrededor de los pozos de gel. Componer el tanque de gel utilizando 1 x solución C (1x TBE) y retirar cuidadosamente el peine. Con una jeringa, aspirar los pozos inmediatamente antes de la carga de la muestra.
- Prepare las muestras de desnaturalización de la siguiente manera. Añadir 2x solución de carga de gel desnaturalizador a las muestras de ARN de interés para hacer que la solución de carga de gel desnaturalizador 1x y la desnaturalización del calor a 95 oC durante 5 min mediante el uso de un termociclador o baño de agua.
- Antes de cargar las muestras, enfríe las muestras durante varios minutos hasta que estén frías al tacto. Cargue las muestras superándolas en capas en la parte inferior de cada pocal, usando puntas de carga de gel.
NOTA: Las puntas redondas se pueden utilizar para geles de 1 mm de espesor o más; puntas planas para geles más delgados. - Ejecuta los geles desnaturalizantes a temperatura ambiente. Asegúrese de que la potencia es lo suficientemente baja para que las placas de vidrio del sistema de gel no se agrieten.
NOTA: En nuestro laboratorio, 28 W para placas de 20 cm x 16 cm era suficiente para este propósito.
3. Preparación y carga de geles nativos
- Prepare un gel nativo con un porcentaje adecuado refiriéndose a la Tabla 3. Considere el sistema específico de fundición de gel, pero tenga en cuenta que los geles de 30 ml se utilizan comúnmente. Mezclar las soluciones de 1x TBE, 40% 29:1 acrilamida:N,N'-metilenbisacrilacrilamida, y glicerol según la Tabla 3, y añadir APS y TEMED inmediatamente antes de verter el gel. Proceda como se describe en los pasos 2.3 y 2.4.
- Preparar muestras nativas añadiendo 2x solución de carga de gel nativo a las muestras de ARN de interés para hacer la solución 1x y permitir que incubaa a temperatura ambiente durante 100 minutos antes de ejecutar el gel para asegurar el plegado completo del ARN.
- Ejecuta el gel nativo en una cámara frigorífica a 4oC, asegurándote de que la potencia sea lo suficientemente baja como para no calentar el gel.
NOTA: En nuestro laboratorio, 14 W para placas de 20 cm x 16 cm era suficiente para este propósito.
4. Preparación del ARN mediante la transcripción T7 de escorrenca
NOTA: Las secuencias de ADN utilizadas para la transcripción de escorrenta10 de las construcciones de ARN Mango fueron ordenadas comercialmente. En este método, los oligonucleótidos de ADN que contienen el complemento inverso (RC) tanto de la secuencia a transcribir como del promotor T7 se hibridan en una secuencia de hebra superior promotora T7 y luego se trancriban in vitro. A continuación, para cada oligonucleótido, el RC de la secuencia del núcleo de Mango se muestra en negrita y el RC de la región promotora del T7 se muestra en cursiva. Los residuos en la fuente regular corresponden a regiones helicoidales complementarias arbitrarias necesarias para permitir que el núcleo de Mango se doble correctamente.
Mango I: GCA CGT ACT CTC CTC TCC GCA CCG TCC CTT CGT ACG TGC CTA TAG TGA GTC GTA TTA AAG
Mango II: GCA CGT ACT CTC CTC TTC CTC TCC TCT CCT CGT ACG TGC CTA TAG TGA GTC GTA TTA AAG
Mango III: GGC ACG TAC GAA TAT ACC ACA TAC CAA TCC TTC CTT CT ACG TGC CTA TAG TGA GTC GTA TTA AAG
Mango IV: GCA CGT ACT CGC CTC CTC ACC ACT CCC TCG GTA CGT GCC TAT AGT GAG TCG TAT TAA AG
T7 Top Strand: CTT TAA TAC GAC TCA CTA TAG G
- Purificación de gel preparativo de los oligonucleótidos de ADN
- Para una síntesis de ADN a escala de 0,2 omol, resuspenda el oligonucleótido de ADN desprotegido en 100 s de ddH2O y 100 l de tinte de carga desnaturalizador 2x (del paso 1.2.2). En este caso, se prefiere incluir los colorantes de carga de gel en la solución de carga a la misma concentración que en el paso 1.2.2.
- Purificar el oligonucleótido de ADN utilizando un gel desnaturalizante de escala preparativa de 50 ml del porcentaje adecuado de poliacrilamida; utilizar la Tabla 2 para elegir el porcentaje de gel. Por ejemplo, utilice un 8% de gel para una secuencia de 50 nt. Idealmente, el azul bromofenol debe funcionar más rápido que el oligonucleótido, y el cianol de xileno debe funcionar más lento.
NOTA: En nuestro laboratorio, estos geles se fundieron utilizando espaciadores de fundición de 1,5 mm de espesor y un peine de carga de gel con pozos de 2-2,5 cm de ancho. - Cargar 100 l de las soluciones de oligonucleótidos de ADN preparadas en el paso 4.1.2 por gel preparativo, así como se describe en los pasos 2.4–2.7.
- Seque cuidadosamente el exterior del gel, retire las placas de vidrio y cubra ambos lados del gel en una envoltura de plástico. Colóquelo en una pantalla de imágenes fluorescentes (las placas de cromatografía de capa delgada con respaldo de aluminio impregnadas con fluoróforo son una solución económica) y utilice una lámpara portátil UV de longitud de onda corta para visualizar las bandas de ADN mediante sombras UV.
- Se debe observar una sombra nítida y bien definida si la síntesis de ADN es de alta calidad. Marque las bandas en la envoltura de plástico, usando un marcador permanente.
NOTA: Proteja la piel y los ojos de la luz UV y mantenga las exposiciones cortas para evitar dañar la muestra de ácido nucleico. - Colocar el gel en una placa de vidrio limpia, cortar cuidadosamente las bandas marcadas y colocar cada fragmento de gel en 400 sL de 300 mM de NaCl. Eluir el ADN durante la noche, usando un rotador a temperatura ambiente.
- Recuperar el eluyente en un tubo de centrífuga limpia y añadir 2.5 equivalentes de etanol para precipitar el ADN. Vortex bien y coloque el tubo a -20 oC durante 30 min.
- Pelet la muestra en una centrífuga de sobremesa a 156.000 x g durante 30 min a 4oC. Retire cuidadosamente el sobrenadante y resuspenda el pellet en ddH2O. Utilice un espectrofotómetro para determinar con precisión la concentración de oligonucleótidos de ADN. Almacene la muestra como un stock de 10 m para mayor comodidad a -20 oC.
- Transcripción de escorrente y purificación en gel de muestras de ARN
- Preparar 5x stock de trifosfato de nucleósidos (NTP) mezclando reservas líquidas de NTP compuestas por una concentración final de trifosfato de guanosina de 40 mM (GTP, 11.400 M-1cm-1), trifosfato de citidina de 25 mM (CTP, 7.600 M-1cm-1), 25 mMM trifosfato de adenosina (ATP, 5.000 M-1cm-1), y trifosfato de uridina de 10 mM (UTP, 10.000 M-1cm-1); todos los coeficientes de extinción a 260 nm.
NOTA: Las existencias líquidas o en polvo se pueden obtener comercialmente. Si prepara las existencias primarias del polvo, ajuste cuidadosamente el pH a un pH final de 7,9, utilizando 1 M NaOH. Alícuota el stock en tubos de microcentrífuga de 1,5 ml y guárdelo a -20 oC. - Preparar 100 ml de tampón de transcripción 10x T7 mezclando 25,6 ml de 1 M Tris-HCl, 14,4ml de base Tris de 1 M, 26 ml de 1 M MgCl 2, 10 ml de 10% de tritón X-100 y 0,637 g de espermadina. Componer la acción a un volumen final de 100 ml añadiendo ddH2O.
NOTA: Se debe confirmar un pH final de 7,9 de la solución 1x utilizando un medidor de pH calibrado. El 10x debe filtrarse estérilmente y almacenarse a -20 oC en alícuotas de tamaño adecuado. - Realice la transcripción de la siguiente manera.
- Añadir los siguientes reactivos para componer una concentración final de 1búfer de transcripción T7 utilizando el stock de tampón de transcripción 10x T7 y ddH2O (del paso 4.2.2), 1x NTPs, 10 mM de ditiothreitol (DTT), 1 M T7 secuencia de hilo superior (ver sección 4 Nota para el secuencia de ADN de mango purificado en gel de 1 M (paso 2.1) y enzima polimerasa de ARN T7 (1 U/L).
- Vórtice, girar hacia abajo, e incubar la solución a 37 oC durante 2 h o hasta que se vuelve turbia y se forma un precipitado blanco en la parte inferior del tubo. Una transcripción de 50 l debe dar como resultado 50 ml de ARN de 50 m después de la purificación del gel. Añadir un volumen igual de 2x tinte desnaturalizante y almacenarlo a -20 oC hasta que esté listo para la purificación del gel.
- Gel-purificar el ARN resultante como se describe en la sección de purificación de gel de oligonucleótidos de ADN siguiendo los pasos 4.1.2-u20124.1.8, sustituyendo la muestra de ARN por el ADN.
NOTA: El ARN es extremadamente sensible a la degradación de RNase, así que asegúrese de que en todos los pasos, guantes y una capa de laboratorio limpia se usen en todo momento. Asegúrese de que todas las muestras se preparen y almacenen en material plástico de un solo uso para protegerlas contra la contaminación por RNase, y lave las placas de vidrio cuidadosamente con agua y jabón caliente antes de su uso, enjuáguelas bien con ddH2O y seque la cristalería con el la asistencia del etanol aplicado a partir de una botella de compresión. - Utilice 1 l de la muestra final de ARN y, utilizando un espectrofotómetro basado en gotas, determine la absorbancia a 260 nm. Usando un coeficiente de extinción determinado por el método de vecino más cercano, calcule la concentración de ARN y ajuste la concentración de la muestra a 10 m con ddH2O para mayor comodidad. Almacene las muestras de ARN a -20 oC.
- Preparar 5x stock de trifosfato de nucleósidos (NTP) mezclando reservas líquidas de NTP compuestas por una concentración final de trifosfato de guanosina de 40 mM (GTP, 11.400 M-1cm-1), trifosfato de citidina de 25 mM (CTP, 7.600 M-1cm-1), 25 mMM trifosfato de adenosina (ATP, 5.000 M-1cm-1), y trifosfato de uridina de 10 mM (UTP, 10.000 M-1cm-1); todos los coeficientes de extinción a 260 nm.
- Escherichia coli extracción total de ácido nucleico bruto
NOTA: El siguiente protocolo es un ejemplo. Este protocolo utiliza ARN 6S con etiqueta I de Mango expresado endógenamente a partir de un plásmido en E.coli, y la inducción de este plásmido se describe en otra parte en detalle4.- A los efectos de este estudio, preparar 500 ml de células inducidas para el pEcoli-RNA Mango y los plásmidos de pEcoli-T1 (las células se inducen a una DO600 nm de 1). Peletizar las células y almacenarlas a -80 oC antes de su uso.
- Realice la extracción de ARN de la siguiente manera.
- Tomar 0,5 ml del pellet de células de pEcoli inducido y añadir 500 ml de fenol equilibrado. A continuación, vórtice la muestra; la solución se volverá blanca lechosa. Centrífuga a máxima velocidad durante 2 minutos para separar las capas.
- Extraiga la capa superior, que será ligeramente amarilla, y repita el paso 4.3.2.1 añadiendo un volumen igual de fenol, centrifugación y extrayendo para un total de 5x (o hasta que la capa media, que es blanca opaca, desaparezca y solo queden dos capas claras).
- Agregue el volumen acuoso extraído de fenol a un volumen igual de cloroformo, vórtice y, a continuación, centrífuga a la velocidad máxima durante 2 min.
- Extrae la capa acuosa y añade NaCl a una concentración final de 300 mM. Precipitar el ácido nucleico resultante mediante la adición de 2,5 equivalentes de etanol, vórtice, espín hacia abajo, y precipitar a -20 oC durante al menos 30 min.
- Pellet en una centrífuga de sobremesa a 15,6 x 1.000 x g a 4 oC durante 30 minutos. Retire cuidadosamente el sobrenadante y vuelva a suspender el pellet en 100 ml de ddH2O.
- Añadir un paso de digestión DNase Ia este procedimiento, siguiendo el protocolo 11 al que se hace referencia, para obtener el ARN total.
NOTA: Vórtice vigorosamente hasta que el pellet se disuelva completamente en ddH2O.
5. Tinción post-gel
- Preparar 1 solución de tinción de gel según la receta en el paso 1.1.3 y añadirlo a un recipiente de vidrio limpio que sea lo suficientemente ancho como para encajar cómodamente el gel.
NOTA: Los recipientes de vidrio borosilicato con tapas de ajuste a presión sirven bien para este propósito. - Añadir suficiente solución de tinción de gel 1x al recipiente para que el gel esté completamente cubierto con la solución y el líquido se sloshes sobre la parte superior del gel cuando se coloca en el rotador orbital.
NOTA: El recipiente debe ser lo suficientemente grande como para caber el gel para que pueda moverse y tener suficiente tampón en el recipiente para cubrir completamente el gel. - Una vez que el gel nativo o desnaturalizante haya terminado de funcionar (sección 2 o 3, respectivamente), retire el gel del aparato y corte sus pozos. Puede ser útil quitar una esquina del gel para la orientación más adelante en el análisis.
- Transfiera cuidadosamente el gel a la solución de tinción de gel 1x preparada en el paso 5.2.
NOTA: Los geles son frágiles y propensos a romperse, así que tenga cuidado al transferir el gel. Mantenga la tapa del recipiente de tinción en todo momento para no contaminar el gel. - Coloque el gel en un rotador orbital a una velocidad de 100 rpm durante 30 minutos a temperatura ambiente.
NOTA: Asegúrese de que el gel no se doble sobre sí mismo; de lo contrario, el ARN puede difundirse fuera del gel y así etiquetar una parte diferente del gel.
6. RNA con etiqueta de Mango de imágenes en gel
- Decantar cuidadosamente el búfer de imágenes. Enjuague el gel rápidamente con agua, asegurando suficientes restos líquidos para mantener el gel ligeramente móvil en el recipiente.
- Transfiera cuidadosamente el gel al imager. Transfiera recogiendo cuidadosamente los lados del gel y colocándolo en la bandeja del imager; alternativamente, el gel se puede verter lentamente en la bandeja. Asegúrese de que no haya burbujas debajo del gel y de que no haya exceso de líquido debajo del gel. Una pipeta enrollada sobre el gel puede ser útil para eliminar cualquier exceso de líquido.
NOTA: Use una toalla de papel para absorber el exceso de líquido. - Tome una imagen de gel, observando la fluorescencia entre la longitud de onda de excitación 510 nm y la longitud de onda de emisión 535 nm (por ejemplo, luz verde a 520 nm ajustes de fluorescencia de longitud de onda en el imager). Asegúrese de que los ajustes estén completamente optimizados en el instrumento y siga cuidadosamente las instrucciones del instrumento utilizado.
Resultados
Los ARN cortos etiquetados con Mango se prepararon como se describe en la sección de protocolo. Suponiendo que la fluorescencia en condiciones de desnaturalización sería más difícil de observar debido a la presencia de urea en los geles, primero estudiamos la resistencia de los aptámeros de mango a la urea, que actúa como un desnaturalizante del ácido nucleico. Encontramos que los aptámeros de mango son sustancialmente resistentes a la desnaturalización hasta una concentración de urea de aproximadamente 1 M (Figura2A). Antes de añadir la solución de tinción de gel a un gel desnaturalizante, la concentración final de urea en el gel es de 6 M. Añadir suficiente solución de tinción para disminuir esta concentración a 1 M sería, por lo tanto, óptimo para asegurar la fluorescencia completa de Mango para los cuatro aptámeros. En la práctica, el protocolo de tinción logró menos que este resultado totalmente óptimo, pero esto simplemente podría ser rectificado si fuera necesario mediante el uso de más solución de tinción o por la simple conveniencia de cambiar la solución una vez durante la tinción.
Una vez que las construcciones de ARN etiquetadas con Mango se encontraron con un gel desnaturalizante, el tiempo de tinción se optimizó para la máxima fluorescencia de gel mediante la carga de tres cantidades diferentes de Mango III en un gel desnaturalizante del 8% (Figura2B). Un curso de tiempo reveló que después de 5 minutos de remojo en la solución de tinción de gel, la fluorescencia era claramente visible. La fluorescencia máxima de la construcción Mango III se obtuvo después de 20-40 minutos de tinción, después de lo cual los pequeños ARN utilizados en este estudio comenzaron a difundirse fuera del gel, resultando en una pérdida de señal fluorescente (Figura2B). En consecuencia, tanto los geles nativos como los desnaturalizantes se teñiron durante 30 minutos cada uno. Las construcciones de ARN más largas podrían tolerar fácilmente tiempos manchados más largos, ya que serían mucho menos propensos a difundirse fuera del gel.
Algunos de los aptámeros de mango de ARN se doblaron más rápidamente que otros. Cada uno de los aptámeros de Mango utilizados en este estudio fue incubado en un tampón de gel complementado con 1.5 M de urea y 100 nM TO1-Biotin dye y analizado usando un fluorómetro. Mango I, II y III se doblaron completamente después de 10 minutos, mientras que Mango IV se dobló sustancialmente sólo después de 40 min (Figura3A). En ausencia de urea, el plegado fue mucho más rápido como se esperaba (Figura3B). Para asegurarnos de que las muestras de gel nativo estaban completamente plegadas, preincubamos muestras durante 100 minutos antes de ejecutarlas en geles nativos. Prácticamente, los datos de la Figura 3 sugieren que este tiempo podría reducirse sustancialmente dependiendo del aptamer de mango utilizado.
Una vez optimizado el protocolo para detectar la fluorescencia de aptámero de ARN Mango, se determinó la sensibilidad del método de postmanchación para cada una de las variantes de Mango en ambos geles nativos. Se observaron bandas individuales correspondientes a ARN bien plegados para cada uno de los cuatro Mangos en geles nativos (Figura4A). Tras la dilución en serie, se pudo observar tan poco como 62,5 fmol de Mango II, mientras que tan poco como 125 fmol de Mango I, III y IV se visualizaron fácilmente. La cuantificación de geles nativos fue log-lineal sobre aproximadamente 1,5 órdenes de magnitud, con Mango I, II y IV comportándose de una manera más lineal que Mango III (Figura4B).
Los resultados de los geles dedesnaturalizantes fueron ligeramente menos sensibles que los geles nativos, pero eran más lineales. Tan solo 125 fmol de Mango II y III se detectaron fácilmente (Figura4C). Curiosamente, la cuantificación (Figura4D) indicaba que los geles desnaturalizantes eran log-lineales o dos órdenes de magnitud. Hipotetizar que, a diferencia de los geles nativos donde los pliegues de ARN tal vez fueron sometidos a desnaturalización parcial durante el proceso de ejecución del gel, la presencia de urea en el gel desnaturalizante podría proporcionar una manera más homogénea de doblar los aptámeros una vez que se colocan en la solución de tinción TO1-Biotina.
Al igual que con todas las metodologías de tinción de gel, si el gel no se transfiere cuidadosamente al recipiente, o la velocidad de rotación es demasiado alta durante el período de tinción, el gel puede plegarse de nuevo sobre sí mismo (Figura5A). Esto puede resultar en la muestra de ARN etiquetada Mango difuminando de un lugar del gel a otro, pero se puede evitar fácilmente en la práctica. En geles nativos y, particularmente, para Mango IV, observamos que el plegado incompleto puede manifestarse en la aparición de múltiples bandas presumiblemente correspondientes a las conformaciones de ARN parcialmente/ mal plegadas (Figura5B)resultantes de tiempos de plegado más cortos. Los problemas de plegado en geles nativos se pueden evitar preincubando muestras de ARN apropiadamente como se describió anteriormente y ejecutando geles nativos en una cámara fría. En geles desnaturalizantes, donde el ARN se pliega in situ dentro del gel, el desdoblamiento no fue un problema significativo. Finalmente, en ausencia de ARN, se observó muy poca fluorescencia de fondo en cualquiera de los sistemas de gel.
A continuación, se estudió la especificidad de la etiqueta DE Mango de ARN al sobreexpresar el ARN regulador 6S en bacterias. Este ARN fue etiquetado previamente usando Mango I (Figura 1E)4. Las células bacterianas se transformaron con el plásmido pecoli-RNA Mango (en adelante M plásmido) o el plásmido pecoli-T1 como un control negativo (en adelante, el plásmido E). Las células transformadas se cultivaron en caldo de lisógeno líquido medio hasta que se alcanzó una OD600 de 1.0. Los cultivos fueron entonces inducidos con isopropilo de 50 m-D-1-thiogalactopyranoside (IPTG) durante 40 min. Las células se cosecharon a través de centrifugación a 6.000 x g durante 15 min. El ARN total se extrajo utilizando la extracción de cloroformo de fenol-fenol de los pellets celulares12 como se describe en la sección de protocolo. Las muestras totales de ARN se concentraron por la precipitación de etanol y luego se trataron con DNase I, siguiendo el protocolo, para eliminar el ADN11. Antes de usar, el ARN se concentró en la precipitación de etanol.
El ARN bacteriano total se enroscócon geles desnaturalizantes del 8% (Figura 6) y se tiñó con SG13 o TO1-Biotina. Para la tinción SG, se añadieron 10 sl de 10.000x SG a 100 ml de tampón de gel; de lo contrario, el protocolo de tinción era idéntico al utilizado para TO1-Biotin. Como era de esperar, se observó una fuerte tinción SG para una multitud de ARN, pero más prominentemente para ribosomal (RRNA) y transferencia de ARN (tRNA) (Figura6, panel izquierdo). Mientras que la tinción dependiente del mango (carriles M) se podía ver en estos geles teñidos de SG, no podían ser identificados de forma única dado el complejo patrón de tinción observado utilizando esta mancha universal. Por el contrario, los geles teñidos con TO1-Biotina destacaron a las bandas dependientes del mango como las bandas más prominentes. Sólo las bandas de ARN ribosomal se ven en competencia con las bandas dependientes de 6S Mango. También se pudieron observar varias bandas manchadas de forma no específica. Sin embargo, las bandas dependientes del Mango fueron de nuevo dominantes, teniendo sólo las bandas rRNA y tRNA como competidores débilmente competidores (Figura6, panel derecho).

Figura 1: Sistema de aptámero de mango. (A) TO1-Fluoróforo de biotina. (B) Mango I. (C) Mango II. (D) Mango III. Los paneles B–D muestran la estructura secundaria de cada aptámero. P1 es un tallo arbitrario. El lazo de tallo similar a GNRA (aquí GAAA) que se encuentra en Mango I y II se muestra en rojo, el motivo triplex de Mango III se muestra en púrpura. (E) El ARN regulador 6S etiquetado con Mango I. Por favor, haga clic aquí para ver una versión más grande de esta figura.
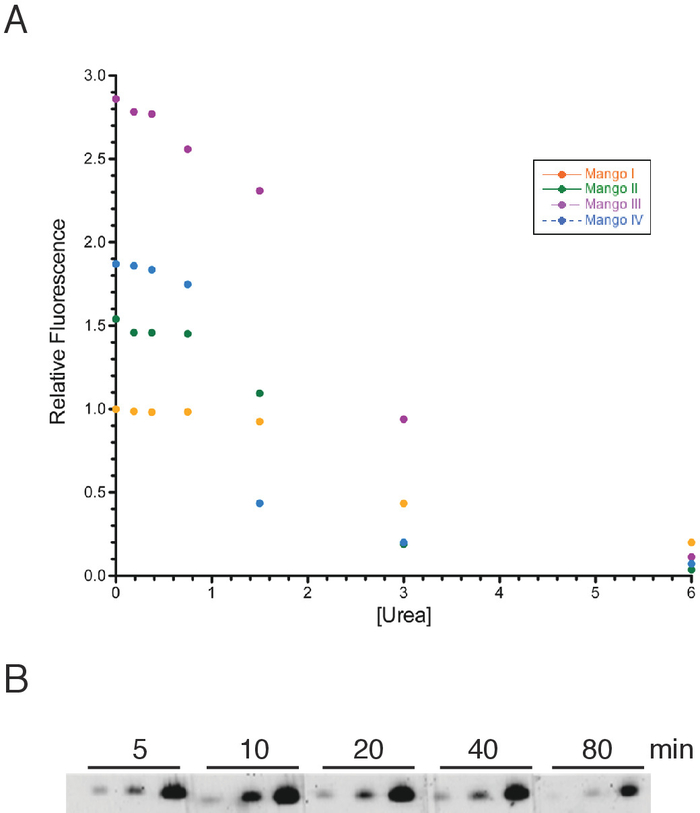
Figura 2: Efecto de la urea en la fluorescencia del afeómero de mango y tiempos de tinción óptimos. (A) Una valoración de urea con 50 nM DE ARN Mango I (círculos naranjas), Mango II (círculos verdes), Mango III (círculos púrpuras) y Mango IV (círculos azules), junto con 100 nM TO1-Biotin dye y en las concentraciones de urea indicadas. Las muestras fueron incubadas durante 40 minutos antes de que se leyera la fluorescencia a una longitud de onda de excitación de 510 nm y una longitud de onda de emisión de 535 nm. (B) El ARN Mango III se cargó en un gel desnaturalizador del 8% y se tiñó con una solución de gel que contenía un tinte TO1-Biotina final de 20 nM. Para cada punto de tiempo indicado, se utilizaron 0,064, 0,32 y 1,6 pmol de ARN de Mango III de izquierda a derecha. La imagen de gel se visualizó con un imager de fluorescencia utilizando un láser de 520 nm y una exposición de 10 minutos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Tiempos de plegado de los aptámeros de mango en presencia y ausencia de urea. (A) En presencia de 1,5 M de urea y 100 nM TO1-Biotina, se realizaron cursos de tiempo de fluorescencia utilizando 50 nM de cada construcción de ARN Mango (RNA Mango I: puntos naranjas, Mango II: puntos verdes, Mango III: puntos morados, y Mango IV: puntos azules). (B) Idéntico al panel A, excepto en ausencia de urea. Todos los cursos de tiempo se realizaron a temperatura ambiente. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Imágenes fluorescentes de PAGE nativo y desnaturalizante con construcciones de Mango de ARN. (A) Un gel nativo del 8% con construcciones de mango de ARN diluido en serie. Los carriles I, II, III y IV contienen 8 cantidades finales de pmol de ARN Mango I, II, III y IV, respectivamente. Los paneles derecho son dos diluciones en serie que contienen 4 pmol, 2 pmol, 1 pmol, 0,5 pmol, 0,25 pmol, 0,125 pmol y 0,0625 pmol de Mango I, II, III o IV, como se indica. El carril 12 no contiene ARN. (B) Cuantificación de tres réplicas del gel nativo (desviación estándar de la media mostrada para cada una). (C) Un gel desnaturalizante del 8% con las mismas muestras cargadas que en el panel A, excepto por el hecho de que se utilizó una solución de carga de gel desnaturalizante en lugar de una solución de carga de gel nativa. (D) Tres réplicas cuantificadas del gel desnaturalizante (desviación estándar de la media mostrada para cada uno). Todas las imágenes de gel se visualizaron un imager de gel con un láser de 520 nm y una exposición de 10 minutos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Geles subóptimos y plegado incompleto de Mango IV en un gel nativo del 8%. (A) Una dilución en serie del tipo que se muestra en la Figura 4 para Mango II pero que muestra el efecto del plegado de gel durante el protocolo de tinción. (B) Muestras de gel nativo mango IV que no se les permitió plegar durante el tiempo suficiente en tampón nativo antes de la carga de gel exhiben bandas dobles. De lo contrario, estos resultados son similares a los resultados de Mango IV mostrados en la Figura 4A. Todas las imágenes de gel se visualizaron utilizando un imager de gel con un láser de 520 nm y una exposición de 10 minutos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Los ARN con etiqueta de mango se pueden detectar en presencia de ARN total, utilizando la tinción TO1-Biotina. 8% geles dedesnaturalizantes fueron cargados con 100 ng de ARN total y se ejecutaron durante 30 min. El gel izquierdo estaba teñido con SG y el panel derecho con TO1-Biotin. Para ambos paneles, los carriles etiquetados como E se cargaron con 100 ng de pEcoli-T1 (sin etiqueta Mango) y los carriles etiquetados M se cargaron con 100 ng de pEcoli-RNA Mango (6S RNA etiquetado con una etiqueta Mango I). Las imágenes de gel teñidas con TO1-Biotina se visualizaron utilizando un imager con un láser de 520 nm y una exposición de 10 minutos. Las imágenes de gel teñidas con SG se visualizaron utilizando un imager de gel utilizando un láser de 460 nm y una exposición de 10 minutos. Haga clic aquí para ver una versión más grande de esta figura.
| Porcentaje | VOLUMEN GEL | |||
| 20 mL | 30 mL | 50 mL | ||
| 5% | Un | 4 | 6 | 10 |
| B | 14 | 21 | 35 | |
| C | 2 | 3 | 5 | |
| 6% | Un | 4.8 | 7.2 | 12 |
| B | 13.2 | 19.8 | 33 | |
| C | 2 | 3 | 5 | |
| 8% | Un | 6.4 | 9.6 | 16 |
| B | 11.6 | 17.4 | 29 | |
| C | 2 | 3 | 5 | |
| 10% | Un | 8 | 12 | 20 |
| B | 10 | 15 | 25 | |
| C | 2 | 3 | 5 | |
| 12% | Un | 9.6 | 14.4 | 24 |
| B | 8.4 | 12.6 | 21 | |
| C | 2 | 3 | 5 | |
| 15% | Un | 12 | 18 | 30 |
| B | 6 | 9 | 15 | |
| C | 2 | 3 | 5 | |
| 20% | Un | 16 | 24 | 40 |
| B | 2 | 3 | 5 | |
| C | 2 | 3 | 5 | |
| APS (L) | 48 | 72 | 120 | |
| TEMED (L) | 20 | 30 | 50 | |
Tabla 1: Desnaturalización de la mesa de fundición de gel PAGE. A - Solución A, B - Solución B, C - Solución C.
| Gel de desnaturalización % | BB (movilidad nt) | XC (movilidad en nt) |
| 5 | 35 | 130 |
| 6 | 26 | 106 |
| 8 | 19 | 70-80 |
| 10 | 12 | 55 |
| 20 | 8 | 28 |
| 23 | 5-6 |
Tabla 2: Movilizaciones aproximadas en gel de gel de bromofenol azul (BB) y gel de xileno cianol (XC) colorantes de carga en geles desnaturalizantes de poliacrilamida.
| Porcentaje | VOLUMEN GEL | |||
| 20 mL | 30 mL | 50 mL | ||
| 5% | 1X TBE | 16.5 | 24.75 | 41.25 |
| 40% 29:1 acrilamida:N,N'-metilenbisacrilamida | 2.5 | 3.75 | 6.25 | |
| Glicerol | 1 | 1.5 | 2.5 | |
| 6% | 1X TBE | 16 | 24 | 40 |
| 40% 29:1 acrilamida:N,N'-metilenbisacrilamida | 3 | 4.5 | 7.5 | |
| Glicerol | 1 | 1.5 | 2.5 | |
| 8% | 1X TBE | 15 | 22.5 | 37,5 |
| 40% 29:1 acrilamida:N,N'-metilenbisacrilamida | 4 | 6 | 10 | |
| Glicerol | 1 | 1.5 | 2.5 | |
| 10% | 1X TBE | 14 | 21 | 35 |
| 40% 29:1 acrilamida:N,N'-metilenbisacrilamida | 5 | 7.5 | 12.5 | |
| Glicerol | 1 | 1.5 | 2.5 | |
| 12% | 1X TBE | 13 | 19.5 | 32.5 |
| 40% 29:1 acrilamida:N,N'-metilenbisacrilamida | 6 | 9 | 15 | |
| Glicerol | 1 | 1.5 | 2.5 | |
| 15% | 1X TBE | 11.5 | 17.25 | 28.75 |
| 40% 29:1 acrilamida:N,N'-metilenbisacrilamida | 7.5 | 11.25 | 18.75 | |
| Glicerol | 1 | 1.5 | 2.5 | |
| 20% | 1X TBE | 9 | 13.5 | 22.5 |
| 40% 29:1 acrilamida:N,N'-metilenbisacrilamida | 10 | 15 | 25 | |
| Glicerol | 1 | 1.5 | 2.5 | |
| APS (L) | 48 | 72 | 120 | |
| TEMED (L) | 20 | 30 | 50 | |
Tabla 3: Tabla de fundición de gel page nativa.
Discusión
Una ventaja significativa de la etiqueta fluorescente Mango es que una sola etiqueta se puede utilizar de múltiples maneras. El alto brillo y la afinidad de estos aptámeros los hacen útiles no sólo para la visualización celular 2, sino también para el ARN in vitro o la purificación RNP4. Por lo tanto, la imagen en gel amplía la versatilidad de la etiqueta Mango de una manera sencilla. La sensibilidad por imágenes de gel de mango es ligeramente menor que la de una mancha norte14, pero puede detectar fácilmente 60-120 fmol de muestra de ARN, sin necesidad de transferencia de membrana y pasos de sondeo largos y tediosos. Esto es comparable a la eficiencia de sondeo basada en hibridación que se encontró anteriormente para pequeños ARN en gel15. Mientras que otras metodologías de aptámero sfluorogénicos, en particular, la Espinaca de ARN, tienen una mayor sensibilidad y especificidad9,ninguna tiene actualmente simultáneamente el alto brillo y la afinidad del sistema de aptámero de mango, que permite que una sola etiqueta de ARN sea imágenes celulares, purificación de RNP y ahora imágenes en gel.
Hay algunos pasos críticos en este protocolo de tinción de gel. Cuando se trabaja con soluciones de ARN, las soluciones deben filtrarse estérilmente y utilizarse plástico de un solo uso. Catuion, geles nativos de funcionamiento como complejos o estructuras de ARN se pueden desnaturalizar fácilmente si los niveles de potencia del gel son demasiado altos y dan lugar a calentamiento de gel. Asegúrese de que cualquier cristalería usada esté limpia y no contaminada con RNases. Además, siempre tenga cuidado al transferir y recoger geles, ya que son frágiles y pueden ser propensos a la rotura.
La mancha TO1-Biotina penetra rápidamente en geles, pero los datos presentados aquí también indican que el plegado de Mango IV, en particular, puede ser limitador de velocidad (Figura2 y Figura 3). Utilizando las condiciones indicadas en la sección de protocolo, observamos el comportamiento log-lineal para los cuatro aptámerossobre dos órdenes de magnitud en geles de desnaturalización, haciendo que el método sea útil para la cuantificación (Figura 4C,D). Dado que los pequeños aptámeros de mango utilizados en este estudio se difunden fácilmente fuera de la matriz de gel, esperamos que la cuantificación mejore para construcciones de ARN más largas.
La metodología de imágenes de gel de etiqueta Mango demostrada aquí es robusta y se prevé que pueda ser simplemente extendida en términos de sensibilidad y especificidad. Mango I, II y III se pliegan de forma fiable, mientras que Mango IV no. Si bien no hemos explorado los protocolos de dilina, anticipamos que ese enfoque también podría simplemente mejorar la especificidad. Mientras que fuera del alcance de este trabajo, la etiqueta de fluorescencia y biotina conferida al ARN con etiqueta Mango cuando se utiliza el fluoróforo TO1-Biotin parece muy probable que agilice aún más el análisis y la purificación del gel. Las técnicas secundarias de etiquetado de biotina disponibles comercialmente, por ejemplo, prometen mejorar aún más los límites de detección de este sencillo sistema de etiquetado de mango de ARN. Del mismo modo, parece probable que los complejos nativos de proteína de ARN etiquetados con Mango puedan eluarse de un gel y recuperarse usando cuentas magnéticas de estreptavidina para capturar el complejo de ARN eludado. Esto simplificaría aún más la purificación rutinaria de ARN y complejos de ARN biológicamente importantes mediante la simple conveniencia de añadir una etiqueta de mango al ARN de interés.
Divulgaciones
Está pendiente una patente sobre el sistema fluorogénico Mango.
Agradecimientos
Los autores agradecen a Razvan Cojocaru y Amir Abdolahzadeh por su asistencia técnica y a Lena Dolgosheina por la corrección del manuscrito. La financiación fue proporcionada para este proyecto por una subvención operativa del Consejo Canadiense de Ciencias Naturales e Ingeniería (NSERC) a P.J.U.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.8mm Thick Comb 14 Wells for 30 mL PAGE gels | LabRepCo | 11956042 | |
| 101-1000 µL tips | Fisher | 02-707-511 | |
| 20-200 µL low retention tips | Fisher Scientific | 02-717-143 | |
| Acrylamide:N,N'-methylenebisacrylamide (40% 19:1) | Bioreagents | BP1406-1 | Acute toxicity |
| Acrylamide:N,N'-methylenebisacrylamide (40% 29:1) | Fisher | BP1408-1 | Acute toxicity |
| Agar | Anachemia | 02116-380 | |
| Aluminium backed TLC plate | Sigma-Aldrich | 1164840001 | |
| Amersham Imager 600 | GE Healthcare Lifesciences | 29083461 | |
| Ammonium Persulfate | Biorad | 161-0700 | Harmful |
| BL21 cells | NEB | C2527H | |
| Boric Acid | ACP | B-2940 | |
| Bromophenol Blue sodium salt | Sigma | B8026-25G | |
| Chloloform | ACP | C3300 | |
| Dithiothreitol | Sigma Aldrich Alcohols | D0632-5G | |
| DNase I | ThermoFisher | EN0525 | |
| EDTA Disodium Salt | ACP | E-4320 | |
| Ethanol | Commerial | P016EAAN | |
| Flat Gel Loading tips | Costar | CS004854 | |
| Formamide 99% | Alfa Aesar | A11076 | |
| Gel apparatus set with spacers and combs | LabRepCo | 41077017 | |
| Glass Dish with Plastic lid | Pyrex | 1122963 | Should be large enough to fit your gel piece |
| Glycerol | Anachemia | 43567-540 | |
| HCl | Anachemia | 464140468 | |
| ImageQuanTL | GE Healthcare Lifesciences | 29000605 | |
| IPTG | Invitrogen | 15529-019 | |
| KCl | ACP | P-2940 | |
| MgCl2 | Caledron | 4903-01 | |
| MgSO4 | Sigma-Aldrich | M3409 | |
| NaCl | ACP | S-2830 | |
| NaOH | BDH | BDH9292 | |
| Orbital Rotator | Lab-Line | ||
| Phenol | Invitrogen | 15513-039 | |
| Round Gel Loading tips | Costar | CS004853 | |
| Sodium Phosphate dibasic | Caledron | 8120-1 | |
| Sodium Phosphate monobasic | Caledron | 8180-01 | |
| SYBRGold | ThermoFisher | S11494 | |
| T7 RNA Polymerase | ABM | E041 | |
| TEMED | Sigma-Aldrich | T7024-50 ml | |
| TO1-3PEG-Biotin Fluorophore | ABM | G955 | |
| Tris Base | Fisher | BP152-500 | |
| Tryptone | Fisher | BP1421-500 | |
| Tween-20 | Sigma | P9496-100 | |
| Urea | Fisher | U15-3 | |
| Xylene Cyanol | Sigma | X4126-10G | |
| Yeast Extract | Bioshop | YEX401.500 |
Referencias
- Dolgosheina, E. V., et al. RNA Mango Aptamer-Fluorophore: A Bright, High-Affinity Complex for RNA Labeling and Tracking. ACS Chemical Biology. , (2014).
- Autour, A., et al. Fluorogenic RNA Mango aptamers for imaging small non-coding RNAs in mammalian cells. Nature Communications. 9, 656(2018).
- Dolgosheina, E. V., Unrau, P. J. Fluorophore-binding RNA aptamers and their applications: Fluorophore-binding RNA aptamers. Wiley Interdisciplinary Reviews: RNA. , (2016).
- Panchapakesan, S. S., et al. Ribonucleoprotein Purification and Characterization using RNA Mango. RNA. , 1592-1599 (2017).
- Trachman, R. J. III, et al. Structural basis for high-affinity fluorophore binding and activation by RNA Mango. Nature Chemical Biology. 13, 807(2017).
- Trachman, R. J., et al. Crystal Structures of the Mango-II RNA Aptamer Reveal Heterogeneous Fluorophore Binding and Guide Engineering of Variants with Improved Selectivity and Brightness. Biochemistry. 57, 3544-3548 (2018).
- Trachman, R., et al. Mango-III is a compact fluorogenic RNA aptamer of unusual structural complexity. Nature Chemical Biology. , in review (2018).
- Panchapakesan, S. S. S., Jeng, S. C. Y., Unrau, P. J. RNA complex purification using high-affinity fluorescent RNA aptamer tags. Annals of the New York Academy of Sciences. , (2015).
- Filonov, G. S., Kam, C. W., Song, W., Jaffrey, S. R. In-gel imaging of RNA processing using Broccoli reveals optimal aptamer expression strategies. Chemistry & Biology. 22, 649-660 (2015).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic Acids Research. 15, 8783-8798 (1987).
- A Typical DNase I Reaction Protocol (M0303). NEB. , Available from: https://www.neb.com/protocols/1/01/01/a-typical-dnase-i-reaction-protocol-m0303 (2018).
- Sambrook, J., Russell, D. W. Purification of Nucleic Acids by Extraction with Phenol:Chloroform. Cold Spring Harbor Protocols. 2006 (1), pdb.prot4455 (2006).
- Tuma, R. S., et al. Characterization of SYBR Gold Nucleic Acid Gel Stain: A Dye Optimized for Use with 300-nm Ultraviolet Transilluminators. Analytical Biochemistry. 268, 278-288 (1999).
- Streit, S., Michalski, C. W., Erkan, M., Kleeff, J., Northern Friess, H. Northern blot analysis for detection and quantification of RNA in pancreatic cancer cells and tissues. Nature Protocols. 4, 37-43 (2009).
- Ebhardt, H. A., Unrau, P. J. Characterizing multiple exogenous and endogenous small RNA populations in parallel with subfemtomolar sensitivity using a streptavidin gel-shift assay. RNA. 15, 724-731 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados