Method Article
Fluoreszierende Visualisierung von Mango-markierter RNA in Polyacrylamidgelen über eine Poststaining-Methode
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier präsentieren wir eine empfindliche, schnelle und diskriminierende Post-Gel-Färbungsmethode, um RNAs mit RNA Mango Aptamern I, II, III oder IV abzubilden, wobei entweder native oder denaturierende Polyacrylamid-Gelelektrophorese (PAGE)-Gelverwendet werden. Nach dem Ausführen von Standard-PAGE-Gelen kann Mango-markierte RNA leicht mit TO1-Biotin gefärbt und dann mit allgemein verfügbaren Fluoreszenzlesern analysiert werden.
Zusammenfassung
Native und denaturierende Polyacrylamid-Gele werden routinemäßig verwendet, um die komplexe Mobilität von Ribonukleoprotein (RNP) zu charakterisieren bzw. die RNA-Größe zu messen. Da viele Gel-Imaging-Verfahren unspezifische Flecken oder teure Fluorophorsonden verwenden, sind empfindliche, diskriminierende und wirtschaftliche Gel-Imaging-Methoden sehr wünschenswert. RNA Mango-Kernsequenzen sind kleine (19–22 nt) Sequenzmotive, die, wenn sie durch einen beliebigen RNA-Stamm geschlossen werden, einfach und kostengünstig an eine RNA von Interesse angehängt werden können. Diese Mango-Tags binden mit hoher Affinität und Spezifität an einen Thiazol-orange Fluorophor-Ligand namens TO1-Biotin, der bei Bindung tausendfach fluoreszierender wird. Hier zeigen wir, dass Mango I, II, III und IV verwendet werden können, um RNA gezielt in Gelen mit hoher Empfindlichkeit abzubilden. Nur 62,5 Fmol RNA in nativen Gelen und 125 Fmol RNA in denaturierenden Gelen können durch Einweichen von Gelen in einem bildgebenden Puffer mit Kalium und 20 nM TO1-Biotin für 30 min nachgewiesen werden. Wir demonstrieren die Spezifität des Mango-markierten Systems, indem wir eine Mit mango-markierte 6S-Bakterien-RNA im Kontext einer komplexen Mischung aus gesamtbakterieller RNA abbilden.
Einleitung
Mango ist ein RNA-Tagging-System, bestehend aus einem Satz von vier kleinen fluoreszierenden RNA-Aptamern, die fest (Nanomolarbindung) an einfache Derivate der Thiazol-Orange (TO1-Biotin, Abbildung 1A)1,2,3 binden . Bei der Bindung wird die Fluoreszenz dieses Liganden je nach spezifischem Aptamer um das 1.000- bis 4.000-fache erhöht. Die hohe Helligkeit des Mango-Systems, das für Mango III die des verbesserten grünen Fluoreszenzproteins (eGFP) in Kombination mit der nanomolaren Bindungsaffinität der RNA Mango-Aptamer übertrifft, ermöglicht es, sowohl bei der Bildgebung als auch bei der Reinigung von RNA eingesetzt zu werden. Komplexe2,4.
Die Röntgenstrukturen von Mango I5, II6und III7 wurden zu hoher Auflösung bestimmt, und alle drei Aptamer verwenden ein RNA-Quadruplex, um TO1-Biotin zu binden (Abbildung 1B–D). Die kompakten Kerne aller drei Aptamer werden über kompakte Adaptermotive aus der externen RNA-Sequenz isoliert. Mango I und II verwenden beide einen flexiblen GNRA-ähnlichen Schleifenadapter, um ihre Mango-Kerne mit einem beliebigen RNA-Duplex zu verbinden (Abbildung 1B, C). Im Gegensatz dazu verwendet Mango III ein starres Triplex-Motiv, um seinen Kern mit einer beliebigen RNA-Helix zu verbinden (Abbildung1D,violette Rückstände), während die Struktur von Mango IV derzeit nicht bekannt ist. Da der Ligandenbindungskern jedes dieser Aptamer durch diese Spiraladapter von der externen RNA-Sequenz getrennt ist, scheint es wahrscheinlich, dass sie alle einfach in eine Vielzahl von RNAs integriert werden können. Die bakterielle 6S-konforme RNA (Mango I), Komponenten des Hefespliceooms (Mango I) und der humanen 5S-RNA, U6-RNA und einerC/D-ScaRNA (Mango II und IV) wurden alle erfolgreich auf diese Weise 2,8getaggt, was darauf hindeutet, dass viele biologische RNAs können mit dem RNA Mango Aptamer System markiert werden.
Denaturing und native Gele werden häufig verwendet, um RNAs zu studieren. Denaturing Gels werden häufig verwendet, um die RNA-Größe oder DIE RNA-Verarbeitung zu beurteilen, aber typischerweise erfordern im Falle eines nördlichen Blots beispielsweise mehrere langsame und sequenzielle Schritte, um ein Bild zu erzeugen. Während andere fluorogene RNA-Aptamer wie RNA Spinat und Brokkoli erfolgreich für die Gelbildgebung9eingesetzt wurden, besitzt bisher kein fluorogenes Aptamersystem die hohe Helligkeit und Affinität des Mango-Systems, was es von erheblichem Interesse macht. um die Gel-Imaging-Fähigkeiten von Mango zu untersuchen. In dieser Studie fragten wir uns, ob das RNA-Mango-System einfach auf die Gel-Bildgebung ausgedehnt werden könnte, da die Anregungs- und Emissionswellenlängen von TO1-Biotin (510 nm bzw. 535 nm) für die Bildgebung im eGFP-Kanal geeignet sind, der den meisten fluoreszierenden Gel-Scanning-Instrumentierung.
Das hier vorgestellte Post-Gel-Färbeprotokoll bietet eine schnelle Möglichkeit, Mango-markierte RNA-Moleküle in nativen und denaturing Polyacrylamid-Gel-Elektrophorese-Gelen (PAGE) gezielt zu detektieren. Bei dieser Färbemethode werden Gele in einem Kalium- und TO1-Biotinpuffer einweichen. RNA Mango Aptamer sind G-Quadruplex-basiert und Kalium wird benötigt, um solche Strukturen zu stabilisieren. Mithilfe von RNA, die aus minimalen Mango-kodierenden DNA-Vorlagen transkribiert wurde (siehe Protokollabschnitt), können wir einfach nur 62,5 Fmol RNA in nativen Gelen und 125 Fmol RNA in denaturierenden Gelen erkennen, indem wir ein einfaches Färbeprotokoll verwenden. Im Gegensatz zu häufigen unspezifischen Nukleinsäureflecken (siehe Tabelle der Materialien, die von nun an auf SG verwiesen werden) können wir Mango-markierte RNA auch dann eindeutig identifizieren, wenn hohe Konzentrationen der gesamten nicht markierten RNA in der Probe vorhanden sind.
Protokoll
1. Herstellung der Reagenzien
-
TO1-Biotin Nachflecklösung (Gelfärbelösung)
- 1 L 1 M Phosphatpuffer bei pH 7,2 bei 25 °C durch Zugabe von 342 ml von 1 M Na2HPO4 und 158 ml von 1 M NaH2PO4machen. Stellen Sie den pH-Wert auf 7,2 bei 50 mM ein, indem Sie die entsprechende Phosphatlösung hinzufügen. Sterilfilter mit einem 0,2-mm-Filter und lagern Sie die Lösung in Kunststoffen bei Raumtemperatur.
- 5x Gelfärbelösung (ohne TO1-Biotin) wie folgt vorbereiten. Bilden Sie eine 1 L-Lösung durch Mischen von 247,5 ml ddH2O, 700 ml von 1 M KCl, 50 ml 1 M Phosphatpuffer (pH 7,2) und 2,5 ml Tween 20. Sterilfilter mit einem 0,2-mm-Filter und lagern Sie die Lösung in Kunststoff. Es kann bei Raumtemperatur gelagert werden.
HINWEIS: MgCl2 kann bei Bedarf zu dieser Lösung hinzugefügt werden, da es POTENZIELL RNA-Komplexe stabilisieren kann. Dies wird einen bescheidenen Einfluss auf das Fluoreszenzsignal2haben. - Erstellen Sie eine 1x Gel-Färbungslösung mit der 5x Gel-Färbungslösung aus Schritt 1.1.2 und ergänzen Sie sie unmittelbar vor der Anwendung mit TO1-Biotin-Fluorophor (siehe Materialtabelle)auf eine Endkonzentration von 20 nM. Zum Beispiel 20 ml 5x Gel-Färbungslösung zu 80 ml entionisiertem Wasser hinzufügen, um 100 ml 1x 1x Gel-Färbungslösung herzustellen, und fügen Sie 2 l eines 1 mM TO1-Biotin-Bestands (in Dimethylformamid zubereitet) hinzu.
ANMERKUNG: Der Aussterbekoeffizient für den TO1-Biotinfarbstoff bei 500 nm beträgt 63.000 M-1cm-1, gemessen in Färbelösung1.
-
Denaturing PAGE (2x Denaturierende Gelladelösung und Lösungen A, B, C, Ammoniumpersulfat und Tetramethylethylendiamin)
- 50 ml 2x Denaturierende Gelladelösung durch Mischen von 40 ml Formamid, 0,5 ml 0,5 M Ethylendiamintetraessigsäure (EDTA, pH 8,0) und 9,5 ml ddH2O vorbereiten. Die Lösung kann bei Raumtemperatur gelagert werden. Ladefarbstoffe werden dieser Lösung nicht hinzugefügt, da sie die fluoreszierende Bildgebung verschleiern kann.
- Bereiten Sie 2x denaturierende Gel-Ladefarbe wie folgt vor. Bei der Reinigung von RNA- und DNA-Oligonukleotiden 0,5 ml von 2,5% (w/v) Bromphenolblau (BB) und 0,5 ml 2,5% (w/v) Xyanol (XC) in die in Schritt 1.2.1 beschriebene Lösung geben und 8,5 ml ddH2O statt 9,5 ml hinzufügen. Die Lösung kann bei Raumtemperatur gelagert werden.
- 100 ml Lösung A vorbereiten, indem man 200,2 g Harnstoff (Formelgewicht: 60,06 g/mol) auswägt und zu 312,5 ml 40% 19:1 Acrylamid:N,N'-Methylenbisacrylamid hinzufügt. Rühren Sie es mit einem magnetischen Rührstab mit maximaler Geschwindigkeit, bis gelöst (ca. 3 h) und bilden Sie die Lösung auf 500 ml mit ddH2O. Beachten Sie, dass die Endkonzentrationen 6.667 M Harnstoff und 25% 19:1 Acrylamid:N,N'-Methylenbisacrylamid sind. Bei 4 °C in sauberer Kunststoffaufbewahrung aufbewahren.
- 1 L Lösung B vorbereiten, indem Sie 400,4 g Harnstoff auswiegen und die Lösung auf 1 L mit ddH2O bilden; rühren, bis sich der Harnstoff aufgelöst hat. Lösung B kann bei Raumtemperatur aufbewahrt werden.
- Bereiten Sie Lösung C (10x Tris-borate-EDTA [TBE]) wie folgt vor. Bereiten Sie 4 L 10x TBE vor, indem Sie 432 g Tris-Basis und 220 g Borsäure auswiegen, 160 ml 0,5 M EDTA mit einem pH-Wert von 8 (gefiltert) hinzufügen und die Lösung auf 4 L mit ddH2O aufstellen. Ein großer Vorrat dieses Puffers wird hergestellt, da er auch als Gellaufpuffer verwendet wird.
- 10% Ammoniumpersulfat (APS) vorbereiten, indem 1 g APS in ein Gesamtvolumen von 10 ml ddH2O. bei 4 °C auflösen.
- Halten Sie Tetramethylethylendiamin (TEMED) griffbereit. Lagern Sie es bei 4 °C zusammen mit der APS-Lösung.
-
Native PAGE (2x native Gel-Ladelösung)
- Bereiten Sie 50 ml 2x native Gel-Ladelösung vor, indem Sie 25 ml 100% Glycerin, 10 ml 5x Gel-Färbungslösung und 15 ml ddH2O mischen, um die Lösung auf 50 ml zu bilden.
HINWEIS: Wie im Denaturierungs-Gel-Bereich können diesem Bestand Ladefarbstoffe hinzugefügt werden, deren Zugabe jedoch potenziell das fluoreszierende Signal im Gel verdecken kann und daher vermieden werden sollte.
- Bereiten Sie 50 ml 2x native Gel-Ladelösung vor, indem Sie 25 ml 100% Glycerin, 10 ml 5x Gel-Färbungslösung und 15 ml ddH2O mischen, um die Lösung auf 50 ml zu bilden.
2. Vorbereitung und Beladung von denaturierenden Gelen
- Bereiten Sie ein denaturierendes Gel mit einem angemessenen Prozentsatz vor, indem Sie Tabelle 1folgen. Betrachten Sie das spezifische Gelgusssystem; 30 ml Gele werden häufig verwendet. Der entsprechende Gelprozentsatz kann mit Tabelle 2geschätzt werden: Wählen Sie den Polyacrylamid-Prozentsatz aus, in dem die BB- und XC-Farbstoffe schneller und langsamer als die RNA von Interesse sind, um eine Hochbandtrennung in der entsprechenden Größe zu gewährleisten. bandbreite.
- Mischen Sie die Lösungen A, B und C gemäß Tabelle 1 und fügen Sie APS und TEMED unmittelbar vor dem Gießen des Gels hinzu. Mischen Sie die Lösungen gut in sauberen Kunststoffen.
- Gießen Sie die Gellösung in ein geeignetes Gelgießgerät, nachdem Sie sichergestellt haben, dass alle Komponenten sorgfältig sauber sind. Heben Sie eine Seite des Geräts so an, dass das Gel beim Gießen leicht geneigt ist, um zu vermeiden, dass Luftblasen im Gel selbst gefangen werden.
- Legen Sie den gewünschten Kamm ein und lassen Sie ihn polymerisieren (ca. 30 min). Beobachten Sie die Polymerisation, indem Sie genau nach einer Änderung des Brechungsindexes um die Gelbrunnen suchen. Den Geltank mit 1x Lösung C (1x TBE) aufstellen und den Kamm vorsichtig entfernen. Mit einer Spritze die Brunnen unmittelbar vor der Probenbelastung aussaugen.
- Bereiten Sie denaturierende Proben wie folgt vor. Fügen Sie den RNA-Proben, die von Interesse sind, 2x Denaturierungs-Gel-Ladelösung hinzu, um die denaturierende Gel-Ladelösung 1x zu machen, und wärmedenaturieren Sie bei 95 °C für 5 min mit einem Thermocycler oder Wasserbad.
- Vor dem Beladen der Proben, kühlen Sie sie einige Minuten, bis sie kühl zum Anfassen sind. Laden Sie die Proben, indem Sie sie auf den Boden jedes Brunnens schichten, indem Sie Gel-Ladespitzen verwenden.
HINWEIS: Rundspitzen können für Gele mit einer Dicke von 1 mm oder mehr verwendet werden; flache Spitzen für dünnere Gele verwenden. - Führen Sie die denaturierenden Gele bei Raumtemperatur aus. Stellen Sie sicher, dass die Wattleistung ausreichend niedrig ist, damit die Glasplatten des Gelsystems nicht reißen.
HINWEIS: In unserem Labor reichten dafür 28 W für 20 cm x 16 cm Platten aus.
3. Vorbereitung und Beladung von nativen Gelen
- Bereiten Sie ein natives Gel mit einem entsprechenden Prozentsatz vor, indem Sie auf Tabelle 3verweisen. Betrachten Sie das spezifische Gelgusssystem, aber denken Sie daran, dass 30 ml Gele häufig verwendet werden. Mischen Sie die Lösungen von 1x TBE, 40% 29:1 Acrylamid:N,N'-methylenbisacrylamid und Glycerin nach Tabelle 3, und fügen Sie APS und TEMED unmittelbar vor dem Gießen des Gels hinzu. Fahren Sie wie in den Schritten 2.3 und 2.4 beschrieben vor.
- Bereiten Sie native Proben vor, indem Sie den VON Interesse interessierten RNA-Proben eine 2x native Gel-Ladelösung hinzufügen, um die Lösung 1x herzustellen und sie 100 min lang bei Raumtemperatur inkubieren zu lassen, bevor sie das Gel ausführt, um eine vollständige RNA-Faltung zu gewährleisten.
- Führen Sie das native Gel in einem 4 °C-Kühlraum aus, um sicherzustellen, dass die Wattleistung ausreichend niedrig ist, um das Gel nicht zu erhitzen.
HINWEIS: In unserem Labor reichten dafür 14 W für 20 cm x 16 cm Platten aus.
4. RNA-Vorbereitung durch Abfluss-T7-Transkription
HINWEIS: DNA-Sequenzen, die für die Abflusstranskription10 von RNA Mango-Konstrukten verwendet wurden, wurden kommerziell bestellt. Bei dieser Methode werden DNA-Oligonukleotide, die das umgekehrte Komplement (RC) sowohl der zu transkribierenden Sequenz als auch des T7-Promotors enthalten, zu einer T7-Promotor-Oberstrangsequenz hybridisiert und dann in vitro transkribiert. Unten wird für jedes Oligonukleotid der RC der Mango-Kernsequenz fett und der RC der T7-Promoterregion kursiv dargestellt. Rückstände in regulärer Schrift entsprechen ansonsten willkürlichen komplementären Spiralbereichen, die erforderlich sind, damit der Mangokern richtig gefaltet werden kann.
Mango I: GCA CGT ACT CTC CTC TCC GCA CCG TCC CTT CGT ACG TGC CTA TAG TGA GTC GTA TTA AAG
Mango II: GCA CGT ACT CTC CTC TTC CTC TCC TCT CCT CGT ACG TGC CTA TAG TGA GTC GTA TTA AAG
Mango III: GGC ACG TAC GAA TAT ACC ACA TAC CAA TCC TTC CTT CGT ACG TGC CTA TAG TGA GTC GTA TTA AAG
Mango IV: GCA CGT ACT CGC CTC ATC CTC ACC ACT CCC TCG GTA CGT GCC TAT AGT GAG TCG TAT TAA AG
T7 Top Strang: CTT TAA TAC GAC TCA CTA TAG G
- Präparative Gelreinigung von DNA-Oligonukleotiden
- Für eine DNA-Synthese im Maßstab 0,2 im Mol-Maßstab das degeschützte DNA-Oligonukleotid in 100 l ddH2O und 100 l 2x denaturing loading dye (ab Schritt 1.2.2) wieder aufsetzen. In diesem Fall wird die Einbeziehung von Gelladefarbstoffen in die Ladelösung bei der gleichen Konzentration wie in Schritt 1.2.2 bevorzugt.
- Reinigen Sie das DNA-Oligonukleotid mit einem 50 ml präparativen Schuppen-Denaturierungsgel des entsprechenden Polyacrylamid-Prozentsatzes; Verwenden Sie Tabelle 2, um den Gelprozentsatz auszuwählen. Verwenden Sie beispielsweise ein 8%-Gel für eine 50-nt-Sequenz. Idealerweise sollte Bromphenolblau schneller als das Oligonukleotid laufen, und Xylolcyanol sollte langsamer laufen.
HINWEIS: In unserem Labor wurden solche Gele mit Guss-Spacern gegossen, die 1,5 mm dick waren, und einem Gel-Ladekamm mit Bohrungen, die 2–2,5 cm breit waren. - Laden Sie 100 l der DNA-Oligonukleotid-Lösungen, die in Schritt 4.1.2 pro präparativem Gel hergestellt wurden, wie in den Schritten 2.4–2.7 beschrieben.
- Trocknen Sie die Außenseite des Gels vorsichtig, entfernen Sie die Glasplatten und bedecken Sie beide Seiten des Gels in Plastikfolie. Legen Sie es auf einen fluoreszierenden Bildschirm (Aluminium-gestützte Dünnschicht-Chromatographieplatten, die mit Fluorophor imprägniert sind, sind eine wirtschaftliche Lösung) und verwenden Sie eine kurzwellige UV-Handlampe, um die DNA-Bänder durch UV-Schatten zu visualisieren.
- Ein gestochen scharf definierter Schatten sollte beobachtet werden, wenn die DNA-Synthese von hoher Qualität ist. Markieren Sie die Bänder auf der Plastikfolie mit einem permanenten Marker.
HINWEIS: Schützen Sie Haut und Augen vor UV-Licht und halten Sie die Exposition enden kurz, um eine Beschädigung der Nukleinsäureprobe zu vermeiden. - Legen Sie das Gel auf eine saubere Glasplatte, schneiden Sie die markierten Bänder sorgfältig aus und legen Sie jedes Gelfragment in 400 l von 300 mM NaCl. Elute die DNA über Nacht, mit einem Rotator bei Raumtemperatur.
- Erholen Sie das Eluent in einem sauberen Zentrifugenrohr und fügen Sie 2,5 Äquivalente Ethanol hinzu, um die DNA auszufällen. Vortex gut und legen Sie das Rohr auf -20 °C für 30 min.
- Pellet die Probe in einer Tischzentrifuge bei 156.000 x g für 30 min bei 4 °C. Entfernen Sie vorsichtig den Überstand und setzen Sie das Pellet in ddH2O wieder auf. Verwenden Sie ein Spektralphotometer, um die DNA-Oligonukleotidkonzentration genau zu bestimmen. Bewahren Sie die Probe als 10-M-Lager für komfortschonend bei -20 °C auf.
- Ablaufzeittranskription und Gelreinigung von RNA-Proben
- 5x Nukleosidtriphosphat (NTP) Vorrat vorbereiten, indem sie flüssige NTP-Bestände aus einer Endkonzentration von 40 mM Guanosintriphosphat (GTP, 11.400 M-1cm-1), 25 mM Cytidintriphosphat (CTP, 7.600 M-1cm-1), 25 mM , mischen Adenosintriphosphat (ATP, 5.000 M-1cm-1) und 10 mM Uridintriphosphat (UTP, 10.000 M-1xcm -1); alle Aussterbekoeffizienten bei 260 nm.
HINWEIS: Flüssige oder pulverförmige Vorräte können kommerziell bezogen werden. Wenn Sie Primärbestände aus Pulver zubereiten, stellen Sie den pH-Wert vorsichtig auf einen endgültigen pH-Wert von 7,9 an, indem Sie 1 M NaOH verwenden. Aliquot den Bestand in 1,5 ml Mikrozentrifugenrohren und lagern Sie ihn bei -20 °C. - Bereiten Sie 100 ml 10x T7 Transkriptionspuffer vor, indem Sie 25,6 ml 1 M Tris-HCl, 14,4 ml 1 M Tris-Basis, 26 ml von 1 M MgCl2, 10 ml 10% Triton X-100 und 0,637 g Spermidin mischen. Machen Sie den Bestand auf ein Endvolumen von 100 ml, indem Sie ddH2O hinzufügen.
HINWEIS: Ein letzter pH-Wert von 7,9 der 1x-Lösung sollte mit einem kalibrierten pH-Messgerät bestätigt werden. Der 10x sollte steril gefiltert und bei -20 °C in entsprechend großen Aliquots gelagert werden. - Führen Sie die Transkription wie folgt durch.
- Fügen Sie die folgenden Reagenzien hinzu, um eine Endkonzentration von 1x T7 Transkriptionspuffer mit dem 10x T7 Transkriptionspuffer und ddH2O (ab Schritt 4.2.2), 1x NTPs, 10 mM Dithiothreitol (DTT), 1 Sequenz), 1 -M gelgereinigte Mango-DNA-Sequenz (Schritt 2.1) und T7-RNA-Polymerase-Enzym (1 U/L).
- Wirbel, Spin-down, und inkubieren Sie die Lösung bei 37 °C für 2 h oder bis es trüb wird und ein weißer Niederschlag bildet sich an der Unterseite des Rohres. Eine 50-L-Transkription sollte nach der Gelreinigung zu einer 50-L-RNA von 50 m RNA führen. Fügen Sie ein gleiches Volumen von 2x Denaturierungfarbstoff hinzu und lagern Sie es bei -20 °C, bis er zur Gelreinigung bereit ist.
- Gel-reine die resultierende RNA, wie sie im Abschnitt zur Gelreinigung von DNA-Oligonukleotiden beschrieben wird, indem sie die Schritte 4.1.2-u20124.1.8 befolgt und die RNA-Probe durch die DNA ersetzt.
HINWEIS: RNA ist extrem empfindlich auf RNase-Abbau, so stellen Sie sicher, dass in allen Schritten, Handschuhe und ein sauberes Labormantel zu jeder Zeit getragen werden. Stellen Sie sicher, dass alle Proben in Einweg-Kunststoffen hergestellt und gelagert werden, um sich vor RNase-Kontamination zu schützen, und waschen Sie Glasplatten vor dem Gebrauch sorgfältig mit heißer Seife und Wasser, spülen Sie sie gründlich mit ddH2O aus und trocknen Sie die Glaswaren mit dem Unterstützung von Ethanol aus einer Squeeze-Flasche aufgetragen. - Verwenden Sie 1 L der endgültigen RNA-Probe und bestimmen Sie mit Hilfe eines tropfenbasierten Spektralphotometers die Absorption bei 260 nm. Berechnen Sie mit Hilfe eines Aussterbekoeffizienten, der durch die Methode des nächsten Nachbarn bestimmt wird, die RNA-Konzentration und passen Sie die Probenkonzentration auf 10 m mit ddH2O an. Bewahren Sie die RNA-Proben bei -20 °C auf.
- 5x Nukleosidtriphosphat (NTP) Vorrat vorbereiten, indem sie flüssige NTP-Bestände aus einer Endkonzentration von 40 mM Guanosintriphosphat (GTP, 11.400 M-1cm-1), 25 mM Cytidintriphosphat (CTP, 7.600 M-1cm-1), 25 mM , mischen Adenosintriphosphat (ATP, 5.000 M-1cm-1) und 10 mM Uridintriphosphat (UTP, 10.000 M-1xcm -1); alle Aussterbekoeffizienten bei 260 nm.
- Escherichia coli gesamter Rohnukleinsäureextraktion
HINWEIS: Das folgende Protokoll ist ein Beispiel. Dieses Protokoll verwendet endogen exprimierte Mango I-markierte 6S-RNA aus einem Plasmid in E. coli, und die Induktion von diesem Plasmid wird an anderer Stelle im Detail beschrieben4.- Für die Zwecke dieser Studie bereiten Sie 500 ml induzierte Zellen sowohl für die pEcoli-RNA Mango als auch für die pEcoli-T1-Plasmide vor (die Zellen werden bei einer OD600nm von 1 induziert). Pellet die Zellen und speichern Sie sie bei -80 °C vor der Verwendung.
- Führen Sie die RNA-Extraktion wie folgt durch.
- Nehmen Sie 0,5 ml des induzierten pEcoli-Zellpellets und fügen Sie 500 l ausgleichiertem Phenol hinzu. Dann wirbeln Sie die Probe aus; die Lösung wird milchig weiß. Zentrifuge mit maximaler Geschwindigkeit für 2 min, um die Schichten zu trennen.
- Extrahieren Sie die obere Schicht, die leicht gelb sein wird, und wiederholen Sie Schritt 4.3.2.1, indem Sie ein gleiches Volumen an Phenol, Zentrifugieren und Extrahieren für insgesamt 5x hinzufügen (oder bis die mittlere Schicht, die undurchsichtig weiß ist, verschwindet und nur noch zwei klare Schichten übrig sind).
- Fügen Sie das phenolextrahierte wässrige Volumen zu einem gleichen Volumen von Chloroform, Wirbel und dann Zentrifuge mit maximaler Geschwindigkeit für 2 min.
- Extrahieren Sie die wässrige Schicht und fügen Sie NaCl zu einer Endkonzentration von 300 mM hinzu. Niederschlagen Sie die resultierende Nukleinsäure durch Zugabe von 2,5 Äquivalenten Ethanol, Wirbel, Spin-down und Ausscheide bei -20 °C für mindestens 30 min.
- Pellet in einer Tischzentrifuge bei 15,6 x 1.000 x g bei 4 °C für 30 min. Entfernen Sie den Überstand vorsichtig und setzen Sie das Pellet in 100 l ddH2O wieder auf.
- Fügen Sie diesem Verfahren einen DNase I-Verdauungsschritt hinzu, der dem referenzierten Protokoll11folgt, um die gesamte RNA zu erhalten.
HINWEIS: Wirbel kräftig, bis das Pellet vollständig in ddH2O gelöst ist.
5. Post-Gel-Färbung
- Bereiten Sie 1x Gel Färbung Lösung nach dem Rezept in Schritt 1.1.3 und fügen Sie es zu einem sauberen Glasbehälter, der breit genug ist, um bequem das Gel passen.
HINWEIS: Borosilikatglasbehälter mit Snap-Fit-Deckel nummieren sich gut. - Fügen Sie genügend 1x Gel-Färbungslösung in den Behälter, so dass das Gel vollständig mit der Lösung bedeckt ist und die Flüssigkeit über der Oberseite des Gels sloshes, wenn es auf den Orbitalrotator gelegt wird.
HINWEIS: Der Behälter muss groß genug sein, um das Gel zu passen, so dass es sich bewegen kann und genügend Puffer im Behälter haben kann, um das Gel vollständig zu bedecken. - Sobald das native oder denaturierende Gel fertig ist (Abschnitt 2 bzw. 3), entfernen Sie das Gel aus dem Gerät und schneiden Sie seine Brunnen ab. Es kann nützlich sein, eine Ecke des Gels zur Orientierung später in der Analyse zu entfernen.
- Das Gel vorsichtig auf die in Schritt 5.2 zubereitete 1x Gel-Färbelösung übertragen.
HINWEIS: Die Gele sind zerbrechlich und anfällig für Bruch, also seien Sie vorsichtig, wenn Sie das Gel übertragen. Halten Sie den Deckel auf dem Färbebehälter zu jeder Zeit, um das Gel nicht zu verunreinigen. - Legen Sie das Gel 30 min bei Raumtemperatur mit einer Geschwindigkeit von 100 Umdrehungen pro Minute auf einen Orbitalrotator.
HINWEIS: Stellen Sie sicher, dass das Gel nicht wieder auf sich selbst faltet; Andernfalls kann die RNA aus dem Gel diffundieren und so einen anderen Teil des Gels kennzeichnen.
6. Imaging Mango-markierte RNAs in Gel
- Dekantieren Sie den Bildgebungspuffer vorsichtig. Spülen Sie das Gel schnell mit Wasser, so dass genügend Flüssigkeit bleibt, um das Gel leicht mobil im Behälter zu halten.
- Übertragen Sie das Gel vorsichtig auf den Imager. Übertragen Sie, indem Sie die Seiten des Gels sorgfältig aufnehmen und auf das Imager-Fach legen; alternativ kann das Gel langsam auf das Tablett gegossen werden. Stellen Sie sicher, dass sich keine Blasen unter dem Gel befinden und dass sich keine überschüssige Flüssigkeit unter dem Gel befindet. Eine Pipetten, die über das Gel gerollt wird, kann nützlich sein, um überschüssige Flüssigkeit zu entfernen.
HINWEIS: Verwenden Sie ein Papiertuch, um die überschüssige Flüssigkeit aufzusaugen. - Nehmen Sie ein Gelbild, beobachten Fluoreszenz zwischen Anregungswellenlänge 510 nm und Emissionswellenlänge 535 nm (z. B. grünes Licht bei 520 nm Wellenlänge Fluoreszenzeinstellungen auf dem Imager). Stellen Sie sicher, dass die Einstellungen auf dem Gerät vollständig optimiert sind, und befolgen Sie sorgfältig die Anweisungen für das verwendete Instrument.
Ergebnisse
Kurze Mit Mango-Tags versehene RNAs wurden wie im Protokollabschnitt beschrieben vorbereitet. Unter der Annahme, dass die Fluoreszenz bei den Denaturierungsbedingungen aufgrund des Vorhandenseins von Harnstoff in den Gelen am schwierigsten zu beobachten wäre, untersuchten wir zunächst die Resistenz der Mango-Aptamer gegen Harnstoff, der als Nukleinsäure-Denaturierungwirkt wirkt. Wir fanden heraus, dass Mango-Aptamer im Wesentlichen resistent gegen Denaturierung bis zu einer Harnstoffkonzentration von ca. 1 M sind (Abbildung 2A). Vor zugabe einer Gelfärbelösung zu einem denaturierenden Gel beträgt die endletzte Harnstoffkonzentration im Gel 6 M. Eine ausreichende Färbelösung, um diese Konzentration auf 1 M zu verringern, wäre daher optimal, um eine vollständige Mangofluoreszenz für alle vier Aptamer zu gewährleisten. In der Praxis hat das Färbeprotokoll weniger als dieses vollständig optimale Ergebnis erzielt, aber dies könnte bei Bedarf einfach durch mehr Färbelösung oder durch den einfachen Zweck, die Lösung einmal während der Färbung zu ändern, behoben werden.
Sobald die Mango-markierten RNA-Konstrukte in ein denaturierendes Gel gelaufen waren, wurde die Färbezeit für maximale Gelfluoreszenz optimiert, indem drei verschiedene Mango III-Mengen in ein 8%-denaturierendes Gel geladen wurden (Abbildung 2B). Ein Zeitverlauf zeigte, dass nach 5 min Einweichen in Gelfärbungslösung die Fluoreszenz deutlich sichtbar war. Die maximale Fluoreszenz des Mango-III-Konstrukts wurde nach 20–40 min Färbung erreicht, danach begannen die in dieser Studie verwendeten kleinen RNAs aus dem Gel zu diffundieren, was zu einem Verlust des fluoreszierenden Signals führte (Abbildung 2B). Folglich wurden sowohl native als auch denaturierende Gele jeweils 30 min lang gefärbt. Längere RNA-Konstrukte könnten längere Färbungen leicht vertragen, da sie viel weniger wahrscheinlich aus dem Gel diffundieren würden.
Einige der RNA Mango Aptamer falteten sich schneller als andere. Jeder der in dieser Studie verwendeten Mango-Aptamer wurde in einem Gelpuffer inkubiert, der mit 1,5 M Harnstoff und 100 nM TO1-Biotinfarbstoff ergänzt und mit einem Fluorometer analysiert wurde. Mango I, II und III wurden nach 10 min vollständig gefaltet, während Mango IV erst nach 40 min im Wesentlichen gefaltet wurde (Abbildung3A). In Ermangelung von Harnstoff war das Falten viel schneller als erwartet (Abbildung3B). Um sicherzustellen, dass die nativen Gelproben vollständig gefaltet wurden, haben wir Proben für 100 min vorbezichtigen, bevor wir sie in native Gele laufen lassen. Praktisch deuten die Daten in Abbildung 3 darauf hin, dass diese Zeit je nach verwendeter Mango-Aptamer erheblich reduziert werden könnte.
Sobald das Protokoll optimiert wurde, um RNA Mango Aptamer Fluoreszenz zu erkennen, wurde die Empfindlichkeit der Poststaining-Methode für jede der Mango-Varianten in beiden nativen Gelen bestimmt. Für jede der vier Mangos in nativen Gelen wurden Einzelbänder beobachtet, die gut gefalteten RNAs entsprachen (Abbildung 4A). Bei serieller Verdünnung konnten nur 62,5 Fmol Mango II beobachtet werden, während nur 125 Fmol Mango I, III und IV leicht visualisiert wurden. Die Quantifizierung der nativen Gele war loglinear über etwa 1,5 Größenordnungen, wobei Mango I, II und IV sich linearer verhielten als Mango III (Abbildung 4B).
Die Ergebnisse der denaturierenden Gele waren etwas weniger empfindlich als die nativen Gele, aber linearer. Nur 125 Fmol mango II und III wurden leicht zu erkennen (Abbildung 4C). Interessanterweise deutete die Quantifizierung (Abbildung 4D) darauf hin, dass denaturierende Gele loglinear oder zwei Größenordnungen waren. Wir gehen davon aus, dass im Gegensatz zu den nativen Gelen, bei denen RNA-Falten während des Gellaufprozesses möglicherweise einer partiellen Denaturierung unterzogen wurden, das Vorhandensein von Harnstoff im denaturierenden Gel eine homogenere Möglichkeit bieten könnte, die Aptamer zu falten, sobald sie in TO1-Biotin Färbelösung.
Wie bei allen Gelfärbemethoden kann das Gel, wenn das Gel nicht sorgfältig in den Behälter überführt wird oder die Drehgeschwindigkeit während der Färbezeit zu hoch ist, auf sich selbst zurückklappen (Abbildung 5A). Dies kann dazu führen, dass die Mango-markierte RNA-Probe von einer Stelle des Gels an eine andere diffundiert, aber in der Praxis leicht vermieden werden kann. In nativen Gelen und insbesondere bei Mango IV haben wir beobachtet, dass sich unvollständige Faltung im Auftreten mehrerer Bänder manifestieren kann, die vermutlich teilweise/fehlgefalteten RNA-Konformationen (Abbildung 5B) entsprechen, die sich aus kürzeren Faltzeiten ergeben. Faltprobleme in nativen Gelen können vermieden werden, indem RNA-Proben entsprechend wie zuvor beschrieben vorinkubiert und native Gele in einem kalten Raum ausgeführt werden. Bei der Denaturierung von Gelen, bei denen SICH RNA in situ innerhalb des Gels faltet, war Fehlfaltung kein signifikantes Problem. Schließlich wurde in Abwesenheit von RNA sehr wenig Hintergrundfluoreszenz in beiden Gel-System beobachtet.
Als nächstes wurde die Spezifität des RNA Mango-Tags untersucht, indem die 6S-konforme RNA in Bakterien überexzessiert wurde. Diese RNA wurde zuvor mit Mango I (Abbildung 1E)4getaggt. Bakterielle Zellen wurden entweder mit dem pEcoli-RNA Mango-Plasmid (daher M-Plasmid) oder dem pEcoli-T1-Plasmid als Negativkontrolle (daher E-Plasmid) transformiert. Transformierte Zellen wurden in flüssiger Lysogenesy-Brühe medium angebaut, bis eine OD600 von 1,0 erreicht wurde. Die Kulturen wurden dann mit 50 M Isopropyl -D-1-Thiogalactopyranosid (IPTG) für 40 min induziert. Die Zellen wurden über Zentrifugation bei 6.000 x g für 15 min geerntet. Total RNA wurde mit Phenol-Phenol-Chloroform-Extraktion aus Zellpellets12 extrahiert, wie im Protokollabschnitt beschrieben. Die gesamten RNA-Proben wurden durch Ethanolfällung konzentriert und dann mit DNase I nach dem Protokoll behandelt, um DNA11zu entfernen. Vor der Verwendung wurde die RNA durch Ethanolfällung konzentriert.
Die gesamte bakterielle RNA wurde in 8% denaturierende Gele (Abbildung6) ausgeführt und entweder mit SG13 oder TO1-Biotin gefärbt. Für SG-Färbung wurden 10 l von 10.000x SG zu 100 ml Gelpuffer hinzugefügt; Andernfalls war das Färbeprotokoll identisch mit dem für TO1-Biotin verwendeten. Wie erwartet, wurde eine starke SG-Färbung für eine Vielzahl von RNAs beobachtet, am deutlichsten für ribosomale (rRNA) und Transfer-RNAs (tRNA) (Abbildung6, linkes Panel). Während die Mango-abhängige Färbung (M-Spuren) in diesen SG-gefärbten Gelen zu sehen war, konnten sie angesichts des komplexen Färbemusters, das mit diesem universellen Fleck beobachtet wurde, nicht eindeutig identifiziert werden. Im Gegensatz dazu haben die TO1-Biotin-gefärbten Gele Mango-abhängige Bänder als die prominentesten Bänder hervorgehoben. Nur die ribosomalen RNA-Bänder stehen im Wettbewerb mit den 6S Mango-abhängigen Bands. Eine Reihe von nicht spezifisch gefärbten Bändern konnte ebenfalls beobachtet werden. Dennoch waren die Mango-abhängigen Bands wieder dominant und hatten nur die rRNA- und tRNA-Bands als schwach konkurrierende Konkurrenten (Abbildung6, rechtes Panel).

Abbildung 1: Mango-Aptamer-System. (A) TO1-Biotin fluorophor. (B) Mango I. (C) Mango II. (D) Mango III. Die Panels B–D zeigen die sekundäre Struktur jedes Aptamers. P1 ist ein beliebiger Stamm. Die GNRA-ähnliche Stammschleife (hier GAAA) aus Mango I und II ist rot dargestellt, das Triplex-Motiv von Mango III in Violett. (E) Die 6S regulatorische RNA mit Mango I getaggt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
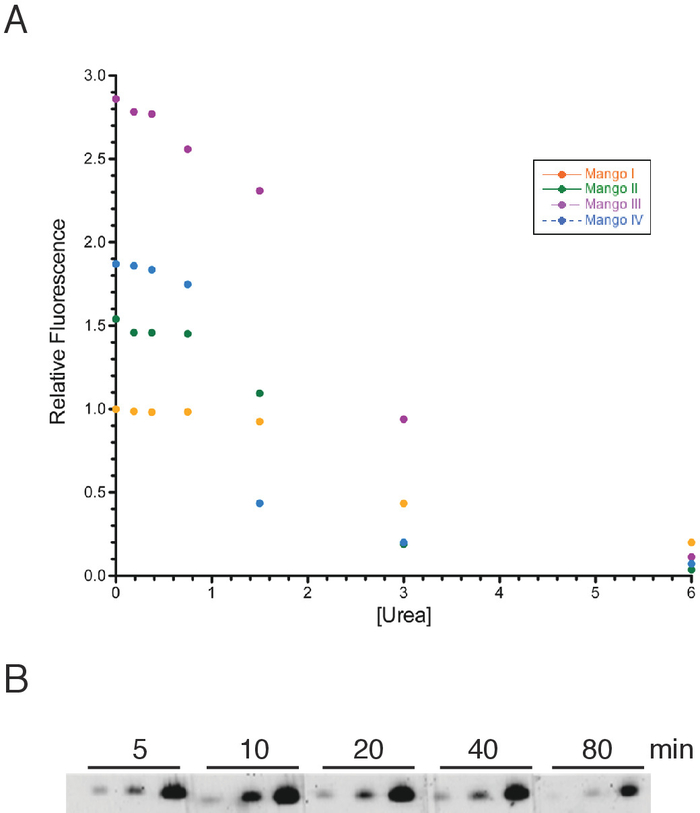
Abbildung 2: Wirkung von Harnstoff auf Mango-Aptamerfluoreszenz und optimale Färbezeiten. (A) Eine Harnstofftitration mit 50 nM RNA Mango I (orange Kreise), Mango II (grüne Kreise), Mango III (lila Kreise) und Mango IV (blaue Kreise), zusammen mit 100 nM TO1-Biotinfarbstoff und bei den angegebenen Harnstoffkonzentrationen. Die Proben wurden 40 min lang inkubiert, bevor die Fluoreszenz bei einer Anregungswellenlänge von 510 nm und einer Emissionswellenlänge von 535 nm gelesen wurde. (B) Mango III RNA wurde in ein 8%-Denaturierungsgel geladen und mit einer Gellösung mit 20 nM endigem TO1-Biotinfarbstoff gebeizt. Für jeden angegebenen Zeitpunkt wurden 0,064, 0,32 und 1,6 pmol Mango III-RNA von links nach rechts verwendet. Das Gelbild wurde mit einem Fluoreszenz-Imager mit einem 520 nm Laser und einer 10 min Belichtung visualisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Faltzeiten der Mango-Aptamer in Gegenwart und Abwesenheit von Harnstoff. (A) In Gegenwart von 1,5 M Harnstoff und 100 nM TO1-Biotin wurden Fluoreszenzzeitkurse mit 50 nM jedes RNA Mango Konstrukts (RNA Mango I: orange Punkte, Mango II: grüne Punkte, Mango III: violette Punkte und Mango IV: blaue Punkte) durchgeführt. (B) Identisch mit Panel A, außer in Ermangelung von Harnstoff. Alle Kurse wurden bei Raumtemperatur durchgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Fluoreszierende Bilder von nativen und denaturierenden PAGE mit RNA Mango-Konstrukten. (A) Ein 8% natives Gel mit seriell verdünnten RNA Mango-Konstrukten. Die Bahnen I, II, III und IV enthalten jeweils 8 pmol Endmengen von RNA Mango I, II, III und IV. Die rechten Paneele sind zweifach serielle Verdünnungen, die 4 pmol, 2 pmol, 1 pmol, 0.5 pmol, 0.25 pmol, 0.125 pmol und 0.0625 pmol von mango I, II, III oder IV enthalten, wie angegeben. Lane 12 enthält keine RNA. (B) Quantifizierung von drei Wiederholungen des nativen Gels (Standardabweichung des jeweils dargestellten Mittelwerts). (C) Ein 8%-Denaturierungsgel mit den gleichen Proben, die wie in Panel Ageladen sind, mit Ausnahme der Tatsache, dass anstelle der nativen Gelladelösung eine Denaturierungslösung für die Gelbeladung verwendet wurde. (D) Drei quantifizierte Wiederholungen des denaturierenden Gels (Standardabweichung des jeweils dargestellten Mittelwerts). Alle Gelbilder wurden mit einem Gel-Imager mit einem 520 nm Laser und einer Belichtung von 10 min visualisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Suboptimale Gele und unvollständige Faltung von Mango IV in einem 8% nativen Gel. (A) Eine serielle Verdünnung der in Abbildung 4 für Mango II dargestellten Art, die jedoch die Wirkung der Gelfaltung während des Färbeprotokolls zeigt. (B) Mango IV native Gelproben, die vor dem Gelladen nicht lange genug in nativem Puffer falten durften, weisen doppel Bänder auf. Andernfalls ähneln diese Ergebnisse den Mango IV-Ergebnissen in Abbildung 4A. Alle Gelbilder wurden mit einem Gel-Imager mit einem 520 nm Laser und einer Belichtung von 10 min visualisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Mango-markierte RNAs können in Gegenwart von Gesamt-RNA mit TO1-Biotin-Färbung nachgewiesen werden. 8% denaturierende Gele wurden mit 100 ng Gesamt-RNA geladen und wurden 30 min lang betrieben. Das linke Gel wurde mit SG und das rechte Panel mit TO1-Biotin befleckt. Für beide Panels wurden Bahnen mit der Bezeichnung E mit 100 ng pEcoli-T1 (kein Mango-Tag) und Bahnen mit der Bezeichnung M mit 100 ng pEcoli-RNA Mango (6S-RNA mit mango I-Tag) beladen. TO1-Biotin-gefärbte Gelbilder wurden mit einem Imager mit einem 520 nm Laser und einer Belichtung von 10 min visualisiert. SG-gefärbte Gelbilder wurden mit einem Gel-Imager mit einem 460 nm Laser und einer Belichtung von 10 min visualisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| prozente | GEL-VOLUMEN | |||
| 20 ml | 30 ml | 50 ml | ||
| 5% | pro | 4 | 6 | 10 |
| B | 14 | 21 | 35 | |
| c | 2 | 3 | 5 | |
| 6% | pro | 4.8 | 7,2 | 12 |
| B | 13,2 | 19,8 | 33 | |
| c | 2 | 3 | 5 | |
| 8% | pro | 6,4 | 9.6 | 16 |
| B | 11,6 | 17,4 | 29 | |
| c | 2 | 3 | 5 | |
| 10% | pro | 8 | 12 | 20 |
| B | 10 | 15 | 25 | |
| c | 2 | 3 | 5 | |
| 12% | pro | 9.6 | 14,4 | 24 |
| B | 8,4 | 12,6 | 21 | |
| c | 2 | 3 | 5 | |
| 15% | pro | 12 | 18 | 30 |
| B | 6 | 9 | 15 | |
| c | 2 | 3 | 5 | |
| 20% | pro | 16 | 24 | 40 |
| B | 2 | 3 | 5 | |
| c | 2 | 3 | 5 | |
| APS (L) | 48 | 72 | 120 | |
| TEMED (L) | 20 | 30 | 50 | |
Tabelle 1: Denaturing PAGE Gelgusstisch. A = Lösung A, B = Lösung B, C = Lösung C.
| Denaturing Gel % | BB (-Mobilität nt) | XC (-Mobilität in nt) |
| 5 | 35 | 130 |
| 6 | 26 | 106 |
| 8 | 19 | 70-80 |
| 10 | 12 | 55 |
| 20 | 8 | 28 |
| 23 | 5-6 |
Tabelle 2: Ungefähre Gelmobilitäten von Bromphenolblau (BB) und Xylolcyanol (XC) Gel-Ladefarbstoffen in Polyacrylamid-Denaturierungsgelen.
| prozente | GEL-VOLUMEN | |||
| 20 ml | 30 ml | 50 ml | ||
| 5% | 1X TBE | 16,5 | 24,75 Uhr | 41,25 |
| 40% 29:1 Acrylamid:N,N'-methylenbisacrylamid | 2,5 | 3,75 | 6.25 | |
| Glycerin | 1 | 1.5 | 2,5 | |
| 6% | 1X TBE | 16 | 24 | 40 |
| 40% 29:1 Acrylamid:N,N'-methylenbisacrylamid | 3 | 4,5 | 7,5 | |
| Glycerin | 1 | 1.5 | 2,5 | |
| 8% | 1X TBE | 15 | 22,5 | 37,5 |
| 40% 29:1 Acrylamid:N,N'-methylenbisacrylamid | 4 | 6 | 10 | |
| Glycerin | 1 | 1.5 | 2,5 | |
| 10% | 1X TBE | 14 | 21 | 35 |
| 40% 29:1 Acrylamid:N,N'-methylenbisacrylamid | 5 | 7,5 | 12,5 | |
| Glycerin | 1 | 1.5 | 2,5 | |
| 12% | 1X TBE | 13 | 19,5 | 32,5 |
| 40% 29:1 Acrylamid:N,N'-methylenbisacrylamid | 6 | 9 | 15 | |
| Glycerin | 1 | 1.5 | 2,5 | |
| 15% | 1X TBE | 11,5 | 17.25 Uhr | 28,75 Uhr |
| 40% 29:1 Acrylamid:N,N'-methylenbisacrylamid | 7,5 | 11.25 Uhr | 18.75 Uhr | |
| Glycerin | 1 | 1.5 | 2,5 | |
| 20% | 1X TBE | 9 | 13,5 | 22,5 |
| 40% 29:1 Acrylamid:N,N'-methylenbisacrylamid | 10 | 15 | 25 | |
| Glycerin | 1 | 1.5 | 2,5 | |
| APS (L) | 48 | 72 | 120 | |
| TEMED (L) | 20 | 30 | 50 | |
Tabelle 3: Native PAGE-Gelgusstisch.
Diskussion
Ein wesentlicher Vorteil des Mango-Fluoreszenz-Tags ist, dass ein einzelnes Tag auf verschiedene Arten verwendet werden kann. Die hohe Helligkeit und Affinität dieser Aptamer machen sie nicht nur für die Zellvisualisierung 2, sondern auch für die In-vitro-RNA- oder RNP-Reinigung4nützlich. Daher erweitert die Gel-Bildgebung die Vielseitigkeit des Mango-Tags auf einfache Weise. Die Empfindlichkeit der Mango-Gel-Bildgebung ist etwas geringer als die eines nördlichen Blots14, kann aber leicht 60–120 Fmol RNA-Proben erkennen, ohne dass langwierige und mühsame Membranübertragungen und Sondierungsschritte erforderlich sind. Dies ist vergleichbar mit der hybridisierungsbasierten Sondierungseffizienz, die zuvor für kleine RNAs in Gel15gefunden wurde. Während andere fluorogene Aptamer-Methoden – insbesondere RNA-Spinat – eine höhere Empfindlichkeit und Spezifität9aufweisen, hat derzeit keine gleichzeitig die hohe Helligkeit und Affinität des Mango-Aptamer-Systems, das es ermöglicht, ein einzelnes RNA-Tag für die zelluläre Bildgebung, DIE RNP-Reinigung und jetzt die Gel-Bildgebung verwendet werden.
Es gibt ein paar kritische Schritte in diesem Gel-Färbungsprotokoll. Bei der Arbeit mit RNA-Lösungen sollten die Lösungen steril gefiltert und Einweg-Kunststoffverwendeter verwendet werden. Catuion, native Gele als Komplexe oder RNA-Strukturen können leicht denaturiert werden, wenn die Leistungsstufen für das Gel zu hoch sind und zu einer Gelerwärmung führen. Stellen Sie sicher, dass alle verwendeten Glaswaren sauber und nicht mit RNases kontaminiert sind. Darüber hinaus seien Sie immer vorsichtig beim Übertragen und Abholen von Gelen, da sie zerbrechlich sind und anfällig für Bruch sein können.
Der TO1-Biotin-Fleck dringt schnell in Gele ein, aber die hier vorgestellten Daten deuten auch darauf hin, dass insbesondere die Mango IV Faltung die Geschwindigkeit begrenzenkann (Abbildung 2 und Abbildung 3). Anhand der im Protokollabschnitt angegebenen Bedingungen beobachteten wir ein loglineares Verhalten für alle vier Aptamer über zwei Größenordnungen bei der Denaturierung von Gelen, was die Methode für die Quantifizierung nützlich macht (Abbildung 4C,D). Da die kleinen Mango-Aptamer, die in dieser Studie verwendet wurden, leicht aus der Gelmatrix diffundiert wurden, erwarten wir, dass sich die Quantifizierung für längere RNA-Konstrukte verbessert.
Die hier gezeigte Mango-Tag-Gel-Bildgebungsmethodik ist robust und soll in Bezug auf Empfindlichkeit und Spezifität einfach erweitert werden können. Mango I, II und III falten zuverlässig, während Mango IV nicht. Obwohl wir die Detaining-Protokolle nicht untersucht haben, gehen wir davon aus, dass ein solcher Ansatz auch einfach die Spezifität verbessern könnte. Während außerhalb des Rahmens dieser Arbeit, die Fluoreszenz und Biotin-Tag an Mango-markierte RNA bei der Verwendung von TO1-Biotin Fluorophor übertragen scheint sehr wahrscheinlich, um die Gelanalyse und -reinigung weiter zu rationalisieren. Kommerziell erhältliche sekundäre Biotin-Etikettierungstechniken versprechen beispielsweise, die Nachweisgrenzen dieses einfachen RNA Mango-Tagging-Systems weiter zu verbessern. Ebenso scheint es wahrscheinlich, dass native Mango-markierte RNA-Proteinkomplexe aus einem Gel eluiert und mit Streptavidin-Magnetperlen zurückgewonnen werden können, um den eluierten RNA-Komplex zu erfassen. Dies würde die routinemäßige Reinigung biologisch wichtiger RNAs und RNA-Komplexe durch den einfachen Zweck, der RNA von Interesse ein Mango-Tag hinzuzufügen, weiter vereinfachen.
Offenlegungen
Ein Patent für das Fluorogensystem Mango ist anhängig.
Danksagungen
Die Autoren danken Razvan Cojocaru und Amir Abdolahzadeh für ihre technische Unterstützung und Lena Dolgosheina für das Korrekturlesen des Manuskripts. Die Finanzierung dieses Projekts wurde von einem Betriebskostenzuschuss des Canadian Natural Sciences and Engineering Research Council (NSERC) an P.J.U. bereitgestellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.8mm Thick Comb 14 Wells for 30 mL PAGE gels | LabRepCo | 11956042 | |
| 101-1000 µL tips | Fisher | 02-707-511 | |
| 20-200 µL low retention tips | Fisher Scientific | 02-717-143 | |
| Acrylamide:N,N'-methylenebisacrylamide (40% 19:1) | Bioreagents | BP1406-1 | Acute toxicity |
| Acrylamide:N,N'-methylenebisacrylamide (40% 29:1) | Fisher | BP1408-1 | Acute toxicity |
| Agar | Anachemia | 02116-380 | |
| Aluminium backed TLC plate | Sigma-Aldrich | 1164840001 | |
| Amersham Imager 600 | GE Healthcare Lifesciences | 29083461 | |
| Ammonium Persulfate | Biorad | 161-0700 | Harmful |
| BL21 cells | NEB | C2527H | |
| Boric Acid | ACP | B-2940 | |
| Bromophenol Blue sodium salt | Sigma | B8026-25G | |
| Chloloform | ACP | C3300 | |
| Dithiothreitol | Sigma Aldrich Alcohols | D0632-5G | |
| DNase I | ThermoFisher | EN0525 | |
| EDTA Disodium Salt | ACP | E-4320 | |
| Ethanol | Commerial | P016EAAN | |
| Flat Gel Loading tips | Costar | CS004854 | |
| Formamide 99% | Alfa Aesar | A11076 | |
| Gel apparatus set with spacers and combs | LabRepCo | 41077017 | |
| Glass Dish with Plastic lid | Pyrex | 1122963 | Should be large enough to fit your gel piece |
| Glycerol | Anachemia | 43567-540 | |
| HCl | Anachemia | 464140468 | |
| ImageQuanTL | GE Healthcare Lifesciences | 29000605 | |
| IPTG | Invitrogen | 15529-019 | |
| KCl | ACP | P-2940 | |
| MgCl2 | Caledron | 4903-01 | |
| MgSO4 | Sigma-Aldrich | M3409 | |
| NaCl | ACP | S-2830 | |
| NaOH | BDH | BDH9292 | |
| Orbital Rotator | Lab-Line | ||
| Phenol | Invitrogen | 15513-039 | |
| Round Gel Loading tips | Costar | CS004853 | |
| Sodium Phosphate dibasic | Caledron | 8120-1 | |
| Sodium Phosphate monobasic | Caledron | 8180-01 | |
| SYBRGold | ThermoFisher | S11494 | |
| T7 RNA Polymerase | ABM | E041 | |
| TEMED | Sigma-Aldrich | T7024-50 ml | |
| TO1-3PEG-Biotin Fluorophore | ABM | G955 | |
| Tris Base | Fisher | BP152-500 | |
| Tryptone | Fisher | BP1421-500 | |
| Tween-20 | Sigma | P9496-100 | |
| Urea | Fisher | U15-3 | |
| Xylene Cyanol | Sigma | X4126-10G | |
| Yeast Extract | Bioshop | YEX401.500 |
Referenzen
- Dolgosheina, E. V., et al. RNA Mango Aptamer-Fluorophore: A Bright, High-Affinity Complex for RNA Labeling and Tracking. ACS Chemical Biology. , (2014).
- Autour, A., et al. Fluorogenic RNA Mango aptamers for imaging small non-coding RNAs in mammalian cells. Nature Communications. 9, 656(2018).
- Dolgosheina, E. V., Unrau, P. J. Fluorophore-binding RNA aptamers and their applications: Fluorophore-binding RNA aptamers. Wiley Interdisciplinary Reviews: RNA. , (2016).
- Panchapakesan, S. S., et al. Ribonucleoprotein Purification and Characterization using RNA Mango. RNA. , 1592-1599 (2017).
- Trachman, R. J. III, et al. Structural basis for high-affinity fluorophore binding and activation by RNA Mango. Nature Chemical Biology. 13, 807(2017).
- Trachman, R. J., et al. Crystal Structures of the Mango-II RNA Aptamer Reveal Heterogeneous Fluorophore Binding and Guide Engineering of Variants with Improved Selectivity and Brightness. Biochemistry. 57, 3544-3548 (2018).
- Trachman, R., et al. Mango-III is a compact fluorogenic RNA aptamer of unusual structural complexity. Nature Chemical Biology. , in review (2018).
- Panchapakesan, S. S. S., Jeng, S. C. Y., Unrau, P. J. RNA complex purification using high-affinity fluorescent RNA aptamer tags. Annals of the New York Academy of Sciences. , (2015).
- Filonov, G. S., Kam, C. W., Song, W., Jaffrey, S. R. In-gel imaging of RNA processing using Broccoli reveals optimal aptamer expression strategies. Chemistry & Biology. 22, 649-660 (2015).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic Acids Research. 15, 8783-8798 (1987).
- A Typical DNase I Reaction Protocol (M0303). NEB. , Available from: https://www.neb.com/protocols/1/01/01/a-typical-dnase-i-reaction-protocol-m0303 (2018).
- Sambrook, J., Russell, D. W. Purification of Nucleic Acids by Extraction with Phenol:Chloroform. Cold Spring Harbor Protocols. 2006 (1), pdb.prot4455 (2006).
- Tuma, R. S., et al. Characterization of SYBR Gold Nucleic Acid Gel Stain: A Dye Optimized for Use with 300-nm Ultraviolet Transilluminators. Analytical Biochemistry. 268, 278-288 (1999).
- Streit, S., Michalski, C. W., Erkan, M., Kleeff, J., Northern Friess, H. Northern blot analysis for detection and quantification of RNA in pancreatic cancer cells and tissues. Nature Protocols. 4, 37-43 (2009).
- Ebhardt, H. A., Unrau, P. J. Characterizing multiple exogenous and endogenous small RNA populations in parallel with subfemtomolar sensitivity using a streptavidin gel-shift assay. RNA. 15, 724-731 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten