Method Article
Imagem latente confocal de RNA Double-Stranded e receptores de reconhecimento padrão na infecção pelo vírus de RNA de sentido negativo
Neste Artigo
Resumo
Double-stranded do RNA produzido durante a replicação de vírus de RNA pode ser reconhecido por receptores de reconhecimento padrão para induzir uma resposta imune inata. Para vírus de RNA de sentido negativo, a interação entre o baixo nível de dsRNA e PRRs permanece obscura. Nós desenvolvemos um método de microscopia confocal para visualizar dsRNA arenavírus e PRR em células individuais.
Resumo
(Ds) do RNA double-stranded é produzido como um intermediário replicative durante a infecção de vírus de RNA. Reconhecimento do dsRNA por receptores de reconhecimento de padrão anfitrião (PRRs) tais como o ácido retinoico (RIG-eu) como receptores (RLRs) RIG-eu e o melanoma proteína associada a diferenciação 5 (MDA-5) leva para a indução da resposta imune inata. A formação e a distribuição intracelular de dsRNA positivo-infecção por vírus de RNA de sentido tem sido bem caracterizada por microscopia. Muitos vírus de RNA de sentido negativo, incluindo alguns arenaviruses, desencadear a resposta imune inata durante a infecção. No entanto, vírus de RNA de sentido negativo foram pensados para produzir baixos níveis de dsRNA, que impede o estudo da imagem do reconhecimento do PRR do dsRNA viral. Além disso, experimentos de infecção com alta patogenicidade arenaviruses devem ser realizados em instalações de alta contenção biossegurança nível (BSL-4). A interação entre viral RNA e PRRs para alta patogenicidade vírus RNA é desconhecida devido os desafios técnicos adicionais que os investigadores precisam enfrentar nas instalações da BSL-4. Recentemente, um anticorpo monoclonal (Mab) (clone 9 5) originalmente usado para detecção de pan-enterovírus foi encontrado para detectar especificamente dsRNA com uma maior sensibilidade do que os tradicionais anticorpos anti-dsRNA J2 ou K1. Neste documento, utilizando o 9 5 anticorpo, descrevemos um protocolo de microscopia confocal que tem sido usado com sucesso para visualizar dsRNA, proteína viral e PRR simultaneamente em células individuais, infectados por arenavírus. O protocolo é também adequado para estudos de dsRNA e distribuição do PRR em células patogênicas arenavírus infectado em instalações de BSL4 de imagem.
Introdução
O passo inicial da indução da resposta imune inata é acolhimento reconhecimento de double-stranded do RNA (ds) por receptores de reconhecimento padrão (PRRs) tais como o ácido retinoico (RIG-eu) como receptores (RLRs) RIG-eu e o melanoma associada a diferenciação proteína 5 (MDA-5)1. Para vírus de RNA de sentido positivo, dsRNA pode geralmente ser prontamente detectado usando o J2 ou K1 anti-dsRNA antibodies monoclonal (Mab)2. Interação entre dsRNA e PRRs em vírus de RNA positivo-vertente, como picornavírus, tem sido caracterizada utilizando microscopia confocal3. No entanto, para vírus de RNA sentido negativo, visualização e caracterização do PRR e dsRNA interação tem sido dificultado pela ausência de anticorpos sensíveis ao dsRNA. Hibridação in situ fluorescente (FISH) de RNA foi aplicada para a visualização de viral RNA e PRRs4. No entanto, a metodologia de peixe requer o conhecimento do destino sequência de RNA e pode não ser compatível com PRR co coloração. Recentemente, a 5 de 9 Mab, que foi originalmente desenvolvido para o diagnóstico da infecção por enterovírus-pan, foi encontrado para ser mais sensível do que o Mab J2 e pode facilmente detectar dsRNA no sentido negativo de5,de infecção de vírus de RNA6. Assim, o Mab 9 5 é um romance e uma ferramenta útil para estudar a replicação viral e a interação entre o PRR e o RNA viral para vírus de RNA sentido negativo.
Arenaviruses é uma família de vírus de RNA de single-stranded, negativo-sentido, que inclui vários agentes patogénicos humanos, tais como vírus de Lassa (LASV), vírus Junín (JUNV) e vírus Machupo (MACV), que causam doenças graves de febre hemorrágica em seres humanos7. Dados clínicos de casos graves e fatais de febre hemorrágica Argentina causada por arenavírus do novo mundo JUNV apresentam níveis anormalmente altos de soro IFN-α8,9. Mostramos que o arenaviruses NW patogénicos (JUNV e MACV), mas não a patogenicidade velho mundo arenavírus, LASV, induzem um tipo eu resposta de interferon (IFN) em humanos células dendríticas derivadas de monócitos10. Além disso, RIG-I é um dos sensores mediando tipo I resposta IFN em células infectadas JUNV11. Também achamos que o receptor de R (PKR) proteína quinase, que é tradicionalmente conhecido por reconhecimento de dsRNA, é ativado em patogenicidade NW arenavírus infecção12. Para entender melhor o mecanismo de resposta IFN vírus específicos durante a infecção arenavírus, visamos desenvolver um protocolo para visualizar a interação entre dsRNA viral e os PRRs citoplasmáticas.
Experimentos de infecção com patogenicidade JUNV, MACV e LASV precisam ser executada em Biossegurança nível 4 instalações (BSL-4). Assim, além do nível baixo presumivelmente de dsRNA formado na infecção por arenavírus, cumprindo as exigências de biossegurança é outro desafio técnica ao realizar estudos de imagiologia para estes vírus de alta patogenicidade. Utilizando o 9 5 anticorpo e Candid1 # estirpe utilizada na vacina de JUNV, um protocolo baseado em microscopia confocal é descrito neste relatório, que tem sido usada com sucesso para visualizar dsRNA, proteína viral e PRR simultaneamente em células infectadas por arenavírus em BSL2 laboratórios. O protocolo é também adequado para visualização da distribuição intracelular de dsRNA e PRR durante infecção patogénica arenavírus em instalações de BSL4.
Protocolo
1. preparação de células A549 e infecção JUNV
- Semente de 2 x 105 pulmão humano A549 células epiteliais em poli-D-lisina (PDL) revestido as lamelas de vidro em placas boas 12 24 horas antes da infecção.
- Preparar alíquotas de 150 mL de JUNV13 em uma multiplicação de infecção (MOI) de 1,0 placa formando unidade por célula diluída em mídia de médio (DMEM) modificado Eagle de Dulbecco suplementada com 2% de soro fetal bovino (FBS) e 1% de penicilina e estreptomicina (P/S ).
- Remova a mídia de cultura de células. Adicionar o inóculo de vírus em cada lamela contendo bem e incubar durante 1,5 h a 37 ° C. Agite as placas cada 15 min.
- Remover o inóculo de vírus, adicionar 1 mL de DMEM suplementado com 5% FBS e 1% P/S. Incubar a placa a 37 ° C para o ponto de tempo desejado.
2. fixação e imunocoloração
- Aspire os meios de comunicação. Enxague as células, adicionando 1 mL de solução salina tamponada fosfato (PBS) suplementada com cálcio e magnésio para cada poço.
- Remova a PBS. Adicione 1 mL de metanol (MeOH) pre-refrigerado a-20 ° C e incubar a-20 º C ou em gelo seco por 15 min.
- Remova MeOH.
- Adicione 1 mL de PBS a cada poço e amostras de lavagens em 4 ° C, com um balanço suave 5 min. repetir a lavagem total de 4 vezes.
- Lave as células fixas em lamelas em 1 mL de 0,2% t-octylphenoxypolyethoxyethanol por 5 min a 4 ° C, com balanço suave.
- Adicione 1 mL de PBS a cada poço. Lave as amostras a 4 ° C por 5 min com balanço suave. Repita a etapa de lavagem para total de 4 vezes.
- Para detectar dsRNA e MDA5, Incube as amostras em 200 mL de anticorpos primários diluído em 3% albumina de soro bovino (BSA). Diluir o anticorpo anti-dsRNA 9 5 a uma diluição de 1:2 e diluir o anticorpo anti-MDA-5 em 1: 250. Incube com suave balançar a 4 ° C durante a noite.
- Remova os anticorpos primários e lave cada uma com 1 mL de PBS por 5 min com suave balançar no RT. repetir esta lavagem quatro vezes mais.
- Adicionar 200 mL de anticorpos secundários (1:2, diluição 000) diluído em 3% BSA e incubar a RT por 1h.
- Remover anticorpos secundários e lave cada uma com 1 mL de PBS por 5 min com suave balançar no RT. repetir esta lavagem quatro vezes mais.
- Para detectar a cadeia JUNV (NP) e RIG-, adicionar 200 mL de anticorpos conjugados diluído em 3% BSA. Diluir o NP conjugado anti-JUNV (AG-12) em 1:1,000 e incubar as amostras por 2 h em RT com balanço suave. Diluir o anti-RIG-I anticorpo conjugado no 1: 500 e incubar a 4 ° C durante a noite com balanço suave.
- Remover anticorpos conjugados e lave cada uma com 1 mL de PBS por 5 min com suave balançar no RT. repetir esta lavagem quatro vezes mais.
- Counterstain as lamelas com DAPI (1:1, 000) por 3 min com balanço suave no RT
- Lave as lamelas 3 vezes, cada vez por 5 min em 1 mL de 0.5% t-octylphenoxypolyethoxyethanol com balanço suave no RT
- Lave duas vezes em 1 mL de PBS durante 5 min cada vez com um balanço suave no RT
- Lave uma vez em 1 mL de DDQ2O por 1 min em RT, balançando suavemente.
- Lamelas de montagem em lâminas de vidro usando a mídia de montagem. Deixe a cura durante a noite.
- Selar os slides com as unhas pintadas e secar por 1h.
- Imagem em microscópio confocal com o x 60 / 1,42 lente de imersão de óleo de abertura numérica usando as mesmas emissões de laser para cada amostra.
- Ao analisar os dados, se necessário, fazer ajustes de brilho e contraste, usando o mesmo ajuste linear para todas as amostras.
Resultados
Este protocolo foi aplicado para estudar a distribuição e o colocalization entre os RLRs (RIG-I e MDA-5) e dsRNA em células infectadas por JUNV. Como mostrado na Figura 1 e Figura 2, a acumulação de dsRNA aumenta ao longo do tempo como progride de infecção viral. Concentrado de MDA-5 (Figura 1) e equipamento-(Figura 2) sinais foram encontrados colocalized com as estruturas punctate do NP e dsRNA.
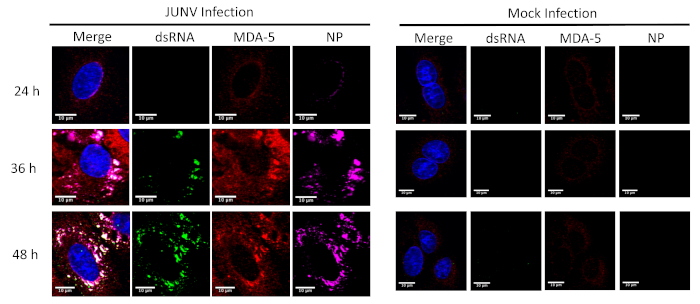
Figura 1: evolução temporal do dsRNA e formação JUNV NP e a distribuição do MDA-5. JUNV-infectados e simulação-infectados A549 células foram fixo, manchadas e criação da imagem de acordo com o protocolo em 24, 36 e 48 horas pós infecção (HPI). Clique aqui para ver uma versão maior desta figura.
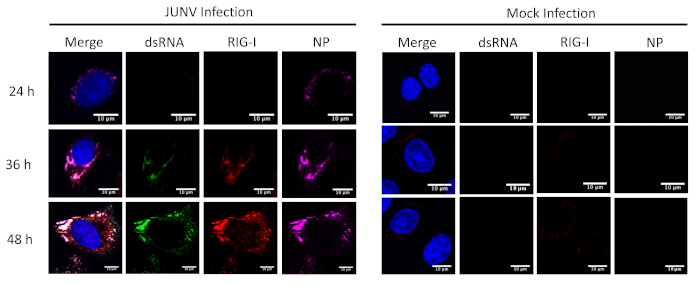
Figura 2: evolução temporal do dsRNA e formação JUNV NP e a distribuição de RIG-I. JUNV-infectados e simulação-infectados A549 células foram fixo, manchadas e criação da imagem de acordo com o protocolo em 24, 36 e 48 HPI. Clique aqui para ver uma versão maior desta figura.
Discussão
Para vírus de RNA de sentido positivo e vírus dsDNA, dsRNA é facilmente detectado com o anticorpo utilizado de anti-dsRNA J2. No entanto, vírus de RNA de sentido negativo acredita-se que produzem o dsRNA em uma baixa ou abaixo do nível de detecção, usando o mesmo anticorpo2. Assim, muitos aspectos da interação viral do RNA e PRR são em grande parte obscuro para vírus de RNA de sentido negativo. Buscou-se mancha para dsRNA na infecção por arenavírus usando o anticorpo J2 mas os sinais de fluorescência não eram diferenciáveis em comparação com a infecção trocista. O Mab 9 5, originalmente usado para detecção de pan-enterovírus, foi encontrado para ser específico para dsRNA e mais sensível do que o J2 anticorpo5. Este anticorpo tem sido usado com sucesso para detectar vírus dsRNA durante a infecção de vírus de RNA, incluindo o protótipo arenavírus coriomeningite vírus5,6,14negativo-sentido. Nesse sentido, nós costumávamos o Mab 9 5 co manchar a presença do dsRNA, NP viral e PRRs a entender sua distribuição e interação em células individuais durante a infecção arenavírus.
Existem vários métodos de fixação que podem ser usados para preservar a estrutura celular. Fixação com MeOH atua por precipitação de proteínas, Considerando que as proteínas de ligações cruzadas paraformaldeído e formol. MeOH é mais eficaz do que os aldeídos em conservar os ácidos nucleicos nas células e fornece imunocoloração de baixo fundo. Metanol também remove lipídios de células e, portanto, permeabilizes as membranas celulares na mesma hora15. Neste protocolo, as células são fixadas usando MeOH gelada. Outros métodos de fixação também foram tentados, incluindo amostras de fixação com metanol seguido por paraformaldeído, formalina, paraformaldeído seguido por metanol e paraformaldeído 4%. No entanto, alta basal nível mancha não específica não foi observada em células infectadas por simulação quando paraformaldeído ou formalina foi usada. O método de fixação ideal é fixação com MeOH a-20 ° C com base na sensibilidade e especificidade dos resultados. Antes mancha do anticorpo primário, amostra de bloqueio com BSA 3% e 5% BSA soro de cabra 10% ou 5% para 30 min a 1 hora foi testado, mas todas resultaram em específico, fundo de coloração. Os melhores resultados foram alcançados sem o bloqueio passo e diretamente usando 3% BSA na diluição anticorpo. Os passos críticos em minimizar os sinais de fundo são o PBS e t-Octylphenoxypolyethoxyethanol lava após a fixação. Enquanto o comercialmente disponível 5 9 anticorpo é diluído pelo vendedor e pronto para direcionar o uso, uma diluição de 1:2 do anticorpo com 3% de BSA também funcionou bem. No caso que o sinal de dsRNA é fraco, o anticorpo pode ser usado sem diluição.
Este protocolo pode ser utilizado para estudar a interação entre dsRNA e PRRs na infecção por arenavírus. Enquanto esta metodologia foi desenvolvida em um ambiente de BSL-2, a fixação de metanol aqui descrita permite completa inactivação do arenavírus. Portanto, o mesmo protocolo pode ser aplicado ao estudo da imagem para arenaviruses de alta patogenicidade (i.e., LASV, JUNV e MACV) em uma facilidade BSL-4. Também é possível aplicar este protocolo para estudos sobre outros vírus de RNA. Para conseguir resultados óptimos, uma modificação do protocolo pode ser necessária.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Gostaríamos de agradecer o UTMB imagem núcleo instalações e Maxim Ivannikov para assistência de microscópio.
Este trabalho foi apoiado pelo serviço de saúde pública conceder RO1AI093445 e RO1AI129198 para SP, premiado UTMB compromisso fundo P84373 de CH e T32 AI007526 para EM.
Materiais
| Name | Company | Catalog Number | Comments |
| APEX Alexa Fluor 647 antibody labeling kit | Invitrogen | A10475 | |
| BSA | Sigma Aldrich | A4503 | |
| DAPI | Cell Signaling | 4083 | 1:1,000 dilution |
| Donkey-anti rabbit Alexa Fluor 594 | Invitrogen | A-11058 | 1:2,000 dilution; Lot #: 1454437 |
| Dulbecco's modified Eagle's medium | Corning | 10-013-CV | |
| Fetal Bovine Serum | Atlanta Bio | S11150 | |
| Glass microscope slides | Fisher | 12-550-15 | |
| Goat-anti mouse Alexa Fluor 488 | Invitrogen | A-11029 | 1:2000 dilution; Lot #: 1874804 |
| Human lung epithelial A549 cells | ATCC | CCL-185 | |
| Methanol | Fisher | A412 | Stored at -20 °C |
| Mouse MAb anti-JUNV NP | BEI | NA05-AG12 | Conjugated to Alexa-647 at 1:1,000 dilution |
| Mouse MAb pan-Enterovirus 9D5 Reagent | Millipore Sigma | 3361 | ready for use; diluted to 1:2; Lot #: 3067445 |
| PBS supplemented with Ca and Mg | Corning | 21-030-CV | |
| PDL Coated coverslips | Neuvitro | H-12-1.5-pdl | |
| ProLong Gold antifade | Invitrogen | P10144 | |
| Rabbit MAb MDA-5 | Abcam | ab126630 | 1:250 dilution; Lot #: GR97758-7 |
| recombiant Candid#1 strain of JUNV | Lab generated | Lab generated | As previously described in reference 13. |
| RIG-I mouse MAb conjugated to Alexa-594 | Santa Cruz | sc-376845 | 1:1000 dilution; Lot #: AO218 |
| Triton X-100 | Sigma Aldrich | T8787 |
Referências
- Jensen, S., Thomsen, A. R. Sensing of RNA viruses: a review of innate immune receptors involved in recognizing RNA virus invasion. Journal of Virology. 86, 2900-2910 (2012).
- Weber, F., Wagner, V., Rasmussen, S. B., Hartmann, R., Paludan, S. R. Double-stranded RNA is produced by positive-strand RNA viruses and DNA viruses but not in detectable amounts by negative-strand RNA viruses. Journal of Virology. 80, 6(2006).
- Triantafilou, K., Vakakis, E., Kar, S., Richer, E., Evans, G. L., Triantafilou, M. Visualisation of direct interaction of MDA5 and the dsRNA replicative intermediate form of positive strand RNA viruses. Journal of Cell Science. 125, 4761-4769 (2012).
- Onomoto, K., et al. Critical role of an antiviral stress granule containing RIG-I and PKR in viral detection and innate immunity. PLoS One. 7, e43031(2012).
- Son, K. N., Liang, Z., Lipton, H. L. Double-stranded RNA is detected by immunofluorescence analysis in RNA and DNA virus infections, including those by negative-sense RNA viruses. Journal of Virology. 89, 9383-9392 (2015).
- Mateer, E. J., Paessler, S., Huang, C. Visualization of double-stranded RNA colocalizing with pattern recognition receptors in arenavirus infected cells. Frontiers Cellular and Infection Microbiology. 8, 251(2018).
- Buchmeier, M. J., de la Torre, J. C., Peters, C. J. Arenavirdae: The viruses and their replication. Fields Virology. Knipe, D. M., Howley, P. M. 2, Lippincott Williams & Wilkins. (2006).
- Levis, S. C., et al. Endogenous interferon in Argentine hemorrhagic fever. The Journal of Infectious Diseases. 149, 428-433 (1984).
- Levis, S. C., et al. Correlation between endogenous interferon and the clinical evolution of patients with Argentine hemorrhagic fever. Journal of Interferon Research. 5, 383-389 (1985).
- Huang, C., et al. Highly pathogenic new world and old world human arenaviruses induce distinct interferon response in human cells. Journal of Virology. 89, 7079-7088 (2015).
- Huang, C., et al. Junin virus infection activates the type I interferon pathway in a RIG-I-dependent manner. PLoS Neglected Tropical Diseases. 6, e1659(2012).
- Huang, C., Kolokoltsova, O. A., Mateer, E. J., Koma, T., Paessler, S. Highly pathogenic new world arenavirus infection activates the pattern recognition receptor protein kinase R without attenuating virus replication in human cells. Journal of Virology. 91, 20(2017).
- Emonet, S. F., et al. Rescue from cloned cDNAs and in vivo characterization of recombinant pathogenic Romero and live-attenuated Candid#1 strains of Junin virus, the causative agent of Argentine hemorrhagic fever disease. Journal of Virology. 85 (4), 1473-1483 (2011).
- Child, S. J., et al. Antagonism of the protein kinase R pathway in human cells by Rhesus Cytomegalovirus. Journal of Virology. 92, 6(2018).
- Hobro, A. J., Smith, N. I. An evaluation of fixation methods: Spatial and compositional cellular changes observed by Raman imaging. Vibrational Spectroscopy. 91, 31-45 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados