Method Article
Análise de ciclo celular no Germline de c. elegans com o EdU analógico de timidina
Neste Artigo
Resumo
É descrito um método baseado em imagens que pode ser usado para identificar a fase S e analisar a dinâmica do ciclo celular no germline hermafrodita de c. elegans , usando a timidina EdU analógico. Este método não requer nenhum transgenes e é compatível com coloração imunofluorescente.
Resumo
Análise de ciclo celular em eucariotos frequentemente utiliza a morfologia do cromossomo, expressão e/ou localização de produtos de genes necessários para várias fases do ciclo celular, ou a incorporação de análogos de nucleosídeos. Durante a fase S, polimerases de DNA incorporam os análogos da timidina como EdU ou BrdU no DNA cromossômico, marcando as células para análise. Para c. elegans, o nucleosídeo EdU analógico é alimentado para os vermes durante a cultura regular e é compatível com as técnicas de imunofluorescência. O germline de c. elegans é um sistema poderoso modelo para os estudos de sinalização de vias, células-tronco, ciclo celular e meiose porque é transparente, geneticamente facile e prófase meiótica e diferenciação celular/gametogênese ocorrem em um forma de montagem, como linear. Estas características fazem EdU, uma ótima ferramenta para estudar aspectos dinâmicos de células mitotically ciclismo e desenvolvimento da linha germinal. Este protocolo descreve como com êxito preparar EdU bactérias, alimentá-los para o selvagem-tipo c. elegans hermafroditas, dissecar a gônada hermafrodita, mancha para incorporação de EdU no DNA, mancha com anticorpos para detectar vários ciclo celular e marcadores do desenvolvimento, a gônada de imagem e analisar os resultados. O protocolo descreve as variações no método e análise para a medição de fase S índice M-fase índice, G2 duração, duração do ciclo celular, taxa de entrada meiótica e taxa de progressão da prófase meiótica. Esse método pode ser adaptado para estudar o ciclo celular ou história de celular em outros tecidos, estágios, origens genéticas e condições fisiológicas.
Introdução
No desenvolvimento do animal, centenas, milhares, milhões, bilhões ou mesmo trilhões de divisões celulares são necessários para formar o organismo adulto. O ciclo celular, o conjunto de eventos celulares compostas de G1 (gap), S (síntese), G2 (gap), e M (mitose) definir a série de eventos que são executados a cada divisão celular. O ciclo celular é dinâmico e melhor apreciada em tempo real, que pode ser tecnicamente difícil. As técnicas apresentadas neste protocolo permitem que se faça as medições das fases e o ciclo celular de imagens fixas.
Rotulagem com análogos de nucleosídeos como 5-ethynyl-2'-desoxiuridina (EdU) ou 5-bromo-2'-desoxiuridina (BrdU) é o padrão-ouro para identificar a fase S nos estudos de dinâmica do ciclo celular no adulto Caenorhabditis elegans (c. elegans) germline hermafrodita1,2,3,4,5. EdU e BrdU tanto podem ser usado em quase qualquer fundo genético, como eles não dependem de qualquer construção genética. Visualizar BrdU requer tratamento químico áspero para expor o antígeno para o anticorpo anti-BrdU coloração, que muitas vezes é incompatível com a avaliação de outros marcadores celulares visualizados por coloração com anticorpos adicionais de co. Em contrapartida, Visualizar o EdU ocorre pela química clique em condições suaves e, portanto, é compatível com o anticorpo que mancha co6,7.
A especificidade do rótulo é clara, desde que os núcleos só incorporam os análogos (5-ethynyl-2'-desoxiuridina) de timidina DNA durante a fase S. Visualização ocorre no tecido fixo. O rótulo de EdU é invisível por si só até um corante contendo azida sódica ou fluoróforo covalentemente reage com o alkyne em EdU por clique cobre-catalisada química8. EdU rotulagem pode fornecer informação imediata, na qual os núcleos estão em fase S, usando um curto pulso de rotulagem. EdU também pode fornecer informações dinâmicas, usando o pulso-chase ou rotulagem contínua; por exemplo, em um experimento de pulso-perseguição, o rótulo é diluído em cada divisão celular ou propagadas como nondividing de células de progresso através de desenvolvimento.
O germline hermafrodita de c. elegans é um sistema poderoso modelo para os estudos de sinalização de vias, células-tronco, ciclo celular e meiose. O adulto germline é uma linha de montagem polarizada com células-tronco encontradas na extremidade distal, seguida por entrada e progressão através da prófase meiótica, coordenado com as fases da gametogênese mais proximal (Figura 1). Na extremidade proximal, oócitos maduros, são ovularam e fertilizados e começam a embriogênese na útero9,10,11. A região de ~ 20 célula-diâmetro longo perto da célula de ponta distal, que inclui o tronco germline mitotically ciclismo, células progenitoras e meiose células em fase S, mas não as células na prófase meiótica, chama-se o progenitor zona2,4 , 9 , 12. as membranas celulares fornecem separação incompleta entre os núcleos no germline distal, mas células progenitoras zona passam por célula mitótica ciclismo em grande parte independente. A duração do ciclo de celular mitótica mediana do germline progenitoras de zona em jovens adultas hermafroditas é ~6.5 h; Fase G1 é curto ou ausente, e quiescência não é observada1,2,13. Diferenciação de células-tronco do germline ocorre através da modulação essencialmente direta e, portanto, carece de trânsito-amplificando divisões4. Durante a diferenciação na fase de paquíteno, aproximadamente 4 em 5 núcleos não formará oócitos, mas em vez disso, passam por apoptose, atuando como enfermeira células doando seu conteúdo citoplasmático para o desenvolvimento oócito12,14 , 15.
Além de rotulagem células em fase S com análogos de nucleosídeos, pode-se identificar as células em mitose e meiose usando anticorpos. Núcleos em mitose são imunorreativas para antifosfo-histona H3 (Ser10) Anticorpo (chamado pH3)7,16. Núcleos na meiose são imunorreativas para anticorpo antiele-3 (uma proteína de eixo de cromossomas)17. Núcleos na zona de progenitor podem ser identificados pela ausência de HIM-3, a presença de auxílio REC-8,18ou a presença de WAPL-119. WAPL-1 intensidade é maior na gônada somática, alta na zona de progenitor e baixa durante cedo meiótica prophases19. Várias medições de ciclo celular são possíveis, com algumas variações no protocolo: eu) identificar os núcleos na fase S e medir o índice de fase S; II) identificar núcleos em M-fase e medir o índice de M-fase; III) determinar se núcleos em S-fase mitótica ou meiótica; IV) medir a duração do G2; V) medir o duartion das fases G1 + G2 + M; VI) medir a taxa de entrada meiótica; VII) estimativa da taxa de progressão meiótica.
Pode-se fazer várias medições de ciclo celular de apenas alguns tipos de experimentos de laboratório-molhado. O protocolo abaixo descreve uma rotulagem de pulso de 30 min por alimentação c. elegans adultas hermafroditas com EdU rotulado de bactérias e células em fase M co rotulagem pela coloração com o progenitor e anticorpo anti-pH3 células zona por coloração com anticorpos anti-WAPL-1. Somente as alterações na duração do EdU alimentar (etapa 2.5), tipo de anticorpos empregados (etapa 5), e análises (passo 8.3) são necessárias para as medições adicionais.
Protocolo
1. preparação de bactérias EdU-etiquetado
-
Cultivar uma cultura de acionador de partida de MG1693. Escherichia coli MG1693 (Escherichia coli) carrega uma mutação em thyA.
- Raia para fora de e. coli MG1693 de um estoque de glicerol congelado em um ágar de caldo (LB) lisogenia 120 mm placa de Petri. Cultura a 37 ° C durante a noite.
- Inocular de duas individuais Escherichia coli MG1693 colônias em dois tubos de 4 mL de líquido cultura lb a 37 ° C para ~ 16 h de duplicar.
Nota: MG1693 cresce bem em LB sem suplementação com timina ou timidina.
-
Cresce MG1693 de Escherichia coli em media mínimos suplementados com EdU.
- Um Erlenmeyer de 500 mL de autoclave.
- Use técnica estéril para adicionar 5 mL de glicose de 20%, 50 μL de 10 mg/mL de tiamina, 120 μL do thymidine 5 mM, 100 μL de 1m MgSO4, 200 μL de 10mm EdU, 100 mL de tampão de M9 e 4 mL de cultura recentemente cultivados de MG1693 durante a noite.
Nota: A concentração final de EdU é 20 μM nesta cultura1,7. Esta concentração leva à prisão de danos e o ciclo celular de DNA se aplicado diretamente às células de mamíferos em cultura20. No entanto, apenas uma fração deste EdU é incorporada na Escherichia coli e, portanto, disponíveis para c. elegans. Há nenhuma evidência de detenção do ciclo celular e nenhuma mudança no tamanho da zona de progenitor ou índice M-fase após o tratamento de EdU de jovens adultos hermafroditas. - Cultura durante a noite, mas não mais de 24 horas a 37 ° C com agitação a 200 rpm.
-
Concentre-se EdU-rotulado e. coli e aplicar a pratos de Petri ágar M9.
- Pré-fixe uma centrífuga de mesa para 4 ° C.
- Use técnica estéril para transferir a cultura para 2 – 4 estéril 50 mL tubos cónicos.
- Centrifugar as culturas a 3.000 x g a 4 ° C por 30 min para o EdU-rotulado Escherichia colide Pelotas.
Nota: Elimine EdU-contendo sobrenadante de acordo com as directrizes locais e institucionais. - Resuspenda as pelotas com 4 mL de fresco M9. Use a ponta da pipeta estéril 1 mL ou uma pipeta sorológica 5ml estéril. Resuspending as pelotas pode demorar vários minutos.
- Use a mesma pipeta para aplicar e espalhar ~ 8 gotas de ressuspensão EdU-rotulado Escherichia coli MG1693 para o centro da temperatura 60mm M9 pratos de Petri ágar. Um lote produz ~ 16 pratos.
- Permitir que os pratos secar durante várias horas ou durante a noite à temperatura ambiente e, em seguida, cada prato com uma tira de filme do laboratório do selo. Pratos podem ser armazenados a 15 ° C ~ 2 semanas e a 4 ° C ~ 2 meses. Use o mesmo lote de pratos do EdU para cada conjunto de experimentos.
2. alimentação EdU para c. elegans
- Sincronize a população por ovo cronometrado hipoclorito alcalino, leigos tratamento seguido de incubação em meio de S com o colesterol, ou escolhendo o estágio apropriado21. Crescem os animais ao estágio desejado (aqui, 24h post-L4) no meio de crescimento de nematódeo semeado com Escherichia coli OP50 a 20 ° C.
Nota: Prepare 50 – 100 animais por experiência, como alguns não podem dissecar bem, e os outros serão perdidos no processo de lavagem e transferência. - Permitir que os pratos de EdU aquecer a 20 ° C (ou a temperatura necessária para o experimento).
- Lave os animais de pratos NGM usando M9 ou tampão fosfato salino (PBS) para um tubo de 1,5 mL. Permitir que os animais a resolver brevemente por gravidade ou um breve spin numa microcentrifuga.
- Remover o sobrenadante e lavar os animais 1 – 2 vezes com ~ 1 mL de M9 ou PBS. Remova o sobrenadante.
- Use um pipeta Pasteur de vidro para transferir os animais em uma minúscula gota de M9 ou PBS no centro do gramado da EdU. Espere alguns minutos para que o líquido a ser absorvido e, em seguida, incubar a 20 ° C por 30 min (para medição direta da fase S) ou mais (para medir a história da fase S), conforme necessário.
Nota: Sinal de EdU é detectável em núcleos germline depois de apenas 15 min do EdU alimentação1. - Lave o prato EdU com ~ 2 mL de M9 ou PBS em um vidro dissecando prato os vermes.
3. dissecção e fixação do Germline de c. elegans
Nota: Este protocolo para a dissecação, fixação e coloração de anticorpo do germline de hermafrodita c. elegans é quase idêntico ao publicado por Gervaise e Arur (2016)22, exceto que os tubos de vidro de 1 mL podem ser centrifugados para acelerar lavagem de passos e um vidro chamou-out pipeta Pasteur pode ser usada para remover o líquido de tubos de vidro de 1 mL mais eficazmente.
-
Lave e dissecar germlines c. elegans .
- Permitir que os animais repousar no fundo do prato dissecação, redemoinho para recolher os animais no centro e usar uma pipeta Pasteur longa para remover PBS. Lave 1 – 2 vezes com ~ 2 mL de PBS.
- Adicione 2 mL de PBS e 4 μL de 100mm levamisole para imobilizar os animais. Agite o prato novamente para recolher os animais no centro do prato.
Nota: Imobilização pode demorar entre alguns segundos e a poucos minutos. Imobilização completa não é necessária para dissecação bem sucedida. Algumas pessoas têm sucesso melhor quando dissecando incompleta imobilizada animais. - Dissecar os animais com um par de agulhas 25 5/8" cortando à frente (aproximadamente entre os dois bulbos da faringe) ou a cauda. Tome cuidado para não cortar o laço do germline. Intestino e germline deve "saltar" da cavidade do corpo devido à pressão interna, mas permanecem ligados. Este protocolo é semelhante ao publicado anteriormente Gervaise e Arur (2016)22.
Nota: Manter o tempo de dissecação para ~ 5 min, certamente não mais que 15 min. tempos mais longos de dissecação podem resultar na perda de sinal de coloração de anticorpo (Sudhir Nayak, comunicação pessoal) e fome em PBS pode afetar a fisiologia dos animais. Aprender a dissecar rapidamente e com precisão pode levar um pouco de prática. - Se necessário, para coletar os animais dissecados no centro do redemoinho e usar uma pipeta Pasteur longa para remover tanto PBS quanto possível.
-
Corrigir e desidratar tecidos
- Adicione 2 mL de 3% paraformaldeído (PFA) em solução de PBS. Cubra o prato frouxamente com filme de laboratório e loja num banco ou numa gaveta por 10 min.
Nota: Descongelar solução PFA em um 37 ° C água de banho e depois arrefecer à temperatura antes da germlines de dissecação.
Atenção: A solução PFA é moderadamente tóxica e um provável carcinogênico e teratógeno. Emissores de soluções paraformaldeído de vapores são inflamáveis. Use luvas de nitrilo. Dilua o PFA de 16% para 3% em uma coifa de química. Quando se trabalha fora da coifa, manter todos os recipientes cobertos. - Transferi as gônadas cuidadosamente para um tubo de centrífuga de vidro limpo 5 mL.
- ~ 3 ml de PBSTw (PBS com 0.1% Tween-20) para o prato que continha as gônadas para ajudar a recuperar o restante das gónadas e diluir a solução PFA.
- Spin-down em uma centrífuga clínica a 870 x g ~ 1 min.. Younger ou animais menores exigem tempos de rotação mais do que animais mais velhos ou maiores.
- Utilizando uma pipeta de vidro longo, transfira o sobrenadante para copo de material indesejado para eventual descarte em garrafa de material indesejada no bairro química.
- Adicionar 2 mL de metanol de primeira qualidade, pre-refrigerado a-20 ° C. Cobrir o tubo de centrifugação firmemente com filme de laboratório.
Nota: O uso de metanol frescos de alta qualidade "gold label" é essencial para morfologia adequada com determinados anticorpos.
Atenção: O metanol é um líquido altamente inflamável e vapor, que é tóxico se ingerido, inalado ou permissão para entrar em contato com a pele. Use luvas e equipamento de protecção adequado. Use um freezer adequado para pequenos volumes de materiais inflamáveis. - Guarde no congelador-20 ° C por 1 h, mesmo durante a noite ou até vários meses.
Nota: O protocolo pode ser pausado aqui.
- Adicione 2 mL de 3% paraformaldeído (PFA) em solução de PBS. Cubra o prato frouxamente com filme de laboratório e loja num banco ou numa gaveta por 10 min.
4. Re-hidratar Germlines
- Encha o tubo de centrifugação de vidro topo com PBSTw para diluir o metanol e Re-hidratar as gônadas. Spin para baixo em uma clínica centrifugar 870 x g por ~ 1 min.
- Usando um copo longo pipeta Pasteur, transfira o sobrenadante para copo de material indesejado para eventual descarte em garrafa de material indesejada no bairro química.
- Lave as gônadas 3 vezes usando ~ 5 mL de PBSTw, girando para baixo em uma clínica centrifugar 870 x g por ~ 1 min cada tempo. Remova o sobrenadante.
- Lave um tubo de vidro de borosilicato de pequena 1 mL e um copo longo pipeta Pasteur com PBSTw.
- Adicionar ~ 700 μL de PBSTw e usar o vidro longo pipeta Pasteur para transferir as gônadas para o pequeno tubo. Use algumas gotas adicionais de PBSTw para garantir que todas as gônadas são transferidas.
- Spin para baixo em uma centrífuga clínica a 870 x g ~ 1 min. usando um copo longo elaborada pipeta Pasteur, remover tanto líquido quanto possível, sem perturbar as gônadas. Embora não mais de 50 μL.
Nota: Se de anticorpos não é necessário, vá para a etapa 6.
5. detectar antígenos com anticorpos
- Dilua os anticorpos no soro de 30% em PBS. No presente exemplo, anticorpo anti-WAPL-1 é 1:2,000 diluído e anticorpo anti-pH3 é diluído 1: 500. Centrifugue o soro fresco descongelado por 10 min em uma microcentrífuga a 20.000 x g a 4 ° C para remover as partículas. Use o sobrenadante, que pode ser armazenado a 4 ° C por vários dias. Use o soro apropriado para coincidir com o organismo hospedeiro de anticorpos secundários (soro de cabra é usado aqui). Uma etapa de bloqueio opcional pode ser adicionada antes da adição de anticorpos primários.
- Aplica 100 μL do anticorpo primário diluído a cada tubo de vidro pequeno. Incube a temperatura ambiente durante 4 h.
Nota: Tempos de incubação variam pelo anticorpo. Para alguns anticorpos, 2 h à temperatura ambiente é suficiente. Incubação do mais longa (ex., durante a noite) são possíveis, mas pode aumentar o fundo. - Encha os tubos para top com PBSTw e spin para baixo em uma centrífuga clínica a 870 x g por ~ 1 min.
- Lave as gônadas 3 vezes usando ~ 1 mL de PBSTw. Incubar por ~ 5 min por lavagem para permitir que o anticorpo primário excesso de lavagem se espalham. Usando um chamou-out longa pipeta Pasteur de vidro, remover tanto líquido quanto possível, sem perturbar as gônadas. Embora não mais de 50 μL.
- Dilua os anticorpos secundários em 30% de soro de cabra em PBS. No presente exemplo, cabra-anticoelho-594 e cabra-antirato-647 são cada diluídos a 1: 400.
Nota: Selecione anticorpos secundários cuidadosamente para certificar-se de corantes são distintos da tintura no kit de EdU. No presente exemplo, o EdU kit continha um corante de excitação 488 nm. - Aplica 100 μL do anticorpo secundário diluído para cada tubo de vidro pequeno. Incube no escuro à temperatura de 2 h.
Nota: Tempos de incubação variam pelo anticorpo. Para alguns anticorpos secundários, 1h à temperatura ambiente é suficiente. Incubação do mais longa (ex., durante a noite) são possíveis, mas pode aumentar o fundo. - Lave as gônadas 3 vezes usando ~ 1 mL de PBSTw. Incubar por ~ 5 min por lavagem para permitir que o anticorpo secundário excesso de lavagem se espalham. Usando um chamou-out longa pipeta Pasteur de vidro, remover tanto líquido quanto possível, sem perturbar as gônadas. Embora não mais de 50 μL.
Nota: Gônadas podem ser armazenadas em 100 µ l de PBS após esta etapa, se necessário, embora isto pode reduzir o sinal. Remova o PBS antes de prosseguir.
6. realizar a reação de EdU clique para detectar EdU
Nota: Executar o EdU clique reação antes o anticorpo que mancha passos (executar a etapa 6 antes do passo 5) é possível, dependendo os anticorpos utilizados7. No entanto, os reagentes de clique podem interferir com determinados antígenos (EG., REC-8 anticorpo é sensível à fixação e permeabilização). A ordem aqui apresentada resulta brilhante anticorpo que mancha com o REC-8, WAPL-1, 3-HIM, pH3, bandeira e CYE-1 anticorpos utilizados, entre outros.
-
Prepare o clique EdU cocktail8 fresco, adicionando o seguinte para um tubo limpo 1,5 mL. A ordem das adições é importante. Proteger da luz e manter todos os reagentes no gelo. Esta receita rende o suficiente para uma amostra (100 μL); multiplica a receita conforme necessário.
- Adicione 2 mL de água ultrapura para o buffer de aditivo. Isso faz com que 10 x aditivo de reserva, que deve ser diluído a 1 x imediatamente antes da utilização.
- Adicione 8,5 μL de tampão de x 10 para 76.5 μL de água ultrapura. Misture bem.
- Adicione 4 μL de 100mm CuSO4 (pode ser rotulada como componente E). Misture bem.
- Adicione 0,25 μL da tintura 488 nm azida. Devem ser descongelado à temperatura ambiente, como seu solvente, dimetilsulfóxido, é sólido a 4 ° C. Misture bem e proteger da luz.
- Mix 9 μL de água ultrapura com 1 μL do buffer aditivo na tampa do tubo. Pipeta de PAC para adicionar ao coquetel restante e misture bem pipetando para cima e para baixo.
-
Realize a reação de clique de EdU.
- Adicione ~ 100 μL de clique EdU cocktail para as gônadas no tubo pequeno. Cubra com filme de laboratório e incube por 30-60 min à temperatura ambiente.
- Lave uma vez com 100 μL de tampão de lavagem de reação.
- Lave as gônadas 4 vezes usando ~ 1 mL de PBSTw. Incubar por ~ 15 min por lavagem para permitir que excesso EdU cocktails componentes difusa na lavagem. Usando um chamou-out longa pipeta Pasteur de vidro, remover tanto líquido quanto possível, sem perturbar as gônadas. Embora não mais de 50 μL.
7. mancha de DNA e preparar Slides
- Adicione 1 gota (~ 25 μL) de meio de montagem antidesgaste com 4' 6-diamidino-2-phenylindole (DAPI, usado para visualizar o DNA) para as gônadas. Aguarde alguns minutos para que ele pode resolver e misturar com as gônadas.
Nota: Alternadamente, numa diluição de 1:1,000 de DAPI (a partir de um estoque de 0,1 mg/mL) em PBS pode ser aplicada por 5 min, seguido por 20 μL de 1,4-diazabiciclo [2.2.2] octano (DABCO) em 90% de glicerol, ou meio de outra montagem antidesgaste. - Prepare uma grande almofada de agarose 2,5% em um padrão de vidro de microscópio.
- Uso um novo limpo livre de poeira vidro longo pipeta Pasteur para transferir as gônadas para a almofada de agarose. Manter todos os líquidos e gônadas no fundo estreito da pipeta para minimizar a perda das gônadas.
Nota: Gônadas preso ao tubo de vidro pequeno ou em vidro longo pipeta Pasteur pode ser "resgatada" por lavagem com PBSTw, recolher o líquido em um prato de dissecação e vamos buscar animais individuais para o slide com um cílio. - Usar uma pestana (ou um laço de cabelo fino) colado a um palito de dente para distribuir as gônadas sobre a almofada de agarose e remover as partículas de poeira.
- Uma lamela de vidro retangular. Baixa lentamente de um lado para evitar bolhas de ar. Use um lenço para remover a solução em excesso, evitando assim a lamela de mover-se livremente.
Nota: Escolha as lamelas que correspondem ao microscópio que será usado. #1 e #1.5 lamelas funcionam bem. - Permitir que os slides para se estabelecer e secar ligeiramente durante a noite em temperatura ambiente ou 4 ° C. Isso ajuda a achatar ligeiramente as gônadas. Slides devem ser armazenadas a 4 ° C.
- Opcional: Sele as bordas da lâmina com esmalte ou outro aferidor de slide. Selando os cantos primeiro, então os lados, impede que a lamela deslocando.
8. análise e imagem confocal
- Imagem a gônada distal com um microscópio fluorescente confocal disco de fiação equipado com uma fonte de luz de alta energia, plano-apocromático objectivos e uma câmera de microscópio de alta eficiência. Capture as imagens com 1 μm ou espaçamento mais apertado entre pilhas de z. Tome nota do tempo de exposição, sensibilidade e poder do laser para todos os canais.
- Use o seguinte: 405 nm laser linha excitação com um filtro de emissão 485 nm (W60) para DAPI, 488 nm laser linha excitação com um filtro de emissão 527 nm (W55) para EdU, 561 nm laser linha da excitação com um filtro de emissão nm (W70) 615 para WAPL-1 e um 640 nm laser linha excita ção com um filtro de emissão 705 nm (W90) para pH3.
Nota: Variará a intensidade do sinal e intensidade de fundo. Da mesma forma, os tempos de exposição necessário variará, possivelmente até 10-fold.
- Use o seguinte: 405 nm laser linha excitação com um filtro de emissão 485 nm (W60) para DAPI, 488 nm laser linha excitação com um filtro de emissão 527 nm (W55) para EdU, 561 nm laser linha da excitação com um filtro de emissão nm (W70) 615 para WAPL-1 e um 640 nm laser linha excita ção com um filtro de emissão 705 nm (W90) para pH3.
- Use o plug-in do contador de célula23 em Fiji24,25 a contagem manualmente cada núcleo. Etiquete cada núcleo individual de acordo com a presença e ausência de pH3, EdU e WAPL-1. Use as classes dos núcleos descritos na tabela 1 e Figura 2, como estas facilitará todos os cálculos descritos abaixo.
Nota: Experimentalistas hábil podem contar com precisão todos os núcleos em uma imagem 3D sem dupla contagem ou faltando qualquer núcleos. Alternadamente, um pode contar cada núcleo em cada plano z, e o roteiro de marcas-de-células R3 pode ser usado para remover núcleos multiplicar-contados. - Calcule o número de células e frequências compreendidas entre as contagens acima, dependendo do tipo de ciclo celular medida necessária. Os tipos de núcleos são definidos na tabela 1 e Figura 2. Os cálculos estão sumarizados na tabela 2.
- (Variação eu) Para identificar os núcleos na fase S, alimente os animais EdU por 30 min. Qualquer núcleos exibindo EdU rótulo são núcleos da fase S. Para calcular, leve os núcleos soma de A e C, veja a tabela 1 e Figura 2.
Nota: Em um pulso de EdU em adultos hermafroditas selvagem-tipo de 30 min, EdU todos rotulados núcleos são co rotulado com o progenitor de marcadores de zona1. - Para medir a zona do progenitor, mancha com um marcador de zona de progenitor como anticorpo REC-8 ou WAPL-1. A zona de progenitor é definida aqui como todos auxílio REC-8,18 ou núcleos de imunorreativas germline WAPL-1. Para calcular, somar todos os núcleos de imunorreativas WAPL-1 (A + B + C + D, ver tabela 1 e Figura 2).
Nota: WAPL-1 também rotula o DTC e núcleos de gônada somática que não devem ser contados. Núcleos somáticos são fáceis de identificar por sinal extremamente intenso de WAPL-1, posição ligeiramente fora do germline e uma morfologia de "ovo frito" dos núcleos. - Para medir o índice de fase S, realizar um experimento de EdU de 30 min e co etiqueta com anticorpo REC-8 ou WAPL-1. O índice de fase S é definido como a proporção da zona progenitor que é na fase S. Para calcular, contar todos os núcleos de fase S e depois dividir pelo número total de núcleos de zona do progenitor (A + C / A + B + C + D, ver tabela 1 e Figura 2).
- (Variação II) Para identificar os núcleos na fase M, mancha com anticorpo pH3. Qualquer núcleos imunorreativas pH3 são núcleos M-fase. Isso funciona independentemente da duração de alimentar o EdU. Para calcular, levar a soma da e núcleos de B, ver tabela 1 e Figura 2.
Nota: WAPL-1 também rotula o DTC e núcleos de gônada somática que não devem ser contados. Núcleos somáticos são fáceis de identificar por sinal extremamente intenso de WAPL-1, posição ligeiramente fora do germline e uma morfologia de "ovo frito" dos núcleos. - Para medir o índice de M-fase, co etiqueta com pH3 e REC-8 ou anticorpos WAPL-1. O índice de M-fase é definido como a proporção da zona progenitor que está numa fase de M. Para calcular, contar todos os núcleos de M-fase e depois dividir pelo número total de núcleos de zona do progenitor (A + B / A + B + C + D, ver tabela 1 e Figura 2).
- (Variação III) Núcleos em S-fase mitótica e meiótica ambos etiqueta com EdU. Para dizer as duas populações separadas, determine se a fase S foi seguida por mitose ou meiose. Para determinar se os núcleos estão em fase S mitótica ou meiótica, alimentar EdU por 4h e co etiqueta para pH3 (um marcador de M-fase) e HIM-3 (uma proteína de eixo de cromossomas) por anticorpos. Gravar os núcleos que exibem tanto EdU e pH3 (tipo A, ver tabela 1 e Figura 2) como S-fase mitótica enquanto núcleos que exibir EdU e HIM-3 (tipo E, ver tabela 1 e Figura 2) como S-fase meiótica.
- (Variação IV) Calcule a duração da fase G2.
Nota: G2-fase separa fase S M-fase. Embora nenhum marcador tem sido relatada a etiqueta G2 no germline de c. elegans , um pode calcular a duração da fase G2, combinando dados de vários experimentos que rotulam M-fase (na época da dissecação) e S-fase (a partir de vários h antes dissecação). Uma célula que exibe marcadores da fase S e M-fase concluída G2-fase durante o curso do experimento. Uma célula que exibe somente o marcador de M-fase e não o marcador de fase S não estava na fase S durante o experimento.- Para calcular a duração da fase G2, alimente EdU por 2h e co etiqueta com anticorpo pH3. Examine a único núcleos que rotulam com pH3 (esses são em M-fase aquando da dissecação) a presença de EdU (estes eram na fase S durante o rótulo de EdU 2 h antes da dissecação). Calcular a fração de núcleos de M-fase que completou a fase G2 (A / A + B, ver tabela 1 e Figura 2).
- Repita esta experiência com um rótulo de EdU de 3h e novamente com um rótulo de EdU 4h (e, opcionalmente, um rótulo de EdU 5h). Traça a porcentagem de pH3 núcleos positivos que são EdU positivo no eixo y e a duração do EdU rótulo no eixo x, como mostrado na Figura 3A.
- Calcule a duração mediana do G2-fase conectando os pontos no gráfico e determinar onde a linha cruza a 50%, conforme mostrado na Figura 3A.
- Calcule a duração máxima da fase G2 conectando os pontos no gráfico e determinar onde a linha cruza a 99%, conforme mostrado na Figura 3A.
- (Variação V) Calcule o duraion do G1 + G2 + M.
Nota: No germline de c. elegans , fase G1 é extraordinariamente curto. Embora nenhum marcador tem sido relatado para rotular G1 no germline de c. elegans , um pode estimar a duração da soma da fase G1, G2 e M e então comparar este tempo com o tempo de G2-fase descrito acima. A duração máxima do G1 + G2 + M é estimada a partir da percentagem de todos os núcleos de zona de progenitor (WAPL-1 imunorreativas) que permanecem EdU negativo (não sejam submetidos a fase S) depois EdU rotulagem durante várias horas.- Para calcular a duração do G1 + G2 + M, alimente EdU por 2h e co etiqueta com anticorpo REC-8 ou WAPL-1. Determinar a fração da zona progenitor que passou por fase S durante este tempo (A + C / A + B + C + D, ver tabela 1 e Figura 2).
- Repita esta experiência com um rótulo de EdU de 3h e novamente com um rótulo de EdU 4h (e, opcionalmente, um rótulo de EdU 5h). Traça a porcentagem de REC-8 ou WAPL-1 núcleos positivos que são EdU positivo no eixo y e a duração do EdU rótulo no eixo x, como mostrado na Figura 3B.
- Calcule a duração máxima do G1 + G2 + M conectando os pontos no gráfico e encontrar onde a linha cruza a 99%, conforme mostrado na Figura 3B.
Nota: É possível realizar os experimentos para determinar a duração do G2 e G1 + G2 + M como um único conjunto de 2, 3, 4 e 5 h EdU experimentos por co etiquetando com anticorpos tanto coelho-anti-WAPL-1 e antirato-pH3.
- (Variação de VI) Para identificar os núcleos que replicados na zona de progenitor, mas desde então, entrou meiose, alimente os animais EdU para 10 h e rótulo co com anticorpos REC-8 ou WAPL-1. Qualquer núcleos exibindo EdU rótulo foram na fase S durante esses 10 h. Qualquer núcleos que não exibem coloração auxílio REC-8 ou WAPL-1 foram na meiose. Simplesmente contar os núcleos com EdU rotulagem que não exibem rotulagem com o marcador de zona de progenitor (E, ver tabela 1).
Nota: Por outro lado, se gônadas estão manchadas para o marcador de prófase meiótica HIM-3 com anticorpos anti-HIM-3, contagem do número de núcleos com EdU rotulagem que são também positivos para HIM-3. - Para calcular a taxa de entrada meiótica, realize o experimento acima com um rótulo de 5 h, 10 h e 15 h de EdU. Plotar o número de núcleos que inseriu meiose no eixo y e a duração do EdU rótulo no eixo x, como mostrado na Figura 3. Em seguida, use uma regressão linear simples para calcular a inclinação (meiose núcleos entrados por h) de y = mx + b.
Nota: É fundamental usar uma regressão linear para calcular a taxa de entrada meiótica. Seria incorrecto é só dividir o número de núcleos que inseriu a meiose pela duração do rótulo do EdU, porque a intercepção de y não é zero. - (Variação VII) Medir a taxa de progressão meiótica.
Nota: Desde que EdU covalentemente é incorporado no DNA, ele pode ser usado para controlar uma população de células através da diferenciação. As células que foram submetidos a fase S na zona de progenitor retêm o rótulo de EdU como eles entram em meiose, progresso através de meiose e submeter-se a ovogênese. Um experimento de pulso-chase com EdU pode ser usado para medir a taxa de progressão meiótica.- Bactérias de EdU-etiquetado alimentação aos animais para 4h (o "pulso"). Transferir os animais para sem rótulo OP50 bactérias por 48h (a "perseguição"), em seguida, dissecar e co rotular com um marcador de zona de progenitor como REC-8 ou WAPL-1 (ou um marcador de prófase meiótica como HIM-3), se desejado.
- Quando a imagem, procure a posição do núcleo EdU-rotulados mais proximal. A taxa de progressão meiótica é a distância (em diâmetros de célula da extremidade da zona de progenitoras) viajou pelo EdU mais proximal rotulado núcleo durante a perseguição de 48 h.
- (Variação eu) Para identificar os núcleos na fase S, alimente os animais EdU por 30 min. Qualquer núcleos exibindo EdU rótulo são núcleos da fase S. Para calcular, leve os núcleos soma de A e C, veja a tabela 1 e Figura 2.
Resultados
Como a síntese de DNA é necessária para incorporar o EdU, pode-se concluir que EdU-rotulado núcleos passou por fase S durante a janela de tempo de criação de etiquetas de EdU. Um pode interpretar os núcleos esse rótulo em uma alimentação de 30 min com EdU rotulado bactérias como núcleos em fase S no momento da dissecação. Núcleos que rotular em uma mais EdU contínua alimentação experimento podem ter rotulado cedo na janela de tempo e desde a fase S esquerdo, ou podem ter identificado na parte final da janela de tempo de EdU. Sinal de EdU co localiza com sinal DAPI. Em alguns núcleos, EdU sinal abrange todos os cromossomos, enquanto em outros núcleos EdU sinal localiza-se a partitura brilhante de 1 – 2 (Figura 4). Estes partitura são provavelmente o cromossomo x, que Replica no final da fase S13.
Aqui, os animais foram alimentados com EdU continuamente por 30 min e dissecados, como descrito acima e na Figura 5. Um exemplo de sucesso EdU coloração em um animal adulto jovem e um exemplo de sucesso EdU coloração em um animal adulto mais velho (ver abaixo) são mostrados na Figura 4. EdU sinal de uma rotulagem de 30min localiza-se a aproximadamente metade dos núcleos na zona de progenitor (definida pelo anticorpo WAPL-1 rotulagem mas aproximadas por DAPI morfologia26,,27,28). Índice de fase S, a proporção da zona progenitor que é EdU positivo, foi anteriormente relatado em 57 ± 5% e de 70% em adultos jovens1,2,3. M-fase índice é aproximadamente 2-3%1,29. Em contínua alimentação para 4 h ou mais, todos os núcleos no rótulo de zona de progenitor com EdU e alguns núcleos que rotulado na zona progenitoras desde que entraram prófase meiótica1.
Enquanto a técnica funciona consistentemente em animais adultos jovens selvagem-tipo, uma fração significativa do acasaladas hermafroditas 5 dias de idade (mesmo aqueles que contém espermatozoides) falha ao rótulo em um 30 min EdU pulso (Figura 4E). No entanto, com um h 4 EdU alimentando, quase todos estes animais rótulo. Falha esporádica de rótulo em genéticas fêmeas com pulsos curtos de EdU também foi relatado30. Pode haver outras situações que resultam em falha esporádica a etiqueta.
Um pode calcular a duração do ciclo celular, realizando vários EdU-rotulagem experimentos com pH3 rotulagem em cada um. A duração da G2 foi estimada analisando a porcentagem de núcleos em M-fase (pH3 imunorreativas) que eram EdU positiva durante o curso do tempo (Figura 6). Essa abordagem fornece mediana e duração máxima de G2 (Figura 3A). O tempo mediano foi interpolado, mostrando uma duração aproximada de G2 de 2,5 h em jovens adultas hermafroditas. A duração do G1 + G2 + M foi estimada da percentagem de todos os núcleos de zona de progenitor (WAPL-1 imunorreativas) que eram EdU positivo (Figura 6). O método G1 + G2 + M fornece uma medida de duração máxima para as fases combinadas (Figura 3B). O tempo de percentil 99 foi interpolado, mostrando uma duração aproximada de G1 + G2 + M de 3,4 h em jovens adultas hermafroditas. Foram utilizados dados das mesmas experiências para calcular a taxa de entrada meiótica (núcleos por h). A taxa é a inclinação da regressão linear do número de núcleos que inseriu a meiose (EdU positivo, positivo negativo ou HIM-3 de WAPL-1) ao longo da duração do rótulo EdU (Figura 3). Os valores para o selvagem-tipo 1 dias de idade adultos hermafroditas são mostrados na tabela 2.
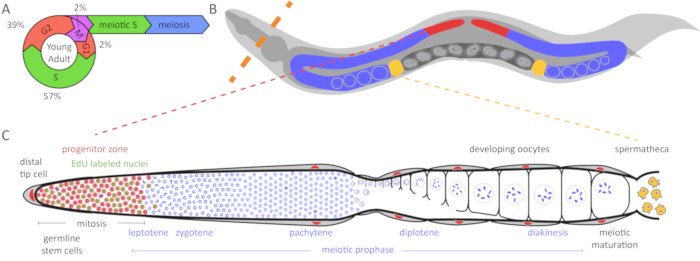
Figura 1: Diagrama do germline de c. elegans e ciclo celular. (A) o ciclo celular das células germinativas no jovem adulto germline hermafrodita. Os números indicam a percentagem aproximada de tempo gasto em cada fase do ciclo celular. (B) c. elegans hermafroditas tem dois germlines em forma de U (vermelho e azul). A espermateca é mostrada em amarelo e o útero com o desenvolvimento de embriões é mostrado em cinza escuro. Linha tracejada laranja indica onde os animais são dissecados para expulsar o germlines. (C) diagrama de uma desdobrada c. elegans germline. DAPI (azul) é um corante de ADN que destaca a morfologia nuclear. A zona distal do progenitor (destacada em vermelho com base na coloração de anticorpo WAPL-1) contém mitotically ciclismo células-tronco e células progenitoras células em fase S meiótica (WAPL-1 também rotula núcleos somáticos gônada). Células em fase S mitótica e meiótica rotular com um pulso de EdU de 30 min e são indicadas em verde. Duas células em fase de M etiqueta com anticorpo pH3 e são mostradas em preto. A célula de ponta distal (DTC) fornece o ligante de GLP-1/entalhe para manter o destino de células-tronco destas células. Como as células migram longe do DTC, eles a zona do progenitor e digite prófase meiótica. Células amarelas são esperma na espermateca. Clique aqui para ver uma versão maior desta figura.
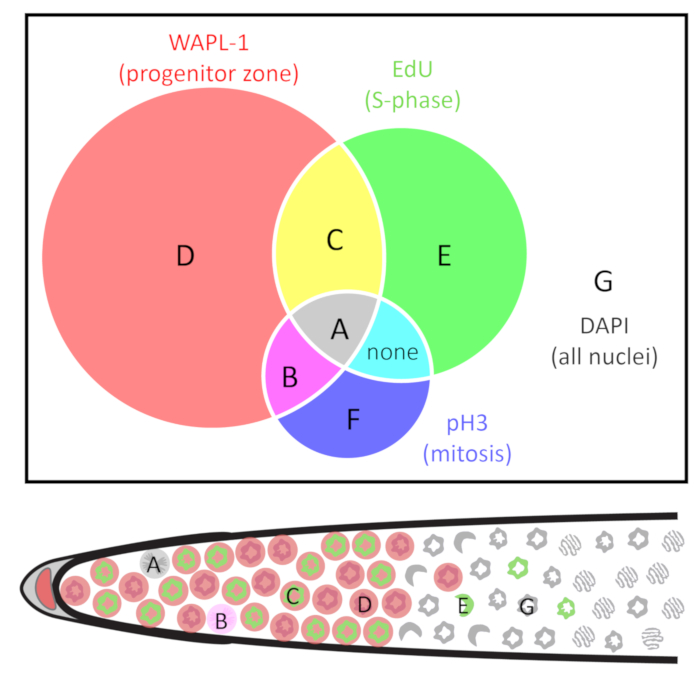
Figura 2: diagrama de classes de núcleos. Núcleos são agrupados pela presença e ausência de três marcadores: WAPL-1 indica células progenitoras de zona (vermelho), EdU indica células em fase S (verde) e pH3 indica células em fase M (azul). Tipos de células são identificados como A-G. Note que no selvagem-tipo jovens adultas hermafroditas não se encontram células do tipo F, e as células co não rotular com EdU e pH3 fora o progenitor (WAPL-1 positivo) zona. O diagrama de gônada distal abaixo indica um exemplo de A-E e G núcleos. Veja a tabela 1 para mais detalhes. Clique aqui para ver uma versão maior desta figura.

Figura 3: apresentação gráfica de duração do ciclo celular e taxa de dados experimentais de entrada meiótica. (A) a duração da G2 fase é interpolada de pH3 e EdU co rotulagem seguinte duração variada EdU pulsos linhas cinzas indicam os percentis 50 e 99 usados em interpolação mediana e máximos durações de G2, indicadas pelas setas. (B) uma célula na fase G1, G2 e M não incorporar EdU. Assim, a duração máxima da fase G1 + G2 + M pode ser estimada medindo-se a duração máxima do rótulo de EdU que produz células de EdU-negativo. A duração da fase G1 + G2 + M é interpolada de EdU e REC-8 co rotulagem seguindo impulsos de EdU de duração variada. Linha cinza indica o percentil 99 usado em duração máxima G1 + G2 + M, indicada por uma seta de interpolação. Não é possível interpolar a duração mediana do G1 + G2 + M. (C) a taxa de entrada meiótica (em núcleos por h – ver quadro 2) é calculado a partir do declive da linha de regressão. Observe que, desde a intercepção de y-intercept não é zero, uma regressão é necessário para um cálculo exacto da taxa de entrada meiótica (C). . Barras de erro indicam o desvio padrão. Figuras modificado e reimpresso com permissão da Fox et al . 20111. Clique aqui para ver uma versão maior desta figura.

Figura 4: exemplo de manchar o EdU bem-sucedidas e malsucedida 30 min. Imagens de microscópio confocal de um 1 dia de idade (A-D) e uma gônada hermafrodita 5 dia de idade (E-H) (esperma não esgotadas) após um EdU rotulagem experimento de 30 min. Linha branca tracejada marca o fim da zona do progenitor. O asterisco marca a posição da ponta distal. Marcas verdes EdU coloração visualizado por clique em química (A). Rotulagem de EdU malsucedido resulta em fundo de baixo nível de coloração mas não brilhante EdU + núcleos (E). Imunofluorescência marcas vermelhas WAPL-1 (B, F). Amarelo indica sobreposição (C, G). Marcas azuis DAPI mancha para DNA (D, H). Pontas de seta única indicam um núcleo com EdU mancha em toda a cromatina. Setas duplas indicam um núcleo com EdU partitura em apenas um par de cromossomos. Imagens foram obtidas com um objetivo X 63. Barra de escala = 10 µm (D, H). Clique aqui para ver uma versão maior desta figura.

Figura 5: fluxo de trabalho Experimental. Um resumo do protocolo experimental para crescer (A), dissecar o rótulo de EdU (B), (C), mancha de anticorpo (D), realizar a reação de clique para anexar um corante para EdU (E), mancha de DNA (F), germlines (G), de imagem e quantificar o EdU rotulado e anticorpo manchado núcleos (H). Clique aqui para ver uma versão maior desta figura.

Figura 6: exemplo de sucesso 4h EdU coloração. Imagens de microscópio confocal de uma gônada hermafrodita adulta 1 dia de idade, após uma EdU rotulagem experimento de 4 h. Linha branca tracejada marca o fim da zona do progenitor. O asterisco marca a posição da ponta distal. Magenta marca pH3 imunofluorescência (A, C). Marcas verdes EdU coloração visualizado por clique em química (B, C). Imunofluorescência marcas vermelhas WAPL-1 (D). Amarelo indica a sobreposição de EdU e WAPL-1 (E). Marcas azuis DAPI coloração de DNA (F). Pontas de seta única indicam núcleos co rotulados com EdU e pH3. Ponta de seta dupla marca um pH3 + EdU-núcleo - uma ocorrência rara em uma rotulagem de EdU de 4h. As setas marcam EdU + WAPL-1 - núcleos que entraram a meiose. Imagens foram obtidas com um objetivo X 63. Uma barra de escala de 10 µm é mostrada (F). Clique aqui para ver uma versão maior desta figura.
| Marcador: | pH3 | EdU * | WAPL-1 ou REC-8 | ELE-3 | |
| Interpretação: | Mitose | S-fase * | Zona de progenitor | Meiose | |
| Classe: | Interpretação combinada: | ||||
| A |  |  |  |  | M-fase, na zona do progenitor, estavam na S-fase mitótica durante EdU rótulo (G2 concluído) |
| B |  |  |  |  | M-fase, na zona do progenitor, não estavam em fase S durante o rótulo de EdU |
| C |  |  |  |  | na interfase, em zona de progenitor, estavam em fase S durante EdU rótulo |
| D |  |  |  |  | na interfase, em zona de progenitor, não estavam em fase S durante o rótulo de EdU |
| E |  |  |  |  | na meiose, estavam em fase S meiótica durante EdU rótulo (núcleos de entrada meiótica) |
| F |  |  |  |  | retornar a mitose (encontrado em alguns mutantes) ou divisões meióticas (em espermatogênese) |
| G |  |  |  |  | na meiose, não estavam em fase S durante o rótulo de EdU |
| soma total pH3 positivo; todas as células na fase M | soma total EdU positiva; todas as células na fase S | soma total WAPL-1 positivo; todas as células na zona do progenitor | soma total positiva do HIM-3; todas as células na prófase meiótica |
Tabela 1: Classes de núcleos. * Observe que 30 min e 4h EdU experiências diferem na interpretação. Na longa duração EdU experimentos, células provavelmente têm progredido além da fase S. Consulte Introdução e passo 8 para duração do EdU rotulagem para experiência relevante.
| Parte do ciclo celular | Definição operacional | Cálculo * | Valor * * |
| Núcleos do progenitor zona | todos WAPL-1 (ou REC-8) núcleos negativos positivos, HIM-3 | A + B + C + D | 231 ± 23 núcleos |
| Núcleos de fase S | núcleos EdU positivo após 30 min EdU rótulo e WAPL-1 positivo | A + C | 133 ± 20 núcleos |
| Núcleos de M-fase | pH3 e núcleos co-positive WAPL-1 | A + B | 5,2 ± 2,3 núcleos |
| Índice de fase S | Núcleos de fase S / núcleos progenitoras zona | A + C / A + B + C + D | 57% do ciclo celular |
| Índice de M-fase | M-fase núcleos / núcleos progenitoras zona | A + B / A + B + C + D | 2% do ciclo celular |
| Células de entrada meióticas | EdU rotulado núcleos na meiose | E | varia de acordo com a duração do rótulo de EdU |
| Taxa de entrada meiótica | Núcleos de entrada meiótica por h do rótulo de EdU | Inclinação da Figura 4 C * * * | 20,3 núcleos por h |
| Duração de G2 (mediana) | interceptação de 50% de Figure4A | 2,5 h | |
| Duração de G2 (máximo) | interceptação de 99% da figura 4A | 3,5 h | |
| Duração do G1 + G2 + M (máximo) | 99% de interceptação de Figura 4B | 3,5 h | |
| Duração do ciclo celular (mediana) | duração mediana do G2 / G2-índice * * * | h 6,5 | |
| Duração do ciclo celular (máximo) | duração máxima do G2 / G2-índice * * * | h 8,1 | |
Tabela 2: cálculos de ciclo celular. * Letras representam as classes de núcleos definidos na tabela 1 e Figura 3. Cálculos são modificados de Fox et al . 20111. * * Valores (± desvio padrão) para o selvagem-tipo hermafroditas levantadas a 20 ° C, com idade para fase de meados-L4 post 24 h. Observe que, desde a intercepção de y-intercept não é zero, uma regressão é necessário para um cálculo exacto da taxa de entrada meiótica. O G2-índice é determinado subtraindo-se o índice de fase S e índice M-fase G1-índice (2%) aproximado de 100%, conforme descrito por Fox et al. 20111.
Discussão
Preparação de bactérias EdU-rotulado (etapa 1) é fundamental para este protocolo e o primeiro ponto para solução de problemas. Rótulo de tipo selvagem jovens adultos hermafroditas muito confiável em um EdU-pulso, inventando um controle útil para cada novo lote de bactérias EdU-rotulado de 4h. Além disso, intactas EdU-rotulado de bactérias que entram no intestino (em animais mais velhos ou certos mutantes defeituosos faringe/moedor) irão rotular com química clique e aparecem como partitura oblonga brilhante no intestino. Uma técnica alternativa para a rotulagem hermafroditas usa um "banho" em alta concentração (1mm) de EdU3. Esta técnica passa fome os animais para a duração da rotulagem, mas fornece uma maneira útil de contornar fazer EdU-rotulado bactérias ao solucionar problemas de fixação e clique em química. Se uma experiência de imersão"EdU" é bem sucedida, enquanto um feed de EdU não é, então prepare-se fresco bactérias EdU-rotulados. Para alcançar uma suficiente densidade bacteriana e também alcançar um alto teor de EdU, um talvez precise ajustar as concentrações de EdU e timidina.
A principal limitação desta técnica para a rotulagem de fase S é a necessidade de alimentar bactérias EdU-etiquetadas com os animais. Os animais que não podem alimentar (devido ao genótipo ou estágio) não podem ser rotulados com esta técnica. Não obstante, os análogos de nucleosídeos são atualmente o único método para identificar os núcleos de fase S no germline de c. elegans , e seu uso não exige que o qualquer transgenes estar presente nos animais. Além disso, uma vez incorporada, EdU permanece em núcleos mesmo enquanto sai fase S, progresso através do ciclo celular, divide ou diferencia. O sinal enfraquece pela metade com cada divisão celular. Isto faz com que seja perfeito para acompanhar a história de uma célula mesmo através de algumas divisões celulares EdU.
A estabilidade do EdU faz experimentos de pulso-perseguição simples; Basta enxaguar o excesso EdU bactérias dos animais após o pulso de duração desejada é terminado e transferir os animais para as bactérias sem rótulo. EdU permanece no DNA e permanece visível mesmo após várias divisões celulares. No entanto, as experiências são limitadas a um único tipo de rótulo de fase S (pulso único do EdU). Co etiquetando com EdU e BrdU em células de mamíferos,31 , é possível, mas não tem sido relatada em c. elegans. Co de rotulagem de IdU e CldU é usada em mamíferos32 mas também não tem sido relatada em c. elegans.
As principais vantagens do EdU rotulagem são que o método não requer nenhum transgenes, EdU pode ser alimentado a c. elegans durante cultura regular, a química é compatível com as técnicas de imunofluorescência e EdU persiste no DNA por muito tempo após a alimentação parou. Estas características fazem EdU, uma ótima ferramenta para estudar muitos aspectos da dinâmica do ciclo celular e células germinativas.
Análise de ciclo celular e células germinativas dinâmica com EdU pode ser aplicado a uma variedade de perguntas de pesquisa. Apenas alguns exemplos de outras aplicações deste método: como alterar a dinâmica do ciclo celular em animais com mutações de genes do ciclo celular? Como condições fisiológicas afetam o ciclo celular em células-tronco, a taxa de entrada de células germinativas em prófase meiótica e a taxa de progressão de células germinativas através da prófase meiótica? Durante o desenvolvimento larval como muda o ciclo celular? Como grandes perturbações via sinalização afeta o ciclo celular, além de alterações no destino da célula (tais como a proliferação de gravidez ectópica)? Este sistema pode ser modificado para estudar o que fazem as células em diversas condições.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Estamos gratos ao centro de estoque de Escherichia coli para MG1693; Wormbase; o centro de genética Caenorhabditis que é financiado pelos institutos de saúde escritório de pesquisa infra-estrutura programas nacionais (P40OD010440) para cepas; Zach Pincus para consultoria estatística; Aiping Feng para reagentes; Luke Schneider, Andrea Scharf, Sandeep Kumar e John Brenner para treinamento, aconselhamento, apoio e discussão útil; e os laboratórios Kornfeld e Schedl para feedback sobre este manuscrito. Este trabalho foi financiado em parte pelo National Institutes of Health [R01 AG02656106A1 à KK, R01 GM100756 TS] e uma bolsa predoctoral National Science Foundation [DGE-1143954 e DGE-1745038 de ZK]. O National Institutes of Health, nem a National Science Foundation tinha qualquer papel no desenho do estudo, coleção, análise e interpretação dos dados, nem por escrito o manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| E. coli MG1693 | Coli Genetic Stock Center | 6411 | grows fine in standard unsupplemented LB |
| E. coli OP50 | Caenorhabditis Genetics Center | OP50 | |
| Click-iT EdU Alexa Fluor 488 Imaging Kit | Thermo Fisher Scientific | C10337 | |
| 5-Ethynyl-2′-deoxyuridine | Sigma | 900584-50MG | or use EdU provided in kit |
| Glucose | Sigma | D9434-500G | D-(+)-Dextrose |
| Thiamine (Vitamin B1) | Sigma | T4625-5G | Reagent Grade |
| Thymidine | Sigma | T1895-1G | BioReagent |
| Magnesium sulfate heptahydrate | Sigma | M1880-1KG | MgSO4, Reagent Grade |
| Sodium Phosphate, dibasic, anhydrous | Fisher | BP332-500G | Na2HPO4 |
| Potassium Phosphate, monobasic | Sigma | P5379-500G | KH2PO4 |
| Ammonium Chloride | Sigma | A4514-500G | NH4Cl, Reagent Plus |
| Bacteriological Agar | US Biological | C13071058 | |
| Calcium Chloride dihydrate | Sigma | C3881-500G | CaCl |
| LB Broth (Miller) | Sigma | L3522-1KG | Used at 25g/L |
| Levamisole | Sigma | L9756-5G | 0.241g/10ml |
| Phosphate buffered saline | Calbiochem Omnipur | 6506 | homemade PBS works just as well |
| Tween-20 | Sigma | P1379-500ML | |
| 16% Paraformaldehyde, EM-grade ampules | Electron Microscopy Sciences | 15710 | 10ml ampules |
| 100% methanol | Thermo Fisher Scientific | A454-1L | Gold-label methanol is critical for proper morphology with certain antibodies |
| Goat Serum | Gibco | 16210-072 | Lot 1671330 |
| rabbit-anti-WAPL-1 | Novus biologicals | 49300002 | Lot G3048-179A02, used at 1:2000 |
| mouse-anti-pH3 clone 3H10 | Millipore | 05-806 | Lot#2680533, used at 1:500 |
| goat-anti-rabbit IgG-conjugated Alexa Fluor 594 | Invitrogen | A11012 | Lot 1256147, used at 1:400 |
| goat-anti-mouse IgG-conjugated Alexa Fluor 647 | Invitrogen | A21236 | Lot 1511347, used at 1:400 |
| Vectashield antifade mounting medium containing 4',6-Diamidino-2-Phenylindole Dihydrochloride (DAPI) | Vector Laboratories | H-1200 | mounting medium without DAPI can be used instead, following a separate DAPI incubation |
| nail polish | Wet n Wild | DTC450B | any clear nail polish should work |
| S-medium | various | see wormbook.org for protocol | |
| M9 buffer | various | see wormbook.org for protocol | |
| M9 agar | various | same recipe as M9 buffer, but add 1.7% agar | |
| Nematode Growth Medium | various | see wormbook.org for protocol | |
| dissecting watch glass | Carolina Biological | 42300 | |
| Parafilm laboratory film | Pechiney Plastic Packaging | PM-996 | 4 inch wide laboratory film |
| petri dishes | 60 mm diameter | ||
| Long glass Pasteur pipettes | |||
| 1ml centrifuge tubes | MidSci Avant | 2926 | |
| Tips | |||
| Serological pipettes | |||
| 500 mL Erlenmyer flask | |||
| Aluminum foil | |||
| 25G 5/8” needles | BD PrecisionGlide | 305122 | |
| 5ml glass centrifuge tube | Pyrex | ||
| Borosilicate glass tubes 1ml | |||
| glass slides | |||
| no 1 coverslips 22 x 40 mm | no 1.5 may work, also | ||
| 37 °C Shaker incubator | |||
| Tabletop Centrifuge | |||
| Clinical Centrifuge | IEC | 428 | with 6 swinging bucket rotor |
| Mini Centrifuge | |||
| 20 °C incubator | |||
| 4 °C refrigerator | |||
| -20 °C freezer | |||
| Observer Z1 microscope | Zeiss | ||
| Plan Apo 63X 1.4 oil-immersion objective lens | Zeiss | ||
| Ultraview Vox spinning disc confocal system | PerkinElmer | Nikon spinning disc confocal system works very well, also, as described here: http://wucci.wustl.edu/Facilities/Light-Microscopy |
Referências
- Fox, P. M., Vought, V. E., Hanazawa, M., Lee, M. -H., Maine, E. M., Schedl, T. Cyclin E and CDK-2 regulate proliferative cell fate and cell cycle progression in the C. elegans germline. Development. 138 (11), Cambridge, England. 2223-2234 (2011).
- Crittenden, S. L., Leonhard, K. A., Byrd, D. T., Kimble, J. Cellular analyses of the mitotic region in the Caenorhabditis elegans adult germ line. Molecular biology of the cell. 17 (7), 3051-3061 (2006).
- Seidel, H. S., Kimble, J. Cell-cycle quiescence maintains Caenorhabditis elegans germline stem cells independent of GLP-1/Notch. eLife. 4, (2015).
- Fox, P. M., Schedl, T. Analysis of Germline Stem Cell Differentiation Following Loss of GLP-1 Notch Activity in Caenorhabditis elegans. Genetics. 201 (9), 167-184 (2015).
- Kocsisova, Z., Kornfeld, K., Schedl, T. Cell cycle accumulation of the proliferating cell nuclear antigen PCN-1 transitions from continuous in the adult germline to intermittent in the early embryo of C. elegans. BMC Developmental Biology. 18 (1), (2018).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proceedings of the National Academy of Sciences. 105 (7), 2415-2420 (2008).
- vanden Heuvel, S., Kipreos, E. T. C. elegans Cell Cycle Analysis. Methods in Cell Biology. , 265-294 (2012).
- ThermoFisher. Click-iT EdU Imaging Kits. , https://assets.thermofisher.com/TFS-Assets/LSG/manuals/mp10338.pdf (2011).
- Pazdernik, N., Schedl, T. Germ Cell Development in C. elegans. , Springer. 1-16 (2013).
- Hirsh, D., Oppenheim, D., Klass, M. Development of the reproductive system of Caenorhabditis elegans. Developmental Biology. 49 (1), 200-219 (1976).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Hansen, D., Schedl, T. Stem cell proliferation versus meiotic fate decision in Caenorhabditis elegans. Advances in Experimental Medicine and Biology. 757, 71-99 (2013).
- Jaramillo-Lambert, A., Ellefson, M., Villeneuve, A. M., Engebrecht, J. Differential timing of S phases, X chromosome replication, and meiotic prophase in the C. elegans germ line. Developmental Biology. 308 (1), 206-221 (2007).
- Agarwal, I., Farnow, C., et al. HOP-1 presenilin deficiency causes a late-onset notch signaling phenotype that affects adult germline function in Caenorhabditis elegans. Genetics. 208 (2), 745-762 (2018).
- Gumienny, T. L., Lambie, E., Hartwieg, E., Horvitz, H. R., Hengartner, M. O. Genetic control of programmed cell death in the Caenorhabditis elegans hermaphrodite germline. Development. 126 (5), Cambridge, England. 1011-1022 (1999).
- Hendzel, M. J., Wei, Y., et al. Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106 (6), 348-360 (1997).
- Zetka, M. C., Kawasaki, I., Strome, S., Mü Ller, F. Synapsis and chiasma formation in Caenorhabditis elegans require HIM-3, a meiotic chromosome core component that functions in chromosome segregation. Genes & development. 13 (17), https://www.ncbi.nlm.nih.gov/pmc/articles/PMC317003/pdf/x8.pdf 2258-2270 (1999).
- Hansen, D., Hubbard, E. J. A., Schedl, T. Multi-pathway control of the proliferation versus meiotic development decision in the Caenorhabditis elegans germline. Developmental Biology. 268 (2), 342-357 (2004).
- Crawley, O., Barroso, C., et al. Cohesin-interacting protein WAPL-1 regulates meiotic chromosome structure and cohesion by antagonizing specific cohesin complexes. eLife. 5 (2), 1-26 (2016).
- Zhao, H., Halicka, H. D., et al. DNA damage signaling, impairment of cell cycle progression, and apoptosis triggered by 5-ethynyl-2′-deoxyuridine incorporated into DNA. Cytometry Part A. 83 (11), 979-988 (2013).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Gervaise, A. L., Arur, S. Spatial and Temporal Analysis of Active ERK. in the C. elegans Germline. Journal of Visualized Experiments. 117 (117), e54901-e54901 (2016).
- Vos, K. De Cell Counter Plugin. , https://imagej.nih.gov/ij/plugins/cell-counter.html (2015).
- Schindelin, J., Arganda-Carreras, I., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Rasband, W. ImageJ. , http://imagej.nih.gov/ij/> (2016).
- Michaelson, D., Korta, D. Z., Capua, Y., Hubbard, E. J. A. Insulin signaling promotes germline proliferation in C. elegans. Development. 137 (4), Cambridge, England. 671-680 (2010).
- Qin, Z., Jane, E., Hubbard, A., Hubbard, E. J. A. Non-autonomous DAF-16/FOXO activity antagonizes age-related loss of C. elegans germline stem/progenitor cells. Nature communications. 6 (5), 7107(2015).
- Luo, S., Kleemann, G. A., Ashraf, J. M., Shaw, W. M., Murphy, C. T. TGFB and Insulin Signaling Regulate Reproductive Aging via Oocyte and Germline Quality Maintenance. Cell. 143 (2), 299-312 (2010).
- Narbonne, P., Maddox, P. S., Labbe, J. -C. daf-18/PTEN locally antagonizes insulin signalling to couple germline stem cell proliferation to oocyte needs in C. elegans. Development. , 4230-4241 (2015).
- Cinquin, A., Chiang, M., et al. Intermittent Stem Cell Cycling Balances Self-Renewal and. Senescence of the C. elegans Germ Line. PLoS Genetics. 12 (4), 1005985(2016).
- Invitrogen EdU (5-ethynyl-2’-deoxyuridine). , https://assets.thermofisher.com/TFS-Assets/LSG/manuals/mp10044.pdf 1-7 (2010).
- Tuttle, A. H., Rankin, M. M., et al. Immunofluorescent Detection of Two Thymidine Analogues (CldU and IdU) in Primary Tissue. Journal of Visualized Experiments. 46 (46), e2166-e2166 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados