Method Article
Análisis de ciclo celular en la línea germinal de C. elegans con la EdU de análogo de la timidina
En este artículo
Resumen
Se describe un método basado en la proyección de imagen que puede utilizarse para identificar la fase S y analizar la dinámica del ciclo celular en el germline hermafrodita C. elegans mediante la timidina EdU analógico. Este método no requiere transgenes y es compatible con la tinción inmunofluorescente.
Resumen
Análisis de ciclo celular en eucariotas con frecuencia utiliza la morfología del cromosoma, expresión y localización de productos génicos necesarios para diferentes fases del ciclo celular, o la incorporación de análogos de nucleósidos. Durante fase de S, polimerasas de la DNA incorporan a análogos de timidina como EdU o BrdU DNA cromosómica, las células para el análisis de la marca. Para C. elegans, el nucleósido análogo EdU se alimenta a los gusanos durante cultura regular y es compatible con técnicas de inmunofluorescencia. Germinales de C. elegans es un sistema potente para los estudios de señalización de vías, las células madre, meiosis y ciclo celular porque es transparente, genéticamente facile y profase meiótica y la diferenciación celular/gametogénesis ocurre en un forma de montaje-como lineal. Estas características hacen de EdU una gran herramienta para el estudio de aspectos dinámicos de células mitotically ciclismo y desarrollo de la línea germinal. Este protocolo describe cómo preparar EdU bacterias con éxito, darles de comer a tipo salvaje hermafroditas de C. elegans , diseccionar la gónada hermafrodita, de la mancha para la incorporación de EdU en el ADN, tinción con los anticuerpos al detectar varios ciclo celular y marcadores del desarrollo, la gónada de la imagen y analizar los resultados. El protocolo describe las variaciones en el método y el análisis para la medición de fase S índice, fase M índice, G2 duración, duración del ciclo celular, tasa de entrada meiótica y tasa de progresión de la profase meiótica. Este método se puede adaptar para estudiar el ciclo celular o la historia de células en otros tejidos, etapas, fondos genéticos y condiciones fisiológicas.
Introducción
En el desarrollo animal, cientos, miles, millones, miles de millones o incluso billones de divisiones celulares se requieren para formar el organismo adulto. El ciclo celular, el conjunto de eventos celulares por G1 (gap), S (síntesis), G2 (gap), y M (mitosis) definir la serie de eventos que son ejecutaron cada división celular. El ciclo celular es dinámica y mejor apreciado en tiempo real, que puede ser técnicamente difícil. Las técnicas presentadas en este protocolo permiten hacer las mediciones de las fases y tiempos del ciclo celular de imágenes fijas.
Etiquetado con análogos de nucleósidos como 5-ethynyl-2'-desoxiuridina (EdU) o 5-bromo-2'-desoxiuridina (BrdU) es el estándar de oro para identificar la fase S en los estudios de la dinámica del ciclo celular en el adulto de Caenorhabditis elegans (C. elegans) hermafroditas del germline1,2,3,4,5. EdU y BrdU pueden utilizarse en casi cualquier fondo genético, como no dependen de ningún concepto genética. Visualización de BrdU requiere tratamiento químico áspero para exponer el antígeno para el anticuerpo anti-BrdU de tinción, que a menudo es incompatible con la evaluación de otros marcadores celulares visualizadas mediante tinción con anticuerpos adicionales conjuntamente. Por el contrario, visualizar EdU se produce por la química del tecleo condiciones suaves y por lo tanto es compatible con el anticuerpo Co manchas6,7.
La especificidad de la etiqueta es clara, puesto que los núcleos sólo incorporan los análogos de timidina (5-ethynyl-2'-desoxiuridina) ADN durante la fase S. Visualización lleva a cabo en tejido fijo. La etiqueta de EdU es invisible por sí mismo hasta un colorante que contiene azida o fluoróforo covalente reacciona con el alquino en EdU de química cobre-catalizada del tecleo8. Etiquetado EdU puede proporcionar información inmediata en la que los núcleos son en fase S, mediante un breve impulso de etiquetado. EdU también puede proporcionar información dinámica, mediante pulso-persiga o etiquetas de continuo; por ejemplo, en un experimento de Pulso-persiga, la etiqueta se diluye en cada división celular o reproducidos como nondividing progreso de células a través del desarrollo.
El germline hermafrodita C. elegans es un sistema potente para los estudios de señalización de vías, las células madre, meiosis y ciclo celular. El germline adulto es una línea de ensamblaje polarizada con células madre en el extremo distal, seguido por la entrada y la progresión a través de la profase meiótica, coordinado con las etapas de la gametogénesis más proximal (figura 1). En el extremo proximal, ovocitos maduran, son ovularon y fertilizan y comienzan la embriogénesis en el útero9,10,11. La región larga de ~ 20 células de diámetro cerca de la célula de la punta distal, que incluye el vástago del germline mitotically ciclismo, células progenitoras y células en fase S meióticas pero no las células en profase meiótica, se llama el progenitor zona2,4 , 9 , 12. las membranas celulares proporcionan separación incompleta entre los núcleos en el germline distal, pero las células de la zona de progenitoras se someten a ciclismo en gran parte independientemente de la célula mitótica. La duración mediana ciclo celular mitótico de las células de zona de progenitor del germline en jóvenes adultos hermafroditas es ~6.5 h; Fase G1 es breve o ausente, y quietud no se observa1,2,13. Diferenciación de la célula de vástago del germline se produce a través de la diferenciación esencialmente directa y carece así de tránsito amplificando las divisiones4. Durante la diferenciación en la fase de paquiteno, aproximadamente 4 de cada 5 núcleos no forma ovocitos pero en cambio sufren apoptosis, actuando como enfermera células donando su contenido citoplasmático al ovocito en desarrollo12,14 , 15.
Además de etiquetado células en fase S con análogos de nucleósidos, uno puede identificar las células en mitosis y meiosis utilizando anticuerpos. Núcleo en la mitosis es inmunorreactivas para anti-phospho-histona H3 (Ser10) anticuerpo (llamado pH3)7,16. Los núcleos en la meiosis son immunoreactive a los anticuerpos anti-lo-3 (una proteína del eje del cromosoma meiótico)17. Núcleos en la zona del progenitor pueden identificarse por la ausencia de él-3, la presencia de nucleoplasmática REC-818o la presencia de WAPL-119. WAPL-1 intensidad es más alta en la gónada somática, alta en la zona de la progenitora y baja durante temprano meiótica prophases19. Varias mediciones de ciclo celular son posibles con algunas variaciones en el protocolo: I) identificar los núcleos en fase S y medir el índice de la fase S; II) identificar los núcleos en fase M y medir el índice de fase M; III) determinar si los núcleos estaban en S-fase mitótica o meiótica; IV) medir la duración de G2; V) medir la duración de las fases G2 + M + G1; VI) medir la velocidad de entrada meiótica; VII) estiman la tasa de progresión meiótica.
Uno puede hacer múltiples mediciones de ciclo celular de sólo algunos tipos de experimentos de laboratorio húmedo. El protocolo a continuación describe un etiquetado de pulso de 30 min por la alimentación de C. elegans adultos hermafroditas con EdU etiquetado como bacterias y células en fase M Co etiquetadas mediante tinción con el progenitor y el anticuerpo anti-pH3 las células de la zona mediante tinción con anticuerpos anti-WAPL-1. Sólo cambios en la duración de EdU de la alimentación (paso 2.5), tipo de anticuerpos emplean (paso 5), y análisis (8,3 de paso) son necesarios para las mediciones adicionales.
Protocolo
1. preparación de bacterias EdU-labeled
-
Cultivar una cultura de arrancador de MG1693. Escherichia coli MG1693 (e. coli) lleva una mutación en thyA.
- Racha a e. coli MG1693 de un stock de glicerol congelado en un agar de caldo (LB) de lisogenia 120 mm plato de Petri. Cultivo a 37 ° C durante la noche.
- Inocular de dos individuales e. coli colonias MG1693 en dos tubos de 4 mL del cultivo líquido lb a 37 ° C para h ~ 16 de duplicar.
Nota: MG1693 crece bien en LB sin suplementar con timina o timidina.
-
Crecen MG1693 de e. coli en medios mínimos suplementados con EdU.
- Autoclave de un erlenmeyer de 500 mL.
- Utilizar técnica estéril para agregar 5 mL de glucosa 20%, 50 μL de 10 mg/mL de tiamina, 120 μL de timidina 5 mM, 100 μL de 1 M MgSO4, 200 μL de 10 mM EdU, 100 mL de buffer M9 y 4 mL de la cultura de MG1693 durante la noche recién crecido.
Nota: La concentración final de EdU es 20 μM en esta cultura1,7. Esta concentración conduce a la detención de daños y el ciclo celular de ADN si se aplica directamente a células de mamífero en cultivo20. Sin embargo, sólo una fracción de este EdU se incorpora en la e. coli y así disponible para C. elegans. Hay ninguna evidencia de la detención del ciclo celular y ningún cambio en el tamaño de la zona de la progenitora o índice de fase M después del tratamiento de EdU de jóvenes adultos hermafroditas. - La cultura durante la noche, pero no más de 24 h a 37 ° C con agitación a 200 rpm.
-
EdU-etiquetados e. coli se concentran y se aplica a platos de Petri agar M9.
- Pre-enfriar una centrifugadora de mesa a 4 ° C.
- Utilizar técnica estéril para transferir la cultura 2-4 tubos cónicos de 50 mL estéril.
- Centrifugue las culturas a 3.000 x g a 4 ° C durante 30 minutos para que sedimenten la EdU-etiquetados e. coli.
Nota: Deseche el sobrenadante que contiene EdU según las pautas locales e institucionales. - Vuelva a suspender los pellets con 4 mL de M9 fresco. Utilice una punta de la pipeta estéril 1 mL o una pipeta serológica de 5 mL estéril. Resuspender el pellet puede tardar varios minutos.
- Utilizar la misma pipeta para aplicar y difundir ~ 8 gotas de resuspendidos EdU-etiquetado de e. coli MG1693 al centro de temperatura 60 mm M9 agar Petri. Un lote da ~ 16 platos.
- Deje que los platos secar por varias horas o durante la noche a temperatura ambiente y luego sellar cada plato con una tira de película de laboratorio. Platos se pueden almacenar a 15 ° C durante 2 semanas y a 4 ° C durante 2 meses. Use el mismo lote de platos de EdU para cada conjunto de experimentos.
2. alimentación de EdU para C. elegans
- Sincronizar población huevo tiempo endecha, alcalinos hipoclorito tratamiento seguido por incubación en medio de S con el colesterol, o escogiendo la etapa adecuada21. Crecen los animales al escenario deseado (aquí, 24 h post-L4) en medio de crecimiento de nematodos con OP50 de e. coli a 20 º C.
Nota: Preparar 50 y 100 animales por experimento, como algunos no pueden diseccionar bien, y otros se perderán en el proceso de lavado y la transferencia. - Deje que los platos de EdU a 20 ° C (o la temperatura deseada para el experimento).
- Lavar los animales de los platos NGM con M9 o tampón fosfato salino (PBS) en un tubo de 1,5 mL. Permitir a los animales resolver brevemente por gravedad o una breve vuelta en una microcentrífuga.
- Quite el sobrenadante y lavar los animales 1 a 2 veces con ~ 1 mL de M9 o PBS. Eliminar el sobrenadante.
- Utilizar un pipeta Pasteur de vidrio para transferir los animales de una pequeña gota de M9 o PBS en el centro del césped de EdU. Espere unos minutos a que el líquido absorbido, luego incubar a 20 ° C durante 30 min (para medición directa de la fase S) o más (para medir la historia de la fase S), según sea necesario.
Nota: Señal de EdU es detectable en los núcleos germinales después de tan sólo 15 min de EdU alimentación1. - Lave los gusanos el plato de EdU con 2 mL de M9 o PBS en un vaso plato de disección.
3. disección y fijación de la línea germinal de C. elegans
Nota: Este protocolo para la disección, fijación y tinción de anticuerpos del germline hermafrodita C. elegans es casi idéntica a la publicada por Gervaise y Arur (2016)22, excepto que los tubos de vidrio de 1 mL pueden ser centrifugados para acelerar lavado de pasos y un vidrio tendida pipeta Pasteur se puede utilizar para quitar el líquido de los tubos de vidrio de 1 mL más con eficacia.
-
Lavar y disecar germlines de C. elegans .
- Permite los animales colocar en la parte inferior de la placa de disección, Remolino para recoger los animales en el centro y utilice una pipeta Pasteur largo para quitar PBS. Lavar 1 – 2 veces con 2 mL de PBS.
- Añadir 2 mL de PBS y 4 μL de levamisole 100 mM para inmovilizar a los animales. Agite el plato otra vez para recoger los animales en el centro del plato.
Nota: La inmovilización puede tardar entre unos segundos y unos minutos. Inmovilización completa no es necesario para la disección exitosa. Algunas personas tienen más éxito cuando incompleto de disección inmovilizados los animales. - Disecar los animales con un par de agujas de 25 5/8" por el corte en la cabeza (aproximadamente entre los dos bulbos faríngeas) o la cola. Tenga cuidado de no para cortar el lazo de la línea germinal. Intestino y línea germinal deben "saltar" de la cavidad del cuerpo debido a la presión interna, pero permanecer Unidos. Este protocolo es similar al previamente publicado Gervaise y Arur (2016)22.
Nota: Mantenga el tiempo de la disección a ~ 5 min, ciertamente no más de 15 minutos tiempo de disección puede resultar en la pérdida de señal tinción de anticuerpo (Sudhir Nayak, comunicación personal) y hambre en PBS puede afectar la fisiología de los animales. Aprender a diseccionar de forma rápida y precisa puede tomar algo de práctica. - Si es necesario, para recoger los animales disecados en el centro del remolino y utilizar una pipeta Pasteur largo para quitar tanta PBS como sea posible.
-
Fijan y deshidratan los tejidos
- Añadir 2 mL de 3% paraformaldehido (PFA) en solución de PBS. Cubrir el plato con laboratorio película y almacenar en un banco o en un cajón durante 10 minutos.
Nota: Descongelar solución de PFA a 37 ° C baño de agua y luego enfriar a temperatura ambiente antes de germlines de disección.
PRECAUCIÓN: Solución PFA es moderadamente tóxico y un probable carcinógeno y teratógeno. Emisión de soluciones paraformaldehido los vapores son inflamables. Guantes nitrilo. Diluir PFA del 16% al 3% en una campana de humos químicos. Cuando se trabaja fuera de la campana, mantenga todos los recipientes cubiertos. - Transferir las gónadas cuidadosamente a un tubo de centrífuga limpio 5 mL vidrio.
- Añadir 3 mL de PBSTw (PBS con un 0.1% Tween-20) al plato que contenía las gónadas para ayudar a recuperar las gónadas restantes y diluir la solución de la PFA.
- Giro en una centrífuga clínica a 870 x g ~ 1 min joven o animales más pequeños requieren tiempos más largos de spin que animales mayores o más grandes.
- Con una pipeta de vidrio larga, transferir el sobrenadante a vaso material no deseado para descartar eventual en botella de material no deseada en la campana química.
- Añadir 2 mL de metanol alto grado previamente enfriada a-20 ° C. Cubrir el tubo de centrífuga con película de laboratorio.
Nota: Uso de metanol fresco alto grado "gold label" es esencial para la adecuada morfología con ciertos anticuerpos.
PRECAUCIÓN: El metanol es un líquido altamente inflamable y el vapor, que es tóxico si se ingiere, inhala o permitido entrar en contacto con la piel. Use guantes y equipo de protección personal. Use un congelador adecuado para pequeñas cantidades de productos inflamables. - Tienda en congelador de-20 ° C por 1 h, incluso durante la noche o incluso varios meses.
Nota: El protocolo se puede detener aquí.
- Añadir 2 mL de 3% paraformaldehido (PFA) en solución de PBS. Cubrir el plato con laboratorio película y almacenar en un banco o en un cajón durante 10 minutos.
4. rehidratar Germlines
- Llene el tubo de centrífuga de vidrio en parte superior con PBSTw para diluir el metanol y rehidratar las gónadas. Girar hacia abajo en una centrífuga clínica a 870 x g ~ 1 min.
- Utilizando un pipeta Pasteur de vidrio largo, transferir el sobrenadante a vaso de material no deseado para descartar eventual en botella de material no deseada en la campana química.
- Lave las gónadas 3 veces con ~ 5 mL de PBSTw, girando hacia abajo en una centrífuga clínica a 870 x g ~ 1 min cada vez. Eliminar el sobrenadante.
- Enjuagar un tubo de vidrio borosilicato de pequeño 1 mL y un vaso largo pipeta Pasteur con PBSTw.
- Añadir ~ 700 μL de PBSTw y utiliza el vidrio larga pipeta Pasteur transferir las gónadas en el tubo pequeño. Utilice unas gotas adicionales de PBSTw para asegurar que se transfieren todas las gónadas.
- Girar hacia abajo en una centrífuga clínica x 870 g ~ 1 min, utilizando un vaso largo tendida pipeta Pasteur, quitar tanto líquido como sea posible sin molestar a las gónadas. Deje no más de 50 μL.
Nota: Si la detección de anticuerpos no es necesaria, vaya al paso 6.
5. detección de antígenos con anticuerpos
- Diluir los anticuerpos primarios en suero de 30% en PBS. En el presente ejemplo, anticuerpo anti-WAPL-1 es 1:2,000 diluido y anticuerpo anti-pH3 es diluido 1: 500. Centrifugar el suero recién descongelado durante 10 minutos en una microcentrífuga a 20.000 x g a 4 ° C para eliminar las partículas. Usar el sobrenadante, que puede ser almacenado a 4 ° C por varios días. Usar el suero adecuado el organismo anfitrión de anticuerpos secundarios (de suero de cabra se utiliza aquí). Un paso de bloqueo opcional puede agregarse antes de la adición de anticuerpos primarios.
- Se aplican 100 μL de anticuerpo primario diluido a cada tubo pequeño de vidrio. Incubar a temperatura ambiente durante 4 horas.
Nota: Tiempos de incubación varían por el anticuerpo. Para algunos anticuerpos, 2 h a temperatura ambiente es suficiente. Incubaciones más largo (por ej., durante la noche) son posibles, pero puede aumentar el fondo. - Llenar los tubos para top con PBSTw y gira hacia abajo en una centrífuga clínica x 870 g ~ 1 min.
- Lave las gónadas 3 veces utilizando ~ 1 mL de PBSTw. Incubar durante ~ 5 min por lavado para permitir exceso de anticuerpo primario difundir en lavado. Usando un dibujado-hacia fuera larga pipeta Pasteur de vidrio, quitar tanto líquido como sea posible sin molestar a las gónadas. Deje no más de 50 μL.
- Diluir los anticuerpos secundarios en suero de cabra 30% en PBS. En el presente ejemplo, cabra-anti-conejo-594 y cabra-anti-mouse-647 cada uno son diluidos al 1: 400.
Nota: Seleccionar anticuerpos secundarios cuidadosamente para asegurarse de que los tintes son distintos de la tintura en el kit de EdU. En el presente ejemplo, el kit de EdU contiene tinte de excitación de 488 nm. - Se aplican 100 μL del anticuerpo secundario diluido a cada tubo pequeño de vidrio. Incubar en la oscuridad a temperatura ambiente 2 h.
Nota: Tiempos de incubación varían por el anticuerpo. Para algunos anticuerpos secundarios, 1 h a temperatura ambiente es suficiente. Incubaciones más largo (por ej., durante la noche) son posibles, pero puede aumentar el fondo. - Lave las gónadas 3 veces utilizando ~ 1 mL de PBSTw. Incubar durante ~ 5 min por lavado para permitir exceso de anticuerpo secundario difuso en lavado. Usando un dibujado-hacia fuera larga pipeta Pasteur de vidrio, quitar tanto líquido como sea posible sin molestar a las gónadas. Deje no más de 50 μL.
Nota: Las gónadas pueden almacenarse en 100 μL de PBS después de este paso, si es necesario, aunque esto puede reducir la señal. Quitar el PBS antes de proceder.
6. realizar la reacción de EdU Click para detectar EdU
Nota: Realizar el EdU click reacción ante el anticuerpo tinción pasos (realizar paso 6 antes de Step 5) es posible, dependiendo de los anticuerpos se usan7. Sin embargo, los reactivos de clic pueden interferir con ciertos antígenos (e.g., REC 8 es sensible a la fijación y permeabilización). La orden presentada aquí produce anticuerpos brillante coloración con la REC-8, WAPL-1, él 3, pH3, bandera y CYE-1 anticuerpos utilizados, entre otros.
-
Preparar el clic EdU coctel8 fresco añadiendo lo siguiente a un tubo limpio de 1.5 mL. El orden de las adiciones es importante. Proteger de la luz y mantener todos los reactivos en el hielo. Esta receta rinde lo suficiente para una muestra (100 μL); multiplicar la receta según sea necesario.
- Añadir 2 mL de agua ultrapura en el búfer de aditivo. Esto hace inmediatamente antes de utilizar 10 x aditivo tampón, que debe ser diluido a 1 x.
- Añadir 8,5 μL de tampón de x 10 a 76,5 μL de agua ultrapura. Mezclar bien.
- Añadir 4 μL del 100 mM CuSO4 (puede ser etiquetado como componente E). Mezclar bien.
- Añadir 0,25 μL de colorante nm 488 azida. Se debe descongelar a temperatura ambiente, como su disolvente, el dimetil sulfóxido, es sólida a 4 ° C. Mezcla bien y proteger de la luz.
- Mix 9 μL de agua ultrapura con 1 μL del buffer añadido en la tapa del tubo. Pipeta de la tapa para añadir al cóctel restantes y mezclar bien mediante pipeteo arriba y abajo.
-
Realizar la reacción de clic de EdU.
- Añadir ~ 100 μL de click EdU coctel a las gónadas en el tubo pequeño. Cubra con película de laboratorio e incube durante 30-60 min a temperatura ambiente.
- Lavar una vez con 100 μL de tampón de reacción enjuague.
- Lave las gónadas 4 veces con ~ 1 mL de PBSTw. Incubar por ~ 15 min por lavado a permitir exceso EdU cóctel que los componentes se difunden en lavado. Usando un dibujado-hacia fuera larga pipeta Pasteur de vidrio, quitar tanto líquido como sea posible sin molestar a las gónadas. Deje no más de 50 μL.
7. ADN de la mancha y preparar diapositivas
- Añada 1 gota (~ 25 μL) antifade de medio de montaje con 4', 6-diamidino-2-phenylindole (DAPI, utilizado para visualizar el ADN) a las gónadas. Espere unos minutos para poder colocar y mezclar con las gónadas.
Nota: Alternativamente, una dilución de 1:1,000 de DAPI (de un stock de 0,1 mg/mL) en PBS puede aplicarse por 5 min, seguido de 20 μL de 1, 4-Diazabiciclo [2.2.2] octano (DABCO) en 90% de glicerol, o medio de montaje antifade otro. - Preparar un gran cojín de agarosa 2,5% en un portaobjetos de microscopio de vidrio estándar.
- Uso un nuevo limpio libre de polvo cristal larga pipeta Pasteur transferir las gónadas a la almohadilla de agarosa. Mantenga todos los líquidos y las gónadas en el estrecha inferior de la pipeta para minimizar la pérdida de las gónadas.
Nota: Las gónadas pegan al tubo pequeño de vidrio o de cristal larga pipeta Pasteur puede ser "rescatado" enjuague con PBSTw, recogiendo el líquido en un plato de disección y recogiendo los animales en el portaobjetos con una pestaña. - Uso una pestaña o un lazo del pelo fino pegado a un palillo de dientes para distribuir las gónadas sobre la base de agarosa y eliminar las partículas de polvo.
- Se aplica un cubreobjetos de vidrio rectangular. Baja lentamente de un lado para evitar burbujas de aire. Utilice un pañuelo de papel para eliminar el exceso de solución evitando el cubreobjetos se mueva libremente.
Nota: Elija cubre-objetos que coinciden con el microscopio que se utilizará. #1 y #1.5 cubreobjetos funcionan bien. - Deje que los portaobjetos y secar ligeramente durante la noche a temperatura ambiente o 4 ° C. Esto ayuda a aplanar un poco las gónadas. Portaobjetos deben almacenarse a 4 ° C.
- Opcional: Sellar los bordes de la diapositiva con esmalte de uñas u otro sellador de diapositiva. Sellado de las esquinas primero, luego los lados, impide que el cubreobjetos desplazamiento.
8. Análisis y proyección de imagen confocal
- Imagen la gónada distal con un microscopio de fluorescencia confocal spinning disco equipado con una fuente de luz de alta energía, objetivos plan-apocromática y una cámara de microscopio de alta eficiencia. Captura las imágenes con 1 μm o mayor espaciamiento entre pilas de z. Tomar nota del tiempo de energía, la sensibilidad y la exposición de láser para todos los canales.
- Utilice el siguiente: 405 nm láser línea excitación con un filtro de emisión de 485 nm (W60) para DAPI, 488 nm láser línea excitación con un filtro de emisión 527 nm (W55) para EdU, 561 excitación de línea láser de nm con un filtro de emisión de 615 nm (W70) WAPL-1 y a 640 nm láser línea excita ción con un filtro de emisión de 705 nm (W90) de pH3.
Nota: Intensidad de la señal y la intensidad de fondo varían. Asimismo, los tiempos de exposición requeridos variará, posiblemente hasta 10 veces.
- Utilice el siguiente: 405 nm láser línea excitación con un filtro de emisión de 485 nm (W60) para DAPI, 488 nm láser línea excitación con un filtro de emisión 527 nm (W55) para EdU, 561 excitación de línea láser de nm con un filtro de emisión de 615 nm (W70) WAPL-1 y a 640 nm láser línea excita ción con un filtro de emisión de 705 nm (W90) de pH3.
- Utilice el plug-in de contador celular23 en Fiji24,25 para contar manualmente cada núcleo. Etiqueta cada núcleo individual según la presencia y la ausencia de pH3, EdU y WAPL-1. Utilice las clases de núcleos que se describen en la tabla 1 y figura 2, como éstos facilitará todos los cálculos que se detallan a continuación.
Nota: Los investigadores calificados pueden contar exactamente todos los núcleos en una imagen 3D sin contar doble o falta cualquier núcleos. Alternativamente, uno puede contar cada núcleo en cada plano de z, y el script de marcas a células R3 se puede usar para eliminar núcleos multiplica-contado. - Calcular el número de células y las frecuencias de las cuentas anteriores dependiendo del tipo de medida del ciclo celular necesitada. Los tipos de núcleos se definen en la tabla 1 y figura 2. Los cálculos se resumen en la tabla 2.
- (Variación I) Para identificar los núcleos en fase S, alimentar los animales EdU durante 30 minutos. Cualquier núcleo Mostrar etiqueta de EdU es núcleos de S-phase. Para calcular, tomar los núcleos suma A y C, ver tabla 1 y figura 2.
Nota: En un minuto 30 EdU pulso en salvaje-tipo adultos hermafroditas, EdU todos etiquetados núcleos son co etiquetados con progenitor zona marcadores1. - Para medir la zona de la progenitora, de la mancha con un marcador de zona de progenitor como REC-8 o WAPL-1 anticuerpo. La zona de progenitoras se define aquí como todos nucleoplasmática REC-818 núcleos de immunoreactive del germline WAPL-1. Para calcular, en definitiva todos los núcleos immunoreactive WAPL-1 (A + B + C + D, ver tabla 1 y figura 2).
Nota: 1 WAPL etiquetas también el DTC y los núcleos somáticos gónada que no deben ser contados. Núcleos somáticos son fáciles de identificar por la señal de WAPL-1 extremadamente intensa, posición ligeramente fuera de la línea germinal y una morfología de "huevo frito" de los núcleos. - Para medir el índice de la fase S, realizar un experimento de EdU de 30 min y co etiqueta con anticuerpo REC-8 o WAPL-1. El índice de fase S se define como la proporción de la zona del progenitor que se encuentra en fase de S. Para calcular, contar todos los núcleos de fase S y luego dividir por el número total de núcleos de la zona de progenitor (A + C / A + B + C + D, ver tabla 1 y figura 2).
- (Variante II) Para identificar los núcleos en fase M, tinción con anticuerpos de pH3. Cualquier pH3 immunoreactive núcleos son núcleos de fase M. Esto funciona independientemente de la duración de la UAE de la alimentación. Para calcular, tomar la suma de la A y núcleos B, ver tabla 1 y figura 2.
Nota: 1 WAPL etiquetas también el DTC y los núcleos somáticos gónada que no deben ser contados. Núcleos somáticos son fáciles de identificar por la señal de WAPL-1 extremadamente intensa, posición ligeramente fuera de la línea germinal y una morfología de "huevo frito" de los núcleos. - Para medir el índice de la fase M, co etiqueta pH3 y REC-8 o anticuerpos WAPL-1. El índice de la fase M se define como la proporción de la zona del progenitor que se encuentra en fase M. Para calcular, contar todos los núcleos de fase M y luego dividir por el número total de núcleos de la zona de progenitor (A + B / A + B + C + D, ver tabla 1 y figura 2).
- (Variación III) Los núcleos en fase S mitotic y meiotic ambos etiqueta con EdU. A decir de las dos poblaciones separadas, determinar si la fase de S fue seguida por mitosis o por meiosis. Para determinar si los núcleos en fase S meiótica o mitótica, alimentar a EdU por 4 h y co etiqueta para pH3 (un marcador de fase M) y él-3 (una proteína del eje del cromosoma meiótico) por anticuerpos. Grabar los núcleos que EdU y pH3 (tipo A, ver cuadro 1 y figura 2) como mitótica fase S mientras que los núcleos que EdU y él-3 (tipo E, ver tabla 1 y figura 2) como S-fase meiótica.
- (Variación IV) Calcular la duración de la fase G2.
Nota: Fase G2 separa fase S fase M. Aunque se ha reportado ningún marcador que etiqueta G2 en el germline de C. elegans , uno puede calcular la duración de la fase G2 al combinar datos de varios experimentos que la fase M (en el momento de la disección) y fase de S (a partir de varios h antes de la etiqueta disección). Una célula que muestra los marcadores de la fase S y fase M había terminado fase G2 durante el curso del experimento. Una célula que muestra sólo el marcador de la fase M y no el marcador de la fase S no estaba en fase S durante el experimento.- Para calcular la duración de la fase G2, pienso EdU para h 2 y co etiqueta con anticuerpo de pH3. Examinar sólo los núcleos que etiqueta con pH3 (estos están en fase de M en el momento de la disección) de la presencia de EdU (eran en fase S en la etiqueta de EdU 2 h antes de la disección). Calcular la fracción de núcleos de fase M que completaron la fase G2 (A / A + B, ver tabla 1 y figura 2).
- Repite este experimento con una etiqueta de EdU 3 h y otra vez con una etiqueta de EdU 4 h (y, opcionalmente, una etiqueta de EdU 5 h). Representar el porcentaje de núcleos positivos pH3 que son positivos en el eje y de EdU y la duración de la etiqueta de EdU en el eje x, como se muestra en la Figura 3A.
- Calcular la mediana de la duración de la fase G2 conectando los puntos de la gráfica y determinar donde la línea cruza el 50%, como se muestra en la Figura 3A.
- Calcular la duración máxima de la fase G2 conectando los puntos de la gráfica y determinar donde la línea cruza el 99%, como se muestra en la Figura 3A.
- (Variación de V) Calcular la duraion de G2 + M + G1.
Nota: En la línea germinal de C. elegans , fase G1 es inusualmente corto. Aunque no se ha reportado ningún marcador para etiquetar G1 en el germline de C. elegans , uno puede estimar la duración de la suma de la fase G1, G2 y M y luego comparar este tiempo con el tiempo de la fase G2 descrito anteriormente. Se estima que la duración máxima de G2 + M + G1 desde el porcentaje de todos los núcleos zona progenitoras (WAPL-1 immunoreactive) que siendo negativo de EdU (fase S no experimentaron) después EdU etiquetado durante varias horas.- Para calcular la duración de G2 + M + G1, pienso EdU para h 2 y co etiqueta con anticuerpo REC-8 o WAPL-1. Determinar la fracción de la zona de progenitoras que experimentó la fase S durante este tiempo (A + C / A + B + C + D, ver tabla 1 y figura 2).
- Repite este experimento con una etiqueta de EdU 3 h y otra vez con una etiqueta de EdU 4 h (y, opcionalmente, una etiqueta de EdU 5 h). Representar el porcentaje de REC-8 o 1 WAPL núcleos positivos que son positivos en el eje y EdU y la duración de la etiqueta de EdU en el eje x, como se muestra en la figura 3B.
- Calcular la máxima duración de G2 + M + G1 conectando los puntos en el gráfico y encontrar donde la línea cruza el 99%, como se muestra en la figura 3B.
Nota: Es posible realizar los experimentos para determinar la duración de G2 y G2 + M + G1 como un conjunto único de 2, 3, 4 y 5 h EdU experimentos Co etiquetado con anticuerpos de conejo anti-WAPL-1 y anti-mouse-pH3.
- (Variación de VI) Para identificar los núcleos que en la zona de la progenitora, pero han entrado meiosis, alimentar los animales EdU durante 10 h y co etiqueta con anticuerpos REC-8 o WAPL-1. Cualquier núcleos Mostrar etiqueta de EdU estaban en fase S durante esos 10 h. Cualquier núcleo que no se muestran tinción REC-8 o 1 WAPL nucleoplasmática fueron en la meiosis. Simplemente contar los núcleos con etiquetado de EdU que no muestran el etiquetado con el marcador de zona de progenitoras (E, ver tabla 1).
Nota: Por el contrario, si las gónadas están manchadas para el marcador de la profase meiótica HIM-3 con anticuerpos anti-él-3, cuenta el número de núcleos con EdU de etiquetado son también positivas para él-3. - Para calcular la tasa de entrada meiótica, realizar el experimento anterior con una marca de 5 h, 10 h y 15 h de EdU. Representar el número de núcleos que en la meiosis en el eje y y la duración de la etiqueta de EdU en el eje x, como se muestra en la figura 3. Luego utilizar una regresión lineal simple para calcular la pendiente (meiosis núcleos entrados por h) desde y = mx + b.
Nota: Es fundamental utilizar una regresión lineal para calcular la tasa de entrada meiótica. Sería incorrecto simplemente divida el número de núcleos que entró en la meiosis por la duración de la etiqueta de EdU, ya que la intersección no es cero. - (Variación VII) Medir la velocidad de progresión meiótica.
Nota: Puesto que EdU se incorpora covalentemente en el ADN, puede utilizarse para realizar un seguimiento de una población de células a través de la diferenciación. Las células que experimentaron la fase S en la zona de progenitor conservan la etiqueta de EdU ya que entra en meiosis, progreso a través de la meiosis y se someten a ovogénesis. Un experimento de Pulso-persiga con EdU puede utilizarse para medir la velocidad de progresión meiótica.- Alimentación etiquetada EdU las bacterias a los animales durante 4 horas (el "pulso"). Transferir los animales a las bacterias sin etiqueta de OP50 durante 48 h ("chase"), a continuación diseccionar y co la etiqueta con un marcador de zona de progenitor como REC-8 o WAPL-1 (o un marcador de la profase meiótica como él-3) si se desea.
- Cuando la proyección de imagen, busque la posición del núcleo más próximo marcado con EdU. La tasa de progresión meiótica es la distancia (en diámetros de celular desde el final de la zona de progenitor) viajó por el EdU más proximal marcado núcleo durante la persecución de 48 h.
- (Variación I) Para identificar los núcleos en fase S, alimentar los animales EdU durante 30 minutos. Cualquier núcleo Mostrar etiqueta de EdU es núcleos de S-phase. Para calcular, tomar los núcleos suma A y C, ver tabla 1 y figura 2.
Resultados
Puesto que la síntesis de ADN es necesaria para incorporar a EdU, se puede concluir que núcleos marcados con EdU experimentaron la fase S durante la ventana Etiquetado EdU. Uno puede interpretar los núcleos etiqueta en una alimentación de 30 min con EdU etiquetado bacterias como núcleos en fase S en el momento de la disección. Núcleos que etiqueta en un EdU continua ya experimento de alimentación indicados en la última parte de la ventana del tiempo EdU o pueden han etiquetado como temprano en la ventana de tiempo y desde la izquierda S-phase. Señal de EdU Co se localiza con la señal de DAPI. En algunos núcleos, EdU señal cubre todos los cromosomas, mientras que en otros núcleos EdU señal se localiza a 1 – 2 brillante orificio (figura 4). Estos puncta probablemente son el cromosoma x, que repeticiones finales de fase S13.
Aquí, los animales fueron alimentados con EdU continuamente por 30 min y disecado, como se describe anteriormente y en la figura 5. Un ejemplo de éxito EdU tinción en un animal adulto joven y un ejemplo de éxito EdU tinción en un animal adulto mayores (véase abajo) se muestran en la figura 4. Señal de EdU de un etiquetado de 30 min se localiza a aproximadamente la mitad de los núcleos en la zona de progenitoras (definido por WAPL-1 anticuerpo etiquetado pero aproximados por DAPI morfología26,27,28). Índice de fase S, la proporción de la zona del progenitor que es EdU positivo, fue previamente divulgados en 57 ± 5% y tan alto como 70% en adultos jóvenes1,2,3. Índice de fase M es de aproximadamente 2 – 3%1,29. En continuo de alimentación para 4 h o más, todos los núcleos en la etiqueta de zona de progenitoras con EdU y algunos núcleos en la zona del progenitor que ya entraron en profase meiótica1.
Si bien la técnica trabaja constantemente en animales adultos jóvenes de tipo salvaje, una fracción significativa de acoplado hermafroditas 5 días de edad (incluso los que contienen espermatozoides) no etiqueta en 30 min EdU pulso (figura 4E). Sin embargo, con un 4 h EdU alimentación, casi todos estos animales etiqueta. Fallo esporádico en etiqueta de hembras genéticas con pulsos cortos de EdU también ha sido reportado30. Puede haber otras situaciones que resultan en fracaso esporádico a etiqueta.
Se puede calcular la duración del ciclo celular mediante la realización de varios experimentos EdU-etiquetado con pH3 etiquetado en cada uno. La duración de G2 se estimó mediante el análisis el porcentaje de núcleos en fase M (pH3 immunoreactive) que fueron EdU positiva durante el curso del tiempo (figura 6). Este enfoque da mediana y máxima duración de G2 (Figura 3A). La mediana de tiempo fue interpolada, con una duración aproximada de la G2 de 2,5 h en jóvenes adultos hermafroditas. La duración de G2 + M + G1 fue estimada del porcentaje de todos los núcleos zona progenitoras (WAPL-1 immunoreactive) que fueron EdU positivo (figura 6). El método de G2 + M + G1 proporciona una medida de la duración máxima para las fases combinadas (figura 3B). El tiempo de percentil 99 fue interpolado, con una duración aproximada de la G2 + M + G1 de 3,4 h en jóvenes adultos hermafroditas. Datos de los mismos experimentos fueron utilizados para calcular la tasa de entrada meiótica (núcleos por h). La velocidad es la pendiente de la regresión lineal del número de núcleos que entrado en meiosis (EdU positivo, positivo negativo o HIM-3 WAPL-1) sobre la duración de la etiqueta de EdU (figura 3). Los valores de tipo salvaje hermafroditas de adulto 1 día de edad se muestran en la tabla 2.
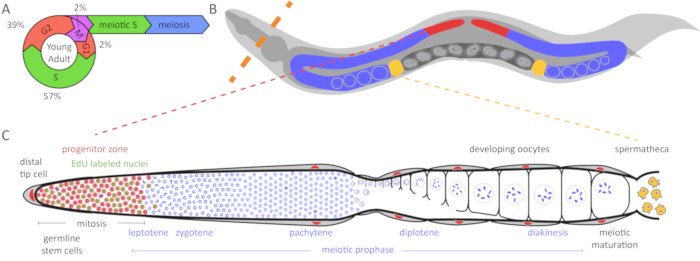
Figura 1: Diagrama de C. elegans del germline y ciclo celular. (A) del ciclo celular de las células germinales en el germline hermafrodita adulto joven. Los números indican el porcentaje aproximado de tiempo de permanencia en cada etapa del ciclo celular. (B) hermafroditas de C. elegans tienen dos germlines en forma de U (rojo y azul). En amarillo se muestra la espermateca y el útero con el desarrollo de embriones se muestra en gris oscuro. La línea naranja discontinua indica donde se disecan animales para sacar el germlines. (C) diagrama de un desplegado de C. elegans germline. DAPI (azul) es un colorante de ADN que resalta la morfología nuclear. La zona distal del progenitor (resaltada en rojo, basado en la tinción de anticuerpos de 1 WAPL) contiene células mitotically ciclismo, células progenitoras y células en fase S meiótica (WAPL-1 etiquetas también núcleos somáticos gónada). Las células en fase S mitotic y meiotic de la etiqueta con un 30 min EdU pulso y están indicadas en verde. Dos células en fase M etiqueta con pH3 anticuerpo y se muestran en negro. La célula de la punta distal (DTC) proporciona el ligando de GLP-1/muesca para mantener el destino de la célula de vástago de estas células. Como las células migran de la DTC, salir de la zona de progenitor y entrar en la profase meiótica. Las células amarillas son espermatozoides de la espermateca. Haga clic aquí para ver una versión más grande de esta figura.
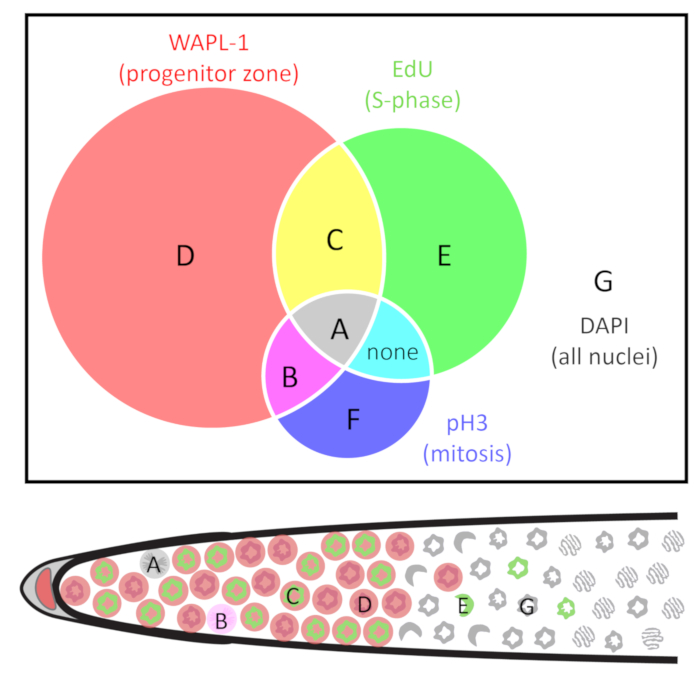
Figura 2: Diagrama de Venn de las clases de núcleos. Los núcleos son agrupados por la presencia y la ausencia de tres marcadores: WAPL-1 indica células progenitoras de la zona (rojo), EdU indica células en fase S (verde) y pH3 indica células en fase M (azul). Tipos de la célula se identifican como A G. Nota que en tipo salvaje jóvenes adultos hermafroditas no se encuentran células de tipo F, y las células no etiqueta junto con EdU y pH3 fuera el progenitor (WAPL-1 positivo) zona. El siguiente diagrama de gónada distal indica un ejemplo de A E y G núcleos. Para más detalle vea la tabla 1 . Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: presentación gráfica de duración del ciclo celular y tasa de datos experimentales meiótica entrada. (A) la duración de G2 fase es interpolada de pH3 y EdU Co etiquetado siguiente duración variada EdU pulsos líneas grises indican los percentiles 50 y 99 usados en la interpolación de mediana y máxima duración de G2, indicado por las flechas. (B) una célula en fase G1, G2 y M no incorpora EdU. Por lo tanto, la duración máxima de la fase G2 + M + G1 puede estimarse mediante la medición de la duración máxima de la etiqueta de EdU que produce las células EdU-negativas. La duración de la fase G2 + M + G1 es interpolada de EdU y los REC-8 Co etiquetado después EdU pulsos de duración variada. Línea gris indica 99 percentil utilizado en interpolación máxima duración de G2 + M + G1, indicada por una flecha. No es posible interpolar la mediana de la duración G2 + M + G1. (C) la tasa de entrada meiótica (en núcleos por h – ver tabla 2) se calcula de la pendiente de la recta de regresión. Tenga en cuenta que desde la intercepción de la intersección no es cero, una regresión es necesaria un cálculo exacto de la tasa de entrada meiótica (C). . Barras de error indican la desviación estándar. Figuras modificado y reproducido con permiso de Fox et al. 20111. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: ejemplo de éxito y fracasado 30 min EdU tinción. Imágenes de microscopio confocal de un 1 día de edad (A-D) y una gónada hermafrodita 5 días de edad (no agotada la esperma) de (E H) después de un minuto 30 experimento etiquetado EdU. La línea blanca discontinua marca el final de la zona de la progenitora. El asterisco marca la posición de la punta distal. Marcas verdes EdU tinción visualizado por haga clic en química (A). Éxito EdU etiquetado resulta en bajo nivel fondo manchas pero no brillante EdU + núcleos (E). Inmunofluorescencia de marcas rojas WAPL-1 (B, F). Amarillo indica superposición (C, G). Marcas azul DAPI de tinción para ADN (D, H). Solo puntas de flecha indican un núcleo con EdU la coloración a lo largo de la cromatina. Doble las puntas de flecha indican un núcleo con EdU puncta en sólo un par de cromosomas. Imágenes fueron obtenidas con un objetivo X 63. Barra de escala = 10 μm (D, H). Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: flujo de trabajo Experimental. Un resumen del protocolo experimental para crecer (A), etiqueta de EdU (B), disección (C), tinción de anticuerpo (D), realizar la reacción de click para colocar un tinte a EdU (E), tinción de DNA (F), germlines (G), la imagen y cuantificar EdU etiquetado y anticuerpo teñido núcleos (H). Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: ejemplo de éxito 4 h EdU tinción. Imágenes de microscopio confocal de una gónada hermafrodita adulto 1 día de edad después de un experimento etiquetado EdU de 4 h. La línea blanca discontinua marca el final de la zona de la progenitora. El asterisco marca la posición de la punta distal. Magenta marca pH3 inmunofluorescencia (A, C). Marcas verdes EdU tinción visualizado por haga clic en química (B, C). Inmunofluorescencia de marcas rojas WAPL-1 (D). Amarillo indica la superposición de EdU y WAPL-1 (E). Marcas azul DAPI de tinción para ADN (F). Solo puntas de flecha indican núcleos marcados conjuntamente con EdU y pH3. Punta de flecha doble marca un pH3 + EdU-núcleo - una ocurrencia rara en un etiquetado de EdU 4 h. Las flechas marcan EdU + WAPL-1 - núcleos que han entrado en meiosis. Imágenes fueron obtenidas con un objetivo X 63. Una barra de escala de 10 μm se muestra (F). Haga clic aquí para ver una versión más grande de esta figura.
| Marcador: | pH3 | EdU * | 1 WAPL o REC-8 | LE-3 | |
| Interpretación: | Mitosis | S-fase * | Zona de progenitor | Meiosis | |
| Clase: | Interpretación combinada: | ||||
| A |  |  |  |  | en la fase M, en zona de progenitor, estaban en S-fase mitótica durante EdU etiqueta (G2 completo) |
| B |  |  |  |  | en la fase M, en zona del progenitor, no estaban en fase S durante etiqueta de EdU |
| C |  |  |  |  | en la interfase, en la zona de la progenitora, estaban en fase S durante etiqueta de EdU |
| D |  |  |  |  | en la interfase, en la zona de la progenitora, no estaban en fase S durante etiqueta de EdU |
| E |  |  |  |  | en la meiosis, estaban en fase S meiótica durante EdU etiqueta (núcleos de entrada meiótica) |
| F |  |  |  |  | volver a la mitosis (se encuentra en algunas mutantes) o divisiones meióticas (en espermatogénesis) |
| G |  |  |  |  | en la meiosis, no fueron en fase S en la etiqueta de EdU |
| suma total pH3 positivo; todas las células en fase M | suma total EdU positiva; todas las células en fase S | suma total WAPL-1 positiva; todas las células en la zona del progenitor | suma total positivo de él-3; todas las células en profase meiótica |
Tabla 1: clases de núcleos. * Tenga en cuenta que 30 min y 4 h EdU experimentos difieren en la interpretación. En una duración más larga EdU experimentos, las células probablemente han progresado más allá de la fase S. Ver introducción y paso 8 para duración de EdU etiquetado para experimento relevante.
| Parte del ciclo celular | Definición operacional | Cálculo * | Valor ** |
| Núcleos de la zona del progenitor | núcleos negativos positivos, él 3 todos WAPL-1 (o REC-8) | A + B + C + D | 231 ± 23 núcleos |
| Núcleos de la fase S | núcleos EdU positivo después de 30 min EdU etiqueta y WAPL-1 positivo | A + C | 133 ± 20 núcleos |
| Núcleos de fase M | pH3 y núcleos co-positive WAPL-1 | A + B | 5,2 ± 2.3 núcleos |
| Índice de fase S | Núcleos de fase S / núcleos de la zona del Progenitor | A + C / A + B + C + D | 57% del ciclo celular |
| Índice de fase M | Núcleos de fase M / núcleos de la zona del Progenitor | A + B / A + B + C + D | 2% del ciclo celular |
| Células meióticas de entrada | Etiquetado como núcleo en la meiosis de la EdU | E | varía según la duración de la etiqueta de EdU |
| Tasa de entrada meiótica | Núcleos de entrada meiótica por h de etiqueta de EdU | Pendiente de la figura 4 C *** | 20,3 núcleos por h |
| Duración de G2 (mediana) | intercepción de 50% de Figure4A | 2,5 h | |
| Duración de G2 (máximo) | 99% de intercepción de Figura 4A | 3,5 h | |
| Duración de G2 + M + G1 (máximo) | 99% de intercepción de la Figura 4B | 3,5 h | |
| Duración del ciclo celular (mediana) | mediana duración de G2 / G2-índice *** | 6,5 h | |
| Duración del ciclo celular (máximo) | máxima duración de G2 / G2-índice *** | h 8,1 | |
Tabla 2: cálculo del ciclo celular. * Letras representan las clases de núcleos definidos en tabla 1 y figura 3. Cálculos son modificados de Fox et al. 20111. ** Los valores (± desviación estándar) de tipo salvaje hermafroditas levantados a 20 º C a etapa de 24 h post mediados de L4 años. Tenga en cuenta que desde la intercepción de la intersección no es cero, una regresión es necesaria un cálculo exacto de la tasa de entrada meiótica. El índice de G2 se determina restando el índice de la fase S, índice de fase M y G1-índice aproximado (2%) de 100%, según lo descrito por Fox et al. 20111.
Discusión
Preparación de bacterias etiquetadas de EdU (paso 1) es fundamental para este protocolo y el primer punto para solucionar problemas. Etiqueta tipo salvaje jóvenes adultos hermafroditas muy confiablemente en un 4 h EdU-pulso, haciendo de este un control útil para cada lote nuevo de bacterias EdU-labeled. Además, bacterias EdU-con la etiqueta intactas que entran en el intestino (en animales más viejos o ciertos mutantes defectuosos de faringe grinder) etiqueta con química del tecleo y aparecen como brillante orificio oblongo en el intestino. Una técnica alternativa para el etiquetado hermafroditas utiliza un "remojo" en una alta concentración (1 mM) de EdU3. Esta técnica muere de hambre los animales para la duración de las etiquetas, pero proporciona una forma útil de bypass que hace EdU-labeled bacterias cuando solución de problemas de fijación y haga clic en química. Si un experimento de EdU "remojo" es correcta aunque no es un feed de EdU, luego preparar frescas bacterias EdU-labeled. Para alcanzar una suficiente densidad bacteriana logrando también un alto contenido de EdU, uno necesita ajustar las concentraciones de EdU y timidina.
La principal limitación de esta técnica para el etiquetado de fase S es en la necesidad de alimentar bacterias etiquetadas de EdU a los animales. Los animales que no se alimentan (debido al genotipo o etapa) no pueden ser etiquetados con esta técnica. Sin embargo, análogos de los nucleósidos análogos son actualmente el único método para identificar los núcleos de fase S en la línea germinal de C. elegans , y su uso no requiere que cualquier transgenes estar presente en los animales. Además, una vez incorporado, restos de EdU en núcleos como salida fase de S, progreso a través del ciclo celular, dividirán o diferenciaran. La señal se debilita por la mitad con cada división celular. Esto hace que EdU perfecto para el seguimiento de la historia de una célula incluso a través de algunos divisiones de célula.
La estabilidad de EdU hace Pulso-persiga los experimentos sencillos; simplemente enjuague exceso EdU bacterias de los animales después de que termine el pulso de la duración deseada y traslado de los animales a las bacterias sin etiqueta. EdU sigue siendo en el ADN y permanece visible incluso después de varias divisiones de célula. Sin embargo, los experimentos se limitan a un único tipo de etiqueta de fase S (un solo pulso de EdU). Conjunto de etiquetado con EdU y BrdU es posible en células de mamíferos31 pero no se ha divulgado en C. elegans. Etiquetado conjunto de IdU y CldU se utiliza en mamíferos32 pero también no se ha divulgado en C. elegans.
Las principales ventajas del etiquetado EdU son que el método no requiere transgenes, EdU puede ser alimentado a C. elegans durante cultura regular, la química es compatible con técnicas de inmunofluorescencia y EdU persiste en el ADN durante mucho tiempo después de la alimentación se detuvo. Estas características hacen de EdU una gran herramienta para el estudio de muchos aspectos de la dinámica del ciclo celular y la célula de germen.
Análisis de dinámica de ciclo celular y la célula de germen con EdU puede aplicarse a una variedad de preguntas de investigación. Algunos ejemplos de otras aplicaciones de este método: ¿Cómo cambiar la dinámica del ciclo celular en los animales con mutaciones de gene del ciclo celular? ¿Cómo afectan las condiciones fisiológicas del ciclo celular en células madre, la tasa de entrada de la célula de germen en profase meiótica y la tasa de progresión de la célula de germen a través de la profase meiótica? ¿Cómo cambia el ciclo celular durante el desarrollo larval? ¿Cómo afectan el ciclo celular, además de los cambios en el destino de la célula (como proliferación ectópica) principales interrupciones de vía señalización? Este sistema puede modificarse para estudiar lo que las células están haciendo en muchas condiciones diferentes.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos al centro común de e. coli MG1693; Wormbase; el centro de genética de Caenorhabditis financiado por los institutos de salud oficina de investigación infraestructura programas nacionales (P40OD010440) para las cepas; Zach Pincus Consejo estadístico; Aiping Feng de reactivos; Luke Schneider, Andrea Scharf, Sandeep Kumar y John Brenner para capacitación, asesoramiento, apoyo y discusión útil; y los laboratorios Kornfeld y Schedl por comentarios sobre este manuscrito. Este trabajo fue financiado en parte por los institutos nacionales de salud [AG02656106A1 R01 a KK, GM100756 R01 a TS] y una beca predoctoral de la Fundación Nacional de Ciencias [DGE-1143954 y DGE-1745038 a ZK]. Los institutos nacionales de salud ni la National Science Foundation tenía cualquier papel en el diseño del estudio, colección, análisis e interpretación de datos, ni por escrito el manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| E. coli MG1693 | Coli Genetic Stock Center | 6411 | grows fine in standard unsupplemented LB |
| E. coli OP50 | Caenorhabditis Genetics Center | OP50 | |
| Click-iT EdU Alexa Fluor 488 Imaging Kit | Thermo Fisher Scientific | C10337 | |
| 5-Ethynyl-2′-deoxyuridine | Sigma | 900584-50MG | or use EdU provided in kit |
| Glucose | Sigma | D9434-500G | D-(+)-Dextrose |
| Thiamine (Vitamin B1) | Sigma | T4625-5G | Reagent Grade |
| Thymidine | Sigma | T1895-1G | BioReagent |
| Magnesium sulfate heptahydrate | Sigma | M1880-1KG | MgSO4, Reagent Grade |
| Sodium Phosphate, dibasic, anhydrous | Fisher | BP332-500G | Na2HPO4 |
| Potassium Phosphate, monobasic | Sigma | P5379-500G | KH2PO4 |
| Ammonium Chloride | Sigma | A4514-500G | NH4Cl, Reagent Plus |
| Bacteriological Agar | US Biological | C13071058 | |
| Calcium Chloride dihydrate | Sigma | C3881-500G | CaCl |
| LB Broth (Miller) | Sigma | L3522-1KG | Used at 25g/L |
| Levamisole | Sigma | L9756-5G | 0.241g/10ml |
| Phosphate buffered saline | Calbiochem Omnipur | 6506 | homemade PBS works just as well |
| Tween-20 | Sigma | P1379-500ML | |
| 16% Paraformaldehyde, EM-grade ampules | Electron Microscopy Sciences | 15710 | 10ml ampules |
| 100% methanol | Thermo Fisher Scientific | A454-1L | Gold-label methanol is critical for proper morphology with certain antibodies |
| Goat Serum | Gibco | 16210-072 | Lot 1671330 |
| rabbit-anti-WAPL-1 | Novus biologicals | 49300002 | Lot G3048-179A02, used at 1:2000 |
| mouse-anti-pH3 clone 3H10 | Millipore | 05-806 | Lot#2680533, used at 1:500 |
| goat-anti-rabbit IgG-conjugated Alexa Fluor 594 | Invitrogen | A11012 | Lot 1256147, used at 1:400 |
| goat-anti-mouse IgG-conjugated Alexa Fluor 647 | Invitrogen | A21236 | Lot 1511347, used at 1:400 |
| Vectashield antifade mounting medium containing 4',6-Diamidino-2-Phenylindole Dihydrochloride (DAPI) | Vector Laboratories | H-1200 | mounting medium without DAPI can be used instead, following a separate DAPI incubation |
| nail polish | Wet n Wild | DTC450B | any clear nail polish should work |
| S-medium | various | see wormbook.org for protocol | |
| M9 buffer | various | see wormbook.org for protocol | |
| M9 agar | various | same recipe as M9 buffer, but add 1.7% agar | |
| Nematode Growth Medium | various | see wormbook.org for protocol | |
| dissecting watch glass | Carolina Biological | 42300 | |
| Parafilm laboratory film | Pechiney Plastic Packaging | PM-996 | 4 inch wide laboratory film |
| petri dishes | 60 mm diameter | ||
| Long glass Pasteur pipettes | |||
| 1ml centrifuge tubes | MidSci Avant | 2926 | |
| Tips | |||
| Serological pipettes | |||
| 500 mL Erlenmyer flask | |||
| Aluminum foil | |||
| 25G 5/8” needles | BD PrecisionGlide | 305122 | |
| 5ml glass centrifuge tube | Pyrex | ||
| Borosilicate glass tubes 1ml | |||
| glass slides | |||
| no 1 coverslips 22 x 40 mm | no 1.5 may work, also | ||
| 37 °C Shaker incubator | |||
| Tabletop Centrifuge | |||
| Clinical Centrifuge | IEC | 428 | with 6 swinging bucket rotor |
| Mini Centrifuge | |||
| 20 °C incubator | |||
| 4 °C refrigerator | |||
| -20 °C freezer | |||
| Observer Z1 microscope | Zeiss | ||
| Plan Apo 63X 1.4 oil-immersion objective lens | Zeiss | ||
| Ultraview Vox spinning disc confocal system | PerkinElmer | Nikon spinning disc confocal system works very well, also, as described here: http://wucci.wustl.edu/Facilities/Light-Microscopy |
Referencias
- Fox, P. M., Vought, V. E., Hanazawa, M., Lee, M. -H., Maine, E. M., Schedl, T. Cyclin E and CDK-2 regulate proliferative cell fate and cell cycle progression in the C. elegans germline. Development. 138 (11), Cambridge, England. 2223-2234 (2011).
- Crittenden, S. L., Leonhard, K. A., Byrd, D. T., Kimble, J. Cellular analyses of the mitotic region in the Caenorhabditis elegans adult germ line. Molecular biology of the cell. 17 (7), 3051-3061 (2006).
- Seidel, H. S., Kimble, J. Cell-cycle quiescence maintains Caenorhabditis elegans germline stem cells independent of GLP-1/Notch. eLife. 4, (2015).
- Fox, P. M., Schedl, T. Analysis of Germline Stem Cell Differentiation Following Loss of GLP-1 Notch Activity in Caenorhabditis elegans. Genetics. 201 (9), 167-184 (2015).
- Kocsisova, Z., Kornfeld, K., Schedl, T. Cell cycle accumulation of the proliferating cell nuclear antigen PCN-1 transitions from continuous in the adult germline to intermittent in the early embryo of C. elegans. BMC Developmental Biology. 18 (1), (2018).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proceedings of the National Academy of Sciences. 105 (7), 2415-2420 (2008).
- vanden Heuvel, S., Kipreos, E. T. C. elegans Cell Cycle Analysis. Methods in Cell Biology. , 265-294 (2012).
- ThermoFisher. Click-iT EdU Imaging Kits. , https://assets.thermofisher.com/TFS-Assets/LSG/manuals/mp10338.pdf (2011).
- Pazdernik, N., Schedl, T. Germ Cell Development in C. elegans. , Springer. 1-16 (2013).
- Hirsh, D., Oppenheim, D., Klass, M. Development of the reproductive system of Caenorhabditis elegans. Developmental Biology. 49 (1), 200-219 (1976).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Hansen, D., Schedl, T. Stem cell proliferation versus meiotic fate decision in Caenorhabditis elegans. Advances in Experimental Medicine and Biology. 757, 71-99 (2013).
- Jaramillo-Lambert, A., Ellefson, M., Villeneuve, A. M., Engebrecht, J. Differential timing of S phases, X chromosome replication, and meiotic prophase in the C. elegans germ line. Developmental Biology. 308 (1), 206-221 (2007).
- Agarwal, I., Farnow, C., et al. HOP-1 presenilin deficiency causes a late-onset notch signaling phenotype that affects adult germline function in Caenorhabditis elegans. Genetics. 208 (2), 745-762 (2018).
- Gumienny, T. L., Lambie, E., Hartwieg, E., Horvitz, H. R., Hengartner, M. O. Genetic control of programmed cell death in the Caenorhabditis elegans hermaphrodite germline. Development. 126 (5), Cambridge, England. 1011-1022 (1999).
- Hendzel, M. J., Wei, Y., et al. Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106 (6), 348-360 (1997).
- Zetka, M. C., Kawasaki, I., Strome, S., Mü Ller, F. Synapsis and chiasma formation in Caenorhabditis elegans require HIM-3, a meiotic chromosome core component that functions in chromosome segregation. Genes & development. 13 (17), https://www.ncbi.nlm.nih.gov/pmc/articles/PMC317003/pdf/x8.pdf 2258-2270 (1999).
- Hansen, D., Hubbard, E. J. A., Schedl, T. Multi-pathway control of the proliferation versus meiotic development decision in the Caenorhabditis elegans germline. Developmental Biology. 268 (2), 342-357 (2004).
- Crawley, O., Barroso, C., et al. Cohesin-interacting protein WAPL-1 regulates meiotic chromosome structure and cohesion by antagonizing specific cohesin complexes. eLife. 5 (2), 1-26 (2016).
- Zhao, H., Halicka, H. D., et al. DNA damage signaling, impairment of cell cycle progression, and apoptosis triggered by 5-ethynyl-2′-deoxyuridine incorporated into DNA. Cytometry Part A. 83 (11), 979-988 (2013).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Gervaise, A. L., Arur, S. Spatial and Temporal Analysis of Active ERK. in the C. elegans Germline. Journal of Visualized Experiments. 117 (117), e54901-e54901 (2016).
- Vos, K. De Cell Counter Plugin. , https://imagej.nih.gov/ij/plugins/cell-counter.html (2015).
- Schindelin, J., Arganda-Carreras, I., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Rasband, W. ImageJ. , http://imagej.nih.gov/ij/> (2016).
- Michaelson, D., Korta, D. Z., Capua, Y., Hubbard, E. J. A. Insulin signaling promotes germline proliferation in C. elegans. Development. 137 (4), Cambridge, England. 671-680 (2010).
- Qin, Z., Jane, E., Hubbard, A., Hubbard, E. J. A. Non-autonomous DAF-16/FOXO activity antagonizes age-related loss of C. elegans germline stem/progenitor cells. Nature communications. 6 (5), 7107(2015).
- Luo, S., Kleemann, G. A., Ashraf, J. M., Shaw, W. M., Murphy, C. T. TGFB and Insulin Signaling Regulate Reproductive Aging via Oocyte and Germline Quality Maintenance. Cell. 143 (2), 299-312 (2010).
- Narbonne, P., Maddox, P. S., Labbe, J. -C. daf-18/PTEN locally antagonizes insulin signalling to couple germline stem cell proliferation to oocyte needs in C. elegans. Development. , 4230-4241 (2015).
- Cinquin, A., Chiang, M., et al. Intermittent Stem Cell Cycling Balances Self-Renewal and. Senescence of the C. elegans Germ Line. PLoS Genetics. 12 (4), 1005985(2016).
- Invitrogen EdU (5-ethynyl-2’-deoxyuridine). , https://assets.thermofisher.com/TFS-Assets/LSG/manuals/mp10044.pdf 1-7 (2010).
- Tuttle, A. H., Rankin, M. M., et al. Immunofluorescent Detection of Two Thymidine Analogues (CldU and IdU) in Primary Tissue. Journal of Visualized Experiments. 46 (46), e2166-e2166 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados