Method Article
Analisi del ciclo cellulare nella linea germinale di c. elegans con la timidina analogico EdU
In questo articolo
Riepilogo
È descritto un metodo basato su formazione immagine che può essere utilizzato per identificare la fase S e analizzare le dinamiche del ciclo cellulare in c. elegans ermafrodita germinale utilizzando la timidina EdU analogico. Questo metodo non richiede nessun transgeni ed è compatibile con la macchiatura immunofluorescente.
Abstract
Analisi del ciclo cellulare negli eucarioti utilizza frequentemente la morfologia del cromosoma, espressione e/o localizzazione di prodotti genici necessari per le varie fasi del ciclo cellulare, o l'incorporazione di analoghi nucleosidici. Durante la fase S, polimerasi del DNA incorporare gli analoghi della timidina come EdU o BrdU nel DNA cromosomico, marcatura delle cellule per l'analisi. Per c. elegans, il nucleoside analogico EdU durante regolare cultura è alimentata con i vermi ed è compatibile con tecniche di immunofluorescenza. La linea germinale di c. elegans è un sistema potente modello per gli studi di segnalazione vie, cellule staminali, meiosi e ciclo cellulare perché è trasparente, geneticamente facile e Profase Meiotica e differenziazione cellulare/gametogenesi si verificano in un lineare Assemblea-come la moda. Queste caratteristiche rendono EdU un ottimo strumento per studiare aspetti dinamici delle cellule mitoticamente ciclismo e sviluppo di germline. Questo protocollo viene descritto come correttamente preparare EdU batteri, nutrirli wild-type c. elegans ermafroditi, sezionare la gonade ermafrodita, macchia per EdU incorporazione nel DNA, macchia con gli anticorpi per rilevare vari ciclo cellulare e gli indicatori dello sviluppo, la gonade di immagine e analizzare i risultati. Il protocollo descrive le variazioni nel metodo e nell'analisi per la misurazione della fase S indice, M-fase indice, G2 durata, durata del ciclo cellulare, tasso di entrata meiotica e tasso di progressione della Profase Meiotica. Questo metodo può essere adattato per studiare il ciclo cellulare o la storia di cella in altri tessuti, fasi, ambiti di provenienza genetici e condizioni fisiologiche.
Introduzione
Nello sviluppo animale, centinaia, migliaia, milioni, miliardi o anche migliaia di miliardi di divisioni cellulari è necessari per formare l'organismo adulto. Il ciclo cellulare, il set di eventi cellulari composto di G1 (gap), S (sintesi), G2 (gap), e M (mitosi) definire la serie di eventi che vengono eseguiti ogni divisione cellulare. Il ciclo cellulare è dinamico e meglio apprezzato in tempo reale, che può essere tecnicamente difficile. Le tecniche presentate in questo protocollo permettono di effettuare le misurazioni delle fasi e la tempistica del ciclo cellulare da immagini fisse.
Etichettatura con analoghi nucleosidici come 5-Etinil-2'-deoxyuridine (EdU) o 5-bromo-2'-deoxyuridine (BrdU) è il gold standard per identificare la fase S negli studi della dinamica del ciclo cellulare nell'adulto Caenorhabditis elegans (c. elegans) germline ermafroditi1,2,3,4,5. EdU sia BrdU utilizzabile in quasi qualsiasi background genetico, come che non si basano su qualsiasi costrutto genetico. Visualizzazione di BrdU richiede duro trattamento chimico per esporre l'antigene per l'anticorpo anti-BrdU colorazione, che spesso è incompatibile con la valutazione di altri markers cellulari visualizzati dalla co-macchiatura con altri anticorpi. Al contrario, visualizzazione EdU si verifica dalla chimica clicca in condizioni blande e quindi è compatibile con l'anticorpo co-macchiatura6,7.
La specificità dell'etichetta è chiara, dal momento che i nuclei solo incorporano gli analoghi (5-Etinil-2'-deoxyuridine) della timidina in DNA durante la fase S. Visualizzazione avviene nel tessuto fisso. L'etichetta di EdU è invisibile da solo fino a un colorante contenenti azide o fluorophore reagisce covalentemente con alchini in EdU di rame-catalizzata clic chimica8. EdU etichettatura può fornire informazioni immediate su cui i nuclei sono in fase S, utilizzando un breve impulso di etichettatura. EdU possono anche fornire informazioni dinamiche, utilizzando pulse-chase o etichettatura continuo; ad esempio, in un esperimento di pulse-chase, l'etichetta è diluita a ogni divisione cellulare o propagate come nondividing cellule progresso attraverso lo sviluppo.
C. elegans ermafrodita germinale è un sistema potente modello per gli studi del ciclo cellulare, cellule staminali, meiosi e vie di segnalazione. La linea germinale adulta è una linea di assemblaggio polarizzata con cellule staminali presenti all'estremità distale, seguita da voce e progressione attraverso Profase Meiotica, coordinato con le fasi della gametogenesi più prossimalmente (Figura 1). L'estremità prossimale, ovociti maturo, sono ovulati e fertilizzato e cominciano l'embriogenesi nel utero9,10,11. La regione lungo ~ 20 cella-diametro accanto alla cella di punta distale, che include il gambo di germline mitotically ciclismo, cellule progenitrici e le cellule di S-fase meiotiche ma non le cellule in Profase Meiotica, viene chiamata il progenitore zona2,4 , 9 , 12. le membrane delle cellule forniscono incompleta separazione tra i nuclei la linea germinale distale, ma la zona cellule progenitrici subiscono mitotica cellulare ciclismo in gran parte in modo indipendente. La durata mediana ciclo cellulare mitotica delle cellule di zona progenitrici di germline in giovane adulti ermafroditi è ~6.5 h; Fase G1 è breve o assente, e quiescenza non è osservato1,2,13. Differenziazione delle cellule staminali germinali si verifica attraverso la differenziazione essenzialmente diretta e quindi manca di transito-amplificando divisioni4. Durante la differenziazione nel pachitene, circa 4 su 5 nuclei non si formano gli ovociti ma invece andare incontro ad apoptosi, in qualità di infermiera cellule donando loro contenuto citoplasmatico di sviluppo dell'ovocita12,14 , 15.
Oltre alle etichette cellule in fase S con analoghi nucleosidici, si possono identificare le cellule in mitosi e meiosi usando la macchiatura dell'anticorpo. Nuclei in mitosi sono immunoreactive all'anti-fosfo-istone H3 (Ser10) anticorpo (chiamato pH3)7,16. Nuclei in meiosi sono immunoreactive all'anticorpo anti-lui-3 (una proteina di asse meiotic del cromosoma)17. I nuclei nella zona del progenitore possono essere identificati dall'assenza di HIM-3, la presenza di adrenocorticale REC-818o la presenza di WAPL-119. WAPL-1 intensità è massima nella gonade somatica, alta nella zona del progenitore e basso durante i primi meiotica prophases19. Diverse misurazioni del ciclo cellulare sono possibili con poche variazioni nel protocollo: I) identificare i nuclei in fase S e misurare l'indice di S-fase; II) identificare i nuclei in fase M e misurare l'indice di fase M; III) determinano se i nuclei erano nella S-fase mitotica o meiotica; IV) misurare la durata di G2; V) misura la duartion delle fasi G1 + G2 + M; VI) misura il tasso di entrata meiotica; VII) stimare il tasso di progressione meiotica.
Si possono fare misurazioni multiple del ciclo cellulare da solo alcuni tipi di esperimenti di laboratorio bagnato. Il protocollo sottostante descrive un 30 min impulso etichettatura alimentandosi di c. elegans adulti ermafroditi con EdU etichettato batteri e cellule co-etichettatura di M-fase macchiando con progenitore e degli anticorpi anti-pH3 zona cellule macchiando con anticorpo anti-WAPL-1. Soltanto i cambiamenti nella durata della EdU feed (punto 2.5), tipo di anticorpi impiegati (punto 5), e sono necessarie per le misure ulteriori analisi (punto 8.3).
Protocollo
1. preparazione di EdU-identificati batteri
-
Crescere una coltura starter di MG1693. Escherichia coli MG1693 (e. coli) trasporta una mutazione in thyA.
- Striscia fuori MG1693 di Escherichia coli da uno stock di glicerolo congelato su un agar di brodo (LB) di 120 mm Lisogenesi di Petri. Coltura a 37 ° C durante la notte.
- Inoculare da due singoli e. coli MG1693 colonie in due duplicare 4 mL provette di coltura liquida lb a 37 ° C per ~ 16 h.
Nota: La MG1693 cresce bene in LB senza completare con timina o della timidina.
-
Crescere e. coli MG1693 in media minimi completati con EdU.
- Autoclave una beuta da 500 mL.
- Usare una tecnica sterile per aggiungere 5 mL di glucosio 20%, 50 μL di 10 mg/mL di tiamina, 120 μL di timidina 5 mM, 100 μL di 1m MgSO4, 200 μL di 10 mM EdU, 100 mL di tampone di M9 e 4 mL di appena cresciuti durante la notte MG1693 cultura.
Nota: La concentrazione finale di EdU è 20 μM in questa cultura1,7. Questa concentrazione conduce ad arresto del ciclo cellulare e danno del DNA, se applicato direttamente a cellule di mammifero in coltura20. Tuttavia, solo una frazione di questo EdU è incorporata in e. coli e quindi disponibile al c. elegans. C'è nessuna prova di arresto del ciclo cellulare e nessun cambiamento nella dimensione della zona di progenitore o Indice M-fase dopo il trattamento di EdU di giovane adulti ermafroditi. - Coltura durante la notte, ma non più di 24 ore a 37 ° C con agitazione a 200 giri/min.
-
Concentrarsi EdU-etichettati e. coli e applicare a M9 agar capsule di Petri.
- Pre-raffreddare una centrifuga da tavolo a 4 ° C.
- Usare una tecnica sterile per trasferire la cultura in 2 – 4 provette coniche da 50 mL sterile.
- Centrifugare le culture a 3.000 x g a 4 ° C per 30 min a pellet la EdU-etichettati e. coli.
Nota: Smaltire il supernatante contenente EdU secondo linee guida locali e istituzionali. - Risospendere il pellet con 4 mL di fresco M9. Utilizzare una punta di pipetta sterile da 1 mL o una pipetta sierologica sterile 5 mL. Risospendere il pellet potrebbe richiedere alcuni minuti.
- Utilizzare la stessa pipetta per applicare e diffondere ~ 8 gocce di sedimento EdU-etichettati e. coli MG1693 al centro delle piastre di Petri di temperatura da 60 mm M9 agar. Un unico lotto produce piatti ~ 16.
- Consentire i piatti asciugare per diverse ore o una notte a temperatura ambiente, quindi sigillare ogni piatto con una striscia di pellicola di laboratorio. Piatti possono essere memorizzati a 15 ° C ~ 2 settimane e a 4 ° C per ~ 2 mesi. Utilizzare la stessa serie di EdU piatti per ogni set di esperimenti.
2. alimentazione EdU al c. elegans
- Sincronizzare popolazione temporizzato uovo laici, alcalini ipoclorito trattamento seguita da Nidoran ♀ medio-S con il colesterolo, o scegliendo la fase appropriata21. Crescere gli animali alla fase desiderata (qui, 24h post-L4), il Medium di crescita Nematode seminato con Escherichia coli OP50 a 20 ° C.
Nota: Preparare 50 – 100 animali per esperimento, come alcuni non possono sezionare bene, e altri saranno persi nel processo di lavaggio e trasferimento. - Consentire i piatti EdU riscaldare a 20 ° C (o la temperatura necessaria per l'esperimento).
- Lavare gli animali dai piatti NGM utilizzando M9 o tampone fosfato salino (PBS) in una provetta da 1,5 mL. Consentire agli animali di risolvere brevemente per gravità o un breve giro in una microcentrifuga.
- Eliminare il surnatante e lavare gli animali 1 – 2 volte con ~ 1 mL di PBS o di M9. Eliminare il surnatante.
- Utilizzare un pipetta Pasteur di vetro per trasferire gli animali in una piccola goccia di M9 o PBS al centro del prato EdU. Attendere qualche minuto per il liquido essere assorbito, quindi incubare a 20 ° C per 30 min (per la misura diretta di S-fase) o più (per misurare la storia della fase S), come necessario.
Nota: EdU segnale è rilevabile nei nuclei di germline dopo appena 15 min di EdU alimentazione1. - Lavare i vermi il piatto di EdU con ~ 2 mL di M9 o PBS in un vetro piatto di dissezione.
3. dissezione e la fissazione di Germline di c. elegans
Nota: Questo protocollo per la dissezione, la fissazione e la macchiatura dell'anticorpo della linea germinale di c. elegans ermafrodita è quasi identico a quello pubblicato da Gervaise e Arur (2016)22, ad eccezione del fatto che i tubi di vetro da 1 mL possono essere centrifugati per accelerare lavatrice passi e un bicchiere di tratte-out pipetta Pasteur può essere utilizzato per rimuovere il liquido da tubi di vetro da 1 mL in modo più efficace.
-
Lavare e sezionare germlines di c. elegans .
- Consentire agli animali di depositarsi sul fondo del piatto per dissezione, ricciolo a raccogliere gli animali nel centro e utilizzare una pipetta Pasteur lunga per rimuovere PBS. Lavare 1 – 2 volte con ~ 2 mL di PBS.
- Aggiungere 2 mL di PBS e 4 μL di levamisole 100 mM per immobilizzare gli animali. Roteare il piatto per raccogliere gli animali al centro del piatto.
Nota: Immobilizzazione può durare da pochi secondi e pochi minuti. L'immobilizzazione completa non è necessaria per la dissezione di successo. Alcune persone hanno successo migliore quando in modo incompleto di dissezione immobilizzato animali. - Sezionare gli animali con una coppia di aghi 25 5/8" da taglio alla testa (circa tra le due lampadine pharyngeal) o la coda. Fare attenzione a non per tagliare il ciclo della linea germinale. Intestino e germinale dovrebbero "saltar fuori" della cavità di corpo dovuta alla pressione interna, ma rimangono attaccati. Questo protocollo è simile a precedentemente pubblicati Gervaise e Arur (2016)22.
Nota: Tenere il tempo di dissezione a ~ 5 min, certamente non più di 15 minuti più lunghi tempi di dissezione possono provocare la perdita del segnale di macchiatura dell'anticorpo (Sudhir Nayak, comunicazione personale) e fame in PBS può influenzare la fisiologia degli animali. Imparare a sezionare rapidamente e con precisione potrebbe richiedere un po' di pratica. - Se necessario, di turbinio per raccogliere gli animali sezionati nel centro e utilizzare una pipetta Pasteur lunga per rimuovere quanto più PBS come possibile.
-
Difficoltà e disidratare i tessuti
- Aggiungere 2 mL di 3% paraformaldeide (PFA) in soluzione PBS. Coprire il piatto senza bloccare con parafilm e negozio su una panchina o in un cassetto per 10 min.
Nota: Scongelare PFA soluzione in un a 37 ° C bagno d'acqua e poi raffreddare a temperatura ambiente prima del germlines di dissezione.
Attenzione: La soluzione PFA è moderatamente tossico e un probabile cancerogeno e teratogeno. Vapori che emettono da soluzioni di paraformaldeide sono infiammabili. Indossare guanti in nitrile. Diluire PFA dal 16% al 3% in una cappa chimica. Quando si lavora di fuori della cappa, mantenere tutti i contenitori coperti. - Trasferire le gonadi attentamente una provetta da centrifuga vetro pulito 5 mL.
- Aggiungere circa 3 mL di PBSTw (PBS con 0,1% Tween-20) per il piatto che conteneva le gonadi di aiutare a recuperare i restanti gonadi e diluire la soluzione PFA.
- Centrifugare in una centrifuga clinica a 870 x g per un minimo di ~ 1 giovane o animali più piccoli richiedono tempi più lunghi di rotazione rispetto agli animali di età superiore o più grandi.
- Utilizzando una pipetta di vetro lunghe, trasferire il surnatante becher di materiale indesiderato per l'eventuale eliminazione in bottiglia materiale indesiderato nella cappa chimica.
- Aggiungere 2 mL di metanolo prima scelta pre-raffreddata a-20 ° C. Coprire la provetta da centrifuga strettamente con parafilm.
Nota: Uso del metanolo fresco alta qualità "gold label" è essenziale per morfologia corretta con alcuni anticorpi.
Attenzione: Il metanolo è un liquido altamente infiammabile e vapore, che è tossico se ingerito, inalato o autorizzati a contatto pelle. Indossare adeguati dispositivi di protezione personali e guanti. Utilizzare un congelatore adatto per piccoli volumi di materiali infiammabili. - Conservare in congelatore a-20 ° C per 1 h, anche durante la notte o anche parecchi mesi.
Nota: Il protocollo può essere messo in pausa qui.
- Aggiungere 2 mL di 3% paraformaldeide (PFA) in soluzione PBS. Coprire il piatto senza bloccare con parafilm e negozio su una panchina o in un cassetto per 10 min.
4. reidratare Germlines
- Riempire la provetta di vetro verso l'alto con PBSTw per diluire il metanolo e reidratare le gonadi. Rotazione verso il basso in una centrifuga clinica a 870 x g per ~ 1 min.
- Utilizzando un pipetta Pasteur di vetro lunghe, trasferire il surnatante becher di materiale indesiderato per l'eventuale eliminazione in bottiglia materiale indesiderato nella cappa chimica.
- Lavare le gonadi 3 volte usando ~ 5 mL di PBSTw, filatura giù in una centrifuga clinica a 870 x g per ~ 1 min ogni volta. Eliminare il surnatante.
- Sciacquare un tubo di vetro borosilicato piccolo 1 mL e un vetro lungo pipetta Pasteur con PBSTw.
- Aggiungere ~ 700 µ l di PBSTw e utilizzare il pipetta Pasteur di vetro lunghe per trasferire le gonadi al piccolo tubo. Utilizzare ulteriori poche gocce di PBSTw per garantire che tutte le gonadi sono trasferiti.
- Rotazione verso il basso in una centrifuga clinica a 870 x g per un minimo di ~ 1 utilizzando un pipetta Pasteur di vetro lunghe tratte-out, rimuovere quanto più liquido possibile senza disturbare le gonadi. Lasciare non più di 50 μL.
Nota: Se la rilevazione dell'anticorpo non è necessario, andare al passaggio 6.
5. rilevare anticorpi-
- Diluire gli anticorpi primari in siero di 30% in PBS. Nell'esempio, anticorpo anti-WAPL-1 è 1:2,000 diluito e dell'anticorpo anti-pH3 è diluito 1: 500. Centrifugare il siero appena scongelato per 10 min in un microfuge a 20.000 x g a 4 ° C per rimuovere il particolato. Utilizzare il surnatante, che può essere conservato a 4 ° C per diversi giorni. Utilizzare il siero appropriato per abbinare l'organismo ospite di anticorpi secondari (siero di capra viene utilizzato qui). Un passo di blocco opzionale può essere aggiunto prima dell'aggiunta di anticorpi primari.
- Applicare 100 µ l di anticorpo primario diluito per ogni piccolo tubo di vetro. Incubare a temperatura ambiente per 4 h.
Nota: Tempi di incubazione variano dall'anticorpo. Per alcuni anticorpi, 2 h a temperatura ambiente è sufficiente. Le incubazioni più lungo (ad es., durante la notte) sono possibili, ma può aumentare la priorità bassa. - Riempire le provette per top con PBSTw e rotazione verso il basso in una centrifuga clinica a 870 x g per ~ 1 min.
- Lavare le gonadi 3 volte utilizzando ~ 1 mL di PBSTw. Incubare per ~ 5 min per lavaggio per consentire in eccesso anticorpo primario di diffondere nel lavaggio. Utilizzando un tratte-out lungo vetro pipetta Pasteur, rimuovere quanto più liquido possibile senza disturbare le gonadi. Lasciare non più di 50 μL.
- Diluire gli anticorpi secondari in siero di capra del 30% in PBS. Nell'esempio, capra-anti-coniglio-594 e capra-anti-mouse-647 sono ciascuno diluiti a 1: 400.
Nota: Selezionare anticorpi secondari con attenzione per assicurarci che i coloranti sono distinte dal colorante nel kit di EdU. Nell'esempio, il kit di EdU conteneva una tintura di eccitazione 488 nm. - Applicare 100 µ l di anticorpo secondario diluito per ogni piccolo tubo di vetro. Incubare al buio a temperatura ambiente 2 h.
Nota: Tempi di incubazione variano dall'anticorpo. Per alcuni anticorpi secondari, è sufficiente 1 ora a temperatura ambiente. Le incubazioni più lungo (ad es., durante la notte) sono possibili, ma può aumentare la priorità bassa. - Lavare le gonadi 3 volte utilizzando ~ 1 mL di PBSTw. Incubare per ~ 5 min per lavaggio per consentire anticorpo secondario in eccesso per diffondersi nell'autolavaggio. Utilizzando un tratte-out lungo vetro pipetta Pasteur, rimuovere quanto più liquido possibile senza disturbare le gonadi. Lasciare non più di 50 μL.
Nota: Gonadi possono essere memorizzati in 100 μL di PBS dopo questo passaggio, se necessario, anche se questo può ridurre il segnale. Rimuovere PBS prima di procedere.
6. eseguire la reazione di EdU Click per rilevare EdU
Nota: Eseguendo l'EdU clic reazione prima l'anticorpo che macchia (eseguire il passaggio 6 fino al passo 5) passaggi è possibile, a seconda gli anticorpi utilizzati7. Tuttavia, i reagenti di clic potrebbero interferire con determinati antigeni (ad es., REC-8 anticorpo è sensibile alla fissazione e permeabilizzazione). L'ordine presentato qui rese luminosa anticorpo che macchia con il REC-8, WAPL-1, HIM-3, pH3, bandiera e CYE-1 gli anticorpi usati, tra gli altri.
-
Preparare il clic EdU cocktail8 fresco aggiungendo quanto segue in una provetta pulita 1,5 mL. L'ordine delle aggiunte è importante. Proteggere dalla luce e conservare tutti i reagenti sul ghiaccio. Questa ricetta produce abbastanza per un campione (100 μL); moltiplicare la ricetta come necessario.
- Aggiungere 2 mL di acqua ultrapura per il buffer di additivo. Questo rende 10x additivo di buffer, che devono essere diluiti a 1 x immediatamente prima dell'uso.
- Aggiungere 8,5 μL di tampone di 10 x 76,5 μL di acqua ultrapura. Mescolare bene.
- Aggiungere 4 μL di 100mm di CuSO4 (possono essere etichettati come componente E). Mescolare bene.
- Aggiungere 0.25 μL della tintura 488 nm Azide. Devono essere scongelato a temperatura ambiente, come il solvente, il solfossido dimetilico, è solido a 4 ° C. Mescolare bene e proteggere dalla luce.
- Mix 9 μL di acqua ultrapura con 1 μL del buffer additivo nel tappo del tubo. Dispensare dal tappo per aggiungere al cocktail rimanente e mescolare bene pipettando su e giù.
-
Eseguire la reazione di clic di EdU.
- Aggiungere ~ 100 μL di clic EdU cocktail per le gonadi nel piccolo tubo. Coprire con la pellicola di laboratorio ed incubare per 30-60 min a temperatura ambiente.
- Lavare una volta con 100 μL di tampone di reazione risciacquo.
- Lavare le gonadi 4 volte utilizzando ~ 1 mL di PBSTw. Incubare per ~ 15 min per lavaggio per consentire in eccesso EdU cocktail componenti si diffondono nella lavata. Utilizzando un tratte-out lungo vetro pipetta Pasteur, rimuovere quanto più liquido possibile senza disturbare le gonadi. Lasciare non più di 50 μL.
7. macchia DNA e preparare i vetrini
- Aggiungere 1 goccia (~ 25 μL) di mezzo di montaggio antifade con 4', 6-diamidino-2-phenylindole (DAPI, utilizzato per visualizzare il DNA) per le gonadi. Attendere qualche minuto in modo che possa depositarsi e mescolare con le gonadi.
Nota: In alternativa, può essere applicata una diluizione di 1:1,000 di DAPI (da uno stock di 0,1 mg/mL) in PBS per 5 min, seguita da 20 μL di 1,4-diazabiciclo [2.2.2] ottano (DABCO) in 90% glicerolo, o un'altra soluzione di montaggio antifade. - Preparare un grande rilievo di agarosio 2,5% su un vetrino per microscopio standard.
- Uso un nuovo pulito privo di polvere di vetro lunghe pipetta Pasteur di trasferire le gonadi per il pad di agarosio. Tenere tutti i liquidi e le gonadi in fondo stretto della pipetta per minimizzare la perdita delle gonadi.
Nota: Gonadi attaccato al piccolo tubo di vetro o in vetro lunga pipetta Pasteur può essere "salvato" da risciacquo con PBSTw, raccogliendo il liquido in un piatto di dissezione e raccogliendo singoli animali sulla diapositiva con un ciglio. - Utilizzare un ciglio (o un ciclo dei capelli sottili) incollato su uno stuzzicadenti per distribuire le gonadi sul riquadro dell'agarosi ed eliminare le particelle di polvere.
- Coprire con un vetrino coprioggetto rettangolare in vetro. Inferiore lentamente da un lato per evitare bolle d'aria. Utilizzare un fazzoletto di carta per rimuovere la soluzione in eccesso, impedendo così il coprioggetto di muoversi liberamente.
Nota: Scegliere le lamelle che corrispondono al microscopio che verrà utilizzato. #1 e #1.5 coprioggetti funzionano bene. - Lasciare i vetrini vivere e trasferirsi a secco leggermente durante la notte a temperatura ambiente o a 4 ° C. Questo aiuta ad per appiattire leggermente le gonadi. I vetrini devono essere conservati a 4 ° C.
- Optional: Sigillare i bordi della diapositiva con smalto, o un'altra diapositiva sealer. Sigillatura degli angoli in primo luogo, quindi i lati, impedisce di spostando il vetrino coprioggetti.
8. analisi e Imaging confocale
- Immagine la gonade distale con un microscopio a fluorescenza confocale filatura disco è dotato di una sorgente di luce ad alta energia, obiettivi plan-apocromatici e una fotocamera di microscopio ad alta efficienza. Catturare le immagini con 1 μm o più stretto spaziatura tra z-stack. Prendere nota del tempo di esposizione, la sensibilità e la potenza del laser per tutti i canali.
- Utilizzare la seguente: 405 nm linea eccitazione laser con un filtro di emissione 485 nm (W60) per DAPI, 488 nm laser linea eccitazione con un filtro di emissione nm (W55) 527 per EdU, 561 nm laser linea di eccitazione con un filtro di emissione nm (L70) 615 per WAPL-1 e un 640 nm laser linea excita zione con un filtro di emissione 705 nm (W90) per pH3.
Nota: L'intensità del segnale e priorità bassa intensità varia. Allo stesso modo, i tempi di esposizione richiesta varierà, possibilmente fino a 10 volte.
- Utilizzare la seguente: 405 nm linea eccitazione laser con un filtro di emissione 485 nm (W60) per DAPI, 488 nm laser linea eccitazione con un filtro di emissione nm (W55) 527 per EdU, 561 nm laser linea di eccitazione con un filtro di emissione nm (L70) 615 per WAPL-1 e un 640 nm laser linea excita zione con un filtro di emissione 705 nm (W90) per pH3.
- Utilizzare il plug-in contatore di cellule23 in Fiji24,25 per contare manualmente ogni nucleo. Etichettare ogni nucleo individuale secondo la presenza e l'assenza di pH3, EdU e WAPL-1. Utilizzare le classi dei nuclei descritti nella tabella 1 e Figura 2, come questi faciliterà tutti i calcoli descritti di seguito.
Nota: Sperimentalisti qualificati possono contare con precisione tutti i nuclei in un'immagine 3D senza duplicazioni o manca qualsiasi nuclei. Alternativamente, uno può contare ogni nucleo in ogni piano z, e i segni di cellule R script3 può essere usato per rimuovere i nuclei si moltiplicano-contato. - Calcolare i numeri di cellulare e frequenze comprese tra i conteggi sopra a seconda del tipo di ciclo cellulare misura necessaria. I tipi di nuclei sono definiti nella tabella 1 e Figura 2. I calcoli sono riassunti nella tabella 2.
- (Variazione mi) Per identificare i nuclei in fase S, nutrire gli animali EdU per 30 min. Qualsiasi visualizzazione etichetta EdU i nuclei sono i nuclei di fase S. Per calcolare, prendere i nuclei somma di A e C, Vedi tabella 1 e Figura 2.
Nota: In un 30 min EdU impulso nel selvaggio-tipo adulti ermafroditi, EdU tutti etichettati i nuclei sono co-etichettati con progenitrici zona marcatori1. - Per misurare la zona di progenitrici, macchia con un indicatore di zona del progenitore come anticorpo REC-8 o WAPL-1. La zona di progenitrici è definita qui come tutti adrenocorticale REC-818 o nuclei immunoreactive germline WAPL-1. Per calcolare, sommare tutti i nuclei immunoreactive di WAPL-1 (A + B + C + D, Vedi tabella 1 e Figura 2).
Nota: WAPL-1 etichette anche il DTC e nuclei di gonade somatico che non devono essere conteggiati. I nuclei somatici sono facili da identificare con segnale estremamente intenso WAPL-1, posizione leggermente fuori la linea germinale e una morfologia "uovo fritto" dei nuclei. - Per misurare l'indice di fase S, eseguire un 30 min EdU esperimento e co-etichetta con anticorpo REC-8 o WAPL-1. L'indice di S-fase è definita come la proporzione della zona progenitore che è in fase S. Per calcolare, contare tutti i nuclei di fase S e poi dividere per il numero totale di nuclei di cellule progenitrici zona (A + C / A + B + C + D, Vedi tabella 1 e Figura 2).
- (Variazione II) Per identificare i nuclei in fase M, macchia con l'anticorpo pH3. Qualsiasi nuclei immunoreactive di pH3 sono nuclei M-fase. Questo funziona indipendentemente dalla durata di EdU feed. Per calcolare, prendere la somma di A e nuclei di B, Vedi tabella 1 e Figura 2.
Nota: WAPL-1 etichette anche il DTC e nuclei di gonade somatico che non devono essere conteggiati. I nuclei somatici sono facili da identificare con segnale estremamente intenso WAPL-1, posizione leggermente fuori la linea germinale e una morfologia "uovo fritto" dei nuclei. - Per misurare l'indice di fase M, co-etichetta con pH3 e REC-8 o gli anticorpi WAPL-1. L'indice di M-fase è definita come la proporzione della zona progenitore che è in fase di M. Per calcolare, contare tutti i nuclei di fase M e poi dividere per il numero totale di nuclei di cellule progenitrici zona (A + B / A + B + C + D, Vedi tabella 1 e Figura 2).
- (Variazione III) I nuclei in fase S mitotica e meiotica entrambi etichetta con EdU. A dire le due popolazioni apart, determinare se la fase di S è stata seguita da mitosi o meiosi. Per determinare se i nuclei sono in fase S mitotica o meiotica, feed EdU per 4h e co-etichetta per pH3 (un indicatore di fase M) e HIM-3 (una proteina di asse meiotic del cromosoma) dalla macchiatura dell'anticorpo. Registrare i nuclei che visualizzano sia EdU e pH3 (tipo A, Vedi tabella 1 e Figura 2) come S-fase mitotica mentre i nuclei che visualizzano sia EdU e HIM-3 (tipo E, Vedi tabella 1 e Figura 2) come S-fase meiotica.
- (Variazione IV) Calcolare la durata della fase G2.
Nota: Fase G2 separa la fase S dalla fase di M. Anche se nessun marcatore è stato segnalato in etichetta G2 nella linea germinale di c. elegans , si può calcolare la durata della fase G2 combinando i dati da diversi esperimenti che etichetta M-fase (al momento della dissezione) e S-fase (a partire da diverse ore prima dissezione). Una cella che visualizza sia M-fase e fase S marcatori completato fase G2 nel corso dell'esperimento. Una cella che visualizza solo l'indicatore di fase M e non l'indicatore di fase S non era in fase S durante l'esperimento.- Per calcolare la durata della fase G2, feed EdU per 2 h e co-etichetta con anticorpo pH3. Esaminare solo i nuclei che etichettare con pH3 (queste sono in fase di M al momento della dissezione) per la presenza di EdU (questi erano in fase S durante l'etichetta di EdU 2 h prima della dissezione). Calcolare la frazione di nuclei di M-fase che completato fase G2 (A / A + B, Vedi tabella 1 e Figura 2).
- Ripetere questo esperimento con un'etichetta di EdU 3h e ancora con un'etichetta di EdU 4h (e, facoltativamente, un'etichetta di EdU 5h). Tracciare la percentuale di nuclei positivi pH3 che sono EdU positivo sull'asse y e la durata dell'EdU etichetta sull'asse x, come mostrato in Figura 3A.
- Calcolare la durata mediana della fase G2 collegando i punti sul grafico e determinare dove la linea attraversa il 50%, come mostrato in Figura 3A.
- Calcolare la durata massima della fase G2 collegando i punti sul grafico e determinare dove la linea attraversa il 99%, come mostrato in Figura 3A.
- (Variazione V) Calcolare il duraion di G1 + G2 + M.
Nota: In c. elegans germinale, fase G1 è insolitamente breve. Anche se nessun marcatore è stato segnalato per etichettare G1 sulla linea germinale di c. elegans , possibile stimare la durata di somma della fase G1 G2 e M e quindi confrontare questa volta con il tempo di fase G2 descritto sopra. La durata massima del G1 + G2 + M è stimata dalla percentuale di tutti i nuclei di zona progenitrici (WAPL-1 immunoreactive) che rimangono EdU negativo (non subissero fase S) dopo l'EdU etichettatura per diverse ore.- Per calcolare la durata del G1 + G2 + M, feed EdU per 2 h e co-etichetta con anticorpo REC-8 o WAPL-1. Determinare la frazione della zona progenitore che ha subito la fase S durante questo periodo (A + C / A + B + C + D, Vedi tabella 1 e Figura 2).
- Ripetere questo esperimento con un'etichetta di EdU 3h e ancora con un'etichetta di EdU 4h (e, facoltativamente, un'etichetta di EdU 5h). Tracciare la percentuale di REC-8 o WAPL-1 nuclei positivi che sono EdU positivo sull'asse y e la durata dell'EdU etichetta sull'asse x, come mostrato nella Figura 3B.
- Calcolare la durata massima del G1 + G2 + M collegando i punti sul grafico e trovare dove la linea attraversa il 99%, come mostrato nella Figura 3B.
Nota: È possibile eseguire gli esperimenti per determinare la durata del G2 e G2 + M + G1 come un singolo set di 2, 3, 4 e 5 h EdU esperimenti di co-etichettatura con anticorpi di coniglio-anti-WAPL-1 sia del mouse-anti-pH3.
- (Variazione VI) Per identificare i nuclei che replicato nella zona progenitrici dal immesso meiosi, nutrire gli animali EdU per 10 h e co-etichetta con anticorpi REC-8 o WAPL-1. Qualsiasi nuclei visualizzazione etichetta EdU erano in fase S durante quei 10 h. Qualsiasi nuclei che non vengono visualizzati adrenocorticale colorazione REC-8 o WAPL-1 erano nella meiosi. Basta contare i nuclei con EdU etichettatura che non vengono visualizzati l'etichettatura con l'indicatore di zona del progenitore (E, Vedi tabella 1).
Nota: Al contrario, se gonadi sono macchiati per il marcatore di Profase Meiotica HIM-3 con gli anticorpi anti-HIM-3, il numero dei nuclei con EdU etichettatura che sono anche positivo per HIM-3. - Per calcolare il tasso di entrata meiotica, eseguire l'esperimento di cui sopra con un'etichetta 5 h, 10 h e 15 h di EdU. Tracciare il numero di nuclei che entrato meiosi sull'asse y e la durata dell'etichetta EdU sull'asse x, come mostrato in Figura 3. Quindi utilizzare una regressione lineare per calcolare la pendenza (meiosi i nuclei entrati a h) da y = mx + b.
Nota: È fondamentale utilizzare una regressione lineare per calcolare il tasso di entrata meiotica. Non sarebbe corretto per dividere semplicemente il numero di nuclei che immesso meiosi dalla durata dell'etichetta EdU, perché l'intercetta y non è zero. - (Variazione VII) Misurare il tasso di progressione meiotica.
Nota: Poiché EdU in covalenza è incorporato nel DNA, può essere utilizzato per tenere traccia di una popolazione di cellule con differenziazione. Le cellule che hanno subito la fase S nella zona di progenitrici conservano l'etichetta EdU entrano in meiosi, progressi attraverso meiosi e subiscono oogenesi. Un esperimento di pulse-chase con EdU può essere utilizzato per misurare la velocità di progressione meiotica.- Batteri di EdU-labeled mangimi agli animali per 4 h (il "polso"). Trasferire gli animali senza etichetta OP50 batteri per 48 h (la "caccia"), poi sezionare e co-etichetta con un indicatore di zona del progenitore come REC-8 o WAPL-1 (o un indicatore di Profase Meiotica come HIM-3), se lo si desidera.
- Quando la formazione immagine, cercare la posizione del nucleo di EdU-labeled più prossimale. Il tasso di progressione meiotica è percorsa la distanza (in diametri cellulari dalla fine della zona di progenitrici) dal più prossimale EdU etichettato nucleo durante l'inseguimento di 48 h.
- (Variazione mi) Per identificare i nuclei in fase S, nutrire gli animali EdU per 30 min. Qualsiasi visualizzazione etichetta EdU i nuclei sono i nuclei di fase S. Per calcolare, prendere i nuclei somma di A e C, Vedi tabella 1 e Figura 2.
Risultati
Poiché per incorporare EdU sono necessaria la sintesi del DNA, si può concludere che EdU-identificati i nuclei hanno subito la fase S durante l'intervallo di tempo EdU-etichettatura. Si può interpretare i nuclei quell'etichetta in un'alimentazione di 30 min con EdU batteri identificati come appartenenti a nuclei in fase S al momento della dissezione. I nuclei che etichettare in un più EdU continuo esperimento di alimentazione possono avere etichettato presto nella finestra temporale e dal sinistro S-fase, o possono avere etichettato nella parte tardiva della finestra temporale di EdU. EdU segnale co-localizza con segnale DAPI. In alcuni nuclei, EdU segnale copre tutti i cromosomi, mentre in altri nuclei EdU segnale si localizza puncta luminoso di 1 – 2 (Figura 4). Questi puncta sono probabilmente il cromosoma x, che replica tardi nella fase S13.
Qui, gli animali sono stati alimentati con EdU continuamente per 30 min e sezionato, come descritto sopra e nella Figura 5. Un esempio di successo EdU macchiatura in un animale adulto giovane e un esempio di riuscita EdU macchiatura in un più vecchio animale adulto (Vedi sotto) sono mostrati in Figura 4. EdU segnale da un'etichettatura 30 min si localizza a circa la metà dei nuclei nella zona del progenitore (definito da WAPL-1 anticorpo etichettatura ma approssimato da DAPI morfologia26,27,28). Indice di fase S, la proporzione della zona progenitore che è EdU positivo, era precedentemente segnalati al 57 ± 5% e addirittura del 70% nei giovani adulti1,2,3. Indice di M-fase è di circa 2 – 3%1,29. In continuo di alimentazione per 4 ore o più, tutti i nuclei nell'etichetta di zona del progenitore con EdU e alcuni nuclei che etichettati nella zona del progenitore sono allora entrati Profase Meiotica1.
Mentre la tecnica funziona in modo coerente nel selvaggio-tipo animali adulti, giovani, una frazione significativa di accoppiate ermafroditi 5 giorno vecchi (anche quelli contenenti sperma) non è riuscito a etichettare in un 30 min impulso EdU (Figura 4E). Tuttavia, con un 4h EdU alimentazione, quasi tutti questi animali etichetta. Sporadico guasto all'etichetta in genetici animali femminili con brevi impulsi di EdU è stato anche segnalato30. Ci possono essere altre situazioni che provocare il guasto sporadico all'etichetta.
Si può calcolare la durata del ciclo cellulare effettuando diverse EdU-etichettatura esperimenti con pH3 etichettatura in ciascuno. La durata del G2 era stimata analizzando la percentuale di nuclei in fase M (pH3 immunoreactive) che erano EdU positivo nel corso del tempo (Figura 6). Questo approccio dà mediana e durata massima di G2 (Figura 3A). Il tempo mediano è stato interpolato, mostrando una durata approssimativa di G2 di 2,5 h in giovane adulti ermafroditi. La durata del G1 + G2 + M è stata stimata dalla percentuale di tutti i nuclei di zona progenitrici (WAPL-1 immunoreactive) che erano EdU positivo (Figura 6). Il metodo G2 + M + G1 fornisce una misura di durata massima per le fasi combinate (Figura 3B). Il tempo 99o percentile è stato interpolato, mostrando una durata approssimativa di G1 + G2 + M di h 3,4 in giovane adulti ermafroditi. Dati dagli stessi esperimenti sono stati utilizzati per calcolare il tasso di entrata meiotica (nuclei a h). La velocità è la pendenza della regressione lineare del numero di nuclei che entrato meiosi (EdU positivo, positivo negativo o HIM-3 WAPL-1) nel corso della durata dell'etichetta EdU (Figura 3). I valori per il selvaggio-tipo 1 giorno di età adulte ermafroditi sono mostrati nella tabella 2.
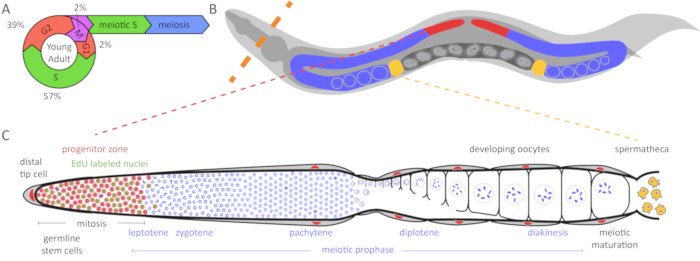
Figura 1: Diagramma del ciclo cellulare e c. elegans germline. (A) il ciclo cellulare delle cellule di germe in linea germinale ermafrodita adulta giovane. I numeri indicano la percentuale approssimativa di tempo trascorso in ogni fase del ciclo cellulare. (B) di c. elegans ermafroditi hanno due germlines a forma di U (rosso e blu). La spermateca è mostrato in giallo e l'utero con lo sviluppo di embrioni è in grigio scuro. La linea arancione tratteggiata indica dove gli animali vengono sezionati per estrudere il germlines. Diagramma (C) di una distesa di c. elegans germinale. DAPI (blu) è un colorante di DNA che evidenzia la morfologia nucleare. La zona distale progenitrici (evidenziata in rosso in base WAPL-1 macchiatura dell'anticorpo) contiene mitotically in bicicletta le cellule staminali, cellule progenitrici e cellule in fase S meiotica (WAPL-1 etichette anche i nuclei gonade somatica). Le cellule in fase S mitotica e meiotica etichettare con un 30 min EdU impulso e sono indicate in verde. Due celle in fase M etichetta con anticorpo pH3 e sono visualizzate in nero. La cella di punta distale (DTC) fornisce il ligando di GLP-1/tacca per mantenere il destino delle cellule staminali di queste cellule. Come le cellule migrano dal DTC, che uscire dalla zona di progenitore e immettere Profase Meiotica. Celle gialle sono spermatozoi nella spermateca. Clicca qui per visualizzare una versione più grande di questa figura.
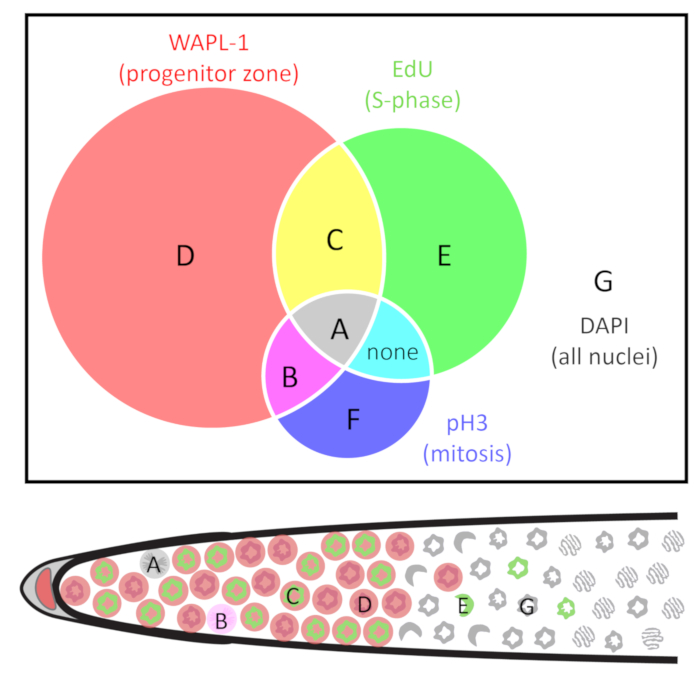
Figura 2: diagramma di Venn delle classi dei nuclei. I nuclei sono raggruppati dalla presenza e dall'assenza di tre indicatori: WAPL-1 indica cellule progenitrici zona (rosso), EdU indica cellule di S-fase (verde) e pH3 indica cellule di M-fase (blu). Tipi di cellule sono identificati come A-G. Nota che nel selvaggio-tipo giovane adulti ermafroditi non si trovano le celle di tipo F, e le cellule non co-etichette con EdU e pH3 fuori il progenitore (WAPL-1 positivo) fuso. Il seguente diagramma di gonade distale indica un esempio di A-E ed i nuclei G. Per ulteriori dettagli, vedere tabella 1 . Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: rappresentazione grafica della durata del ciclo cellulare e di dati sperimentali voce meiotic. (A) la durata del G2 fase viene interpolato da pH3 ed EdU co-etichettatura seguente durata varia EdU pulsa linee grigie indicano 50th e 99th percentili utilizzati in interpolazione mediana e durata massima di G2, indicata dalle frecce. (B) una cellula in fase G1 e G2, M o non incorpora EdU. Quindi, la durata massima della fase G1 + G2 + M può essere valutata misurando la durata massima dell'etichetta di EdU che produce cellule EdU-negative. La durata della fase G1 + G2 + M viene interpolata da EdU e REC-8 co-etichettatura seguendo gli impulsi di durata varia EdU. Linea grigia indica 99o percentile utilizzato in interpolazione durata massima G1 + G2 + M, indicata da una freccia. Non è possibile interpolare la durata mediana di G1 + G2 + M. (C) il tasso di entrata meiotica (nei nuclei a h – Vedi tabella 2) viene calcolata dalla pendenza della retta di regressione. Si noti che, poiché l'intercetta y-intercettazione non è zero, una regressione è necessaria per un calcolo preciso del tasso di entrata meiotica (C). . Barre di errore indicano deviazione standard. Cifre per volta e ristampato con il permesso da Fox et al. 20111. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: esempio di successo e insuccesso 30 min EdU macchiatura. Immagini al microscopio confocale di un 1 giorno di vita (A-D) e una 5 giorno vecchia gonade hermaphrodite (E-H) (non sperma impoverito) dopo un 30 min EdU esperimento d'etichettatura. La linea tratteggiata bianca segna la fine della zona di progenitrici. L'asterisco contrassegna la posizione della punta distale. Segni verdi EdU macchiatura visualizzata da scegliere chimica (A). Infruttuoso EdU etichettatura conseguente basso livello sfondo macchiatura ma nessun frammento luminoso EdU + (E). Immunofluorescenza di segni rossi WAPL-1 (B, F). Il giallo indica la sovrapposizione (C, G). Segni blu DAPI macchiatura per DNA (D, H). Punte di freccia singole indicare un nucleo con EdU macchiatura in tutta la cromatina. Doppie punte di freccia indicano un nucleo con EdU puncta su solo una coppia di cromosomi. Le immagini sono state ottenute con un obiettivo X 63. Barra della scala = 10 µm (D, H). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: flusso di lavoro sperimentale. Una sintesi del protocollo sperimentale a crescere (A), etichetta EdU (B), sezionare (C), macchia di anticorpo (D), eseguire la reazione di clic per fissare un colorante alla EdU (E), macchia di DNA (F), germlines (G), di immagine e quantificare EdU etichettati e anticorpo macchiato i nuclei (H). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: esempio di successo 4h EdU macchiatura. Immagini al microscopio confocale di una gonade ermafrodita adulta 1 giorno di vita dopo un 4h EdU etichettatura esperimento. La linea tratteggiata bianca segna la fine della zona di progenitrici. L'asterisco contrassegna la posizione della punta distale. Magenta segna pH3 immunofluorescenza (A, C). Segni verdi EdU macchiatura visualizzata da scegliere chimica (B, C). Immunofluorescenza di segni rossi WAPL-1 (D). Il giallo indica la sovrapposizione di EdU e WAPL-1 (E). Segni blu DAPI macchiatura per DNA (F). Punte di freccia singole indicare i nuclei co-etichettati con EdU e pH3. Doppia freccia segna un pH3 + EdU-nucleo - un avvenimento raro in un 4h EdU etichettatura. Le frecce contrassegno EdU + WAPL-1 - i nuclei che sono entrati in meiosi. Le immagini sono state ottenute con un obiettivo X 63. (F) viene visualizzata una barra di scala di 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.
| Marcatore: | pH3 | EdU * | WAPL-1 o REC-8 | LUI-3 | |
| Interpretazione: | Mitosi | S-fase * | Progenitrici zona | Meiosi | |
| Classe: | Interpretazione congiunta: | ||||
| A |  |  |  |  | in fase M, nella zona di progenitrici, erano in fase S mitotico durante etichetta EdU (completato G2) |
| B |  |  |  |  | in fase M, nella zona di progenitore, non erano in fase S durante etichetta EdU |
| C |  |  |  |  | in interfase, nella zona di progenitrici, erano in fase S durante etichetta EdU |
| D |  |  |  |  | in interfase, nella zona di progenitore, non erano in fase S durante etichetta EdU |
| E |  |  |  |  | nella meiosi, erano in fase S meiotica durante etichetta EdU (nuclei di voce meiotica) |
| F |  |  |  |  | tornare alla mitosi (trovato in alcuni mutanti) o divisioni meiotiche (nella spermatogenesi) |
| G |  |  |  |  | nella meiosi, non erano in fase S durante etichetta EdU |
| somma totale pH3 positivo; tutte le cellule in fase M | somma totale EdU positivo; tutte le cellule in fase S | somma totale positivo di WAPL-1; tutte le celle nella zona di progenitrici | somma totale HIM-3 positivo; tutte le celle nella profase meiotica |
Tabella 1: classi di nuclei. * Notare che esperimenti di EdU 30 min e 4 h differiscono nella interpretazione. Durata più lunga EdU esperimenti, le cellule hanno probabilmente superata fase S. Vedere Introduzione e passaggio 8 per durata di EdU etichettatura per esperimento pertinente.
| Parte del ciclo cellulare | Definizione operativa | Calcolo * | Valore * * |
| Nuclei di cellule progenitrici zona | tutte le WAPL-1 (o REC-8), positivi, HIM-3 negativo ai nuclei | A + B + C + D | 231 ± 23 nuclei |
| Nuclei di fase S | nuclei EdU positivo dopo 30 min EdU etichetta e WAPL-1 positivo | A + C | 133 ± 20 nuclei |
| Nuclei fase M | pH3 e nuclei co-positive WAPL-1 | A + B | 5,2 ± 2.3 nuclei |
| Indice di fase S | I nuclei di fase S / nuclei di cellule progenitrici zona | A + C / A + B + C + D | 57% del ciclo cellulare |
| Indice di fase M | I nuclei M-fase / i nuclei progenitrici zona | A + B / A + B + C + D | 2% del ciclo cellulare |
| Meiotiche cellule di voce | EdU etichettato i nuclei nella meiosi | E | varia a seconda della durata dell'etichetta EdU |
| Meiotica tasso di entrata | Nuclei di voce meiotica per h di etichetta EdU | Pendio da figura 4 C * * * | 20,3 nuclei a h |
| Durata G2 (mediana) | intercetta il 50% da Figure4A | h 2,5 | |
| Durata G2 (massimo) | 99% intercetta da figura 4A | h 3,5 | |
| G1 + G2 + M durata (massimo) | 99% intercetta da figura 4B | h 3,5 | |
| Durata del ciclo cellulare (mediana) | durata mediana di G2 / G2-indice * * * | h 6,5 | |
| Durata del ciclo cellulare (massimo) | durata massima G2 / G2-indice * * * | h 8,1 | |
Tabella 2: ciclo cellulare calcoli. * Lettere rappresentano le classi dei nuclei definiti nella tabella 1 e nella figura 3. I calcoli vengono modificati da Fox et al. 20111. * * Valori (± deviazione standard) per ermafroditi di selvaggio-tipo alzati a 20 ° C a fase di metà-L4 post h 24 anni. Si noti che, poiché l'intercetta y-intercettazione non è zero, una regressione è necessaria per un calcolo preciso del tasso di entrata meiotica. G2-indice è determinato sottraendo l'indice di S-fase, fase M indice e approssimativo G1-indice (2%) da 100%, come descritto da Fox et al. 20111.
Discussione
Preparazione di EdU-identificati batteri (passaggio 1) è critica per questo protocollo e il primo punto della risoluzione dei problemi. Etichetta di selvaggio-tipo giovane adulto ermafroditi molto affidabile in un 4h EdU-impulso, rendendo questo un controllo utile per ogni nuovo lotto di EdU-identificati batteri. Inoltre, intatti EdU-etichetta per i batteri che entrano l'intestino (in più vecchi animali o alcuni mutanti difettosi faringe/smerigliatrice) verranno etichettare con clic chimica e appaiono come brillante puncta oblunga nell'intestino. Una tecnica alternativa per l'etichettatura ermafroditi utilizza un "ammollo" in alta concentrazione (1 mM) di EdU3. Questa tecnica affama gli animali per la durata di etichettatura, ma fornisce un utile modo per bypassare facendo EdU-identificati batteri durante la risoluzione di fissazione e fare clic su chimica. Se un esperimento di EdU "ammollo" è successo mentre un feed di EdU non è, quindi preparare freschi EdU-identificati batteri. Per raggiungere una sufficiente densità batterica ottenendo anche un alto contenuto di EdU, uno potrebbe essere necessario regolare le concentrazioni di EdU e timidina.
La limitazione principale di questa tecnica per l'etichettatura di fase S è nella necessità di alimentare EdU-identificati batteri agli animali. Gli animali che non possono nutrire (a causa di genotipo o fase) non possono essere etichettati con questa tecnica. Tuttavia, analoghi nucleosidici sono attualmente l'unico metodo per identificare i nuclei di fase S sulla linea germinale di c. elegans , e loro utilizzo non richiede che qualsiasi transgeni essere presente negli animali. Inoltre, una volta incorporato, EdU resti nei nuclei anche come uscita S-fase, progressi attraverso il ciclo cellulare, dividono o differenziano. Il segnale si indebolisce di metà ad ogni divisione cellulare. Questo rende EdU perfetto per tracciare la storia di una cella anche attraverso poche divisioni cellulari.
La stabilità di EdU fa esperimenti di pulse-chase semplice; basta sciacquare in eccesso EdU batteri dagli animali dopo l'impulso di durata desiderata è finito e trasferire gli animali ai batteri senza etichetta. EdU rimane nel DNA e rimane visibile anche dopo molteplici divisioni cellulari. Tuttavia, gli esperimenti sono limitati a un singolo tipo di etichetta di fase S (un singolo impulso di EdU). Co-etichettatura con EdU e BrdU in cellule di mammifero31 è possibile ma non è stata segnalata in c. elegans. Co-etichettatura di IdU e CldU viene utilizzato in mammiferi32 ma anche non è stata segnalata in c. elegans.
I principali vantaggi di EdU etichettatura sono che il metodo non richiede nessun transgeni, EdU possono essere alimentati al c. elegans durante la coltura di regolare, la chimica è compatibile con le tecniche di immunofluorescenza ed EdU persiste nel DNA per lungo tempo dopo alimentazione ha Ci siamo fermati. Queste caratteristiche rendono EdU un ottimo strumento per studiare molti aspetti della dinamica del ciclo cellulare e delle cellule di germe.
Analisi delle dinamiche del ciclo cellulare e delle cellule di germe con EdU può essere applicata ad una varietà di domande di ricerca. Solo alcuni esempi di ulteriori applicazioni di questo metodo: come cambiano le dinamiche del ciclo cellulare in animali con le mutazioni di gene del ciclo cellulare? Come influiscono le condizioni fisiologiche del ciclo cellulare in cellule staminali, il tasso di entrata delle cellule di germe in Profase Meiotica e la velocità di progressione delle cellule di germe attraverso Profase Meiotica? Come cambia il ciclo cellulare durante lo sviluppo larvale? Come segnalazione via gravi perturbazioni influenzano il ciclo cellulare, oltre a modifiche nel destino della cellula (ad esempio proliferazione ectopico)? Questo sistema può essere modificato per studiare che cosa stanno facendo le cellule in diverse condizioni.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Siamo grati al centro stock di e. coli per MG1693; WormBase; il centro di genetica di Caenorhabditis che è finanziato dai nazionali istituti di salute di ricerca infrastrutture programmi di Office (P40OD010440) per i ceppi; Zach Pincus per consulenza statistica; Aiping Feng per reagenti; Luke Schneider, Andrea Scharf, Sandeep Kumar e John Brenner per formazione, consulenza, supporto e discussioni utili; e i laboratori Kornfeld e Schedl per feedback su questo manoscritto. Questo lavoro è stato supportato in parte dal National Institutes of Health [R01 AG02656106A1 a KK, R01 GM100756 TS] e una borsa di dottorato National Science Foundation [DGE-1143954 e DGE-1745038 a ZK]. Il National Institutes of Health, né la National Science Foundation ha avuto alcun ruolo nella progettazione di studio, raccolta, analisi e interpretazione dei dati, né per iscritto il manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| E. coli MG1693 | Coli Genetic Stock Center | 6411 | grows fine in standard unsupplemented LB |
| E. coli OP50 | Caenorhabditis Genetics Center | OP50 | |
| Click-iT EdU Alexa Fluor 488 Imaging Kit | Thermo Fisher Scientific | C10337 | |
| 5-Ethynyl-2′-deoxyuridine | Sigma | 900584-50MG | or use EdU provided in kit |
| Glucose | Sigma | D9434-500G | D-(+)-Dextrose |
| Thiamine (Vitamin B1) | Sigma | T4625-5G | Reagent Grade |
| Thymidine | Sigma | T1895-1G | BioReagent |
| Magnesium sulfate heptahydrate | Sigma | M1880-1KG | MgSO4, Reagent Grade |
| Sodium Phosphate, dibasic, anhydrous | Fisher | BP332-500G | Na2HPO4 |
| Potassium Phosphate, monobasic | Sigma | P5379-500G | KH2PO4 |
| Ammonium Chloride | Sigma | A4514-500G | NH4Cl, Reagent Plus |
| Bacteriological Agar | US Biological | C13071058 | |
| Calcium Chloride dihydrate | Sigma | C3881-500G | CaCl |
| LB Broth (Miller) | Sigma | L3522-1KG | Used at 25g/L |
| Levamisole | Sigma | L9756-5G | 0.241g/10ml |
| Phosphate buffered saline | Calbiochem Omnipur | 6506 | homemade PBS works just as well |
| Tween-20 | Sigma | P1379-500ML | |
| 16% Paraformaldehyde, EM-grade ampules | Electron Microscopy Sciences | 15710 | 10ml ampules |
| 100% methanol | Thermo Fisher Scientific | A454-1L | Gold-label methanol is critical for proper morphology with certain antibodies |
| Goat Serum | Gibco | 16210-072 | Lot 1671330 |
| rabbit-anti-WAPL-1 | Novus biologicals | 49300002 | Lot G3048-179A02, used at 1:2000 |
| mouse-anti-pH3 clone 3H10 | Millipore | 05-806 | Lot#2680533, used at 1:500 |
| goat-anti-rabbit IgG-conjugated Alexa Fluor 594 | Invitrogen | A11012 | Lot 1256147, used at 1:400 |
| goat-anti-mouse IgG-conjugated Alexa Fluor 647 | Invitrogen | A21236 | Lot 1511347, used at 1:400 |
| Vectashield antifade mounting medium containing 4',6-Diamidino-2-Phenylindole Dihydrochloride (DAPI) | Vector Laboratories | H-1200 | mounting medium without DAPI can be used instead, following a separate DAPI incubation |
| nail polish | Wet n Wild | DTC450B | any clear nail polish should work |
| S-medium | various | see wormbook.org for protocol | |
| M9 buffer | various | see wormbook.org for protocol | |
| M9 agar | various | same recipe as M9 buffer, but add 1.7% agar | |
| Nematode Growth Medium | various | see wormbook.org for protocol | |
| dissecting watch glass | Carolina Biological | 42300 | |
| Parafilm laboratory film | Pechiney Plastic Packaging | PM-996 | 4 inch wide laboratory film |
| petri dishes | 60 mm diameter | ||
| Long glass Pasteur pipettes | |||
| 1ml centrifuge tubes | MidSci Avant | 2926 | |
| Tips | |||
| Serological pipettes | |||
| 500 mL Erlenmyer flask | |||
| Aluminum foil | |||
| 25G 5/8” needles | BD PrecisionGlide | 305122 | |
| 5ml glass centrifuge tube | Pyrex | ||
| Borosilicate glass tubes 1ml | |||
| glass slides | |||
| no 1 coverslips 22 x 40 mm | no 1.5 may work, also | ||
| 37 °C Shaker incubator | |||
| Tabletop Centrifuge | |||
| Clinical Centrifuge | IEC | 428 | with 6 swinging bucket rotor |
| Mini Centrifuge | |||
| 20 °C incubator | |||
| 4 °C refrigerator | |||
| -20 °C freezer | |||
| Observer Z1 microscope | Zeiss | ||
| Plan Apo 63X 1.4 oil-immersion objective lens | Zeiss | ||
| Ultraview Vox spinning disc confocal system | PerkinElmer | Nikon spinning disc confocal system works very well, also, as described here: http://wucci.wustl.edu/Facilities/Light-Microscopy |
Riferimenti
- Fox, P. M., Vought, V. E., Hanazawa, M., Lee, M. -H., Maine, E. M., Schedl, T. Cyclin E and CDK-2 regulate proliferative cell fate and cell cycle progression in the C. elegans germline. Development. 138 (11), Cambridge, England. 2223-2234 (2011).
- Crittenden, S. L., Leonhard, K. A., Byrd, D. T., Kimble, J. Cellular analyses of the mitotic region in the Caenorhabditis elegans adult germ line. Molecular biology of the cell. 17 (7), 3051-3061 (2006).
- Seidel, H. S., Kimble, J. Cell-cycle quiescence maintains Caenorhabditis elegans germline stem cells independent of GLP-1/Notch. eLife. 4, (2015).
- Fox, P. M., Schedl, T. Analysis of Germline Stem Cell Differentiation Following Loss of GLP-1 Notch Activity in Caenorhabditis elegans. Genetics. 201 (9), 167-184 (2015).
- Kocsisova, Z., Kornfeld, K., Schedl, T. Cell cycle accumulation of the proliferating cell nuclear antigen PCN-1 transitions from continuous in the adult germline to intermittent in the early embryo of C. elegans. BMC Developmental Biology. 18 (1), (2018).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proceedings of the National Academy of Sciences. 105 (7), 2415-2420 (2008).
- vanden Heuvel, S., Kipreos, E. T. C. elegans Cell Cycle Analysis. Methods in Cell Biology. , 265-294 (2012).
- ThermoFisher. Click-iT EdU Imaging Kits. , https://assets.thermofisher.com/TFS-Assets/LSG/manuals/mp10338.pdf (2011).
- Pazdernik, N., Schedl, T. Germ Cell Development in C. elegans. , Springer. 1-16 (2013).
- Hirsh, D., Oppenheim, D., Klass, M. Development of the reproductive system of Caenorhabditis elegans. Developmental Biology. 49 (1), 200-219 (1976).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Hansen, D., Schedl, T. Stem cell proliferation versus meiotic fate decision in Caenorhabditis elegans. Advances in Experimental Medicine and Biology. 757, 71-99 (2013).
- Jaramillo-Lambert, A., Ellefson, M., Villeneuve, A. M., Engebrecht, J. Differential timing of S phases, X chromosome replication, and meiotic prophase in the C. elegans germ line. Developmental Biology. 308 (1), 206-221 (2007).
- Agarwal, I., Farnow, C., et al. HOP-1 presenilin deficiency causes a late-onset notch signaling phenotype that affects adult germline function in Caenorhabditis elegans. Genetics. 208 (2), 745-762 (2018).
- Gumienny, T. L., Lambie, E., Hartwieg, E., Horvitz, H. R., Hengartner, M. O. Genetic control of programmed cell death in the Caenorhabditis elegans hermaphrodite germline. Development. 126 (5), Cambridge, England. 1011-1022 (1999).
- Hendzel, M. J., Wei, Y., et al. Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106 (6), 348-360 (1997).
- Zetka, M. C., Kawasaki, I., Strome, S., Mü Ller, F. Synapsis and chiasma formation in Caenorhabditis elegans require HIM-3, a meiotic chromosome core component that functions in chromosome segregation. Genes & development. 13 (17), https://www.ncbi.nlm.nih.gov/pmc/articles/PMC317003/pdf/x8.pdf 2258-2270 (1999).
- Hansen, D., Hubbard, E. J. A., Schedl, T. Multi-pathway control of the proliferation versus meiotic development decision in the Caenorhabditis elegans germline. Developmental Biology. 268 (2), 342-357 (2004).
- Crawley, O., Barroso, C., et al. Cohesin-interacting protein WAPL-1 regulates meiotic chromosome structure and cohesion by antagonizing specific cohesin complexes. eLife. 5 (2), 1-26 (2016).
- Zhao, H., Halicka, H. D., et al. DNA damage signaling, impairment of cell cycle progression, and apoptosis triggered by 5-ethynyl-2′-deoxyuridine incorporated into DNA. Cytometry Part A. 83 (11), 979-988 (2013).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Gervaise, A. L., Arur, S. Spatial and Temporal Analysis of Active ERK. in the C. elegans Germline. Journal of Visualized Experiments. 117 (117), e54901-e54901 (2016).
- Vos, K. De Cell Counter Plugin. , https://imagej.nih.gov/ij/plugins/cell-counter.html (2015).
- Schindelin, J., Arganda-Carreras, I., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Rasband, W. ImageJ. , http://imagej.nih.gov/ij/> (2016).
- Michaelson, D., Korta, D. Z., Capua, Y., Hubbard, E. J. A. Insulin signaling promotes germline proliferation in C. elegans. Development. 137 (4), Cambridge, England. 671-680 (2010).
- Qin, Z., Jane, E., Hubbard, A., Hubbard, E. J. A. Non-autonomous DAF-16/FOXO activity antagonizes age-related loss of C. elegans germline stem/progenitor cells. Nature communications. 6 (5), 7107(2015).
- Luo, S., Kleemann, G. A., Ashraf, J. M., Shaw, W. M., Murphy, C. T. TGFB and Insulin Signaling Regulate Reproductive Aging via Oocyte and Germline Quality Maintenance. Cell. 143 (2), 299-312 (2010).
- Narbonne, P., Maddox, P. S., Labbe, J. -C. daf-18/PTEN locally antagonizes insulin signalling to couple germline stem cell proliferation to oocyte needs in C. elegans. Development. , 4230-4241 (2015).
- Cinquin, A., Chiang, M., et al. Intermittent Stem Cell Cycling Balances Self-Renewal and. Senescence of the C. elegans Germ Line. PLoS Genetics. 12 (4), 1005985(2016).
- Invitrogen EdU (5-ethynyl-2’-deoxyuridine). , https://assets.thermofisher.com/TFS-Assets/LSG/manuals/mp10044.pdf 1-7 (2010).
- Tuttle, A. H., Rankin, M. M., et al. Immunofluorescent Detection of Two Thymidine Analogues (CldU and IdU) in Primary Tissue. Journal of Visualized Experiments. 46 (46), e2166-e2166 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon