Method Article
Uma combinatória célula única abordagem para caracterizar o Molecular e imunofenotípica heterogeneidade de tronco humano e populações de Progenitor
Neste Artigo
Resumo
Granel gene expressão medições nuvem célula individual diferenças nas populações de células heterogêneas. Aqui, descrevemos um protocolo para análise de expressão do gene como unicelular e índice classificação por Florescence ativado celular classificação (FACS) pode ser combinado para delinear a heterogeneidade e immunophenotypically caracterizam populações de células molecularmente distinta.
Resumo
Caracterização imunofenotípica e análise molecular tem sido muito utilizados para delinear a heterogeneidade e definir populações de células distintas. FACS é inerentemente um ensaio de célula única, no entanto antes da análise molecular, as células de destino são isoladas frequentemente prospectivamente a granel, perdendo assim a resolução de célula única. Análise da expressão de gene de célula única fornece um meio para compreender diferenças moleculares entre células individuais em populações de células heterogêneas. Na análise de células em massa uma sobre-representação de um tipo de células distintas resulta em preconceitos e oclusões de sinais de células raras com importância biológica. Utilizando o FACS índice classificação acoplado a análise da expressão de gene de célula única, populações podem ser investigadas sem perda de resolução de célula única enquanto células com expressão de marcador de superfície de célula intermediária também são capturadas, permitindo a avaliação de a relevância da expressão do marcador de superfície contínua. Aqui, descrevemos uma abordagem que combina célula única transcrição reversa quantitativo PCR (RT-qPCR) e FACS índice classificação para caracterizar simultaneamente o molecular e imunofenotípica heterogeneidade dentro de populações de células.
Em contraste com os métodos de sequenciamento de RNA célula única, o uso de qPCR com amplificação de destino específico permite para medições robustas de baixa abundância transcrições com menos interrupções, enquanto isso não é confundido por questões relacionadas às variações de célula para célula em leitura profundidade. Além disso, por diretamente esse método de buffer de ordenação de índice único-células em Lise, permite para a síntese do cDNA e pré-amplificação alvo específico para ser executada em uma única etapa, bem como quanto à correlação de assinaturas moleculares posteriormente derivadas com superfície celular expressão do marcador. A abordagem descrita foi desenvolvida para investigar single-células hematopoiéticas, mas também têm sido utilizados com sucesso em outros tipos de células.
Em conclusão, a abordagem aqui descrita permite medição sensível da expressão de RNAm para um painel de genes pré-selecionados com a possibilidade para desenvolver protocolos para isolamento prospectivo subsequente de subpopulações molecularmente distintas.
Introdução
Cada célula individual do sangue é acreditada para residir em uma hierarquia de celular, onde as células-tronco formam o ápice em cima de uma série de progenitores intermediários cada vez mais comprometidos que eventualmente terminal se diferenciar em células efetoras final carregando específicas funções biológicas1. Muito do conhecimento sobre como os sistemas de células-tronco são organizados foi gerado no sistema hematopoiético, em grande parte devido a capacidade de isolar prospectivamente distintas populações hematopoiéticas altamente enriquecidas para células-tronco ou progenitoras vários2 , classificando o FACS. Isto permitiu para muitas dessas populações a serem analisados funcionalmente ou molecularmente, predominantemente através da expressão do gene de perfil3,4. No entanto quando analisando a expressão gênica das diferenças individuais de em massa das populações entre as células são em média para fora e perdeu5. Assim, incapacidade para detectar variações de célula para célula dentro frações heterogêneas célula pode confundir nosso entendimento de processos biológicos críticos se pequenos subconjuntos de células esclarecem a função biológica inferida do que a população6, 7. Por outro lado, investigação das assinaturas de expressão do gene na célula única resolução oferecem uma possibilidade de delinear a heterogeneidade e contornar a sombra influências de sobre-representados subconjuntos de células8.
Até à data têm sido desenvolvidos muitos protocolos para análise de expressão do gene de célula única; com cada abordagem tendo suas próprias limitações. O método mais antigo foi RNA fluorescente em situ hibridação (RNA-FISH), que mede um número limitado de transcrições de cada vez, mas é o único que permite a investigação de RNA localização9,11. Início métodos usando PCR e qPCR para detectar que um único ou poucos transcrições foram também desenvolvidos12. No entanto, estas ultimamente foram substituídas por métodos baseados em microfluídica que podem analisar simultaneamente a expressão de centenas de transcrições por célula em centenas de células através da qPCR e assim permitir para o uso de análise dimensional elevada heterogeneidade Pre-determinado gene painéis10,13. Recentemente tecnologias baseadas em sequenciamento de RNA tem tornar-se amplamente utilizadas para análise de célula única, como estes teoricamente podem medir a transcriptoma inteira de uma célula e, assim, adicionar uma dimensão exploratória a heterogeneidade análise10, 14. análise de qPCR multiplexados e sequenciamento de RNA de célula única têm características diferentes, assim, a justificativa para usar um dos métodos depende a pergunta, bem como o número de células na população-alvo. A alta produtividade e baixo custo por célula juntamente com características imparciais, exploratórias de sequenciamento de RNA de célula única são desejáveis quando célula desconhecida ou grandes populações são investigadas. No entanto, a sequenciação do ARN de célula única também é inclinada para sequenciamento transcrições abundantes altas mais frequentemente enquanto transcrições com baixa abundância são propensas a desistentes. Isso pode levar a dados consideravelmente complexos que coloca exigências de alta na análise de bioinformatic revelar importantes sinais moleculares que são muitas vezes sutis ou ocultas em ruído técnica15. Assim, para tecidos bem caracterizados, qPCR célula única análise usando pre-determinada painéis cartilha selecionados para funcionalmente importantes genes ou marcadores moleculares podem servir como uma abordagem sensível, simples para determinar a heterogeneidade de um população. No entanto, deve notar-se que em relação à célula única RNA-seq, o custo de cada célula é geralmente maior para métodos de célula única qPCR. Aqui, descrevemos uma abordagem que combina monocelulares RT-qPCR (modificado de J. Teles et al 16), índice de FACS classificação17 e bioinformática análise18 para simultaneamente caracterizam a heterogeneidade molecular e imunofenotípica dentro das populações.
Nesta abordagem, a população de células de interesse está manchada, e single-células são classificadas por FACS diretamente em tampão de Lise em placas de PCR de 96 poços. Simultaneamente, os níveis de expressão de um conjunto adicional de marcadores de superfície celular são registrados para cada célula única durante FACS-classificação, um método que é conhecido como índice de classificação. O material de lisados celulares é posteriormente amplificado e a expressão do gene de um conjunto selecionado de genes analisados com RT-qPCR, usando uma plataforma microfluidic. Esta estratégia permite a análise molecular da caracterização de célula única, bem como simultânea classificada de expressão do cada célula marcador da pilha-superfície. Mapeando diretamente molecularmente distintos subconjuntos de células para a expressão dos marcadores classificadas de indexado, as subpopulações podem ser vinculadas a um imunofenótipo específico que pode ser usado para seu isolamento em perspectiva. O método é descrito passo a passo na Figura 1. Um painel pre-determinado gene mais contribui para uma maior resolução da expressão do gene alvo, desde que contorna a medição dos genes abundantes irrelevantes que caso contrário pode ocluir os sinais de expressão do gene sutil. Além disso, a amplificação de destino específico, transcrição reversa de um passo e amplificação permite a medição robusta de baixas transcrições expressadas, como factores de transcrição ou RNAs não-poli-adenylated. Importante, qPCR métodos permitem a medição de mRNA de proteínas da fusão, que são importantes na investigação de determinadas doenças malignas,19. Finalmente, o foco número dos genes investigados, baixas taxas de abandono, e limitado as diferenças técnicas entre as células fazem este método facilmente analisado em comparação com métodos dimensionais mais elevados, como célula única RNA-Seq Seguindo o protocolo, pode ser realizada uma experiência inteira, de classificação células para resultados analisados, no prazo de três dias, tornando este um método simples e rápido para análise de expressão do gene de célula única sensível, elevado-throughput.
Protocolo
1. preparação das placas de Lise
- Usando um banco livre de RNA/DNA, prepare o suficiente do lysis para 96 poços, com 10% extra, misturando 390 água livre da nuclease µ l, 17 µ l de 10% NP-40, 2,8 µ l 10 mM dNTP, 10 µ l 0,1 M DTT e inibidor de RNAse 5,3 µ l (ver Tabela de materiais). Vórtice e spin-down.
- Distribuir 4 µ l de tampão de Lise para cada poço de uma placa PCR bem 96 e selar as placas com película adesiva. Spin para baixo os tubos para coletar o líquido na parte inferior das placas. Mantém as placas no gelo até a célula classificação (máximo 24 h).
2. preparação de células para triagem de célula
- Descongele o número adequado de células (aqui, CD34 enriquecido tronco hematopoiético e células progenitoras) para o experimento. 1 x 106 células são apropriadas para aproximadamente três placas de 96 poços de single-células com controles de classificação.
- Transferência de células descongeladas para um tubo cônico de 15 mL e adicionar 1 mL FBS cada 30 s até um volume total de 8 mL é atingido. Girar as células em uma centrífuga a 350 x g durante 10 minutos a 4 ° C e remover o sobrenadante.
- Ressuspender as células em 8 mL de tampão de coloração (PBS com 2% FBS) e centrifugar a 350 x g durante 10 minutos a 4 ° C e remover o sobrenadante.
- Os eritrócitos de coloração, 200 µ l de buffer e remover células para manchas de controle.
- Fazer fluorescência menos um controles (FMO) para cada fluoróforo, manchando uma fração das células em 50 µ l de tampão de coloração. Neste exemplo, 6 tubos de microcentrifuga com 20.000 células são usados como FMOSs. Observe que o número de células deve ser ajustado dependendo da população investigada. Adicione todos os anticorpos na mesma concentração como a mancha de amostra, com exceção de um para cada tubo.
- Fazer manchas única para cada fluoróforo manchando uma fração de células no buffer de 50 µ l de cada fluoróforo usado. Neste exemplo são utilizados 6 tubos microcentrifuga com 20.000 células. Observe que o destino de cada anticorpo precisa ser expresso pelas células usadas para controles. Adicione cada anticorpo na mesma concentração como a mancha de amostra em tubos individuais. Além disso, manter 20.000 células imaculadas em 50 µ l como um controle imaculado.
- Para a amostra de célula, acrescente os anticorpos em sua concentração adequada. Usados aqui são CD34-FITC numa concentração de 1/100, 1/50, CD38-APC CD90-PE 1/10, 1/50 CD45RA-bv421, CD49F-PECy7 1/50 e Mix de linhagem: CD3-PECy5 1/50, CD2-PECy5 1/50, CD19-PECy5 1/50, 1/50 de CD56-PECy5, CD123-PECy5 1/50, CD14-PECy5 1/50, CD16-PECy5 1/50, e CD235a-PECy5 1/1000.
- Incube as células com anticorpos por 30 min no gelo no escuro.
- Lavam-se células com 3 mL de tampão de coloração. Centrifugar as células a 350 x g durante 10 minutos a 4 ° C e remover o sobrenadante.
- Ressuspender as células e repita o passo 2.9.
- Resuspenda amostra em 500 µ l e FMO em 100 µ l de tampão com células 7AAD e filtro de 1/100 de coloração através de um filtro de 50 µm para obter uma suspensão de célula única.
3. célula de triagem
- Certifique-se de que a máquina de FACS é definida corretamente com atraso de entrega e instalação do citômetro e rastreamento (CST) que recentemente foram executados de acordo com as instruções do fabricante, para garantir que as células adequadas são classificadas. Para células hematopoiéticas, é recomendado o uso do mícron 85 bocal e máxima velocidade de 4, enquanto a taxa de evento ideal é entre 800 e 2000 eventos/s.
- Corrigir para sobreposição espectral, realizando a compensação de fluorescência e conjunto de portões de acordo com FMO controles ou controles internos negativos.
- Execute a reanálise da população alvo, classificando a pelo menos 100 células de destino em um novo tubo de microcentrifuga com 100 µ l de tampão de mancha. FACS analisar as células classificadas pela gravação da amostra classificada e certifique-se de que eles acabam na porta do tipo.
- Instalação placa de célula simples classificação por centrar a queda no poço A1 em um prato bem 96. Quando está centrado, tipo 50 – 100 6 µm de partículas em todos os poços em torno da borda de um prato vazio de 96 bem para garantir que todos os poços terá uma cela no centro de cada poço.
- Se possível, um controle adicional para garantir que as células viáveis são classificadas pode ser adicionado, classificando single-células para crescimento em vitro . As células hematopoiéticas podem ser cultivadas em placas bem de fundo U 96 em 100 µ l SFEM com estreptomicina de penicilina de 1%, 100 ng/mL, FLT3L, TPO e SCF. Analise cada poço após 3 dias na cultura para colônias de célula, usando um microscópio.
- Remova a película adesiva de placas. Tipo uma única célula de interesse (aqui Lin-CD34 + CD38-células) em 92 de 96 poços, ativar índice-classificação-nos FACS classificação software para salvar o perfil imunofenotípica para outros marcadores de interesse (aqui CD45RA, CD49f e CD90) para cada célula única.
- Classificar dois poços com células de 10 e 20, respectivamente para controles de linearidade na amplificação por PCR. Poços de H1 e H2 são usados geralmente.
- Manter dois poços sem quaisquer células como não-modelo controla, geralmente poços H3 e H4.
- Selar as placas com película adesiva e girar as placas a 300 x g por 1 min antes snap congelando em gelo seco.
- Armazenar as placas congeladas a-80 ° C.
Nota: Ponto de parada segura. Classificada e lisadas células podem ser mantidas a-80 ° C para armazenamento a longo prazo.
4. inverter a transcrição e a amplificação de destino específico
- Prepare a mistura de cartilha para todos os destinos do gene 96 adicionando 2 µ l de cada par de primer, incluindo limpeza gene primers e primers para cravado no controle do RNA, em um 1,5 mL tubo livre de RNAse num banco livre de RNA/DNA. Se usar menos de 96 cartilhas, adicione uma quantidade equivalente de água livre de nuclease para os primers faltando. As primeiras demão são ordenadas separadamente para combinar com o painel do gene desejado.
- Fazer a transcrição reversa e amplificação de destino específico mistura adicionando 632.5 µ l 2 x a mistura de reação, 101.2 µ l Taq/SuperscriptIII, mistura de Primer 151.8 µ l e 0,7 µ l cravado no controle do RNA. Pré-forma esta etapa em um banco de DNA gratuito. Mix por num Vortex e spin para baixo para coletar o líquido no fundo do tubo. Manter-se no gelo até adição à amostra.
- Faça transcrição reversa-não mistura de controle por quatro poços misturando 27,5 µ l da mistura de reação de 2x, 1.76 µ l da enzima Taq, 6.6 µ l de mistura da primeira demão e 2.64 µ l de água livre de nuclease. Spike no controle do RNA. Execute esta etapa em um banco de DNA gratuito. Vórtice e spin para baixo para coletar o líquido na parte inferior do tubo e manter em gelo até adição de amostra.
- Descongele o lisado placas no gelo. Adicione 8,75 µ l da transcrição reversa previamente preparada e mistura de amplificação de destino específico para 92 poços, incluindo os controles de linearidade e não-modelo. Adicione 8,75 µ l do mix de controle não-reverso transcrição nos quatro poços restantes. Selar as placas com película adesiva e rotação para baixo para coletar líquido na parte inferior das placas.
- Pré-forma transcrição reversa e amplificação de destino específico, executando a placa em uma máquina PCR de acordo com o programa do pré-amplificador; Passo 01:50 ° C por 60 min, passo 2: 95 ° C por 2 min, passo 3: 95 ° C por 15 s, passo 4: 60 ° C por 4 min, repetir os passos 3 – 4 24 vezes e finalmente passo 5: 8 ° C para sempre.
- Depois de PCR está completo, manter a placa a 8 ° C para armazenamento de curto prazo e -20 ° C para armazenamento a longo prazo.
Nota: Ponto de parada segura. Material amplificado pode ser mantido a 8 ° C para armazenamento de curto prazo e a-20 ° C para armazenamento a longo prazo.
5. preparação da amostra e placas de ensaio para análise da expressão de Gene de Microfluidic Multiplex
- Preparar o ensaio carregando a placa por pipetagem 3 reagente de carregamento do ensaio µ l de cada poço de uma placa bem 96. Adicione 3 µ l de cada primer para poços individuais no ensaio de placa de carregamento.
- Placa com película adesiva e spin para baixo para coletar líquido na parte inferior das placas do selo.
- Prepare a placa de diluição por pipetagem µ l 8 de nuclease água livre em todos os poços de uma placa bem 96. Adicione 2 µ l de amostra amplificada para a placa de diluição, tornando uma diluição final de 1:5.
- Placa com película adesiva do selo, misture por placa num Vortex para 10 s e finalmente girar para baixo para coletar líquido na parte inferior das placas.
- Prepare a mistura de carregamento da amostra misturando cuidadosamente 352 µ l do mix mestre com 35,2 µ l de reagente de carregamento de amostra. Prepare a placa de carregamento da amostra por alíquotas 3.3 µ l de mistura para cada poço de uma placa bem 96 a carregar.
- Adicione 2,7 µ l da amostra diluída em cada poço da amostra carregando a placa.
- Placa com película adesiva e spin para baixo para coletar líquido na parte inferior das placas do selo.
6. carregamento de Microfluidic Chip
- Tire um novo chip de microfluidic 96 x 96. Prepare entradas, picando-os com uma seringa com tampa em ter a certeza de que eles podem ser movidos.
- Remova as bolhas de seringas. Adicione o volume completo de seringas para cada válvula enquanto o chip de inclinação de 45 graus e pressionando para baixo a válvula. O primeiro chip com o controlador da IFC.
- Carrega a cada entrada de ensaio com 4,25 µ l de cada um dos poços no ensaio de placa de carregamento. Evite bolhas. Se as bolhas aparecem no poço, removê-los com a ponta da pipeta.
- Continuar a carregar a cada entrada de amostra com 4,25 µ l de cada um dos poços na amostra carregando a placa, evitar bolhas e se aparecem bolhas, remova-os com a ponta da pipeta.
- Carregar o chip com o controlador da IFC.
- Verifique se o chip parece mesmo e que todas as câmaras foram carregadas. Remova o pó da superfície do chip por tocá-lo com fita adesiva. Execute o chip na plataforma de expressão do gene microfluidic multiplex.
7. executando o Chip na plataforma de expressão do Gene de Microfluidic Multiplex
- Depois de carregar o chip para a plataforma de expressão do gene microfluidic multiplex, o nome da amostra.
- Definir ROX como corante passiva. Conjunto único teste e FAM-MGB como fluorescência. Use padrão 96x96 v2 como protocolo. Inicie-executar.
- Chip de remover quando executar é completa.
8. preliminar análise da gerência de Chip
- Carregar dados no software de análise de PCR em tempo real.
- Carregar o gene nomes e nomes de células colando layouts de célula e o gene de um arquivo delimitado por tabulação.
- Abra o Visualizador de imagens e selecione ROX como corante. Verifique se todos os poços tem tintura passiva ROX.
- Investiga se todas as parcelas de amplificação olhem okey, com uma curva suave de amplificação com sem espinhos (semelhantes a Figura 3E).
- Certifique-se de que todos os single-células têm expressão de cravado no controle do RNA para certificar-se de que todos foram carregados corretamente.
- Certifique-se de que todas as células têm expressão de gene das tarefas domésticas e, portanto, foram classificadas corretamente.
- Certifique-se que 10 a 20 controles de linearidade de célula tem aproximadamente 1 CT diferença para validar a amplificação linear.
- Verifique se há expressão nas amostras controle noRT. Se a expressão for detectado no noRT considere mudar sondas para pontas de prova que não detecta o DNA genômico para execuções subsequentes.
- Exporte dados em ficheiros csv para posterior análise.
9. célula única análise usando SCExV
Nota: Um filme introdutório é presente20 para introduzir a ferramenta. Aqui, uma recomendação curta de como fazer a análise usando os controles introduzidos no protocolo é apresentada.
- Ligue para o SCexV site20.
- Upload de arquivos CSV exportados.
- Escolheu o controle cravado no RNA como controle positivo. Remova qualquer célula que tem um controle de RNA CT acima de 25. Normalize os dados de expressão mediana do controle do RNA.
- Clique em "aqui -> Analyze". Remova noRT, notemplate, controles de célula de 10 e 20 na opção excluir células.
- Aglomerado de células com a abordagem de clusterização de escolha com o número esperado de clusters. Valores de exportação analisado.
10. classificação de índice de análise
- Abra o software de análise de FACS e carregar as amostras indexadas.
- Abra o editor de scripts e execute o script disponível de Quinn J. et al . 21
- Agora que o software de análise de FACS deveria ter feito um portão para cada uma das células simples, abra o editor de layout e cor das células de acordo com o agrupamento de SCexV (disponível no arquivo chamado "Sample_complete_Data.xls").
Resultados
O protocolo descrito é rápida, facilmente realizada e altamente confiável. Uma visão geral sobre a montagem experimental está apresentada na Figura 1. O protocolo inteiro, desde a triagem de single-células, a amplificação de destino específico, medições de expressão de gene e análise preliminar pode ser realizado em três dias. Um exemplo dos resultados analisados sob a forma de um mapa de calor que representa dados analisados preliminares da análise da expressão de gene de célula única utilizando 96 primers e 96 nas células de um paciente de leucemia mieloide crônica (LMC) ou 96 de correspondência um idoso saudável controle é apresentado na Figura 2. Usando agrupamento hierárquico, as células analisadas podem ser divididas em quatro subgrupos com base em suas assinaturas de expressão do gene. A visualização de mapa de calor é uma maneira conveniente de obter uma visão geral dos dados, bem como para controle de poços que devem ser excluídos da análise (por exemplo, poços de controle). Figura 2B é uma análise de componentes principais (PCA), visualizando como semelhantes as células de cada grupo são uns aos outros usando redução de dimensão. Aqui, outliers distinguem-se facilmente do resto das células. Para analisar como genes individuais são distribuídos entre os clusters, bem como diferem entre si, parcelas de violino são úteis. Na Figura 2, a expressão dos quatro genes representativos são mostrados: o gene de fusão BCR-ABL1, que marca todas as células leucêmicas; o mKI67 de marcador do ciclo celular, que é expresso em um grupo ativamente divisória; o marcador de diferenciação mieloide MPO, que é restrito para o subgrupo de ciclismo; e finalmente a casa mantendo o gene RPS18, que é expressa em todas as células. Finalmente, na Figura 2D as assinaturas moleculares tem sido correlacionadas com immunophenotyped, como as identidades de cada célula em cada cluster são usados para colorir as células individuais em uma trama de FACS gerado a partir do índice de classificação. Mostrada é a expressão do marcador de superfície celular para os marcadores de células-tronco hematopoiéticas CD90 e CD45RA que neste caso poderia ser usado para discriminar entre alguns dos clusters e prospectivamente isolá-los para análise funcional.
A microfluidics usado para executar o qPCR de célula única é muito sensível à introdução de ar ou partículas no sistema. Portanto, é fundamental para fazer uma verificação de qualidade pós execução dos dados. A Figura 3 mostra o representante dados e o contraste entre um sucesso e uma falha executam devido ao carregamento de amostra pobre. A figura 3A mostra como os níveis de Rox, depois de uma bem sucedida atropelar espalhar uniformemente todos os poços que tranquilizar o carregamento tem sido bem sucedida. Em contraste, a Figura 3B ilustra como a amostra e a carga de ensaio falharam como indicado pela falta de ROX em grandes partes do chip. Uma vez que a qualidade de Rox tenha sido avaliada, a qualidade dos sinais de expressão de gene cru pode ser avaliada. Na Figura 3, é apresentado um mapa de calor de uma execução bem sucedida, onde expressão é uniformemente distribuída do amostras com sinal forte ao redor 7-25 Ct:s. em contraste, não figura 3D mostra um mapa de calor de uma execução falha onde muitas amostras tem nenhum sinal ou expressão de uma quantidade muito limitada de genes. Os resultados na Figura 3D são provavelmente devido a um erro na FACS triagem antes da análise de expressão do gene de célula única, uma vez que muitas células têm sinais de clara expressão enquanto outros faltam expressão completamente. Finalmente, a Figura 3E exibe a curva de amplificação esperado de cravado no controle do RNA com todos os poços, tendo clara expressão de cerca de 10 CT com pouca ou nenhuma variação. Esse controle positivo mede a eficiência de todas as etapas da análise de célula única qPCR. Para garantir que a amplificação é alcance dinâmico, números de telemóvel diferentes são incluídos como controles de linearidade; aqui, 10 - e 20-célula controles são usados. Diferenças entre os controles de linearidade CT devem refletir as diferenças no número de células. Finalmente, a não transcrição reversa (noRT) e os controles negativo de nenhum modelo certifique-se de que não há sinais positivos falsos são detectados de genomic DNA ou contaminações, respectivamente.

Figura 1: visão esquemática do protocolo. As células são coradas, classificados com índice-tipo aplicativo ativado para gravar a expressão do marcador de superfície e lysed para liberar o mRNA. mRNA é posteriormente reverso transcrito de cDNA e amplificado usando amplificação de destino específico. Em seguida, as amostras são carregadas juntamente com iniciadores individuais em um chip microfluídicos, onde o perfil de expressão do gene de cada célula é analisado usando RT-qPCR. Após a coleta de dados, os dados são pré-processados para remover células de baixa qualidade e de cluster é executado para definir populações de células molecularmente distinta. Finalmente, subpopulações molecularmente definidas estão correlacionadas com expressão de marcador de superfície de FACS índice classificando dados de immunophenotypically caracterizam a heterogeneidade das populações. Clique aqui para ver uma versão maior desta figura.
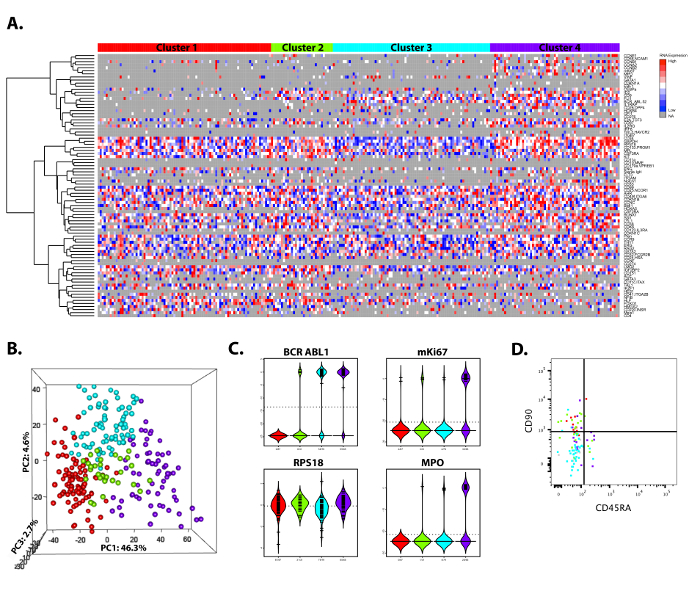
Figura 2: resultados representativos três bem sucedidos célula única da expressão do gene analisa de células-tronco hematopoiéticas normais e leucêmicas. (A) calor mapa mostrando célula única gene medições de expressão de 270 células única de um paciente de leucemia mieloide crônica, bem como um controle saudável correspondência idade analisado por usando SCExV 18, uma ferramenta de análise de cluster hierárquica projetado para este tipo de dados. Vermelho representa alta expressão, expressão de baixo azul e cinza sem expressão. As células foram divididas em quatro grupos diferentes com base em suas assinaturas de expressão do gene. É evidente a partir dos dados que a população de células leucêmicas do paciente pode ser dividida em uma população quiescente (cluster de 3), com expressão de apenas alguns marcadores de diferenciação e uma população ativamente divisória que iniciou a expressão de genes associados com diferenciação mieloide (cluster 4). Os dados utilizados aqui são de um paciente selecionado incluído no trabalho publicado anteriormente por Warfvinge et al . 22 (B) principais (PCA) da análise de componentes dos dados exibidos em A, onde quatro separados grupos pode ser mostrada. (C) violino parcelas de quatro genes com expressão específica do cluster, BCR-ABL1, mKI67, RPS18 e MPO. Cada parcela de violino representa a expressão em cada cluster e a linha pontilhada do outro lado representa o valor médio de expressão para todas as células. (D) análise de ordenação de índice de um dos representados no mapa de calor, onde cada célula é marcada com a cor do seu subpopulação molecular das amostras. Clique aqui para ver uma versão maior desta figura.

Figura 3: controles de qualidade da qPCR multiplex em software PCR em tempo real. (A) Rox carregando imagem de amostra bem sucedida e carregamento do ensaio. (B) Rox carregando imagem de amostra malsucedida e ensaio de carregamento, onde falhadas cargas das amostras são mostradas por uma falta de ROX em linhas horizontais. Falha de carregamento de ensaios iria ser mostrado em linhas verticais. (C) exemplo de um êxito executar na visualização de mapa de calor, onde single-células representam linhas e genes representam colunas. Expressão gênica alta é indicada na expressão de amarelo, baixo em azul e não detectado em preto. (D) exemplo de uma falha executar na visualização de mapa de calor, onde single-células representam linhas e genes representam colunas. Expressão elevada do gene é representado pelo amarelo, baixa expressão é representado pelo azul e nenhuma expressão detectado como preto. (E) normalizado intensidade (à esquerda) e enredo de amplificação (à direita) de um controle cravado no RNA. CT-valores de aproximadamente 10 e curvas com variação limitada indicam sucesso e robusta reverso transcrição, pré-amplificação e qPCR análise dentro de todos os poços. (F) normalizado intensidade (à esquerda) e enredo de amplificação (à direita) de um gene falhou. As intensidades do normalizada não formam uma curva e o enredo de amplificação mostra grandes picos em fluorescência. Clique aqui para ver uma versão maior desta figura.
Discussão
Nos últimos anos, a análise da expressão de gene de célula única tornou-se uma adição valiosa para definir a heterogeneidade de várias populações de nível23. O advento das tecnologias de sequenciamento de RNA teoricamente fornece a possibilidade de medir a transcriptoma inteira de uma célula, no entanto, esses métodos são complicados por variações em profundidades de sequenciamento de célula para célula e abandono. Célula única qPCR oferece uma análise sensível e robusta da expressão de centenas de genes críticos onde todas as células são tratadas da mesma forma, reduzindo o ruído de técnico. A análise focada de um número limitado de transcrições Adicionalmente permite análise simplificada, sem a interferência de genes altamente expressos.
Desde que a abordagem investiga apenas um subconjunto dos genes expressado nas células é altamente importante escolher o conjunto correto de genes ao projetar o painel do gene. A escolha dos genes pode ser feita de várias maneiras; através da literatura, conclusões anteriores por análise em massa e bioinformática de análise de dados publicamente disponíveis. Isto não é trivial; no entanto, com o conjunto correto de genes, a abordagem pode ser muito poderosa. Quando o painel de gene foram projetado, é importante validar que as primeiras demão não interferindo durante a multiplexação. Aqui recomendamos analisar se os primers são lineares por para plástico uma série de diluição com o material amplo; uma diluição de série de primers utilizados neste estudo é mostrado em suplementar Figura 2D em Warfvinge et al . 22
As duas principais limitações do protocolo aqui apresentado são o número limitado de células e genes investigados. No entanto, apesar de apenas 96 células são investigadas em cada corrida, este protocolo relativamente simples pode ser concluído em um dia e, assim, centenas de células podem ser analisadas em uma semana. Além disso, o número de genes investigados poderia ser aumentado se mais primeiras demão são incluídas na etapa de pré-amplificação de destino específico. Neste caso, várias fichas são necessários para investigar cada conjunto de 96 primers.
Se altamente expressa genes ou células com alto teor de RNA-estão sendo analisados, o número de ciclos otimizados para células-tronco hematopoiéticas com baixa geral de expressão de gene23 deve ser modificado. Mudança de ciclos de amplificação também é importante quando investigando em massa das populações. Para análise em massa de células-tronco hematopoiéticas (100 células) recomendamos reduzindo a quantidade de ciclos de Pre-amplificação de 25 a 18.
Os mais comuns razões para falha é executado é devido à classificação sem sucesso de single-células dentro do poço da placa de Lise, classificação de células não-viáveis, suboptimal pré-amplificação ou chip de falha de carregamento. O nível de Rox em cada câmara de reação é um indicador do carregamento da amostra ou do ensaio, e se estas são baixas, é recomendável que as amostras são re-executadas em um chip novo, desde Rox baixo níveis são provavelmente causados pela introdução de bolhas durante o carregamento. Descontrole cravado na expressão do RNA é um indicador de que os pré-amplificadores falharam, provavelmente devido a problemas com pré-amplificação- ou reverter os reagentes de transcrição. Neste caso, é recomendável que um novo chip é executado com células recentemente classificadas e reagentes de novos. Deteção de controle RNA mas nenhuma indicação de outra expressão de gene de sinais é uma indicação de FACS malsucedido de célula única classificação antes da análise de expressão do gene. Para evitar erros durante FACS classificação é importante garantir que o single-células são classificadas para o centro de cada poço. Isso pode ser feito pela classificação de miçangas em um prato vazio e inspecionar visualmente onde o droplet terras. Certifique-se de um tipo de teste em todos os poços nas bordas da placa para garantir que a configuração não é apenas correta para os primeiros poucos poços. Quando aplicável, single-células podem, em paralelo, ser classificadas para o crescimento in vitro e analisaram antes da análise de qPCR para controle para protocolos de classificação bem sucedidos. Informações de ordenação de índice não só permite investigar a immunophenotyped de uma subpopulação molecularmente distinta, mas também podem fornecer introspecções úteis quando solução de problemas, se você está classificando uma população indefinida e muitas células de baixa qualidade são presente na análise da expressão do gene de célula única. As informações de ordenação de índice podem ser usadas para otimizar a estratégia associada e evitar futuras classificação de células de baixa qualidade.
Análise da expressão de gene de célula única está revolucionando a célula como heterogênea populações são investigadas e pavimentando o caminho para a compreensão mais de diferenciação celular. Na hematopoiese, análise da expressão de gene de célula única têm sido utilizados para refinar estratégias de classificação para subconjuntos de linfócitos7, para resolver a heterogeneidade de progenitoras multipotentes populações6,24,25 e identificar a diferenciação de células leucêmicas de terapia a populações22,26. Combinação do índice de classificação com a expressão de gene de célula única - e análise funcional anteriormente foi mostrada para ser confiável e eficiente quando investigando imunofenótipo e heterogeneidade das diferentes populações27, 28,29. Aqui, uma abordagem para investigação molecular e imunofenotípica heterogeneidade de célula populações têm sido descritos onde combinar qPCR célula única com a classificação de índice permite resultados rápidos e podem ser reproduzidos sem análise complicada em um curto período de tempo.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho é apoiado por subsídios da sociedade sueca de câncer, o Conselho Sueco de pesquisa, a sociedade sueca para pesquisa médica, o sueco Childhood Cancer Foundation, a Fundação de Ragnar Söderberg e The Knut e Alice Wallenberg Foundation
Materiais
| Name | Company | Catalog Number | Comments |
| CD14 PECY5 | eBioscience | 15-0149-42 | Clone: 61D3 |
| CD16 PECY5 | Biolegend | 302010 | Clone: 3G8 |
| CD56 PECY5 | Biolegend | 304608 | Clone: MEM-188 |
| CD19 PECY5 | Biolegend | 302210 | Clone: HIB19 |
| CD2 PECY5 | Biolegend | 300210 | Clone: RPA-2.10 |
| CD3 PECY5 | Biolegend | 300310 | Clone: HIT3a |
| CD123 PECY5 | Biolegend | 306008 | Clone: 6H6 |
| CD235A PECY5 | BD Pharma | 559944 | Clone GAR2 |
| CD34 FITC | Biolegend | 343604 | Clone: 561 |
| CD38 APC | Biolegend | 303510 | Clone: Hit2 |
| CD90 PE | Biolegend | 328110 | Clone: 5E10 |
| CD45RA BV421 | BD bioscience | 560362 | Clone: HI100 |
| CD49f Pecy7 | eBioscience | 25-0495-82 | Clone: eBioGOH3 |
| FBS | HyClone | SV30160.3 | |
| PBS | HyClone | SH30028.02 | |
| 96-well u-bottom Plate | VWR | 10861-564 | |
| SFEM | Stem cell technologies | 9650 | |
| Penicillin streptomycin | HyClone | SV30010 | |
| TPO | Peprotech | 300-18 | |
| SCF | Peprotech | 300-07 | |
| FLT3L | Peprotech | 300-19 | |
| Falcon Tube 15 mL | Sarstedt | 62.554.502 | |
| Eppendorph tube | Sarstedt | 72.690.001 | |
| CST beads | BD | 642412 | |
| Accudrop Beads | BD | 345249 | 6 µm particles |
| Adhesive film Clear | Thermo scientific | AB-1170 | |
| Adhesive film Foil | Thermo scientific | AB-0626 | |
| 96 well PCR plate | Axygen | PCR-96M2-HS-C | |
| PCR 1.5 mL tube | Axygen | MCT-150-L-C | |
| T100 PCR cycler | BioRad | 186-1096 | |
| 10% NP40 | Thermo scientific | 85124 | |
| 10 mM dNTP | Takara | 4030 | |
| 0.1 M DTT | Invitrogen | P2325 | |
| RNAsout | Invitrogen | 10777-019 | RNAse inhibitor |
| CellsDirect One-Step qRT-PCR Kit | Invitrogen | 11753-100 | |
| Neuclease free water | Invitrogen | 11753-100 | from CellsDirect kit |
| 2x Reaction Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| SuperScript III RT/Platinum Taq Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| Platinum Taq DNA Polymerase | Invitrogen | 10966026 | |
| TaqMan Cells-to-CT Control Kit | Invitrogen | 4386995 | |
| Xeno RNA Control | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 20x Xeno RNA Control Taqman Gene Expression Assay | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 96.96 Sample/Loading Kit—10 IFCs | Fluidigm | BMK-M10-96.96 | |
| 2x Assay Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| 20x GE Sample Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| Control line fluid | Fluidigm | From 96.96 Sample/Loading Kit | |
| TaqMan Gene Expression Master Mix | Applied Biosystems | 4369016 | |
| BioMark HD | Fluidigm | BMKHD-BMKHD | |
| 96.96 Dynamic Array IFC | Fluidigm | BMK-M10-96.96GT | |
| Excel | Microsoft | Microsoft | |
| FlowJo V10 | TreeStar | TreeStar | |
| Fluidigm real time PCR analysis | Fluidigm | Fluidigm | |
| CD179a.VPREB1 | Thermofisher scientific | Hs00356766_g1 | |
| ACE | Thermofisher scientific | Hs00174179_m1 | |
| AHR | Thermofisher scientific | Hs00169233_m1 | |
| BCR_ABL.52 | Thermofisher scientific | Hs03043652_ft | |
| BCR_ABL41 | Thermofisher scientific | Hs03024541_ft | |
| BMI1 | Thermofisher scientific | Hs00995536_m1 | |
| CCNA2 | Thermofisher scientific | Hs00996788_m1 | |
| CCNB1 | Thermofisher scientific | Hs01030099_m1 | |
| CCNB2 | Thermofisher scientific | Hs01084593_g1 | |
| CCNC | Thermofisher scientific | Hs01029304_m1 | |
| CCNE1 | Thermofisher scientific | Hs01026535_g1 | |
| CCNF | Thermofisher scientific | Hs00171049_m1 | |
| CCR9 | Thermofisher scientific | Hs01890924_s1 | |
| CD10.MME | Thermofisher scientific | Hs00153510_m1 | |
| CD11a | Thermofisher scientific | Hs00158218_m1 | |
| CD11c.ITAX | Thermofisher scientific | Hs00174217_m1 | |
| CD123.IL3RA | Thermofisher scientific | Hs00608141_m1 | |
| CD133.PROM1 | Thermofisher scientific | Hs01009250_m1 | |
| CD151 | Thermofisher scientific | Hs00911635_g1 | |
| CD220.INSR | Thermofisher scientific | Hs00961554_m1 | |
| CD24.HSA | Thermofisher scientific | Hs03044178_g1 | |
| NCOR1 | Thermofisher scientific | Hs01094540_m1 | |
| CD26.DPP4 | Thermofisher scientific | Hs00175210_m1 | |
| CD274 | Thermofisher scientific | Hs01125301_m1 | |
| CD276 | Thermofisher scientific | Hs00987207_m1 | |
| CD32.FCGR2B | Thermofisher scientific | Hs01634996_s1 | |
| CD33 | Thermofisher scientific | Hs01076281_m1 | |
| CD34 | Thermofisher scientific | Hs00990732_m1 | |
| CD344.FZD4 | Thermofisher scientific | Hs00201853_m1 | |
| CD352.SLAMF6 | Thermofisher scientific | Hs01559920_m1 | |
| CD38 | Thermofisher scientific | Hs01120071_m1 | |
| CD4 | Thermofisher scientific | Hs01058407_m1 | |
| CD41.ITGA2B | Thermofisher scientific | Hs01116228_m1 | |
| CD49f.ITGA6 | Thermofisher scientific | Hs01041011_m1 | |
| CD56.NCAM1 | Thermofisher scientific | Hs00941830_m1 | |
| CD9 | Thermofisher scientific | Hs00233521_m1 | |
| CD97 | Thermofisher scientific | Hs00173542_m1 | |
| CD99 | Thermofisher scientific | Hs00908458_m1 | |
| CDK6 | Thermofisher scientific | Hs01026371_m1 | |
| CDKN1A | Thermofisher scientific | Hs00355782_m1 | |
| CDKN1B | Thermofisher scientific | Hs01597588_m1 | |
| CDKN1C | Thermofisher scientific | Hs00175938_m1 | |
| CEBPa | Thermofisher scientific | Hs00269972_s1 | |
| CSF1r | Thermofisher scientific | Hs00911250_m1 | |
| CSF2RA | Thermofisher scientific | Hs00531296_g1 | |
| CSF3RA | Thermofisher scientific | Hs01114427_m1 | |
| E2A.TCF3 | Thermofisher scientific | Hs00413032_m1 | |
| EBF1 | Thermofisher scientific | Hs01092694_m1 | |
| ENG | Thermofisher scientific | Hs00923996_m1 | |
| EPOR | Thermofisher scientific | Hs00959427_m1 | |
| ERG | Thermofisher scientific | Hs01554629_m1 | |
| FLI1 | Thermofisher scientific | Hs00956711_m1 | |
| FLT3 | Thermofisher scientific | Hs00174690_m1 | |
| FOXO1 | Thermofisher scientific | Hs01054576_m1 | |
| GAPDH | Thermofisher scientific | Hs02758991_g1 | |
| GATA1 | Thermofisher scientific | Hs00231112_m1 | |
| GATA2 | Thermofisher scientific | Hs00231119_m1 | |
| GATA3 | Thermofisher scientific | Hs00231122_m1 | |
| GFI1 | Thermofisher scientific | Hs00382207_m1 | |
| HES1 | Thermofisher scientific | Hs01118947_g1 | |
| HLF | Thermofisher scientific | Hs00171406_m1 | |
| HMGA2 | Thermofisher scientific | Hs00171569_m1 | |
| HOXA5 | Thermofisher scientific | Hs00430330_m1 | |
| HOXB4 | Thermofisher scientific | Hs00256884_m1 | |
| ID2 | Thermofisher scientific | Hs04187239_m1 | |
| IGF2BP1 | Thermofisher scientific | Hs00198023_m1 | |
| IGF2BP2 | Thermofisher scientific | Hs01118009_m1 | |
| IKZF1 | Thermofisher scientific | Hs00172991_m1 | |
| IL1RAP | Thermofisher scientific | Hs00895050_m1 | |
| IL2RG | Thermofisher scientific | Hs00953624_m1 | |
| IRF8 | Thermofisher scientific | Hs00175238_m1 | |
| ITGB7 | Thermofisher scientific | Hs01565750_m1 | |
| KIT | Thermofisher scientific | Hs00174029_m1 | |
| Lin28B | Thermofisher scientific | Hs01013729_m1 | |
| LMO2 | Thermofisher scientific | Hs00153473_m1 | |
| LYL1 | Thermofisher scientific | Hs01089802_g1 | |
| Meis1 | Thermofisher scientific | Hs01017441_m1 | |
| mKi67 | Thermofisher scientific | Hs01032443_m1 | |
| MPL | Thermofisher scientific | Hs00180489_m1 | |
| MPO | Thermofisher scientific | Hs00924296_m1 | |
| NFIB | Thermofisher scientific | Hs01029175_m1 | |
| Notch1 | Thermofisher scientific | Hs01062011_m1 | |
| Pten | Thermofisher scientific | Hs02621230_s1 | |
| RAG2 | Thermofisher scientific | Hs01851142_s1 | |
| RPS18 | Thermofisher scientific | Hs01375212_g1 | |
| RUNX1 | Thermofisher scientific | Hs00231079_m1 | |
| Shisa2 | Thermofisher scientific | Hs01590823_m1 | |
| Spi1 | Thermofisher scientific | Hs02786711_m1 | |
| Sterile.IgH | Thermofisher scientific | Hs00378435_m1 | |
| TAL1 | Thermofisher scientific | Hs01097987_m1 | |
| THY1 | Thermofisher scientific | Hs00264235_s1 | |
| Tim.3.HAVCR2 | Thermofisher scientific | Hs00958618_m1 | |
| VWF | Thermofisher scientific | Hs00169795_m1 |
Referências
- Seita, J., Weissman, I. L. Hematopoietic stem cell: Self-renewal versus differentiation. Wiley Interdiscip Rev Syst Biol Med. 2 (6), 640-653 (2010).
- Orkin, S. H., Zon, L. I. Hematopoiesis: An evolving paradigm for stem cell biology. Cell. 132 (4), 631-644 (2008).
- Ye, F., Huang, W., Guo, G. Studying hematopoiesis using single-cell technologies. Journal of Hematology & Oncology. 10, 27 (2017).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nature Cell Biology. 16 (10), 919-927 (2014).
- Wills, Q. F., et al. Single-cell gene expression analysis reveals genetic associations masked in whole-tissue experiments. Nature Biotechnol. 31 (8), 748-752 (2013).
- Velten, L., et al. Human haematopoietic stem cell lineage commitment is a continuous process. Nat Cell Biol. 19 (4), 271-281 (2017).
- Wilson, N. K., Nicola, K., et al. Combined single-cell functional and gene expression analysis resolves heterogeneity within stem cell populations. Cell Stem Cell. 16 (6), 712-724 (2015).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: Advances and future challenges. Nucleic Acids Research. 42 (14), 8845-8860 (2014).
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of Single RNA Transcripts in Situ. Science. 280 (5363), 585-590 (1998).
- Kalisky, T., et al. A brief review of single-cell transcriptomic technologies. Briefings in Functional Genomics. , elx019 (2017).
- Crosetto, N., Bienko, M., van Oudenaarden, A. Spatially resolved transcriptomics and beyond. Nature Reviews Genetics. 16, 57 (2014).
- Bengtsson, M., Ståhlberg, A., Rorsman, P., Kubista, M. Gene expression profiling in single cells from the pancreatic islets of Langerhans reveals lognormal distribution of mRNA levels. Genome Research. 15 (10), 1388-1392 (2005).
- Bengtsson, M., Hemberg, M., Rorsman, P., Ståhlberg, A. Quantification of mRNA in single cells and modelling of RT-qPCR induced noise. BMC Molecular Biology. 9 (1), 63 (2008).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nature Methods. 10, 1096 (2013).
- Tung, P. -. Y., et al. Batch effects and the effective design of single-cell gene expression studies. Scientific Reports. 7, 39921 (2017).
- Teles, J., Enver, T., Pina, C. Single-cell PCR profiling of gene expression in hematopoiesis. Methods in Molecular Biology. , 21-42 (2014).
- Hayashi, T., et al. Single-cell gene profiling of planarian stem cells using fluorescent activated cell sorting and its "index sorting" function for stem cell research. Development, Growth, & Differentiation. 52 (1), 131-144 (2010).
- Lang, S., et al. SCExV: A webtool for the analysis and visualisation of single cell qRT-PCR data. BMC Bioinformatics. 16 (1), 320 (2015).
- de Klein, A., et al. A cellular oncogene is translocated to the Philadelphia chromosome in chronic myelocytic leukaemia. Nature. 300 (5894), 765-767 (1982).
- . indexed-sorting Available from: https://github.com/FlowJo-LLC/indexed-sorting (2016)
- Warfvinge, R., et al. Single-cell molecular analysis defines therapy response and immunophenotype of stem cell subpopulations in CML. Blood. 129 (17), 2384-2394 (2017).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Breton, G., et al. Human dendritic cells (DCs) are derived from distinct circulating precursors that are precommitted to become CD1c+ or CD141+ DCs. The Journal of Experimental Medicine. 213 (13), 2861-2870 (2016).
- Alberti-Servera, L., et al. Single-cell RNA sequencing reveals developmental heterogeneity among early lymphoid progenitors. The EMBO Journal. 36 (24), 3619-3633 (2017).
- Giustacchini, A., et al. Single-cell transcriptomics uncovers distinct molecular signatures of stem cells in chronic myeloid leukemia. Nature Medicine. 23, 692 (2017).
- Hansmann, L., Han, A., Penter, L., Liedtke, M., Davis, M. M. Clonal expansion and interrelatedness of distinct B-lineage compartments in multiple myeloma bone marrow. Cancer Immunology Research. 5 (9), 744-754 (2017).
- Psaila, B., et al. Single-cell profiling of human megakaryocyte-erythroid progenitors identifies distinct megakaryocyte and erythroid differentiation pathways. Genome Biology. 17 (1), 83 (2016).
- Schulte, R., et al. Index sorting resolves heterogeneous murine hematopoietic stem cell populations. Experimental Hematology. 43 (9), 803-811 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados