Method Article
組合せの単一セルは分子を特徴付ける方法・肺浸潤異質性ひと幹と前駆細胞集団
要約
一括遺伝子発現測定クラウド個々 のセル違い異種細胞集団で。ここでは、プロトコルについて述べる方法単一細胞遺伝子発現解析とインデックスの不均一性の線引きによって蛍光活性化セルの並べ替え (FACS) を並べ替えを結合でき、immunophenotypically 分子細胞集団を特徴付けます。
要約
肺浸潤の評価と分子解析長い不均一性を表し、明確な細胞集団を定義に使用されています。FACS は本質的に単一セルの試金、しかし分子解析する前に、標的細胞は、一括で分離されたよく前向き単一セルの解像度を失うこと。単一細胞の遺伝子発現解析は、異種細胞集団の個々 のセルの分子の違いを理解するための手段を提供します。一括細胞分析で異なる細胞型の overrepresentation 結果バイアスと希少な細胞からの信号の咬合の生物学的重要性。FACS インデックス並べ替え結合した単一細胞遺伝子発現解析を活用することにより集団調査できる単一セルの解像度の損失なし中間細胞表面マーカーの発現と細胞もキャプチャされます, 中の評価を有効にします。連続表面マーカー発現の関連性。ここでは、逆のトランスクリプションの単一セルを組み合わせたアプローチについて述べる定量的 PCR (RT qPCR) および FACS インデックスを同時に分子を特徴付ける並べ替えおよび細胞集団内で肺浸潤の不均一性。
単一細胞 RNA 配列方法と対照をなして特定のターゲットの増幅と qPCR を使用少ない落ちこぼれで低豊富な転写産物の堅牢な測定のためにはしない読み取りの細胞間の変動に関連する問題によって混同される中深さ。また、直接インデックス並べ替え単一セル換散には、このメソッドをバッファーにより cDNA を合成して特定のターゲット中古増幅の細胞表面で分子の署名の後派生の相関に関しては同様に 1 つのステップで実行するにはマーカー式。造血の単一細胞を調べるのにも他のセルタイプで正常に使用されている記述方法をしました。
結論としては、記載方法は明確な分子集団のそれに続く分離のためのプロトコルを開発するパネルの可能性は、事前に選択された遺伝子の mRNA 発現の高感度に測定できます。
概要
それぞれ個々 の白血球細胞が幹細胞が最終的に末期特定を運ぶ最終的なエフェクター細胞に分化ますますコミットの中間の前駆細胞の一連の上に頂点を形成、細胞の階層内に存在すると考えられています。生体機能1。幹細胞システムの構成方法に関する知識の多くは前向きの高濃縮幹細胞や前駆細胞様々 な2 異なる造血集団を切り離すことができるため主造血系で発生しています。FACS を並べ替えています。これが分析する機能や分子、主に遺伝子発現プロファイリング3,4を介してこれらの人口の多くはできました。ただし一括集団個体差細胞の遺伝子発現解析は、平均し、5を失った。したがって、異種細胞内細胞の変化を検出する無能を混同するかもしれない重要な生物学的プロセスの私達の理解場合細胞の小さなサブセットは、人口6の推定される生物学的機能の 7。逆に、単一セルの解像度で遺伝子発現シグネチャの調査は、不均一性を表し、セル8のあるサブセットから覆っの影響を回避する可能性を提供しています。
日付 1 細胞遺伝子発現解析のための多くのプロトコルが開発されました。それぞれの方法で、独自の注意事項があります。最も早い方法であった RNA 蛍光その場で交配 (RNA-魚)、一度に転写産物の限られた数を測るが、RNA 局在9,11の調査のことでユニークです。単一または非常に少数の成績証明書も検出する PCR と qPCR 使用初期の方法は、12を開発しました。ただし、これらが最近交換済み qPCR を通して細胞の何百もの細胞 1 個あたりの成績の何百もの式を同時に分析でき、したがって高次元不均一性解析の使用を許可するマイクロ流体法によるあらかじめ決められた遺伝子パネル10,13。最近 RNA シーケンス技術普及して単一細胞解析、これらは理論的にセルの全体のトランスクリプトームを測定することができ、それにより不均一性解析10、に探索的次元を追加14. 多重 qPCR 解析と単一細胞 RNA シーケンスが異なる機能を持っている、従ってメソッドのいずれかを使用するための理論的根拠はターゲット人口のセルの数だけでなく、質問に決まります。高スループットと単一細胞 RNA シーケンスの公平な探索特性と共にセルあたりの低コストは、不明なセルまたは大規模な集団を調査したときに望ましいです。ただし、単一細胞 RNA シーケンスは低豊富で成績証明書が脱落しやすいより頻繁高い豊富な成績証明書のシーケンスに偏っても。これはバイオ情報解析は、しばしば微妙なや技術的ノイズ15に隠された重要なの分子シグナルを明らかにするために高需要を置くかなり複雑なデータにつながることができます。したがって、よ特徴付けられた組織の単一セル qPCR による解析済みプライマー パネルの機能的に重要な遺伝子や分子マーカーの選択することができますの不均一性を決定するための敏感な簡単なアプローチとして、人口。しかし、それは比較するに注意する必要があります単一細胞 RNA シーケンス、セルあたりのコストが単一セル qPCR 法一般的に高くなっています。ここでは、単一セル Rt-qpcr (Teles J.らから変更を組み合わせたアプローチについて述べる16)、FACS インデックスに集団内で分子と肺浸潤の不均質性を特徴付けるために17バイオインフォマティクス解析18を同時に並べ替え。
このアプローチで興味の細胞を染色すると、単一セルは、96 ウェル PCR プレートの換散バッファーに直接 FACS によって並べ替えられます。同時に、細胞表面マーカーの追加セットの表現のレベルは、FACS-並べ替え、インデックス並べ替えと呼ばれますメソッド中に単一セルごとに記録されます。物質分離細胞は増幅されその後、RT qPCR、マイクロ流体プラットフォームを使用して、選択した一連の遺伝子の遺伝子発現分析します。この方法により、個々 のセルのセル表面のマーカー式の並べ替えられた同時と同様シングル セル特性の分子分析です。細胞の分子特定サブセットをインデックス付きの並べ替えられたマーカーの発現に直接マップによって subpopulations は、将来の分離に使用できます特定イムノフェノ タイプにリンクできます。メソッドの説明図 1で一歩一歩。微妙な遺伝子式信号を見えなくことができますそれ以外の場合は無関係の豊富な遺伝子の測定を回避できますので、さらにパネルであらかじめ決められた遺伝子は目標とされた遺伝子の表現のより高い解像度に貢献します。さらに、特定のターゲットの増幅、1 つのステップの逆のトランスクリプションおよび増幅できます低表現された成績証明書、転写因子や非ポリ adenylated Rna のような堅牢な測定。重要なは、qPCR 法19特定の悪性疾患を調査するときに重要である融合蛋白質から mRNA の測定を可能にします。最後に、遺伝子の数は集中を調査した、低ドロップ アウト率と単一細胞 RNA シーケンスより高い次元のメソッドと比較して限られた細胞をこのメソッドを簡単に分析した技術的な差プロトコルに従うことによって実験全体から実行できます、このシンプルで簡単な高感度、高スループットの単一細胞遺伝子発現解析法を作って 3 日以内、分析結果にセルを並べ替えします。
プロトコル
1. 溶解プレートの準備
- 390 μ L ヌクレアーゼ フリー水、10% の 17 μ L を混合することによって、余分な 10% の 96 ウェルの十分な換散バッファーを準備 RNA/DNA 無料ベンチを使用して NP 40 2.8 μ 10 mM dNTP 10 μ L 0.1 M DTT、5.3 μ L RNAse 阻害剤 (材料の表を参照してください)。渦とスピン ・ ダウン。
- 96 ウェル PCR プレートの各ウェルに換散バッファーの 4 μ L を配布し、接着フィルムでプレートをシールします。スピン ・ ダウン チューブ、プレートの下部に液体を収集します。ソーティング (最大 24 時間) まで氷の上プレートを保ちます。
2. 細胞選別の細胞の調製
- (ここで、CD34 濃縮造血幹・前駆細胞) 細胞実験の適切な数を解凍します。1 x 10 の6セルは、コントロールと単一細胞の約 3 つの 96 ウェル プレートを並べ替えに適しています。
- 15 mL の円錐管に解凍のセルを転送し、1 mL FBS すべて 30 を追加 s 8 mL の容量に達するまで。350 x gで 4 ° C で 10 分間遠心することで細胞をスピンし、上清を除去します。
- 8 mL のバッファーを染色に細胞を再懸濁します (PBS 2 %fbs) と 4 ° C で 10 分間 350 × gで遠心し、上清を除去。
- 200 μ L 染色バッファーと削除セル コントロールの汚れのために細胞を再懸濁します。
- マイナス 1 つコントロール (FMOs) 各 fluorophore の 50 のセルの割合を染色して蛍光を作る染色バッファー μ L。この例では 20,000 セルと 6 の遠心管は、FMOSs として使用されます。セルの数が人口調査に応じて調整されることに注意してください。各管に 1 つを除いてサンプル染色のように同じ濃度で全ての抗体を追加します。
- 使用各 fluorophore の 50 μ L バッファーのセルの割合の汚損によって各 fluorophore の単一の汚れを確認します。この例では 20,000 セル 6 遠心管を使用します。各抗体のターゲットがコントロールに使用される細胞によって表現される必要があることに注意してください。個々 のチューブのサンプルの汚れのように同じ濃度で各抗体を追加します。無染色のコントロールとして 50 μ L に 20,000 無染色の細胞をさらにしてください。
- セルのサンプルするには、適切な濃度で抗体を追加します。CD90 PE CD45RA bv421 1/50、1/50 CD49F-PECy7 とリネージュ ミックス 1/10 CD38 APC 1/50、1/100 濃度で CD34 FITC をここでは使用される: CD3 PECy5 1/50、CD2 PECy5 1/50、CD19 PECy5 1/50、CD56 PECy5 1/50、CD123 PECy5 1/50、CD14 PECy5 1/50、CD16 PECy5 1/50 とCD235a PECy5 1/1000。
- 暗闇の中で氷の上で 30 分間抗体と細胞を孵化させなさい。
- 3 ml のバッファーを汚すセルを洗浄します。4 ° C で 10 分間 350 x gで細胞を遠心し、上清を除去します。
- 細胞を再懸濁します、2.9 の手順を繰り返します。
- 500 μ L のサンプルと 100 FMOs を再懸濁します μ L 単一細胞懸濁液を得るために 50 μ m フィルターを通して 1/100 7AAD とフィルターのセルを持つバッファーを染色します。
3. セルの並べ替え
- FACS マシンが設定されていることを確認正しくとドロップ遅延の cytometer セットアップ追跡 (CST) 適切なセルが並べ替えられていることを確認するための製造元の指示に従って実行された最近のアップ。最適なイベント率が 800、2000 年イベント/秒の間は、造血細胞の 4 の 85 ミクロン ノズルと最大速度の使用をお勧めします。
- 蛍光補正を実行することによりスペクトルの重複の修正、FMO コントロールまたは内部のマイナス コントロールによるとゲートを設定します。
- ターゲット人口の再解析を実行するには、染色バッファーの 100 μ L を新しい微量遠心チューブに少なくとも 100 の標的細胞を選別します。FACS は並べ替えられたサンプルを記録することによって、細胞を分析し、彼らは並べ替え門で終わるかどうかを確認します。
- 96 ウェル プレートにも A1 にドロップを中心として並べ替え設定単セル板。それが中央寄せの場合は、すべての井戸で各ウェルの中央のセルを得るが空 96 well プレートのエッジの周りのすべての井戸に 50-100 6 μ m の粒子を並べ替えます。
- 可能であれば、体外で成長の単一セルを並べ替えることにより実行可能なセルが並べ替えられていることを確認する追加のコントロールを追加できます。FLT3L、TPO と SCF、造血細胞を 100 μ l 論文 1% ペニシリン ストレプトマイシン、100 ng/mL で U 下 96年ウェル プレートで栽培できます。顕微鏡を用いた細胞コロニーのため 3 日間の培養後各ウェルを分析します。
- プレートから粘着フィルムを削除します。96 ウェルから 92 への興味 (ここで林-cd 34 + CD38-細胞) の並べ替え 1 つのセルは、各単一セルの (CD90、CD49f、ここ CD45RA) 興味の他のマーカーの肺浸潤プロファイルを保存するソフトウェアを並べ替え FACS でインデックス ソートを有効にします。
- PCR 増幅直線性コントロールのそれぞれ 10 と 20 のセルを持つ 2 つの井戸を並べ替えます。井戸 H1 と H2 は通常使用されます。
- いいえテンプレート コントロール、通常 H3 と H4 の井戸として任意のセルのない 2 つの井戸を保ちます。
- 透明粘着フィルムでプレートをシールし、300 x gスナップ ドライアイスで凍結する前に 1 分でプレートをスピンします。
- 冷凍板-80 ° C で保存します。
注: 安全な停止ポイント。並べ替えや分離細胞は長期保存用-80 ° C で保存できます。
4. 逆転写と特定のターゲットの増幅
- 96 遺伝子のすべてのターゲットのプライマー ミックスを準備するには、1.5 mL RNA/DNA 無料ベンチに RNAse フリー チューブでハウスキーピング遺伝子のプライマーおよびスパイクの制御、RNA のプライマーを含む各プライマーの 2 μ L を追加します。未満 96 プライマーを使用している場合は、不足しているプライマーのヌクレアーゼ フリー水の相当量を追加します。プライマーは、目的遺伝子のパネルに合わせて個別に並べられます。
- 逆のトランスクリプションとミックス 632.5 μ L 2 x 反作用の組合せ、101.2 μ L を追加することによって特定のターゲット増幅 Taq/SuperscriptIII、151.8 μ L プライマー ミックス、およびコントロール RNA のスパイク 0.7 μ L。DNA 無料ベンチ上のこの手順をプリフォームします。ボルテックスし、スピンダウン機能収集管の底で液体をミックス。サンプルに加えまで氷の上を保ちます。
- 2 x 反作用の組合せの 27.5 μ L、Taq の酵素の 1.76 μ L、プライマー ミックスの 6.6 μ L、ヌクレアーゼ フリー水の 2.64 μ L を混合することにより 4 つの井戸を作るない逆のトランスクリプション コントロール ミックス。コントロール RNA をスパイクします。DNA 無料ベンチでこの手順を実行します。渦とダウン サンプルを収集、チューブや追加まで氷の下に液体のスピン。
- 雪解けの氷のライセート プレート。直線性といいえテンプレート コントロールを含む 92 の井戸に事前に準備された逆のトランスクリプションおよび特定のターゲット増幅ミックスの 8.75 μ L を追加します。4 つの残りのウェルにない逆転写制御ミックスの 8.75 μ L を追加します。透明粘着フィルムとプレートの下部に収集の液体までスピン プレートをシールします。
- プリアンプ プログラムに応じて PCR マシンでプレートを実行して逆のトランスクリプションおよび特定のターゲットの増幅をプリフォームします。ステップ 1:50 2:95 ° C を 2 分間ステップ 15 3:95 ° C、60 分の ° C s、ステップ 4:60 ° C で 4 分、繰り返し手順 3-4 24 回し、最後にステップ 5:8 ° C を永遠に。
- PCR が完了したら後、は、8 ° C、短期記憶と長期記憶の-20 ° C でプレートを保ちます。
注: 安全な停止ポイント。短期的なストレージの 8 ° c と長期保存のための-20 ° C で、増幅された材料を保つことができます。
5. 多重マイクロ遺伝子発現解析のためのサンプルとアッセイ プレート準備
- 注 3 によって積載面の試金を準備 96 well プレートの各ウェルに μ L 測定荷重試薬。各プライマーの 3 μ L を積載面分析で個々 の井戸に追加します。
- 粘着フィルム ・ プレートの下部に収集の液体までスピン プレートをシールします。
- 96 well プレートのすべてのウェルに無料水ヌクレアーゼの 8 μ L 分注、希釈プレートを準備します。1:5 の最終希釈を作る希釈プレートに増幅されたサンプルの 2 μ L を追加します。
- シールの粘着フィルム、プレート、10 のボルテックス板によるミックス s と最後にプレートの下部に収集の液体までスピンします。
- 慎重にサンプル読み込み試薬の 35.2 μ L でのマスターの組合せの 352 μ L を混合することによってサンプル読み込みミックスを準備します。96 well プレートの各ウェルにミックスの読み込みの早かった 3.3 μ L、サンプル板を準備します。
- 試料プレートの各ウェルに、希釈したサンプルから 2.7 μ L を追加します。
- 粘着フィルム ・ プレートの下部に収集の液体までスピン プレートをシールします。
6. マイクロ流体チップの読み込み
- 新しい 96 x 96 マイクロ流体チップを取り出してください。彼らを移動できることを確認するためにキャップのついた注射器で彼らを突き、入り江を準備します。
- 注射器から泡を削除します。チップを 45 度傾けるとバルブを押しながら各バルブにシリンジのフル ・ ボリュームを追加します。IFC コント ローラー チップを首相します。
- 積載面試金で井戸のそれぞれから 4.25 μ L で各分析の入口をロードします。泡を避けるため。井戸の中に泡が表示される場合、は、ピペット チップを削除します。
- 積載面サンプルでは井戸の各から 4.25 μ L で各サンプル入口の読み込みを続行、泡を避けるため、泡が表示される場合は、ピペット チップとそれらを削除します。
- IFC のコント ローラー チップを読み込みます。
- チップにも見えることと、すべての部屋が読み込まれたことを確認します。テープでそれに触れることにより、チップ表面からほこりを削除します。マイクロ多重遺伝子式プラットフォームでチップを実行します。
7. マルチプレックス マイクロ遺伝子式プラットフォームでチップを実行します。
- マイクロ多重遺伝子式プラットフォームにチップをロードした後、サンプルを名前します。
- 受動的な染料として ROX を設定します。蛍光として単一のプローブと FAM MGB を設定します。96 x 96 標準 v2 をプロトコルとして使用します。実行を開始します。
- 実行すると削除チップは完了です。
8 チップの予備的分析
- リアルタイム PCR 解析ソフトウェアにデータを読み込みます。
- タブ区切りのファイルから細胞、遺伝子のレイアウトを貼り付けることで、遺伝子名とセル名を読み込みます。
- イメージ ビューを開き、染料として ROX を選択します。チェックすべての井戸が ROX 受動的な色素を持っているかどうか。
- すべて増幅プロット見て ok、スパイク (図 3 eに類似) で滑らかな増幅曲線を場合を調査します。
- スパイクのコントロールすべてが正常に読み込まれたことを確認する RNA の発現があるすべての単一セルを確認します。
- すべてのセル ハウスキーピング遺伝子発現がある、従って正しく並べ替えられていることを確認します。
- 10 と 20 のセル直線コントロールがある線形増幅を検証する約 1 CT 違いを確認します。
- 水資源コントロールのサンプル式があるかどうかはチェックしてください。水資源の式が検出された場合は、以降の実行のゲノム DNA を検出するプローブにプローブを変更してみます。
- さらに分析は、csv ファイルのデータをエクスポートします。
9 SCExV を使用して単一細胞解析
注: 入門映画は現在20ツールを紹介します。ここでは、プロトコルに導入されたコントロールを使用して分析を行う方法の短い勧告が表示されます。
- 20SCexV のウェブサイトに接続します。
- エクスポートした CSV ファイルをアップロードします。
- 肯定的な制御としてスパイクのコントロール RNA を選んだ。25 上コントロール RNA CT のあるセルを削除します。中央コントロール RNA 発現データを正規化します。
- "分析を -> 実行ここで"をクリックします。セルのオプションを除外で水資源、notemplate、10 と 20 のセル コントロールを削除します。
- クラスター数の期待値で任意のクラスタ リング手法とクラスターのセルです。分析値をエクスポートします。
10. インデックス ソートの分析
- FACS 解析ソフトウェアを開き、インデックスのサンプルをロードします。
- スクリプト エディターを開き、j ・ クインらから利用可能なスクリプトを実行21
- 今では FACS 解析ソフトウェアは、単一のセルの各ゲートをしなければならないで、レイアウト エディターを開き、SCexV ("Sample_complete_Data.xls"という名前のファイルで利用可能) からグループ化による細胞の色します。
結果
説明されたプロトコルは、簡単に実行、迅速かつ信頼性の高いです。実験のセットアップの概要については、図 1にされます。特定のターゲットの増幅、遺伝子発現測定および予備的な分析に、単一セルの並べ替えから、プロトコル全体は、3 日間で実行できます。96 プライマーといずれかの慢性骨髄性白血病 (CML) 患者から 96 セルまたは健康な高齢者一致から 96 セルを用いた単一細胞遺伝子発現解析からの予備的な分析データを表す熱マップの形で解析結果の例コントロールは、図 2で示されます。階層的クラスタ リングを使用して、分析された細胞は、その遺伝子発現シグネチャに基づいて 4 つのサブグループに分けることが。熱マップの視覚エフェクトは、解析 (制御井戸など) から除外すべき井戸のコントロールだけでなく、データの概要を取得する便利な方法です。図 2 bは各グループの細胞が次元縮約を使用して互いに類似の可視化主成分分析 (PCA)。ここでは、外れ値がセルの残りの部分から容易に識別します。分析するバイオリン プロットが役にどのように個々 の遺伝子クラスターに分散だけでなく、それらの間で異なります。図 2、4 つの代表的な遺伝子の発現が表示されます: 融合遺伝子 BCR ABL1、マークすべての白血病細胞;積極的に分割、グループで表現される細胞周期のマーカー mKI67骨髄性の分化マーカー MPO サイクリング サブグループに制限されています最後に家を保つ遺伝子 RPS18 は、すべてのセルで表現されます。最後に、図 2 D immunophenotyped に関連した分子シグネチャ、各クラスター内の各セルの id として使ってインデックス並べ替えから生成された FACS プロットの個々 のセルの色します。CD90 とここでいくつかのクラスターの間で区別し、前向きの機能解析、それらを分離する使用できる CD45RA 造血幹細胞マーカーの細胞表面マーカーの発現を示します。
単一セルの qPCR の実行に使用するマイクロ流体は空気または粒子のシステムへの導入に非常に敏感です。したがって、データのポスト実行の品質チェックを行うことが重要です。データと成功と失敗の間コントラスト悪いサンプル負荷による実行代表を図 3に示します。図 3 aは、Rox レベル成功実行後広がり均等に安心すべての井戸で読み込みが成功している方法を示しています。対照的に、図 3 bは、チップの大部分で ROX の欠乏によって示されているように、サンプルとアッセイの読み込みが失敗した方法を示しています。Rox の品質が評価されて、生遺伝子式信号の品質を評価できます。成功した実行のヒート マップの提示図 3で、周りの強力な信号のサンプルのに均等に式を分散 7-25 Ct:s. でコントラスト、図 3 Dマップを示しています熱失敗した実行の多くのサンプルがある信号がないまたは遺伝子の非常に限られた量の式。図 3 Dの結果が多くの細胞表現に完全に欠けて他人ながら明確な表現の信号を持っているので単一細胞遺伝子発現の分析の前に FACS 並べ替えのエラー可能性があります。最後に、図 3Eほとんど差のない約 10 CT の明確な表現を持つすべての井戸でスパイク コントロール RNA の予想される増幅曲線を表示します。この肯定的な制御は、単一セルの qPCR の分析のすべてのステップの効率を測定します。増幅がダイナミック レンジ内にあるためには、別のセル番号が直線コントロールとして含まれるここでは、10 と 20 のセル コントロールを使用します。直線コントロールの CT の違いは、細胞数の違いを反映してください。最後に、ないのテンプレートの陰性対照およびない逆のトランスクリプション (水資源) は、偽の肯定的な信号が検出されないことからゲノム DNA や汚染、それぞれを確認します。

図 1: プロトコルの概略図。細胞が染色、mRNA を解放するアプリケーション表面マーカー式を記録するために活性化され、分離をインデックス並べ替えで並べ替え。cDNA に転写され、特定のターゲットの増幅を使用して増幅された mRNA はその後逆です。次に、サンプルはマイクロ流体チップに個々 のプライマーと一緒に読み込まれます Rt-qpcr を用いて各セルの遺伝子発現プロファイルが。データ収集後のデータはあらかじめ処理された低品質のセルを削除して分子細胞集団を定義するクラスタ リングが実行されます。最後に、分子定義された集団は、FACS インデックス並べ替えから表面マーカーの発現に相関データ immunophenotypically を特徴付ける人口の不均一性。この図の拡大版を表示するのにはここをクリックしてください。
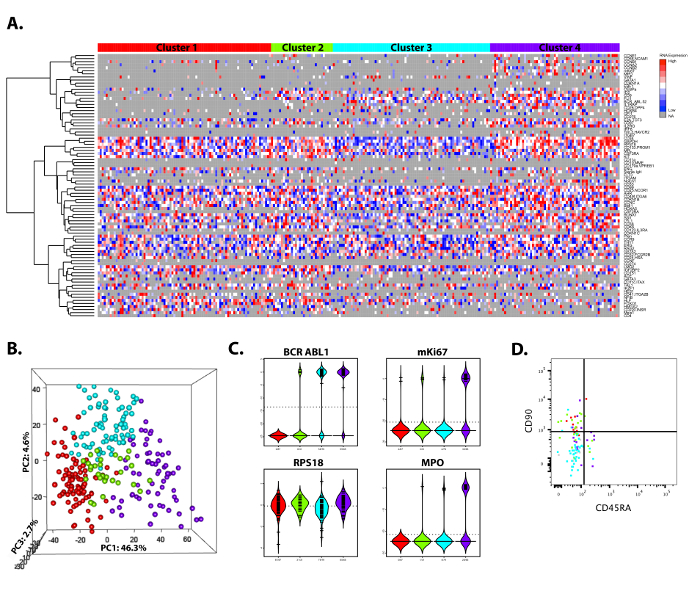
図 2: 通常、白血病の造血幹細胞の 3 つの成功した単一細胞遺伝子発現の代表の結果を分析します。(A) 熱マップ表示の単一細胞遺伝子 SCExV 18、分析ツールを用いた階層的クラスタ リングによって分析、年齢をマッチさせた健康な制御と同様、一人の慢性骨髄性白血病患者から 270 の単一セルの式測定このタイプのデータのために設計されています。赤、青の低発現の高い表現を表し、灰色のない式。細胞は、その遺伝子発現シグネチャに基づいて 4 つの異なるクラスターに分けられました。1 静止人口 (クラスター 3)、のみいくつかの分化マーカーの発現との式を開始している 1 つの積極的に分割人口この患者から白血病幹細胞の人口を分けることができますデータから明らかです。(クラスター 4) 骨髄分化に関連する遺伝子。ここで使用されるデータは、Warfvingeらで以前に公開された作品に含まれて選択した患者から22 (B) プリンシパル グループを区切って 4 A に表示されるデータの主成分分析 (PCA) を示すことができます。(C) バイオリン プロット クラスター特定の表現と 4 つの遺伝子の BCR ABL1、mKI67、RPS18 と MPO。各バイオリン プロットは各クラスター内の式を表し、点線で式のすべてのセルの平均値。(D) 個々 のセルはその分子集団の色でマーク、ヒート マップで表される患者のサンプルの 1 つのインデックスの並べ替えの分析。この図の拡大版を表示するのにはここをクリックしてください。

図 3: リアルタイム PCR ソフトウェアで多重の qPCR の品質コントロール。(A) 成功したサンプルと試験荷重の Rox 読み込みイメージ。(B) Rox に失敗したサンプルと載荷試験のイメージを読み込み、どこのサンプルの失敗した荷重は、水平線で ROX の不足によって示されます。試金のロードの失敗が垂直線で表示されます。成功の (C) の例は、ヒート マップ ビュー、単一セルを表す行、遺伝子を表す列で実行します。遺伝子の高発現は青で黄色、低発現と黒で検出された式が示されます。失敗の例を (D) は、ヒート マップ ビュー、単一セルを表す行、列を表す遺伝子で実行します。遺伝子の高発現が表される黄色、低式は青と黒として検出された式が表されます。(左) (E) 正規化された強度とスパイクの RNA 制御の増幅プロット (右)。約 10 と限られた変化曲線の ct 値は、成功と堅牢なリバース転写、中古増幅と qPCR 分析すべての井戸の内でを示します。(F) 正規化 (左) 強度と失敗した遺伝子の増幅プロット (右)。正規化された強度曲線を形成しないし、増幅プロットは蛍光性の大きなスパイクを示しています。この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
近年では、1 細胞遺伝子発現解析は様々 な細胞集団23の不均一性を定義する貴重な追加になりました。RNA シーケンス テクノロジーの出現は、しかし、これらのメソッドは、セルのセル配列の深さやドロップ アウトの変化によって複雑な理論的にセルの全体のトランスクリプトームを測定する可能性を提供します。何百ものすべてのセルが重要な遺伝子の発現の敏感でロバストな解析は技術的な騒音低減に同様に、扱われる単一セル qPCR を提供します。成績証明書の限られた数の焦点を絞った分析また高発現遺伝子から干渉されることがなく簡易解析できます。
アプローチのみ調査する細胞で発現する遺伝子のサブセットから高い遺伝子パネルをデザインするときに、適切な遺伝子のセットを選択することが重要です。遺伝子の選択は、いくつかの方法で行うことができます。文学、一括解析と公開されているデータのバイオインフォマティクス解析による前の調査結果。これに足らない;しかし、遺伝子の正しいセット アプローチは非常に強力することができます。遺伝子パネルに設計されている、プライマーが多重化中に妨害していないことを検証することが重要です。ここでお勧めします分析線形のプライマーが十分な材料の希釈系列を予備成形による場合補足のように本研究で使用されるプライマーの系列希釈 Warfvingeらの 2 D を図します。22
ここで提示されたプロトコルの 2 つの主要な制限は、細胞、遺伝子調査の限られた数です。しかし、にもかかわらず、各走行の 96 だけ細胞を調べたこの比較的単純なプロトコルは 1 日で完了することができます、従って何百もの細胞を 1 週間に分析できます。さらより多くのプライマーはターゲット固有中古増幅ステップに含まれている場合、調査した遺伝子の数は高めることができます。この場合、96 プライマーの各セットを調査するいくつかのチップが必要です。
遺伝子や細胞に高発現 RNA 含量を分析している、低い全体的な遺伝子表現23の造血幹細胞に最適のサイクル数を変更する必要があります。一括集団を調査するとき、重要の拡大のサイクルを変更することです。造血幹細胞 (100) のバルク分析 25 から 18 に中古増幅サイクル数を削減することをお勧めします。
最も一般的な理由は失敗した実行は換散プレートのウェルに単一セルの並べ替え失敗したため、非実行可能なセル、最適の中古増幅またはロード障害チップの並べ替え。各チャンバーの Rox レベルはサンプルまたはアッセイのいずれかの読み込みのインジケーター、これらが低い場合は、サンプルが新しいチップでは、レベルがロード中に気泡の導入によって引き起こされた可能性が高い低ロックスから再実行をお勧めします。スパイクの RNA 発現のコントロールの欠如はそのインジケーター失敗した中古アンプを中古増幅との問題が原因の可能性が高い-転写試薬に戻したり。この場合、新しいチップを新しく細胞と新しい反応を実行することをお勧めします。コントロール RNA の検出が他の遺伝子発現の兆候は失敗した単一細胞の FACS を示す遺伝子発現解析の前に並べ替えを通知します。FACS のそれの並べ替え中にエラーを回避するには、各井戸の中心に単一セルが並べ替えられていることを確認することが重要です。これは空のプレートにビーズを並べ替えと液滴が土地を目視で行うことができます。セットアップが最初のいくつかの井戸のために正しいのみれないように板のエッジのすべての井戸にテスト ソートしてください。該当する場合、単一セル並列の体外発育の並べ替え可能し、前に成功した並べ替えプロトコル制御する qPCR 分析を分析します。インデックス並べ替え情報分子の異なる亜種の immunophenotyped を調査するためだけことはできませんが、未定義の人口を並べ替えは、多くの低品質の電池をトラブルシューティングする場合、有用な洞察力を提供することも単一細胞の遺伝子発現解析に存在します。ゲートの戦略を最適化し、将来の低品質のセルの並べ替えを避けるインデックス並べ替え情報を使用できます。
単一細胞の遺伝子発現解析は異種細胞集団を調査に革命を起こす、細胞分化のさらなる理解のための道を舗装します。造血系で単一細胞遺伝子発現解析は、多能性前駆細胞集団6,24,25の不均一性を解決する HSCs7のサブセットの並べ替えの戦略を絞り込むに使用されています。、治療に依存しない白血病幹細胞集団22,26を識別します。単一細胞遺伝子発現 - と機能解析の両方の並べ替えインデックスの組み合わせは、以前イムノフェノ タイプと異なる個体群27,の不均一性を調査する場合に、信頼性と効率性に示されている28,29。ここでは、分子の調査のためのアプローチと集団がされている細胞の肺浸潤を不均一性説明どこインデックス ソートと単一セル qPCR を組み合わせることにより、短期間で複雑な分析をせず高速かつ再現性のある結果時間枠。
開示事項
著者が明らかに何もありません。
謝辞
この仕事、医学研究、スウェーデン小児がん財団、ラグナル協会財団と、クヌートとアリス バレンベリー財団でスウェーデンの癌協会、スウェーデン研究評議会、スウェーデン会からの助成金によってサポートします。
資料
| Name | Company | Catalog Number | Comments |
| CD14 PECY5 | eBioscience | 15-0149-42 | Clone: 61D3 |
| CD16 PECY5 | Biolegend | 302010 | Clone: 3G8 |
| CD56 PECY5 | Biolegend | 304608 | Clone: MEM-188 |
| CD19 PECY5 | Biolegend | 302210 | Clone: HIB19 |
| CD2 PECY5 | Biolegend | 300210 | Clone: RPA-2.10 |
| CD3 PECY5 | Biolegend | 300310 | Clone: HIT3a |
| CD123 PECY5 | Biolegend | 306008 | Clone: 6H6 |
| CD235A PECY5 | BD Pharma | 559944 | Clone GAR2 |
| CD34 FITC | Biolegend | 343604 | Clone: 561 |
| CD38 APC | Biolegend | 303510 | Clone: Hit2 |
| CD90 PE | Biolegend | 328110 | Clone: 5E10 |
| CD45RA BV421 | BD bioscience | 560362 | Clone: HI100 |
| CD49f Pecy7 | eBioscience | 25-0495-82 | Clone: eBioGOH3 |
| FBS | HyClone | SV30160.3 | |
| PBS | HyClone | SH30028.02 | |
| 96-well u-bottom Plate | VWR | 10861-564 | |
| SFEM | Stem cell technologies | 9650 | |
| Penicillin streptomycin | HyClone | SV30010 | |
| TPO | Peprotech | 300-18 | |
| SCF | Peprotech | 300-07 | |
| FLT3L | Peprotech | 300-19 | |
| Falcon Tube 15 mL | Sarstedt | 62.554.502 | |
| Eppendorph tube | Sarstedt | 72.690.001 | |
| CST beads | BD | 642412 | |
| Accudrop Beads | BD | 345249 | 6 µm particles |
| Adhesive film Clear | Thermo scientific | AB-1170 | |
| Adhesive film Foil | Thermo scientific | AB-0626 | |
| 96 well PCR plate | Axygen | PCR-96M2-HS-C | |
| PCR 1.5 mL tube | Axygen | MCT-150-L-C | |
| T100 PCR cycler | BioRad | 186-1096 | |
| 10% NP40 | Thermo scientific | 85124 | |
| 10 mM dNTP | Takara | 4030 | |
| 0.1 M DTT | Invitrogen | P2325 | |
| RNAsout | Invitrogen | 10777-019 | RNAse inhibitor |
| CellsDirect One-Step qRT-PCR Kit | Invitrogen | 11753-100 | |
| Neuclease free water | Invitrogen | 11753-100 | from CellsDirect kit |
| 2x Reaction Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| SuperScript III RT/Platinum Taq Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| Platinum Taq DNA Polymerase | Invitrogen | 10966026 | |
| TaqMan Cells-to-CT Control Kit | Invitrogen | 4386995 | |
| Xeno RNA Control | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 20x Xeno RNA Control Taqman Gene Expression Assay | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 96.96 Sample/Loading Kit—10 IFCs | Fluidigm | BMK-M10-96.96 | |
| 2x Assay Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| 20x GE Sample Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| Control line fluid | Fluidigm | From 96.96 Sample/Loading Kit | |
| TaqMan Gene Expression Master Mix | Applied Biosystems | 4369016 | |
| BioMark HD | Fluidigm | BMKHD-BMKHD | |
| 96.96 Dynamic Array IFC | Fluidigm | BMK-M10-96.96GT | |
| Excel | Microsoft | Microsoft | |
| FlowJo V10 | TreeStar | TreeStar | |
| Fluidigm real time PCR analysis | Fluidigm | Fluidigm | |
| CD179a.VPREB1 | Thermofisher scientific | Hs00356766_g1 | |
| ACE | Thermofisher scientific | Hs00174179_m1 | |
| AHR | Thermofisher scientific | Hs00169233_m1 | |
| BCR_ABL.52 | Thermofisher scientific | Hs03043652_ft | |
| BCR_ABL41 | Thermofisher scientific | Hs03024541_ft | |
| BMI1 | Thermofisher scientific | Hs00995536_m1 | |
| CCNA2 | Thermofisher scientific | Hs00996788_m1 | |
| CCNB1 | Thermofisher scientific | Hs01030099_m1 | |
| CCNB2 | Thermofisher scientific | Hs01084593_g1 | |
| CCNC | Thermofisher scientific | Hs01029304_m1 | |
| CCNE1 | Thermofisher scientific | Hs01026535_g1 | |
| CCNF | Thermofisher scientific | Hs00171049_m1 | |
| CCR9 | Thermofisher scientific | Hs01890924_s1 | |
| CD10.MME | Thermofisher scientific | Hs00153510_m1 | |
| CD11a | Thermofisher scientific | Hs00158218_m1 | |
| CD11c.ITAX | Thermofisher scientific | Hs00174217_m1 | |
| CD123.IL3RA | Thermofisher scientific | Hs00608141_m1 | |
| CD133.PROM1 | Thermofisher scientific | Hs01009250_m1 | |
| CD151 | Thermofisher scientific | Hs00911635_g1 | |
| CD220.INSR | Thermofisher scientific | Hs00961554_m1 | |
| CD24.HSA | Thermofisher scientific | Hs03044178_g1 | |
| NCOR1 | Thermofisher scientific | Hs01094540_m1 | |
| CD26.DPP4 | Thermofisher scientific | Hs00175210_m1 | |
| CD274 | Thermofisher scientific | Hs01125301_m1 | |
| CD276 | Thermofisher scientific | Hs00987207_m1 | |
| CD32.FCGR2B | Thermofisher scientific | Hs01634996_s1 | |
| CD33 | Thermofisher scientific | Hs01076281_m1 | |
| CD34 | Thermofisher scientific | Hs00990732_m1 | |
| CD344.FZD4 | Thermofisher scientific | Hs00201853_m1 | |
| CD352.SLAMF6 | Thermofisher scientific | Hs01559920_m1 | |
| CD38 | Thermofisher scientific | Hs01120071_m1 | |
| CD4 | Thermofisher scientific | Hs01058407_m1 | |
| CD41.ITGA2B | Thermofisher scientific | Hs01116228_m1 | |
| CD49f.ITGA6 | Thermofisher scientific | Hs01041011_m1 | |
| CD56.NCAM1 | Thermofisher scientific | Hs00941830_m1 | |
| CD9 | Thermofisher scientific | Hs00233521_m1 | |
| CD97 | Thermofisher scientific | Hs00173542_m1 | |
| CD99 | Thermofisher scientific | Hs00908458_m1 | |
| CDK6 | Thermofisher scientific | Hs01026371_m1 | |
| CDKN1A | Thermofisher scientific | Hs00355782_m1 | |
| CDKN1B | Thermofisher scientific | Hs01597588_m1 | |
| CDKN1C | Thermofisher scientific | Hs00175938_m1 | |
| CEBPa | Thermofisher scientific | Hs00269972_s1 | |
| CSF1r | Thermofisher scientific | Hs00911250_m1 | |
| CSF2RA | Thermofisher scientific | Hs00531296_g1 | |
| CSF3RA | Thermofisher scientific | Hs01114427_m1 | |
| E2A.TCF3 | Thermofisher scientific | Hs00413032_m1 | |
| EBF1 | Thermofisher scientific | Hs01092694_m1 | |
| ENG | Thermofisher scientific | Hs00923996_m1 | |
| EPOR | Thermofisher scientific | Hs00959427_m1 | |
| ERG | Thermofisher scientific | Hs01554629_m1 | |
| FLI1 | Thermofisher scientific | Hs00956711_m1 | |
| FLT3 | Thermofisher scientific | Hs00174690_m1 | |
| FOXO1 | Thermofisher scientific | Hs01054576_m1 | |
| GAPDH | Thermofisher scientific | Hs02758991_g1 | |
| GATA1 | Thermofisher scientific | Hs00231112_m1 | |
| GATA2 | Thermofisher scientific | Hs00231119_m1 | |
| GATA3 | Thermofisher scientific | Hs00231122_m1 | |
| GFI1 | Thermofisher scientific | Hs00382207_m1 | |
| HES1 | Thermofisher scientific | Hs01118947_g1 | |
| HLF | Thermofisher scientific | Hs00171406_m1 | |
| HMGA2 | Thermofisher scientific | Hs00171569_m1 | |
| HOXA5 | Thermofisher scientific | Hs00430330_m1 | |
| HOXB4 | Thermofisher scientific | Hs00256884_m1 | |
| ID2 | Thermofisher scientific | Hs04187239_m1 | |
| IGF2BP1 | Thermofisher scientific | Hs00198023_m1 | |
| IGF2BP2 | Thermofisher scientific | Hs01118009_m1 | |
| IKZF1 | Thermofisher scientific | Hs00172991_m1 | |
| IL1RAP | Thermofisher scientific | Hs00895050_m1 | |
| IL2RG | Thermofisher scientific | Hs00953624_m1 | |
| IRF8 | Thermofisher scientific | Hs00175238_m1 | |
| ITGB7 | Thermofisher scientific | Hs01565750_m1 | |
| KIT | Thermofisher scientific | Hs00174029_m1 | |
| Lin28B | Thermofisher scientific | Hs01013729_m1 | |
| LMO2 | Thermofisher scientific | Hs00153473_m1 | |
| LYL1 | Thermofisher scientific | Hs01089802_g1 | |
| Meis1 | Thermofisher scientific | Hs01017441_m1 | |
| mKi67 | Thermofisher scientific | Hs01032443_m1 | |
| MPL | Thermofisher scientific | Hs00180489_m1 | |
| MPO | Thermofisher scientific | Hs00924296_m1 | |
| NFIB | Thermofisher scientific | Hs01029175_m1 | |
| Notch1 | Thermofisher scientific | Hs01062011_m1 | |
| Pten | Thermofisher scientific | Hs02621230_s1 | |
| RAG2 | Thermofisher scientific | Hs01851142_s1 | |
| RPS18 | Thermofisher scientific | Hs01375212_g1 | |
| RUNX1 | Thermofisher scientific | Hs00231079_m1 | |
| Shisa2 | Thermofisher scientific | Hs01590823_m1 | |
| Spi1 | Thermofisher scientific | Hs02786711_m1 | |
| Sterile.IgH | Thermofisher scientific | Hs00378435_m1 | |
| TAL1 | Thermofisher scientific | Hs01097987_m1 | |
| THY1 | Thermofisher scientific | Hs00264235_s1 | |
| Tim.3.HAVCR2 | Thermofisher scientific | Hs00958618_m1 | |
| VWF | Thermofisher scientific | Hs00169795_m1 |
参考文献
- Seita, J., Weissman, I. L. Hematopoietic stem cell: Self-renewal versus differentiation. Wiley Interdiscip Rev Syst Biol Med. 2 (6), 640-653 (2010).
- Orkin, S. H., Zon, L. I. Hematopoiesis: An evolving paradigm for stem cell biology. Cell. 132 (4), 631-644 (2008).
- Ye, F., Huang, W., Guo, G. Studying hematopoiesis using single-cell technologies. Journal of Hematology & Oncology. 10, 27 (2017).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nature Cell Biology. 16 (10), 919-927 (2014).
- Wills, Q. F., et al. Single-cell gene expression analysis reveals genetic associations masked in whole-tissue experiments. Nature Biotechnol. 31 (8), 748-752 (2013).
- Velten, L., et al. Human haematopoietic stem cell lineage commitment is a continuous process. Nat Cell Biol. 19 (4), 271-281 (2017).
- Wilson, N. K., Nicola, K., et al. Combined single-cell functional and gene expression analysis resolves heterogeneity within stem cell populations. Cell Stem Cell. 16 (6), 712-724 (2015).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: Advances and future challenges. Nucleic Acids Research. 42 (14), 8845-8860 (2014).
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of Single RNA Transcripts in Situ. Science. 280 (5363), 585-590 (1998).
- Kalisky, T., et al. A brief review of single-cell transcriptomic technologies. Briefings in Functional Genomics. , elx019 (2017).
- Crosetto, N., Bienko, M., van Oudenaarden, A. Spatially resolved transcriptomics and beyond. Nature Reviews Genetics. 16, 57 (2014).
- Bengtsson, M., Ståhlberg, A., Rorsman, P., Kubista, M. Gene expression profiling in single cells from the pancreatic islets of Langerhans reveals lognormal distribution of mRNA levels. Genome Research. 15 (10), 1388-1392 (2005).
- Bengtsson, M., Hemberg, M., Rorsman, P., Ståhlberg, A. Quantification of mRNA in single cells and modelling of RT-qPCR induced noise. BMC Molecular Biology. 9 (1), 63 (2008).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nature Methods. 10, 1096 (2013).
- Tung, P. -. Y., et al. Batch effects and the effective design of single-cell gene expression studies. Scientific Reports. 7, 39921 (2017).
- Teles, J., Enver, T., Pina, C. Single-cell PCR profiling of gene expression in hematopoiesis. Methods in Molecular Biology. , 21-42 (2014).
- Hayashi, T., et al. Single-cell gene profiling of planarian stem cells using fluorescent activated cell sorting and its "index sorting" function for stem cell research. Development, Growth, & Differentiation. 52 (1), 131-144 (2010).
- Lang, S., et al. SCExV: A webtool for the analysis and visualisation of single cell qRT-PCR data. BMC Bioinformatics. 16 (1), 320 (2015).
- de Klein, A., et al. A cellular oncogene is translocated to the Philadelphia chromosome in chronic myelocytic leukaemia. Nature. 300 (5894), 765-767 (1982).
- . indexed-sorting Available from: https://github.com/FlowJo-LLC/indexed-sorting (2016)
- Warfvinge, R., et al. Single-cell molecular analysis defines therapy response and immunophenotype of stem cell subpopulations in CML. Blood. 129 (17), 2384-2394 (2017).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Breton, G., et al. Human dendritic cells (DCs) are derived from distinct circulating precursors that are precommitted to become CD1c+ or CD141+ DCs. The Journal of Experimental Medicine. 213 (13), 2861-2870 (2016).
- Alberti-Servera, L., et al. Single-cell RNA sequencing reveals developmental heterogeneity among early lymphoid progenitors. The EMBO Journal. 36 (24), 3619-3633 (2017).
- Giustacchini, A., et al. Single-cell transcriptomics uncovers distinct molecular signatures of stem cells in chronic myeloid leukemia. Nature Medicine. 23, 692 (2017).
- Hansmann, L., Han, A., Penter, L., Liedtke, M., Davis, M. M. Clonal expansion and interrelatedness of distinct B-lineage compartments in multiple myeloma bone marrow. Cancer Immunology Research. 5 (9), 744-754 (2017).
- Psaila, B., et al. Single-cell profiling of human megakaryocyte-erythroid progenitors identifies distinct megakaryocyte and erythroid differentiation pathways. Genome Biology. 17 (1), 83 (2016).
- Schulte, R., et al. Index sorting resolves heterogeneous murine hematopoietic stem cell populations. Experimental Hematology. 43 (9), 803-811 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved