Method Article
Una combinatoria unicelular enfoque para caracterizar Molecular y heterogeneidad de Immunophenotypic de madre humana y de las poblaciones progenitoras
En este artículo
Resumen
A granel gene expresión medidas nube célula individual diferencias en poblaciones celulares heterogéneas. Aquí, describimos un protocolo para análisis de expresión génica e índice cómo sola célula clasificación de Florescence activa célula clasificación (FACS) puede combinarse para delinear la heterogeneidad y immunophenotypically caracterizar poblaciones celulares molecularmente distintos.
Resumen
Immunophenotypic caracterización y análisis molecular han utilizado durante mucho tiempo heterogeneidad de delinear y definir poblaciones celulares distintas. FACS es inherentemente un sola célula ensayo, sin embargo antes de análisis molecular, las células diana se aíslan a menudo prospectivamente a granel, perdiendo resolución unicelular. Análisis de expresión génica sola célula proporciona un medio para entender las diferencias moleculares entre las células individuales en poblaciones celulares heterogéneas. En análisis de la célula a granel una sobrerrepresentación de un tipo de células distinto resultado sesgos y oclusiones de las señales de las células raras con importancia biológica. Utilizando FACS índice clasificación acoplada a análisis de expresión génica unicelular, poblaciones pueden ser investigadas sin pérdida de resolución unicelular aunque también se capturan las células con expresión de marcadores de superficie intermedia de la célula, permitiendo la evaluación de la importancia de la expresión del marcador de superficie continua. Aquí, describimos un acercamiento que combina unicelular transcripción reversa cuantitativa PCR (RT-qPCR) y FACS índice de clasificación para al mismo tiempo caracterizar molecular y heterogeneidad de immunophenotypic dentro de poblaciones celulares.
En contraste con los métodos de secuenciación de RNA unicelular, el uso de qPCR con amplificación específicos permite medidas robustas de transcripciones de baja abundancia con menos abandonos, mientras que no se confunde por cuestiones relacionadas con las variaciones de célula a célula en lectura profundidad. Por otra parte, directamente índice-clasificación individual-células en lisis de búfer este método, permite para que síntesis de cDNA y pre-amplificación específicos a realizar en un solo paso, así en cuanto a la correlación de firmas moleculares derivadas posteriormente con la superficie de la célula expresión del marcador. El enfoque descrito se ha desarrollado para investigar células hematopoyéticas de solo, pero también se han utilizado con éxito en otros tipos celulares.
En conclusión, el enfoque descrito en el presente documento permite medida sensible de la expresión del mRNA de un panel de genes previamente seleccionados con la posibilidad de desarrollar protocolos para el aislamiento posterior prospectivo de molecularmente diferentes subpoblaciones.
Introducción
Cada célula individual de la sangre se cree que residen en una jerarquía de celular, donde las células madre forman el ápice en la parte superior una serie de progenitores intermedias cada vez más comprometidos que eventualmente terminal se diferencian en las células effector final transporte específico funciones biológicas1. Mucho del conocimiento sobre cómo están organizados los sistemas de la célula de vástago se ha generado en el sistema hematopoyético, en gran parte debido a la capacidad de prospectiva aislar a poblaciones hematopoyéticas distintas altamente enriquecidas de células madre o progenitores diferentes2 por clasificación de FACS. Esto ha permitido muchas de estas poblaciones a analizarse funcionalmente o molecularmente, predominante a través de la expresión génica perfiles3,4. Sin embargo cuando análisis de expresión génica de las diferencias individuales de las poblaciones de bulto entre las células se promediaron y perdió5. Por lo tanto, incapacidad para detectar variaciones de célula a célula en fracciones celulares heterogéneas puede confundir nuestra comprensión de los procesos biológicos críticos si pequeños subconjuntos de células responsables de la función biológica inferida población6, 7. Por el contrario, investigación de firmas de expresión génica en unicelulares resolución ofrecen la posibilidad a delinear la heterogeneidad y eludir influencias precede de subconjuntos excesivamente de la células8.
Hasta la fecha se han desarrollado muchos protocolos para análisis de expresión génica unicelular; cada enfoque tiene sus propias advertencias. El método más temprano fue el RNA fluorescente en situ hibridación RNA (peces), que mide un número limitado de las transcripciones a la vez, pero es el único que permite la investigación de localización de RNA9,11. Primeros métodos usando PCR y qPCR para detectar que una sola o muy pocas transcripciones fueron también desarrollaron12. Sin embargo, estos últimamente han sido sustituidos por métodos basados en microfluídica que pueden analizar simultáneamente la expresión de cientos de transcripciones por la célula en cientos de células mediante qPCR y así permiten la heterogeneidad multidimensional utilizando análisis predeterminado de10,de paneles de gen13. Recientemente las tecnologías basadas en la secuencia de RNA han convertido en ampliamente utilizadas para el análisis de la célula, ya que teóricamente pueden medir el transcriptoma completo de una célula y así añadir una dimensión exploratoria a heterogeneidad análisis10, 14. Análisis de qPCR de multiplexado y la secuencia de RNA unicelulares tienen características diferentes, así la justificación para el uso de cualquiera de los métodos depende de la pregunta así como el número de células en la población objetivo. El alto rendimiento y bajo costo por la célula con características imparciales, exploratorias de la secuencia de RNA unicelulares son deseables cuando investigan celular desconocido o grandes poblaciones. Sin embargo, la secuencia de RNA unicelular también está sesgada hacia alta abundantes transcripciones de secuenciación más con frecuencia mientras las transcripciones con baja abundancia son propensas a la deserción. Esto puede conducir a los datos considerablemente complejos que pone altas demandas en análisis Bioinformático para revelar importantes señales moleculares que son a menudo sutiles u ocultas en ruido técnica15. Así, para los tejidos bien caracterizados, unicelular qPCR utilizando análisis predeterminados paneles primer seleccionados para los genes funcionalmente importantes o marcadores moleculares pueden servir como un acercamiento sensible, simple para determinar la heterogeneidad de un población. Sin embargo, cabe señalar que, en comparación a unicelular RNA-seq, el costo por la célula es generalmente más alto para qPCR unicelular métodos. Aquí, describimos un acercamiento que combina unicelular RT-qPCR (modificado de Teles J., et al. 16), al índice de FACS clasificación simultáneamente17 y bioinformática análisis18 a fin de caracterizar la heterogeneidad molecular e immunophenotypic dentro de las poblaciones.
En este enfoque, la población de la célula de interés se tiñe, y solo las células se clasifican por FACS directamente en tampón de lisis en placas de PCR de 96 pocillos. Al mismo tiempo, los niveles de expresión de un conjunto de marcadores de superficie celular se registran para cada celda individual durante FACS-clasificación, un método que se conoce como índice de clasificación. Posteriormente se amplifica el material sometidas a lisis celular y la expresión génica de un conjunto seleccionado de genes analizados con RT-qPCR, utilizando una plataforma de microfluidos. Esta estrategia permite el análisis molecular de la caracterización ordenada unicelular como simultáneo de la expresión de marcadores de superficie celular de cada célula individual. Mediante la asignación directamente molecularmente diferentes subconjuntos de células a la expresión de los marcadores clasificados indexadas, las subpoblaciones pueden vincularse con un immunophenotype específico que puede utilizarse para su aislamiento prospectivo. El método se describe paso a paso en la figura 1. Un panel determinado gen adicional contribuye a una mayor resolución de la expresión de genes específicos, ya que elude la medición de genes abundantes irrelevantes que de lo contrario puede ocluir gene sutiles señales de expresión. Por otra parte, la amplificación de la Diana, transcripción reversa un paso y amplificación permite medición robusta de baja transcripciones expresadas, como factores de transcripción o RNAs poli-adenylated. Lo importante, qPCR métodos permiten medición de mRNA de las proteínas de fusión, que es importante la investigación de ciertas enfermedades malignas19. Por último, el número centrado de genes investigados, bajas tasas de abandono, y limitadas diferencias técnicas entre las células hacen este método fácilmente analizado compararon con métodos dimensionales más alta, como unicelular RNA-seq. Siguiendo el protocolo, puede realizarse un experimento todo, de la clasificación de las células a los resultados analizados, dentro de tres días, haciendo de este un método sencillo y rápido para análisis de expresión génica sola célula sensible, de alto rendimiento.
Protocolo
1. preparación de placas de lisis
- Un banco libre de ARN/ADN, preparar suficiente buffer de lisis de 96 pocillos, con 10% extra, mezclando 390 agua libre de nucleasa μl, 17 μl de 10% NP-40, 2,8 μl 10 mM dNTP, 10 μl de 0,1 M TDT e inhibidor de Rnasa de 5.3 μl (véase Tabla de materiales). Vórtice y desactivación.
- Distribuir 4 μL de tampón de lisis en cada pocillo de una placa PCR bien 96 y sellar las placas con la película adhesiva. Desactivación de tubos para recoger el líquido en la parte inferior de las placas. Manten las placas de hielo hasta la célula clasificación (máximo 24 h).
2. preparación de células para la clasificación de la célula
- Descongelar el número apropiado de células (aquí, enriquecido madre hematopoyética CD34 y células progenitoras) para el experimento. 1 x 106 células son apropiadas para clasificar aproximadamente tres placas de 96 pocillos de solo las células con los controles.
- Transferencia de células descongeladas a un tubo cónico de 15 mL y añadir 1 mL de SBF cada 30 s hasta alcanzar un volumen total de 8 mL. Girar las células en una centrifugadora a 350 x g por 10 min a 4 ° C y eliminar el sobrenadante.
- Resuspender las células en 8 mL de tampón de tinción (PBS con 2% SBF) y centrifugar a 350 x g durante 10 min a 4 ° C y eliminar el sobrenadante.
- Resuspender las células en 200 μL tinción de buffer y eliminar células para control de manchas.
- Hacen fluorescencia menos uno controla (consistente) para cada fluoróforo, manchando de una fracción de células en 50 μl de tampón de tinción. En este ejemplo, se utilizan 6 tubos de microcentrífuga con 20.000 células como FMOSs. Tenga en cuenta que el número de células debe ajustarse dependiendo de la población investigada. Añadir anticuerpos todos en la misma concentración como en la tinción de la muestra excepto uno en cada tubo.
- Hacer las manchas individuales para cada fluoróforo manchando de una fracción de células en 50 μl de tampón de cada fluoróforo utilizado. En este ejemplo se utilizan 6 tubos de microcentrífuga con 20.000 células. Tenga en cuenta que el objetivo para cada anticuerpo debe ser expresada por las células que se utiliza para los controles. Añadir cada anticuerpo en la misma concentración como en la tinción de la muestra en tubos individuales. Además tenga 20.000 células sin manchas en 50 μl como control sin manchas.
- Añadir a la muestra de células, anticuerpos en su concentración adecuada. Usados aquí son CD34-FITC a una concentración 1/100, 1/50, CD38-APC CD90-PE 1/10, CD45RA-bv421 1/50, CD49F-PECy7 1/50 y linaje mezcla: 1/50 de CD3-PECy5 CD2-PECy5 1/50, CD19-PECy5 1/50, CD56-PECy5 1/50, CD123-PECy5 1/50, CD14-PECy5 1/50, CD16-PECy5 1/50, y CD235a-PECy5 1/1000.
- Incubar las células con anticuerpos por 30 min en hielo en la oscuridad.
- Lavar las células con 3 mL de tampón de tinción. Centrifugar las células a 350 x g por 10 min a 4 ° C y eliminar el sobrenadante.
- Resuspender las células y repetir el paso 2.9.
- Agite la muestra de 500 μl y consistente en 100 μl de buffer con células 7AAD y filtro de 1/100 de tinción a través de un filtro de 50 μm para obtener una suspensión unicelular.
3. célula clasificación
- Asegúrese que la máquina de FACS está correctamente con retardo de caída y configuración citómetro y seguimiento (CST) que recientemente se han realizado según las instrucciones del fabricante, para asegurar que se ordenan las células adecuadas. De células hematopoyéticas, se recomienda el uso de la velocidad de la boquilla y máximo de 85 micras de 4, mientras que la tasa de evento óptima es entre 800 y 2000 eventos/s.
- Correcta superposición espectral mediante la realización de compensación de fluorescencia y ajustar puertas según FMO controles o controles internos negativos.
- Realizar nuevo análisis de la población objetivo por clasificar por lo menos 100 células de destino en un nuevo tubo de microcentrífuga con 100 μl de tampón de tinción. FACS analizar las células clasificadas mediante el registro de la muestra ordenada y asegúrese de que terminan en la puerta de clase.
- Instalación placa celular única clasificación centrando la gota en el pocillo A1 en una placa bien 96. Cuando se centra, ordenar las partículas del μm 50 – 100 6 en todos los pozos alrededor del borde de un plato bien 96 vacío para asegurar que todos los pocillos Obtén una celda en el centro de cada pozo.
- Si es posible, puede agregarse un control adicional para asegurarse de que las células viables se ordenan por ordenar solo las células para el crecimiento en vitro . Células hematopoyéticas pueden cultivarse en placas bien inferior U 96 en 100 μl SFEM con 1% estreptomicina penicilina, 100 ng/mL FLT3L, TPO y SCF. Analizar cada bien después de 3 días en la cultura para las colonias de células utilizando un microscopio.
- Retire la película de placas. Una sola célula de tipo de interés (aquí Lin-CD34 + CD38-células) en 92 de 96 pocillos, activar ordenación de índice en el FACS clasificación software para guardar el perfil immunophenotypic para otros marcadores de interés (aquí CD45RA, CD49f y CD90) para cada célula.
- Ordenar dos pozos con 10 y 20 células respectivamente para controles de linealidad de la amplificación por PCR. Pozos H1 y H2 se utilizan generalmente.
- Mantener dos pozos sin cualquier célula como plantilla de no controles, generalmente pozos H3 y H4.
- Sellar las placas con la película transparente adhesiva y girar las placas a 300 x g durante 1 min antes de presión cero en hielo seco.
- Almacenar las placas congeladas a-80 ° C.
Nota: Punto seguro. Las células sometidas a lisis y ordenadas pueden conservarse a-80 ° C para almacenamiento a largo plazo.
4. Invierta la transcripción y la amplificación de la Diana
- Preparar la mezcla de cartilla para todos los objetivos gen 96 agregando 2 μl de cada primer, incluyendo par limpieza gene cartillas y cartillas para el control de tacon en RNA, de 1,5 mL tubo libre de ARNasas en un banco libre de ARN/ADN. Si usando las cartillas menos de 96, añadir un volumen equivalente de agua libre de nucleasa para los iniciadores que falta. Cartillas se ordenan por separado para que coincida con el panel del gene deseado.
- Hacer la transcripción inversa y amplificación específicos mezclar agregando 632.5 μl 2 x la mezcla de reacción, 101.2 μl Taq/SuperscriptIII, 151.8 μl mezclar de cartilla y 0,7 μl con control RNA. Preforma de este paso en un banco de ADN gratuito. Mezcla por Vortex y centrifugar hasta recoger el líquido en la parte inferior del tubo. Mantenga en hielo hasta que además de la muestra.
- Hacer no-reverso transcripción control mezcla de cuatro pozos mezclando 27,5 μl de 2 x la mezcla de reacción, 1.76 μl de enzima Taq, 6,6 μl de mezcla de cartilla y 2.64 μl de agua libre de nucleasa. Punto de control de RNA. Realizar este paso en un banco de ADN gratuito. Vórtice y vuelta hacia abajo para recoger el líquido en la parte inferior del tubo y mantener en hielo hasta la adición a la muestra.
- Descongelar las placas lisadas en hielo. Añadir μl 8,75 de la transcripción reversa previamente preparada y mezcla de amplificación específicos a 92 pozos, incluyendo los controles de linealidad y no es de plantilla. Añadir 8,75 μl de mezcla de control no-reverso transcripción a los cuatro pozos restantes. Sellar las placas con la película adhesiva del claro y vuelta a recoger líquido en la parte inferior de las placas.
- Preforma de transcripción inversa y amplificación específicos mediante la ejecución de la placa en una máquina PCR según el programa previo; Paso 1:50 ° C durante 60 min, paso 2: 95 ° C por 2 min, paso 3: 95 ° C por 15 s, paso 4: 60 ° C por 4 min, repita los pasos 3-4 24 veces y finalmente el paso 5: 8 ° C para siempre.
- Después de PCR es completa, mantenga la placa en 8 ° C para almacenamiento a corto plazo y -20 ° C para almacenamiento a largo plazo.
Nota: Punto seguro. Material amplificado puede conservarse a 8 ° C para almacenamiento a corto plazo y a-20 ° C para almacenamiento a largo plazo.
5. preparación de muestra y las placas de ensayo para análisis de expresión génica de microfluidos Multiplex
- Preparar ensayo placa de carga por pipeteo 3 μl de reactivo de ensayo de carga en cada pocillo de una placa bien 96. Agregar 3 μl de cada cebador a pocillos individuales en el ensayo de placa de carga.
- Selle la placa con película adhesiva y vuelta a recoger líquido en la parte inferior de las placas.
- Preparar placa de dilución por pipeteo 8 μl de nucleasa agua libre en todos los pocillos de una placa bien 96. Añadir 2 μl de la muestra ampliada a la placa de la dilución, realizar una dilución final de 1:5.
- La placa con la película adhesiva del sello, de la mezcla por la placa de Vortex por 10 s y por último girar hacia abajo para recoger líquido en la parte inferior de las placas.
- Preparar mezcla de carga de la muestra mezclando cuidadosamente 352 μl de mezcla principal con 35,2 μl de muestra carga reactivo. Preparar placa de carga de la muestra tomar 3.3 μl de mezcla a cada pocillo de una placa bien 96 de carga.
- Añadir 2,7 μl de la muestra diluida en cada pocillo de la muestra de placa de carga.
- Selle la placa con película adhesiva y vuelta a recoger líquido en la parte inferior de las placas.
6. carga del Chip de microfluidos
- Sacar un nuevo chip de microfluidos 96 x 96. Preparar las entradas por meter con una jeringa con la tapa en asegurarse de que se puedan mover.
- Elimine las burbujas de la jeringa. Añadir volumen completo de jeringas a cada válvula mientras el chip de inclinación 45 grados y presionar la válvula. Primer chip con el controlador IFC.
- Cargar cada entrada ensayo con 4.25 μl de cada uno de los pozos en el ensayo de placa de carga. Evitar las burbujas. Si aparecen burbujas en el pozo, elimínelos con una punta de pipeta.
- Continuar cargando cada entrada muestra con 4.25 μl de cada uno de los pozos en la muestra de placa de carga, evitar burbujas y si aparecen burbujas, retirar con la punta de la pipeta.
- Carga de viruta con el controlador IFC.
- Verifique que el chip se ve aún y que las cámaras hayan sido cargadas. Eliminar el polvo de la superficie del chip tocando con cinta. El chip se ejecutan en la plataforma de expresión de gene de microfluidos multiplex.
7. ejecución de Chip en Multiplex microfluídicos Gene expresión plataforma
- Después de cargar el chip en la plataforma de expresión de gene de microfluidos multiplex, nombre de la muestra.
- Establecer ROX como tinte pasiva. Fijar sonda individual y FAM-MGB como fluorescencia. 96 x 96 estándar v2 como el protocolo en uso. Empezar a correr.
- Quitar chip cuando se ejecuta es completa.
8. preliminar análisis del funcionamiento de la viruta
- Cargar datos en el software de análisis de PCR en tiempo real.
- Cargar nombres de genes y células pegar diseños celular y génica de un archivo delimitado por tabuladores.
- Abrir la vista de la imagen y seleccione ROX como tinte. Compruebe si todos los pozos tienen tinte pasiva ROX.
- Investigar si todas las parcelas de amplificación mira bien, con una curva de amplificación liso sin púas (similar a la figura 3E).
- Asegúrese de que todas las células solo tienen expresión de tacon en control RNA para asegurarse de que todos hayan sido cargados correctamente.
- Asegúrese de que todas las células tienen genes housekeeping y así han sido clasificadas correctamente.
- Aseguran control de linealidad de célula 10 y 20 aproximadamente 1 CT diferencia a validar linear de la amplificación.
- Compruebe si hay expresión en las muestras de control norte. Si se detecta la expresión en noRT considere cambiar sondas sondas que no detectan ADN genómico para posteriores carreras.
- Exportación de datos en archivos csv para su posterior análisis.
9. unicelular análisis con SCExV
Nota: Una película introductoria está presente20 para introducir la herramienta. Aquí, se presenta una breve recomendación de cómo hacer uso de los controles introducidos en el protocolo de análisis.
- Conecte a la Página Web de SCexV20.
- Subir archivos CSV exportados.
- Eligió el tacon en control RNA como control positivo. Eliminar cualquier célula que tiene un control ARN CT por encima de 25. Normalizar los datos mediana expresión de control RNA.
- Haga clic en "aquí -> analizar". Eliminar noRT, notemplate, 10 y 20 controles de celular en la opción de excluir de la célula.
- Se agrupan las células con el enfoque de clusterización de elección con el número esperado de racimos. Valores de exportación analizado.
10. ordenación de índice análisis
- Abra el software de análisis FACS y cargar las muestras indexadas.
- Abra el editor de secuencias de comandos y ejecute el script disponible de J. Quinn et al. 21
- Ahora que el software de análisis FACS si han hecho una puerta para cada una de las células, abrir el editor de diseño y color de las células según la agrupación de SCexV (disponible en el archivo llamado "Sample_complete_Data.xls").
Resultados
El protocolo descrito es rápido, fácilmente realizado y altamente confiable. Una descripción del montaje experimental se presenta en la figura 1. El conjunto del Protocolo, de separación de células individuales, amplificación del objetivo específico, las mediciones de expresión génica y análisis preliminar se puede realizar en tres días. Un ejemplo de los resultados analizados en forma de un mapa de calor que representa datos preliminares del análisis de la expresión de gene unicelular utilizando cebadores de 96 y 96 células de un paciente leucemia mieloide crónica (LMC) o 96 de una edad comparable sanos control se presenta en la figura 2. Mediante clustering jerárquico, las células analizadas pueden dividirse en cuatro subgrupos basados en sus firmas de expresión génica. La visualización de mapa de calor es una forma conveniente para obtener un resumen de los datos, así como para control de pozos que deben ser excluidos del análisis (por ejemplo, pozos de control). Figura 2B es un análisis de componentes principales (PCA) visualizar cómo las células de cada grupo son mutuamente mediante reducción de dimensión. Aquí, afloramientos se distinguen fácilmente del resto de las células. Analizar cómo los genes individuales se distribuyen entre los grupos difieren entre ellos, como parcelas de violín son útiles. En la figura 2, se muestran la expresión de cuatro genes representativos: el gen de fusión BCR-ABL1, que marca todas las células leucémicas; el ciclo celular marcador mKI67, que se expresa en un grupo activamente divisorio; el marcador de diferenciación mieloide MPO, restringido para el subgrupo de ciclismo; y por último la casa gen RPS18, que se expresa en todas las células. Finalmente, en la Figura 2D las firmas moleculares se han correlacionado a immunophenotyped, como las identidades de cada celda de cada cluster se utilizan para las celdas individuales en una parcela de FACS generados a partir de la clasificación de índice de color. Es la expresión de marcador superficial de la célula para los marcadores de células madre hematopoyéticas CD90 y CD45RA que en este caso se podría utilizar para discriminar entre algunos de los clusters y aislarlos prospectiva para el análisis funcional.
La microfluídica utilizada para llevar a cabo la qPCR unicelular es muy sensible a la introducción de aire o partículas en el sistema. Por lo tanto, es fundamental hacer una verificación de post ejecución de calidad de los datos. La figura 3 muestra a representante de datos y el contraste entre un éxito y no funcionan a causa de carga de la muestra pobre. Figura 3A muestra cómo los niveles de Rox después de una exitosa propagación uniformemente en todos los pocillos tranquilizador que la carga ha sido un éxito. En cambio, figura 3B ilustra cómo han fallado muestra y la carga de ensayo según lo indicado por la falta de ROX en grandes partes del chip. Una vez que se ha evaluado la calidad de Rox, puede evaluarse la calidad de las señales de expresión del gen crudo. En la figura 3, se presenta un mapa de calor de una carrera exitosa, donde expresión es uniformemente repartida por muestras con señal fuerte de alrededor de 7-25 Ct:s. en contraste, no figura 3D muestra un mapa de calor de una gestión fallida donde muchas muestras tienen ninguna señal o expresión de una cantidad muy limitada de los genes. Los resultados en la figura 3D están probable que debido a un error en la clasificación de la FACS antes de análisis de la expresión del gene de unicelulares, ya que muchas células tienen señales de expresión clara mientras que otros carecen totalmente de expresión. Por último, figura 3E muestra la curva de amplificación esperados de tacon en control RNA con todos los pozos con expresión clara de alrededor 10 CT con poca o ninguna variación. Este control positivo mide la eficiencia de todos los pasos en el análisis de qPCR unicelular. Para asegurarse de que la amplificación está dentro de rango dinámico, un número diferente de células está incluido como controles de linealidad; aquí, se utilizan controles de 10 y 20 células. Las diferencias de CT entre controles de linealidad deben reflejar las diferencias en el número de células. Finalmente, la no transcripción inversa (norte) y los controles negativos de ninguna plantilla aseguran que no hay señales positivas falsas son detectadas de genomic DNA o contaminaciones, respectivamente.

Figura 1: vista esquemática del protocolo. Las células se tiñen, con aplicación de tipo índice activado para grabar la expresión superficial del marcador y lisis para liberar el ARNm. mRNA es posteriormente atrás transcrito a cDNA amplificado usando la amplificación de la Diana. A continuación, las muestras se cargan junto con cartillas individuales en un chip de microfluidos, donde se analiza el perfil de expresión génica de cada celda usando RT-qPCR. Después de la recolección de datos, los datos previamente procesados para eliminar las células de baja calidad y agrupamiento se realiza para definir poblaciones celulares molecularmente distintos. Finalmente, las subpoblaciones molecular definidas están correlacionadas con la expresión superficial del marcador de FACS índice clasificación datos immunophenotypically caracterizan la heterogeneidad de las poblaciones. Haga clic aquí para ver una versión más grande de esta figura.
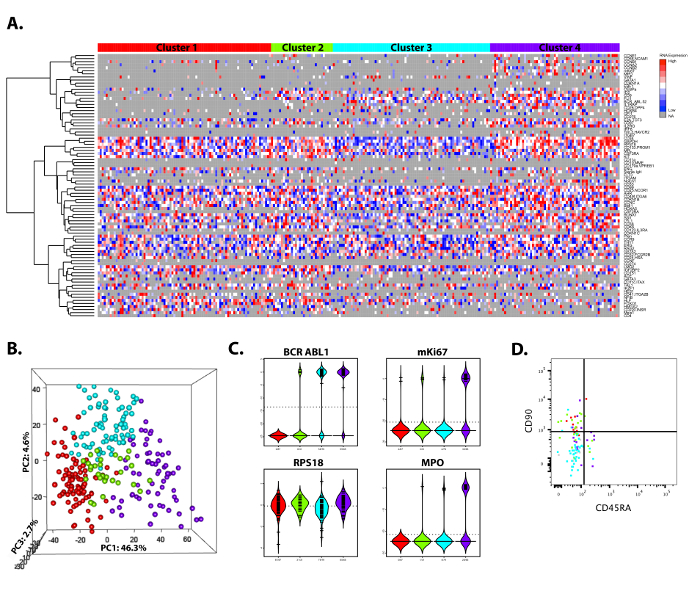
Figura 2: analiza resultados representativos de tres de éxito sola célula genes de las células madre hematopoyéticas normales y leucémicas. (A) calor mapa mostrando unicelular gen medidas de expresión de 270 celdas individuales de un paciente de leucemia mieloide crónica, así como un edad comparable del control sano por agrupamiento jerárquico con SCExV 18, una herramienta de análisis diseñado para este tipo de datos. Rojo representa alta expresión, expresión bajo azul y no gris expresión. Las células se dividieron en cuatro grupos diferentes, basados en sus firmas de expresión génica. Está claro de los datos que la población de células leucémicas de este paciente puede dividirse en una población quieta (cluster 3), con la expresión de sólo unos cuantos marcadores de diferenciación y una población activamente divisoria que ha iniciado la expresión de genes asociados con la diferenciación mieloide (cluster 4). Los datos utilizados aquí están de un paciente seleccionado dentro de trabajos previamente publicados por Warfvinge et al. 22 (B) principales análisis de componentes (PCA) de los datos mostrados en el A, donde los cuatro separados grupos pueden ser demostrado. (C) violín parcelas de cuatro genes con expresión específica de cluster, BCR-ABL1, mKI67, RPS18 y MPO. Cada parcela de violín representa la expresión en cada grupo y a través de la línea de puntos representa el valor medio de expresión para todas las células. (D) análisis de ordenación de índice de una de las muestras en el mapa de calor, donde cada elemento está marcado con el color de su subpoblación molecular. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: controles de calidad de qPCR multiplex en el software PCR en tiempo real. (A) carga de Rox imagen de éxito muestra y la carga de ensayo. Rox (B) carga de imagen de éxito muestra y prueba de carga, donde se muestran cargas fallidas de muestras por la falta de ROX en líneas horizontales. Carga fallida de ensayos se mostrarán en líneas verticales. (C) ejemplo de un exitoso funcionamiento en la vista de mapa de calor, donde solo células representan filas y genes columnas. Expresión génica alta está indicado en amarilla, baja expresión en azul y ninguna expresión detectada en negro. (D) ejemplo de no ejecutar en vista del mapa de calor, donde las células solo representan filas y genes columnas. Expresión génica alta está representada por amarillo, baja expresión está representado por el azul y ninguna expresión detectada como negro. (E) intensidad normalizada (izquierda) y parcela de amplificación (derecha) de un tacon en el control de RNA. Valores de CT de aproximadamente 10 y curvas con limitada variación indican éxito robusto reverso transcripción, pre-amplificación y qPCR análisis y dentro de todos los pozos. (F) normalizado intensidad (izquierda) y parcela de amplificación (derecha) de un gen fallido. Las intensidades normalizadas no forman una curva y la amplificación muestra grandes picos de fluorescencia. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
En los últimos años, análisis de expresión génica de la célula solo se ha convertido en un valioso aporte para definir la heterogeneidad de las diferentes poblaciones de células23. El advenimiento de las tecnologías de secuencias de RNA ofrece teóricamente una posibilidad para medir el transcriptoma completo de una célula, sin embargo estos métodos son complicados por las variaciones de profundidad de secuenciación de célula a célula y abandono. Sola célula qPCR ofrece un análisis sensible y robusto de la expresión de cientos de genes críticos en todas las células son tratadas del mismo modo, reduce el ruido del técnico. Además el análisis enfocado de un número limitado de expedientes permite análisis simplificado sin la interferencia de genes altamente expresados.
Desde el enfoque investiga sólo un subconjunto de genes expresados en las células es muy importante elegir el sistema correcto de genes al diseñar el panel de genes. La elección de genes se puede hacer de varias maneras; a través de la literatura, los resultados anteriores por análisis de bulto y bioinformática de datos públicamente disponibles. Esto no es trivial; sin embargo, con el conjunto correcto de genes el enfoque puede ser muy poderoso. Cuando el panel de genes ha sido diseñado, es importante validar que los cebadores no están interfiriendo en la multiplexación. Aquí te recomendamos analizar si los iniciadores son lineales por preforma una serie de diluciones con amplio material; una dilución en serie de los primers utilizados en este estudio se muestra en el suplementario Figura 2D en Warfvinge et al. 22
Las dos principales limitaciones del protocolo presentado aquí son el número limitado de células y genes investigados. Sin embargo, a pesar de solamente 96 células se investigan en cada plazo, este protocolo relativamente simple puede ser completado en un día y así cientos de células que pueden analizarse en una semana. Además, el número de genes investigados podría aumentar si más cartillas están incluidos en la etapa de pre-Amplificacion de específico del destino. En este caso, se necesitan varios chips para investigar cada juego de 96 iniciadores.
Si altamente expresado genes o células con alto contenido de RNA están siendo analizados, debe modificarse el número de ciclos optimizados para las células madre hematopoyéticas con baja total gene expresión23 . Cambio de ciclos de amplificación, también es importante la investigación de las poblaciones a granel. Para el análisis de volumen de células madre hematopoyéticas (100 células) se recomienda reducir la cantidad de ciclos de amplificación previa de 25 a 18.
Razones más comunes de funciona es debido a la clasificación de células solo en el pozo de la placa de lisis, clasificación de las células no viables, pre-amplificación subóptima o chip carga del fracaso. El nivel de Rox en cada cámara de reacción es un indicador de la carga de muestra o ensayo y si éstos son bajos, se recomienda que las muestras se vuelva a ejecutar en un nuevo chip, desde Rox bajos niveles son causados probablemente por la introducción de burbujas durante la carga. Falta de control de tacon en expresión de RNA es un indicador de que los pre-amps han fracasado, probablemente debido a problemas con pre-amplificación- o reactivos de transcripción. En este caso, se recomienda que un nuevo chip se ejecuta con las células recién ordenadas y nuevos reactivos. Detección de RNA del control pero no hay indicación de otros genes de señales es una indicación de éxito unicelulares FACS clasificación antes de análisis de expresión génica. Para evitar errores durante el FACS clasificación es importante asegurar que las células solo se clasifican en el centro de cada pozo. Esto puede hacerse por clasificar los granos en un plato vacío y visualmente inspeccionar donde las tierras de la gota. Asegúrese de que ordenar prueba en todos los pozos en los bordes de la placa para asegurarse de que la configuración no sólo es correcta para los primeros pocos pozos. En su caso, solo las células pueden ser clasificadas en paralelo para el crecimiento in vitro y analizan a antes de su análisis de qPCR para controlar protocolos de clasificación acertados. Índice-clasificación de la información no sólo permite investigar immunophenotyped de una subpoblación molecularmente distinta pero también puede proporcionar información útil al solucionar problemas, si se clasifica una población definida y muchas células de baja calidad presente en el análisis de la expresión de gene de unicelulares. La información de ordenación de índice puede utilizarse para optimizar la estrategia de bloquea y evitar la selección futura de las células de baja calidad.
Análisis de expresión génica unicelular es revolucionando la célula cómo heterogénea poblaciones son investigadas y allanando el camino para la comprensión adicional de la diferenciación celular. En la hematopoyesis, análisis de expresión génica de la célula solo se han utilizado para afinar estrategias de ordenación para subconjuntos de HSCs7, para resolver la heterogeneidad de las poblaciones de progenitoras multipotentes6,24,25 e identificar la terapia células leucémicas insensible poblaciones22,26. Combinación del índice de clasificación unicelulares gene expresión - tanto el análisis funcional se ha demostrado previamente para ser confiable y eficiente al investigar el immunophenotype y heterogeneidad de las diferentes poblaciones27, 28,29. Aquí, un enfoque para la investigación molecular y heterogeneidad de immunophenotypic de célula poblaciones han sido descritos donde combina unicelular qPCR con índice de clasificación permite resultados rápidos y reproducibles sin análisis complicado en un corto período de tiempo.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo es apoyado por becas de la sociedad sueca de cáncer, el Consejo de investigación sueco, sociedad sueca para la investigación médica, la Fundación sueca de cáncer infantil, la Fundación de Söderberg de Ragnar y el Knut y Alice Wallenberg Foundation
Materiales
| Name | Company | Catalog Number | Comments |
| CD14 PECY5 | eBioscience | 15-0149-42 | Clone: 61D3 |
| CD16 PECY5 | Biolegend | 302010 | Clone: 3G8 |
| CD56 PECY5 | Biolegend | 304608 | Clone: MEM-188 |
| CD19 PECY5 | Biolegend | 302210 | Clone: HIB19 |
| CD2 PECY5 | Biolegend | 300210 | Clone: RPA-2.10 |
| CD3 PECY5 | Biolegend | 300310 | Clone: HIT3a |
| CD123 PECY5 | Biolegend | 306008 | Clone: 6H6 |
| CD235A PECY5 | BD Pharma | 559944 | Clone GAR2 |
| CD34 FITC | Biolegend | 343604 | Clone: 561 |
| CD38 APC | Biolegend | 303510 | Clone: Hit2 |
| CD90 PE | Biolegend | 328110 | Clone: 5E10 |
| CD45RA BV421 | BD bioscience | 560362 | Clone: HI100 |
| CD49f Pecy7 | eBioscience | 25-0495-82 | Clone: eBioGOH3 |
| FBS | HyClone | SV30160.3 | |
| PBS | HyClone | SH30028.02 | |
| 96-well u-bottom Plate | VWR | 10861-564 | |
| SFEM | Stem cell technologies | 9650 | |
| Penicillin streptomycin | HyClone | SV30010 | |
| TPO | Peprotech | 300-18 | |
| SCF | Peprotech | 300-07 | |
| FLT3L | Peprotech | 300-19 | |
| Falcon Tube 15 mL | Sarstedt | 62.554.502 | |
| Eppendorph tube | Sarstedt | 72.690.001 | |
| CST beads | BD | 642412 | |
| Accudrop Beads | BD | 345249 | 6 µm particles |
| Adhesive film Clear | Thermo scientific | AB-1170 | |
| Adhesive film Foil | Thermo scientific | AB-0626 | |
| 96 well PCR plate | Axygen | PCR-96M2-HS-C | |
| PCR 1.5 mL tube | Axygen | MCT-150-L-C | |
| T100 PCR cycler | BioRad | 186-1096 | |
| 10% NP40 | Thermo scientific | 85124 | |
| 10 mM dNTP | Takara | 4030 | |
| 0.1 M DTT | Invitrogen | P2325 | |
| RNAsout | Invitrogen | 10777-019 | RNAse inhibitor |
| CellsDirect One-Step qRT-PCR Kit | Invitrogen | 11753-100 | |
| Neuclease free water | Invitrogen | 11753-100 | from CellsDirect kit |
| 2x Reaction Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| SuperScript III RT/Platinum Taq Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| Platinum Taq DNA Polymerase | Invitrogen | 10966026 | |
| TaqMan Cells-to-CT Control Kit | Invitrogen | 4386995 | |
| Xeno RNA Control | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 20x Xeno RNA Control Taqman Gene Expression Assay | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 96.96 Sample/Loading Kit—10 IFCs | Fluidigm | BMK-M10-96.96 | |
| 2x Assay Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| 20x GE Sample Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| Control line fluid | Fluidigm | From 96.96 Sample/Loading Kit | |
| TaqMan Gene Expression Master Mix | Applied Biosystems | 4369016 | |
| BioMark HD | Fluidigm | BMKHD-BMKHD | |
| 96.96 Dynamic Array IFC | Fluidigm | BMK-M10-96.96GT | |
| Excel | Microsoft | Microsoft | |
| FlowJo V10 | TreeStar | TreeStar | |
| Fluidigm real time PCR analysis | Fluidigm | Fluidigm | |
| CD179a.VPREB1 | Thermofisher scientific | Hs00356766_g1 | |
| ACE | Thermofisher scientific | Hs00174179_m1 | |
| AHR | Thermofisher scientific | Hs00169233_m1 | |
| BCR_ABL.52 | Thermofisher scientific | Hs03043652_ft | |
| BCR_ABL41 | Thermofisher scientific | Hs03024541_ft | |
| BMI1 | Thermofisher scientific | Hs00995536_m1 | |
| CCNA2 | Thermofisher scientific | Hs00996788_m1 | |
| CCNB1 | Thermofisher scientific | Hs01030099_m1 | |
| CCNB2 | Thermofisher scientific | Hs01084593_g1 | |
| CCNC | Thermofisher scientific | Hs01029304_m1 | |
| CCNE1 | Thermofisher scientific | Hs01026535_g1 | |
| CCNF | Thermofisher scientific | Hs00171049_m1 | |
| CCR9 | Thermofisher scientific | Hs01890924_s1 | |
| CD10.MME | Thermofisher scientific | Hs00153510_m1 | |
| CD11a | Thermofisher scientific | Hs00158218_m1 | |
| CD11c.ITAX | Thermofisher scientific | Hs00174217_m1 | |
| CD123.IL3RA | Thermofisher scientific | Hs00608141_m1 | |
| CD133.PROM1 | Thermofisher scientific | Hs01009250_m1 | |
| CD151 | Thermofisher scientific | Hs00911635_g1 | |
| CD220.INSR | Thermofisher scientific | Hs00961554_m1 | |
| CD24.HSA | Thermofisher scientific | Hs03044178_g1 | |
| NCOR1 | Thermofisher scientific | Hs01094540_m1 | |
| CD26.DPP4 | Thermofisher scientific | Hs00175210_m1 | |
| CD274 | Thermofisher scientific | Hs01125301_m1 | |
| CD276 | Thermofisher scientific | Hs00987207_m1 | |
| CD32.FCGR2B | Thermofisher scientific | Hs01634996_s1 | |
| CD33 | Thermofisher scientific | Hs01076281_m1 | |
| CD34 | Thermofisher scientific | Hs00990732_m1 | |
| CD344.FZD4 | Thermofisher scientific | Hs00201853_m1 | |
| CD352.SLAMF6 | Thermofisher scientific | Hs01559920_m1 | |
| CD38 | Thermofisher scientific | Hs01120071_m1 | |
| CD4 | Thermofisher scientific | Hs01058407_m1 | |
| CD41.ITGA2B | Thermofisher scientific | Hs01116228_m1 | |
| CD49f.ITGA6 | Thermofisher scientific | Hs01041011_m1 | |
| CD56.NCAM1 | Thermofisher scientific | Hs00941830_m1 | |
| CD9 | Thermofisher scientific | Hs00233521_m1 | |
| CD97 | Thermofisher scientific | Hs00173542_m1 | |
| CD99 | Thermofisher scientific | Hs00908458_m1 | |
| CDK6 | Thermofisher scientific | Hs01026371_m1 | |
| CDKN1A | Thermofisher scientific | Hs00355782_m1 | |
| CDKN1B | Thermofisher scientific | Hs01597588_m1 | |
| CDKN1C | Thermofisher scientific | Hs00175938_m1 | |
| CEBPa | Thermofisher scientific | Hs00269972_s1 | |
| CSF1r | Thermofisher scientific | Hs00911250_m1 | |
| CSF2RA | Thermofisher scientific | Hs00531296_g1 | |
| CSF3RA | Thermofisher scientific | Hs01114427_m1 | |
| E2A.TCF3 | Thermofisher scientific | Hs00413032_m1 | |
| EBF1 | Thermofisher scientific | Hs01092694_m1 | |
| ENG | Thermofisher scientific | Hs00923996_m1 | |
| EPOR | Thermofisher scientific | Hs00959427_m1 | |
| ERG | Thermofisher scientific | Hs01554629_m1 | |
| FLI1 | Thermofisher scientific | Hs00956711_m1 | |
| FLT3 | Thermofisher scientific | Hs00174690_m1 | |
| FOXO1 | Thermofisher scientific | Hs01054576_m1 | |
| GAPDH | Thermofisher scientific | Hs02758991_g1 | |
| GATA1 | Thermofisher scientific | Hs00231112_m1 | |
| GATA2 | Thermofisher scientific | Hs00231119_m1 | |
| GATA3 | Thermofisher scientific | Hs00231122_m1 | |
| GFI1 | Thermofisher scientific | Hs00382207_m1 | |
| HES1 | Thermofisher scientific | Hs01118947_g1 | |
| HLF | Thermofisher scientific | Hs00171406_m1 | |
| HMGA2 | Thermofisher scientific | Hs00171569_m1 | |
| HOXA5 | Thermofisher scientific | Hs00430330_m1 | |
| HOXB4 | Thermofisher scientific | Hs00256884_m1 | |
| ID2 | Thermofisher scientific | Hs04187239_m1 | |
| IGF2BP1 | Thermofisher scientific | Hs00198023_m1 | |
| IGF2BP2 | Thermofisher scientific | Hs01118009_m1 | |
| IKZF1 | Thermofisher scientific | Hs00172991_m1 | |
| IL1RAP | Thermofisher scientific | Hs00895050_m1 | |
| IL2RG | Thermofisher scientific | Hs00953624_m1 | |
| IRF8 | Thermofisher scientific | Hs00175238_m1 | |
| ITGB7 | Thermofisher scientific | Hs01565750_m1 | |
| KIT | Thermofisher scientific | Hs00174029_m1 | |
| Lin28B | Thermofisher scientific | Hs01013729_m1 | |
| LMO2 | Thermofisher scientific | Hs00153473_m1 | |
| LYL1 | Thermofisher scientific | Hs01089802_g1 | |
| Meis1 | Thermofisher scientific | Hs01017441_m1 | |
| mKi67 | Thermofisher scientific | Hs01032443_m1 | |
| MPL | Thermofisher scientific | Hs00180489_m1 | |
| MPO | Thermofisher scientific | Hs00924296_m1 | |
| NFIB | Thermofisher scientific | Hs01029175_m1 | |
| Notch1 | Thermofisher scientific | Hs01062011_m1 | |
| Pten | Thermofisher scientific | Hs02621230_s1 | |
| RAG2 | Thermofisher scientific | Hs01851142_s1 | |
| RPS18 | Thermofisher scientific | Hs01375212_g1 | |
| RUNX1 | Thermofisher scientific | Hs00231079_m1 | |
| Shisa2 | Thermofisher scientific | Hs01590823_m1 | |
| Spi1 | Thermofisher scientific | Hs02786711_m1 | |
| Sterile.IgH | Thermofisher scientific | Hs00378435_m1 | |
| TAL1 | Thermofisher scientific | Hs01097987_m1 | |
| THY1 | Thermofisher scientific | Hs00264235_s1 | |
| Tim.3.HAVCR2 | Thermofisher scientific | Hs00958618_m1 | |
| VWF | Thermofisher scientific | Hs00169795_m1 |
Referencias
- Seita, J., Weissman, I. L. Hematopoietic stem cell: Self-renewal versus differentiation. Wiley Interdiscip Rev Syst Biol Med. 2 (6), 640-653 (2010).
- Orkin, S. H., Zon, L. I. Hematopoiesis: An evolving paradigm for stem cell biology. Cell. 132 (4), 631-644 (2008).
- Ye, F., Huang, W., Guo, G. Studying hematopoiesis using single-cell technologies. Journal of Hematology & Oncology. 10, 27 (2017).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nature Cell Biology. 16 (10), 919-927 (2014).
- Wills, Q. F., et al. Single-cell gene expression analysis reveals genetic associations masked in whole-tissue experiments. Nature Biotechnol. 31 (8), 748-752 (2013).
- Velten, L., et al. Human haematopoietic stem cell lineage commitment is a continuous process. Nat Cell Biol. 19 (4), 271-281 (2017).
- Wilson, N. K., Nicola, K., et al. Combined single-cell functional and gene expression analysis resolves heterogeneity within stem cell populations. Cell Stem Cell. 16 (6), 712-724 (2015).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: Advances and future challenges. Nucleic Acids Research. 42 (14), 8845-8860 (2014).
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of Single RNA Transcripts in Situ. Science. 280 (5363), 585-590 (1998).
- Kalisky, T., et al. A brief review of single-cell transcriptomic technologies. Briefings in Functional Genomics. , elx019 (2017).
- Crosetto, N., Bienko, M., van Oudenaarden, A. Spatially resolved transcriptomics and beyond. Nature Reviews Genetics. 16, 57 (2014).
- Bengtsson, M., Ståhlberg, A., Rorsman, P., Kubista, M. Gene expression profiling in single cells from the pancreatic islets of Langerhans reveals lognormal distribution of mRNA levels. Genome Research. 15 (10), 1388-1392 (2005).
- Bengtsson, M., Hemberg, M., Rorsman, P., Ståhlberg, A. Quantification of mRNA in single cells and modelling of RT-qPCR induced noise. BMC Molecular Biology. 9 (1), 63 (2008).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nature Methods. 10, 1096 (2013).
- Tung, P. -. Y., et al. Batch effects and the effective design of single-cell gene expression studies. Scientific Reports. 7, 39921 (2017).
- Teles, J., Enver, T., Pina, C. Single-cell PCR profiling of gene expression in hematopoiesis. Methods in Molecular Biology. , 21-42 (2014).
- Hayashi, T., et al. Single-cell gene profiling of planarian stem cells using fluorescent activated cell sorting and its "index sorting" function for stem cell research. Development, Growth, & Differentiation. 52 (1), 131-144 (2010).
- Lang, S., et al. SCExV: A webtool for the analysis and visualisation of single cell qRT-PCR data. BMC Bioinformatics. 16 (1), 320 (2015).
- de Klein, A., et al. A cellular oncogene is translocated to the Philadelphia chromosome in chronic myelocytic leukaemia. Nature. 300 (5894), 765-767 (1982).
- . indexed-sorting Available from: https://github.com/FlowJo-LLC/indexed-sorting (2016)
- Warfvinge, R., et al. Single-cell molecular analysis defines therapy response and immunophenotype of stem cell subpopulations in CML. Blood. 129 (17), 2384-2394 (2017).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Breton, G., et al. Human dendritic cells (DCs) are derived from distinct circulating precursors that are precommitted to become CD1c+ or CD141+ DCs. The Journal of Experimental Medicine. 213 (13), 2861-2870 (2016).
- Alberti-Servera, L., et al. Single-cell RNA sequencing reveals developmental heterogeneity among early lymphoid progenitors. The EMBO Journal. 36 (24), 3619-3633 (2017).
- Giustacchini, A., et al. Single-cell transcriptomics uncovers distinct molecular signatures of stem cells in chronic myeloid leukemia. Nature Medicine. 23, 692 (2017).
- Hansmann, L., Han, A., Penter, L., Liedtke, M., Davis, M. M. Clonal expansion and interrelatedness of distinct B-lineage compartments in multiple myeloma bone marrow. Cancer Immunology Research. 5 (9), 744-754 (2017).
- Psaila, B., et al. Single-cell profiling of human megakaryocyte-erythroid progenitors identifies distinct megakaryocyte and erythroid differentiation pathways. Genome Biology. 17 (1), 83 (2016).
- Schulte, R., et al. Index sorting resolves heterogeneous murine hematopoietic stem cell populations. Experimental Hematology. 43 (9), 803-811 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados