Method Article
분자 특성 조합 단일 셀 접근 및 인간의 줄기 및 조상 인구 Immunophenotypic이
요약
대량 유전자 식 측정 클라우드 개별 셀 차이 이종 세포 인구에서. 여기, 우리는 프로토콜 설명 어떻게 단일 셀 유전자 표정 분석 및 인덱스에 대 한 분류로 개화 활성화 셀 정렬 (FACS)이 윤곽을 그리 다 결합 될 수 있다 및 immunophenotypically 특성 분자로 뚜렷한 세포 인구.
초록
Immunophenotypic 특성 및 분자 분석이 윤곽을 그리 다 뚜렷한 세포 인구를 정의 하 오랫동안 사용 되어 왔습니다. 그러나 FACS은 본질적으로 단일 세포 분석 실험, 분자 분석 하기 전에, 대상 셀은 종종 prospectively 절연 되어 대량, 따라서 단일 셀 해상도 잃고. 단일 셀 유전자 표정 분석 이종 세포 인구에 있는 개별 셀 사이의 분자 차이 이해 하는 수단을 제공 합니다. 대량 세포 분석에서 고유한 셀 형식의 overrepresentation 생물 학적 중요성으로 편견 및 희소 한 세포에서 신호 않기에 결과. FACS 인덱스 정렬 단일 셀 유전자 표정 분석을 결합 하 여 인구 조사 수 단일 셀 해상도의 손실 없이 중간 세포 표면 마커 식 세포 또한 점령 하 고, 하는 동안 평가 활성화 연속 표면 마커 식의 관련성입니다. 여기, 우리는 단일 셀 반전 녹음 방송 결합 하는 접근 방식을 설명 정량 PCR (RT-정량) 및 FACS 색인 동시에 분자의 특성을 정렬 하 고 셀 인구 내의 immunophenotypic이.
단일 셀 RNA 시퀀싱 방법, 달리 특정 대상 증폭 정량 Pcr 사용 하 여 동안 수 있습니다 적은 낙후, 낮은 풍부 성적표의 강력한 측정 셀 변화에 읽기에 관련 된 문제에 의해 혼동 하지는 깊이입니다. 또한,에 의해 직접 인덱스 정렬 단일-세포 세포의 용 해로,이 방법은 버퍼 수 있게 cDNA 합성 및 특정 대상 사전 증폭 세포 표면 분자 서명 이후에 파생된의 상관 관계 뿐만 아니라 한 단계에서 수행할 수 표식 식입니다. 기술된 접근, 조 혈 단일 세포를 조사 하지만 또한 다른 세포 유형에 성공적으로 사용 되었습니다 개발 되었습니다.
끝으로, 여기에 설명 된 접근 분자로 별개 부분 모집단의 후속 예비 격리 프로토콜을 개발 하 패널에 대 한 가능성과 함께 미리 선택 된 유전자의 mRNA 표현의 중요 한 측정에 대 한 수 있습니다.
서문
각 개별 혈액 세포 어디 줄기 세포 형성 결국 말기 특정 들고 최종 효과 기 세포로 분화 하는 점점 더 최선을 다하고 중간 창시자의 시리즈 위에 정점 세포 계층 구조에서 것으로 생각 됩니다. 생물 학적 기능1. 줄기 세포 시스템 구성 방식에 대 한 지식을 많이 크게 능력을 고도로 농축 줄기 세포 또는 다양 한 창시자2 가지 조 혈 인구 prospectively 분리 때문에 조 혈 시스템에 생성 된 FACS 정렬. 이 분석 기능 또는 분자로, 주로 유전자 발현 프로 파일링3,4를 통해 이러한 인구의 많은 수 있다. 그러나 때 대량 인구 개별 차이 세포의 유전자 발현을 분석은 평균 밖으로 하 고5를 잃었다. 따라서, 이기종 셀 분수 내 셀 변화를 감지 하는 무능 경우 셀의 작은 하위 집합 그 인구6,의 유추 생물 학적 기능에 대 한 중요 한 생물 학적 과정에 대 한 우리의 이해 혼동 수 있습니다. 7. 반대로, 유전자 식 서명 단일 셀 해상도 조사가 윤곽을 그리 다 셀8overrepresented 하위 집합에서가 영향을 회피 하는 가능성을 제공.
현재까지 단일 셀 유전자 표정 분석을 위한 많은 프로토콜 개발 되었습니다; 각 접근 자체 주의 데. 가장 이른 방법은 이었다 RNA 형광 제자리에서 교 잡 (RNA-물고기), 한 번에 제한 된 수의 성적을 측정 하지만 독특한 RNA 지역화9,11의 조사에 대 한 수 있습니다. 단일 또는 거의 기록 했다 또한 감지를 PCR과 정량 Pcr를 사용 하 여 초기 방법 개발12. 그러나,이 요즘 대체 되었습니다 동시에 정량 셀의 수백에 셀 당 증명서의 수백의 식을 분석 하 고 따라서 높은 차원이 분석을 사용 하 여 허용 수 있는 마이크로-기반 방법으로 사전 결정 유전자 패널10,13. 최근 RNA 시퀀싱 기반 기술을 널리 사용이 될 단일 세포 분석을 위해 이러한 이론적으로 수 셀의 전체 transcriptome 측정 하 고 그로 인하여 추가 답사 차원이 분석10, 14. Multiplexed 정량 분석 및 단일 셀 RNA 시퀀싱 다른 기능을가지고, 따라서 방법 중 하나를 사용 하 여에 대 한 근거는 타깃 셀의 수 뿐만 아니라 질문에 따라 달라 집니다. 높은 처리량 및 단일 셀 RNA 시퀀싱의 편견, 탐구 특성 함께 셀 당 저가 알 수 없는 셀 또는 큰 인구 조사 때 바람직한. 그러나, 단일 셀 RNA 시퀀싱은 또한 낮은 풍부와 성적 증명서는 중단 하는 경향이 높은 풍부한 성적표를 더 자주 시퀀싱으로 편 파. 이 종종 미묘한 또는 기술적인 잡음15에 숨겨진 중요 한 분자 신호를 공개 bioinformatic 분석에 높은 수요를 상당히 복잡 한 데이터 발생할 수 있습니다. 따라서, 잘 특징이 조직에 대 한 단일 셀 정량 분석을 사용 하 여 미리 결정 뇌관 패널 기능적으로 중요 한 유전자 또는 분자 마커 선택의 질을 결정 하는 민감한, 간단한 접근 될 수 있는 인구입니다. 그러나, 주목 해야 한다 그에 비해 단일 셀 RNA-seq, 셀 당 비용은 단일 셀 정량 방법에 대 한 일반적으로 더 높은. 여기, 우리가 결합 하 여 단일 셀 실시간 정량 Pcr (Teles J. 그 외 여러분 에서 수정 하는 방법 설명 16), FACS 인덱스 인구 내에서 분자와 immunophenotypic이 특징에17 및 생물 정보학 분석18 를 동시에 정렬.
이 방법에서는, 관심사의 세포 인구, 스테인드 그리고 단일 세포 세포의 용 해 버퍼 96 잘 PCR 접시에서에 직접 FACS에 의해 정렬 됩니다. 동시에, 세포 표면 마커 추가 집합 식 수준의 FACS 라고 인덱스 정렬 방법-정렬, 하는 동안 각 단일 셀에 대 한 기록 됩니다. Lysed 세포 물자는 연속적으로 증폭 하 고 실시간 정량, 미세 플랫폼을 사용 하 여 선택된 된 유전자의 유전자 발현 분석. 이 전략에는 각 개별 셀의 세포 표면 마커 식의 정렬 된 단일 셀으로 동시 특성화의 분자 분석 수 있습니다. 직접 인덱스 정렬 된 표시자의 식 세포의 분자로 고유 하위 집합을 매핑하여,를 모집단 그들의 잠재 격리에 대 한 사용할 수 있는 특정 immunophenotype에 연결할 수 있습니다. 메서드를 설명 하는 그림 1에서 단계적으로. 미리 정해진된 유전자 패널 더 미묘한 유전자 식 신호를 가리고 그렇지 않으면 수 없는 풍부한 유전자의 측정 circumvents 이후 타겟된 유전자 발현의 높은 해상도에 기여 한다. 또한, 특정 대상 확대, 한 단계 반전 녹음 방송, 그리고 증폭 녹음 방송 요인 또는 비 폴 리 adenylated RNAs 같은 낮은 표현된 성적표의 강력한 측정에 대 한 수 있습니다. 중요 한 것은, 정량 방법에서 특정 악성 질병19를 조사할 때 중요 한 융해 단백질 mRNA의 측정에 대 한 수 있습니다. 마지막으로, 유전자의 집중된 수 조사, 낮은 드롭 아웃 요금, 및 제한 된 셀 확인이이 방법은 쉽게 분석 기술 차이 단일 셀 RNA-이 같은 높은 차원 방법에 비해 프로토콜에 따라, 전체 실험 수행할 수 있습니다, 정렬 셀 분석된 결과를 3 일 이내 중요 한, 높은 처리량 단일 셀 유전자 표정 분석에 대 한 단순 하 고 빠른 방법 만들기에서.
프로토콜
1입니다. 세포 접시의 준비
- 390 µ L nuclease 무료 물, 10%의 17 µ L를 혼합 하 여 10% 추가, 96 웰 스에 대 한 충분 한 세포의 용 해 버퍼 준비 RNA/DNA 무료 벤치를 사용 하 여 NP-40, 2.8 µ L 10 mM dNTP, 10 µ L 0.1 M DTT와 5.3 µ L RNAse 억제제 ( 재료의 표참조). 와 동 그리고 아래로 회전입니다.
- 96 잘 PCR 플레이트의 각 음에 세포의 용 해 버퍼의 4 µ L 고 접착 필름으로 접시를 봉인. 접시의 바닥에 액체를 수집 하는 튜브 아래로 회전 합니다. 셀 정렬 (최대 24 시간)까지 얼음에 플레이트를 유지.
2입니다. 셀 정렬 셀의 준비
- 적절 한 수의 실험에 대 한 (여기, CD34 풍부한 조 혈 줄기와 조상 세포) 셀 동 1 x 106 세포는 약 3 96 잘 접시 컨트롤에 포함 된 단일 셀의 정렬 적합 합니다.
- 15 mL 원뿔 튜브에 해 동된 셀을 전송 하 고 1 mL FBS 모든 30 추가 s 8 mL의 총 볼륨에 도달할 때까지. 셀 4 ° C에서 10 분 동안 350 x g 에서 원심 분리기에서 회전 하 고 상쾌한을 제거 합니다.
- Resuspend 8 mL 버퍼 얼룩에 셀 (PBS 2 %FBS) 4 ° C에서 10 분 동안 350 x g 에서 원심과 상쾌한 제거.
- 200 µ L 얼룩에서 resuspend 셀 버퍼링 하 고 셀 제어 얼룩을 제거.
- 50에 세포의 일부분을 얼룩에 의해 한 컨트롤 (FMOs) 각 fluorophore 마이너스 형광을 확인 µ L 버퍼 얼룩. 이 예제에서 20000 셀 6 microcentrifuge 튜브는 FMOSs로 사용 됩니다. 참고 셀의 수는 인구 조사에 따라 조정 되어야 한다. 각 튜브를 하나 제외 샘플 얼룩 처럼 동일한 농도에서 모든 항 체를 추가 합니다.
- 각 fluorophore 사용에 대 한 50 µ L 버퍼에 셀의 일부를 착 색 하 여 각 fluorophore에 대 한 하나의 얼룩을 확인 합니다. 이 예제에서 20000 셀 6 microcentrifuge 튜브 사용 됩니다. 참고 각 항 체에 대 한 대상 컨트롤에 사용 되는 세포에 의해 표현 될 필요가. 개별 튜브에서 샘플 얼룩 처럼 동일한 농도에서 각 항 체를 추가 합니다. 또한 흠 없는 컨트롤로 50 µ L에 20000 흠 없는 셀을 계속.
- 셀 샘플을 그들의 적절 한 농도에서 항 체를 추가 합니다. 사용 1/100 농도, CD38-APC 1/50, CD90-PE 1/10, CD45RA-bv421 1/50, CD49F-PECy7 1/50 및 혈통 믹스에서 CD34-FITC 운항: CD3-PECy5 1/50, c d 2-PECy5 1/50, CD19-PECy5 1/50, CD56-PECy5 1/50, CD123-PECy5 1/50, CD14-PECy5 1/50, CD16-PECy5 1/50, 및 CD235a-PECy5 1/1000입니다.
- 어둠 속에서 얼음에 30 분을 위한 항 체와 세포를 품 어.
- 셀 3 mL 버퍼 얼룩이 씻어. 4 ° C에서 10 분 동안 350 x g 에서 세포를 원심 고 상쾌한을 제거 합니다.
- Resuspend 셀 고 2.9 단계를 반복 합니다.
- 500 µ L에서 샘플 및 100에서 FMOs resuspend µ L 버퍼 1/100 7AAD 및 필터 세포를 단일 세포 현 탁 액을 50 µ m 필터를 통해 얼룩.
3. 셀 정렬
- FACS 기계 설정 되어 있는지 확인 드롭 지연 및 cytometer 설치 및 추적 (중부 표준시) 적절 한 셀 정렬 있도록 제조업체의 지침에 따라 수행 되었습니다 최근 그와 제대로. 조 혈 모 세포에 대 한 4의 85 미크론 노즐과 최대 속도의 사용은 권장, 최적의 이벤트 속도가 800 및 2000 이벤트/s 사이.
- 형광 보상을 수행 하 여 스펙트럼 중첩에 대 한 수정 하 고 게이츠 FMO 컨트롤 또는 내부 부정적인 컨트롤 설정.
- 대상 인구의 해도 얼룩 버퍼의 100 µ L 가진 새로운 microcentrifuge 관으로 적어도 100 대상 셀을 정렬 하 여 수행 합니다. FACS 정렬된 셀 정렬된 샘플을 기록 하 여 분석 하 고 그들은 정렬 게이트에서 결국 다는 것을 확인 한다.
- 설정 단일 셀 플레이트 96 잘 접시에 잘 A1 드롭 중심으로 정렬. 중심은 50-100 6 µ m 입자 모든 우물 우물 모든 셀의 각 센터에 얻을 것 이다 있도록 빈 96 잘 접시의 가장자리 주위에 정렬 됩니다.
- 만약에 가능 하다 면, 가능한 셀 정렬 되도록 추가 제어 생체 외에 성장에 대 한 단일 셀을 정렬 하 여 추가할 수 있습니다. Haematopoietic 셀 FLT3L, TPO, SCF 1% 페니실린 스, 100 ng/mL로 100 µ l SFEM에서에서 U-하단 96 잘 접시에서 자란 수 있습니다. 3 일 후에 문화는 현미경을 사용 하 여 셀 식민지를 위한 각 잘 분석.
- 플레이트에서 접착 필름을 제거 합니다. 96 웰 스에서 92에 (여기에 린-CD34 + CD38-세포)의 단일 셀 정렬 각 단일 셀에 대 한 (여기 CD45RA, CD49f, 및 CD90) 다른 표식에 대 한 immunophenotypic 프로필을 저장 하는 소프트웨어를 정렬 FACS에 인덱스 정렬 활성화 합니다.
- 10 및 20 각각 선형성 컨트롤 PCR 증폭에 대 한 세포와 2 개의 우물을 정렬 합니다. 웰 스 h 1과 h 2 일반적으로 사용 됩니다.
- 아니오-템플릿 컨트롤, 일반적으로 우물 H3 및 H4 셀 없이 2 개의 우물을 유지.
- 명확한 접착제 필름으로 접시를 봉인 하 고 스냅 드라이 아이스에 냉동 하기 전에 1 분 동안 300 x g 에서 접시를 회전.
- -80 ° c.에 냉동된 번호판을 저장
참고: 안전 정지 지점입니다. 정렬 및 lysed 세포는 장기 저장을 위한-80 ° C에 보관 수 있습니다.
4. 반전 전사 및 특정 대상 확대
- 등 내부 관리 유전자 뇌관 뇌관 제어용 아군에서 RNA, RNA/DNA 무료 벤치에 RNAse 무료 튜브 1.5 mL에 각 뇌관 쌍의 2 µ L을 추가 하 여 모든 96 유전자 표적을 위해 뇌관 믹스를 준비 합니다. 미만 96 뇌관을 사용 하 여, nuclease 무료 물 없는 뇌관에 대 한 해당 볼륨을 추가 합니다. 프라이 머는 별도로 원하는 유전자 패널에 맞게 정렬 됩니다.
- 반전 녹음 방송 및 혼합 반응 혼합 x 632.5 µ L 2, 101.2 µ L을 추가 함으로써 특정 대상 확대 Taq/SuperscriptIII, 151.8 µ L 뇌관 믹스, 그리고 0.7 µ L 제어 RNA에에서 아군. 이 단계는 DNA 무료 벤치에 예비적 형성. Vortexing와 수집 튜브의 하단에 있는 액체 아래로 회전 급강하에 의해 혼합. 샘플에 추가 될 때까지 얼음에 계속.
- 반응 혼합 x 2의 27.5 µ L, Taq 효소의 1.76 µ L, 뇌관 믹스의 6.6 µ L, nuclease 무료 물 2.64 µ L를 혼합 하 여 4 개의 우물에 대 한 만들기 없음 반전 전사 제어 믹스. 제어 RNA에에서 스파이크. DNA 무료 벤치에이 단계를 수행 합니다. 와 동 그리고 샘플 수집 튜브 및 추가까지 얼음에 계속의 하단에 있는 액체 아래로 회전.
- Lysate 번호판 얼음에 녹여 92 웰 스, 선형성 및 아니오-템플릿 컨트롤을 포함 하 여 이전에 준비 된 반전 녹음 방송 및 특정 대상 확대 믹스 8.75 µ L를 추가 합니다. 4 나머지 우물을 아니 반전 전사 제어 믹스의 8.75 µ L를 추가 합니다. 명확한 접착제 필름 및 수집 액체 접시의 바닥에 아래로 회전 접시를 봉인.
- 반전 녹음 방송 및 특정 대상 증폭 앰프 프로그램;에 따라 PCR 기계에서 접시를 실행 하 여 예비적 형성 단계 1시 50분 ° C 60 분, 2 분 동안 2: 95 ° C 단계, 단계 3: 95 ° C 15에 대 한 s, 단계 4: 60 ° C 4 분, 반복 단계 3-4 24 시간 및 마지막으로 영원히 5: 8 ° C 단계.
- PCR를 완료 한 후 단기 저장을 위해 8 ° C 및 장기 저장을 위한-20 ° C에서 접시를 유지.
참고: 안전 정지 지점입니다. 단기 저장을 위해 8 ° C에 그리고 장기 저장을 위한-20 ° C에서 증폭 된 자료를 보관할 수 있습니다.
5. 다중 미세 유전자 표정 분석에 대 한 샘플 및 시험 접시의 준비
- 분석 결과 pipetting 3 접시를 로드 준비 µ L 분석 결과 로딩 시 약 96 잘 접시의 각 음에. 플레이트 로드 분석 결과에서 개별 우물에 각 뇌관의 3 µ L를 추가 합니다.
- 접착 필름 및 플레이트의 하단에 수집 액체 아래로 회전 접시를 봉인.
- 준비 희석 플레이트 nuclease의 pipetting 8 µ L에 의해 무료 물 96 잘 접시의 모든 우물에. 1: 5의 최종 희석 하는 희석 접시에 증폭 된 샘플의 2 µ L를 추가 합니다.
- 접착 필름 판 인감, 10 vortexing 격판덮개에 의해 혼합 s, 그리고 마지막으로 수집 액체 접시의 바닥에 아래로 회전.
- 샘플 로드 믹스 마스터 믹스의 352 µ L의 샘플 로딩 시 약 35.2 µ L 신중 하 게 혼합 하 여 준비 합니다. Aliquoting 3.3 µ L 로드 96 잘 접시의 각 잘 혼합 하 여 샘플 로드 접시를 준비 합니다.
- 접시를 로딩 하는 샘플의 각 음에 희석된 샘플에서 2.7 µ L를 추가 합니다.
- 접착 필름 및 플레이트의 하단에 수집 액체 아래로 회전 접시를 봉인.
6. 미세 칩의 로드
- 새로운 96 x 96 미세 칩을 가져가 라. 파고 그들 모자와 주사기에 이동 될 수 있다 다는 것을 확인 하 여 후미를 준비 합니다.
- 주사기에서 거품을 제거 합니다. 기울이기 칩 45도 하면서 밸브를 누르고 각 밸브를 주사기의 전체 볼륨을 추가 합니다. IFC 컨트롤러 칩을 프라임.
- 플레이트 로드 분석 결과에서 우물의 각에서 각 분석 결과 입구 4.25 µ L로를 로드 합니다. 거품을 하지 마십시오. 거품에는 잘 표시, 피 펫 팁 그들을 제거 합니다.
- 플레이트 로드 샘플에 우물의 각에서 각 샘플 입구 4.25 µ L로 로드 continue, 거품을 방지 하 고 거품 표시, 피 펫 팁 그들을 제거.
- IFC 컨트롤러 칩을 로드 합니다.
- 칩도 외모와 모든 챔버 로드 된 확인 하십시오. 테이프와 그것으로 칩 표면에서 먼지를 제거 합니다. 멀티플렉스 미세 유전자 식 플랫폼에 칩을 실행 합니다.
7. 다중 미세 유전자 식 플랫폼에 칩 실행
- 멀티플렉스 미세 유전자 식 플랫폼으로 칩을 로드 한 후 샘플을 이름을 지정 합니다.
- 이용한 수동 염료로 설정 합니다. 형광으로 단일 프로브 및 팸-MGB를 설정 합니다. 96 x 96 표준 v 2를 사용 하 여 프로토콜. 실행 시작.
- 제거 칩 실행 하면 완료 됩니다.
8. 예비 분석 칩 실행의
- 실시간 PCR 분석 소프트웨어에 데이터를 로드 합니다.
- 세포 및 유전자 레이아웃 탭으로 구분 된 파일에서 풀 칠해서 유전자 이름 및 셀 이름을 로드 합니다.
- 이미지 보기를 열고 고 염료를 이용한 선택 합니다. 모든 웰 스를 이용한 수동 염료 있다면 확인 하십시오.
- 모든 증폭 플롯 확인 ( 그림 3E비슷합니다) 없이 스파이크와 부드러운 증폭 곡선으로 본다면 조사.
- 모든 단일 세포 아군에 제어 모두 제대로 로드는 다는 것을 확인 하는 RNA의 식을 확인 합니다.
- 모든 셀 내부 관리 유전자 발현 있고 따라서 제대로 정렬 된 것을 확인 합니다.
- 10 및 20 셀 선형성 제어 선형 증폭을 확인 하기 위해 약 1 CT 차이가 있는지 확인 합니다.
- NoRT 컨트롤 샘플 식 인지 확인 합니다. 식을 noRT에 감지 되 면 후속 실행에 대 한 게놈 DNA를 검색 하지 않습니다 프로브에 프로브를 변경 하는 것이 좋습니다.
- 추가 분석을 위해 csv 파일에서 데이터를 내보냅니다.
9. 단일 세포 분석 SCExV를 사용 하 여
참고: 입문 영화 존재20 도구를 소개 하는. 여기, 프로토콜에 도입 하는 컨트롤을 사용 하 여 분석을 수행 하는 방법의 짧은 추천 제공 됩니다.
- SCexV 웹사이트20에 연결 합니다.
- 내보낸된 CSV 파일을 업로드.
- 긍정적인 통제로 제어 RNA에서 아군을 선택 했다. 25 위 제어 RNA CT는 모든 셀을 제거 합니다. 중간 식 제어 RNA의 데이터를 정상화.
- "다 여기-> 분석"을 클릭 합니다. 제외 셀 옵션에서 noRT, notemplate, 10 및 20 셀 컨트롤을 제거 합니다.
- 클러스터의 예상된 번호 선택의 클러스터링 방법으로 클러스터 셀. 분석된 값을 내보냅니다.
10. 인덱스 정렬 분석
- FACS 분석 소프트웨어 열고 인덱싱된 샘플을 로드 합니다.
- 스크립트 편집기를 열고 퀸 제이 외 에서 사용할 수 있는 스크립트를 실행 21
- FACS 분석 소프트웨어 단일 셀의 각 게이트를 만든 해야, 레이아웃 편집기를 열고 고 ("Sample_complete_Data.xls" 라는 파일에서 사용할 수 있는) SCexV에서 그룹화에 따라 셀 색.
결과
설명 하는 프로토콜은 빠르고 쉽게 수행 하 고 신뢰할 수 있는. 실험 설정에 대 한 개요는 그림 1에 표시 됩니다. 전체 프로토콜 특정 대상 확대, 유전자 식 측정 및 예비 분석 단일 셀의 정렬에서 3 일 동안에서 수행할 수 있습니다. 96 뇌관 및 만성 골수성 백혈병 (CML) 환자에서 96 셀 이나 건강 세 일치에서 96 셀을 사용 하 여 단일 셀 유전자 표정 분석에서 예비 분석 된 데이터를 나타내는 열 지도 형태로 분석된 결과의 예 제어는 그림 2에 표시 됩니다. 계층적 클러스터링을 사용 하 여, 분석 된 세포는 그들의 유전자 식 서명 기반 4 개의 하위 그룹으로 나눌 수 있습니다. 열 지도 시각화 분석 (예를 들어, 제어 우물)에서 제외 해야 하는 웰 스에 대 한 제어로 뿐만 아니라 데이터에 대 한 개요를 얻을 수 있는 편리한 방법입니다. 그림 2B 는 얼마나 비슷한 각 그룹의 세포는 서로 차원 감소를 사용 하 여 시각화 하는 주성분 분석 (PCA). 여기, outliers 셀의 나머지 부분에서 쉽게 고유 있습니다. 분석을 어떻게 개별 유전자 클러스터 간에 분산으로 그들 사이 다, 바이올린 플롯은 유용 하다. 그림 2C, 4 개의 대표적인 유전자의 표현을 표시 됩니다: 융합 유전자 BCR-ABL1, 표시 모든 leukemic 세포; 적극적으로 나누어 그룹;에서 표현 되는 세포 주기 마커 mKI67 골수성 분화 마커 MPO는 자전거 네; 그리고 마지막으로 집 유지 유전자 RPS18, 모든 셀에 표시 됩니다. 마지막으로, 그림 2D 분자 서명 immunophenotyped에 상관 되어 있다, 각 클러스터에 있는 각 셀의 id로 사용 됩니다 인덱스 정렬에서 생성 된 FACS 플롯에서 개별 셀을. 표시 된 CD90 및이 경우에 클러스터의 일부 구분 및 prospectively 기능 분석에 대 한 격리를 사용할 수 있는 CD45RA 조 혈 줄기 세포 표식에 대 한 세포 표면 마커 식이입니다.
단일 셀 정량 Pcr을 수행 하는 데 사용 하는 마이크로 시스템에 공기 또는 입자의 도입에 아주 과민 하다. 따라서, 그것은 데이터의 게시물 실행된 품질 검사 중요입니다. 그림 3 데이터와 성공와 실패는 가난한 샘플 로드 인해 실행 대표를 보여준다. 그림 3A 어떻게 성공적인 실행 후 확산 균등 하 게 모든 우물을 안심 위에 로드를 이용한 레벨 성공 했다 보여줍니다. 반면, 그림 3B 어떻게 샘플 및 분석 결과 로딩 실패를 이용한 칩의 큰 부분에의 부족에 의해 표시 된 대로 보여 줍니다. 이용한의 품질을 평가 하는 일단의 원시 유전자 식 신호 품질을 평가할 수 있습니다. 그림 3C, 성공적인 실행의 열 지도 제시, 주위의 강한 신호를 샘플링 어디 식 균등 하 게 확산 밖으로 7-25 Ct:s.에 대비, 3D 그림 표시 실패 한 실행의 열 지도 많은 샘플 있는 신호 또는 매우 제한 된 양의 유전자의 표현입니다. 그림 3d에서 결과 때문에 많은 세포 분명 식 신호 다른 완전히 식 부족 하는 동안 이전에 단일 셀 유전자 표정 분석, FACS 정렬 오류로 인해 높습니다. 마지막으로, 거의 아무 변이 약 10 CT의 명확한 표현 하는 데 모든 우물 그림 3E 아군에 제어 RNA의 예상된 증폭 곡선을 표시 됩니다. 이 긍정적인 컨트롤 단일 셀 정량 분석에서 모든 단계의 효율성을 측정합니다. 증폭 동적 범위는 되도록 다른 핸드폰 번호는 선형성 컨트롤;으로 포함 여기, 10 및 20 셀 컨트롤 사용 됩니다. 선형성 컨트롤의 CT 차이점 셀 숫자의 차이 반영 해야 합니다. 마지막으로, 아무 역 전사 (noRT) 및 아무 템플릿 부정적인 컨트롤 아무 거짓 긍정적인 신호 게놈 DNA 또는 오염에서 각각 검색 됩니다 확인 합니다.

그림 1: 프로토콜의 회로도 보기. 출시는 mRNA 인덱스 정렬 응용 표면 마커 식 기록 활성화 및 lysed 정렬 셀은 얼룩이 진다. mRNA는 이후 역 cDNA를 변하게 하 고 특정 대상 확대를 사용 하 여 증폭. 다음으로, 샘플 미세 칩에 개별 뇌관 함께 로드 됩니다 실시간 정량 Pcr를 사용 하 여 각 셀의 유전자 식 프로필을 분석 하는 곳. 데이터 수집 후 데이터 전처리 된 낮은 질 세포를 제거 하 고 분자로 뚜렷한 세포 인구를 정의 하는 클러스터링 수행. 마지막으로, 분자로 정의 된 모집단 상관 표면 마커 식 FACS 인덱스 정렬에서 데이터 immunophenotypically 인구가 특징. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
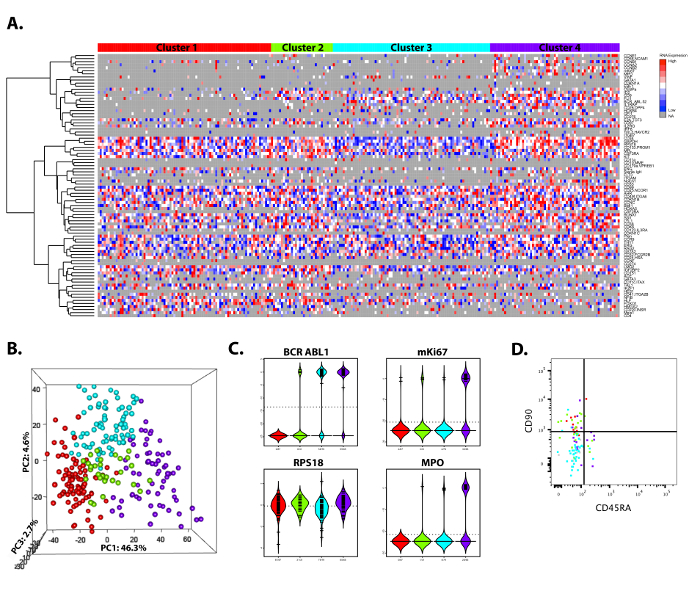
그림 2: 정상과 leukemic 조 혈 줄기 세포의 3 개의 성공적인 단일 셀 유전자 발현의 대표적인 결과 분석. (A) 열 식 측정 한 나이 일치 하는 건강 한 제어 SCExV 18, 분석 도구를 사용 하 여 계층적 클러스터링에 의해 분석 한 만성 골수성 백혈병 환자에서 270 단일 셀의 단일 셀 유전자를 보여주는 지도 데이터의이 유형에 대 한 설계 되었습니다. 레드 블루 낮은 식 높은 식 나타내고 식이 없는 회색. 세포는 그들의 유전자 식 서명을 기반으로 4 개의 다른 클러스터로 분할 되었다. 그것은 분명이 환자에서 줄기 세포의 leukemic 인구만 몇 분화 마커의 식이 한 무부하 인구 (클러스터 3), 그리고의 식을 시작 했습니다 한 적극적으로 나누어 인구에 분할 될 수 있다 데이터에서 골수성 차별화 (클러스터 4)와 관련 하는 유전자 여기에 사용 되는 데이터는 Warfvinge 그 외 여러분 에 의해 이전에 게시 작업에 포함 선택 된 환자에서 22 (B) 주 A, 4 그룹을 구분 하는 어디에 표시 되는 데이터의 구성 요소 분석 (PCA) 표시 될 수 있습니다. (C) 바이올린 플롯 클러스터 특정 식, 4 개의 유전자의 BCR-ABL1, mKI67, RPS18 및 MPO. 각 바이올린 플롯 각 클러스터에 식을 나타내고 걸쳐 점선 식 모든 셀의 평균 값을 나타냅니다. (D) 각 개별 셀의 분자 부분 모집단의 색상으로 표시 된 열 지도에 표현 하는 환자 샘플 중 하나의 인덱스 정렬 분석. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3: 다중 정량 실시간 PCR 소프트웨어에서의 품질 컨트롤. (A)를 이용한 로딩 이미지 성공적인 샘플 및 분석 결과 로딩. (B)를 이용한 실패 샘플 및 로드 분석 결과의 이미지 로드, 어디 샘플의 실패 한 선적 수평 라인에를 이용한의 부족에 의해 표시 됩니다. 분석 실험의 실패 한 로드 수직 라인 표시 될 것 이다. 성공의 (C) 예 열 지도 보기, 행을 대표 하는 단일 세포 및 유전자 대표 열에서에서 실행 합니다. 높은 유전자 발현 파란색에서 노란색, 낮은 식과 블랙에 없는 감지 식에 표시 됩니다. 실패는의 (D) 예 열 지도 보기, 행을 대표 하는 단일 세포 및 유전자 대표 열에서에서 실행 합니다. 높은 유전자 발현 표현 노란색, 낮은 식 파란색과 검은색으로 아무 검색된 식에 의해 표현 됩니다. (E) 정규화 강도 (왼쪽) 고 아군에서 RNA 제어의 증폭 플롯 (오른쪽). Ct 값 약 10 및 한정 된 변이 가진 곡선의 성공적이 고 강력한 반전 녹음 방송, 사전 증폭 및 정량 분석을 모든 우물 내에서 나타냅니다. (F) 정규화 (왼쪽)와 실패 한 유전자의 증폭 플롯 (오른쪽). 정규화 된 농도 곡선을 형성 하지 않는다 하 고 증폭 플롯 형광에 큰 스파이크를 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
토론
최근 몇 년 동안, 단일 셀 유전자 표정 분석은 다양 한 세포 인구23의 정의에 가치 추가 되었다. 그러나 이러한 메서드는 셀 시퀀싱 깊이 탈락에 의해 복잡 하 게 RNA 시퀀싱 기술의 출현 이론적으로 셀의 전체 transcriptome 측정 하는 가능성을 제공 합니다. 단일 셀 정량 제공 중요 한 유전자에 있는 모든 세포의 수백의 식의 과민 하 고 강력한 분석 기술 노이즈 감소 마찬가지로 처리 됩니다. 성적 증명서의 제한 된 수의 집중된 분석 또한 매우 표현된 유전자에서 간섭 없이 단순화 된 분석을 위해 수 있습니다.
만 셀에 표현 하는 유전자의 하위 집합을 조사 하는 접근 그것 이므로 매우 유전자 패널을 설계 할 때 유전자의 정확한 세트를 선택 하는 것이 중요. 유전자의 선택; 여러 가지 방법으로 할 수 있습니다. 통해 문학, 대량 분석 및 공개적으로 사용 가능한 데이터의 생물 정보학 분석에 의해 이전 연구 결과. 이것은 간단 합니다. 그러나, 유전자의 정확한 세트 접근은 매우 강력한 수 있습니다. 유전자 패널 설계 되었습니다, 그것은 뇌관 멀티플렉싱 하는 동안 방해 하지는 유효성을 검사 하는 것이 중요입니다. 여기 좋습니다 분석 뇌관 preforming 충분 한 소재; 희석 시리즈 선형은 경우 이 연구에 사용 된 뇌관의 시리즈 보충 에서처럼 희석 Warfvinge 외 에 2D 그림 22
여기에 제시 된 프로토콜의 두 가지 주요 제한 셀과 유전자 조사의 제한 된 수 있습니다. 그러나,만 96 셀 각 실행에서 조사는, 비록이 비교적 간단한 프로토콜은 하루에 완료 될 수 있다 고 따라서 셀의 수백 1 주일에 분석 될 수 있다. 또한, 더 뇌관 특정 대상 사전 증폭 단계에 포함 된 경우 조사 하는 유전자의 수를 증가 될 수 있습니다. 이 경우에, 여러 칩 96 뇌관의 각 집합을 조사 하기 위해 필요 합니다.
높은 유전자 또는 세포를 표현 하는 경우 높은 RNA-콘텐츠 분석 되, 낮은 전반적인 유전자 식23 조 혈 줄기 세포에 대 한 최적화 하는 사이클의 수를 수정 해야 합니다. 대량 인구를 조사할 때에 확대 주기를 변경도 중요 하다. 조 혈 줄기 세포 (100)의 대량 분석 18 25에서 사전 증폭의 양을 줄이는 것이 좋습니다.
가장 일반적인 이유는 실패에 대 한 실행은 실패 한 세포 격판덮개의 우물으로 단일 셀의 정렬 가능한 비 셀, 차선의 사전 증폭, 또는 실패를 로드 하는 칩의 정렬. 각 반응 챔버에서를 이용한 수준 분석 결과, 또는 샘플의 로드의 표시기 이며이 낮은 경우에, 것이 좋습니다 샘플 레벨은 로드 하는 동안 거품의 도입으로 발생 가능성이 낮은 이용한 이후 새로운 칩에서 다시 실행 됩니다. 아군에서 제어 RNA 식의 부족은 그 사전 앰프 때문에 실패, 가능성이 문제 사전 증폭-또는 반전 녹음 시 약. 이 경우에, 새로운 칩은 새로 정렬된 셀 및 새로운 반응 실행 되도록 하는 것이 좋습니다. 제어 RNA의 검출 하지만 다른 유전자 발현의 징후 실패 단일 셀 FACS의 표시 유전자 표정 분석 전에 정렬 신호. FACS에 정렬 하는 동안 오류가 발생 하지 않도록 하려면 단일 세포 각 우물의 중앙으로 정렬 됩니다 있도록 중요 하다. 이 빈 접시에 비즈를 정렬 하 고 시각적으로 물방울 토지 검사 하 여 수행할 수 있습니다. 테스트-정렬 되도록 설정 하지만 처음 몇 웰 스에 대 한 올바른 접시의 가장자리에 있는 모든 우물에 게 확인 하십시오. 해당 되는 경우 단일 셀 동시에 분류 될 수 있다 생체 외에 성장 하 고 성공적인 정렬 프로토콜에 대 한 제어를 정량 분석 하기 전에 분석. 인덱스 정렬 정보 분자로 별개 부분 모집단의 immunophenotyped 조사만 허용 하지 않습니다 하지만 문제 해결, 경우 정의 되지 않은 인구를 정렬 하는 많은 낮은-품질 셀 때 유용한 통찰력을 제공할 수 있습니다. 단일 셀 유전자 표정 분석에. 인덱스 정렬 정보 제어 전략을 최적화 하 고 품질이 낮은 세포의 미래 정렬 방지를 사용할 수 있습니다.
단일 셀 유전자 표정 분석 인구 조사는 어떻게 다른 유형의 셀을 혁신적 이며 세포 분화의 더 이해를 위한 방법을 포장. Haematopoiesis에서 단일 셀 유전자 표정 분석 multipotent 조상 인구6,,2425 의 해결 하려면 HSCs7의 하위 집합에 대 한 정렬 전략을 수정 하는 데 사용 되었습니다. 치료 구분 leukemic 세포 인구22,26를 식별 하 고. 단일 셀 진 식-및 기능 분석 정렬 하는 인덱스의 조합 이전 immunophenotype 및 다른 인구27, 의 조사할 때 안정적이 고 효율적인 것 보였다 28,29. 여기, 분자의 조사에 대 한 접근 인구 되었습니다 셀의 immunophenotypic이 어디 짧은에서 복잡 한 분석 없이 신속 하 고 재현성 결과 수 있습니다 단일 셀 정량 결합 인덱스 정렬 설명 기간입니다.
공개
저자는 공개 없다.
감사의 말
이 작품은 의료 연구, 스웨덴어 유년기 암 재단, 라 그 나 Söderberg 재단, 그리고는 크누트와 앨리스 발 렌 버그 재단에 대 한 스웨덴의 암 사회, 스웨덴 연구 위원회, 스웨덴의 사회에서 교부 금에 의해 지원
자료
| Name | Company | Catalog Number | Comments |
| CD14 PECY5 | eBioscience | 15-0149-42 | Clone: 61D3 |
| CD16 PECY5 | Biolegend | 302010 | Clone: 3G8 |
| CD56 PECY5 | Biolegend | 304608 | Clone: MEM-188 |
| CD19 PECY5 | Biolegend | 302210 | Clone: HIB19 |
| CD2 PECY5 | Biolegend | 300210 | Clone: RPA-2.10 |
| CD3 PECY5 | Biolegend | 300310 | Clone: HIT3a |
| CD123 PECY5 | Biolegend | 306008 | Clone: 6H6 |
| CD235A PECY5 | BD Pharma | 559944 | Clone GAR2 |
| CD34 FITC | Biolegend | 343604 | Clone: 561 |
| CD38 APC | Biolegend | 303510 | Clone: Hit2 |
| CD90 PE | Biolegend | 328110 | Clone: 5E10 |
| CD45RA BV421 | BD bioscience | 560362 | Clone: HI100 |
| CD49f Pecy7 | eBioscience | 25-0495-82 | Clone: eBioGOH3 |
| FBS | HyClone | SV30160.3 | |
| PBS | HyClone | SH30028.02 | |
| 96-well u-bottom Plate | VWR | 10861-564 | |
| SFEM | Stem cell technologies | 9650 | |
| Penicillin streptomycin | HyClone | SV30010 | |
| TPO | Peprotech | 300-18 | |
| SCF | Peprotech | 300-07 | |
| FLT3L | Peprotech | 300-19 | |
| Falcon Tube 15 mL | Sarstedt | 62.554.502 | |
| Eppendorph tube | Sarstedt | 72.690.001 | |
| CST beads | BD | 642412 | |
| Accudrop Beads | BD | 345249 | 6 µm particles |
| Adhesive film Clear | Thermo scientific | AB-1170 | |
| Adhesive film Foil | Thermo scientific | AB-0626 | |
| 96 well PCR plate | Axygen | PCR-96M2-HS-C | |
| PCR 1.5 mL tube | Axygen | MCT-150-L-C | |
| T100 PCR cycler | BioRad | 186-1096 | |
| 10% NP40 | Thermo scientific | 85124 | |
| 10 mM dNTP | Takara | 4030 | |
| 0.1 M DTT | Invitrogen | P2325 | |
| RNAsout | Invitrogen | 10777-019 | RNAse inhibitor |
| CellsDirect One-Step qRT-PCR Kit | Invitrogen | 11753-100 | |
| Neuclease free water | Invitrogen | 11753-100 | from CellsDirect kit |
| 2x Reaction Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| SuperScript III RT/Platinum Taq Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| Platinum Taq DNA Polymerase | Invitrogen | 10966026 | |
| TaqMan Cells-to-CT Control Kit | Invitrogen | 4386995 | |
| Xeno RNA Control | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 20x Xeno RNA Control Taqman Gene Expression Assay | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 96.96 Sample/Loading Kit—10 IFCs | Fluidigm | BMK-M10-96.96 | |
| 2x Assay Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| 20x GE Sample Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| Control line fluid | Fluidigm | From 96.96 Sample/Loading Kit | |
| TaqMan Gene Expression Master Mix | Applied Biosystems | 4369016 | |
| BioMark HD | Fluidigm | BMKHD-BMKHD | |
| 96.96 Dynamic Array IFC | Fluidigm | BMK-M10-96.96GT | |
| Excel | Microsoft | Microsoft | |
| FlowJo V10 | TreeStar | TreeStar | |
| Fluidigm real time PCR analysis | Fluidigm | Fluidigm | |
| CD179a.VPREB1 | Thermofisher scientific | Hs00356766_g1 | |
| ACE | Thermofisher scientific | Hs00174179_m1 | |
| AHR | Thermofisher scientific | Hs00169233_m1 | |
| BCR_ABL.52 | Thermofisher scientific | Hs03043652_ft | |
| BCR_ABL41 | Thermofisher scientific | Hs03024541_ft | |
| BMI1 | Thermofisher scientific | Hs00995536_m1 | |
| CCNA2 | Thermofisher scientific | Hs00996788_m1 | |
| CCNB1 | Thermofisher scientific | Hs01030099_m1 | |
| CCNB2 | Thermofisher scientific | Hs01084593_g1 | |
| CCNC | Thermofisher scientific | Hs01029304_m1 | |
| CCNE1 | Thermofisher scientific | Hs01026535_g1 | |
| CCNF | Thermofisher scientific | Hs00171049_m1 | |
| CCR9 | Thermofisher scientific | Hs01890924_s1 | |
| CD10.MME | Thermofisher scientific | Hs00153510_m1 | |
| CD11a | Thermofisher scientific | Hs00158218_m1 | |
| CD11c.ITAX | Thermofisher scientific | Hs00174217_m1 | |
| CD123.IL3RA | Thermofisher scientific | Hs00608141_m1 | |
| CD133.PROM1 | Thermofisher scientific | Hs01009250_m1 | |
| CD151 | Thermofisher scientific | Hs00911635_g1 | |
| CD220.INSR | Thermofisher scientific | Hs00961554_m1 | |
| CD24.HSA | Thermofisher scientific | Hs03044178_g1 | |
| NCOR1 | Thermofisher scientific | Hs01094540_m1 | |
| CD26.DPP4 | Thermofisher scientific | Hs00175210_m1 | |
| CD274 | Thermofisher scientific | Hs01125301_m1 | |
| CD276 | Thermofisher scientific | Hs00987207_m1 | |
| CD32.FCGR2B | Thermofisher scientific | Hs01634996_s1 | |
| CD33 | Thermofisher scientific | Hs01076281_m1 | |
| CD34 | Thermofisher scientific | Hs00990732_m1 | |
| CD344.FZD4 | Thermofisher scientific | Hs00201853_m1 | |
| CD352.SLAMF6 | Thermofisher scientific | Hs01559920_m1 | |
| CD38 | Thermofisher scientific | Hs01120071_m1 | |
| CD4 | Thermofisher scientific | Hs01058407_m1 | |
| CD41.ITGA2B | Thermofisher scientific | Hs01116228_m1 | |
| CD49f.ITGA6 | Thermofisher scientific | Hs01041011_m1 | |
| CD56.NCAM1 | Thermofisher scientific | Hs00941830_m1 | |
| CD9 | Thermofisher scientific | Hs00233521_m1 | |
| CD97 | Thermofisher scientific | Hs00173542_m1 | |
| CD99 | Thermofisher scientific | Hs00908458_m1 | |
| CDK6 | Thermofisher scientific | Hs01026371_m1 | |
| CDKN1A | Thermofisher scientific | Hs00355782_m1 | |
| CDKN1B | Thermofisher scientific | Hs01597588_m1 | |
| CDKN1C | Thermofisher scientific | Hs00175938_m1 | |
| CEBPa | Thermofisher scientific | Hs00269972_s1 | |
| CSF1r | Thermofisher scientific | Hs00911250_m1 | |
| CSF2RA | Thermofisher scientific | Hs00531296_g1 | |
| CSF3RA | Thermofisher scientific | Hs01114427_m1 | |
| E2A.TCF3 | Thermofisher scientific | Hs00413032_m1 | |
| EBF1 | Thermofisher scientific | Hs01092694_m1 | |
| ENG | Thermofisher scientific | Hs00923996_m1 | |
| EPOR | Thermofisher scientific | Hs00959427_m1 | |
| ERG | Thermofisher scientific | Hs01554629_m1 | |
| FLI1 | Thermofisher scientific | Hs00956711_m1 | |
| FLT3 | Thermofisher scientific | Hs00174690_m1 | |
| FOXO1 | Thermofisher scientific | Hs01054576_m1 | |
| GAPDH | Thermofisher scientific | Hs02758991_g1 | |
| GATA1 | Thermofisher scientific | Hs00231112_m1 | |
| GATA2 | Thermofisher scientific | Hs00231119_m1 | |
| GATA3 | Thermofisher scientific | Hs00231122_m1 | |
| GFI1 | Thermofisher scientific | Hs00382207_m1 | |
| HES1 | Thermofisher scientific | Hs01118947_g1 | |
| HLF | Thermofisher scientific | Hs00171406_m1 | |
| HMGA2 | Thermofisher scientific | Hs00171569_m1 | |
| HOXA5 | Thermofisher scientific | Hs00430330_m1 | |
| HOXB4 | Thermofisher scientific | Hs00256884_m1 | |
| ID2 | Thermofisher scientific | Hs04187239_m1 | |
| IGF2BP1 | Thermofisher scientific | Hs00198023_m1 | |
| IGF2BP2 | Thermofisher scientific | Hs01118009_m1 | |
| IKZF1 | Thermofisher scientific | Hs00172991_m1 | |
| IL1RAP | Thermofisher scientific | Hs00895050_m1 | |
| IL2RG | Thermofisher scientific | Hs00953624_m1 | |
| IRF8 | Thermofisher scientific | Hs00175238_m1 | |
| ITGB7 | Thermofisher scientific | Hs01565750_m1 | |
| KIT | Thermofisher scientific | Hs00174029_m1 | |
| Lin28B | Thermofisher scientific | Hs01013729_m1 | |
| LMO2 | Thermofisher scientific | Hs00153473_m1 | |
| LYL1 | Thermofisher scientific | Hs01089802_g1 | |
| Meis1 | Thermofisher scientific | Hs01017441_m1 | |
| mKi67 | Thermofisher scientific | Hs01032443_m1 | |
| MPL | Thermofisher scientific | Hs00180489_m1 | |
| MPO | Thermofisher scientific | Hs00924296_m1 | |
| NFIB | Thermofisher scientific | Hs01029175_m1 | |
| Notch1 | Thermofisher scientific | Hs01062011_m1 | |
| Pten | Thermofisher scientific | Hs02621230_s1 | |
| RAG2 | Thermofisher scientific | Hs01851142_s1 | |
| RPS18 | Thermofisher scientific | Hs01375212_g1 | |
| RUNX1 | Thermofisher scientific | Hs00231079_m1 | |
| Shisa2 | Thermofisher scientific | Hs01590823_m1 | |
| Spi1 | Thermofisher scientific | Hs02786711_m1 | |
| Sterile.IgH | Thermofisher scientific | Hs00378435_m1 | |
| TAL1 | Thermofisher scientific | Hs01097987_m1 | |
| THY1 | Thermofisher scientific | Hs00264235_s1 | |
| Tim.3.HAVCR2 | Thermofisher scientific | Hs00958618_m1 | |
| VWF | Thermofisher scientific | Hs00169795_m1 |
참고문헌
- Seita, J., Weissman, I. L. Hematopoietic stem cell: Self-renewal versus differentiation. Wiley Interdiscip Rev Syst Biol Med. 2 (6), 640-653 (2010).
- Orkin, S. H., Zon, L. I. Hematopoiesis: An evolving paradigm for stem cell biology. Cell. 132 (4), 631-644 (2008).
- Ye, F., Huang, W., Guo, G. Studying hematopoiesis using single-cell technologies. Journal of Hematology & Oncology. 10, 27(2017).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nature Cell Biology. 16 (10), 919-927 (2014).
- Wills, Q. F., et al. Single-cell gene expression analysis reveals genetic associations masked in whole-tissue experiments. Nature Biotechnol. 31 (8), 748-752 (2013).
- Velten, L., et al. Human haematopoietic stem cell lineage commitment is a continuous process. Nat Cell Biol. 19 (4), 271-281 (2017).
- Wilson, N. K., Nicola, K., et al. Combined single-cell functional and gene expression analysis resolves heterogeneity within stem cell populations. Cell Stem Cell. 16 (6), 712-724 (2015).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: Advances and future challenges. Nucleic Acids Research. 42 (14), 8845-8860 (2014).
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of Single RNA Transcripts in Situ. Science. 280 (5363), 585-590 (1998).
- Kalisky, T., et al. A brief review of single-cell transcriptomic technologies. Briefings in Functional Genomics. , elx019(2017).
- Crosetto, N., Bienko, M., van Oudenaarden, A. Spatially resolved transcriptomics and beyond. Nature Reviews Genetics. 16, 57(2014).
- Bengtsson, M., Ståhlberg, A., Rorsman, P., Kubista, M. Gene expression profiling in single cells from the pancreatic islets of Langerhans reveals lognormal distribution of mRNA levels. Genome Research. 15 (10), 1388-1392 (2005).
- Bengtsson, M., Hemberg, M., Rorsman, P., Ståhlberg, A. Quantification of mRNA in single cells and modelling of RT-qPCR induced noise. BMC Molecular Biology. 9 (1), 63(2008).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nature Methods. 10, 1096(2013).
- Tung, P. -Y., et al. Batch effects and the effective design of single-cell gene expression studies. Scientific Reports. 7, 39921(2017).
- Teles, J., Enver, T., Pina, C. Single-cell PCR profiling of gene expression in hematopoiesis. Methods in Molecular Biology. , 21-42 (2014).
- Hayashi, T., et al. Single-cell gene profiling of planarian stem cells using fluorescent activated cell sorting and its "index sorting" function for stem cell research. Development, Growth, & Differentiation. 52 (1), 131-144 (2010).
- Lang, S., et al. SCExV: A webtool for the analysis and visualisation of single cell qRT-PCR data. BMC Bioinformatics. 16 (1), 320(2015).
- de Klein, A., et al. A cellular oncogene is translocated to the Philadelphia chromosome in chronic myelocytic leukaemia. Nature. 300 (5894), 765-767 (1982).
- Lang, S., et al. SCExV. , Available from: http://stemsysbio.bmc.lu.se/SCexV/ (2015).
- Quinn, J. indexed-sorting. , Available from: https://github.com/FlowJo-LLC/indexed-sorting (2016).
- Warfvinge, R., et al. Single-cell molecular analysis defines therapy response and immunophenotype of stem cell subpopulations in CML. Blood. 129 (17), 2384-2394 (2017).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Breton, G., et al. Human dendritic cells (DCs) are derived from distinct circulating precursors that are precommitted to become CD1c+ or CD141+ DCs. The Journal of Experimental Medicine. 213 (13), 2861-2870 (2016).
- Alberti-Servera, L., et al. Single-cell RNA sequencing reveals developmental heterogeneity among early lymphoid progenitors. The EMBO Journal. 36 (24), 3619-3633 (2017).
- Giustacchini, A., et al. Single-cell transcriptomics uncovers distinct molecular signatures of stem cells in chronic myeloid leukemia. Nature Medicine. 23, 692(2017).
- Hansmann, L., Han, A., Penter, L., Liedtke, M., Davis, M. M. Clonal expansion and interrelatedness of distinct B-lineage compartments in multiple myeloma bone marrow. Cancer Immunology Research. 5 (9), 744-754 (2017).
- Psaila, B., et al. Single-cell profiling of human megakaryocyte-erythroid progenitors identifies distinct megakaryocyte and erythroid differentiation pathways. Genome Biology. 17 (1), 83(2016).
- Schulte, R., et al. Index sorting resolves heterogeneous murine hematopoietic stem cell populations. Experimental Hematology. 43 (9), 803-811 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
ISSN 2578-6326
Copyright © 2025 MyJoVE Corporation. 판권 소유
당사 웹 사이트에서는 사용자의 경험을 향상시키기 위해 쿠키를 사용합니다.
당사 웹 사이트를 계속 사용하거나 '계속'을 클릭하는 것은 당사 쿠키 수락에 동의하는 것을 의미합니다.