Method Article
הגישה קומבינטורית תא בודד כדי לאפיין את מולקולרית והטרוגניות Immunophenotypic של גזע האדם ואוכלוסיות קדמון
In This Article
Summary
תפזורת ג'ין ביטוי מדידות ענן תא בודד הבדלים באוכלוסיות הטרוגניות תא. כאן, אנו מתארים את פרוטוקול עבור ביטוי מפענוח ואינדקס איך החד-תאיים מיון על-ידי Florescence מופעל התא מיון (FACS) יכול להיות משולב על הטרוגניות, immunophenotypically מאפיינות אוכלוסיות תאים נפרדים מולקולרי.
Abstract
Immunophenotypic אפיון וניתוח מולקולרית זמן רב השתמשו ניסחו הטרוגניות ולהגדיר אוכלוסיות תאים נפרדים. FACS הוא מיסודו assay מתא בודד, אולם לפני ניתוח מולקולרית, תאי היעד מבודדים לעיתים קרובות פרוספקטיבי בצובר, ובכך לאבד רזולוציה תא בודד. תא בודד מפענוח ביטוי מספק אמצעים כדי להבין את ההבדלים מולקולרי בין תאים בודדים באוכלוסיות הטרוגניות תא. בניתוח תא בכמות גדולה overrepresentation מסוג תאים נפרדים התוצאה הטיות occlusions של אותות מתאי נדיר עם חשיבות ביולוגית. על ידי ניצול FACS אינדקס מיון מצמידים ניתוח ביטוי גנטי מתא בודד, אוכלוסיות יכול ייחקרו ללא האובדן של תא בודד רזולוציה בזמן תאים עם ביטוי סמן משטח תאים ביניים גם נלכדים, המאפשרת הערכת הרלוונטיות של הביטוי סמן משטח רציף. כאן, אנו מתארים גישה המשלבת שעתוק במהופך תא בודד PCR (RT-qPCR) וסטטיסטי FACS אינדקס מיון במקביל לאפיין את מולקולרית והטרוגניות immunophenotypic בתוך התא אוכלוסיות.
לעומת שיטות רצף של RNA מתא בודד, השימוש qPCR עם הגברה ביעד מסוים מאפשר למדידות חזקים של הפרוטוקולים נמוך-שפע עם פחות תקלות, בזמן זה הוא לא מבולבל על ידי נושאים הקשורים לתא וריאציות בלקריאה עומק. יתר על כן, על-ידי ישירות מיון אינדקס חד-תאים לתוך פירוק מאגר בשיטה זו, מאפשר cDNA סינתזה הגברה קדם ביעד מסוים שיש לבצע צעד גם לגבי המתאם של חתימות מולקולרית נגזר לאחר מכן עם משטח תא סמן ביטוי. הגישה המתוארת פותחה כדי לחקור hematopoietic חד-תאים, אך גם שימשו בהצלחה על סוגי תאים אחרים.
לסיכום, הגישה המתוארים בזאת מאפשרת מדידה רגיש של mRNA הביטוי של הגנים שנבחרו מראש עם האפשרות על פאנל לפתח פרוטוקולים עבור בידוד פוטנציאליים עוקבות של subpopulations מולקולרי ייחודי.
Introduction
כל תא דם בודדים הוא האמין להתגורר בהיררכיה של הסלולר, שבו תאי גזע טופס השיא על סדרת מחויב יותר ויותר אבות ביניים, כי בסופו של דבר סופני להבדיל לתוך התאים אפקטור הסופי נושא ספציפי תפקודים ביולוגיים1. רוב הידע על איך תא גזע מערכות מאורגנות נוצר במערכת hematopoietic, בעיקר בגלל היכולת לבודד פרוספקטיבי אוכלוסיות hematopoietic ברורים מאוד מועשר בתאי גזע או אבות שונים2 על-ידי מיון FACS. זו אפשרה עבור רבים של אוכלוסיות אלה כדי להיות מנותח באופן פונקציונלי או מולקולרי, בעיקר דרך ביטוי גנים פרופילים3,4. אולם כאשר ניתוח ביטוי גנים של צובר אוכלוסיות הבדלים אינדיבידואליים בין התאים שהגוף, איבדתי5. לפיכך, חוסר היכולת לזהות וריאציות לתא בתוך התא הטרוגנית שברים עשויים לבלבל את ההבנה שלנו של תהליכים ביולוגיים קריטי אם קבוצות משנה קטנה של תאים חשבון עבור הפונקציה הנגזרת הביולוגי של האוכלוסייה הזאת6, 7. לעומת זאת, החקירה של ג'ין חתימות ביטוי ברזולוציה תא בודד מציעים אפשרות ניסחו הטרוגניות, לעקוף מאפילה השפעות של קבוצות משנה overrepresented של תאים8.
עד כה פותחו פרוטוקולים רבים לניתוח ביטוי גנים מתא בודד; עם כל גישה יש אזהרות משלו. השיטה המוקדמת ביותר היה RNA פלורסנט בחיי עיר הכלאה (RNA-דג), אשר מודד את מספר מוגבל של הפרוטוקולים בכל פעם אבל הוא ייחודי בכך שהוא מאפשר לחקירה של RNA לוקליזציה9,11. שיטות מוקדמות באמצעות PCR qPCR כדי לזהות שיחיד או תעתיקים מעטים מאוד היו גם פיתח12. עם זאת, אלה לאחרונה הוחלפו על-ידי שיטות מבוססות מיקרופלואידיקה אשר ניתן בו זמנית לנתח את הביטוי של מאות תעתיקים בכל תא מאות תאים דרך qPCR, ובכך לאפשר הטרוגניות גבוהה-ממדי באמצעות ניתוח מראש נקבע ג'ין לוחות10,13. לאחרונה טכנולוגיות מבוססות רצפי RNA יש להיות בשימוש נרחב עבור ניתוח תא בודד, ככל אלה תיאורטית יכול למדוד את כל transcriptome של התא ובכך להוסיף מימד גישוש הטרוגניות ניתוח10, 14. ניתוח qPCR Multiplexed ורצף RNA בתא יחיד יש תכונות שונות, ובכך הרציונל לשימוש באחת מהשיטות תלוי השאלה כמו גם מספר התאים באוכלוסיית היעד. תפוקה גבוהה ועלות נמוכה בכל תא יחד עם מאפיינים לא משוחד, גישוש של רצפי RNA בתא יחיד בעת רצוי התא לא ידוע או אוכלוסיות גדולות נחקרות. עם זאת, רצפי RNA בתא יחיד גם מוטה לטובת קביעת רצף תעתיקים שופע גבוהה בתדירות גבוהה יותר, בעוד תעתיקים עם שפע נמוכה נוטים נשירה. זה יכול להוביל נתונים מורכבים במידה ניכרת שמעבירות גבוהה-דרישות על ניתוח bioinformatic לגלות אותות מולקולריים חשוב כי הם לעיתים קרובות עדינים או מוסתרים רעש טכני15. לכן, בשביל לרקמות מאופיין היטב, qPCR תא בודד באמצעות ניתוח שנקבע מראש לוחות פריימר שנבחר עבור גנים חשיבות תפקודית או סמנים מולקולריים יכול לשמש גישה רגיש פשוטה כדי לקבוע את הטרוגניות של אוכלוסייה. עם זאת, יצוין כי לעומת העלות לכל תא RNA בתא יחיד-seq, הוא בדרך כלל גבוה יותר עבור שיטות qPCR תא בודד. כאן, אנו מתארים גישה המשלבת תא בודד RT-qPCR (שונה Teles ג'יי. et al. 16), לאינדקס FACS מיון במקביל17 ו ביואינפורמטיקה ניתוח18 על מנת לאפיין את הטרוגניות המולקולריים immunophenotypic בתוך אוכלוסיות.
בגישה זו, האוכלוסייה תא עניין היא מוכתמת, חד-תאים ממוינים על-ידי FACS ישירות לתוך מאגר פירוק ב 96-ובכן PCR צלחות. בו זמנית, רמות הביטוי של קבוצה נוספת של התא-פני סמני נרשמים לכל תא יחיד במהלך FACS-מיון, שיטה שבה מתייחסים כאל מיון אינדקס. החומר תא lysed לאחר מכן מוגבר וניתח בביטוי הגן של קבוצת גנים שנבחרו עם RT-qPCR, באמצעות פלטפורמה microfluidic. אסטרטגיה זו מאפשרת ניתוח מולקולרית של מיון מתא בודד, כמו גם בו זמנית האפיון תא-פני סמן ביטוי של תא בודד כל. על-ידי ישירות מיפוי מולקולרי ייחודי קבוצות משנה של תאים לביטוי סמני אינדקס ממוינים, subpopulations יכולים להיות מקושרים כדי immunophenotype ספציפי שיכול לשמש לבידוד פוטנציאליים שלהם. השיטה המותווה צעד אחר צעד באיור1. פאנל ג'ין שנקבע מראש עוד תורמת רזולוציה גבוהה יותר של ביטוי גנים יישוב, מאז זה עוקף מדידה של הגנים שופע לא רלוונטי יכול אחרת occlude אותות ביטוי הגן מעודן. יתר על כן, הגברה יעד ספציפיות, שעתוק במהופך צעד אחד, הגברה מאפשרת מדידה חזקים של תעתיקים ביטוי נמוך, כמו גורמי שעתוק או הלא-פולי-adenylated RNAs. חשוב לציין, שיטות qPCR מאפשרים מדידה של mRNA של פיוז'ן חלבונים, אשר חשובים כאשר חוקרים מסוימים במחלות ממאירות19. בסופו של דבר, המספר ממוקד של גנים חקרו, שיעורי הנשירה נמוך, ולא מוגבל הבדלים טכניים בין תאים להפוך שיטה זו נותחו בקלות לעומת שיטות מימד גבוה יותר, כגון RNA בתא יחיד-תת סעיף לפי הפרוטוקול, ניסוי כולו יכול להתבצע, מיון תאי לתוצאות שנותחה, בתוך שלושה ימים, ממציא זה שיטה מסובכת ומהיר עבור ניתוח ביטוי גנטי תא בודד רגיש, תפוקה גבוהה.
Protocol
1. הכנת פירוק לוחות
- באמצעות ספסל חינם RNA/DNA, להכין מאגר מספיק פירוק וולס 96, עם 10% נוספים, על ידי ערבוב מים חינם נוקלאז µL 390, 17 µL של 10% NP-40, 2.8 dNTP 10 מ מ µL, µL 10 0.1 M DTT, 5.3 µL RNAse מעכב (ראה טבלה של חומרים). מערבולת, ספין מטה.
- פזר µL 4 פירוק המאגר כדי כל טוב של צלחת PCR טוב 96, לאטום את הצלחות עם סרט דביק. ספין למטה צינורות לאיסוף נוזלים בתחתית של הלוחות. לשמור על צלחות על הקרח עד התא מיון (מקסימום 24 שעות).
2. הכנה של תאים מיון תא
- להפשיר מספר מתאים של תאים (כאן, גזע hematopoietic מועשר CD34 ו ובתאים) לניסוי. עונה 1 פרק 106 תאים מתאימים לצורך מיון כ שלושה 96-ובכן צלחות חד-תאים עם פקדים.
- להעביר תאים המופשרים צינור חרוטי 15 מ"ל ולהוסיף 1 מ"ל FBS כל 30 s עד הנפח הכולל של 8 מ ל. לסובב תאים ומפרידה ב x 350 גר' 10 דקות ב 4 ° C ולהסיר תגובת שיקוע.
- Resuspend התאים ב- mL 8 מכתים מאגר (PBS עם 2% FBS), צנטריפוגה ב x 350 גר' 10 דקות ב 4 ° C, להסיר את תגובת שיקוע.
- תאים resuspend מכתים µL 200 מאגר ולהסיר תאים עבור פקד כתמים.
- להפוך פלורסצנטיות מינוס אחד פקדים (FMOs) עבור כל fluorophore, על-ידי מכתימה שבריר של תאים ב- 50 µL מכתים מאגר. בדוגמה זו, צינורות microcentrifuge 6 עם 20,000 תאים משמשים FMOSs. שימו לב כי מספר התאים צריך להיות מותאם בהתאם האוכלוסייה חקר. להוסיף לכל הנוגדנים ריכוז זהה כמו הכתם מדגם חוץ מאחד את כל שפופרת.
- הפוך כתמי יחיד עבור כל fluorophore על ידי צביעת שבריר של תאים במאגר µL 50 עבור כל fluorophore בשימוש. בדוגמה זו נעשה שימוש 6 צינורות microcentrifuge עם תאים 20,000. שימו לב כי היעד עבור כל נוגדן צריך לבוא לידי ביטוי על ידי התאים המשמש עבור פקדים. להוסיף כל נוגדן ריכוז זהה כמו הכתם מדגם צינורות בודדים. בנוסף לשמור תאים וללא רבב 20,000 50 µL כפקד של וללא רבב.
- לדגימת תא, להוסיף נוגדנים שלהם הריכוז המתאים. בשימוש להלן CD34-FITC-ריכוז 1/100, CD38-APC 1/50, CD90-PE 1/10, CD45RA-bv421 1/50, CD49F-PECy7 1/50, שושלת היוחסין מיקס: CD3-PECy5 1/50, CD2-PECy5 1/50, CD19-PECy5 1/50, CD56-PECy5 1/50, CD123-PECy5 1/50, CD14-PECy5 1/50, CD16-PECy5 1/50, ו CD235a-PECy5 1/1000.
- דגירה תאים עם נוגדנים במשך 30 דקות על קרח בחושך.
- לשטוף תאים עם 3 מ"ל מכתים מאגר. צנטריפוגה תאים ב x 350 גר' 10 דקות ב 4 ° C, להסיר את תגובת שיקוע.
- Resuspend תאים וחזור על שלב 2.9.
- Resuspend מדגם 500 µL ו- FMOs 100 µL מכתים מאגר עם תאים 7AAD ומסנן 1/100 דרך מסנן 50 מיקרומטר לקבל השעיה תא בודד.
3. מיון תא
- ודא כי המכשיר FACS מוגדר עד כראוי עם טיפה עיכוב ו cytometer ההתקנה ומעקב (CST) שבוצעו לאחרונה על פי הוראות היצרן, כדי להבטיח כי התאים המתאים ממוינים. עבור תאים hematopoietic, מומלץ השימוש של מיקרון 85 זרבובית, מקסימום מהירות 4, בעוד שיעור אירוע האופטימלי הוא בין 800 ל- 2000 אירועים/s.
- ולתיקון ספקטרלי חפיפה על-ידי ביצוע קרינה פלואורסצנטית פיצויים וקבע השערים על-פי FMO בפקדי או פנימי שלילי.
- לבצע בצמחייה של אוכלוסיית היעד על-ידי מיון תאי היעד לפחות 100 לתוך צינור microcentrifuge עם 100 µL הכתם המאגר. FACS לנתח את התאים ממוינות על ידי רישום המדגם ממוינים ולוודא כי הם מסיימים השער מיון.
- הגדרת תא הלוח היחיד מיון לפי מרכוז ירידה A1 טוב בצלחת טוב 96. כאשר, ממורכזת לסווג חלקיקים מיקרומטר 50 – 100 6 לתוך כל הבארות שמסביב לקצה צלחת ריקה טוב 96 להבטיח כי כל בארות יקבלו תא במרכז של כל טוב.
- במידת האפשר, ניתן להוסיף פקד נוספים כדי להבטיח כי התאים קיימא ממוינים על-ידי מיון חד-תאים לצמיחה במבחנה . יכול להיות גדל התאים Haematopoietic U-התחתון 96 הצלחות טוב 100 µl SFEM עם 1% פניצילין סטרפטומיצין, 100 ננוגרם למ"ל FLT3L, TPO ו- SCF. לנתח כל טוב אחרי 3 ימים בתרבות על מושבות תאים באמצעות מיקרוסקופ.
- הסר את סרט דביק של צלחות. סוג הריבית (כאן לין-CD34 + CD38-תאים) לתוך 92 מתוך הבארות 96, תא בודד להפעיל אינדקס-מיון FACS מיון תוכנה לשמירת הפרופיל immunophenotypic עבור סמנים אחרים עניין (כאן CD45RA, CD49f ו CD90) עבור כל תא יחיד.
- למיין שתי בארות עם תאים 10 ל- 20 בהתאמה עבור פקדים ליניאריות הגברה PCR. וולס H1 ו- H2 משמשים בדרך כלל.
- שמרו שתי בארות ללא תאים לא-תבנית שולט, בדרך כלל בארות H3 ו- H4.
- לאטום את הצלחות עם סרט דביק ברור ולסובב את הצלחות ב x 300 גרם במשך 1 דקה לפני snap קפוא בקרח יבש.
- חנות צלחות קפוא ב-80 מעלות צלזיוס.
הערה: לנקודת עצירה בטוח. ניתן לשמור תאים ממוינים ו lysed ב-80 מעלות צלזיוס לאחסון לטווח ארוך.
4. הפוך שעתוק ו ביעד מסוים הגברה
- להכין את תערובת פריימר כל המטרות גנים 96 על-ידי הוספת 2 µL של כל זוג פריימר, כולל ניקיון הגן תחל ותחל לבקרת עלה ב- RNA, 1.5 מ ל RNAse צינור חינם על ספסל חינם RNA/DNA. אם משתמש תחל פחות מ 96, להוסיף אמצעי מקביל של נוקלאז מים חינם עבור תחל החסר. תחל מסודרים בנפרד כדי להתאים את לוח הגן הרצוי.
- להפוך את שעתוק במהופך הגברה ביעד מסוים לערבב על-ידי הוספת 632.5 µL 2 x תערובת התגובה, 101.2 µL Taq/SuperscriptIII, 151.8 מיקס פריימר µL ו- 0.7 µL עלה בפקד ה-RNA. Preform שלב זה על ספסל ללא ה-DNA. מיקס על ידי vortexing ו ספין למטה כדי לאסוף הנוזל בתחתית של הצינור. לשמור בקירור עד בנוסף הדגימה.
- להפוך ללא היפוך שעתוק שליטה מיקס עבור מארבע בארות על ידי ערבוב 27.5 µL של 2 x תערובת התגובה, 1.76 µL של האנזים Taq, µL 6.6 של פריימר מיקס µL 2.64 נוקלאז מים חינם. ספייק בפקד ה-RNA. יש לבצע פעולה זו על ספסל ללא ה-DNA. מערבולת, ספין למטה כדי לאסוף הנוזל בתחתית של התחתית, לשמור על הקרח עד תוספת מדגם.
- הפשרת צלחות lysate על הקרח. להוסיף µL 8.75 של שעתוק במהופך בעבר מוכן, יעד ספציפיות הגברה מיקס וולס 92, כולל את הפקדים ליניאריות, לא-תבנית. להוסיף µL 8.75 של מיקס בקרת שעתוק ללא היפוך הבארות ארבעת הנותרים. חותם צלחות עם סרט דביק ברורה ו ספין למטה כדי לאסוף נוזלים בתחתית של הלוחות.
- Preform שעתוק במהופך, הגברה ביעד מסוים על-ידי הפעלת המשטח במכונת ה-PCR על-פי התוכנית preamp; שלב 1:50 ° C עבור 60 דקות, שלב 2: 95 ° C למשך 2 דקות, צעד 3: 95 ° C 15 s, שלב 4: 60 ° C למשך 4 דקות, חזור על שלבים 3-4 24 פעמים, ולבסוף שלב 5: 8 ° C לנצח.
- בתום ה-PCR, לקחת את הצלחת ב 8 ° C לאחסון לטווח הקצר ו-20 ° C לאחסון לטווח ארוך.
הערה: לנקודת עצירה בטוח. ניתן לשמור חומר מוגבר 8 מעלות לאחסון לטווח קצר וכן ב-20 ° C לאחסון לטווח ארוך.
5. הכנה של הדגימה ואת וזמינותו צלחות ניתוח ביטוי גנטי Microfluidic מולטיפלקס
- להכין assay טעינת צלחת על ידי pipetting 3 µL Assay הטעינה מגיב על כל טוב של צלחת טוב 96. להוסיף 3 µL של פריימר כל בארות בודדים וזמינותו טעינת צלחת.
- חותם צלחת עם סרט דביק, ספין למטה כדי לאסוף נוזלים בתחתית של הלוחות.
- להכין צלחת דילול על ידי pipetting 8 µL של נוקלאז מים חינם לתוך בארות כל צלחת טוב 96. להוסיף 2 µL מדגם מוגבר לצלחת דילול, גורם לדילול הסופי של 1:5.
- לאטום את הצלחת עם סרט דביק, לערבב מאת vortexing צלחת 10 s, סוף סוף ספין למטה כדי לאסוף נוזלים בתחתית של הלוחות.
- הכן תערובת טעינה הדגימה על ידי ערבוב µL 352 של מיקס מאסטר עם 35.2 µL של ריאגנט טעינה הדגימה. להכין צלחת טעינה הדגימה על-ידי µL aliquoting 3.3 בטעינה שילוב כל טוב של צלחת טוב 96.
- להוסיף 2.7 µL מתוך המדגם מדולל לבאר כל המדגם טעינת צלחת.
- חותם צלחת עם סרט דביק, ספין למטה כדי לאסוף נוזלים בתחתית של הלוחות.
6. טעינה של שבב Microfluidic
- להוציא שבב microfluidic, 96x96 חדש. הכן אינלטס על ידי לדקור אותם בעזרת מזרק עם מכסה על כדי לוודא כי הם ניתנים להעברה.
- הסר בועות מזרקים. להוסיף נפח מלא של מזרקים כל שסתום תוך כדי הטיית השבב 45 מעלות, לוחץ השסתום. השבב העיקרי עם בקר IFC.
- לטעון כל כניסת assay עם 4.25 µL מכל הנוגע לבארות וזמינותו טעינת צלחת. להימנע בועות. אם שהבועות מופיעות הבאר, להסיר אותם עם טיפ פיפטה.
- להמשיך לטעון כל כניסה מדגם עם 4.25 µL מכל הבארות במדגם טעינת צלחת להימנע בועות, אם מופיעות בועות, להסיר אותם עם טיפ פיפטה.
- לטעון שבב עם בקר IFC.
- בדוק כי השבב נראה אפילו כי טעינת כל צ'יימברס. להסיר אבק מפני השטח שבב על ידי נגיעה בו עם קלטת. להפעיל את השבב פלטפורמת ביטוי של גנים מולטיפלקס microfluidic.
7. מפעיל את שבב על פלטפורמת ביטוי גנים Microfluidic מולטיפלקס
- לאחר טעינת השבב לתוך פלטפורמת ביטוי של גנים microfluidic מולטיפלקס, שם את הדגימה.
- הגדר רוקס לצבוע פסיבי. הגדר החללית יחיד ואת FAM-MGB זריחה. השתמש v2 סטנדרטי, 96x96 פרוטוקול. תתחיל לרוץ.
- הסר שבב בעת הפעלה הושלמה.
8. ניתוח ראשוני של צ'יפ בניהול
- לטעון את הנתונים לתוך תוכנת ניתוח בזמן אמת PCR.
- לטעון ג'ין שמות ושמות תא על-ידי הדבקת התא ואת הגן פריסות מתוך קובץ מופרד באמצעות טאבים.
- פתח תצוגת תמונה ובחר רוקס כמו לצבוע. בדוק אם כל בארות יש לצבוע פסיבי רוקס.
- לחקור אם כל העלילות הגברה נראית בסדר, עם עקומה חלקה הגברה עם אין קוצים (בדומה איור 3E).
- ודא שכל יחיד-התאים שיש של שליטה עלה ב- RNA כדי לוודא כי כל מה יש נטען כראוי.
- ודא כי כל התאים יש ביטוי גנים משק הבית, ובכך מוינו כראוי.
- ודא 10 ל- 20 תא ליניאריות פקדים שיש כ CT 1 ההבדל לאימות הגברה ליניארי.
- בדוק אם יש ביטוי הדגימות שליטה צפונית. אם הביטוי הוא זוהה צפונית שקול לשנות הגששים הגששים אשר מזהה את ה-DNA גנומי של עוקבות.
- לייצא נתונים קבצי csv עבור ניתוח נוסף.
9. החד-תאיים ניתוח באמצעות SCExV
הערה: סרט מבוא הוא נוכח20 להציג את הכלי. כאן, מוצג מכתב המלצה קצרה של איך לעשות ניתוח באמצעות הפקדים הציג בפרוטוקול.
- לחבר האתר SCexV20.
- להעלות קבצי CSV המיוצא.
- בחר הפקד עלה ב- RNA בקרה חיובית. הסר תא כלשהו אשר פקד RNA CT מעל 25. לנרמל את הנתונים לביטוי החציוני של פקד ה-RNA.
- לחץ על "כאן -> ניתוח". הסר צפונית, notemplate, פקדים תא 10 ל- 20 האפשרות תא אל תכלול.
- אשכול תאים עם הגישה קיבוץ באשכולות הבחירה עם המספר הצפוי של אשכולות. ערכי ייצוא מנותח.
10. אינדקס-מיון ניתוח
- פתח את תוכנת ניתוח FACS וטען הדגימות באינדקס.
- לפתוח את עורך קובץ script ולהפעיל קובץ ה-script הזמינים מ. ג'יי קווין ואח 21
- זה התוכנה ניתוח FACS צריך לעשות שער לכל אחד התאים, לפתוח פריסה עורך וצבע את התאים לפי הקיבוץ מן SCexV (זמין בקובץ בשם "Sample_complete_Data.xls").
תוצאות
פרוטוקול המתואר הוא מהיר, שבוצעו בקלות ואמינים ביותר. סקירה כללית של הסידור ניסיוני מוצג באיור1. פרוטוקול כולו, ממיון של חד-תאים, הגברה יעד ספציפיות, מדידות ביטוי גנים, ניתוח ראשוני יכול להתבצע בשלושה ימים. דוגמה של תוצאות שנותחה בדמות מפת החום מייצג ראשוני ניתחו נתונים מניתוח ביטוי גנים מתא בודד באמצעות תחל 96 96 תאים ממטופל או לוקמיה מיאלואידית כרונית (CML) או תאים 96 מ בגילאי-מתאימים בריא שליטה מוצגת באיור2. בעזרת קיבוץ באשכולות הירארכי, התאים שנותחה ניתן לחלק ארבע קבוצות בהתבסס על החתימות שלהם ביטוי גנים. הפריט החזותי מפה חום הוא דרך נוחה לקבלת סקירה כללית של הנתונים, כמו גם ניתן לשלוט בארות אשר לא להיות כלולים הניתוח (למשל, שליטה וולס). איור 2B הוא ניתוח גורמים ראשיים (PCA) להמחיש עד כמה דומה בתאים של כל קבוצה סובלים אחד את השני באמצעות ממד אלגוריתמית. . הנה, ליניאריים כאן בקלות להבחין בין שאר התאים. כדי לנתח כמה גנים יחידניים מתחלקים בין האשכולות, כמו גם נבדלים ביניהם, כינור חלקות שימושיים. איור 2C, הביטוי של גנים נציג ארבעה מוצגים: פיוז'ן הגן BCR-ABL1, אשר מסמנת את כל התאים לוקמיה; mKI67 סמן מחזור התא, הבאה לידי ביטוי קבוצת החלוקה באופן פעיל; דה מרקר התמיינות תאים מיאלואידים MPO, אשר מוגבל קבוצת המשנה אופניים; ושמירת בסופו של דבר הבית ג'ין RPS18, הבאה לידי ביטוי כל התאים. לבסוף, ב איור דו-ממדי יש היה בקורלציה החתימות מולקולרית immunophenotyped, כמו זהותם של כל תא באשכול כל משמשים צבע התאים הבודדים בחלקה FACS שנוצר מתוך האינדקס-המיון. המוצג הוא הביטוי סמן משטח תאים עבור הסמנים תאי גזע hematopoietic CD90, CD45RA במקרה זה יכול לשמש כדי להפלות בין חלק האשכולות ולבודד אותם פרוספקטיבי לניתוח פונקציונלי.
מיקרופלואידיקה המשמש לביצוע qPCR של תא בודד רגישה מאוד החדרת אוויר או חלקיקים לתוך המערכת. לכן, חיוני לבצע בדיקה איכות פוסט ריצה של הנתונים. איור 3 מראה נציג נתונים ולהפעיל את הניגוד בין הצלחה שנכשלה בשל טעינה הדגימה המסכן. איור 3A מראה כמה רמות רוקס לאחר מוצלחת לדרוס את התפשטות באופן שווה כל בארות שאכן הטעינה הושלמה בהצלחה. לעומת זאת, דמות 3B מדגימה איך מדגם וטעינה assay נכשלו כמצוין על-ידי חוסר רוקס בחלקים גדולים של השבב. ברגע יש היה להעריך את האיכות של תאגיד לוקאס סולאס, ניתן להעריך איכות האותות ביטוי גנים raw. ב- איור 3C, מוצגת מפת החום של ריצה מוצלח, שבו הביטוי הוא אחיד להתפרס על דגימות עם קליטה טובה של סביב 7-25 Ct:s., ניגוד, איור תלת-ממד מראה מפת החום של התמודדות שבו יש דוגמאות רבות אין אות או ביטוי כמות מוגבלת מאוד של גנים. התוצאות דמות תלת-ממד הן הסבירות עקב שגיאה ב- FACS המיון לפני ניתוח ביטוי גנטי מתא בודד, שכן הרבה תאים יש ביטוי ברור אותות בזמן שאחרים חוסר בביטוי לחלוטין. לבסוף, איור 3E מציג עקומת ההגברה הצפויה של שליטה עלה ב- RNA עם כל וולס יש ביטוי ברור של בסביבות 10 CT עם מעט אין וריאציה. שליטה חיובית זו מודדת את היעילות של כל השלבים בניתוח qPCR תא בודד. כדי להבטיח כי הגברה בטווח דינמי, מספרי הטלפון הנייד שונים נכללים כפקדי ליניאריות; כאן, 10 ו- 20-תאים פקדים משמשים. CT ההבדלים בין הפקדים ליניאריות לשקף את ההבדלים מספרי הטלפון הנייד. לבסוף, אין הפוך שעתוק (צפונית) אין תבנית הפקדים שלילי על מנת להבטיח כי אין אותות חיובי כוזב. מזוהים דנ א גנומי או מציג, בהתאמה.

איור 1: הצג סכימתי של הפרוטוקול. תאים מוכתמים, ממוינים עם יישום אינדקס-מיון מופעל כדי להקליט סמן משטח הביטוי, ואת lysed כדי לשחרר את ה-mRNA. ה-mRNA הוא לאחר מכן הפוך עיבד כדי cDNA מוגבר באמצעות הגברה יעד ספציפיות. בשלב הבא, הדגימות נטענים עם תחל בודדים לתוך שבב microfluidic, איפה פרופיל ביטוי גנטי של כל תא הוא נותחה באמצעות RT-qPCR. לאחר איסוף הנתונים, הנתונים הם מעובדים מראש כדי להסיר תאים באיכות נמוכה, clustering מבוצע כדי להגדיר אוכלוסיות תאים נפרדים מולקולרי. לבסוף, subpopulations מולקולרי מוגדר נמצאים בקורלציה לביטוי סמן משטח ממיון אינדקס FACS נתונים immunophenotypically לאפיין את הטרוגניות של האוכלוסיות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
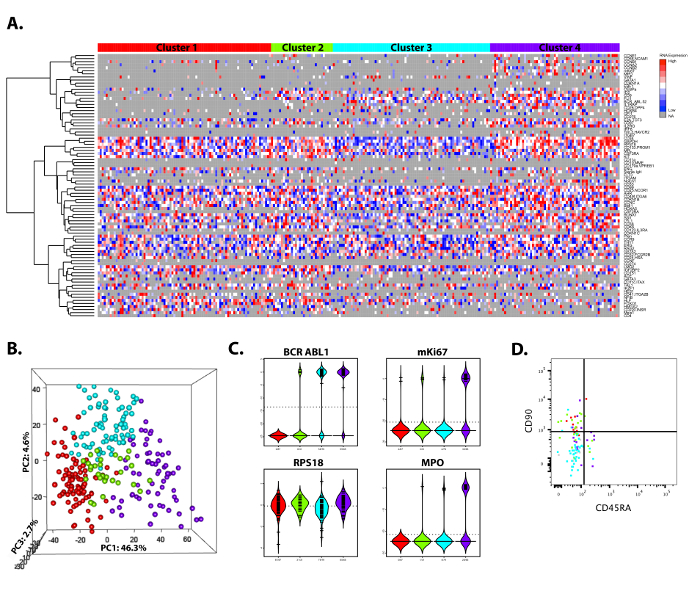
איור 2: נציג התוצאות של ביטוי גנים מתא בודד מוצלחת שלושה מנתח של תאי גזע נורמלי של לוקמיה hematopoietic. (א) חום מפה המציגה תא בודד הגן ביטוי מדידות של תאים בודדים 270 אחד חולה לוקמיה מיאלואידית כרונית, כמו גם תואם גיל בריא פקד נותחו על ידי קיבוץ באשכולות היררכי באמצעות SCExV 18, כלי ניתוח עוצב עבור סוג נתונים זה. האדום מייצג ביטוי גבוהה, ביטוי נמוך כחול, אפור ללא הבעה. התאים היו מחולקים 4 אשכולות שונים בהתבסס על החתימות שלהם ביטוי גנים. ברור מתוך הנתונים כי האוכלוסייה לוקמיה בתאי גזע של המטופל הזה ניתן לחלק אוכלוסיה השבתה הדרגתית אחת (מקבץ 3), עם ביטוי של סמנים בידול ספורים בלבד, אוכלוסיית החלוקה באופן פעיל אחד יזם ביטוי גנים הקשורים מיאלואידית בידול (מקבץ 4). הנתונים המשמשים כאן הוא ממטופל שנבחר הכלולים העבודה שפורסמו על ידי. Warfvinge et al. 22 (B) העיקרי ניתן להציג ניתוח גורמים (PCA) של הנתונים המוצגים ב- A, שבו מופרדים ארבע קבוצות. (C) כינור מתווה של ארבעה גנים עם אשכול ביטוי ספציפי, BCR-ABL1, mKI67, RPS18 ו- MPO. כל חלקה כינור מייצג את הביטוי באשכול כל, הקו המקווקו מעבר מייצג את הערך הממוצע של הביטוי עבור כל התאים. (ד) ניתוח מיון אינדקס של אחת הדגימות המטופל מיוצג במפה חום, שבו כל תא בודד מסומן עם הצבע של שלה subpopulation מולקולרית. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 3: בקרת איכות של qPCR מולטיפלקס בתוכנת ה-PCR בזמן אמת- (א) רוקס טעינת תמונה של דגימה מוצלחת וטעינה וזמינותו. (B) רוקס טעינת התמונה דוגמה לא מוצלחת ואת וזמינותו טעינה, איפה loadings כושל של דגימות מוצגים על ידי חוסר של תאגיד לוקאס סולאס בקווים אופקיים. נכשלה טעינת מבחני להיות מוצג קווים אנכיים. (ג) דוגמה מוצלחת הפעל בתצוגת מפה חום, שבו תאי-יחיד מייצג שורות וגנים מייצג עמודות. ביטוי גנים גבוהה מציינים ביטוי נמוך, צהוב, כחול, אין ביטוי שזוהו בשחור. (ד) דוגמא-הצלחתי להפעיל בתצוגת מפה חום, שבו תאי-יחיד מייצג שורות וגנים מייצג עמודות. ביטוי גנים גבוהה מיוצג על ידי צהוב, ביטוי נמוך מיוצג על-ידי כחול, אין ביטוי שזוהו כשחור. עוצמת מנורמל (E) (משמאל), הגברה מגרש (מימין) של פקד עלה ב- RNA. Ct-ערכים של 10 ועיקולים עם וריאציה מוגבל מציינים מוצלח וחזק הפוכה שעתוק, הגברה קדם qPCR ניתוח בתוך כל בארות. (F) מנורמל בעוצמה (משמאל) עלילה הגברה (מימין) של גנים שנכשלו. עוצמות מנורמל לא בצורת עקומה, העלילה הגברה מציג קוצים גדולים בזריחה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Discussion
בשנים האחרונות, תא בודד מפענוח הביטוי הפך תוספת רבת ערך כדי להגדיר את הטרוגניות של אוכלוסיות שונות תא23. כניסתו של טכנולוגיות רצף הרנ א תיאורטית מספק אפשרות למדוד את transcriptome שלם של תא, עם זאת, שיטות אלה הן מסובך וריאציות של רצף לתא לבין נשירה. QPCR תא בודד מציע ניתוח רגיש, איתנה של הביטוי של מאות גנים קריטי איפה כל התאים מטופלים באופן דומה, הפחתת רעש טכני. ניתוח ממוקד של מספר מוגבל של התמלילים מאפשר בנוסף לבדיקה פשוטה ללא הפרעה מאוד ביטוי גנים.
מאז הגישה חוקר רק תת-ערכה של גנים ביטוי בתאים חשוב מאוד לבחור את קבוצת הגנים הנכונים בעת עיצוב לוח ג'ין. הבחירה של גנים יכול להיעשות במספר דרכים; דרך הספרות, ממצאים קודמים על-ידי ניתוח בצובר ולאחר ביואינפורמטיקה ניתוח של נתונים זמין לציבור. . זה לא טריוויאלי; עם זאת, כאשר ערכת הנכון של גנים הגישה יכולות להיות חזקות מאוד. כאשר החלונית הגן עוצבו, חשוב לאמת כי תחל שמפריעה במהלך ריבוב. כאן אנו ממליצים ניתוח אם צבעי בסיס לינארי על-ידי preforming סדרה דילול עם חומר בשפע; לדילול סדרת תחל השתמשו במחקר זה מוצג משלימה איור דו-ממדי ב. Warfvinge et al. 22
המגבלות העיקריות שני של פרוטוקול המובאים כאן הם מספר מוגבל של תאים, גנים חקרו. עם זאת, למרות התאים רק 96 נחקרות ב כל הפעלה, פרוטוקול פשוט יחסית זה יכול להסתיים ביום אחד, ובכך ניתן לנתח מאות תאים בשבוע אחד. בנוסף, יכול להיות עלה מספר גנים חקרו אם תחל עוד כלולים בשלב קדם הגברה במטרה. במקרה זה, מספר שבבי דרושים כדי לחקור כל ערכה של צבעי יסוד 96.
אם מאוד בא לידי ביטוי גנים או תאים עם RNA-תכולה גבוהה נבחנים, מספר מחזורים אופטימיזציה עבור תאי גזע hematopoietic עם נמוך הכולל גנים ביטוי23 צריך להיות שונה. שינוי מחזורים הגברה חשוב גם כאשר חוקרים אוכלוסיות בצובר. לניתוח כמות גדולה של תאי גזע hematopoietic (תאים 100) מומלץ להפחית את כמות מחזורי קדם הגברה מ 25 ל-18.
הנפוצות ביותר סיבות כי נכשל פועל נובע מיון לא מוצלח של חד-תאים לתוך הבאר של פירוק צלחת, מיון של תאים כלכלית, שיוצרת הגברה מראש, או שבב כשל בטעינת. רמת רוקס בכל אחד התגובה הוא מחוון הטעינה של הדגימה או assay, אם אלה נמוכה, מומלץ כי הדגימות מחדש לרוץ על שבב חדש, מאז רוקס נמוך רמות נגרמים כנראה הקדמה של בועות במהלך טעינה. חוסר שליטה עלה ב- RNA הביטוי הוא מחוון זה קדם המגברים נכשלו, ככל הנראה עקב בעיות עם הגברה טרום- או להפוך ריאגנטים שעתוק. במקרה זה, מומלץ כי שבב חדש מנוהל עם תאים ממוינים לאחרונה, המגיבים חדש. זיהוי של פקד ה-RNA אבל אין אינדיקציה של ביטוי גנים אחרים אותות הוא אינדיקציה FACS תא בודד לא מוצלח מיון לפני ניתוח ביטוי גנטי. כדי למנוע שגיאות במהלך FACS מיון שזה חשוב להבטיח כי חד-התאים ממוינים לתוך המרכז של כל טוב. ניתן לבצע זאת על-ידי מיון חרוזים לתוך צלחת ריקה של בודק באופן חזותי איפה מכניסה את ה-droplet. הקפידו על הבדיקה-מיון לתוך כל בארות בקצוות של צלחת כדי להבטיח הסידור לא רק נכונות עבור מספר הבארות הראשונים. בעת הצורך, חד-תאים במקביל ניתן למיין לצמיחה חוץ גופית בתוך וניתח לפני ניתוח qPCR כדי לשלוט עבור פרוטוקולים המיון בהצלחה. מיון-אינדקס מידע אינה מאפשרת רק עבור חוקרים את immunophenotyped של subpopulation מולקולרי ברורים אך יכולים גם לספק תובנות שימושיות בעת פתרון בעיות, אם המיון מתבצע אוכלוסיה לא מוגדר והם הרבה תאים באיכות נמוכה נוכח בניתוח ביטוי גנים מתא בודד. אינדקס-מיון המידע יכול לשמש כדי למטב את האסטרטגיה המגביל ולהימנע מיון העתידי של תאים באיכות נמוכה.
ניתוח ביטוי גנטי תא בודד לעשות מהפכה תא הטרוגנית כמה אוכלוסיות נחקרות, לסלול את הדרך להבנת נוספת של תאית התמיינות. ב- haematopoiesis, תא בודד ביטוי מפענוח שימשו כדי לשכלל אסטרטגיות מיון עבור קבוצות משנה של HSCs7, כדי לפתור את הטרוגניות של24,6,אוכלוסיות קדמון multipotent25 , וכדי לזהות טיפול רגישות תאי לוקמיה אוכלוסיות22,26. שילוב של מדד מיון עם ביטוי גנים מתא בודד - וגם אנליזה פונקציונלית שהוצג בעבר להיות אמין ויעיל כאשר חוקרים immunophenotype והטרוגניות של אוכלוסיות שונות27, 28,29. הנה, גישה לחקירה של מולקולרית ותיאר immunophenotypic הטרוגניות של תא אוכלוסיות היה בו שילוב של תא בודד qPCR עם אינדקס מיון מאפשר תוצאות לשחזור ומהירה ללא ניתוח מורכב קצר פרק זמן.
Disclosures
המחברים אין לחשוף.
Acknowledgements
עבודה זו נתמכת מענקים השוודית החברה לסרטן, המועצה למחקר השבדי, החברה השוודית למחקר רפואי, קרן סרטן ילדות שוודית, קרן Söderberg רגנר, ו קנוט, אליס ולנברג קרן
Materials
| Name | Company | Catalog Number | Comments |
| CD14 PECY5 | eBioscience | 15-0149-42 | Clone: 61D3 |
| CD16 PECY5 | Biolegend | 302010 | Clone: 3G8 |
| CD56 PECY5 | Biolegend | 304608 | Clone: MEM-188 |
| CD19 PECY5 | Biolegend | 302210 | Clone: HIB19 |
| CD2 PECY5 | Biolegend | 300210 | Clone: RPA-2.10 |
| CD3 PECY5 | Biolegend | 300310 | Clone: HIT3a |
| CD123 PECY5 | Biolegend | 306008 | Clone: 6H6 |
| CD235A PECY5 | BD Pharma | 559944 | Clone GAR2 |
| CD34 FITC | Biolegend | 343604 | Clone: 561 |
| CD38 APC | Biolegend | 303510 | Clone: Hit2 |
| CD90 PE | Biolegend | 328110 | Clone: 5E10 |
| CD45RA BV421 | BD bioscience | 560362 | Clone: HI100 |
| CD49f Pecy7 | eBioscience | 25-0495-82 | Clone: eBioGOH3 |
| FBS | HyClone | SV30160.3 | |
| PBS | HyClone | SH30028.02 | |
| 96-well u-bottom Plate | VWR | 10861-564 | |
| SFEM | Stem cell technologies | 9650 | |
| Penicillin streptomycin | HyClone | SV30010 | |
| TPO | Peprotech | 300-18 | |
| SCF | Peprotech | 300-07 | |
| FLT3L | Peprotech | 300-19 | |
| Falcon Tube 15 mL | Sarstedt | 62.554.502 | |
| Eppendorph tube | Sarstedt | 72.690.001 | |
| CST beads | BD | 642412 | |
| Accudrop Beads | BD | 345249 | 6 µm particles |
| Adhesive film Clear | Thermo scientific | AB-1170 | |
| Adhesive film Foil | Thermo scientific | AB-0626 | |
| 96 well PCR plate | Axygen | PCR-96M2-HS-C | |
| PCR 1.5 mL tube | Axygen | MCT-150-L-C | |
| T100 PCR cycler | BioRad | 186-1096 | |
| 10% NP40 | Thermo scientific | 85124 | |
| 10 mM dNTP | Takara | 4030 | |
| 0.1 M DTT | Invitrogen | P2325 | |
| RNAsout | Invitrogen | 10777-019 | RNAse inhibitor |
| CellsDirect One-Step qRT-PCR Kit | Invitrogen | 11753-100 | |
| Neuclease free water | Invitrogen | 11753-100 | from CellsDirect kit |
| 2x Reaction Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| SuperScript III RT/Platinum Taq Mix | Invitrogen | 11753-100 | from CellsDirect kit |
| Platinum Taq DNA Polymerase | Invitrogen | 10966026 | |
| TaqMan Cells-to-CT Control Kit | Invitrogen | 4386995 | |
| Xeno RNA Control | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 20x Xeno RNA Control Taqman Gene Expression Assay | Invitrogen | 4386995 | From TaqMan Cells-to-CT Control Kit |
| 96.96 Sample/Loading Kit—10 IFCs | Fluidigm | BMK-M10-96.96 | |
| 2x Assay Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| 20x GE Sample Loading Reagent | Fluidigm | From 96.96 Sample/Loading Kit | |
| Control line fluid | Fluidigm | From 96.96 Sample/Loading Kit | |
| TaqMan Gene Expression Master Mix | Applied Biosystems | 4369016 | |
| BioMark HD | Fluidigm | BMKHD-BMKHD | |
| 96.96 Dynamic Array IFC | Fluidigm | BMK-M10-96.96GT | |
| Excel | Microsoft | Microsoft | |
| FlowJo V10 | TreeStar | TreeStar | |
| Fluidigm real time PCR analysis | Fluidigm | Fluidigm | |
| CD179a.VPREB1 | Thermofisher scientific | Hs00356766_g1 | |
| ACE | Thermofisher scientific | Hs00174179_m1 | |
| AHR | Thermofisher scientific | Hs00169233_m1 | |
| BCR_ABL.52 | Thermofisher scientific | Hs03043652_ft | |
| BCR_ABL41 | Thermofisher scientific | Hs03024541_ft | |
| BMI1 | Thermofisher scientific | Hs00995536_m1 | |
| CCNA2 | Thermofisher scientific | Hs00996788_m1 | |
| CCNB1 | Thermofisher scientific | Hs01030099_m1 | |
| CCNB2 | Thermofisher scientific | Hs01084593_g1 | |
| CCNC | Thermofisher scientific | Hs01029304_m1 | |
| CCNE1 | Thermofisher scientific | Hs01026535_g1 | |
| CCNF | Thermofisher scientific | Hs00171049_m1 | |
| CCR9 | Thermofisher scientific | Hs01890924_s1 | |
| CD10.MME | Thermofisher scientific | Hs00153510_m1 | |
| CD11a | Thermofisher scientific | Hs00158218_m1 | |
| CD11c.ITAX | Thermofisher scientific | Hs00174217_m1 | |
| CD123.IL3RA | Thermofisher scientific | Hs00608141_m1 | |
| CD133.PROM1 | Thermofisher scientific | Hs01009250_m1 | |
| CD151 | Thermofisher scientific | Hs00911635_g1 | |
| CD220.INSR | Thermofisher scientific | Hs00961554_m1 | |
| CD24.HSA | Thermofisher scientific | Hs03044178_g1 | |
| NCOR1 | Thermofisher scientific | Hs01094540_m1 | |
| CD26.DPP4 | Thermofisher scientific | Hs00175210_m1 | |
| CD274 | Thermofisher scientific | Hs01125301_m1 | |
| CD276 | Thermofisher scientific | Hs00987207_m1 | |
| CD32.FCGR2B | Thermofisher scientific | Hs01634996_s1 | |
| CD33 | Thermofisher scientific | Hs01076281_m1 | |
| CD34 | Thermofisher scientific | Hs00990732_m1 | |
| CD344.FZD4 | Thermofisher scientific | Hs00201853_m1 | |
| CD352.SLAMF6 | Thermofisher scientific | Hs01559920_m1 | |
| CD38 | Thermofisher scientific | Hs01120071_m1 | |
| CD4 | Thermofisher scientific | Hs01058407_m1 | |
| CD41.ITGA2B | Thermofisher scientific | Hs01116228_m1 | |
| CD49f.ITGA6 | Thermofisher scientific | Hs01041011_m1 | |
| CD56.NCAM1 | Thermofisher scientific | Hs00941830_m1 | |
| CD9 | Thermofisher scientific | Hs00233521_m1 | |
| CD97 | Thermofisher scientific | Hs00173542_m1 | |
| CD99 | Thermofisher scientific | Hs00908458_m1 | |
| CDK6 | Thermofisher scientific | Hs01026371_m1 | |
| CDKN1A | Thermofisher scientific | Hs00355782_m1 | |
| CDKN1B | Thermofisher scientific | Hs01597588_m1 | |
| CDKN1C | Thermofisher scientific | Hs00175938_m1 | |
| CEBPa | Thermofisher scientific | Hs00269972_s1 | |
| CSF1r | Thermofisher scientific | Hs00911250_m1 | |
| CSF2RA | Thermofisher scientific | Hs00531296_g1 | |
| CSF3RA | Thermofisher scientific | Hs01114427_m1 | |
| E2A.TCF3 | Thermofisher scientific | Hs00413032_m1 | |
| EBF1 | Thermofisher scientific | Hs01092694_m1 | |
| ENG | Thermofisher scientific | Hs00923996_m1 | |
| EPOR | Thermofisher scientific | Hs00959427_m1 | |
| ERG | Thermofisher scientific | Hs01554629_m1 | |
| FLI1 | Thermofisher scientific | Hs00956711_m1 | |
| FLT3 | Thermofisher scientific | Hs00174690_m1 | |
| FOXO1 | Thermofisher scientific | Hs01054576_m1 | |
| GAPDH | Thermofisher scientific | Hs02758991_g1 | |
| GATA1 | Thermofisher scientific | Hs00231112_m1 | |
| GATA2 | Thermofisher scientific | Hs00231119_m1 | |
| GATA3 | Thermofisher scientific | Hs00231122_m1 | |
| GFI1 | Thermofisher scientific | Hs00382207_m1 | |
| HES1 | Thermofisher scientific | Hs01118947_g1 | |
| HLF | Thermofisher scientific | Hs00171406_m1 | |
| HMGA2 | Thermofisher scientific | Hs00171569_m1 | |
| HOXA5 | Thermofisher scientific | Hs00430330_m1 | |
| HOXB4 | Thermofisher scientific | Hs00256884_m1 | |
| ID2 | Thermofisher scientific | Hs04187239_m1 | |
| IGF2BP1 | Thermofisher scientific | Hs00198023_m1 | |
| IGF2BP2 | Thermofisher scientific | Hs01118009_m1 | |
| IKZF1 | Thermofisher scientific | Hs00172991_m1 | |
| IL1RAP | Thermofisher scientific | Hs00895050_m1 | |
| IL2RG | Thermofisher scientific | Hs00953624_m1 | |
| IRF8 | Thermofisher scientific | Hs00175238_m1 | |
| ITGB7 | Thermofisher scientific | Hs01565750_m1 | |
| KIT | Thermofisher scientific | Hs00174029_m1 | |
| Lin28B | Thermofisher scientific | Hs01013729_m1 | |
| LMO2 | Thermofisher scientific | Hs00153473_m1 | |
| LYL1 | Thermofisher scientific | Hs01089802_g1 | |
| Meis1 | Thermofisher scientific | Hs01017441_m1 | |
| mKi67 | Thermofisher scientific | Hs01032443_m1 | |
| MPL | Thermofisher scientific | Hs00180489_m1 | |
| MPO | Thermofisher scientific | Hs00924296_m1 | |
| NFIB | Thermofisher scientific | Hs01029175_m1 | |
| Notch1 | Thermofisher scientific | Hs01062011_m1 | |
| Pten | Thermofisher scientific | Hs02621230_s1 | |
| RAG2 | Thermofisher scientific | Hs01851142_s1 | |
| RPS18 | Thermofisher scientific | Hs01375212_g1 | |
| RUNX1 | Thermofisher scientific | Hs00231079_m1 | |
| Shisa2 | Thermofisher scientific | Hs01590823_m1 | |
| Spi1 | Thermofisher scientific | Hs02786711_m1 | |
| Sterile.IgH | Thermofisher scientific | Hs00378435_m1 | |
| TAL1 | Thermofisher scientific | Hs01097987_m1 | |
| THY1 | Thermofisher scientific | Hs00264235_s1 | |
| Tim.3.HAVCR2 | Thermofisher scientific | Hs00958618_m1 | |
| VWF | Thermofisher scientific | Hs00169795_m1 |
References
- Seita, J., Weissman, I. L. Hematopoietic stem cell: Self-renewal versus differentiation. Wiley Interdiscip Rev Syst Biol Med. 2 (6), 640-653 (2010).
- Orkin, S. H., Zon, L. I. Hematopoiesis: An evolving paradigm for stem cell biology. Cell. 132 (4), 631-644 (2008).
- Ye, F., Huang, W., Guo, G. Studying hematopoiesis using single-cell technologies. Journal of Hematology & Oncology. 10, 27 (2017).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nature Cell Biology. 16 (10), 919-927 (2014).
- Wills, Q. F., et al. Single-cell gene expression analysis reveals genetic associations masked in whole-tissue experiments. Nature Biotechnol. 31 (8), 748-752 (2013).
- Velten, L., et al. Human haematopoietic stem cell lineage commitment is a continuous process. Nat Cell Biol. 19 (4), 271-281 (2017).
- Wilson, N. K., Nicola, K., et al. Combined single-cell functional and gene expression analysis resolves heterogeneity within stem cell populations. Cell Stem Cell. 16 (6), 712-724 (2015).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: Advances and future challenges. Nucleic Acids Research. 42 (14), 8845-8860 (2014).
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of Single RNA Transcripts in Situ. Science. 280 (5363), 585-590 (1998).
- Kalisky, T., et al. A brief review of single-cell transcriptomic technologies. Briefings in Functional Genomics. , elx019 (2017).
- Crosetto, N., Bienko, M., van Oudenaarden, A. Spatially resolved transcriptomics and beyond. Nature Reviews Genetics. 16, 57 (2014).
- Bengtsson, M., Ståhlberg, A., Rorsman, P., Kubista, M. Gene expression profiling in single cells from the pancreatic islets of Langerhans reveals lognormal distribution of mRNA levels. Genome Research. 15 (10), 1388-1392 (2005).
- Bengtsson, M., Hemberg, M., Rorsman, P., Ståhlberg, A. Quantification of mRNA in single cells and modelling of RT-qPCR induced noise. BMC Molecular Biology. 9 (1), 63 (2008).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nature Methods. 10, 1096 (2013).
- Tung, P. -. Y., et al. Batch effects and the effective design of single-cell gene expression studies. Scientific Reports. 7, 39921 (2017).
- Teles, J., Enver, T., Pina, C. Single-cell PCR profiling of gene expression in hematopoiesis. Methods in Molecular Biology. , 21-42 (2014).
- Hayashi, T., et al. Single-cell gene profiling of planarian stem cells using fluorescent activated cell sorting and its "index sorting" function for stem cell research. Development, Growth, & Differentiation. 52 (1), 131-144 (2010).
- Lang, S., et al. SCExV: A webtool for the analysis and visualisation of single cell qRT-PCR data. BMC Bioinformatics. 16 (1), 320 (2015).
- de Klein, A., et al. A cellular oncogene is translocated to the Philadelphia chromosome in chronic myelocytic leukaemia. Nature. 300 (5894), 765-767 (1982).
- . indexed-sorting Available from: https://github.com/FlowJo-LLC/indexed-sorting (2016)
- Warfvinge, R., et al. Single-cell molecular analysis defines therapy response and immunophenotype of stem cell subpopulations in CML. Blood. 129 (17), 2384-2394 (2017).
- Nestorowa, S., et al. A single-cell resolution map of mouse hematopoietic stem and progenitor cell differentiation. Blood. 128 (8), e20-e31 (2016).
- Breton, G., et al. Human dendritic cells (DCs) are derived from distinct circulating precursors that are precommitted to become CD1c+ or CD141+ DCs. The Journal of Experimental Medicine. 213 (13), 2861-2870 (2016).
- Alberti-Servera, L., et al. Single-cell RNA sequencing reveals developmental heterogeneity among early lymphoid progenitors. The EMBO Journal. 36 (24), 3619-3633 (2017).
- Giustacchini, A., et al. Single-cell transcriptomics uncovers distinct molecular signatures of stem cells in chronic myeloid leukemia. Nature Medicine. 23, 692 (2017).
- Hansmann, L., Han, A., Penter, L., Liedtke, M., Davis, M. M. Clonal expansion and interrelatedness of distinct B-lineage compartments in multiple myeloma bone marrow. Cancer Immunology Research. 5 (9), 744-754 (2017).
- Psaila, B., et al. Single-cell profiling of human megakaryocyte-erythroid progenitors identifies distinct megakaryocyte and erythroid differentiation pathways. Genome Biology. 17 (1), 83 (2016).
- Schulte, R., et al. Index sorting resolves heterogeneous murine hematopoietic stem cell populations. Experimental Hematology. 43 (9), 803-811 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved