Method Article
Isolamento e extração de RNA de neurônios, macrófagos e Microglia de Zebrafish Larval cérebros
Neste Artigo
Resumo
Apresentamos um protocolo para isolar os neurônios, macrófagos e microglia do zebrafish larval cérebros sob condições fisiológicas e patológicas. Após o isolamento, RNA é extraído dessas células para analisar seu perfil de expressão do gene. Este protocolo permite para a coleção de RNA de alta qualidade para a realização de análise a jusante como qPCR e transcriptomics.
Resumo
Para obter uma compreensão detalhada do papel de células diferentes do CNS durante o desenvolvimento ou o estabelecimento e progressão do patologias do cérebro, é importante isolar essas células sem alterar seu perfil de expressão do gene. O modelo de zebrafish fornece um grande número de linhas de peixe transgênico em que tipos de células específicas são rotulados; por exemplo, neurônios na linha de NBT:DsRed ou macrófagos/microglia na linha de mpeg1:eGFP. Além disso, os anticorpos têm sido desenvolvidos para manchar a células específicas, tais como microglia com o 4-4 anticorpo.
Aqui, descrevemos o isolamento de neurônios, macrófagos e microglia do zebrafish larval cérebros. Central para este protocolo é evitar uma digestão enzimática do tecido a 37 ° C, o que poderia modificar perfis celulares. Em vez disso, é usado um sistema mecânico de homogeneização do tecido a 4 ° C. Este protocolo envolve homogeneização dos cérebros em suspensão de células, sua imuno-coloração e o isolamento de neurônios, macrófagos e microglia por FACS. Depois, extraído do RNA essas células e avaliada a sua qualidade/quantidade. Conseguimos obter RNA de alta qualidade (número de integridade do RNA (RIN) > 7) para realizar qPCR em análise macrófagos/microglia e neurônios e transcriptomic na microglia. Essa abordagem permite uma melhor caracterização destas células, bem como uma compreensão mais clara sobre o seu papel no desenvolvimento e patologias.
Introdução
Conhecimento sobre o desenvolvimento do cérebro e doenças cerebrais melhorou significativamente na última década desde a primeira quantificação de cérebro de rato transcriptomes1. Com efeito, análise de expressão de gene ampla do genoma nos dá acesso para detalhadas informações genéticas no tecido cerebral e as células que podem complementar e melhorar as observações feitas com outras técnicas e ferramentas.
O peixe-zebra é um potente modelo biológico, fáceis de criar e modificar geneticamente; sua transparência óptica em estágios larvas permite de observações de imagens ao vivo2. Infelizmente, em comparação ao humano e mouse, o número de anticorpos disponíveis para executar imuno-coloração é um pouco baixa. Para corrigir isso, linhas de peixe zebrafish transgênicos são feitas facilmente modificando geneticamente o peixe para expressar proteínas fluorescentes sob os promotores específicos de tipo de célula. Linhas de zebrafish transgênicos têm sido utilizadas no passado para estudar o papel de macrófagos e microglia durante o desenvolvimento do sistema nervoso central (SNC) e doença3,4,5,6. No entanto, para obter uma compreensão detalhada destes processos, precisamos entender mudanças na expressão gênica nos tipos de célula respectivos. Para esse fim, nós desenvolvemos um método experimental para isolar especificamente células como os neurônios, macrófagos e microglia de 3 a 8 dias cérebros de zebrafish larval pós fertilização (dpf). Para o estabelecimento do protocolo, trabalhamos com linhas de peixe transgênico que expressam a proteína verde fluorescente (GFP) em macrófagos/microglia sob o macrófago-expressa o gene promotor (mpeg1:eGFP) e DsRed nos neurônios sob a ß-tubulina neural promotor (NBT:DsRed)7,8,9. Além disso, realizamos imuno-coloração de micróglia usando 4 4, um anticorpo monoclonal de rato que especificamente manchas zebrafish microglia10,11. Depois, ácido ribonucleico (RNA) é extraído dessas células para mais quantitativas cadeia da polimerase (qPCR) ou análises de transcriptoma. Este protocolo foi projetado para homogeneizar eficientemente o tecido cerebral de zebrafish larvas; coletar os neurônios, macrófagos/microglia e microglia sem alteração de sua integridade de membrana plasmática e finalmente extrair RNA destas células em alta qualidade (RIN > 7) e a quantidade para executar análise genômica. Ao contrário de estudos anteriormente publicados que usam o tratamento de tripsina a 37 ° C para digerir o cérebro tecido12,13, este protocolo promove o trabalho em 4 ° C até a etapa de extração de RNA para reduzir as modificações do perfil de expressão do gene. Esta etapa é crucial como microglia e os macrófagos são células altamente sensíveis que respondem às mudanças em seu microambiente imediatamente, alterando seu gene expressão perfil e polarização14,15, 16.
O protocolo, aqui descrito em detalhe, mostra que o isolamento de neurônios, macrófagos e microglia do zebrafish larval cérebros, mas virtualmente, pode ser adaptado a qualquer outra célula presentes dentro do cérebro - usando linhas de peixe transgénicos ou rotulados com anticorpos específicos. Este método irá permitir uma melhor caracterização das células CNS através de suas análises de expressão de gene ampla de genoma e ajudará a compreender o seu papel durante o desenvolvimento e doenças cerebrais.
Protocolo
1. amostra e preparação de meios de comunicação
- Prepare o suporte de embrião (E3) pela dissolução de 6,4 mM KCl, 0,22 mM NaCl, 0,33 mM CaCl2 2 H2O, 0,33 mM MgSO4 7 H2O em H2O.
- Colete embriões zebrafish imediatamente após a fertilização (0 dpf).
- Dividi o zebrafish embriões em 50 por prato de petri de 90 mm. Criar embriões a 28,5 ° C em 50 mL de meio de embrião (E3) tratado com 200 µM 1-fenil-2-tioureia (PTU), do final do primeiro dia do desenvolvimento (0 dpf) para a duração do experimento para inibir a pigmentação.
- Muda o E3 + médio PTU diariamente para a duração do experimento.
- Tela mpeg1:eGFP e larvas de NBT:DsRed no dpf 2 usando um microscópio fluorescente para a expressão do transgene positivo, macrófagos GFP+ /microglia e DsRed+ neurônios.
- Preparem-se todos os meios no dia anterior o experimento sob uma capa de cultura de tecidos para evitar a contaminação e, em seguida, armazene-os em 4 ° C.
- Preparar a mídia A dissolvendo Hepes de 15 mM e 25 mM D-glicose em HBSS 1 x.
- Prepare o médio gradiente de densidade (100%) misturando 9 volumes do meio de gradiente de densidade de 1 volume de HBSS 10 x.
- Prepare o suporte de gradiente de densidade (22%) diluindo 22 mL de meio de gradiente de densidade (100%) em 88 mL de DPBS 1 x.
- Preparar DPBS 1 x.
- Prepare E3 médio + tricaina misturando E3 médio com 450 μM tricaina.
2. homogeneização
Nota: Todas as etapas são realizadas 4 ° C.
- Adicione 1,5 mL metanosulfonato (15 mM) de 90 mm placas de Petri contendo 50 larvas em 50 mL de meio de embrião E3 tratado com 200 µM PTU para anestesia terminal.
- Puxa-saco 10 larvas anestesiadas desde os pratos de petri com uma mL 3 pipetas de plástico grosso.
- Larvas de transferência anestesiada 10 por 10 em uma placa de Petri 55mm repleto de gelada médio de embrião de E3 + tricaina.
- Sob um estereomicroscópio, alinhe 10 larvas no centro da caixa de Petri. Então transecto larvas cabeças acima o saco vitelino-usando microtesoura cirúrgica (excluir a bexiga natatória para evitar flutuação cabeças).
- Puxa-saco cabeças desde os pratos de petri com uma mL 3 pipetas de plástico grosso. Espere até que todas as cabeças se reúnem dentro a ponta da pipeta e em seguida, transferi-los para um homogeneizador de vidro contendo 1 mL gelada Media A (transferência em um volume mínimo para reduzir a mídia uma diluição por E3 + tricaina). Manter o homogenizador de vidro no gelo. Use um homogeneizador por condição experimental.
- Substituir cada pequeno prato de petri contendo gelo frio E3 + tricaina com um novo cada 30 min para assegurar essa transecção é executada no frio E3 + tricaina médio.
- Substitua o gelado A mídia no homogenizador de vidro quando a cor começa a desvanecer-se.
Nota: Diluição de A mídia pode alterar o tecido cabeça devido a mudanças de temperatura. - Uma vez que todos os chefes foram coletados (600 cabeças/condição), remover o volume máximo da mídia A partir do homogenizador de vidro e substituí-lo com 1 mL da gelada fresco Media.
- Perturbar o tecido do cérebro com um homogeneizador de vidro apertado no gelo. Execute 40 rodadas de esmagamento e voltas para larvas de dpf de 3-5 e 50 para larvas de dpf 7 e 8.
- Adicionar 2 mL da mídia à suspensão de células (1 mL A de mídia / 200 cabeças), que irá diluir as células e reduzir sua aglomeração com mielina para facilitar sua separação durante a centrifugação em gradiente médio de densidade.
- Para eliminar a aglomeração de célula, execute a suspensão de células através de um filtro de célula de 40 µm colocado em cima de um tubo de Falcão frio 50ml mantido no gelo. Repita esta operação 3 vezes.
- Transferir 1 mL da suspensão de células para tubos de frio 1,5 mL e girá-los a 300 g por 10 min a 4 ° C.
- Remover o sobrenadante utilizando uma seringa de 10 mL + agulha 23 x 1 '.
- Ressuspender o centrifugado com 1 mL de meio de gradiente gelada 22% densidade levemente sobreposto por 0,5 mL de DPBS gelada 1 x (do não mistura-los, uma interfase entre ambas as soluções serão vistos).
- Girar os tubos em 950 g sem freio e aceleração lenta por 30 min a 4 ° C.
Nota: Este passo separa a mielina de outras células pelo sequestro de no período do intérfase de DPBS 1 x e 22% médio gradiente de densidade, Considerando que células serão sedimento no fundo do tubo. Remoção de mielina é mais eficiente quando a concentração de célula não é muito alta. - Usando uma seringa de 10 mL + agulha 23 x 1 ' descartar o máximo de DPBS, médio gradiente de densidade e mielina preso na sua interfase.
- Lavam-se células com 0,5 mL da mídia A + 2% de soro de cabra normal (NGS) e, em seguida, girar tubos a 300 g por 10 min a 4 ° C.
- Descartar o máximo do líquido sobrenadante e, em seguida, juntar todos os chumbos de célula da mesma condição experimental junto em 1 mL de mídia A + 2% NGS.
- Girar os tubos em 950 g sem freio e aceleração lenta por 30 min a 4 ° C.
- Se as células de interesse expressam uma proteína fluorescente como macrófagos/microglia de mpeg1:eGFP ou neurônios de NBT:DsRed peixe transgénicos (screening de peixe transgénicos ver 1.1.3), executar a suspensão de células através de um filtro de célula de 35 µm cap e transferi-los para uma tubos de frio 5 mL FACS no gelo, protegidos da luz.
Nota: Alternativamente, a imuno-coloração da microglia pode ser executada.
3. Microglia Immuno-coloração
Nota: Todas as etapas são executadas a 4 ° C.
- Ressuspender o centrifugado com 0,3 mL de mídia A + 2% NGS. Dividi-los em tubos de 3 x 1.5 mL: um para células imaculadas medir a autofluorescência de células de interesse, em segundo lugar para o anticorpo secundário (1/200) medir a ligação não-específica do anticorpo secundário para microglia e, terceiro, como um teste (4 4 mouse monoclonal anticorpo (microglia específico) (1/20) + (1/200) de anticorpo secundário).
- Adicione baixa endotoxina, livre de azida sódica (folha) em 1% para as células (todos os tubos) para bloquear CD16/CD32 interações com o domínio do Fc de imunoglobulinas. Incube as células por 10 min com agitação suave cada 5 min.
- Adicionar 4 a 4 anticorpo (1/20) para as células (tubo 3) e incube por 30 min com agitação suave a cada 10 min.
- Girar os tubos a 300 g por 10 min a 4 ° C e, em seguida, descartar o sobrenadante.
- Lave uma vez com 0,5 mL da mídia A + 2% NGS, então girar tubos a 300 g por 10 min a 4 ° C.
- Ressuspender o centrifugado com 0,5 ml de mídia A + 2% NGS e incubar as células com folha em 1% por 10 min com agitação suave cada 5 min.
- Adicione o anticorpo secundário (1/200) para as células (tubo 2 e 3). Incube as células por 30 min com agitação suave cada 10 min e protecção leve.
- Girar os tubos a 300 g por 10 min a 4 ° C e, em seguida, descartar o sobrenadante.
- Lave duas vezes com 0,5 mL de mídia A + 2% NGS, então a re-suspender centrifugado com 1 mL de mídia A + 2% NGS.
- Executar a suspensão de células através de uma tampa de filtro de célula µm 35 e transferi-los para tubos de 5ml frio FACS no gelo, protegido da luz.
4. célula de triagem (FACS)
Nota: Executar todas as etapas, a 4 ° C.
- Classificar os neurônios, macrófagos/microglia e microglia usando um FACS.
Nota: Esta etapa é geralmente realizada por um membro do pessoal da instalação de FACS e configurações dependem do tipo de equipamento utilizado.- Adicione DAPI em uma concentração de 1 µ g/mL em cada tubo de FACS para rotular as células mortas.
- Configurar o FACS e tipo de neurônios, macrófagos/microglia ou microglia de todas as células do cérebro. Separar células de detritos em função do seu tamanho e granulosidade e, em seguida, single-células por dispersão para a frente e dispersão do lado da porta. Exclua células mortas por DAPI rotulagem de células vivas. Identifica os neurônios, macrófagos/microglia ou microglia por seus respectivos coloração positiva.
- Recolher pilhas em tubos de 1,5 mL contendo 1 mL de gelado A mídia + 2% NGS em gelo. Use tubos diferentes para cada tipo de célula.
- Girar os tubos a 300 g por 10 min a 4 ° C e em seguida, descartar o sobrenadante.
- Lave uma vez com 0,5 mL de mídia A então descartar o sobrenadante no máximo.
5. extração do RNA
- Extraia o RNA dos diferentes tipos de células separados por FACS. Para a extração de RNA usar um kit específico e siga as orientações do fabricante. Certifique-se para trabalhar em um ambiente livre de RNase por pipetas com um produto de descontaminação de RNase e limpeza do espaço de trabalho e usar filtro dicas.
- Recentemente Prepare-se 1 mL de tampão de Lise suplementado com 50 µM β-Mercaptoetanol.
Nota: Aqui, β-Mercaptoetanol é adicionado para reduzir a degradação de RNA. - Prepare a solução de etanol 70% e 80% usando RNase água livre.
- Lyse pilhas com 75 µ l de tampão de Lise suplementado com 50 µM β-Mercaptoetanol. Em seguida, use uma máquina trituradora para melhorar o rompimento da pilha.
- Continue manual de extração de RNA de acordo com o fabricante.
- No final do protocolo, Eluir o RNA com 14 µ l de água livre de RNase para obter uma concentração suficiente de RNA.
- Recentemente Prepare-se 1 mL de tampão de Lise suplementado com 50 µM β-Mercaptoetanol.
Resultados
O protocolo descrito é uma abordagem simples para isolar os neurônios, macrófagos e microglia do zebrafish larval cérebros. A partir desses isolados de células, uma quantidade significativa de alta qualidade (RIN > 7) RNA foram extraídos. O objectivo do presente protocolo é isolar diferentes tipos de células do SNC, com mínima modificação do seu perfil de expressão do gene para analisar e caracterizar as funções e propriedades da célula. Portanto, o protocolo inteiro é realizado a 4 ° C, com uma homogeneização do tecido de cérebro mecânico. Este método tem sido utilizado com sucesso para dois estudos realizados em laboratório. No primeiro estudo, os neurônios e macrófagos/microglia foram isoladas de 8 dpf mpeg1:eGFP+/NBT:DsRed+ larva (Figura 1). FACS permitida a separação da pilha de detritos em função do seu tamanho (FSC-A) e granularidade (SSC-A) (Figura 1A). Células únicas foram então separadas de parelhas ou aglomerados de células (Figura 1B). Da população única célula, um portão foi desenhado para eliminar as células mortas (DAPI+). O enredo de ponto correspondente revelou que este protocolo experimental preserva a integridade da membrana plasmática celular, como a taxa de células mortas é apenas 26,7% (Figura 1C). Finalmente, neurônios (DsRed+) e macrófagos/microglia (GFP+) eram facilmente separavam os portões de população de células vivas. A população de neurônio (23,1%) parecia ser mais proeminente do que a população de macrófagos/microglia (1,56%) dentro do cérebro (Figura 1D). Este protocolo permitiu para isolar o RNA dessas células para realizar análises subsequentes qPCR para comparar a expressão de genes específicos entre os neurônios e macrófagos/microglia. A Figura 2 mostra neuronal e os níveis de expressão do gene de macrófagos/microglia do antígeno nuclear de proliferação celular (pcna)contra gene β-actina domésticas, por exemplo.
O segundo estudo este método focado em microglia isolamento de 3, 5 e 7 dpf cérebros larval. Em contraste com o experimento descrito acima, as células foram isoladas por imuno-coloração usando 4 4, um anticorpo que rotula especificamente microglia (Figura 3 A-D). Como descrito anteriormente, microglia (4 4+) foram selecionados a partir de células vivas e coletados (Figura 3-D). Microglia números dentro zebrafish larval cérebros são variáveis (tabela 1) e muito baixa no dpf 3 (∼ 25 por peixes). Qualidade e quantidade de RNA extraído dessas células foram medidos utilizando um sistema de eletroforese capilar micro baseado. Resultados obtidos do RNA extraído de micróglia 5 dpf zebrafish larval cérebros foram fornecidos para ilustrar um exemplo de análise de RNA (tabela 1 (dpf 5; experimento 4)). A Figura 4 mostra o rastreamento de eletroforese e sua representação gráfica obtidos para esta amostra com uma visualização clara do RNA ribossomal (28s e 18s). Estes dados são necessários para calcular a amostra RIN e determinar a concentração de RNA. A tabela 1 resume o número de isolados microglia por peixes, a quantidade de RNA por microglia e o escore de RIN obtido para cada experimento diferente em 3, 5 e 7 dpf. A quantidade e a qualidade do RNA extraído de micróglia isolado usando este método permitiram a amplificar o RNA em cDNA usando um kit. Testes de qualidade e quantidade fornecidos por Edimburgo genómica confirmam que o cDNA amplificado de qualidade suficiente para a preparação de biblioteca e posterior sequenciamento. A Figura 5 mostra a distribuição de tamanho de fragmentos de cDNA e sua quantidade medido utilizando um sistema eletroforético. Neste exemplo, o cDNA tinha um tamanho médio de 299bp em uma concentração de 36100 pmol/l. tabela 2 ilustra, respectivamente, qualidade e quantidade de testes feitos no cDNA amplificado de amostras do RNA (tabela 1 (dpf 5; experimento 4)). O cDNA amplificado tem sido usado com sucesso para o sequenciamento.
Vários estudos realizados em laboratório confirmaram que a qualidade e a quantidade de RNA extraído de neurônios, macrófagos e microglia podem ser usados para qPCR subsequente e as análises de expressão de gene ampla do genoma. Portanto, este protocolo experimental pode ser usado para isolar confiantemente diferentes tipos de células do CNS sem alterar sua integridade de membrana e limitando a modificação do seu perfil de expressão do gene.

Figura 1 : FACS 8 dpf zebrafish larvas de classificação para os neurônios e macrófagos/microglia do /NBT:DsRed mpeg1:GFP++ . (A-C) Gating sucessivas mostra seleção sequencial de todo cérebro células (A), dispersão de células únicas por dispersão para a frente e lateral (B). Células (C) mortos foram excluídas pela rotulagem DAPI. (D) os neurônios e macrófagos/microglia foram identificados respectivamente pela coloração positiva DsRed e GFP. Clique aqui para ver uma versão maior desta figura.
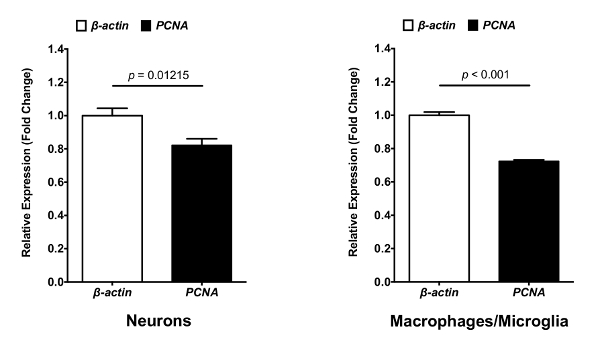
Figura 2 : Análise de expressão do Gene para pcna e β-actina nos neurônios e macrófagos/microglia. RNA de neurônios isolados e macrófagos/microglia pode ser transcrito em cDNA para uso em análises quantitativas de PCR. níveis de expressão de RNAm de pcna contra gene β-actina domésticas em neurônios isolados e macrófagos/microglia determinado pelo qPCR (N = 3). Mudança de dobra foi medida utilizando o método comparativo (ΔΔCT). Erro da barra representam média ± SEM. clique aqui para ver uma versão maior desta figura.

Figura 3 : FACS classificação para microglia de 3 larvas de zebrafish dpf. (A-C) Sucessivas sequencial Mostrar seleção associada do cérebro todas as células (A), dispersão de células únicas por dispersão para a frente e lateral (B). Células (C) mortos foram excluídas pela rotulagem DAPI. (D) Microglia foram identificados por 4 4 coloração positiva. Clique aqui para ver uma versão maior desta figura.

Figura 4 : Resultados de eletroforese capilar-micro do RNA extraído das 5 dpf zebrafish microglia. Os dois picos altos são o RNA ribosomal 18S e 28S. Número de integridade do RNA (RIN) foi calculado automaticamente pelo software bioanalyzer usando a relação gerada da 18S e 28S subunidades ribossomais e a análise do rastreamento eletroforética inteira. Microglia RNA tem um RIN 8,6. Clique aqui para ver uma versão maior desta figura.

Figura 5 : Testes de qualidade e quantidade do cDNA amplificado de amostra de RNA de 5 dpf zebrafish microglia. A imagem mostra a distribuição de tamanho de fragmento do cDNA da amostra analisada com um tamanho médio de 299 BP clique aqui para ver uma versão maior desta figura.
| Condição | Experimento | Número de peixes | Número do celular | Número de células por peixes | Concentração de RNA (pg/ul) | RNA total (pg) | Quantidade de RNA por célula (pg) | Pontuação de RIN |
| 3dpf | 1 | 700 | 11922 | 17,03 | 126 | 1512 | 0.13 | 8 |
| 3dpf | 2 | 600 | 22527 | 37.55 | 253 | 3036 | 0.13 | 7,9 |
| 3dpf | 3 | 600 | 18688 | 31.15 | 255 | 3060 | 0.16 | 7.7 |
| 3dpf | 4 | 600 | 11121 | 18.54 | 189 | 2268 | 0.20 | 7,8 |
| 3dpf | 5 | 600 | 15581 | 25.97 | 131 | 1572 | 0,10 | 8.4 |
| 3dpf | 6 | 600 | 11965 | 19,94 | 256 | 3072 | 0,26 | 8.2 |
| 5dpf | 1 | 600 | 58629 | 97.72 | 362 | 4344 | 0.07 | 7.4 |
| 5dpf | 2 | 600 | 32510 | 54,18 | 348 | 4176 | 0.13 | 8.1 |
| 5dpf | 3 | 600 | 77884 | 129.81 | 594 | 7128 | 0,09 | 8.3 |
| 5dpf | 4 | 600 | 50755 | 84.59 | 305 | 3660 | 0.07 | 8.6 |
| 5dpf | 5 | 600 | 44967 | 74.95 | 134 | 1608 | 0,04 | 7.6 |
| 5dpf | 6 | 600 | 51031 | 85.05 | 163 | 1956 | 0,04 | 7,9 |
| 7dpf | 1 | 600 | 60496 | 100.83 | 183 | 2196 | 0,04 | 7.6 |
| 7dpf | 2 | 450 | 55517 | 123.37 | 183 | 2196 | 0,04 | 7,8 |
| 7dpf | 3 | 600 | 88897 | 148.16 | 465 | 5580 | 0,06 | 8.1 |
| 7dpf | 4 | 600 | 63008 | 105.01 | 356 | 4272 | 0.07 | 8.4 |
| 7dpf | 5 | 350 | 34956 | 99.87 | 245 | 2940 | 0.08 | 8.1 |
| 7dpf | 6 | 600 | 63887 | 106.48 | 341 | 4092 | 0,06 | 7,8 |
Tabela 1: Resumo de isolamento microglia e dados de extração de RNA de 3, 5 e 7 dpf zebrafish larvas.
| ID de amostra interna | ID de amostra externo | Qubit (ng/ul) | Qubit(ng/ul) | Concentração média (ng/ul) | Volume (ul) | UG recebido | Aprovação/reprovação para concentração mínima | Aprovação/reprovação para quantidade de minimim | Aprovação/reprovação para quantidade recomendada |
| 10907SD0010 | 5 dpf (experimento 4) | 58,8 | 58,4 | 58,6 | 30 | 1.76 | Passar | Passar | Passar |
| Requisitos de amostra para a preparação da biblioteca: | |||||||||
| Biblioteca Prep | Quantidade mínima (ng) | Quantidade recomendada (ng) | Mínima concentração ng/uL | ||||||
| TruSeq Nano gel biblioteca inserção livre bp 350 de cDNA | 600 | 1100 | 10 | ||||||
Tabela 2: Testes de quantidade de cDNA amplificado de amostra de RNA de 5 dpf zebrafish microglia.
Discussão
O protocolo experimental descrito aqui representa um método robusto e eficiente para isolar as células do cérebro de zebrafish larvas de 3 a 8 dpf. Importante, este é o primeiro protocolo que permite o isolamento específico da microglia do zebrafish larval cérebros. O protocolo é projetado para preservar a integridade da membrana celular e para minimizar possíveis modificações da expressão genética ocorrem durante o processamento. Este último ponto é crucial para a relevância dos resultados com base na análise desses perfis genômicos celulares isolados. Com efeito, polarização microglia e macrófagos são fortemente influenciados pelo seu microambiente. A 37 ° C, estas células teria mudado seu perfil de expressão de gene em resposta a condições experimentais (resposta de lesão (transecção)). Portanto, foi crucial para realizar este experimento a 4 ° C antes da extração de RNA, para retardar processos celulares e atividades metabólicas. Além disso, a homogeneização do tecido de cérebro mecânico a 4 ° C foi escolhida em vez de digestão enzimática do tecido a 37 ° C para evitar qualquer impacto sobre perfis de expressão de gene.
É importante salientar que este método é muito rápido; dentro de um dia de várias populações de células do cérebro podem ser isoladas de pelo menos duas diferentes condições experimentais e seu RNA extraído. O comprimento total do protocolo depende do número de larvas usados para cada condição como a transecção de cabeças larvas é o passo limitante (∼ 350 cabeças/h). Em geral, para trabalhar com microglia de 3, 5 e 7 dpf é aconselhável transecto 600 cabeças ∼ por teste para obter suficiente RNA para extrair (tabela 1). Como micróglia representam o tipo de célula com o menor rendimento (aproximadamente 112 células per capita no dpf 7), o número de cabeças pode ser reduzido para outros tipos de células, incluindo os macrófagos (aproximadamente 170 células per capita em 7 dpf).
Outra vantagem deste protocolo é que, uma vez que se estabeleceram as configurações no classificador de FACS, as mesmas configurações podem ser usadas para diferentes experiências. Tem sido observado que populações de células se encaixam perfeitamente de um experimento para outro com portões previamente concebidos, mostrando a reprodutibilidade de experimentos usando esse método.
Uma ligeira desvantagem desse método é a relativamente baixa quantidade de RNA que é colhido. No entanto, esta limitação é mais uma questão biológica do que um problema técnico, como o número de micróglia é muito baixo em estágios iniciais de desenvolvimento do cérebro (tabela 1). Por causa desta baixa quantidade de RNA coletado, etapas de amplificação precisam ser considerados para executar análise de expressão de gene ampla do genoma. Felizmente, estas etapas de amplificação produzem quantidades suficientes de cDNA de alta qualidade. Assim, mudanças globais nos perfis de expressão do gene de células isoladas podem ser estudadas.
Em conclusão, este protocolo fornece um método robusto para isolar e estudar vários tipos de células do CNS de cérebros de zebrafish larval. Isto pode ser aplicado para ganhar uma compreensão mais profunda dessas células durante o desenvolvimento, bem como para estudar o seu papel na doença.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Agradecemos o Dr. Claire Davis e Dr. Miron Veronique (a rainha Instituto de investigação médica, Edimburgo, Kingdome Unidos) pela ajuda inicial e discussões sobre a abordagem experimental, a estrutura de classificação de células e a citometria de fluxo QMRI. Este trabalho foi apoiado por um Cancer Research UK carreira estabelecimento Award para Dr. Dirk Sieger.
Materiais
| Name | Company | Catalog Number | Comments |
| 1-phenyl 2-thiourea (PTU) | Sigma | P7629 | |
| Hepes | Gibco | 15630-056 | |
| D-Glucose | Sigma | G8644-100ML | |
| HBSS 1X | Gibco | 14170-088 | |
| Percoll | GE Healthcare | 17-0891-02 | |
| HBSS 10X | Gibco | 14180-046 | |
| DPBS 1X | Gibco | 14190-094 | |
| Tricaine (MS222) | Sigma | A5040 | |
| Sterilin standard 90mm petri dishes | ThermoFisher | 101VIRR | |
| Surgical micro-scissors | Fine Science Tools | 15000-00 | |
| 3 mL Pasteur plastic bulk pipette | SLS | PIP4206 | |
| Glass homogenizer | Wheaton | 357538 | |
| Sterilin standard 55mm petri dishes | ThermoFisher | P55V | |
| Percoll | GE Healthcare | 17-0891-02 | |
| 40 µm cell strainer | Falcon | 352340 | |
| 50 ml polypropylen conical tube | Falcon | 352070 | |
| Centrifuge | Eppendorf | 5804 R | |
| 10 mL syringe | BD | 302188 | |
| Needle 23G x 1'' | BD | 300800 | |
| Normal goat serum (NGS) | Cell Signalling | 5425S | |
| 35 μm cell strainer cap | BD | 352235 | |
| FACS tubes | BD | 352063 | |
| Low Endotoxin, Azide-Free (LEAF) | Biolegend | 101321 | |
| Alexa Fluor 647 Goat Anti-Mouse IgG (H+L) | Life Technologies | A11008 | |
| Anti-4C4 | Courtesy of Catherina Becker (University of Edinburgh) | ||
| FACS sorter FACSAria II | BD | QMRI, FACS facility | |
| RNeasy Plus Micro Kit | QIAGEN | 74034 | |
| β-mercaptoethanol | Gibco | 31350-010 | |
| QIAshredder | QIAGEN | 79654 | |
| SsoAdvanced Universal SYBR Green Supermix | Bio-Rad | 1725271 | |
| SuperScript III First-Strand Synthesis System | Invitrogen | 18080-051 | |
| LightCycler 96 Real-Time PCR System | Roche | ||
| Ovation RNA-Seq System V2 | NuGEN | 7102-32 | |
| Agilent RNA 6000 Pico reagents | Agilent | 5067-1513 | |
| 2100 Bioanalyzer | Agilent | ||
| RNaseZap | Ambion | AM9780 |
Referências
- Chrast, R., Scott, H. S., et al. The Mouse Brain Transcriptome by SAGE: Differences in Gene Expression between P30 Brains of the Partial Trisomy 16 Mouse Model of Down Syndrome (Ts65Dn) and Normals. Genome Research. 10 (12), 2006-2021 (2000).
- Beis, D., Stainier, D. Y. R. In vivo cell biology: following the zebrafish trend. Trends in Cell Biology. 16 (2), 105-112 (2006).
- Shiau, C. E., Kaufman, Z., Meireles, A. M., Talbot, W. S. Differential Requirement for irf8 in Formation of Embryonic and Adult Macrophages in Zebrafish. PLoS ONE. 10 (1), 0117513-0117515 (2015).
- Mazaheri, F., Breus, O., et al. Distinct roles for BAI1 and TIM-4 in the engulfment of dying neurons by microglia. Nature Communications. 5, 1-11 (2014).
- Oosterhof, N., Holtman, I. R., et al. Identification of a conserved and acute neurodegeneration-specific microglial transcriptome in the zebrafish. Glia. 65 (1), 138-149 (2016).
- Hamilton, L., et al. A Zebrafish Live Imaging Model Reveals Differential Responses of Microglia Toward Glioblastoma Cells In Vivo. Zebrafish. , (2016).
- Ellett, F., Pase, L., Hayman, J. W., Andrianopoulos, A., Lieschke, G. J. mpeg1 promoter transgenes direct macrophage-lineage expression in zebrafish. Blood. 117 (4), 49-56 (2011).
- Peri, F., Nüsslein-Volhard, C. Live Imaging of Neuronal Degradation by Microglia Reveals a Role for v0-ATPase a1 in Phagosomal Fusion In Vivo. Cell. 133 (5), 916-927 (2008).
- Sieger, D., Moritz, C., Ziegenhals, T., Prykhozhij, S., Peri, F. Long-range Ca2+ waves transmit brain-damage signals to microglia. Developmental cell. 22 (6), 1138-1148 (2012).
- Becker, T., Becker, C. G. Regenerating descending axons preferentially reroute to the gray matter in the presence of a general macrophage/microglial reaction caudal to a spinal transection in adult zebrafish. The Journal of comparative neurology. 433 (1), 131-147 (2001).
- Ohnmacht, J., Yang, Y., et al. Spinal motor neurons are regenerated after mechanical lesion and genetic ablation in larval zebrafish. Development. 143 (9), Cambridge, England. 1464-1474 (2016).
- Roy-Carson, S., Natukunda, K., et al. Defining the transcriptomic landscape of the developing enteric nervous system and its cellular environment. BMC Genomics. 18 (1), 1-24 (2017).
- Khuansuwan, S., Gamse, J. T. Identification of differentially expressed genes during development of the zebrafish pineal complex using RNA sequencing. Developmental biology. 395 (1), 144-153 (2014).
- Schmid, C. D., Melchior, B., et al. Differential gene expression in LPS/IFNγ activated microglia and macrophages: in vitroversus in vivo. Journal of Neurochemistry. 109, 117-125 (2009).
- Bohlen, C. J., Bennett, F. C., Tucker, A. F., Collins, H. Y., Mulinyawe, S. B., Barres, B. A. Diverse Requirements for Microglial Survival, Specification, and Function Revealed by Defined- Medium Cultures. Neuron. 94 (4), 759-773 (2017).
- Khan, A., Ju, F., et al. Transcriptomic analysis reveals differential activation of microglial genes after ischemic stroke in mice. Neuroscience. 348, 212-227 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados