Method Article
절연 및 애벌레 Zebrafish 두뇌에서 신경 세포, 대 식 세포 및 Microglia RNA 추출
요약
우리는 신경 세포, 대 식 세포 및에서 생리 및 병 적인 조건 하에서 애벌레 zebrafish 두뇌 microglia 분리 프로토콜을 제시. 절연, 시 RNA 유전자 식 프로필 분석 하이 셀에서 추출 됩니다. 이 프로토콜 정량 및 transcriptomics 같은 다운스트림 분석에 대 한 높은-품질 RNA의 컬렉션에 대 한 수 있습니다.
초록
개발 또는 설립 중 다른 CNS 세포의 역할에 대 한 상세한 이해 및 두뇌 병 리의 진행을 얻으려면, 그것은 그들의 유전자 식 프로 파일을 변경 하지 않고 이러한 세포를 분리 하는 것이 중요. Zebrafish 모델 제공 많은 유전자 변형 물고기 라인 있는 특정 세포 유형 표시 됩니다; NBT:DsRed 선 또는 대 식 세포/microglia mpeg1:eGFP 라인에서 하는 예를 들어 신경에 대 한 또한, 항 체 개발 되었습니다 microglia 4C 4와 같은 특정 세포를 얼룩이 항 체.
여기, 우리 신경 세포, 대 식 세포에서 애벌레 zebrafish 두뇌 microglia의 설명. 이 프로토콜은 셀룰러 프로필을 수정할 수 있는 37 ° C에서 효소 조직 소화의 회피입니다. 4 ° C에서 조직 균질의 기계 시스템 대신 사용 됩니다. 이 프로토콜 세포 현 탁 액으로 두뇌의 균질, 그들의 immuno 얼룩이 지 고 신경 세포, 대 식 세포 및 FACS에 의해 microglia의 격리를 의미 한다. 이후에, 우리는 그 세포에서 RNA를 추출 하 고 그들의 품질/수량 평가. 우리는 고품질의 RNA를 관리 (RNA 무결성 번호 (린) > 7) 세포/microglia 및 신경, 그리고 transcriptomic microglia 분석에서 정량 수행 하. 이 방법은 개발 및 pathologies에 그들의 역할에 대해 명확 하 게 이해로 서이 세포의 더 나은 특성을 수 있습니다.
서문
두뇌 개발 및 뇌 질환에 대 한 지식을 마우스 뇌 transcriptomes1의 첫 번째 정량화 이후 지난 10 년간에서 크게 개선 했다. 실제로, 게놈 넓은 유전자 표정 분석 우리에 액세스할 뇌 조직 및 보완 하 고 관측 다른 기술 및 도구를 향상 시킬 수 있는 셀에 대 한 자세한 유전 정보.
제 브라는 쉽게 번 식 하 고 유전자; 수정 강력한 생물 학적 모델 애벌레 단계에서 광학 투명도 라이브 이미징 관찰2수 있습니다. 불행 하 게도, 인간을 비교 하 고 마우스, immuno 얼룩이 수행에 사용할 수 있는 항 체의 수는 오히려 낮은. 이 문제를 해결 하려면 유전자 변형 zebrafish 물고기 라인 쉽게 만들어집니다 유전자 물고기를 수정 하 여 셀 형식 특정 발기인에서 형광 단백질을 표현 하. 유전자 변형 zebrafish 라인 중앙 신경 시스템 (CNS) 개발 및 질병3,4,,56동안 microglia 및 대 식 세포의 역할을 공부 하는 과거에 사용 되었습니다. 그러나, 이러한 프로세스의 상세한 이해를 얻기 위해 우리가 해당 셀 형식에서 유전자 발현에 변화를 이해 해야 합니다. 이 목표를 우리는 특별히 신경 세포, 대 식 세포 및 8 일 3에서 microglia 같은 세포를 분리 하는 실험 방법 개발 후 수정 (dpf) 애벌레 zebrafish 두뇌. 프로토콜의 설립에 대 한 우리 신경 신경 ß-tubulin 아래에 있는 대 식 세포 표현 유전자 발기인 (mpeg1:eGFP) 및 DsRed 세포/microglia에서 녹색 형광 단백질 (GFP)를 표현 하는 유전자 변형 물고기 라인 근무 발기인 (NBT:DsRed)7,,89. 또한, 우리는 immuno 얼룩이 microglia 4C 4, 특별히 zebrafish microglia10,11얼룩 마우스 단일 클론 항 체를 사용 하 여의 수행. 이후에, ribonucleic 산 (RNA) 추가 정량적 중 합 효소 연쇄 반응 (정량) 또는 transcriptome 분석에 대 한 이러한 세포에서 추출 됩니다. 이 프로토콜 zebrafish 애벌레;에서 뇌 조직을 효율적으로 균질 하도록 설계 되었습니다. 신경 세포, 세포/microglia 및 그들의 원형질 막 무결성의 변경 없이 microglia를 수집 하 고 마지막으로 높은 품질의 이러한 세포에서 RNA 추출 (린 > 7) 및 게놈 분석을 수행 하는 수량. 뇌 조직12,13소화를 트립 신 처리 37 ° C에서 사용 하는 연구에 대 한 이전 게시, 달리이 프로토콜 RNA 추출 단계를 줄이기 위해 유전자 식 프로필의 수정까지 4 ° C에서 작업을 촉진 합니다. 이 단계는 결정적으로 microglia 및 대 식 세포는 매우 중요 한 세포는 그들의 진 식 프로필과 분극14,15, 를 변경 하 여 즉시 그들의 microenvironment에 있는 변화에 응답 16.
세부 사항에, 여기에 설명 된 프로토콜 신경 세포, 대 식 세포 및 microglia zebrafish 애벌레 두뇌에서 거의 그것의 다른 셀에 적용할 수 있습니다 보여줍니다 뇌-유전자 변형 물고기 라인을 사용 하 여 내 제시 또는 함께 표시 특정 항 체입니다. 이 방법은 그들의 게놈 넓은 유전자 표정 분석을 통해 CNS 세포의 더 나은 특성화 고 개발 및 뇌 질환 중 그들의 역할을 이해 하는 데 도움이 됩니다.
프로토콜
1. 샘플 및 미디어 준비
- 6.4 m KCl m, 0.22 m m NaCl, 0.33 m m CaCl2 2 H2O, 0.33 m m MgSO4 7 H2O H2o.에에서 용 해 하 여 배아 매체 (E3)를 준비
- 수정 (0 dpf) 후 즉시 zebrafish 태아를 수집 합니다.
- Zebrafish 태아 90 mm 페 트리 접시 당 50 분할. 착 색을 억제 하는 실험의 기간에 대 한 개발 (0 dpf)의 첫번째 날의 끝에서 배아 매체 (E3) 200 µ M 1-페 닐 2-thiourea (PTU), 치료의 50 mL에 28.5 ° C에서 배아를 올립니다.
- 변화는 E3 + 실험 기간 동안 매일 통해 매체.
- Mpeg1:eGFP 및 NBT:DsRed 애벌레 2 dpf 긍정적인 transgene 식, GFP+ 세포/microglia 및 DsRed+ 뉴런에 대 한 형광 stereomicroscope를 사용 하 여에서 화면.
- 모든 미디어, 오염을 피하기 위하여 조직 문화 후드 실험 전날 준비 다음 4 ° c.에서 그들을 저장합니다
- 15 mM Hepes, HBSS 1에서에서 25 m m D-포도 당을 용 해 하 여 준비 하는 미디어 A x.
- HBSS 10의 1 볼륨에 밀도 그라데이션 매체의 9 볼륨을 혼합 하 여 밀도 그라데이션 매체 (100%)를 준비 x.
- 밀도 그라데이션 매체 (22%)을 준비 하는 DPBS 1의 88 ml에서 밀도 그라데이션 매체 (100%)의 22 mL를 diluting 하 여 x.
- 준비 하는 DPBS 1 x.
- E3 매체 + Tricaine 450 μ M으로 E3 매체를 혼합 하 여 준비 Tricaine.
2입니다. 균질
참고: 모든 단계는 수행된 4 ° c.
- 90 mm 페 트리 요리 E3 배아 매체 200 µ M 말기 그들을 anesthetize를 통해 치료의 50 ml 50 애벌레를 1.5 mL Tricaine (15 m m)를 추가 합니다.
- 3 mL 파스퇴르 대량 플라스틱 피 펫과 페 트리 디쉬에서 10 마 취 애벌레 빨 아.
- 취 전송 애벌레 55 mm 페 트리 접시에 10 10 얼음 E3 배아 매체 + Tricaine로 가득합니다.
- Stereomicroscope, 아래 10 애벌레 페 트리 접시의 중앙에 맞춥니다. 다음 노 른 자-sac 수술 마이크로-가 위 (수영 방광을 피하기 위해 머리를 부동 제외)를 사용 하 여 위에 애벌레 머리 transect.
- 3 mL 파스퇴르 대량 플라스틱 피 펫과 페 트리 디쉬에서 빨 아. 모든 머리 피 펫 팁 내에서 수집 하 고 다음 1 mL 차가운 미디어 A를 포함 하는 유리 균질 화기로 전송 될 때까지 기다립니다 (전송 미디어를 줄이기 위해 최소 볼륨에서 희석 e 3 + Tricaine). 얼음에 유리 균질 화기를 유지. 실험 조건 당 한 균질 화기를 사용 합니다.
- 각 작은 페 트리 접시를 포함 하는 얼음 교체 차가운 E3 + Tricaine 새 것으로 그 transection 보장 하기 위해서 모든 30 분 차가운 E3 + Tricaine에서 수행 하는 매체.
- 색상 페이딩 시작 유리 균질 화기에 얼음 처럼 차가운 미디어 A를 교체 합니다.
참고: 미디어 A 희석 온도 변화 머리 조직을 변경할 수 있습니다. - 모든 머리 수집 된 일단 (600 머리/조건), 유리 균질 화기에서 미디어의 최대 볼륨을 제거 하 고 신선한 얼음 처럼 차가운 미디어 a 1 mL와 함께 그것을 대체합니다
- 얼음 꽉 유리 균질 화기와 뇌 조직 방해. 3-5 dpf 애벌레 및 7 및 8 dpf 애벌레에 대 한 50의 분쇄 40 라운드를 수행 합니다.
- 세포 현 탁 액에 미디어 A의 2 개 mL를 추가 (1 mL 미디어 A 200 / 머리), 세포를 희석 하 고 수 초 밀도 그라데이션 매체에서 원심 분리 하는 동안 그들의 분리를 촉진 하기 위하여와 그들의 덩어리를 줄이기 위해.
- 세포 덩어리를 제거 하려면 얼음에 유지 찬 50 mL 팔 콘 튜브 위에 40 µ m 셀 스 트레이너를 통해 세포 현 탁 액을 실행 합니다. 3 번이이 작업을 반복 합니다.
- 차가운 1.5 mL 튜브에 세포 현 탁 액 1 mL를 전송 및 4 ° c.에서 10 분 300 g에서 그들을 회전합니다
- 10 mL 주사기를 사용 하 여 상쾌한 제거 + 바늘 23G x 1'.
- 다시 1 mL의 얼음 처럼 차가운 22% 밀도 그라데이션 부드럽게 차가운 DPBS 1의 0.5 mL에 의해 중첩 된 셀 펠 릿을 일시 중단 (그들, 두 솔루션 사이의 interphase 나타납니다 혼합 하지 마십시오) x.
- 4 ° c.에 30 분 동안 느린 가속 및 브레이크 없이 950 g에서 튜브를 회전
참고: 이 단계는 DPBS 1의 interphase에 격리 하 여 다른 셀에서 myelin를 분리 x와 22% 밀도 그라데이션 매체, 반면 셀 튜브의 하단에 작은 것입니다. 수 초 제거 세포 농도가 너무 높은 경우 더 효율적입니다. - 그들의 interphase 10 mL 주사기 + 바늘 23G x 1' 삭제 DPBS, 밀도 그라데이션 매체 및 myelin의 최대 갇혀 사용 하.
- 미디어의 0.5 mL + 2% 정상 염소 혈 청 (NGS)의 세포를 씻어 다음 4 ° c.에서 10 분 300 g에서 튜브를 회전
- 상쾌한, 최대 삭제 다음 미디어 A + 2%의 동일한 실험 조건 함께 1 mL에서에서 모든 셀 펠 릿 풀 NGS.
- 4 ° c.에 30 분 동안 느린 가속 및 브레이크 없이 950 g에서 튜브를 회전
- 관심사의 세포 대 식 세포/microglia mpeg1:eGFP에서 처럼 형광 단백질 또는 NBT:DsRed 유전자 변형 물고기 (유전자 변형 물고기 1.1.3의 심사), 35 µ m 셀 스 트레이너를 통해 세포 현 탁 액을 실행에서 뉴런 뚜껑과에 그들을 전송 하는 경우는 차가운 5 mL FACS 빛 으로부터 보호과 얼음, 튜브.
참고: Microglia의 면역 얼룩을 또는, 수행할 수 있습니다.
3. Microglia Immuno 얼룩이
참고: 4 ° c.에 모든 단계를 수행
- 다시 미디어 A + 2%의 0.3 mL와 함께 셀 펠 릿을 일시 중단 NGS. 3 x 1.5 mL 튜브에 그들을 분할: 흠 없는 세포 비 특정 바인딩 microglia 및 세 번째 시험으로 이차 항 체의 측정을 이차 항 체 (1/200)에 대 한 관심의 셀에서 자동 형광 두 번째 측정 한 (4C 4 마우스 단일 클로 널 (해당 microglia)는 항 체 (1/20) + 2 차 항 체 (1/200)).
- 낮은, 아 지 드 무료 (잎) 세포 (모든 튜브)를 1%에서 면역 글로불린의 Fc 도메인 CD16/CD32 상호 작용을 차단 하를 추가 합니다. 부드러운 동요 매 5 분으로 10 분에 대 한 셀을 품 어.
- 4C 4 추가 항 체 (1/20) 셀 (관 3)와 품 어 부드러운 교 반으로 30 분 마다 10 분.
- 4 ° C에서 10 분 300 g에서 튜브를 회전 다음 삭제는 상쾌한.
- 일단 미디어 A + 2 %0.5 mL와 NGS, 다음 4 ° c.에서 10 분 300 g에서 튜브 회전 세척
- 미디어 A + 2%의 0.5 ml와 함께 셀 펠 릿 다시 중단 NGS와 부드러운 동요 매 5 분으로 10 분에 대 한 1%에서 잎 세포를 품 어.
- 셀 (튜브 2와 3)를 이차 항 체 (1/200)를 추가 합니다. 매 10 분 및 가벼운 보호 부드러운 동요와 30 분에 대 한 셀을 품 어.
- 4 ° C에서 10 분 300 g에서 튜브를 회전 한 다음 상쾌한 삭제.
- 미디어 A + 2%의 1 mL와 함께 미디어 A + 2%, NGS 다음 다시 일시 중지 셀 펠 릿의 0.5 mL로 두 번 세척 NGS.
- 35 µ m 셀 스 트레이너 모자를 통해 세포 현 탁 액을 실행 하 고 빛 으로부터 보호 하는 얼음에 차가운 5 mL FACS 튜브로 그들을 전송.
4입니다. 셀 정렬 (FACS)
참고: 4 ° c.에 모든 단계를 수행
- 신경 세포, 대 식 세포/microglia 및 microglia를 FACS를 사용 하 여 정렬 합니다.
참고: 이 단계는 일반적으로 FACS 시설의 직원에 의해 수행 하 고 설정을 사용 하는 장비의 유형에 따라 다릅니다.- 죽은 세포를 각 FACS 튜브에 1 µ g/mL의 농도에서 DAPI를 추가 합니다.
- 모든 뇌 세포에서 FACS 및 정렬 신경 세포, 대 식 세포/microglia 또는 microglia를 설정 합니다. 그들의 크기, 단위, 그리고 앞으로 분산형 및 사이드 분산형 게이트 단일 세포의 기능에 있는 파편에서 별도 셀. DAPI에서 라이브 셀 라벨에 의해 죽은 세포를 제외 합니다. 그들의 각각 긍정적인 얼룩에 의해 신경 세포, 대 식 세포/microglia 또는 microglia를 식별 합니다.
- 얼음 처럼 차가운 미디어 A + 2%의 1 mL를 포함 하는 1.5 mL 튜브에 세포를 수집 NGS 얼음에. 다른 튜브를 사용 하 여 각 세포 유형에 대 한.
- 4 ° C에서 10 분 300 g에서 튜브를 회전 하 고는 상쾌한 다음 삭제.
- 미디어의 0.5 mL로 한 다음 삭제 상쾌한 최대 한 번 씻는 다.
5. RNA 추출
- FACS를 구분 하는 다른 세포 유형에서 RNA를 추출 합니다. RNA 추출에 대 한 특정 키트를 사용 하 고 제조업체의 지침을 따릅니다. 청소 작업 영역 및 RNase 오염 제품 펫 RNase 무료 환경에서 작동 하는 필터 팁을 사용 하 여 확인 합니다.
- 갓 50 µ M β-mercaptoethanol 보충 하는 세포의 용 해 버퍼의 1 mL를 준비 합니다.
참고: 여기서, β-mercaptoethanol RNA 저하를 줄이기 위해 추가 됩니다. - 무료 물 70%와 80% 에탄올 솔루션 RNase를 사용 하 여 준비 합니다.
- 세포 세포의 용 해 버퍼 50 µ M β-mercaptoethanol 보충의 75 µ L lyse 다음 세포 파쇄를 분쇄기를 사용.
- 제조 업체에 따라 RNA 추출의 매뉴얼을 계속 한다.
- 프로토콜의 끝에, 충분 한 RNA 농도를 RNase 무료 물 14 µ L로 RNA elute.
- 갓 50 µ M β-mercaptoethanol 보충 하는 세포의 용 해 버퍼의 1 mL를 준비 합니다.
결과
설명된 프로토콜은 신경 세포, 대 식 세포 및에서 zebrafish 애벌레 두뇌 microglia 분리 간단 접근 이다. 이들에서 고립 세포, 높은 품질의 상당수 (린 > 7) RNA 추출 되었다. 이 프로토콜의 목표 분석 및 셀 속성 및 기능 특성에 그들의 유전자 식 프로 파일의 최소 수정 CNS에서 다른 종류의 세포를 분리 하는. 따라서, 전체 프로토콜 기계적 뇌 조직 균질 화와 4 ° C에서 수행 됩니다. 이 방법은 실험실에서 수행 하는 두 개의 연구 성공적으로 사용 되었습니다. 첫 번째 연구에서 신경 세포와 대 식 세포/microglia 8 dpf mpeg1:eGFP+/NBT:DsRed+ 애벌레 (그림 1)에서 분리 했다. 그들의 크기의 기능에서 파편에서 셀 분리를 허용 하는 FACS (FSC-A) 및 세분성 (SSC-A) (그림 1A). 단일 셀 다음 남자 또는 세포 응집 체 (그림 1B)에서 분리 되었다. 단일 세포 인구에서 죽은 세포 (DAPI+)를 제거 하는 게이트 그려 했다. 해당 점 플롯으로 죽은 세포의 비율은 26.7% (그림 1C)만이 실험 프로토콜 세포 원형질 막 무결성을 보존 공개 했다. 마지막으로, 신경 (DsRed+)와 대 식 세포/microglia (GFP+) 쉽게 라이브 셀 인구 게이츠에서 차별 했다. 신경 인구 (23.1%) (그림 1D) 뇌 내 세포/microglia 인구 (1.56%) 보다 더 두드러진 것으로 나타났다. 이 프로토콜을 신경 세포와 대 식 세포/microglia 사이 특정 유전자의 표정을 비교할 후속 정량 분석을 수행 하려면 그 세포에서 RNA를 분리를 허용 했다. 그림 2 쇼 신경 고 증식 세포 핵 항 원 (pcna)예를 들어 β-걸 집 지키는 유전자에 대 한의 대 식 세포/microglia 유전자 식 수준.
두 번째 연구에 대 한이 방법은 3, 5, 7 dpf 애벌레 두뇌 microglia 절연에 집중 했다. 위에서 설명한 실험과 달리 세포 면역 염색에 의해 고립 되었다 4C 4, 특히 라벨 microglia (그림 3 A D)는 항 체를 사용 하 여. 앞에서 설명한, microglia (4C 4+) 라이브 셀에서 선정 됐다 고 (그림 3D) 수집. Zebrafish 애벌레 두뇌 내의 Microglia 번호는 변수 (표 1), 3 dpf (물고기 당 25 ∼)에서 매우 낮은. 그 세포에서 추출 된 RNA의 양과 품질 마이크로 모 세관 전기 이동 법 기반 시스템을 사용 하 여 측정 되었다. 결과 5 dpf 애벌레 zebrafish 두뇌 microglia에서 추출 된 RNA의 RNA 분석 (표 1 (5 dpf; 실험 4))의 예를 보여 주기 위해 제공 되었습니다. 그림 4 는 전기 추적 및 리보솜 RNA (28s와 18s)의 명확한 시각화로이 샘플에 대 한 얻은 그것의 그래픽 표현. 이 데이터는 샘플 린 계산 하 고 RNA 농도 결정 하는 데 필요한. 표 1 절연된 microglia 생선과 microglia 당 RNA의 양을 각각 다른 실험 3, 5, 7 dpf에 대해 린 점수 당 수를 요약 합니다. 이 메서드를 사용 하 여 격리 microglia에서 추출 된 RNA의 품질과 양을 키트를 사용 하 여 cDNA로 RNA를 증폭 수 있었습니다. 에 딘 버 러 유전체학에서 제공 하는 품질 및 수량 테스트 증폭된 cDNA 라이브러리 준비 및 후속 시퀀싱에 대 한 충분 한 품질의 확인. 그림 5 는 cDNA 조각 및 전기 이동 시스템을 사용 하 여 측정 하는 그들의 금액의 크기 분포를 보여준다. 이 샘플에는 cDNA 36100의 농도에 299bp의 평균 크기를 했다 pmol/l. 표 2 는 각각 RNA 샘플 (표 1 (5 dpf; 실험 4))에서 증폭 된 cDNA에서 품질과 수량 테스트를 보여 줍니다. 증폭된 cDNA는 시퀀싱을 위해 성공적으로 사용 되었습니다.
실험실에서 수행 하는 여러 연구 확인 품질 신경 세포, 대 식 세포 및 microglia에서 추출 된 RNA의 수량 및 후속 정량 및 게놈 넓은 진 식 분석에 사용할 수 있습니다. 따라서,이 실험 프로토콜 안정적으로 막 무결성을 변경 하 고 그들의 유전자 식 프로 파일의 수정에 제한 하지 않고 CNS 세포의 다른 종류를 분리 사용할 수 있습니다.

그림 1 : 신경 세포와 대 식 세포/microglia mpeg1:GFP+/NBT:DsRed+ 에서 8 dpf zebrafish 애벌레 정렬 FACS. (A-C) 연속 게이팅의 모든 뇌 세포 () 순차 선택, 앞으로 분산형과 측면에 의해 단일 셀 분산형 (B). (C) 죽은 세포 DAPI 라벨에 의해 제외 되었습니다. (D) 신경 세포 및 대 식 세포/microglia 각각 DsRed와 GFP 긍정적인 얼룩에 의해 확인 되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
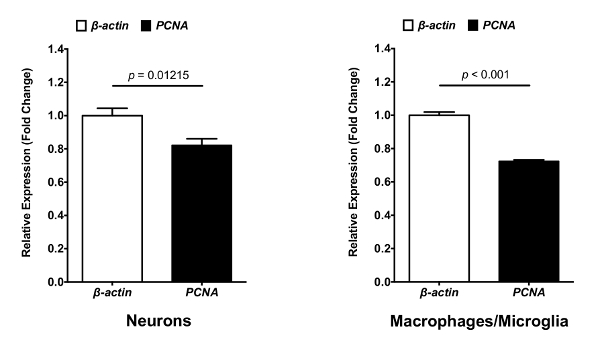
그림 2 : 유전자 표정 분석 pcna 및 신경 세포와 대 식 세포/microglia β-말라 . 격리 된 신경 세포와 대 식 세포/microglia RNA 정량 PCR 분석에 사용 하기 위해 cDNA에는 복사할 수 수 있습니다. 격리 된 신경 세포와 대 식 세포/microglia 정량에 의해 결정에서 β-걸 집 지키는 유전자에 대 한 pcna 의 mRNA 식 레벨 (N = 3). 배 변화 비교 (ΔΔCT) 메서드를 사용 하 여 측정 했다. 오차 막대 대표 평균 ± SEM. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3 : FACS microglia 3 dpf zebrafish 애벌레에서에 대 한 정렬. (A-C) 연속 제어 순차 선택 모든 뇌의 세포 (A), 앞으로 분산형과 측면에 의해 단일 셀 분산형 (B). (C) 죽은 세포 DAPI 라벨에 의해 제외 되었습니다. (D) Microglia 4C 4에 의해 확인 되었다 긍정적인 얼룩이 지기. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 4 : 5 dpf zebrafish microglia에서 추출 된 RNA의 마이크로 모 세관 전기 이동 법 결과. 두 개의 큰 봉우리는 18와 28S ribosomal RNA. RNA 무결성 번호 (린)은 28S ribosomal 소 단위는 18의 생성된 비율 및 전체 전기 이동 추적의 분석을 사용 하 여 bioanalyzer 소프트웨어에 의해 자동으로 계산 됩니다. Microglia RNA 린 8.6 있다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 5 : 5 dpf zebrafish microglia의 RNA 샘플에서 증폭 된 cDNA의 품질과 수량 테스트. 화면은 299의 평균 크기를 가진 cDNA 조각 크기 분포 분석된 샘플의 bp. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| 조건 | 실험 | 물고기 수 | 휴대폰 번호 | 물고기 당 휴대폰 번호 | RNA 농도 (pg/ul) | 총 RNA (pg) | 셀 (pg) 당 RNA 금액 | 린 점수 |
| 3dpf | 1 | 700 | 11922 | 17.03 | 126 | 1512 | 0.13 | 8 |
| 3dpf | 2 | 600 | 22527 | 37.55 | 253 | 3036 | 0.13 | 7.9 |
| 3dpf | 3 | 600 | 18688 | 31.15 | 255 | 3060 | 0.16 | 7.7 |
| 3dpf | 4 | 600 | 11121 | 18.54 | 189 | 2268 | 0.20 | 7.8 |
| 3dpf | 5 | 600 | 15581 | 25.97 | 131 | 1572 | 0.10 | 8.4 |
| 3dpf | 6 | 600 | 11965 | 19.94 | 256 | 3072 | 0.26 | 8.2 |
| 5dpf | 1 | 600 | 58629 | 97.72 | 362 | 4344 | 0.07 | 7.4 |
| 5dpf | 2 | 600 | 32510 | 54.18 | 348 | 4176 | 0.13 | 8.1 |
| 5dpf | 3 | 600 | 77884 | 129.81 | 594 | 7128 | 0.09 | 8.3 |
| 5dpf | 4 | 600 | 50755 | 84.59 | 305 | 3660 | 0.07 | 8.6 |
| 5dpf | 5 | 600 | 44967 | 74.95 | 134 | 1608 | 0.04 | 7.6 |
| 5dpf | 6 | 600 | 51031 | 85.05 | 163 | 1956 | 0.04 | 7.9 |
| 7dpf | 1 | 600 | 60496 | 100.83 | 183 | 2196 | 0.04 | 7.6 |
| 7dpf | 2 | 450 | 55517 | 123.37 | 183 | 2196 | 0.04 | 7.8 |
| 7dpf | 3 | 600 | 88897 | 148.16 | 465 | 5580 | 0.06 | 8.1 |
| 7dpf | 4 | 600 | 63008 | 105.01 | 356 | 4272 | 0.07 | 8.4 |
| 7dpf | 5 | 350 | 34956 | 99.87 | 245 | 2940 | 0.08 | 8.1 |
| 7dpf | 6 | 600 | 63887 | 106.48 | 341 | 4092 | 0.06 | 7.8 |
표 1: microglia 격리 및 3, 5, 7 dpf zebrafish 애벌레에서 RNA 추출 데이터의 요약.
| 내부 샘플 ID | 외부 샘플 ID | 큐 비트 (ng/ul) | Qubit(ng/ul) | 평균 농도 (ng/ul) | 볼륨 (ul) | ug 받은 | 최소 농도 대 한 합격/불합격 | Minimim 수량에 대 한 합격/불합격 | 권장된 수량에 대 한 합격/불합격 |
| 10907SD0010 | 5 dpf (실험 4) | 58.8 | 58.4 | 58.6 | 30 | 1.76 | 패스 | 패스 | 패스 |
| 샘플 라이브러리 준비에 대 한 요구 사항: | |||||||||
| 라이브러리 준비 | 최소 수량 (ng) | 권장된 수량 (ng) | 최소 농도 ng/uL | ||||||
| TruSeq 나노 젤 cDNA에서 무료 350 bp 삽입 라이브러리 | 600 | 1100 | 10 | ||||||
표 2: 5의 dpf zebrafish microglia의 RNA 샘플에서 증폭 된 cDNA의 수량 테스트 합니다.
토론
여기에 설명 된 실험 프로토콜 8 dpf에 3에서 zebrafish 애벌레에서 뇌 세포를 분리 하는 강력 하 고 효율적인 방법을 나타냅니다. 중요 한 것은, 이것은 첫 번째 프로토콜에서 애벌레 zebrafish 두뇌 microglia의 특정 격리를 허용입니다. 프로토콜은 세포 막의 무결성을 유지 하 고 처리 하는 동안 발생 하는 유전자 발현의 잠재적인 수정 최소화 하기 위해 설계 되었습니다. 이 마지막 점은 그 고립 된 세포 게놈 프로 파일의 분석에 따라 결과의 관련성에 대 한 중요 한 있습니다. 실제로, microglia 및 macrophage 분극 강하게 그들의 microenvironment에 의해 영향을 받습니다. 37 ° C에서이 세포 실험 조건 (부상 (transection) 응답)에 자신의 유전자 식 프로필을 변경 것입니다. 따라서, 그것은 이전 세포 프로세스 및 신진 대사 활동을 천천히 RNA 추출, 4 ° C에서이 실험을 수행 하는 중요 한 했다. 또한, 기계적 뇌 조직 균질 4 ° C에서 37 ° C에서 효소 조직 소화 대신 유전자 식 프로필에 어떤 영향을 피하기 위해 선정 되었다.
이 메서드는 매우 빠른; 강조 하는 것이 중요 하다 하루 내 여러 뇌 세포 인구는 적어도 두 개의 다른 실험 조건 및 그들의 RNA 추출에서 격리 될 수 있습니다. 프로토콜의 총 길이 애벌레 머리의 transection 제한 단계 (∼ 350 머리/h)으로 각 조건에 대해 사용 하는 애벌레의 수에 따라 달라 집니다. 일반적으로 3에서 microglia를 작업할 5 및 7 dpf이 좋습니다 transect ∼ 600 머리 (표 1)를 추출 하는 충분 한 RNA를 시험 당 하. Microglia 셀 형식을 나타내는 낮은 산출 (7 dpf에 인당 약 112 세포), 대 식 세포 (7 dpf에 인당 약 170 세포)를 포함 하 여 다른 세포 유형 머리의 수를 줄일 수 있습니다.
이 프로토콜의 또 다른 장점은 FACS 정렬 설정이 설정 된 후 동일한 설정을 사용할 수 있는 다양 한 실험에 대 한. 그것은 세포 인구 맞는 완벽 하 게 한 실험에서 다른 이전 설계, 게이츠와 함께이 메서드를 사용 하 여 실험의 재현성을 보여주는 관찰 되었습니다.
이 방법의 약간의 불리는 수확 하는 RNA의 상대적으로 낮은 금액입니다. 그러나,이 제한은 microglia 수로 두뇌 개발 (표 1)의 초기 단계에서 매우 낮은 기술적인 문제 보다는 더 많은 생물 학적 문제입니다. 수집 하는 RNA의이 낮은 양 때문에 증폭 단계 게놈 넓은 유전자 표정 분석을 수행 하기 위해 고려 될 필요가 있다. 다행히도, 이러한 증폭 단계 고품질 cDNA의 충분 한 양을 생산. 따라서, 고립 된 세포의 유전자 식 프로필에 글로벌 변화는 공부 될 수 있다.
결론적으로,이 프로토콜에는 격리 하 고 애벌레 zebrafish 두뇌에서 다양 한 CNS 셀 유형을 공부 강력한 방법을 제공 한다. 이것은 또한 질병에 그들의 역할을 연구로 개발 하는 동안이 세포의 깊은 이해를 얻기 위해 적용할 수 있습니다.
공개
저자는 공개 없다.
감사의 말
우리 초기 도움말 및 실험적인 접근 및 QMRI Flow Cytometry 셀 정렬 시설에 대 한 토론에 대 한 박사 클레어 데이비스와 닥터 베로 Miron (여왕의 의료 연구소, 에든버러, 유나이티드 Kingdome) 감사합니다. 이 작품은 박사 더크 Sieger에 암 연구 영국 경력 설립 수상에 의해 지원 되었다.
자료
| Name | Company | Catalog Number | Comments |
| 1-phenyl 2-thiourea (PTU) | Sigma | P7629 | |
| Hepes | Gibco | 15630-056 | |
| D-Glucose | Sigma | G8644-100ML | |
| HBSS 1X | Gibco | 14170-088 | |
| Percoll | GE Healthcare | 17-0891-02 | |
| HBSS 10X | Gibco | 14180-046 | |
| DPBS 1X | Gibco | 14190-094 | |
| Tricaine (MS222) | Sigma | A5040 | |
| Sterilin standard 90mm petri dishes | ThermoFisher | 101VIRR | |
| Surgical micro-scissors | Fine Science Tools | 15000-00 | |
| 3 mL Pasteur plastic bulk pipette | SLS | PIP4206 | |
| Glass homogenizer | Wheaton | 357538 | |
| Sterilin standard 55mm petri dishes | ThermoFisher | P55V | |
| Percoll | GE Healthcare | 17-0891-02 | |
| 40 µm cell strainer | Falcon | 352340 | |
| 50 ml polypropylen conical tube | Falcon | 352070 | |
| Centrifuge | Eppendorf | 5804 R | |
| 10 mL syringe | BD | 302188 | |
| Needle 23G x 1'' | BD | 300800 | |
| Normal goat serum (NGS) | Cell Signalling | 5425S | |
| 35 μm cell strainer cap | BD | 352235 | |
| FACS tubes | BD | 352063 | |
| Low Endotoxin, Azide-Free (LEAF) | Biolegend | 101321 | |
| Alexa Fluor 647 Goat Anti-Mouse IgG (H+L) | Life Technologies | A11008 | |
| Anti-4C4 | Courtesy of Catherina Becker (University of Edinburgh) | ||
| FACS sorter FACSAria II | BD | QMRI, FACS facility | |
| RNeasy Plus Micro Kit | QIAGEN | 74034 | |
| β-mercaptoethanol | Gibco | 31350-010 | |
| QIAshredder | QIAGEN | 79654 | |
| SsoAdvanced Universal SYBR Green Supermix | Bio-Rad | 1725271 | |
| SuperScript III First-Strand Synthesis System | Invitrogen | 18080-051 | |
| LightCycler 96 Real-Time PCR System | Roche | ||
| Ovation RNA-Seq System V2 | NuGEN | 7102-32 | |
| Agilent RNA 6000 Pico reagents | Agilent | 5067-1513 | |
| 2100 Bioanalyzer | Agilent | ||
| RNaseZap | Ambion | AM9780 |
참고문헌
- Chrast, R., Scott, H. S., et al. The Mouse Brain Transcriptome by SAGE: Differences in Gene Expression between P30 Brains of the Partial Trisomy 16 Mouse Model of Down Syndrome (Ts65Dn) and Normals. Genome Research. 10 (12), 2006-2021 (2000).
- Beis, D., Stainier, D. Y. R. In vivo cell biology: following the zebrafish trend. Trends in Cell Biology. 16 (2), 105-112 (2006).
- Shiau, C. E., Kaufman, Z., Meireles, A. M., Talbot, W. S. Differential Requirement for irf8 in Formation of Embryonic and Adult Macrophages in Zebrafish. PLoS ONE. 10 (1), 0117513-0117515 (2015).
- Mazaheri, F., Breus, O., et al. Distinct roles for BAI1 and TIM-4 in the engulfment of dying neurons by microglia. Nature Communications. 5, 1-11 (2014).
- Oosterhof, N., Holtman, I. R., et al. Identification of a conserved and acute neurodegeneration-specific microglial transcriptome in the zebrafish. Glia. 65 (1), 138-149 (2016).
- Hamilton, L., et al. A Zebrafish Live Imaging Model Reveals Differential Responses of Microglia Toward Glioblastoma Cells In Vivo. Zebrafish. , (2016).
- Ellett, F., Pase, L., Hayman, J. W., Andrianopoulos, A., Lieschke, G. J. mpeg1 promoter transgenes direct macrophage-lineage expression in zebrafish. Blood. 117 (4), 49-56 (2011).
- Peri, F., Nüsslein-Volhard, C. Live Imaging of Neuronal Degradation by Microglia Reveals a Role for v0-ATPase a1 in Phagosomal Fusion In Vivo. Cell. 133 (5), 916-927 (2008).
- Sieger, D., Moritz, C., Ziegenhals, T., Prykhozhij, S., Peri, F. Long-range Ca2+ waves transmit brain-damage signals to microglia. Developmental cell. 22 (6), 1138-1148 (2012).
- Becker, T., Becker, C. G. Regenerating descending axons preferentially reroute to the gray matter in the presence of a general macrophage/microglial reaction caudal to a spinal transection in adult zebrafish. The Journal of comparative neurology. 433 (1), 131-147 (2001).
- Ohnmacht, J., Yang, Y., et al. Spinal motor neurons are regenerated after mechanical lesion and genetic ablation in larval zebrafish. Development. 143 (9), Cambridge, England. 1464-1474 (2016).
- Roy-Carson, S., Natukunda, K., et al. Defining the transcriptomic landscape of the developing enteric nervous system and its cellular environment. BMC Genomics. 18 (1), 1-24 (2017).
- Khuansuwan, S., Gamse, J. T. Identification of differentially expressed genes during development of the zebrafish pineal complex using RNA sequencing. Developmental biology. 395 (1), 144-153 (2014).
- Schmid, C. D., Melchior, B., et al. Differential gene expression in LPS/IFNγ activated microglia and macrophages: in vitroversus in vivo. Journal of Neurochemistry. 109, 117-125 (2009).
- Bohlen, C. J., Bennett, F. C., Tucker, A. F., Collins, H. Y., Mulinyawe, S. B., Barres, B. A. Diverse Requirements for Microglial Survival, Specification, and Function Revealed by Defined- Medium Cultures. Neuron. 94 (4), 759-773 (2017).
- Khan, A., Ju, F., et al. Transcriptomic analysis reveals differential activation of microglial genes after ischemic stroke in mice. Neuroscience. 348, 212-227 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유