Method Article
分離およびゼブラフィッシュ稚魚の脳から神経細胞、大食細胞、ミクログリアの RNA の抽出
要約
ニューロン、マクロファージやミクログリア ゼブラフィッシュ稚魚の脳生理学的および病理学的条件の下でからを分離するためのプロトコルを提案します。分離時にこれらの細胞の遺伝子発現プロファイルを分析するから RNA を抽出します。このプロトコルは、qPCR とトランスクリプトミクスのような下流の分析を実行するため高品質の RNA のコレクションをできます。
要約
開発または設立時に別の中枢神経系の細胞の役割の解明と脳の病態の進行を得るためには、彼らの遺伝子発現プロファイルを変更することがなくこれらのセルを隔離することが重要です。ゼブラフィッシュ モデル提供多数の遺伝子導入魚ラインに特定の細胞型が分類される;たとえば NBT:DsRed ラインや mpeg1:eGFP ラインにおけるマクロファージ/ミクログリアのニューロン。さらに、抗体は、4 4 ミクログリアなどの特定の細胞を染色する開発されている抗体です。
ここでは、神経細胞、マクロファージやミクログリア ゼブラフィッシュ稚魚の脳からの分離について述べる。このプロトコルの中心は、37 ° c では、携帯電話のプロファイルを変更可能性があります組織の酵素消化の回避です。代わりに 4 ° C で組織の均質化の機械システムが使用されます。このプロトコルは、脳の細胞懸濁液に均質化、彼らの免疫染色と細胞、マクロファージ、FACS によるミクログリアの分離を伴います。その後、それらの細胞から RNA を抽出し、その質・量を評価しました。高品質の RNA を取得管理して (RNA 整合性数 (鈴) > 7) ミクログリアのマクロファージ/ミクログリアとニューロン ・ トランスクリプトーム解析の qPCR を実行します。このアプローチにより、開発と病態におけるその役割についての明確な理解と同様、これらの細胞のより良い評価です。
概要
脳の発達と脳の病気に関する知識は、マウス脳トランスクリプトーム1の最初の定量化から過去 10 年間で大幅に改善します。確かに、ゲノム広い遺伝子発現解析私たちは、脳組織と細胞を補完でき、他の手法やツールを使った観測を向上させる詳細な遺伝情報。
ゼブラフィッシュは強力な生物学的モデルで、簡単に繁殖と遺伝的に変更するのには幼虫の段階で、光の透過性は、ライブ イメージング観測2をことができます。残念ながら、比較する人間、マウス、入手可能な抗体免疫染色を実行する数はかなり低い。この問題を解決するには、トランスジェニックゼブラフィッシュ魚行は簡単に携帯型特定プロモーターの下で蛍光蛋白質を表現する魚に遺伝子改変によって行われます。トランスジェニックゼブラフィッシュ ラインは、中枢神経系 (CNS) の開発と疾患3,4,5,6時にマクロファージやミクログリアの役割を研究する過去に使用されています。しかし、これらのプロセスの詳細な理解を得るためには、各種細胞の遺伝子発現の変化を理解する必要があります。この目的に特にのように神経細胞、マクロファージ、3 から 8 日にミクログリア細胞を分離する実験手法を開発した後受精 (dpf) ゼブラフィッシュ稚魚脳。プロトコルの確立のため我々 は神経 β-チューブリンの下で神経細胞のマクロファージ表現遺伝子プロモーター (mpeg1:eGFP) と下流の下マクロファージ/ミクログリアで緑色蛍光タンパク質 (GFP) を表す遺伝子導入魚ラインで働いてください。プロモーター (NBT:DsRed)7,8,9。さらに、ミクログリア 4 4、特に汚れのゼブラフィッシュ ミクログリア10,11マウスのモノクローナル抗体を用いた免疫染色を行った.その後、リボ核酸 (RNA) はさらに量的なポリメラーゼの連鎖反応 (qPCR) またはトランスクリプトーム解析のためのこれらの細胞から抽出されます。このプロトコルは効率的にゼブラフィッシュ幼虫; から脳組織を均一に設計されています神経細胞、マクロファージ/ミクログリアとミクログリア細胞膜完全性の変化なしを収集し、最終的に高品質のこれらの細胞から RNA を抽出 (凛 > 7) とゲノム解析を実行する数量。脳組織12,13を消化する 37 ° C でトリプシン処理を使用して、以前に発行された研究とは異なりは、このプロトコルは、遺伝子発現プロファイルの変更を抑える RNA 抽出工程まで 4 ° c の作業を推進しています。このステップがミクログリアとして重要とマクロファージが非常に敏感な細胞の遺伝子発現プロファイルと偏波14,15,を変えることによってすぐに彼らの微小環境の変化に対応します。16。
詳しくは、ここで説明したプロトコル神経細胞、マクロファージやミクログリア ゼブラフィッシュ仔の脳が、事実上、それからの分離は、他のセルに合わせることができるショー脳内 - トランスジェニック魚行を使用して、いずれかを提示または付けられて特定の抗体。このメソッドは、中枢神経系細胞のゲノム広い遺伝子発現解析を通してのより良い特性を許可し、発展と脳の病気の中に自分の役割を理解するのに役立ちます。
プロトコル
1. サンプルとメディアの準備
- 6.4 mM KCl、0.22 mM の NaCl、0.33 mM CaCl2 2 H2O、0.33 mM MgSO4 7 H2O H2o. を溶解することにより胚メディア (E3) の準備します。
- 受精 (0 dpf) の直後にゼブラフィッシュ胚を収集します。
- 90 mm シャーレあたり 50 にゼブラフィッシュの胚を分割します。開発 (0 dpf) 色素沈着を抑制する実験の期間の最初の日の終わりから胚胚中 (E3) 200 μ M 1-フェニル-2-チオ尿素 (PTU) で治療の 50 ml 28.5 ° c を上げます。
- 変更、E3 + PTU 媒体毎日実験の持続期間のため。
- 画面 mpeg1:eGFP と蛍光顕微鏡を用いた遺伝子発現の肯定的な GFP+マクロファージ/ミクログリアとした DsRed+ニューロン 2 dpf で NBT:DsRed 幼虫。
- すべてのメディアの汚染を避けるためにティッシュ文化フードの下で実験の前に一日を準備し、4 ° C で保存
- 15 mM Hepes と 25 mM グルコース HBSS 1 を溶解することによりメディア A の準備 x。
- 密度勾配 HBSS 10 の 1 ボリューム中の 9 つの容積を混合することによって密度勾配媒体 (100%) を準備 x。
- 密度勾配媒体 (100%) の 22 mL DPBS 1 の 88 mL で希釈することによって密度勾配媒体 (22%) を準備 x。
- 準備 DPBS 1 x。
- 450 μ M と E3 媒体を混合して E3 媒体 + Tricaine を準備 Tricaine。
2. 均質化
注:すべての手順が実行される 4 ° C です。
- 200 μ M 末期それらを麻酔する PTU で治療 E3 胚中の 50 mL の 50 の幼虫を含む 90 mm シャーレに 1.5 mL Tricaine (15 mM) を追加します。
- 3 ml のプラスチック製バルクのパスツール ピペット シャーレから 10 麻酔幼虫を吸います。
- 伝達麻酔幼虫 55 mm のペトリ皿に 10 の 10 は、冷たい E3 胚中 + Tricaine でいっぱい。
- 、顕微鏡下でペトリ皿の中心 10 幼虫に合わせます。その後、トランセクト卵黄嚢の外科マイクロはさみ (フローティング ヘッドを避けるために浮袋を除く) を使用して上記の幼虫の頭。
- 3 ml のプラスチック製バルクのパスツール ピペット シャーレから頭を吸います。すべてのヘッド ピペット チップ内で収集し、1 mL 冷たいメディア A を含んでいるガラス ホモジナイザーに転送まで待つ (メディアを抑える最小限のボリュームで希釈して転送 E3 + Tricaine)。氷の上のガラスのホモジェナイザーを維持します。実験条件ごとの 1 つのホモジナイザーを使用します。
- 氷が入っている各小さいペトリ皿を置き換える冷たい E3 + Tricaine 冷たい E3 + Tricaine で新しいものとその断裂を確保するために 30 分ごとを実行中。
- 色が退色を起動時ガラス ホモジナイザの冷たいメディア A を取り付けます。
注:メディアの希釈は、温度変化による頭部の組織を変更できます。 - (600 頭/条件)、ガラスのホモジェナイザーからメディア A の最大ボリュームを削除し、1 ml の新鮮な冷たいメディア A の交換のすべてのヘッドが収集された後
- 氷の上タイトなガラス ホモジナイザーを用いた脳組織を混乱させます。3-5 dpf 幼虫と 7 と 8 の dpf 幼虫 50 の 40 回のラウンドを粉砕し、回転を実行します。
- 細胞懸濁液にメディア A 2 mL を追加 (1 mL メディア A/200 頭)、それは細胞を希釈し、密度勾配媒体の遠心分離の間に彼らの分離を容易にするミエリンとその凝集を低減します。
- 細胞の集積を避けるためには、氷の上を維持 50 mL の冷たいファイティングファルコン管の上に配置された 40 μ m 携帯こし器を通って細胞懸濁液を実行します。この操作を 3 回繰り返します。
- 冷たい 1.5 mL チューブに細胞懸濁液の 1 mL を移すし、4 ° c. で 10 分の 300 g でそれらをスピン
- 10 mL のシリンジを使用して上清を除去 + 針 23 x 1」。
- 優しく冷たい DPBS 1 の 0.5 mL でオーバーレイ冷たい 22% の密度勾配媒体の 1 mL の細胞ペレットを再停止 (は見られるそれらは、両方のソリューションの間中間期) x。
- スピンで 950 g ブレーキと 4 ° C で 30 分間低速加速なしチューブ
注:この手順は DPBS 1 の相間で貯留することで他のセルからの髄を分離 x と 22% 密度勾配媒体、細胞が管の底にペレットに対し。髄鞘の除去は、細胞濃度が高すぎるではない場合に効率的です。 - 相間で 10 mL のシリンジ + 最大 DPBS、密度勾配媒体および髄鞘が閉じ込められた針 23 × 1 ' 破棄を使用します。
- メディア A 0.5 mL + ヤギ血清 (NGS) の 2% 血球を洗浄し、スピンで 4 ° C で 10 分間 300 g チューブ
- 培養上清の最大値を破棄し、プールのメディア A + 2 %1 mL で一緒に同一の実験条件からすべての細胞ペレット NGS。
- スピンで 950 g ブレーキと 4 ° C で 30 分間低速加速なしチューブ
- 興味のセル表現 mpeg1:eGFP からマクロファージ/ミクログリアのような蛍光蛋白質、または NBT:DsRed 遺伝子導入魚 (遺伝子導入魚を参照してください 1.1.3 のスクリーニング)、35 μ m 携帯こし器を通って細胞懸濁液を実行からニューロン キャップしにそれらを転送します。光から保護されている氷の上の冷たい 5 mL FACS 管。
注:また、ミクログリアの免疫染色を実行できます。
3. ミクログリア免疫染色
注:4 ° C ですべての手順を実行します。
- メディア + 2% の 0.3 ml 細胞ペレットを再停止 NGS。3 × 1.5 mL チューブにそれらを分割: 無染色細胞ミクログリアと第 3 テストの二次抗体の非特異的結合を測定する二次抗体 (1/200) の第二に興味のセルから自動蛍光を測定するための 1 つ (4 4 マウス モノクローナル抗体 (ミクログリア特定) (1/20) + 二次抗体 (1/200))。
- 低エンドトキシン、アジ化無料 (リーフ) 細胞 (すべての管) に 1% で免疫グロブリンの Fc ドメイン CD16/CD32 相互作用をブロックするを追加します。穏やかな攪拌 5 分ごとに 10 分のセルを孵化させなさい。
- 4 4 を追加抗体 (1/20) (管 3) のセルに 10 分毎穏やかな撹拌で 30 分間インキュベートし、。
- 4 ° C で 10 分の 300 g でチューブをスピンし、上澄みを廃棄します。
- メディア A + 2 %0.5 mL と NGS、それからスピン チューブ 4 ° c. で 10 分の 300 g で一度洗う
- メディア + 2% の 0.5 ml の細胞ペレットの再停止 NGS と穏やかな攪拌 5 分ごとに 10 分間の 1% で葉の細胞を孵化させなさい。
- 二次抗体 (1/200) を (2 と 3 の管) のセルに追加します。すべての 10 分と軽い保護は、穏やかな攪拌と 30 分のための細胞を孵化させなさい。
- 4 ° C で 10 分の 300 g でチューブをスピンし、上澄みを廃棄します。
- メディア + 2 %1 mL でメディア A + 2 %ngs、細胞ペレットの再停止の 0.5 mL で 2 回洗浄 NGS。
- 35 μ m の細胞のストレーナー キャップを介して細胞懸濁液を実行し、氷、光から保護に冷たい 5 mL FACS の管にそれらを転送します。
4. セル (FACS) を並べ替え
注:4 ° C ですべての手順を実行します
- 神経細胞、マクロファージ/ミクログリアおよび、FACS を用いたミクログリアを並べ替えます。
注:この手順は通常 FACS 施設のスタッフ メンバーが実行し、使用機器の種類に依存する設定。- 死んだ細胞をラベルする各 FACS 管で 1 μ G/ml の濃度の DAPI を追加します。
- すべての脳細胞の FACS と並べ替えの神経細胞、マクロファージ/ミクログリアやミクログリアを設定します。破片のサイズと細かさで、関数の中から細胞を分離し、前方散乱、側方散乱による単一細胞のゲートします。DAPI は生きた細胞からラベリングによって死んだ細胞を除外します。彼らのそれぞれ肯定的な染色による神経細胞やマクロファージ/ミクログリア ミクログリアを識別します。
- 冷たいメディア + 2 %1 mL を含む 1.5 mL チューブに細胞を収集 NGS 氷の上。セル タイプごとに別のチューブを使用します。
- スピンで 4 ° C で 10 分間 300 g チューブと、上澄みを廃棄します。
- メディアの 0.5 mL とし、破棄上清の最大一度洗ってください。
5. RNA の抽出
- FACS で区切られた異なった細胞のタイプから RNA を抽出します。RNA の抽出特定のキットを使用し、製造元のガイドラインに従ってください。クリーニング ワークスペースと RNase 除染製品、ピペットによる RNase フリー環境で動作して、フィルターのヒントを確認します。
- たて 50 μ M β-メルカプトエタノールを添加した換散バッファーの 1 つの mL を準備します。
注:ここでは、RNA の劣化を抑える β-メルカプトエタノールが追加されます。 - 無料水 RNase を使用して 70% と 80% のエタノール溶液を準備します。
- 50 μ M β-メルカプトエタノールを添加した換散バッファーの 75 μ L で細胞を溶解させます。セル中断を改善するためにシュレッダーを使用しています。
- 取扱説明書メーカーによると RNA の抽出を続行します。
- プロトコルの最後で、RNA 濃度が十分な RNase 自由水の 14 μ L の RNA を溶出します。
- たて 50 μ M β-メルカプトエタノールを添加した換散バッファーの 1 つの mL を準備します。
結果
記述されていたプロトコルは、神経細胞、マクロファージやミクログリア ゼブラフィッシュ仔脳から分離する簡単な方法です。これらから大量高品質のセルを分離 (凛 > 7) RNA を抽出しました。このプロトコルの目的は、さまざまな種類の細胞から中枢神経系、変更は最小限での彼らの遺伝子発現プロファイルを分析し、細胞の性質と機能を特徴付けるを分離することです。したがって、プロトコル全体が機械的脳組織の均質化と 4 ° C で実行されます。このメソッドは、正常に研究室で実行される 2 つの研究のために使用されています。最初の研究では、神経細胞とマクロファージ/ミクログリアは 8 dpf mpeg1:eGFP+/NBT:DsRed+幼虫 (図 1) から分離されました。FACS のサイズの関数で残骸からの細胞分離を許可 (FSC A) と粒度 (SSC-A) (図 1A)。単一のセルは、ダブレットまたは細胞塊 (図 1B) から、分かれていた。単一のセルの人口から (DAPI+) の死んだ細胞を除去するためにゲートを描いた。対応するドット プロットでは、死んだ細胞の率は 26.7% (図 1C) としてこの実験的プロトコルが細胞膜の完全性を維持すること明らかにしました。最後に、ニューロン (+した DsRed) とマクロファージ/ミクログリア (GFP+) 簡単に生きているセル人口のゲートから隔離されました。ニューロン集団 (23.1%) は、脳 (図 1D) 内のマクロファージ/ミクログリア人口 (1.56%) よりもより顕著なように見えた。このプロトコルは、その後 qPCR 神経細胞とマクロファージ/ミクログリアの間特定の遺伝子発現を比較する解析を実行するそれらの細胞からの RNA を隔離するできました。図 2ショー神経細胞やマクロファージ/ミクログリア遺伝子発現例としてβ-アクチン-ハウスキーピング遺伝子に対する増殖細胞核抗原 (pcna)の。
2 番目の研究のためこのメソッドはミクログリア 3、5、7 の dpf 幼虫の脳から分離に焦点を当てた。免疫染色で隔離されたセル、上記実験と対照をなして 4 4、特にミクログリア (図 3 A ~ D) と分類する抗体を使用しています。前述したように、ミクログリア (4 4+) 生きているセルから選ばれ、(図 3D) を収集します。ゼブラフィッシュ仔脳のミクログリア番号が変数 (表 1) と 3 dpf (∼ 25 魚あたり) で非常に低い。それらの細胞から抽出した RNA の量と質は、基づくマイクロ キャピラリー電気泳動システムを使用して測定しました。5 dpf ゼブラフィッシュ稚魚脳のミクログリアから抽出した RNA の得られた結果は、RNA 分析 (表 1 (5 dpf; 実験 4)) の例を説明するために提供されています。図 4は、電気泳動トレースとリボソーム RNA (28 秒・ 18 秒) を明確に可視化でこのサンプルの得られたそのグラフィック表現を示します。このデータは、凛のサンプルを計算して RNA 濃度を決定するため必要です。表 1は、魚、ミクログリアの RNA の量、3、5、7 の dpf でそれぞれ異なる実験のため得られた鈴スコアごと分離ミクログリアの数をまとめたものです。このメソッドを使用して分離のミクログリアから抽出した RNA の品質と量キットを使用して cDNA に RNA を増幅することができました。エジンバラ ゲノミクスによって提供される質と量のテストは、増幅 cDNA ライブラリの準備および後続のシーケンスのための十分な品質のであることを確認します。図 5は、cDNA 断片の電気泳動システムを使用して測定量のサイズ分布を示しています。このサンプルでは cDNA 36100 の濃度の 299bp の平均サイズがあった pmol/l.テーブル 2はそれぞれ (表 1 (5 dpf; 実験 4)) の RNA のサンプルからの増幅された cDNA は、質と量のテストを示しています。増幅 cDNA は、シーケンスの正常に使用されています。
研究室で実行されるいくつかの研究は、神経細胞、マクロファージやミクログリアから抽出した RNA の量と質を後続の qPCR のゲノム広い遺伝子発現解析使用できることを確認しました。したがって、この実験的なプロトコルは、膜の完全性の変更とその遺伝子発現プロファイルの変更を制限することがなく中枢神経系細胞の種類を確実に分離する使用できます。

図 1: 8 dpf ゼブラフィッシュ幼虫神経細胞とマクロファージ/ミクログリア mpeg1:GFP+/NBT:DsRed+からの並べ替え FACS 。(A ~ C)前方散乱、側による単一細胞が散布図 (B)、連続ゲーティングすべて脳からの細胞 (A) 連続しない選択を示しています。(C) 死んだ細胞は、DAPI ラベリングによって除外されました。(D) 神経細胞とマクロファージ/ミクログリアは、下流と GFP 陽性によってそれぞれ識別されました。この図の拡大版を表示するのにはここをクリックしてください。
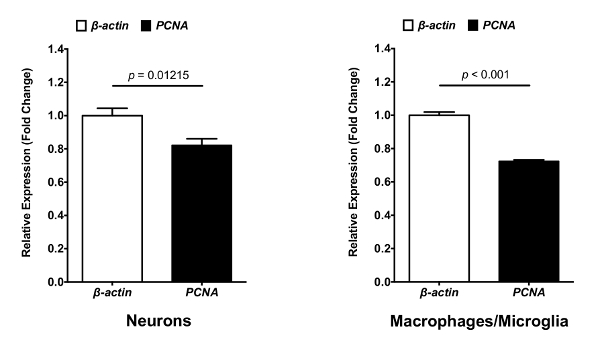
図 2: Pcnaと神経細胞とマクロファージ/ミクログリアにおけるβ-アクチン遺伝子発現解析します。分離ニューロンとマクロファージ/ミクログリアからの RNA は、定量的 PCR 解析で使用するための cDNA に転写できます。β-アクチンの家は維持遺伝子分離神経細胞とマクロファージ/ミクログリア qPCR による決定に対するpcnaの mRNA 発現レベル (N = 3)。フォールドの変更を比較 (ΔΔCT) メソッドを使用して測定しました。誤差範囲を表す平均 ± SEM.この図の拡大版を表示するのにはここをクリックしてください。

図 3: 3 dpf ゼブラフィッシュ幼虫からミクログリアの並べ替え FACS 。(A ~ C)連続ゲーティングを逐次すべて脳の細胞 (A)、前方散乱、側による単一細胞が散布図 (B)。(C) 死んだ細胞は、DAPI ラベリングによって除外されました。(D) ミクログリアが 4 4 によって識別された陽性。この図の拡大版を表示するのにはここをクリックしてください。

図 4: 5 dpf ゼブラフィッシュ ミクログリアから抽出した RNA のマイクロ キャピラリー電気泳動結果。2 つの背の高いピークは、18 s、28 s リボソーム RNA です。RNA の整合性の数 (鈴) だった全体の電気泳動のトレースの分析と 18 秒と 28 秒の ribosomal 亜単位の生成比率を用いてバイオアナライザー ソフトウェアによって自動的に計算されます。ミクログリアの RNA が鈴 8.6 です。この図の拡大版を表示するのにはここをクリックしてください。

図 5: 5 dpf ゼブラフィッシュ ミクログリアの RNA のサンプルからの増幅された cDNA の質と量のテスト。平均サイズが 299 の分析サンプルの cDNA フラグメント サイズの分布の図は跪くこの図の拡大版を表示するのにはここをクリックしてください。
| 条件 | 実験 | 魚の数 | セル数 | 魚あたりのセル数 | RNA 濃度 (pg/ul) | 総 RNA (pg) | 1 つのセル (pg) RNA 量 | 凛のスコア |
| 3dpf | 1 | 700 | 11922 | 17.03 | 126 | 1512 | 0.13 | 8 |
| 3dpf | 2 | 600 | 22527 | 37.55 | 253 | 3036 | 0.13 | 7.9 |
| 3dpf | 3 | 600 | 18688 | 31.15 | 255 | 3060 | 0.16 | 7.7 |
| 3dpf | 4 | 600 | 11121 | 18.54 | 189 | 2268 | 0.20 | 7.8 |
| 3dpf | 5 | 600 | 15581 | 25.97 | 131 | 1572 | 0.10 | 8.4 |
| 3dpf | 6 | 600 | 11965 | 19.94 | 256 | 3072 | 0.26 | 8.2 |
| 5dpf | 1 | 600 | 58629 | 97.72 | 362 | 4344 | 0.07 | 7.4 |
| 5dpf | 2 | 600 | 32510 | 54.18 | 348 | 4176 | 0.13 | 8.1 |
| 5dpf | 3 | 600 | 77884 | 129.81 | 594 | 7128 | 0.09 | 8.3 |
| 5dpf | 4 | 600 | 50755 | 84.59 | 305 | 3660 | 0.07 | 8.6 |
| 5dpf | 5 | 600 | 44967 | 74.95 | 134 | 1608 | 0.04 | 7.6 |
| 5dpf | 6 | 600 | 51031 | 85.05 | 163 | 1956 | 0.04 | 7.9 |
| 7dpf | 1 | 600 | 60496 | 100.83 | 183 | 2196 | 0.04 | 7.6 |
| 7dpf | 2 | 450 | 55517 | 123.37 | 183 | 2196 | 0.04 | 7.8 |
| 7dpf | 3 | 600 | 88897 | 148.16 | 465 | 5580 | 0.06 | 8.1 |
| 7dpf | 4 | 600 | 63008 | 105.01 | 356 | 4272 | 0.07 | 8.4 |
| 7dpf | 5 | 350 | 34956 | 99.87 | 245 | 2940 | 0.08 | 8.1 |
| 7dpf | 6 | 600 | 63887 | 106.48 | 341 | 4092 | 0.06 | 7.8 |
表 1: ミクログリア分離と 3、5、7 の dpf ゼブラフィッシュ幼虫からの RNA 抽出データの概要。
| サンプルの内部 ID | 外部サンプル ID | 量子ビット (ng/ul) | Qubit(ng/ul) | 平均濃度 (ng/ul) | ボリューム (ul) | 受信した ug | 最小濃度の合否を判定 | 最小量の合否を判定 | 推奨量の合否を判定 |
| 10907SD0010 | 5 dpf (実験 4) | 58.8 | 58.4 | 58.6 | 30 | 1.76 | 渡す | 渡す | 渡す |
| サンプル ライブラリの準備のための要件: | |||||||||
| ライブラリの準備 | 最小量 (ng) | 推奨量 (ng) | 最小濃度 ng/uL | ||||||
| TruSeq ナノ ゲル cDNA から無料 350 bp 挿入ライブラリ | 600 | 1100 | 10 | ||||||
表 2:5 の dpf ゼブラフィッシュ ミクログリアの RNA のサンプルからの増幅された cDNA の数量をテストします。
ディスカッション
ここで記述されている実験のプロトコルは、3 から 8 dpf にゼブラフィッシュ幼虫から脳細胞を分離する堅牢で効率的なメソッドを表します。重要なは、ゼブラフィッシュ稚魚の脳のミクログリアの特定の分離を可能にする最初のプロトコルです。プロトコルは、細胞膜の完全性を維持するために、処理中に発生する遺伝子発現の潜在的な変更を最小限に抑えるために設計されています。この最後の点は、これらの孤立した細胞ゲノムのプロファイルの解析に基づく結果の関連性にとって重要です。確かに、小グリア細胞およびマクロファージの偏光は、その微小環境に左右され。37 ° C でこれらの細胞が実験条件 (損傷 (断裂) の応答) への応答での遺伝子発現プロファイルを変わっていたでしょう。したがって、RNA の抽出、細胞プロセスと代謝活動を遅く前に 4 ° C でこの実験を実行することが重要だった。さらに、4 ° C で機械的脳組織の均質化は、37 ° C で酵素のティッシュの消化ではなく遺伝子発現への影響を避けるために選ばれました。
このメソッドは非常に簡単です。 強調表示することが重要です。日内でいくつかの脳細胞集団は、少なくとも 2 つの異なる実験条件から抽出した RNA の分離できます。プロトコルの長さの合計は、幼虫の頭の断裂が制限ステップ (∼ 350 頭/h) 条件ごとに使用される幼虫の個体数に依存します。一般に、3 からミクログリアを使用する 5 と 7 の dpf は勧めトランセクト ∼ 600 頭 (表 1) を抽出するのに十分な RNA を取得するテストごとにします。ミクログリア細胞型を表す最も低い収量 (7 dpf で一人当たり約 112 細胞)、マクロファージ (7 dpf で一人当たり約 170 細胞) を含む他の細胞型のヘッドの数を減らすことが。
このプロトコルの別の利点は、FACS 選別機の設定が確立されるは、異なる実験の同じ設定を使用できます。その細胞集団に完璧にフィット 1 つの実験から別以前に設計された、ゲートでこのメソッドを使用して実験の再現性を示すことが観察されています。
このメソッドの欠点はわずかは、収穫された RNA の比較的低いです。ただし、この制限は、脳開発 (表 1) の初期段階でミクログリア数が非常に少ない技術的な問題よりもより多くの生物学的問題です。収集された RNA のこの少量のため増幅手順はゲノム広い遺伝子発現解析に考慮する必要が.幸いなことに、増幅手順は高品質 cDNA の十分な量を生成します。したがって、単離細胞の遺伝子発現プロファイルのグローバルな変化を学ぶことができます。
結論としては、このプロトコルは、分離し、各種ゼブラフィッシュ稚魚の脳から中枢神経系細胞を研究する手法を提供します。これは、についてその疾患における役割を研究開発中にこれらの細胞のより深い理解を得るために適用できます。
開示事項
著者が明らかに何もありません。
謝辞
初期ヘルプと実験的アプローチと QMRI フローサイトメトリーと細胞選別設備に関する議論、博士クレア デイビスと博士ヴェロニク ミロン (女王の医療研究所、エジンバラ、ユナイテッド キングドーム) に感謝します。この作品は、博士ダーク シエガー癌研究英国のキャリア確立賞によって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| 1-phenyl 2-thiourea (PTU) | Sigma | P7629 | |
| Hepes | Gibco | 15630-056 | |
| D-Glucose | Sigma | G8644-100ML | |
| HBSS 1X | Gibco | 14170-088 | |
| Percoll | GE Healthcare | 17-0891-02 | |
| HBSS 10X | Gibco | 14180-046 | |
| DPBS 1X | Gibco | 14190-094 | |
| Tricaine (MS222) | Sigma | A5040 | |
| Sterilin standard 90mm petri dishes | ThermoFisher | 101VIRR | |
| Surgical micro-scissors | Fine Science Tools | 15000-00 | |
| 3 mL Pasteur plastic bulk pipette | SLS | PIP4206 | |
| Glass homogenizer | Wheaton | 357538 | |
| Sterilin standard 55mm petri dishes | ThermoFisher | P55V | |
| Percoll | GE Healthcare | 17-0891-02 | |
| 40 µm cell strainer | Falcon | 352340 | |
| 50 ml polypropylen conical tube | Falcon | 352070 | |
| Centrifuge | Eppendorf | 5804 R | |
| 10 mL syringe | BD | 302188 | |
| Needle 23G x 1'' | BD | 300800 | |
| Normal goat serum (NGS) | Cell Signalling | 5425S | |
| 35 μm cell strainer cap | BD | 352235 | |
| FACS tubes | BD | 352063 | |
| Low Endotoxin, Azide-Free (LEAF) | Biolegend | 101321 | |
| Alexa Fluor 647 Goat Anti-Mouse IgG (H+L) | Life Technologies | A11008 | |
| Anti-4C4 | Courtesy of Catherina Becker (University of Edinburgh) | ||
| FACS sorter FACSAria II | BD | QMRI, FACS facility | |
| RNeasy Plus Micro Kit | QIAGEN | 74034 | |
| β-mercaptoethanol | Gibco | 31350-010 | |
| QIAshredder | QIAGEN | 79654 | |
| SsoAdvanced Universal SYBR Green Supermix | Bio-Rad | 1725271 | |
| SuperScript III First-Strand Synthesis System | Invitrogen | 18080-051 | |
| LightCycler 96 Real-Time PCR System | Roche | ||
| Ovation RNA-Seq System V2 | NuGEN | 7102-32 | |
| Agilent RNA 6000 Pico reagents | Agilent | 5067-1513 | |
| 2100 Bioanalyzer | Agilent | ||
| RNaseZap | Ambion | AM9780 |
参考文献
- Chrast, R., Scott, H. S., et al. The Mouse Brain Transcriptome by SAGE: Differences in Gene Expression between P30 Brains of the Partial Trisomy 16 Mouse Model of Down Syndrome (Ts65Dn) and Normals. Genome Research. 10 (12), 2006-2021 (2000).
- Beis, D., Stainier, D. Y. R. In vivo cell biology: following the zebrafish trend. Trends in Cell Biology. 16 (2), 105-112 (2006).
- Shiau, C. E., Kaufman, Z., Meireles, A. M., Talbot, W. S. Differential Requirement for irf8 in Formation of Embryonic and Adult Macrophages in Zebrafish. PLoS ONE. 10 (1), 0117513-0117515 (2015).
- Mazaheri, F., Breus, O., et al. Distinct roles for BAI1 and TIM-4 in the engulfment of dying neurons by microglia. Nature Communications. 5, 1-11 (2014).
- Oosterhof, N., Holtman, I. R., et al. Identification of a conserved and acute neurodegeneration-specific microglial transcriptome in the zebrafish. Glia. 65 (1), 138-149 (2016).
- Hamilton, L., et al. A Zebrafish Live Imaging Model Reveals Differential Responses of Microglia Toward Glioblastoma Cells In Vivo. Zebrafish. , (2016).
- Ellett, F., Pase, L., Hayman, J. W., Andrianopoulos, A., Lieschke, G. J. mpeg1 promoter transgenes direct macrophage-lineage expression in zebrafish. Blood. 117 (4), 49-56 (2011).
- Peri, F., Nüsslein-Volhard, C. Live Imaging of Neuronal Degradation by Microglia Reveals a Role for v0-ATPase a1 in Phagosomal Fusion In Vivo. Cell. 133 (5), 916-927 (2008).
- Sieger, D., Moritz, C., Ziegenhals, T., Prykhozhij, S., Peri, F. Long-range Ca2+ waves transmit brain-damage signals to microglia. Developmental cell. 22 (6), 1138-1148 (2012).
- Becker, T., Becker, C. G. Regenerating descending axons preferentially reroute to the gray matter in the presence of a general macrophage/microglial reaction caudal to a spinal transection in adult zebrafish. The Journal of comparative neurology. 433 (1), 131-147 (2001).
- Ohnmacht, J., Yang, Y., et al. Spinal motor neurons are regenerated after mechanical lesion and genetic ablation in larval zebrafish. Development. 143 (9), Cambridge, England. 1464-1474 (2016).
- Roy-Carson, S., Natukunda, K., et al. Defining the transcriptomic landscape of the developing enteric nervous system and its cellular environment. BMC Genomics. 18 (1), 1-24 (2017).
- Khuansuwan, S., Gamse, J. T. Identification of differentially expressed genes during development of the zebrafish pineal complex using RNA sequencing. Developmental biology. 395 (1), 144-153 (2014).
- Schmid, C. D., Melchior, B., et al. Differential gene expression in LPS/IFNγ activated microglia and macrophages: in vitroversus in vivo. Journal of Neurochemistry. 109, 117-125 (2009).
- Bohlen, C. J., Bennett, F. C., Tucker, A. F., Collins, H. Y., Mulinyawe, S. B., Barres, B. A. Diverse Requirements for Microglial Survival, Specification, and Function Revealed by Defined- Medium Cultures. Neuron. 94 (4), 759-773 (2017).
- Khan, A., Ju, F., et al. Transcriptomic analysis reveals differential activation of microglial genes after ischemic stroke in mice. Neuroscience. 348, 212-227 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved