Method Article
Aislamiento y extracción de ARN de las neuronas, macrófagos y Microglia de cerebros de larvas de pez cebra
En este artículo
Resumen
Presentamos un protocolo para aislar a las neuronas, macrófagos y microglia del cerebro de pez cebra larvas bajo condiciones fisiológicas y patológicas. Al aislamiento, el RNA se extrae de estas células a analizar su perfil de expresión génica. Este protocolo permite la colección de ARN de alta calidad para realizar análisis posteriores como qPCR y transcriptómica.
Resumen
Para obtener una comprensión detallada del papel de diferentes células del SNC durante el desarrollo o el establecimiento y progresión de las patologías del cerebro, es importante aislar estas células sin modificar su perfil de expresión génica. El modelo de pez cebra ofrece un gran número de líneas de peces transgénicos en los que se etiquetan tipos celulares específicos; por ejemplo las neuronas de la línea de NBT:DsRed o los macrófagos/microglía en la línea de mpeg1:eGFP. Además, se han desarrollado anticuerpos para teñir células específicas, tales como la microglia con el 4 4 anticuerpos.
Aquí, describimos el aislamiento de las neuronas, macrófagos y microglia del cerebro larvas de pez cebra. Este protocolo es la evitación de una digestión enzimática del tejido a 37 ° C, que podría modificar perfiles celulares. En su lugar se utiliza un sistema mecánico de homogeneización de tejidos a 4 º C. Este protocolo implica homogeneización de cerebros en suspensión de células, su inmuno-tinción y el aislamiento de las neuronas, macrófagos y microglía por FACS. Luego, se extrajeron RNA de esas células y evaluaron su calidad o cantidad. Logramos obtener ARN de alta calidad (número de integridad del RNA (RIN) > 7) para llevar a cabo la qPCR en el análisis de los macrófagos/microglía y neuronas y transcriptómicos de microglia. Este enfoque permite una mejor caracterización de estas células, así como una comprensión más clara sobre su papel en el desarrollo y patologías.
Introducción
Conocimiento en el desarrollo del cerebro y enfermedades cerebrales ha mejorado significativamente en la última década desde la primera cuantificación de transcriptomas de cerebro de ratón1. De hecho, análisis de expresión génica amplia genoma nos da acceso a toda la información genética en el tejido cerebral y las células que pueden complementar y mejorar observaciones con otras técnicas y herramientas.
El pez cebra es un modelo biológico potente, fácil de criar y de modificar genéticamente; su transparencia óptica en estadios larvarios permite en proyección de imagen de observaciones2. Por desgracia, en comparación al humano y ratón, el número de anticuerpos disponibles para realizar tinción de inmuno es bastante baja. Para remediar esto, líneas de pesca de pez cebra transgénico se hacen fácilmente modificando genéticamente pescado para expresar proteínas fluorescentes bajo promotores específicos de tipo celular. Líneas de pez cebra transgénico se han utilizado en el pasado para estudiar la función de macrófagos y microglia durante el desarrollo del sistema nervioso central (SNC) y enfermedad3,4,5,6. Sin embargo, para obtener una comprensión detallada de estos procesos tenemos que entender los cambios en la expresión génica en los tipos respectivos de la célula. Para ello, hemos desarrollado un método experimental para aislar específicamente células como neuronas, macrófagos y microglia de 3 a 8 días cerebros de larvas de pez cebra de la fertilización (dpf). Para el establecimiento del Protocolo, hemos trabajado con líneas de peces transgénicos que expresan la proteína verde fluorescente (GFP) en los macrófagos/microglía bajo el promotor del gen expresado en macrófagos (mpeg1:eGFP) y DsRed en las neuronas bajo la ß-tubulina neural promotor (NBT:DsRed)7,8,9. Además, se realizó tinción de inmuno de microglia 4 en 4, un anticuerpo monoclonal de ratón que tiñe específicamente el pez cebra microglia10,11. Luego, el ácido ribonucleico (ARN) se extrae de estas células más reacción en cadena de la polimerasa cuantitativa (qPCR) o análisis de transcriptoma. Este protocolo ha sido diseñado eficientemente homogeneizar el tejido de cerebro de larvas de pez cebra; recoger las neuronas, los macrófagos/microglía y microglia sin alteración de su integridad de la membrana plasmática y finalmente extraer RNA de estas células en alta calidad (RIN > 7) y la cantidad para realizar el análisis genómico. A diferencia de los estudios previamente publicados que utilizan tratamiento de tripsina a 37 ° C para digerir tejido de cerebro12,13, este protocolo promueve el trabajo a 4 ° C hasta el paso de extracción de RNA para reducir modificaciones del perfil de expresión génica. Este paso es crucial como microglia y macrófagos son las células sensibles que responden a los cambios en su microambiente inmediato alterando sus genes expresión perfil y polarización14,15, 16.
El protocolo, que se describe aquí en detalle, muestra que el aislamiento de las neuronas, macrófagos y microglia del cerebro larvas de pez cebra, que prácticamente, se puede adaptar a cualquier otra célula presente en el cerebro - mediante líneas de peces transgénicos o etiquetados con anticuerpos específicos. Este método permite una mejor caracterización de células del SNC a través de sus análisis de expresión del gen amplia de genoma y ayudará a entender su papel durante el desarrollo y enfermedades cerebrales.
Protocolo
1. la muestra y la preparación de los medios de comunicación
- Preparar medio embrión (E3) disolviendo 6,4 mM KCl, NaCl de 0,22 mM, 0,33 mM CaCl2 2 H2O, 0,33 mM MgSO4 7 H2O H2O.
- Recolectar embriones de pez cebra inmediatamente después de la fertilización (PD 0).
- Embriones de pez cebra se dividió en 50 por plato de petri de 90 mm. Crear embriones a 28,5 ° C en 50 mL de medio de embrión (E3) tratado con 200 μm 1-fenil 2-tiourea (PTU), desde el final del primer día de desarrollo (PD 0) para la duración del experimento para inhibir pigmentación.
- Cambio el E3 + PTU mediana diariamente para la duración del experimento.
- Pantalla mpeg1:eGFP y NBT:DsRed larvas en 2 PD con un estereomicroscopio fluorescente para la expresión de transgenes positivo, GFP+ los macrófagos/microglía y DsRed+ las neuronas.
- Preparar todos los medios el día antes del experimento bajo una campana de cultivo de tejidos para evitar la contaminación, a continuación, almacenar a 4 ° C.
- Preparar los medios de comunicación A disolviendo 15 mM Hepes y 25 mM D-glucosa en HBSS 1 x.
- Preparar medio de gradiente de densidad (100%) mediante la mezcla de 9 volúmenes de medio de gradiente de densidad en 1 volumen de HBSS 10 x.
- Preparar el medio de gradiente de densidad (22%) diluyendo 22 mL de medio de gradiente de densidad (100%) de 88 mL de DPBS 1 x.
- Preparar DPBS 1 x.
- Preparar medio de E3 + Tricaine mezclando medio E3 con 450 μM Tricaine.
2. homogeneización
Nota: Todos los pasos están realizados 4 ° C.
- Añadir 1.5 mL Metanosulfonato (15 mM) a platos de Petri de 90 mm que contienen larvas de 50 en 50 mL de medio de embrión de E3 tratado con 200 μm de PTU para anestesiar terminal les.
- Chupar hasta 10 larvas anestesiadas de los platos de petri con una pipeta de plástico a granel Pasteur de 3 mL.
- Transferencia anestesiado larvas 10 por 10 en un plato de petri de 55 mm llenan de helado medio embrión de E3 + Tricaine.
- Bajo un estereomicroscopio, alinear 10 larvas en el centro de la caja Petri. Entonces transecto cabezas larvas sobre el saco vitelino mediante micro-tijeras quirúrgicas (excluir vejiga natatoria para evitar flotación cabezas).
- Chupar cabezas de los platos de petri con una pipeta de plástico a granel Pasteur de 3 mL. Espere hasta que todas las cabezas se reúnen en la punta de la pipeta y luego transfieren en un homogeneizador de vidrio que contengan 1 mL A helada de los medios de comunicación (transferencia de un volumen mínimo para reducir los medios de comunicación una dilución por E3 + Metanosulfonato). Mantenga el homogenizador de vidrio en el hielo. Uso de un homogeneizador por condición experimental.
- Reemplazar cada pequeño placa de Petri que contiene hielo frío E3 + Metanosulfonato con uno nuevo cada 30 minutos para asegurar que corte transversal se realiza en frío E3 + Tricaine medio.
- Vuelva a colocar la helada A los medios de comunicación en el homogenizador de vidrio cuando el color se descolora.
Nota: Dilución de A los medios de comunicación puede alterar el tejido de la cabeza debido a los cambios de temperatura. - Una vez que se han recogido todas las cabezas (600 cabezas/condición), retire el volumen máximo de la multimedia al homogeneizador de vidrio y sustituirla por 1 mL fresca helada a los medios de comunicación.
- Afectar el tejido cerebral con un homogeneizador de vidrio firmemente en el hielo. Realizar 40 rondas de trituración y vueltas para 3-5 larvas de PD y 50 para las larvas de dpf 7 y 8.
- Añadir 2 mL del medio A suspensión de la célula (1 mL A los medios de comunicación / 200 cabezas), que se diluya las células y reducir su aglomeración con mielina para facilitar su separación durante la centrifugación en gradiente medio de densidad.
- Para eliminar la aglomeración de células, ejecutar la suspensión de células a través de 40 μm células colador colocado encima de un tubo de frío 50 mL falcon mantenido en hielo. Repetir esta operación 3 veces.
- Transfiera 1 mL de la suspensión celular en tubos de 1,5 mL frío y centrifugado a 300 g durante 10 min a 4 ° C.
- Eliminar el sobrenadante utilizando una jeringa de 10 mL + aguja 23 x 1''.
- Resuspenda el sedimento celular con 1 mL de helado 22% densidad gradiente medio suavemente overlaid por 0,5 mL de DPBS helada 1 x (do no mezcla de ellos, una interfase entre ambas soluciones se verá).
- Girar los tubos a 950 g sin freno y aceleración lento durante 30 min a 4 ° C.
Nota: Este paso separa del myelin de otras células por secuestrar en la interfase de DPBS 1 x y 22% medio gradiente de densidad, mientras que células se de pellets en la parte inferior del tubo. Eliminación de la mielina es más eficiente cuando la concentración de células no es demasiado alto. - Utilizando una jeringa de 10 mL + descarte aguja 23 x 1'' el máximo de DPBS, medio de gradiente de densidad y mielina atrapado en su interfase.
- Lavar las células con 0,5 mL del medio A + 2% de suero normal de cabra (NGS), luego gire tubos a 300 g durante 10 min a 4 ° C.
- Deseche el sobrenadante al máximo, entonces todos los pellets de celular de la misma condición experimental en 1 mL de medio A + 2% de la piscina NGS.
- Girar los tubos a 950 g sin freno y aceleración lento durante 30 min a 4 ° C.
- Si las células de interés expresan una proteína fluorescente como los macrófagos/microglía de mpeg1:eGFP o las neuronas de NBT:DsRed peces transgénicos (proyección de peces transgénicos ver 1.1.3), ejecutar la suspensión de células a través de un tamiz de 35 μm celular tapón y transfieren a un tubos de 5 mL frío FACS en el hielo, protegidos de la luz.
Nota: Alternativamente, se puede realizar inmuno-tinción de microglia.
3. Microglia inmuno-tinción
Nota: Todas las medidas se realizan a 4 ° C.
- Resuspenda el sedimento celular con 0,3 mL de medios de comunicación A + 2% NGS. Dividir en tubos de 3 x 1,5 mL: uno de células sin manchas medir auto fluorescencia de las células de interés, en segundo lugar para que el anticuerpo secundario (1/200) medir la Unión inespecífica del anticuerpo secundario a la microglia y el tercero como prueba (4 de 4 ratón monoclonal anticuerpo (microglia específica) (1/20) + (1/200) de anticuerpo secundario).
- Agregar baja endotoxina, libre de azida (hoja) al 1% a las células (los tubos) para bloquear las interacciones CD16/CD32 con el dominio Fc de las inmunoglobulinas. Incube las células por 10 min con suave agitación cada 5 min.
- Añadir los 4 4 anticuerpos (1/20) a las células (tubo 3) e incubar por 30 min con suave agitación cada 10 minutos.
- Girar los tubos a 300 g durante 10 min a 4 ° C y descarte el sobrenadante.
- Lavar una vez con 0,5 mL del medio A + 2% NGS, luego girar tubos a 300 g durante 10 min a 4 ° C.
- Resuspenda el sedimento celulares con 0,5 ml de medios de comunicación A + 2% NGS e incubar las células con la hoja en el 1% por 10 min con suave agitación cada 5 min.
- Añadir el anticuerpo secundario (1/200) a las células (tubo de 2 y 3). Incube las células por 30 min con suave agitación cada 10 min y la protección de la luz.
- Girar los tubos a 300 g durante 10 min a 4 ° C, luego deseche el sobrenadante.
- Lave dos veces con 0,5 mL de medios de comunicación A + 2% NGS, luego vuelva a suspender el sedimento celulares con 1 mL de medio A + 2% NGS.
- Ejecutar la suspensión de células a través de una tapa de filtro 35 μm células y transferirlos en tubos de 5 mL frío FACS en el hielo, protegido de la luz.
4. clasificación (FACS) de la célula
Nota: Realizar todos los pasos a 4 ° C.
- Clasificar las neuronas, los macrófagos/microglía y microglia usando un FACS.
Nota: Este paso generalmente se realiza por un miembro del personal de la instalación del FACS y ajustes dependen del tipo de equipo utilizado.- Añadir DAPI en una concentración de 1 μg/mL en cada tubo de FACS para etiquetar las células muertas.
- Configurar las neuronas FACS y tipo, los macrófagos/microglía o microglia de todas las células del cerebro. Separar las células de residuos en función de su tamaño y granularidad, a continuación solo las células por dispersión hacia delante y la dispersión lateral de la puerta. Excluir las células muertas, DAPI etiquetado de células vivas. Identificar las neuronas, los macrófagos/microglía o microglía por su respectiva coloración positiva.
- Recoger las células en tubos de 1,5 mL que contiene 1 mL de helado A Media + 2% NGS en hielo. Utilizar tubos diferentes para cada tipo de célula.
- Girar los tubos a 300 g durante 10 min a 4 ° C y luego descartar el sobrenadante.
- Lavar una vez con 0,5 mL de medios de comunicación un entonces deseche el máximo de sobrenadante.
5. extracción RNA
- Extraiga el RNA de los diferentes tipos de células separados por FACS. Para la extracción de RNA utiliza un kit específico y siga las instrucciones del fabricante. Asegúrese de trabajar en un ambiente libre de Rnasa por espacio de trabajo de limpieza y pipetas con un producto de descontaminación de RNasa y utilizar las puntas de filtro.
- Recién preparar 1 mL de tampón de lisis suplementado con 50 μm β-mercaptoetanol.
Nota: Aquí, β-mercaptoetanol es añadido para reducir la degradación del RNA. - Preparar solución de etanol 70% y 80% usando Rnasa agua libre.
- Lyse las células con 75 μl de tampón de lisis suplementado con 50 μm β-mercaptoetanol. Utilizar una trituradora para mejorar la interrupción de la célula.
- Seguir el manual de extracción de RNA según fabricante.
- Al final del Protocolo, eluir el RNA con 14 μl de agua libre de ARNasa para obtener una concentración suficiente de RNA.
- Recién preparar 1 mL de tampón de lisis suplementado con 50 μm β-mercaptoetanol.
Resultados
El protocolo descrito es un método sencillo para aislar a las neuronas, macrófagos y microglia del cerebro larvas de pez cebra. De estos aislados células, cantidades significativas de alta calidad (RIN > 7) se obtuvieron RNA. El objetivo de este protocolo es aislar a diferentes tipos de células del SNC, con la mínima modificación de su perfil de expresión génica para analizar y caracterizar las funciones y propiedades de celda. Por lo tanto, el conjunto del Protocolo se lleva a cabo a 4 ° C con una homogeneización del tejido de cerebro mecánico. Este método se ha utilizado con éxito para dos estudios realizados en el laboratorio. En el primer estudio, las neuronas y los macrófagos/microglía fueron aislados de 8 PD mpeg1:eGFP+/NBT:DsRed+ larvas (figura 1). FACS permitida la separación de células de residuos en función de su tamaño (FSC-A) y detalle (SSC-A) (figura 1A). Las células fueron separadas luego de Dobletes o aglomeraciones de células (figura 1B). De la población de la célula, una puerta fue dibujada para eliminar células muertas (DAPI+). La trama de punto correspondiente reveló que este protocolo experimental conserva la integridad de membrana plasmática de la célula, como la tasa de células muertas es sólo el 26.7% (figura 1C). Finalmente, las neuronas (DsRed+) y los macrófagos/microglía (GFP+) fácilmente fueron separados de las puertas de la población de células vivas. La población de neuronas (23,1%) parece ser más prominente que la población de macrófagos/microglía (1.56%) dentro del cerebro (figura 1D). Este protocolo ha permitido para aislar ARN de las células para llevar a cabo análisis de qPCR posteriores para comparar la expresión de genes específicos entre las neuronas y los macrófagos/microglía. Figura 2 se muestra neuronal y niveles de expresión de gene de los macrófagos/microglía del antígeno nuclear de proliferación celular (pcna)contra el gen de mantenimiento de la casa β-actina como ejemplo.
Para el segundo estudio este método centrado en aislamiento microglia del cerebro larvas de 3, 5 y 7 PD. En contraste con el experimento descrito anteriormente, las células fueron aisladas por inmuno-tinción 4 en 4, un anticuerpo que específicamente etiquetas microglia (figura 3 A-D). Como se describió anteriormente, microglía (4 de 4+) fueron seleccionados de células vivas y recogidas (figura 3D). Números de la microglia en cerebros de larvas de pez cebra son variables (tabla 1) y muy baja en PD 3 (∼ 25 por peces). Calidad y cantidad de ARN extraído de las células se midieron utilizando un sistema de electroforesis capilar en. Resultados obtenidos del ARN extraído de microglía del cerebro de larvas de pez cebra de PD 5 se han proporcionado para ilustrar un ejemplo de análisis de ARN (tabla 1 (PD 5, experimento 4)). La figura 4 muestra el rastro de la electroforesis y su representación gráfica obtenida para esta muestra con una visualización clara del ARN ribosomal (28s y 18s). Este dato es necesario para calcular la muestra de RIN y para determinar la concentración de RNA. La tabla 1 resume el número de microglía aislado por el pescado, la cantidad de RNA por la microglia y la puntuación de RIN obtenida para cada experimento diferentes en PD 3, 5 y 7. La cantidad y la calidad del ARN extraído de microglia aislado usando este método nos permitieron amplificar el RNA en cDNA usando un kit. Pruebas de calidad y la cantidad proporcionadas por genómica de Edimburgo confirman que el cDNA amplificado es de calidad suficiente para la preparación de la biblioteca y posterior secuenciación. La figura 5 muestra la distribución de tamaño de fragmentos de cDNA y su cantidad medida usando un sistema electroforético. En este ejemplo, el cDNA tenía un tamaño medio de 299bp a una concentración de 36100 pmol/l. tabla 2 ilustra respectivamente pruebas de calidad y la cantidad de cDNA amplificado de muestras de RNA (tabla 1 (PD 5, experimento 4)). El cDNA amplificado se ha utilizado con éxito para la secuencia.
Varios estudios realizados en el laboratorio confirmaron que la calidad y la cantidad de ARN extraído de las neuronas, macrófagos y microglia pueden utilizarse para qPCR posteriores y análisis de expresión del gene amplia de genoma. Por lo tanto, este protocolo experimental puede utilizarse para aislar confiablemente diferentes tipos de células del SNC sin alterar su integridad de la membrana y limitar la modificación de su perfil de expresión génica.

Figura 1 : FACS clasificación de las neuronas y los macrófagos/microglía de /NBT:DsRed mpeg1:GFP++ 8 larvas de pez cebra PD. (A-C) Gating sucesivas muestra selección secuencial de todo cerebro células (A), dispersión de las células por dispersión hacia delante y lateral (B). Las células c muertos fueron excluidas por DAPI etiquetado. (D) las neuronas y los macrófagos/microglía se identificaron respectivamente por la coloración positiva DsRed y GFP. Haga clic aquí para ver una versión más grande de esta figura.
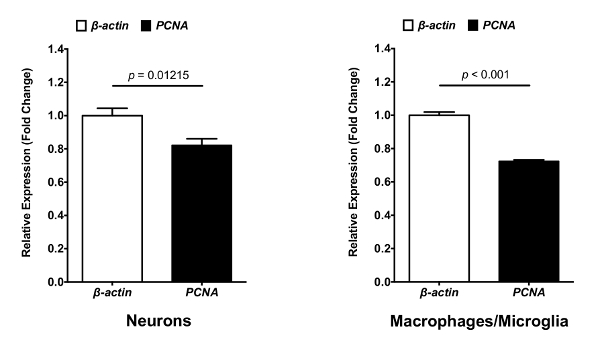
Figura 2 : Análisis de expresión génica para pcna y β-actina en las neuronas y los macrófagos/microglía. ARN de neuronas aisladas y los macrófagos/microglía se puede transcribir en cDNA para el uso en análisis cuantitativo por PCR. niveles de expresión de mRNA de pcna contra β-actina mantenimiento gene en neuronas aisladas y los macrófagos/microglía determinado por qPCR (N = 3). Doble cambio se midió utilizando el método comparativo (ΔΔCT). Error de la barra representan media ± SEM. haga clic aquí para ver una versión más grande de esta figura.

Figura 3 : FACS clasificación de microglia de 3 larvas de pez cebra PD. (A-C) Bloquea sucesivas Mostrar selección secuencial del cerebro todas las células (A), dispersión de las células por dispersión hacia delante y lateral (B). Las células c muertos fueron excluidas por DAPI etiquetado. (D) Microglia se identificaron 4 4 tinción positiva. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4 : Resultados de electroforesis capilar de ARN extraído de microglia de pez cebra de PD 5. Los dos altos picos son el ARN ribosomal 18S y 28S. Número de integridad del RNA (RIN) fue calculado automáticamente por el software bioanalyzer usando la relación generada entre el 18S y 28S ribosomal subunidades y el análisis de la traza entera electroforético. Microglia ARN tiene un 8.6 de RIN. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5 : Pruebas de calidad y la cantidad de cDNA amplificado de muestras de RNA de la microglía de pez cebra de PD 5. La imagen muestra la distribución de tamaño fragmento de cDNA de la muestra analizada con un tamaño promedio de 299 BP haga clic aquí para ver una versión más grande de esta figura.
| Condición | Experimento | Número de los pescados | Número de células | Número de células por pescado | Concentración de RNA (pg/ul) | ARN total (pg) | Cantidad de RNA por la célula (pg) | Puntuación de RIN |
| 3dpf | 1 | 700 | 11922 | 17.03 | 126 | 1512 | 0.13 | 8 |
| 3dpf | 2 | 600 | 22527 | 37,55 | 253 | 3036 | 0.13 | 7.9 |
| 3dpf | 3 | 600 | 18688 | 31.15 | 255 | 3060 | 0.16 | 7.7 |
| 3dpf | 4 | 600 | 11121 | 18.54 | 189 | 2268 | 0.20 | 7.8 |
| 3dpf | 5 | 600 | 15581 | 25.97 | 131 | 1572 | 0.10 | 8.4 |
| 3dpf | 6 | 600 | 11965 | 19.94 | 256 | 3072 | 0.26 | 8.2 |
| 5dpf | 1 | 600 | 58629 | 97.72 | 362 | 4344 | 0.07 | 7.4 |
| 5dpf | 2 | 600 | 32510 | 54.18 | 348 | 4176 | 0.13 | 8.1 |
| 5dpf | 3 | 600 | 77884 | 129.81 | 594 | 7128 | 0.09 | 8.3 |
| 5dpf | 4 | 600 | 50755 | 84.59 | 305 | 3660 | 0.07 | 8.6 |
| 5dpf | 5 | 600 | 44967 | 74.95 | 134 | 1608 | 0.04 | 7.6 |
| 5dpf | 6 | 600 | 51031 | 85.05 | 163 | 1956 | 0.04 | 7.9 |
| 7dpf | 1 | 600 | 60496 | 100.83 | 183 | 2196 | 0.04 | 7.6 |
| 7dpf | 2 | 450 | 55517 | 123.37 | 183 | 2196 | 0.04 | 7.8 |
| 7dpf | 3 | 600 | 88897 | 148.16 | 465 | 5580 | 0.06 | 8.1 |
| 7dpf | 4 | 600 | 63008 | 105.01 | 356 | 4272 | 0.07 | 8.4 |
| 7dpf | 5 | 350 | 34956 | 99.87 | 245 | 2940 | 0.08 | 8.1 |
| 7dpf | 6 | 600 | 63887 | 106.48 | 341 | 4092 | 0.06 | 7.8 |
Tabla 1: Resumen de microglia aislamiento y datos de extracción de RNA de las larvas de pez cebra de PD 3, 5 y 7.
| ID de muestra interna | ID de muestra externo | Qubit (ng/ul) | Qubit(NG/UL) | Concentración media (ng/ul) | Volumen (ul) | recibido de la UG | Pasa/no pasa para la concentración mínima | Pasa/no pasa para la mínimo cantidad | Pasa/no pasa para la cantidad recomendada |
| 10907SD0010 | 5 PD (experimento 4) | 58.8 | 58.4 | 58.6 | 30 | 1.76 | Pasar | Pasar | Pasar |
| Requisitos de la muestra para la preparación de la biblioteca: | |||||||||
| Preparación de la biblioteca | Cantidad mínima (ng) | Cantidad recomendada (ng) | Mínima concentración ng/uL | ||||||
| TruSeq Nano gel biblioteca de insertar bp 350 libre de cDNA | 600 | 1100 | 10 | ||||||
Tabla 2: Pruebas de la cantidad de cDNA amplificado de muestras de RNA de la microglía de pez cebra de PD 5.
Discusión
El protocolo experimental descrito aquí representa un método robusto y eficiente para aislar las células del cerebro de las larvas de pez cebra de 3 a 8 PD. Lo importante, este es el primer protocolo que permite el aislamiento específico de microglía del cerebro larvas de pez cebra. El protocolo está diseñado para preservar la integridad de la membrana de la célula y para minimizar posibles modificaciones de la expresión génica que ocurren durante el proceso. Este último punto es crucial para la relevancia de los resultados basados en el análisis de los perfiles genómicas celulares aisladas. De hecho, la polarización microglia y macrófagos están fuertemente influenciadas por su microambiente. A 37 ° C, estas células habrían cambiado su perfil de expresión génica en respuesta a las condiciones experimentales (respuesta de la lesión (corte transversal)). Por lo tanto, era crucial para llevar a cabo este experimento a 4 ° C antes de la extracción de RNA, frenar actividades metabólicas y procesos celulares. Además, la homogeneización del tejido de cerebro mecánico a 4 ° C fue elegida en lugar de digestión enzimática del tejido a 37 ° C para evitar cualquier impacto en los perfiles de expresión génica.
Es importante destacar que este método es muy rápido; en un día varias poblaciones de la célula de cerebro pueden ser aisladas de al menos dos diferentes condiciones experimentales y su ARN extraído. La longitud total del protocolo depende del número de larvas que se utiliza para cada condición como el transection de cabezas larvales es el paso limitante (∼ 350 cabezas por hora). En general, para trabajar con microglia de 3, 5 y 7 PD se recomienda transecto 600 cabezas ∼ por prueba para conseguir suficiente RNA para extraer (tabla 1). Microglía representan el tipo de célula con el más bajo rendimiento (aprox. 112 células por cabeza en dpf 7), puede reducir el número de cabezas para otros tipos de células incluyendo los macrófagos (aprox. 170 células por cabeza en dpf 7).
Otra ventaja de este protocolo es que, una vez que se han establecido parámetros en el clasificador de FACS, la misma configuración puede utilizarse para diversos experimentos. Se ha observado que poblaciones celulares encajan perfectamente de un experimento a otro con puertas previamente diseñados, que muestra la reproducibilidad de los experimentos con este método.
Una leve desventaja de este método es la cantidad relativamente baja de RNA que se cosecha. Sin embargo, esta limitación es más una cuestión biológica que una cuestión técnica, como el número de microglía es muy escaso en las primeras etapas del desarrollo del cerebro (tabla 1). Debido a esta baja cantidad de RNA recogido, pasos de amplificación deben ser considerados para llevar a cabo análisis de genoma amplio gene expresión. Afortunadamente, estos pasos de amplificación producen cantidades suficientes de cDNA de alta calidad. Por lo tanto, pueden estudiarse los cambios globales en los perfiles de expresión génica de células aisladas.
En conclusión, este protocolo proporciona un método robusto para aislar y estudiar varios tipos de la célula de la CNS de cerebros de larvas de pez cebra. Esto se puede aplicar para obtener una comprensión más profunda de estas células durante el desarrollo, así como para estudiar su papel en la enfermedad.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos Dr. Claire Davis y el Dr. Veronique Miron (de la reina Instituto de investigación médica, Edimburgo, Reino Unido) ayuda inicial y debates sobre el enfoque experimental y la citometría de flujo de QMRI y facilidad de clasificación celular. Este trabajo fue apoyado por un cáncer Research UK carrera establecimiento Premio a Dr. Dirk Sieger.
Materiales
| Name | Company | Catalog Number | Comments |
| 1-phenyl 2-thiourea (PTU) | Sigma | P7629 | |
| Hepes | Gibco | 15630-056 | |
| D-Glucose | Sigma | G8644-100ML | |
| HBSS 1X | Gibco | 14170-088 | |
| Percoll | GE Healthcare | 17-0891-02 | |
| HBSS 10X | Gibco | 14180-046 | |
| DPBS 1X | Gibco | 14190-094 | |
| Tricaine (MS222) | Sigma | A5040 | |
| Sterilin standard 90mm petri dishes | ThermoFisher | 101VIRR | |
| Surgical micro-scissors | Fine Science Tools | 15000-00 | |
| 3 mL Pasteur plastic bulk pipette | SLS | PIP4206 | |
| Glass homogenizer | Wheaton | 357538 | |
| Sterilin standard 55mm petri dishes | ThermoFisher | P55V | |
| Percoll | GE Healthcare | 17-0891-02 | |
| 40 µm cell strainer | Falcon | 352340 | |
| 50 ml polypropylen conical tube | Falcon | 352070 | |
| Centrifuge | Eppendorf | 5804 R | |
| 10 mL syringe | BD | 302188 | |
| Needle 23G x 1'' | BD | 300800 | |
| Normal goat serum (NGS) | Cell Signalling | 5425S | |
| 35 μm cell strainer cap | BD | 352235 | |
| FACS tubes | BD | 352063 | |
| Low Endotoxin, Azide-Free (LEAF) | Biolegend | 101321 | |
| Alexa Fluor 647 Goat Anti-Mouse IgG (H+L) | Life Technologies | A11008 | |
| Anti-4C4 | Courtesy of Catherina Becker (University of Edinburgh) | ||
| FACS sorter FACSAria II | BD | QMRI, FACS facility | |
| RNeasy Plus Micro Kit | QIAGEN | 74034 | |
| β-mercaptoethanol | Gibco | 31350-010 | |
| QIAshredder | QIAGEN | 79654 | |
| SsoAdvanced Universal SYBR Green Supermix | Bio-Rad | 1725271 | |
| SuperScript III First-Strand Synthesis System | Invitrogen | 18080-051 | |
| LightCycler 96 Real-Time PCR System | Roche | ||
| Ovation RNA-Seq System V2 | NuGEN | 7102-32 | |
| Agilent RNA 6000 Pico reagents | Agilent | 5067-1513 | |
| 2100 Bioanalyzer | Agilent | ||
| RNaseZap | Ambion | AM9780 |
Referencias
- Chrast, R., Scott, H. S., et al. The Mouse Brain Transcriptome by SAGE: Differences in Gene Expression between P30 Brains of the Partial Trisomy 16 Mouse Model of Down Syndrome (Ts65Dn) and Normals. Genome Research. 10 (12), 2006-2021 (2000).
- Beis, D., Stainier, D. Y. R. In vivo cell biology: following the zebrafish trend. Trends in Cell Biology. 16 (2), 105-112 (2006).
- Shiau, C. E., Kaufman, Z., Meireles, A. M., Talbot, W. S. Differential Requirement for irf8 in Formation of Embryonic and Adult Macrophages in Zebrafish. PLoS ONE. 10 (1), 0117513-0117515 (2015).
- Mazaheri, F., Breus, O., et al. Distinct roles for BAI1 and TIM-4 in the engulfment of dying neurons by microglia. Nature Communications. 5, 1-11 (2014).
- Oosterhof, N., Holtman, I. R., et al. Identification of a conserved and acute neurodegeneration-specific microglial transcriptome in the zebrafish. Glia. 65 (1), 138-149 (2016).
- Hamilton, L., et al. A Zebrafish Live Imaging Model Reveals Differential Responses of Microglia Toward Glioblastoma Cells In Vivo. Zebrafish. , (2016).
- Ellett, F., Pase, L., Hayman, J. W., Andrianopoulos, A., Lieschke, G. J. mpeg1 promoter transgenes direct macrophage-lineage expression in zebrafish. Blood. 117 (4), 49-56 (2011).
- Peri, F., Nüsslein-Volhard, C. Live Imaging of Neuronal Degradation by Microglia Reveals a Role for v0-ATPase a1 in Phagosomal Fusion In Vivo. Cell. 133 (5), 916-927 (2008).
- Sieger, D., Moritz, C., Ziegenhals, T., Prykhozhij, S., Peri, F. Long-range Ca2+ waves transmit brain-damage signals to microglia. Developmental cell. 22 (6), 1138-1148 (2012).
- Becker, T., Becker, C. G. Regenerating descending axons preferentially reroute to the gray matter in the presence of a general macrophage/microglial reaction caudal to a spinal transection in adult zebrafish. The Journal of comparative neurology. 433 (1), 131-147 (2001).
- Ohnmacht, J., Yang, Y., et al. Spinal motor neurons are regenerated after mechanical lesion and genetic ablation in larval zebrafish. Development. 143 (9), Cambridge, England. 1464-1474 (2016).
- Roy-Carson, S., Natukunda, K., et al. Defining the transcriptomic landscape of the developing enteric nervous system and its cellular environment. BMC Genomics. 18 (1), 1-24 (2017).
- Khuansuwan, S., Gamse, J. T. Identification of differentially expressed genes during development of the zebrafish pineal complex using RNA sequencing. Developmental biology. 395 (1), 144-153 (2014).
- Schmid, C. D., Melchior, B., et al. Differential gene expression in LPS/IFNγ activated microglia and macrophages: in vitroversus in vivo. Journal of Neurochemistry. 109, 117-125 (2009).
- Bohlen, C. J., Bennett, F. C., Tucker, A. F., Collins, H. Y., Mulinyawe, S. B., Barres, B. A. Diverse Requirements for Microglial Survival, Specification, and Function Revealed by Defined- Medium Cultures. Neuron. 94 (4), 759-773 (2017).
- Khan, A., Ju, F., et al. Transcriptomic analysis reveals differential activation of microglial genes after ischemic stroke in mice. Neuroscience. 348, 212-227 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados