Method Article
Isolamento e l'estrazione di RNA dei neuroni, macrofagi e Microglia da cervelli di Zebrafish larvale
In questo articolo
Riepilogo
Vi presentiamo un protocollo per isolare i neuroni, i macrofagi e microglia da cervelli di zebrafish larvale in condizioni fisiologiche e patologiche. All'isolamento, RNA è Estratto da queste cellule per analizzare il loro profilo di espressione genica. Questo protocollo consente la raccolta di RNA di alta qualità per l'esecuzione dell'analisi a valle come qPCR e trascrittomica.
Abstract
Per ottenere una comprensione dettagliata del ruolo delle diverse cellule di CNS durante lo sviluppo o l'istituzione e la progressione delle patologie del cervello, è importante isolare queste cellule senza modificare il loro profilo di espressione genica. Il modello di zebrafish fornisce un gran numero di linee di pesci transgenici in cui sono etichettati tipi cellulari specifici; ad esempio i neuroni nella riga di NBT:DsRed o macrofagi/microglia nella linea mpeg1:eGFP. Inoltre, gli anticorpi sono stati sviluppati per macchiare le cellule specifiche, come la microglia con 4 4 anticorpo.
Qui, descriviamo l'isolamento dei neuroni, macrofagi e microglia da cervelli di zebrafish larvale. Centrale di questo protocollo è l'evitare una digestione enzimatica del tessuto a 37 ° C, che potrebbe modificare i profili di cellulari. Viene invece utilizzato un sistema meccanico di omogeneizzazione del tessuto a 4 ° C. Questo protocollo comporta omogeneizzazione dei cervelli in sospensione cellulare, loro immunocolorazione e l'isolamento dei neuroni, i macrofagi e microglia di FACS. In seguito, abbiamo estratto il RNA da quelle cellule ed abbiamo valutato la loro qualità/quantità. Siamo riusciti a ottenere RNA di alta qualità (RNA integrità numero (RIN) > 7) eseguire qPCR analisi trascrittomica e neuroni e microglia/macrofagi, microglia. Questo approccio consente una migliore caratterizzazione di queste cellule, come pure una più chiara comprensione circa il loro ruolo nello sviluppo e patologie.
Introduzione
Conoscenza per lo sviluppo del cervello e malattie cerebrali ha migliorato significativamente nell'ultimo decennio dal prima quantificazione del cervello del mouse trascrittomi1. Infatti, analisi dell'espressione genica vasta genoma ci dà accesso a dettagliate informazioni genetiche sul tessuto cerebrale e le cellule che possono integrare e migliorare le osservazioni fatte con altre tecniche e strumenti.
Zebrafish è un modello biologico potente, facile da allevare e di modificare geneticamente; la relativa trasparenza ottica a stadi larvali permette live imaging osservazioni2. Purtroppo, rispetto a umano e mouse, il numero di anticorpi disponibili per eseguire immunocolorazione è piuttosto basso. Per risolvere questo problema, linee pesce zebrafish transgenici sono facilmente realizzati modificando geneticamente i pesci per esprimere le proteine fluorescenti sotto promotori specifici di tipo cellulare. Linee di zebrafish transgenici sono stati utilizzati in passato per studiare il ruolo dei macrofagi e microglia durante lo sviluppo del sistema nervoso centrale (SNC) e malattia3,4,5,6. Tuttavia, per ottenere una comprensione dettagliata di questi processi abbiamo bisogno di capire i cambiamenti nell'espressione genica nei tipi rispettivi cellulari. A questo scopo, abbiamo sviluppato un metodo sperimentale per isolare in modo specifico le cellule come i neuroni, i macrofagi e microglia da 3 a 8 giorni cervelli larvale zebrafish post-fertilizzazione (dpf). Per l'istituzione del protocollo, abbiamo lavorato con linee di pesci transgenici che esprimono la proteina fluorescente verde (GFP) in macrofagi/microglia sotto il promotore del gene del macrofago-espresso (mpeg1:eGFP) e DsRed in neuroni sotto il ß-tubulina neurale promotore (NBT:DsRed)7,8,9. Inoltre, abbiamo effettuato immunocolorazione della microglia utilizzando 4 4, un anticorpo monoclonale di topo che specificamente le macchie zebrafish microglia10,11. In seguito, l'acido ribonucleico (RNA) è estratta da queste cellule per ulteriore reazione a catena della polimerasi quantitativa (qPCR) o analisi del trascrittoma. Questo protocollo è stato progettato per omogeneizzare in modo efficiente il tessuto cerebrale da larve di zebrafish; raccogliere i neuroni, microglia/macrofagi e microglia senza alterazione della loro integrità della membrana plasmatica e infine estrarre RNA da queste cellule in alta qualità (RIN > 7) e la quantità di eseguire analisi genomica. A differenza di studi precedentemente pubblicati che utilizzano il trattamento di tripsina a 37 ° C per digerire cervello tessuto12,13, questo protocollo promuove il lavoro a 4 ° C fino alla fase di estrazione RNA per ridurre le modifiche del profilo di espressione genica. Questo passaggio è cruciale come microglia e macrofagi sono cellule altamente sensibili che rispondono ai cambiamenti nella loro microambiente immediatamente alterando il loro gene expression profile e polarizzazione14,15, 16.
Il protocollo qui descritto in dettaglio, spettacoli che l'isolamento dei neuroni, macrofagi e microglia da zebrafish larvale cervelli, ma virtualmente, può essere adattato a qualsiasi altra cella presentano all'interno del cervello - tramite linee di pesci transgenici o etichettati con anticorpi specifici. Questo metodo permetterà una migliore caratterizzazione delle cellule di CNS attraverso loro analisi dell'espressione genica vasta genoma e aiuterà a comprendere il loro ruolo durante lo sviluppo e malattie del cervello.
Protocollo
1. campione e preparazione di terreni
- Preparare il supporto di embrione (E3) sciogliendo 6,4 mM KCl, 0,22 mM NaCl, 0,33 mM CaCl2 2 H2O, 0,33 mM MgSO4 7 H2O H2O.
- Raccogliere gli embrioni di zebrafish immediatamente dopo la fecondazione (0 dpf).
- Dividere gli embrioni di zebrafish in 50 a capsula di petri di 90 mm. Sollevare gli embrioni a 28,5 ° C in 50 mL di mezzo di embrione (E3) trattati con 200 µM 1-fenil 2-tiourea (PTU), dalla fine del primo giorno di sviluppo (0 dpf) per tutta la durata dell'esperimento per inibire la pigmentazione.
- Cambia l'E3 + PTU medio giornaliero per tutta la durata dell'esperimento.
- Schermo mpeg1:eGFP e larve di NBT:DsRed al dpf 2 utilizzando uno stereomicroscopio fluorescente per l'espressione del transgene positivo, macrofagi GFP+ /microglia e DsRed+ i neuroni.
- Preparare tutti i media il giorno prima dell'esperimento sotto una cappa di coltura del tessuto per evitare la contaminazione, quindi conservarli a 4 ° C.
- Preparare i supporti A sciogliendo 15 mM Hepes e 25 mM D-glucosio in HBSS 1 x.
- Preparare mezzo gradiente di densità (100%) con 9 volumi del mezzo gradiente di densità in 1 volume di HBSS 10 x.
- Preparare il supporto di gradiente di densità (22%) diluendo 22 mL di terreno gradiente di densità (100%) in 88 mL di DPBS 1 x.
- Preparare DPBS 1 x.
- Preparare il medium E3 + tricaina mescolando E3 media con 450 μM tricaina.
2. omogeneizzazione
Nota: Tutti i passaggi sono eseguiti 4 ° C.
- Aggiungere 1,5 mL tricaina (15 mM) contenente 50 larve in 50 mL di mezzo di embrione E3 trattati con 200 µM PTU per anestetizzare terminalmente li di Petri di 90 mm.
- Aspirare 10 larve anestetizzate dalle capsule di Petri con una pipetta Pasteur massa plastica da 3 mL.
- Trasferimento anestetizzato larve 10 per 10 in una capsula di Petri 55mm riempito con gelida E3 embrione medium + tricaina.
- Sotto un microscopio stereoscopico, allineano 10 larve al centro del piatto petri. Quindi transetto larvale teste sopra il vitellino utilizzando micro-forbici chirurgiche (escludere la vescica natatoria per evitare teste di galleggiamento).
- Aspirare con teste dalle capsule di Petri con una pipetta Pasteur massa plastica da 3 mL. Attendere fino a quando tutte le teste si riuniscono all'interno la punta della pipetta e poi trasferiscono in un omogeneizzatore di vetro contenenti 1 mL ghiacciata Media A (trasferimento in un volume minimo di ridurre Media una diluizione in E3 + tricaina). Mantenere l'omogeneizzatore di vetro sul ghiaccio. Utilizzare un omogeneizzatore a condizione sperimentale.
- Sostituire ogni piccolo piatto di petri contenente ghiaccio freddo E3 + tricaina con uno nuovo ogni 30 min per assicurare che il transection viene eseguita a freddo E3 + tricaina medio.
- Sostituire la gelida A Media nell'omogeneizzatore di vetro quando il colore inizia dissolvenza.
Nota: Diluizione A media può alterare il tessuto testa a causa di sbalzi di temperatura. - Una volta che tutte le teste sono stati raccolti (600 capi/condizione), togliere il volume massimo di supporto all'omogeneizzatore di vetro e sostituirlo con 1 mL della ghiacciata a Media fresco.
- Distruggere il tessuto di cervello con un omogeneizzatore di vetro stretto sul ghiaccio. Eseguire 40 giri di frantumazione e giri per 3-5 dpf larve e 50 per le larve di dpf 7 e 8.
- Aggiungere 2 mL di supporto A sospensione cellulare (1 mL di supporto A / 200 capi), che in grado di diluire le cellule e ridurre la loro agglomerazione con mielina per facilitare la loro separazione durante la centrifugazione in gradiente medio densità.
- Per eliminare agglomerazione di cella, è possibile eseguire la sospensione cellulare attraverso un colino di cella di 40 µm posizionato sopra un tubo falcon freddo 50ml mantenuto sul ghiaccio. Ripetere questa operazione 3 volte.
- Trasferire 1 mL di sospensione cellulare in provette da 1,5 mL freddo e li spin a 300 g per 10 min a 4 ° C.
- Rimuovere il surnatante usando una siringa da 10 mL + ago 23 x 1 '.
- Risospendere il pellet cellulare con 1 mL di 22% densità ghiacciata terreno sfumatura delicatamente overlaid da 0,5 mL di DPBS ghiacciata 1 x (do non mix loro, un'interfase tra entrambe le soluzioni saranno visibili).
- Girare le provette a 950 g senza freno e lenta accelerazione per 30 min a 4 ° C.
Nota: Questo passaggio separa mielina da altre cellule sequestrando esso all'interfase di DPBS 1 x e 22% densità gradiente medio, considerando che le cellule saranno a pellet nella parte inferiore del tubo. Rimozione di mielina è più efficiente quando la concentrazione delle cellule non è troppo alto. - Usando una siringa da 10 mL + ago 23 x 1 ' scartare il massimo di DPBS, densità gradiente medio e mielina intrappolato al loro interfase.
- Lavare le cellule con 0,5 mL di mezzi A + 2% di siero di capra normale (NGS), poi fai girare tubi a 300 g per 10 min a 4 ° C.
- Scartare il massimo del surnatante, poi piscina tutte le palline delle cellule dalla stessa condizione sperimentale insieme in 1 mL di Media A + 2% NGS.
- Girare le provette a 950 g senza freno e lenta accelerazione per 30 min a 4 ° C.
- Se le cellule di interesse esprimono una proteina fluorescente come macrofagi/microglia da mpeg1:eGFP o neuroni dai pesci transgenici NBT:DsRed (proiezione di pesci transgenici Vedi 1.1.3), eseguire la sospensione cellulare attraverso un colino di cella 35 µm di cap e trasferirli in un tubi freddi 5ml FACS su ghiaccio, al riparo dalla luce.
Nota: In alternativa, può essere eseguita la immunocolorazione della microglia.
3. Microglia Immuno-colorazione
Nota: Tutti i passaggi sono eseguiti a 4 ° C.
- Risospendere il pellet cellulare con 0,3 mL di Media A + 2% NGS. Dividerli in tubi 3 x 1,5 mL: uno per le celle non colorate su misura auto-fluorescenza da celle di interesse, in secondo luogo per l'anticorpo secondario (1/200) misurare il legame non specifico dell'anticorpo secondario di microglia e terzo come test (4 4 mouse monoclonale anticorpo (microglia specifica) (1/20) + anticorpo secondario (1/200)).
- Aggiungere basso endotossina, privo di Azide (foglia) all'1% delle cellule (tutti i tubi) per bloccare CD16/CD32 interazioni con il dominio Fc delle immunoglobuline. Incubare le cellule per 10 min con movimentazione delicata ogni 5 min.
- Aggiungere i 4 4 anticorpo (1/20) alle cellule (tubo 3) ed incubare per 30 min con agitazione delicata ogni 10 min.
- Girare i tubi a 300 g per 10 min a 4 ° C, quindi scartare il surnatante.
- Lavare una volta con 0,5 mL di mezzi A + 2% NGS, poi girare le provette a 300 g per 10 min a 4 ° C.
- Pellet e risospendere con 0,5 ml di Media A + 2% NGS e incubare le cellule con foglia all'1% per 10 minuti con agitazione delicata ogni 5 min.
- Aggiungere anticorpo secondario (1/200) alle celle (filmato 2 e 3). Incubare le cellule per 30 min con agitazione delicata ogni 10 min e protezione dalla luce.
- Gira tubi a 300 g per 10 min a 4 ° C, quindi eliminare il surnatante.
- Lavare due volte con 0,5 mL di mezzi A + 2% NGS, poi pellet e risospendere con 1 mL di Media A + 2% NGS.
- Eseguire la sospensione cellulare attraverso un tappo di colino di 35 µm cellulare e trasferirli in provetta freddo 5 mL FACS su ghiaccio, al riparo dalla luce.
4. cell Sorting (FACS)
Nota: Eseguire tutti i passaggi a 4 ° C.
- Ordinare i neuroni, microglia/macrofagi e microglia utilizzando un FACS.
Nota: Questa operazione viene solitamente eseguita da un membro del personale della struttura FACS e impostazioni dipendono dal tipo di attrezzatura utilizzata.- Aggiungere DAPI ad una concentrazione di 1 µ g/mL in ciascun tubo di FACS per etichettare le cellule morte.
- Impostare FACS e sorta di neuroni, microglia/macrofagi o microglia da tutte le cellule del cervello. Celle separate dai detriti in funzione della loro dimensione e granularità, quindi cancello singolo-cellule di forward scatter e dispersione laterale. Escludere le cellule morte di DAPI etichettatura da cellule vive. Identificare i neuroni, microglia/macrofagi o microglia dalla loro rispettiva macchiatura positiva.
- Raccogliere le cellule in provette da 1,5 mL contenente 1 mL di gelida Media A + 2% NGS su ghiaccio. Utilizzare tubi diversi per ogni tipo di cellula.
- Gira tubi a 300 g per 10 min a 4 ° C e quindi scartare il surnatante.
- Lavare una volta con 0,5 mL di Media un allora scartare il massimo del surnatante.
5. estrazione del RNA
- Estrarre RNA dai tipi differenti delle cellule separati da FACS. Estrazione del RNA utilizzare un kit specifico e attenersi alle indicazioni del produttore. Garantire per lavorare in un ambiente privo di RNAsi di pulizia dell'area di lavoro e le pipette con un prodotto di decontaminazione di RNAsi e utilizzare puntali con filtro.
- Appena preparare 1 mL di tampone di lisi completati con 50 µM β-mercaptoetanolo.
Nota: Qui, β-mercaptoetanolo è aggiunto per ridurre la degradazione del RNA. - Preparare la soluzione di etanolo 70% e 80% utilizzando RNasi acqua gratuita.
- Lisare cellule con 75 µ l di tampone di lisi completati con 50 µM β-mercaptoetanolo. Quindi utilizzare un trituratore per migliorare la distruzione cellulare.
- Continuare manuale dell'estrazione di RNA secondo produttore.
- Alla fine del protocollo, eluire il RNA con 14 µ l di acqua libera RNasi per ottenere una sufficiente concentrazione di RNA.
- Appena preparare 1 mL di tampone di lisi completati con 50 µM β-mercaptoetanolo.
Risultati
Il protocollo descritto è un approccio diretto per isolare i neuroni, macrofagi e microglia dai cervelli larvale zebrafish. Da questi isolato le cellule, una quantità significativa di alta qualità (RIN > 7) RNA sono stati estratti. L'obiettivo di questo protocollo è quello di isolare i diversi tipi di cellule dallo SNC, con modifiche minime del loro profilo di espressione genica per analizzare e caratterizzare funzioni e proprietà delle celle. Pertanto, l'intero protocollo viene eseguita a 4 ° C con un'omogeneizzazione del tessuto di cervello meccanico. Questo metodo è stato utilizzato con successo per due studi condotti in laboratorio. Nel primo studio, i neuroni e microglia/macrofagi sono stati isolati da 8 dpf mpeg1:eGFP+/NBT:DsRed+ larve (Figura 1). FACS ha permesso la separazione delle cellule dai detriti in funzione della loro dimensione (FSC-A) e granularità (SSC-A) (Figura 1A). Singole cellule poi sono state separate da doppietti o agglomerati di cellule (Figura 1B). Della popolazione di singola cellula, un cancello è stato disegnato per eliminare le cellule morte (DAPI+). La trama di dot corrispondente ha rivelato che questo protocollo sperimentale preserva l'integrità della membrana plasmatica delle cellule, come il tasso di cellule morte è solo il 26,7% (Figura 1C). Infine, i neuroni (DsRed+) e macrofagi/microglia (GFP+) è stati segregati facilmente dalle porte della popolazione di cellule vive. La popolazione di neurone (23,1%) è sembrato essere più prominente rispetto alla popolazione di microglia/macrofagi (1,56%) all'interno del cervello (Figura 1D). Questo protocollo ha permesso di isolare RNA da quelle cellule per eseguire analisi successive qPCR per confrontare l'espressione di specifici geni tra neuroni e microglia/macrofagi. Figura 2 Mostra un neurone e livelli di espressione genica di microglia/macrofagi dell' antigene nucleare delle cellule di proliferazione (pcna)contro il gene di mantenimento della casa β-actina come esempio.
Per il secondo studio questo metodo focalizzata sulla microglia isolamento da 3, 5 e 7 dpf larvale brains. In contrasto con l'esperimento descritto in precedenza, le cellule sono state isolate mediante immuno-colorazione utilizzando 4 4, un anticorpo che specificamente etichette microglia (Figura 3 A-D). Come precedentemente descritto, microglia (4 4+) sono stati selezionati da cellule vive e raccolti (Figura 3D). Microglia numeri all'interno di zebrafish larvale cervelli sono variabili (tabella 1) e molto basso alle 3 dpf (∼ 25 al pesce). Qualità e quantità di RNA Estratto da quelle cellule sono stati misurati utilizzando un sistema di elettroforesi di micro-capillare basato. Risultati ottenuti di RNA Estratto da microglia di 5 dpf larvale zebrafish cervelli sono stati forniti per illustrare un esempio di analisi di RNA (tabella 1 (dpf 5; esperimento 4)). La figura 4 Mostra la traccia di elettroforesi e sua rappresentazione grafica ottenuta per questo esempio con una chiara visualizzazione di RNA ribosomiale (28s e 18s). Questi dati sono necessari per calcolare il campione RIN e per determinare la concentrazione di RNA. La tabella 1 riassume il numero di microglia isolato al pesce, la quantità di RNA per microglia e il Punteggio di RIN ottenuto per ogni esperimento diverso al dpf 3, 5 e 7. La quantità e la qualità del RNA Estratto da microglia isolato utilizzando questo metodo ci ha permesso di amplificare il RNA in cDNA usando un kit. Test di qualità e quantità fornite da Edimburgo genomica confermano che il cDNA amplificato è di qualità sufficiente per la preparazione di biblioteca e successivo sequenziamento. La figura 5 Mostra la distribuzione delle dimensioni dei frammenti di cDNA e loro quantità misurata utilizzando un sistema elettroforetico. In questo esempio, il cDNA aveva una dimensione media di un 299bp ad una concentrazione di 36100 pmol/l. la tabella 2 illustra rispettivamente test di qualità e quantità effettuata il cDNA amplificato da campioni di RNA (tabella 1 (dpf 5; esperimento 4)). Il cDNA amplificato è stato utilizzato con successo per il sequenziamento.
Numerosi studi condotti in laboratorio hanno confermato che la qualità e la quantità di RNA Estratto da neuroni, macrofagi e microglia può essere utilizzati per successive qPCR e analisi dell'espressione genica vasta genoma. Di conseguenza, questo protocollo sperimentale consente di isolare in modo affidabile diversi tipi di cellule CNS senza alterare l'integrità della membrana e limitando la modifica del loro profilo di espressione genica.

Figura 1 : FACS ordinamento per i neuroni e macrofagi/microglia da /NBT:DsRed mpeg1:GFP++ 8 larve di zebrafish dpf. (A-C) Gating successivi Mostra selezione sequenziale di tutte le cellule di cervello (A), disperdono di singole cellule di forward scatter e lato (B). Cellule morte (C) sono stati esclusi dall'etichettatura DAPI. (D) neuroni e microglia/macrofagi sono stati identificati rispettivamente dalla macchiatura positiva DsRed e GFP. Clicca qui per visualizzare una versione più grande di questa figura.
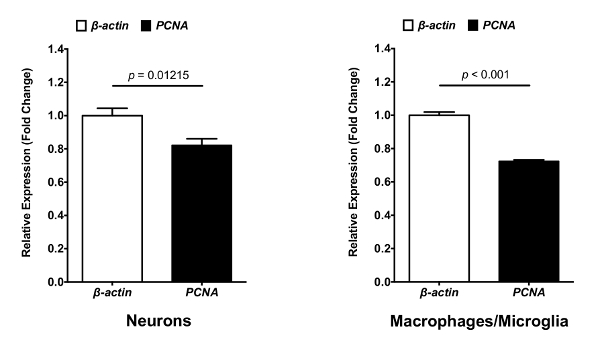
Figura 2 : Analisi di espressione genica per pcna e β-actina nei neuroni e microglia/macrofagi. RNA da isolato neuroni e microglia/macrofagi può essere trascritto in cDNA per uso nell'analisi quantitativa di PCR. livelli di espressione di mRNA di pcna contro gene house-keeping β-actina in neuroni isolati e macrofagi/microglia determinato da qPCR (N = 3). Cambiamento di piega è stato misurato usando il metodo comparativo (ΔΔCT). Errore bar rappresentano media ± SEM. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3 : FACS ordinamento per microglia da 3 larve di zebrafish dpf. (A-C) Successivi gating Visualizza selezione sequenziale del cervello tutte le cellule (A), disperdono di singole cellule di forward scatter e lato (B). Cellule morte (C) sono stati esclusi dall'etichettatura DAPI. (D) Microglia sono stati identificati da 4 4 macchiatura positiva. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4 : Risultati di elettroforesi di micro-capillare di RNA Estratto da 5 dpf zebrafish microglia. Le due alte cime sono il RNA ribosomiale 18S e 28S. Numero di RNA integrità (RIN) è stato calcolato automaticamente dal software bioanalyzer utilizzando il rapporto generato il 18S e 28S subunità ribosomali e l'analisi di tutto il tracciato elettroforetico. Microglia RNA ha un 8.6 RIN. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5 : Test di qualità e quantità di cDNA amplificato dal campione di RNA di 5 dpf zebrafish microglia. L'immagine mostra la distribuzione di dimensione del frammento del cDNA del campione analizzato con una dimensione media di 299 BP. Clicca qui per visualizzare una versione più grande di questa figura.
| Condizione | Esperimento | Numero di pesci | Numero di cellulare | Numero di cellulare per pesce | Concentrazione di RNA (pg/ul) | RNA totale (pg) | Quantità di RNA per cella (pg) | Punteggio di RIN |
| 3dpf | 1 | 700 | 11922 | 17.03 | 126 | 1512 | 0.13 | 8 |
| 3dpf | 2 | 600 | 22527 | 37,55 | 253 | 3036 | 0.13 | 7.9 |
| 3dpf | 3 | 600 | 18688 | 31,15 | 255 | 3060 | 0.16 | 7.7 |
| 3dpf | 4 | 600 | 11121 | 18,54 | 189 | 2268 | 0.20 | 7,8 |
| 3dpf | 5 | 600 | 15581 | 25,97 | 131 | 1572 | 0.10 | 8.4 |
| 3dpf | 6 | 600 | 11965 | 19,94 | 256 | 3072 | 0,26 | 8.2 |
| 5dpf | 1 | 600 | 58629 | 97.72 | 362 | 4344 | 0.07 | 7.4 |
| 5dpf | 2 | 600 | 32510 | 54,18 | 348 | 4176 | 0.13 | 8.1 |
| 5dpf | 3 | 600 | 77884 | 129.81 | 594 | 7128 | 0,09 | 8.3 |
| 5dpf | 4 | 600 | 50755 | 84,59 | 305 | 3660 | 0.07 | 8.6 |
| 5dpf | 5 | 600 | 44967 | 74,95 | 134 | 1608 | 0,04 | 7.6 |
| 5dpf | 6 | 600 | 51031 | 85,05 | 163 | 1956 | 0,04 | 7.9 |
| 7dpf | 1 | 600 | 60496 | 100.83 | 183 | 2196 | 0,04 | 7.6 |
| 7dpf | 2 | 450 | 55517 | 123.37 | 183 | 2196 | 0,04 | 7,8 |
| 7dpf | 3 | 600 | 88897 | 148.16 | 465 | 5580 | 0,06 | 8.1 |
| 7dpf | 4 | 600 | 63008 | 105.01 | 356 | 4272 | 0.07 | 8.4 |
| 7dpf | 5 | 350 | 34956 | 99,87 | 245 | 2940 | 0.08 | 8.1 |
| 7dpf | 6 | 600 | 63887 | 106.48 | 341 | 4092 | 0,06 | 7,8 |
Tabella 1: Sintesi delle microglia isolamento e dati di estrazione di RNA da larve di zebrafish dpf 3, 5 e 7.
| ID campione interno | ID campione esterno | Qubit (ng/ul) | Qubit(ng/UL) | Media concentrazione (ng/ul) | Volume (ul) | UG ha ricevuto | Pass/fail per concentrazione minima | Pass/fail per quantitativo minimo | Pass/fail per quantità consigliata |
| 10907SD0010 | 5 dpf (esperimento 4) | 58,8 | 58,4 | 58,6 | 30 | 1.76 | Passare | Passare | Passare |
| Requisiti del campione per libreria Prep: | |||||||||
| Biblioteca Prep | Quantità minima (ng) | Quantità consigliata (ng) | Minima concentrazione ng/uL | ||||||
| TruSeq Nano libreria gratuita del inserto bp 350 da cDNA del gel | 600 | 1100 | 10 | ||||||
Tabella 2: Test quantità di cDNA amplificato dal campione di RNA di 5 dpf zebrafish microglia.
Discussione
Il protocollo sperimentale descritto qui rappresenta un metodo affidabile ed efficiente per isolare le cellule cerebrali da larve di zebrafish da 3 a 8 dpf. D'importanza, questo è il primo protocollo che permette l'isolamento specifico di microglia da cervelli di zebrafish larvale. Il protocollo è progettato per preservare l'integrità della membrana delle cellule e ridurre al minimo le potenziali modifiche di espressione genica che si verificano durante l'elaborazione. Quest'ultimo punto è fondamentale per la rilevanza dei risultati sulla base dell'analisi di tali profili genomici cellulari isolati. Infatti, la polarizzazione di microglia e macrofagi sono fortemente influenzati dalla loro microambiente. A 37 ° C, queste cellule avrebbero cambiato il loro profilo di espressione genica in risposta a condizioni sperimentali (risposta di lesione (transection)). Pertanto, è stato cruciale eseguire questo esperimento a 4 ° C prima dell'estrazione di RNA, di rallentare i processi cellulari e attività metaboliche. Inoltre, l'omogeneizzazione del tessuto di cervello meccanico a 4 ° C è stato scelto invece di digestione enzimatica del tessuto a 37 ° C per evitare qualsiasi impatto su profili di espressione genica.
È importante sottolineare che questo metodo è molto veloce; entro un giorno diverse popolazioni di cellule del cervello possono essere isolate da almeno due diverse condizioni sperimentali e loro RNA estratto. La lunghezza totale del protocollo dipende dal numero delle larve utilizzato per ogni condizione come il transection del teste larvale è il passaggio limitante (∼ 350 capi/h). In generale, per lavorare con microglia da 3, 5 e 7 dpf è consigliabile transetto teste di ∼ 600 per ogni test per ottenere abbastanza RNA per estrarre (tabella 1). Come microglia rappresentano il tipo di cella con il più basso rendimento (circa 112 cellule pro capite alle 7 dpf), il numero di teste può essere ridotta per altri tipi di cellule tra cui i macrofagi (circa 170 cellule pro capite al dpf 7).
Un altro vantaggio di questo protocollo è che dopo aver definite le impostazioni ordinatore FACS, le stesse impostazioni possono essere utilizzate per diversi esperimenti. È stato osservato che le popolazioni delle cellule perfettamente da un esperimento a altro con cancelli precedentemente progettati, mostrando la riproducibilità degli esperimenti utilizzando questo metodo.
Un leggero svantaggio di questo metodo è la quantità relativamente bassa di RNA che viene raccolto. Tuttavia, questa limitazione è più un problema biologico di un problema tecnico, come il numero di microglia è molto basso nelle fasi iniziali di sviluppo del cervello (tabella 1). A causa di questa bassa quantità di RNA raccolto, amplificazione passaggi devono essere considerati per eseguire analisi di espressione genica vasta di genoma. Fortunatamente, questi passaggi di amplificazione producono una quantità sufficiente di cDNA di alta qualità. Così, cambiamenti globali in profili di espressione genica delle cellule isolate possono essere studiati.
In conclusione, questo protocollo fornisce un metodo affidabile per isolare e studiare vari tipi di cellule di CNS dai cervelli di zebrafish larvale. Questo può essere applicato per ottenere una più profonda comprensione di queste cellule durante lo sviluppo anche per studiare il loro ruolo nella malattia.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo il Dr. Claire Davis e Dr. Veronique Miron (The Queen Medical Research Institute, Edinburgh, Regno Kingdome) per aiuto iniziale e discussioni sull'approccio sperimentale e QMRI citometria a flusso e Cell Sorting Facility. Questo lavoro è stato supportato da un Cancer Research UK carriera stabilimento Award al Dr. Dirk Sieger.
Materiali
| Name | Company | Catalog Number | Comments |
| 1-phenyl 2-thiourea (PTU) | Sigma | P7629 | |
| Hepes | Gibco | 15630-056 | |
| D-Glucose | Sigma | G8644-100ML | |
| HBSS 1X | Gibco | 14170-088 | |
| Percoll | GE Healthcare | 17-0891-02 | |
| HBSS 10X | Gibco | 14180-046 | |
| DPBS 1X | Gibco | 14190-094 | |
| Tricaine (MS222) | Sigma | A5040 | |
| Sterilin standard 90mm petri dishes | ThermoFisher | 101VIRR | |
| Surgical micro-scissors | Fine Science Tools | 15000-00 | |
| 3 mL Pasteur plastic bulk pipette | SLS | PIP4206 | |
| Glass homogenizer | Wheaton | 357538 | |
| Sterilin standard 55mm petri dishes | ThermoFisher | P55V | |
| Percoll | GE Healthcare | 17-0891-02 | |
| 40 µm cell strainer | Falcon | 352340 | |
| 50 ml polypropylen conical tube | Falcon | 352070 | |
| Centrifuge | Eppendorf | 5804 R | |
| 10 mL syringe | BD | 302188 | |
| Needle 23G x 1'' | BD | 300800 | |
| Normal goat serum (NGS) | Cell Signalling | 5425S | |
| 35 μm cell strainer cap | BD | 352235 | |
| FACS tubes | BD | 352063 | |
| Low Endotoxin, Azide-Free (LEAF) | Biolegend | 101321 | |
| Alexa Fluor 647 Goat Anti-Mouse IgG (H+L) | Life Technologies | A11008 | |
| Anti-4C4 | Courtesy of Catherina Becker (University of Edinburgh) | ||
| FACS sorter FACSAria II | BD | QMRI, FACS facility | |
| RNeasy Plus Micro Kit | QIAGEN | 74034 | |
| β-mercaptoethanol | Gibco | 31350-010 | |
| QIAshredder | QIAGEN | 79654 | |
| SsoAdvanced Universal SYBR Green Supermix | Bio-Rad | 1725271 | |
| SuperScript III First-Strand Synthesis System | Invitrogen | 18080-051 | |
| LightCycler 96 Real-Time PCR System | Roche | ||
| Ovation RNA-Seq System V2 | NuGEN | 7102-32 | |
| Agilent RNA 6000 Pico reagents | Agilent | 5067-1513 | |
| 2100 Bioanalyzer | Agilent | ||
| RNaseZap | Ambion | AM9780 |
Riferimenti
- Chrast, R., Scott, H. S., et al. The Mouse Brain Transcriptome by SAGE: Differences in Gene Expression between P30 Brains of the Partial Trisomy 16 Mouse Model of Down Syndrome (Ts65Dn) and Normals. Genome Research. 10 (12), 2006-2021 (2000).
- Beis, D., Stainier, D. Y. R. In vivo cell biology: following the zebrafish trend. Trends in Cell Biology. 16 (2), 105-112 (2006).
- Shiau, C. E., Kaufman, Z., Meireles, A. M., Talbot, W. S. Differential Requirement for irf8 in Formation of Embryonic and Adult Macrophages in Zebrafish. PLoS ONE. 10 (1), 0117513-0117515 (2015).
- Mazaheri, F., Breus, O., et al. Distinct roles for BAI1 and TIM-4 in the engulfment of dying neurons by microglia. Nature Communications. 5, 1-11 (2014).
- Oosterhof, N., Holtman, I. R., et al. Identification of a conserved and acute neurodegeneration-specific microglial transcriptome in the zebrafish. Glia. 65 (1), 138-149 (2016).
- Hamilton, L., et al. A Zebrafish Live Imaging Model Reveals Differential Responses of Microglia Toward Glioblastoma Cells In Vivo. Zebrafish. , (2016).
- Ellett, F., Pase, L., Hayman, J. W., Andrianopoulos, A., Lieschke, G. J. mpeg1 promoter transgenes direct macrophage-lineage expression in zebrafish. Blood. 117 (4), 49-56 (2011).
- Peri, F., Nüsslein-Volhard, C. Live Imaging of Neuronal Degradation by Microglia Reveals a Role for v0-ATPase a1 in Phagosomal Fusion In Vivo. Cell. 133 (5), 916-927 (2008).
- Sieger, D., Moritz, C., Ziegenhals, T., Prykhozhij, S., Peri, F. Long-range Ca2+ waves transmit brain-damage signals to microglia. Developmental cell. 22 (6), 1138-1148 (2012).
- Becker, T., Becker, C. G. Regenerating descending axons preferentially reroute to the gray matter in the presence of a general macrophage/microglial reaction caudal to a spinal transection in adult zebrafish. The Journal of comparative neurology. 433 (1), 131-147 (2001).
- Ohnmacht, J., Yang, Y., et al. Spinal motor neurons are regenerated after mechanical lesion and genetic ablation in larval zebrafish. Development. 143 (9), Cambridge, England. 1464-1474 (2016).
- Roy-Carson, S., Natukunda, K., et al. Defining the transcriptomic landscape of the developing enteric nervous system and its cellular environment. BMC Genomics. 18 (1), 1-24 (2017).
- Khuansuwan, S., Gamse, J. T. Identification of differentially expressed genes during development of the zebrafish pineal complex using RNA sequencing. Developmental biology. 395 (1), 144-153 (2014).
- Schmid, C. D., Melchior, B., et al. Differential gene expression in LPS/IFNγ activated microglia and macrophages: in vitroversus in vivo. Journal of Neurochemistry. 109, 117-125 (2009).
- Bohlen, C. J., Bennett, F. C., Tucker, A. F., Collins, H. Y., Mulinyawe, S. B., Barres, B. A. Diverse Requirements for Microglial Survival, Specification, and Function Revealed by Defined- Medium Cultures. Neuron. 94 (4), 759-773 (2017).
- Khan, A., Ju, F., et al. Transcriptomic analysis reveals differential activation of microglial genes after ischemic stroke in mice. Neuroscience. 348, 212-227 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon