Method Article
Usando três cores único-molécula FRET para estudar a correlação de interações da proteína
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para obter dados de três cores de smFRET e sua análise com um ensemble 3D Hidden Markov modelo. Com esta abordagem, cientistas podem extrair informações cinéticas de sistemas complexos proteína, incluindo cooperatividade ou interações correlacionadas.
Resumo
Transferência de energia de ressonância do único-molécula Förster (smFRET) tornou-se uma técnica amplamente utilizada biofísica para estudar a dinâmica de biomoléculas. Para muitas máquinas moleculares em proteínas uma célula tem de agir em conjunto com parceiros de interação em um ciclo funcional a cumprir sua tarefa. A extensão de duas cores para cor multi smFRET possibilita a sonda simultaneamente mais de uma interação ou mudança conformacional. Isto não só acrescenta uma nova dimensão às experiências de smFRET, mas também oferece a possibilidade única de estudar diretamente a sequência de eventos e para detectar interações correlacionadas ao usar uma amostra de imobilizado e uma fluorescência de reflexão interna total microscópio (TIRFM). Portanto, multi cor smFRET é uma ferramenta versátil para estudar biomolecular complexos de forma quantitativa e em um detalhe anteriormente inatingível.
Aqui, demonstramos como superar os desafios especiais de multi-cor smFRET experimentos sobre proteínas. Apresentamos a protocolos detalhados para obtenção dos dados e para extrair as informações cinéticas. Isso inclui os critérios de seleção de rastreamento, separação do estado e a recuperação de trajetórias de estado a partir dos dados ruidosos usando um ensemble 3D Hidden modelo de Markov (HMM). Em comparação com outros métodos, as informações cinéticas não são recuperadas de histogramas de tempo de interrupção mas diretamente do HMM. O quadro de máxima verossimilhança nos permite avaliar criticamente o modelo cinético e prever as taxas significativas incertezas.
Através da aplicação de nosso método para a proteína de choque do calor 90 (Hsp90), somos capazes de separar a ligação de nucleotídeos e as globais mudanças conformacionais da proteína. Isso nos permite observar diretamente a cooperatividade entre os dois bolsos de vinculação do nucleotide do dímero Hsp90.
Introdução
Muitas proteínas cumprem sua função em dinâmicos complexos com outras moléculas, mediadas por mudanças conformacionais e associações transientes em uma ampla gama de escalas temporais1,2,3. Acoplado a uma fonte de energia externa (por exemplo, ATP) essas interações dinâmicas podem levar a direcionalidade de um ciclo funcional e, finalmente, manter o estado de desequilíbrio constante em uma célula, o pré-requisito para a vida.
A fim de compreender plenamente essas máquinas moleculares, uma descrição estática guiada por estudos estruturais não é suficiente. Além disso, é essencial ter conhecimento do modelo cinético subjacente e determinar as constantes de velocidade cinética. Vários métodos existentes permitem aos pesquisadores estudar a dinâmica de interações de binárias entre duas moléculas de interesse, por exemplo, ressonância de plasmon de superfície, métodos de relaxamento com uma leitura espectroscópico (por exemplo, o salto ou fluxo parou técnicas) e ressonância magnética nuclear. No entanto, sua aplicabilidade é na maioria dos casos limitados a simples sistemas de dois Estados (por exemplo, um limite e um estado desacoplado) devido a média de inerente às experiências em massa. Em casos onde a mais Estados ou intermediários estão envolvidos, rendem apenas uma mistura complexa das constantes de velocidade. Único-molécula métodos tais como Pinça óptica ou magnética ou smFRET de duas cores, ou seja, um doador e um aceitador fluoróforo, com uma amostra de superfície-imobilizado podem recuperar as constantes de taxa para todos observadas mudanças conformacionais. No entanto, quando se trata de interações que afetam o sítio de ligação a mais de um, esses métodos permanecem limitados e as informações sobre as interações de possível correlação dos dois (ou mais) só será acessíveis via indirectas conclusões de um conjunto de experiências.
Multi cor smFRET4,5,6,7,8,9 oferece a oportunidade de estudar a interação entre esses componentes directamente, em tempo real e sob condições fisiológicas perto de10. Isso permite que um para investigar, por exemplo, a vinculação dependente da conformação de um ligante ou outra proteína8,9,11. A abordagem global apresentada aqui é para rotular as somáticas de interesse em posições específicas, para anexar uma proteína para a superfície da câmara de medição e para controlar a intensidade da fluorescência ao longo do tempo em um prisma-tipo TIRFM (para detalhes veja 9 , 12). a proximidade espacial das tinturas diferentes então pode ser determinada a partir a transferência de energia entre eles. Estratégias de rotulagem pode variar de proteína a proteína (revista em 13) e orientações para evitar artefatos nas medições smFRET existem14.
Desde que um corante doador pode transferir energia para aceitador diferentes corantes em um experimento smFRET multi cor, a posição relativa de todos os corantes não é acessível a partir da excitação de um corante sozinho15,16. Mas em combinação com a alternância de excitação do laser (ALEX17e revisto em 18) este método fornece todas as informações de espaço-temporal em segundo e resolução de sub nanômetros.
No principal, de alta resolução, informações estruturais podem ser conseguidas usando-se as distâncias de tintura inter calculadas a partir da combinação de todas as intensidades de fluorescência em um smFRET de cor multi experiência com ALEX. No entanto, aqui focamos na identificação de estado e de separação, bem como a extração de modelos cinéticos, onde smFRET multi cor é indispensável. Quando "só" determinação de estrutura por triangulação é desejada, um conjunto de experiências mais simples de duas cores smFRET com alta relação sinal-ruído pode ser realizada de12,19.
Nós usamos a fluorescência parcial ( ) como um proxy para a transferência de energia entre dois fluorophores7. A PF é calculado a partir da intensidade de fluorescência análoga à eficiência de traste de um experimento de duas cores:
) como um proxy para a transferência de energia entre dois fluorophores7. A PF é calculado a partir da intensidade de fluorescência análoga à eficiência de traste de um experimento de duas cores:

Onde,  é a intensidade de emissão canal em após excitação com cor ex, e c é o aceitador com o comprimento de onda mais longo. Canais de deteção representam a mesma posição na câmara de amostra, mas registros diferentes intervalos espectrais da luz da fluorescência. O mesmo identificador para excitação e emissão são utilizados neste protocolo (ou seja, "azul", "verde" e "vermelho").
é a intensidade de emissão canal em após excitação com cor ex, e c é o aceitador com o comprimento de onda mais longo. Canais de deteção representam a mesma posição na câmara de amostra, mas registros diferentes intervalos espectrais da luz da fluorescência. O mesmo identificador para excitação e emissão são utilizados neste protocolo (ou seja, "azul", "verde" e "vermelho").
Por causa de deficiências experimentais as intensidades de fluorescência medido dependem não só sobre a transferência de energia mas também Propriedades fluoróforo e instalação. Para obter a eficiência de transferência de energia verdadeira entre dois fluorophores, as intensidades medidas tem que ser corrigido. O procedimento a seguir baseia-se na referência9. Fatores de correção para vazamento aparente (LC, ou seja, a detecção de fótons de um fluoróforo num canal designada para outro corante) e aparente gama (ag, ou seja, o rendimento quântico de fluorescência do corante e a eficiência de deteção do canal) são obtidos a partir de vestígios de único-molécula que mostram um aceitador evento de branqueamento.
O escapamento do doador corante em todos os canais possíveis aceitador é calculado a partir de todos os pontos de dados nos vestígios de fluorescência gravada onde o corante aceitador branqueada, mas o doador é ainda fluorescente ( ):
):

A mediana do histograma escapamento é usada como o fator de vazamento aparente. Após a correção para detecção de fugas, o fator gama aparente é determinado a partir do mesmo conjunto de traços. É calculado dividindo-se a mudança da fluorescência no canal aceitador pela mudança de fluorescência no canal ao descoramento do corante aceitador de doador:

Onde c é novamente o canal de deteção para o aceitador com o comprimento de onda mais longo. A mediana da distribuição resultante é usada como o fator de correção aparente.
As intensidades corrigidas em cada canal são obtidas por:

A PF é então calculado de acordo com:

Diferentes populações podem ser separadas no espaço multidimensional ocupado pela s da PF. A posição e a largura de cada Estado é determinado pelo ajuste os dados com funções Gaussian multidimensionais. Otimização subsequente de um HMM global com base em todos os vestígios PF fornece uma descrição quantitativa de cinética observada. Mesmo as pequenas alterações das taxas são detectáveis.
HMMs fornecem uma maneira de se inferir um modelo de estado de uma coleção de traços de tempo barulhento. O sistema é considerado em parte de um conjunto de discreta, Estados ocultos em um determinado momento e a observação real (ou seja, a emissão) é uma função probabilística deste estado oculto20. No caso dos dados de smFRET TIRFM, a emissão probabilidades beu por Estado, pode ser modelado por funções de densidade de probabilidade gaussiana contínua. Em pontos regularmente espaçados de tempo discreto, transições de um para outro Estado podem ocorrer de acordo com a probabilidade de transição que é invariante no tempo e só depende do estado atual. A matriz de transição A contém estas probabilidades de transição umij entre todos os Estados-Membros escondidos. A distribuição de estado inicial  dá as probabilidades específicas de estado
dá as probabilidades específicas de estado  para o primeiro ponto de tempo de um rastreamento de tempo. Usando uma abordagem de máxima probabilidade, estes parâmetros podem ser otimizados para melhor descrevem os dados com o para diante-para trás e Baum-Welch algoritmos20,21. Isso produz os estimadores de máxima verossimilhança (MLE). Finalmente, a sequência de estado que provavelmente produzido a trajetória de observações pode ser inferida com o algoritmo de Viterbi. Em contraste com outras analises HMM smFRET dados24,25,26 não usamos o HMM como um mero "suavização" da dados, mas o modelo de estado cinético extraído do conjunto de dados sem a necessidade para caber o tempo de permanência histogramas de27. HMM análise é feita com in-house scripts usando Igor Pro. Aplicação do código baseia-se na referência21. Nós fornecemos um kit de software e dados exemplares na nossa página da Web para seguir seções 5 e 6 do presente protocolo (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). Software completo está disponível mediante pedido.
para o primeiro ponto de tempo de um rastreamento de tempo. Usando uma abordagem de máxima probabilidade, estes parâmetros podem ser otimizados para melhor descrevem os dados com o para diante-para trás e Baum-Welch algoritmos20,21. Isso produz os estimadores de máxima verossimilhança (MLE). Finalmente, a sequência de estado que provavelmente produzido a trajetória de observações pode ser inferida com o algoritmo de Viterbi. Em contraste com outras analises HMM smFRET dados24,25,26 não usamos o HMM como um mero "suavização" da dados, mas o modelo de estado cinético extraído do conjunto de dados sem a necessidade para caber o tempo de permanência histogramas de27. HMM análise é feita com in-house scripts usando Igor Pro. Aplicação do código baseia-se na referência21. Nós fornecemos um kit de software e dados exemplares na nossa página da Web para seguir seções 5 e 6 do presente protocolo (https://www.singlemolecule.uni-freiburg.de/software/3d-fret). Software completo está disponível mediante pedido.
Pontos nos dados com PF < -1 ou PF > 2 em qualquer canal de deteção são atribuídos a probabilidade de emissão mínima para todos os Estados (10-200). Isso impede que transições artificiais nestes pontos de dados.
Os parâmetros para as probabilidades de emissão são obtidos a partir do ajuste do histograma PF 3D com funções Gaussian conforme descrito na etapa 5.7. Esses parâmetros são mantidos fixos durante a otimização do HMM.
Na abordagem apresentada, o vetor de distribuição do estado inicial e a matriz de transição são utilizados mundialmente para descrever todo o conjunto de traços. Eles são atualizados com base em todas as moléculas de N do conjunto de dados de acordo com a referência27.
Parâmetros para a distribuição de estado inicial são determinados de projeções 2D do histograma PF (etapa 5.3) e as probabilidades de transição são definidas como 0,05 com exceção as probabilidades de permanecer no mesmo estado, que são escolhidos tais que a probabilidade de sair de um determinado estado é normalizada à unidade.
Um método de criação de perfil de probabilidade é usado para dar intervalos de confiança (CIs) para todos os transição taxas21,22, que servem como estimativas significativas para sua incerteza. Para calcular os limites da IC para uma taxa específica, a probabilidade de transição de juros é fixa para um valor diferente do MLE. Isto rende o teste modelo λ'. Um teste de ratio (LR) probabilidade da probabilidade  dado o conjunto de dados 0 é realizada de acordo com:
dado o conjunto de dados 0 é realizada de acordo com:

A confiança de 95% vinculada para o parâmetro é alcançado quando LR excede 3,841, o Quantil de 95% de um X2-distribuição com um grau de liberdade22,23.
O poder do método é demonstrado usando o Hsp90. Esta proteína abundante é encontrada em bactérias e eucariotas e é parte do estresse celular resposta28. É um alvo de droga promissora no tratamento de câncer29. Hsp90 é um homodímero com um bolso de ligação de nucleotídeos do domínio N-terminal de cada subunidade30. Ele pode sofrer transições pelo menos duas conformações distintas globalmente, um fechado e um N-terminal conformação aberta, em forma de V19,31,32. A natureza dimérica diretamente a questão da interação entre os dois locais de ligação de nucleotídeos em Hsp90.
A seguir, fornecemos um protocolo passo a passo para a aquisição de dados e análise de um experimento de smFRET três cores na levedura Hsp90 e nucleotídeos. A vinculação dependente da conformação do AMP-PNP fluorescente etiquetadas (AMP-PNP *, um analog não hidrolisável do ATP) é analisada. A aplicação do procedimento descrito permite o estudo da ligação de nucleótidos e ao mesmo tempo as mudanças conformacionais de Hsp90 e, assim, revela a cooperatividade entre os dois bolsos de vinculação de nucleotídeo de Hsp90.
Protocolo
1. instalação e pré-requisitos
- Realizar as medições de cor multi smFRET em um TIRFM de prisma-tipo. Uma descrição de uma configuração de duas cores como uma publicação de JoVE é dado referência a12.
- Construa um TIRFM de várias cor. Um layout geral é detalhado em 9.
- Uso comutável, diodo bombeou onda contínua lasers de estado sólido, que tornam a utilização de obturadores mecânicos nos caminhos de excitação desnecessárias.
- Emprega um prisma assimétrico, alongado que impede a reflexão traseira do feixe da excitação da parte traseira para entrar o objetivo.
- Use lentes de silicone acromática de esféricos fundido de 2 polegadas nos caminhos de deteção que coletar tanta luz quanto possível e evitar autofluorescência e aberrações, por exemplo, as distorções nas regiões fora do centro da imagem.
- Focar cada caminho de deteção do chip do EMCCD com uma lente separada. Isso permite que o foco ideal de cada canal de deteção.
Atenção: Lasers de classe 3B são usados na TIRFM. Isto significa que eles são perigosos, se o olho é exposto diretamente, mas reflexões difusas não são prejudiciais. Garantir a conformidade com laser precauções de segurança, de acordo com as regulamentações governamentais locais antes que o sistema é operado.
- Determine os factores de correcção para propriedades de configuração e fluoróforo previamente usando amostras de dsDNA.
- Usar um alto-FRET dsDNA amostra para cada corante em combinação com o aceitador tendo o mais longo comprimento de onda de excitação (para a configuração apresentada: Atto488-Atto647N, Atto550-Atto647N, Atto594-Atto647N). Certifique-se de que o DNA é modificado adicionalmente com uma biotina.
- Diluir a amostra a 5 nM com tampão TNM (5 mM Tris pH 7,5, 5 mM de NaCl, 20 mM MgCl2) e 2 mM Trolox (use também esse buffer para medição).
- Imobilize o dsDNA conforme descrito para Hsp90 em etapas 2.5 e 2.7.
- Calcule os fatores de correção para vazamento aparente (LC) e aparente gama (ag) de traços de único-molécula que mostram um evento de branqueamento do aceitador.
- Construa uma câmara de fluxo que é um sanduíche de uma lâmina de quartzo passivadas PEG/biotina-PEG, uma película fina que é adesivo em ambos os lados e uma lamínula. Para protocolos de detalhe para a limpeza do quartzo slides e passivação consulte referência9.
- Uso de espessura (3mm) quartzo slides para evitar geometricamente a coleção do laser espalhados na interface entre o prisma e o slide funcionalizados quartzo quartzo-glicerol-quartzo.
- Use uma fina (40 µm) filme que é pulverizado com adesivo no lado não-adesivo de vedação. A película fina reduz a distância entre as moléculas de superfície-anexado e objectivo. Aquecer a 80 ° C e pressione.
- Coloca uma lamínula por cima. Aquecer a 80 ° C e pressione. Use uma gota de glicerol, ao colocar a câmara de fluxo sobre o prisma.
Nota: Os materiais de prisma e os slides de quartzo, bem como o glicerol são índice de correspondência.
- Express Hsp90 de Saccharomyces cerevisiae na forma de dois mutantes de ponto único de cisteína nas posições D61 ou Q385. Adicione um motivo de bobina coiled do C-terminal para evitar a dissociação do dímero em concentrações picomolar. Rotular as proteínas mutantes separadamente e trocar os monómeros para obter heterodímeros rotulados com Atto488 na posição do ácido aminado 61 e Atto550 no ácido aminado posição 3859.
2. medição
- Inicie o software da câmera e definir os parâmetros de imagem conforme especificado abaixo:
- Regule a temperatura para o sensor de resfriamento mais baixo possível (-95 ° C com refrigeração a água externa) para diminuir o ruído de corrente escuro.
- Usar as configurações da câmera que são otimizadas para a gravação do único-molécula: velocidade de deslocamento vertical 3.3 µs, tensão de pulso de disparo vertical normal, 17 horizontal MHz 16 bits lidos, pré-amplificação ganho 3, ganho de elétron multiplicador 1.000.
- Defina o acionamento do alarme a aquisição "Externo" e o tempo de exposição para 70 filmes de registro MS., com um comprimento de 750 ciclos de aquisição.
Nota: Desliga a luz do quarto quando a câmera está adquirindo para evitar a saturação do sensor EMCCD.
- Crie uma pasta no local SSD para a medição. No software configurações vão para o piloto < Auto-Save > habilitar < Auto-save > em escolher o formato de arquivo "Tiff" para aquisição de filme. Selecione a pasta sobre o SSD como local de salvamento automático.
- Inicie o software que controles o acústico-Óptico Sintonizável filtro (AOTF), o software que controla a operação dos lasers e o software de gatilho que sincroniza os lasers, AOTF, persianas no caminho de deteção e câmeras. Ajustar a potência do laser com o AOTF (ca. 3 mW antes de entrar o prisma) e carregar o padrão correto de disparo.
- Montar o porta-amostras com o prisma e a câmara de fluxo, conecte a tubulação, coloque o tubo de entrada em um copo de microcentrifuga e conecte o tubo de saída de uma bomba de seringa. Lave a câmara com cerca de 150 buffer µ l, alinhe a excitação feixe e foco. Bleach todos os contaminadores na superfície fluorescentes por movendo-se lentamente ao longo do intervalo de detecção completa do slide com um poder do laser de cerca de 10 mW para todos os lasers. Isso leva cerca de 1h.
Nota: se não for indicado o contrário, o buffer usado contém 40 mM HEPES pH 7,5, KCl, de 150 mM e 10 mM MgCl2. - Descarga de aproximadamente 300 µ l de uma solução de NeutrAvidin (0,25 mg/mL em buffer) para a câmara e incubar durante 1 min. Flush fora NeutrAvidin desacoplado com tampão e flush aproximadamente 300 ml de uma solução de BSA (0,5 mg/mL em buffer) através da câmara.
Nota: Este bloco restante superfície functionalization defeitos por adsorção inespecíficas da BSA para a superfície. - Imobilizar a amostra por carregar a câmara de fluxo com aproximadamente 150 µ l do biotinilado e rotulado Hsp90 em concentrações crescentes (diluídas em tampão + 0,5 mg/mL BSA) até uma densidade de superfície suficiente é atingida, que é geralmente o caso numa concentração de 5-22:00. Lave desvinculado da proteína com buffer de aproximadamente 300 µ l + 0,5 mg/mL BSA.
- Descarga em 150 µ l de 25 nM AMP-PNP * no tampão + 0,5 mg/mL BSA. Deixe incubar durante 5 min e repita este passo uma vez para garantir a concentração correta do nucleotide.
Nota: Para experiências na presença de AMP-PNP adicionais, sem rótulo, também adicionar 250 µM AMP-PNP. - Começam com a aquisição de dados. Para uma quantidade adequada de dados adquira cerca de 20 filmes, o que leva cerca de 1,5 h.
- Mova a posição da câmara de amostra perpendicular ao feixe de excitação com um seletor de piezo para alterar o campo de visão.
- Ajuste o foco com o z-piezo que controla a altura do objectivo, se necessário. Isso não deve ser necessário muitas vezes, quando a câmara de medição é montada sem inclinação.
- Preparar a gravação das câmeras pressionando < leve sinal > no software da câmera e iniciar os ciclos de excitação/aquisição usando o botão < Iniciar > no software do gatilho. Isso inicia a aquisição da intensidade da fluorescência.
- Execute um registo de canal pela primeira gravação de um filme com grânulos fluorescentes que mostram a emissão de fluorescência na faixa espectral de todos os canais de deteção da instalação. Em seguida, detectar as posições do grânulo do filme de calibração por procurar os pontos mais brilhantes e determinar que a posição central de uma gaussiana se encaixam no perfil de intensidade. Salve as coordenadas dos grânulos que são encontrados em todos os canais e ajuste os dois o deslocamento mapeamento em x - e y-direção com uma 2D polinomial de grau três9.
3. selecção dos traços de único-molécula
- A análise dos dados é feita com in-house scripts usando Igor Pro. Carregar todos os scripts necessários através da abertura de "iniTIRF.ipf" e selecione o tipo correto de experiência.
Nota: no seguinte, < botão > especifica elementos clicáveis no menu ou a interface do usuário. Chamadas de função são indicadas entre aspas, por exemplo, "Print"Hello World"". Esses comandos podem ser colados na linha de comando do Igor Pro (sem as aspas de fechamento). - Verifique se que os parâmetros para o registo do canal de deteção são carregados.
- Inicie a GUI clicando < smFRET novo | Análise GUI >.
- Carrega os filmes (ou seja, a sequência de quadros com 512 x 512 pixels armazenados como pilhas TIFF de 16 bits) que seguram a intensidade nos respectivos canais. Fazer isso pressionando o botão < filme de carga > e selecionando os arquivos das câmeras "mestre" e "escravo" so-called um após o outro.
- Identifica as posições dos potenciais moléculas simples, procurando os pontos mais brilhantes na soma dos cinco primeiros quadros em um determinado canal de deteção. Calcule as posições correspondentes em todos os outros canais de deteção de mapeamento do canal. Para obter o rastreamento de intensidade de fluorescência, soma-se a intensidade de um quadrado de pixel em torno da posição central para cada quadro. Fazer isso pressionando o botão < encontrar vestígios > no GUI.
Nota: O comprimento do lado do quadrado (em pixel) é dada por: 2 * < soma Pxs > + 1. - Para cada molécula, calcule um traço comum intensidade crua como a soma de todos os vestígios deste local com a mesma cor de excitação. Avalie o perfil de intensidade da molécula em todos os canais para os seguintes critérios:
- Um planalto aproximadamente plano em intensidade crua conjunta e um branqueamento único passo para todas as cores de excitação, comportamento anticorrelated nos canais apropriados de deteção, detecção de fluorescência vermelha (indicando FRET para um limite AMP-PNP *) pelo menos uma vez dentro do rastreamento e não há várias etapas na fluorescência vermelha, que indicaria a presença de dois AMP-PNP * acoplado a um dímero de Hsp90.
- Salve os traços de fluorescência do local para posterior análise, se estes critérios forem satisfeitos. Fazer isso selecionando o rastreamento com o cursor e pressionando o botão < salvar > no gráfico de "prazos". Inspecione manualmente os traços de intensidade em todos os três canais de deteção após excitação azul para cerca de 200 moléculas por filme.
4. cálculo dos traços fluorescência parcial
- Exiba todos os vestígios de intensidade de fluorescência para uma das moléculas salvas. Use os cursores no gráfico para selecionar intervalos de tempo.
- Selecione um intervalo de tempo onde todos os fluorophores são clareados já. A intensidade média do fundo é calculada a partir desse intervalo e subtraída o rastreamento de intensidade em cada canal. Fazer isso pressionando o botão < Background >.
- Selecione o intervalo de eficiência FRET, onde pelo menos ambos os corantes anexados a Hsp90 (Atto488 e Atto550) estão presentes. Certifique-se de excluir rastreamentos que contêm um evento piscando (ver Figura 3B). Esses eventos são caracterizados por uma intensidade de fluorescência de drop-in em um canal sem um aumento que acompanha qualquer outro canal.
- Calcule os vestígios PF . Fazer isso pressionando o botão < PF Calc >. Os factores de correcção predefinidos para vazamento aparente (LC) e aparente gama (ag) são aplicados a intensidade crua para corrigir para foto-física e propriedades de configuração.
5. população seleção e ajuste de histograma 3D
- Remova moléculas que mostram uma baixa relação sinal-ruído nos vestígios PF . Moléculas que excedem o intervalo [-1; 2] em qualquer vestígio PF para mais de 10% dos quadros são removidas do conjunto de dados. Fazer isso, executar o "RemoveTracesLowSNR()" na janela de comando.
- Calcule binned projeções 2D de dados da PF . Lote
 sobre
sobre 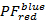 e
e 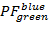 sobre
sobre 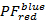 no intervalo [-0.5; 1.5] com uma resolução de 100 x 100 caixas. Para fazer isso, execute:
no intervalo [-0.5; 1.5] com uma resolução de 100 x 100 caixas. Para fazer isso, execute:- "HistFret2D ("r_b","r_g", binHist = 100)"
- "HistFret2D ("r_b","g_b", binHist = 100); MoveWindow 553.5, 42,5, 1055.25, 508,25"
- Determine a população relativa de cada Estado distinguível nas projecções 2D.
- Trazer o gráfico apropriado para a frente e executar o "panelHist2DCount()".
- Pressione a tecla < Init > e desenhar um polígono de mão livre em volta do pico.
- Clique no botão < contagem >. O número de pontos de dados no polígono e o número total de pontos de dados na projeção são impressos na janela de comando.
- Preparar um histograma 3D dos dados PF executando "HistFret3D ("g_b","r_b","r_g")".
- Normalize o histograma 3D para uma integral de 1. Execute o seguinte:
- "Fit0 de NewDataFolder/S"
- "Duplicata/O:: FRET: Hist3D, Hist3D"
- "Div variável/g = sum(Hist3D)*(DimDelta(Hist3D,0)) ^ 3"
- "Hist3D = div; Impressão div"
- Fornece parâmetros iniciais para o Gaussian 3D se encaixa e preparem estruturas de dados necessárias.
- Execute "Gauss3D_initParam(); editar W_coef_old."
- Adicione as populações do estado até o fim do vetor parâmetro.
- Executar "Gauss3D_prepareFit()."
Nota: W_coef_old é um vetor que contém os parâmetros iniciais para o ajuste. Por Estado, isso significa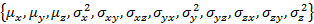 e a população do estado, que é concatenada ao final do vetor. Certifique-se que a matriz de covariância é simétrica.
e a população do estado, que é concatenada ao final do vetor. Certifique-se que a matriz de covariância é simétrica.
- Cabe a soma de S funções Gaussian 3D para o histograma PF 3D, com S , sendo o número de Estados distinguíveis.
- Executar "do3D()." Isto pode demorar uma hora ou mais em um escritório normal PC, dependendo da qualidade dos parâmetros iniciais.
- Execute "postprocessFitMultiGauss3D(); evalFitMultiGauss3D(); editar W_coef."
- Exiba o resultado de apto. Para cada uma das duas projeções 2D, use os seguintes comandos:
- "contourPF3D_new(0); contourPF3D_new(1); contourPF3D_new(2); contourPF3D_new(3); contourPF3D_new(4)"
- "contourPF3D_colorize()"
6. cinética análise 3D Ensemble HMM
- Prepare um ensemble HMM executar para extrair as informações cinéticas. Um HMM é otimizado de todas as moléculas no conjunto de dados. Use as informações obtidas na etapa anterior para definir a posição e a largura de cada Estado no espaço 3D PF .
- Inicializar a interface do usuário HMM (< HMM | Init HMM >) e escolha o número apropriado de Estados-Membros (no caso dos dados Hsp90 isso significa < NumStates > = 5), o número de dimensões do sinal de entrada (< NumDims > = 3) e o tipo da entrada (< Input Type > = "FRET bgr 3D").
- Otimizar os parâmetros do HMM, deixando o software convergem a probabilidade do HMM (execute "prepENS_CONVERGE_gB(GetDataFolder(1), -14)") até que a mudança na matriz de transição, em comparação com a iteração anterior cai abaixo de um limite (10-14 para a soma da mudança absoluta para cada probabilidade de transição). Isto rende o MLE para as probabilidades de transição em cerca de uma hora em um PC normal do escritório.
- Repita a seleção da população, encaixe gaussiana e a otimização de HMM para subconjuntos de dados (por exemplo, 75% do conjunto de dados completo). Se fundiu-se o conjunto de dados completo de experiências diferentes, repita a otimização também para cada um dos experimentos simples. Análise de subconjuntos permite estimar a incerteza da seleção manual da população e a variabilidade dentro do conjunto de dados.
- Calcule o CI para as probabilidades de transição, que o relatório sobre a heterogeneidade do conjunto de dados e a precisão do HMM.
- Obter uma estimativa aproximada dos limites CI executando "$cd (raiz: path3Dimport +"HMM"); loop_getCI_estimate_limits()."
- Calcule os limites exatos da IC, executando:
- "loop_getCI_HMM_converge(1)"
- "CIresults_conv_new()"
- "cd:: HMM_CIresult; reportCI_conv()"
- "cd:: cmp_CI_conv; CI_plot2 ("HMM", doAppend = 0) "
- Condense as informações cinéticas disponíveis para simplificar a interpretação.
- Colete informações sobre o tempo um nucleotídeo rotulado permanece ligado à Hsp90. Para fazer isso, o colapso dos Estados que são obrigados a AMP-PNP * (ou seja, S1e S2 , S3, ver também a Figura 2) e compilar o histograma de tempo de permanência. Execute o "cd $(raiz: path3Dimport +"HMM"); collapse_states_get_DT({0,1,1,1,0}) ", que combina habita de Estados S0 e S4 bem como S1e S2S3.
- Extrair os tempos de permanência do caminho de Viterbi para cada Estado de interesse e comparar o histograma resultante de tempo Habitai para diferentes condições experimentais. Execute "plot_collapsed_DT_Hist (wDTo_01110_record1)."
- Para uma imagem mais detalhada, colapso dos Estados que têm distintas PF mas são funcionalmente idênticos para facilitar a análise de dados adicional, por exemplo, no caso de um experimento de três cores com Hsp90 etiquetados e nucleotídeos, Estados S2 e S 3 pode ser recolhido. Executar "collapse_states_get_DT({0,1,2,2,4})."
Resultados
Medições de cor multi smFRET permitem a detecção directa de correlação entre dois ou mais sites de interação distintos. Isso torna a técnica exclusiva para investigar sistemas multicomponentes, tais como os complexos da proteína. Focalizamos a apresentação de um experimento de três cores de smFRET aqui, que serve como um exemplo ilustrativo.
O fluxo de trabalho geral do método é mostrado na Figura 1. A primeira parte compreende a gravação de dados multi-cor smFRET em um microscópio TIRF prisma-tipo. A estratégia de superfície de fixação e os esquemas da instalação estão representados na Figura 2A. Para uma descrição mais detalhada da instalação, consulte a referência9. A segunda parte do método apresentado concentra-se na análise dos dados. Vestígios de intensidade de fluorescência exemplar são mostrados na Figura 2B. Tempo apropriado traça mostrar: (i) um claro passo branqueamento para ambos os fluorophores anexado a Hsp90, platôs de intensidade (ii) plana, (iii) anticorrelacionados comportamento nos canais correspondentes e evento de ligação (iv) pelo menos um de AMP-PNP * (Figura 3). Mais de 400 moléculas que atendem aos critérios de seleção foram selecionadas para produzir estatísticas fiáveis. No sistema estudado, cinco Estados podem ser distinguidos pelas intensidades de fluorescência, com quatro Estados, sendo funcionalmente distintos (Figura 2).
A intensidade da fluorescência vestígios que da fluorescência parcial (PF, a extensão da eficiência FRET para experiências multi cor smFRET) pode ser calculadas (Figura 4A). A PF está relacionada com a proximidade das tinturas. Em um experimento de três cores de smFRET, os dados abrange um espaço 3D (Figura 5B). 2D projecções do histograma 3D tem provado para ser útil para a separação do estado (Figura 4B, C). Em uma experiência bem sucedida, todos os Estados que teoricamente são esperados nas condições experimentais aplicadas são distinguíveis pelos seus PF nas projecções 2D.
As populações relativas dos Estados são determinadas das projeções 2D pelo desenho a mão livre polígonos que cercam o pico (Figura 5). Esta abordagem foi encontrada para ser exato e confiável9. As probabilidades de emissão para o HMM são obtidas por encaixe o histograma PF 3D com a soma de funções Gaussian 3D, onde S é o número de Estados distinguíveis (cinco no caso apresentado; A Figura 5). Para este ajuste convergir corretamente, apenas a população relativa peu de cada estado deve ser mantida constante enquanto a posição e a largura da Gaussians são livres.
Sobre o conjunto completo de dados, um ensemble HMM é otimizado com as probabilidades de emissão fixadas para os parâmetros obtidos a partir do ajuste Gaussian (Figura 6). A fim de obter uma medida para a incerteza das probabilidades de transição extraídos, 95% CI, para cada transição é determinada (Figura 7).
Além disso, a média tempo que o nucleotídeo repórter rotulado AMP-PNP * permanece vinculado a Hsp90 pode ser extraído sob diferentes condições experimentais (Figura 8A). Isso ajuda a reduzir ainda mais a complexidade dos resultados. Para fazê-lo, Estados-Membros que representam AMP-PNP * acopladas e desacopladas conformações são recolhidas em trajetórias do estado, respectivamente. Do Habitai médio tempo para o AMP-PNP * dissociação pode ser calculado (Figura 8B).
Comparando-se a cinética observada na ausência e presença de adicionais, podem ser adquiridas sem rótulo informações AMP-PNP, exclusivas sobre a correlação entre as alterações conformacionais de Hsp90 e o estado de nucleotídeo. Isto torna possível estudar diretamente a cooperatividade entre os dois bolsos de vinculação de nucleotídeo de Hsp90. Além disso, ele contorna a necessidade de experimentos de titulação que medem a ocupação do local de ligação em função da concentração de substrato (por exemplo, lotes de Hill). Para sistemas de proteínas altamente dinâmico como o Hsp90, esta abordagem também é sensível a pequenas mudanças em taxas11.

Figura 1 : Fluxo de trabalho geral de aquisição de dados e análise. Dados são adquiridos em um microscópio de fluorescência de reflexão interna total multi cor smFRET (TIRFM). Após a seleção de rastreamento e cálculo da fluorescência parcial (PF), o histograma PF 3D e 2D projeções respectivas são compiladas. Usando as 2D projecções, a população de todos os Estados-Membros distinguíveis pode ser determinada. Estes são usados como uma restrição para um 3D Gaussian apto para o histograma PF . As funções de densidade de probabilidade gaussiana servem como probabilidades de emissão para o ensemble subsequente 3D Hidden Markov Modeling (HMM). Isso produz a descrição cinética do sistema. Clique aqui para ver uma versão maior desta figura.

Figura 2 : Regime de aquisição de dados. (A) pictograma do sistema estudado, consistindo de um dímero de Hsp90 (ovais amarelas representam a estrutura de domínio) ligado à superfície com as etiquetas Atto488 (azul) e Atto550 (verde) e o nucleotídeo repórter AMP-PNP * em solução, etiquetado com Atto647N (vermelho). Dados são gravados em um microscópio TIRF prisma-tipo com a alternância de excitação do laser (ALEX). (B) vestígios de intensidade (FL. Int.) exemplar de fluorescência após a excitação de azul e verde. (C) pictogramas dos Estados conformacionais distinguíveis da Hsp90 (S0S1, S2, S3, S-4) e seu respectivo identificador para o estado funcional (O, C, O *, C *) usado neste trabalho. Fluoróforo posições estão indicadas em vermelho, verde e azul. Duas populações representam o mesmo estado funcional, ou seja, abrir Hsp90 com AMP-PNP * vinculado (S-2 e S-3). Clique aqui para ver uma versão maior desta figura.

Figura 3 : Critérios de seleção. (A), A molécula seleccionada para uma análise mais aprofundada. (B, C) Moléculas não selecionados para uma análise mais aprofundada. (B) nenhum chapadas planas e Atto550 está em um estado escuro cerca de 30 s após a excitação verde (indicada pelas setas). (C) branqueamento várias etapas após a excitação verde (indicada pelas setas). FL. Int.: intensidade de fluorescência, azul: Atto488, verde: Atto550, Atto647N vermelho, ex: excitação. Clique aqui para ver uma versão maior desta figura.
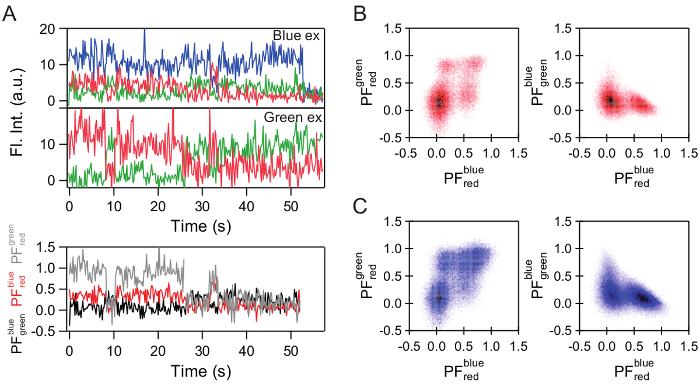
Figura 4 : Cálculo dos vestígios PF e histogramas representativas. Vestígios de intensidade (FL. Int.) (A) representante da fluorescência e os vestígios de fluorescência parcial (PF) correspondente. (B) duas projeções 2D do histograma PF 3D para a medição na ausência do nucleotide adicional, sem rótulo. (C) as mesmas projeções para o experimento na presença adicional de 250 µM AMP-PNP. Clique aqui para ver uma versão maior desta figura.

Figura 5 : Processo de seleção da população. (A) a posição das cinco populações distinguíveis nas duas projecções 2D. (B) cópia dos pictogramas na Figura 2. (C) representação 3D de dispersão dos dados PF . A coloração dos pontos de dados é somente para visualização. É usado o mesmo código de cor, como em B. (D) determinação das populações relativas é feita pelo desenho a mão livre de polígonos em torno dos picos no histograma 2D. Usando uma combinação das duas projeções retratado na Figura 3, todas as cinco populações podem ser distinguidas. (E) resultados do Gaussian 3D apto para o histograma dos dados PF mostrados na r. Depicted são o isosuperfícies em FWHM, que representam as cinco populações diferentes. Clique aqui para ver uma versão maior desta figura.

Figura 6 : Fluxograma da otimização ensemble 3D HMM. Um modelo é otimizado para todas as moléculas do conjunto de dados. Os valores de início são fornecidos pelo modelo de entrada (com um número pré-definido de Estados). A probabilidade do modelo dado os dados (os traços PF 3D) é avaliada com o algoritmo para diante-para trás (FB). O algoritmo Baum-Welch (BW) produz uma estimativa local probabilidade máxima (MLE) dos parâmetros. O MLE global então encontram-se iterativamente. O algoritmo de Viterbi calcula a trajetória de estado mais provável dada um modelo. Clique aqui para ver uma versão maior desta figura.
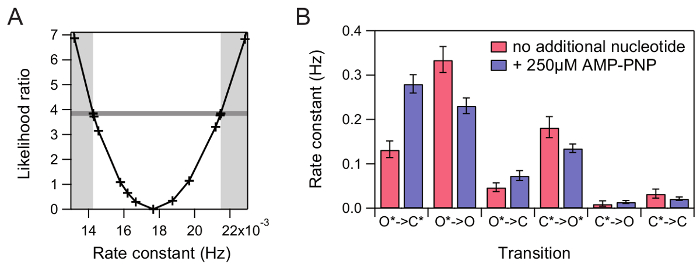
Figura 7 : Estimativa de incerteza significativa com CI. (A) determinação da IC para uma constante de velocidade exemplar. O rácio de probabilidade (LR) do modelo de teste em comparação com o modelo do estimador (MLE) de máxima verossimilhança é calculado na região em torno do MLE para a constante de velocidade. A limite de confiança de 95% é atingida quando o LR excede 3,841 (linha horizontal de cinza escura). (B) a taxa extraída constantes sem adicional nucleotídeo (vermelho) e com AMP-PNP (azul) e seu 95% CI. Clique aqui para ver uma versão maior desta figura.

Figura 8 : A média habitar o tempo do nucleotide repórter AMP-PNP * limite para Hsp90 é prolongada na presença do nucleotide adicional. (A) pictograma da dissociação observada de AMP-PNP rotulada * do dímero Hsp90, com uma média ao longo de todas as conformações de Hsp90. A estrutura de domínio de Hsp90 é representada pelo amarelos ovais e a flexibilidade conformacional é indicada pela sobreposição de um dímero de aberto e fechado. (B) média habitam tempo de AMP-PNP * limite a Hsp90 ausência do nucleotide adicional (vermelho) e na presença de 250 µM unlabeled AMP-PNP (azul). Clique aqui para ver uma versão maior desta figura.
Discussão
Apresentamos o procedimento experimental para obter dados de três cores smFRET para um sistema complexo de proteínas e uma descrição passo a passo da análise dessas medições. Essa abordagem oferece a possibilidade única de avaliar diretamente a correlação entre vários sites de interação ou mudanças conformacionais.
A fim de obter dados de único-molécula multi cores adequados em proteínas é importante realizar as medições podem ser reproduzidas em um baixo nível de ruído. Isto pode ser conseguido usando um protocolo de passivação superfície eficiente e confiável para a câmara de fluxo9. Uma densidade suficiente de moléculas imobilizadas para a superfície deve ser usada para aumentar o rendimento de moléculas por filme gravado. Isto significa que luz de moléculas simples de fluorescência deve cair sobre a cerca de 5-10% dos pixels nas imagens gravadas. Ao mesmo tempo sobrecarga deve ser prevenida, pois resultaria em uma sobreposição dos vizinhos moléculas. Quando uma espécie etiquetada é livre em solução (AMP-PNP * no estudo apresentado), certifique-se que a concentração é baixa o suficiente para não interferir com a medição por excitação direta de lasers com comprimento de onda menor (ou seja, tipicamente bem abaixo um micromolar concentração). Também, certifique-se que a concentração destes compostos não é menor do que a prevista devido à vinculação inespecíficas para interfaces. Além disso, o poder de excitação ideal representa um trade-off entre a relação sinal-ruído e o fotobranqueamento do fluorophores.
Sem oxigênio, sistema de eliminação é empregado no protocolo apresentado. Os corantes usados de Atto-Tec não apresentam significativos piscando na escala de tempo do experimento (70 ms iluminação por ciclo de excitação) e sem diminuição da taxa de lixívia de foto foi observada por um sistema de limpador consistindo de glicose oxidase, catalase e Trolox33 ,34. Também, ausência de scavenger de oxigênio evita artefatos devido às interações proteína inespecíficos, como sistemas de eliminação de oxigênio geralmente contêm componentes proteicos até uma concentração micromolar35.
Outro passo crítico é a seleção de rastreamento. O tamanho da área para somar a intensidade de um fluoróforo único é escolhido para ser tão pequena quanto possível mas ainda maior do que o ponto de espalhar a função da instalação do. Esteja ciente de que um platô plano na intensidade comum traços só podem ser esperados se os canais de deteção tem gama aparente semelhante fatores, uma vez que os traços não são corrigidos nesta fase da análise. Somente moléculas que atendam a critérios bem definidos para os vestígios de intensidade de fluorescência são incluídas na análise (secção 3). Moléculas com uma pobre relação sinal-ruído em rastreamentos o PF excluem-se de uma análise mais aprofundada e não afetam a avaliação de dados como clara seleção critérios são utilizados.
Dependendo da qualidade de dados, abordagens alternativas fornecem alocação de estado ideal. Um enciclopédia ensemble multidimensional HMM baseado também sobre a intensidade de fluorescência traços27 ou os traços PF poderiam ser aplicados para alocar os Estados subjacentes, otimizando as probabilidades de emissão correspondente (por exemplo, Gaussian PDFs) enquanto ao mesmo tempo otimizando o modelo cinético. Ambas as abordagens têm suas vantagens e desvantagens. Vestígios PF podem conter espinhos desfavoráveis enquanto os níveis de intensidade de fluorescência variam de molécula a molécula. O protocolo apresentado resolve esse problema evitando transições artificiais quando ocorrem picos e estimar que as probabilidades de emissão de uma gaussiana 3D apto para o histograma PF e só otimizando as probabilidades de transição no subsequente Ensemble HMM executar.
A abordagem apresentada é limitada pela necessidade de um parceiro de interação rotulado que vincula-se com uma afinidade elevada comparativamente a fim de ser compatível com as necessário para medições convencionais do único-molécula de baixas concentrações. Isto pode ser superado por estratégias para aumentar a concentração local (tethering da interação parceiros36, encapsulamento em vesículas37,38) ou métodos para diminuir o volume animado (por exemplo, zero-modo guias de onda39). Além disso, etiquetas posições tem que ser escolhido com cuidado e experimentos de controle são necessários para excluir os efeitos secundários graves do fluorophores. Em caso de Hsp90, isto geralmente é verificado por um ensaio de ATPase, que prova que a atividade enzimática de40. Se disponível, deve ser considerada informação estrutural do cristal ou estrutura NMR. Posições preferenciais em loops de superfície exposta, longe de interfaces de interação e não enterraram em um bolso de superfície da proteína. Isso garante um grande volume acessível para a tintura.
O quadro é adequado para um experimento de multi cor smFRET com um número arbitrário de cores. Focamos em três cores medidas aqui, pois estas já fornecem o recurso que processa-los distintos de outros experimentos de único-molécula, ou seja, a possibilidade de observar diretamente a correlação entre dois processos (por exemplo, a ligação de um parceiro de interação e mudanças conformacionais). Esta informação é inacessível em um experimento de smFRET padrão de duas cores, mas é de interesse fundamental para a compreensão da função de qualquer máquina molecular.
O experimento apresentado e análise é capaz de caracterizar sistemas de proteína, que podem exibir dinâmica em uma ampla gama de escalas de tempo entre os Estados são altamente flexíveis se3. Isto contrasta publicados anteriormente multi cor smFRET experimentos, que incidiu sobre sistemas de modelo de DNA ou RNA, como o Holliday entroncamentos4, exibindo as transições entre Estados bem definidos (geralmente dois). Em sistemas de proteínas, a relação sinal-ruído é inferior e extrair o conjunto completo de probabilidades de transição é difícil por causa da largura de banda limitada temporal em experimentos de smFRET devido ao fotobranqueamento. Aqui, nós mostrar-se aplicada com cuidado e com restrições razoáveis - os obstáculos em proteína sistemas de medição podem ser superados com multi cor smFRET usando alocação estado multidimensional e ensemble escondido Markov análise9.
Divulgações
Os autores declaram não há conflitos de interesse.
Agradecimentos
Este trabalho é financiado pela Fundação de pesquisa alemã (INST 39/969-1) e o Conselho Europeu de investigação através da Convenção de subvenção ERC n. 681891.
Materiais
| Name | Company | Catalog Number | Comments |
| Setup | |||
| vibration-damped optical table | Newport, Irvine, CA, USA | RS2000 | |
| OBIS 473nm LX 75mW LASER | Coherent Inc, Santa Clara, CA, USA | 1185052 | |
| OBIS 532nm LS 50mW LASER | Coherent Inc, Santa Clara, CA, USA | 1261779 | |
| OBIS 594nm LS 60mW LASER | Coherent Inc, Santa Clara, CA, USA | 1233470 | |
| OBIS 637nm LX 140mW LASER | Coherent Inc, Santa Clara, CA, USA | 1196625 | |
| laser control unit | Coherent Inc, Santa Clara, CA, USA | 1234465 | Scientific Remote |
| aspheric telescope lenses | Thorlabs Inc, Newton, New Jersey, USA | d=25.4mm, f=50mm and f=100mm | |
| CF ex1 | AHF analysentechnik AG, Tübingen, Germany | ZET 473/10 | cleanup filter excitation |
| CF ex2 | AHF analysentechnik AG, Tübingen, Germany | ZET 532/10 | cleanup filter excitation |
| CF ex3 | AHF analysentechnik AG, Tübingen, Germany | ZET 594/10 | cleanup filter excitation |
| CF ex4 | Thorlabs Inc, Newton, New Jersey, USA | FL635-10 | cleanup filter excitation |
| DM ex1 | AHF analysentechnik AG, Tübingen, Germany | ZQ594RDC | dichroic mirror excitation |
| DM ex2 | AHF analysentechnik AG, Tübingen, Germany | 570DCXR | dichroic mirror excitation |
| DM ex3 | AHF analysentechnik AG, Tübingen, Germany | ZQ491RDC | dichroic mirror excitation |
| AOTFnC-Vis | AA Opto-Electronic, Orsay, France | ||
| λ/4 plate | Thorlabs Inc, Newton, New Jersey, USA | AQWP05M-600 | |
| CFI Apo TIRF 100x | Nikon Instruments Inc, Melville, NY, USA | high-NA objective | |
| piezo focus positioner MIPOS 250 CAP | piezosystem jena GmbH, Jena, Germany | Piezo Controller NV 40/1 CLE | |
| piezo stepper | Newport, Irvine, CA, USA | PZA12 | PZC200-KT NanoPZ Actuator Kit |
| achromatic aspheric lenses | Qioptiq Photonics GmbH & Co. KG, Göttingen, Germany | G322-304-000 | d=50mm, f=200mm |
| adjustable optical slit | Owis GmbH, Staufen i. Br., Germany | 27.160.1212 | max. aperture 12 x 12 mm |
| DM det1 | AHF analysentechnik AG, Tübingen, Germany | T 600 LPXR | dichroic mirror detection |
| DM det2 | AHF analysentechnik AG, Tübingen, Germany | H 560 LPXR superflat | dichroic mirror detection |
| DM det3 | AHF analysentechnik AG, Tübingen, Germany | HC BS R635 | dichroic mirror detection |
| BP det1 | AHF analysentechnik AG, Tübingen, Germany | 525/40 BrightLine HC | bandpass filter detection |
| BP det2 | AHF analysentechnik AG, Tübingen, Germany | 586/20 BrightLine HC | bandpass filter detection |
| BP det3 | AHF analysentechnik AG, Tübingen, Germany | 631/36 BrightLine HC | bandpass filter detection |
| BP det4 | AHF analysentechnik AG, Tübingen, Germany | 700/75 ET Bandpass | bandpass filter detection |
| optical shutters detection | Vincent Associates, Rochester, NY, USA | Uniblitz VS25S2T0 | |
| EMCCD iXon Ultra 897 | Andor Technology Ltd, Belfast, Northern Ireland | ||
| digital I/O card, PCIe-6535 | National Instruments, Austin, Texas, USA | ||
| syringe pump | Harvard Apparatus, Holliston, MA, USA | PHD22/2000 | |
| Name | Company | Catalog Number | Comments |
| Flow chamber | |||
| quartz slides | G. Finkenbeiner Inc, Waltham, MA, USA | Spectrosil2000, h=3mm | |
| TEGADERM film | 3M Deutschland GmbH, Neuss, Germany | 1626W | 10 x 12cm |
| spray adhesive | 3M Deutschland GmbH, Neuss, Germany | Photo Mount 050777 | |
| glycerol | Carl Zeiss AG, Oberkochen, Germany | Immersol G | |
| immersion oil | OLYMPUS EUROPA SE & CO. KG, Hamburg, Germany | IMMOIL-F30CC | |
| prism | Vogelsberger Quarzglastechnik GmbH, Hauzenberg, Germany | Suprasil1 | |
| aluminium prism holder | custom built | ||
| hollow setscrews | Thorlabs Inc, Newton, New Jersey, USA | with custom drilling | |
| Tygon S3 E-3603 tubing | neoLab Migge GmbH, Heidelberg, Germany | 2-4450 | ACF00001 |
| PTFE tubing | Bohlender GmbH, Grünsfeld, Germany | S1810-08 | |
| Name | Company | Catalog Number | Comments |
| Sample | |||
| yeast Hsp90 D61C, Q385C_biotin | UniProt ID P02829 | ||
| Maleimide derivatives of Atto488, Atto550 | ATTO-TEC GmbH, Siegen, Germany | ||
| AMP-PNP* | Jena Bioscience, Jena, Germany | γ-[(6-Aminohexyl)-imido]-AMP-PNP-Atto647N | |
| Fluospheres | Thermo Fisher Scientific, Waltham, MA, USA | F8764 | amine-modified, 0.2 μm, yellow-green fluorescent |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Andor Solis | Andor Technology Ltd, Belfast, Northern Ireland | version 4.30 | |
| LabVIEW | National Instruments, Austin, Texas, USA | version 2012, 32bit; misc. hardware control | |
| MDS control software | AA Opto-Electronic, Orsay, France | version 2.03a | |
| Coherent Connection | Coherent Inc, Santa Clara, CA, USA | version 3 | |
| Igor Pro | WaveMetrics Inc, Portland, OR, USA | version 6.37 |
Referências
- Nooren, I. M. A., Thornton, J. M. Diversity of protein-protein interactions. EMBO J. 22 (14), 3486-3492 (2003).
- Marsh, J. A., Teichmann, S. A. Structure, dynamics, assembly, and evolution of protein complexes. Annu Rev Biochem. 84, 551-575 (2015).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Hohng, S., Joo, C., Ha, T. Single-Molecule Three-Color FRET. Biophys J. 87 (2), 1328-1337 (2004).
- Person, B., Stein, I. H., Steinhauer, C., Vogelsang, J., Tinnefeld, P. Correlated movement and bending of nucleic acid structures visualized by multicolor single-molecule spectroscopy. ChemPhysChem. 10 (9-10), 1455-1460 (2009).
- Lee, J., Lee, S., Ragunathan, K., Joo, C., Ha, T., Hohng, S. Single-molecule four-color FRET. Angew Chem Int Ed. 49 (51), 9922-9925 (2010).
- Ratzke, C., Berkemeier, F., Hugel, T. Heat shock protein 90's mechanochemical cycle is dominated by thermal fluctuations. Proc Natl Acad Sci U S A. 109 (1), 161-166 (2012).
- Ratzke, C., Hellenkamp, B., Hugel, T. Four-colour FRET reveals directionality in the Hsp90 multicomponent machinery. Nat Commun. 5, 4192 (2014).
- Götz, M., Wortmann, P., Schmid, S., Hugel, T. A Multicolor Single-Molecule FRET Approach to Study Protein Dynamics and Interactions Simultaneously. Methods Enzymol. 581, 487-516 (2016).
- Yengo, C. M., Berger, C. L. Fluorescence anisotropy and resonance energy transfer: Powerful tools for measuring real time protein dynamics in a physiological environment. Curr Opin Pharmacol. 10 (6), 731-737 (2010).
- Wortmann,P , ., Götz M, ., Hugel T , . Cooperative Nucleotide Binding in Hsp90 and Its Regulation by Aha1. Biophys J. 113, 1711-1718 (2017).
- Dörfler, T., Eilert, T., Röcker, C., Nagy, J., Michaelis, J. Structural Information from Single-molecule FRET Experiments Using the Fast Nano-positioning System. J Vis Exp. (120), e54782 (2017).
- Stephanopoulos, N., Francis, M. B. Choosing an effective protein bioconjugation strategy. Nature chemical biology. 7 (12), 876-884 (2011).
- Sánchez-Rico, C., Voith von Voithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of Fluorophore Attachment on Protein Conformation and Dynamics Studied by spFRET and NMR Spectroscopy. Chemistry (Weinheim an der Bergstrasse, Germany). , (2017).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nat Methods. 5 (6), 507-516 (2008).
- Lee, N. K., et al. Three-color alternating-laser excitation of single molecules: monitoring multiple interactions and distances. Biophys J. 92 (1), 303-312 (2007).
- Kapanidis, A. N., Lee, N. K., Laurence, T. A., Doose, S., Margeat, E., Weiss, S. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proc Natl Acad Sci U S A. 101 (24), 8936-8941 (2004).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chem Soc Rev. 43 (4), 1156-1171 (2014).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nat Methods. 14, 174-180 (2017).
- Rabiner, L. R. A tutorial on hidden Markov models and selected applications in speech recognition. Proc IEEE. 77 (2), 257-286 (1989).
- Fink, G. A. . Markov Models for Pattern Recognition. , (2014).
- Giudici, P., Ryden, T., Vandekerkhove, P. Likelihood-Ratio Tests for Hidden Markov Models. Biometrics. 56 (3), 742-747 (2000).
- Visser, I., Raijmakers, M. E. J., Molenaar, P. C. M. Confidence intervals for hidden Markov model parameters. Br J Math Stat Psychol. 53 (2), 317-327 (2000).
- McKinney, S. A., Joo, C., Ha, T. Analysis of Single-Molecule FRET Trajectories Using Hidden Markov Modeling. Biophys J. 91 (5), 1941-1951 (2006).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L., Wiggins, C. H. Learning Rates and States from Biophysical Time Series: A Bayesian Approach to Model Selection and Single-Molecule FRET Data. Biophys J. 97 (12), 3196-3205 (2009).
- Greenfeld, M., Pavlichin, D. S., Mabuchi, H., Herschlag, D. Single Molecule Analysis Research Tool (SMART): an integrated approach for analyzing single molecule data. PLoS ONE. 7 (2), e30024 (2012).
- Schmid, S., Götz, M., Hugel, T. Single-Molecule Analysis beyond Dwell Times: Demonstration and Assessment in and out of Equilibrium. Biophys J. 111 (7), 1375-1384 (2016).
- Taipale, M., Jarosz, D. F., Lindquist, S. HSP90 at the hub of protein homeostasis: emerging mechanistic insights. Nat Rev Mol Cell Biol. 11 (7), 515-528 (2010).
- Trepel, J., Mollapour, M., Giaccone, G., Neckers, L. Targeting the dynamic HSP90 complex in cancer. Nat Rev Cancer. 10 (8), 537-549 (2010).
- Wayne, N., Bolon, D. N. Dimerization of Hsp90 is required for in vivo function. Design and analysis of monomers and dimers. J Biol Chem. 282 (48), 35386-35395 (2007).
- Ali, M. M. U., et al. Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex. Nature. 440 (7087), 1013-1017 (2006).
- Southworth, D. R., Agard, D. A. Species-dependent ensembles of conserved conformational states define the Hsp90 chaperone ATPase cycle. Mol Cell. 32 (5), 631-640 (2008).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proc Natl Acad Sci U S A. 101 (35), 12893-12898 (2004).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophys J. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Rognoni, L., Stigler, J., Pelz, B., Ylänne, J., Rief, M. Dynamic force sensing of filamin revealed in single-molecule experiments. Proc Natl Acad Sci U S A. 109 (48), 19679-19684 (2012).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in Surface-Tethered Lipid Vesicles as a New Tool for Single Biomolecule Spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode waveguides for single-molecule analysis at high concentrations. Science. 299 (5607), 682-686 (2003).
- Panaretou, B., et al. ATP binding and hydrolysis are essential to the function of the Hsp90 molecular chaperone in vivo. EMBO J. 17 (16), 4829-4836 (1998).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados