Method Article
Verwendung von dreifarbigen Einzelmolekül-Bund, die Korrelation von Protein-Interaktionen zu studieren
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen ein Protokoll zur dreifarbigen SmFRET Daten und deren Auswertung mit einem 3D-Ensemble Hidden-Markov-Modell zu erhalten. Mit diesem Ansatz können Wissenschaftler kinetische Informationen aus komplexen Protein Systeme, einschließlich der Kooperativität oder korrelierte Interaktionen extrahieren.
Zusammenfassung
Einzelmolekül-Förster Resonance Energietransfer (SmFRET) ist eine weit verbreitete biophysikalische Technik, die Dynamik von Biomolekülen zu studieren geworden. Für viele molekulare Maschinen in einer Zelle Proteine zusammen mit Interaktionspartner in einem funktionalen Kreislauf, ihre Aufgabe zu erfüllen müssen. Die Verlängerung der Zweifarben-, Multi-Color SmFRET macht es möglich, gleichzeitig mehrere Interaktion oder Konformationsänderung Sonde. Dies gibt nicht nur eine neue Dimension für SmFRET Experimente, sondern es bietet auch die einzigartige Möglichkeit, die Abfolge der Ereignisse direkt zu studieren und korrelierte Interaktionen zu erkennen, bei der Verwendung einer immobilisierten Probe und eine Totalreflexion Fluoreszenz Mikroskop (TIRFM). Multi-Color SmFRET deshalb ein vielseitiges Werkzeug für die Untersuchung biomolekularer komplexe in einer quantitativen Weise und in einem zuvor unerreichbar Detail.
Hier zeigen wir wie die besonderen Herausforderungen der Multi-Color SmFRET Experimente an Proteinen zu überwinden. Wir präsentieren Ihnen ausführliche Protokolle für die Beschaffung der Daten und kinetische Informationen extrahieren. Dazu gehören Spur Auswahlkriterien, Trennung von Staat und die Rückgewinnung von staatlichen Bahnen aus dem verrauschten Daten mit Hilfe einer 3D Ensemble Hidden Markov Model (HMM). Im Vergleich zu anderen Methoden, ist die kinetische Informationen nicht aus wohnen Zeit Histogramme, sondern direkt aus der HMM wiederhergestellt. Die maximum-Likelihood-Rahmen ermöglicht es uns, die kinetische Modell kritisch zu bewerten und sinnvolle Unsicherheiten für die Tarife zu bieten.
Unsere Methode auf die Hitze-Schock-Protein 90 (Hsp90) anwenden, können wir die Nukleotid-Bindung und die globale Konformationsänderungen des Proteins zu entwirren. Dies ermöglicht uns die Kooperativität zwischen den zwei Nukleotid verbindliche Taschen von Hsp90 Dimer direkt zu beobachten.
Einleitung
Viele Proteine erfüllen ihre Funktion in dynamische komplexe mit anderen Molekülen, vermittelt durch Konformationsänderungen und transiente Verbände auf ein breites Spektrum von zeitlichen1,2,3. Gekoppelt an eine externe Energiequelle (z. B. ATP) diese dynamische Interaktionen zu Direktionalität in einem funktionalen Kreislauf führen und letztlich nicht-Gleichgewichts-stationären in eine Zelle, die Voraussetzung für das Leben zu erhalten.
Um diese molekularen Maschinen zu verstehen, ist eine statische Beschreibung geführt durch strukturelle Studien nicht ausreichend. Darüber hinaus ist es wichtig, Kenntnisse der zugrunde liegenden kinetischen Modell haben und die kinetischen Geschwindigkeitskonstanten bestimmen. Mehrere vorhandene Methoden können Forscher die Dynamik der binäre Interaktionen zwischen zwei Molekülen von Interesse, z. B. Oberflächenplasmonenresonanz, Entspannungsmethoden mit einer spektroskopischen Anzeige (z. B. Sprung oder gestoppt-Flow zu studieren Techniken) und magnetischen Kernresonanz. Ihre Anwendbarkeit ist jedoch in den meisten Fällen beschränkt auf einfache zwei-Staaten-Systeme (z. B. einer gebundenen und einer ungebundenen Zustand) durch die Mittelung in loser Schüttung Experimente. In Fällen wo mehrere Staaten oder Zwischenprodukte beteiligt sind, führen sie nur eine komplexe Mischung aus der Geschwindigkeitskonstanten. Einzelmolekül-Methoden wie optische oder magnetische Pinzette oder Zweifarben-SmFRET, d. h. eine Spenderin und einem Akzeptor Fluorophor, mit einer Oberfläche immobilisiert Probe können die Geschwindigkeitskonstanten für alle beobachteten Konformationsänderungen wiederherstellen. Allerdings geht es um Interaktionen beeinflussen mehr als eine Bindungsstelle, diese Methoden beschränkt bleiben und die Informationen über die mögliche Korrelation der beiden (oder mehr) Wechselwirkungen werden nur über indirekte Schlussfolgerungen aus einer Reihe von Experimenten.
Multi-Color-SmFRET4,5,6,7,8,9 bietet die Möglichkeit, das Zusammenspiel dieser Komponenten direkt in Echtzeit und unter studieren in der Nähe von physiologischen Bedingungen10. Dies erlaubt zum Beispiel die Konformation-abhängige Bindung eines Liganden oder ein anderes Protein8,9,11untersuchen. Das hier vorgestellte Gesamtkonzept ist benutzt von Interesse an bestimmten Positionen, ein Protein auf der Oberfläche der Messkammer zu befestigen und die Fluoreszenzintensität im Laufe der Zeit auf ein Prisma-Typ TIRFM (für Details siehe 9 verfolgen beschriften , 12). die räumliche Nähe der verschiedenen Farbstoffe kann dann aus der Energieübertragung zwischen ihnen ermittelt werden. Kennzeichnung Strategien von Protein zu Protein (rezensiert in 13) abweichen und Richtlinien, um Artefakte in SmFRET Messungen zu vermeiden gibt es14.
Da ein Spender-Farbstoff zu verschiedenen Akzeptor Farbstoffe in einem Multi-Color-SmFRET-Experiment Energie übertragen kann, ist die relative Position der alle Farbstoffe nicht Erregung einen Farbstoff allein15,16erreichbar. Aber in Kombination mit abwechselnden Laseranregung (ALEX17und bewertete in 18) bietet diese Methode alle räumlich-zeitliche Informationen bei unter einer Sekunde und Sub-Nanometer Auflösung.
Im Prinzip hochauflösende strukturelle Informationen erreicht werden kann, mithilfe der Inter Farbstoff Entfernungen berechnet aus der Kombination aller Fluoreszenz-Intensitäten in einem Multi-Color-SmFRET-Experiment mit ALEX. Aber, hier konzentrieren wir uns auf staatliche Identifizierung und Trennung sowie die Gewinnung von kinetische Modelle, wo Multi-Color SmFRET unerlässlich ist. Wenn "nur" Strukturaufklärung durch Triangulation gewünscht wird, kann eine Reihe einfacher Zweifarben-SmFRET-Experimente mit hohen Signal-Rausch-Verhältnis durchgeführten12,19.
Wir verwenden teilweise Fluoreszenz ( ) als Proxy für die Energieübertragung zwischen zwei Fluorophore7. Die PF errechnet sich aus der Fluoreszenzintensität analog zu der FRET-Effizienz eines zwei-Farben-Experiments:
) als Proxy für die Energieübertragung zwischen zwei Fluorophore7. Die PF errechnet sich aus der Fluoreszenzintensität analog zu der FRET-Effizienz eines zwei-Farben-Experiments:

Wo,  ist die Intensität in Emission Kanal Em nach Anregung mit Farbe abund c ist der Akzeptor mit der längsten Wellenlänge. Erkennung-Kanäle stellen die gleiche Position in der Probenkammer aber erfassen unterschiedliche Spektralbereiche des Lichts Fluoreszenz dar. Der gleiche Bezeichner für Anregung und Emission werden in diesem Protokoll verwendet (z.B. "blau", "grün" und "rot").
ist die Intensität in Emission Kanal Em nach Anregung mit Farbe abund c ist der Akzeptor mit der längsten Wellenlänge. Erkennung-Kanäle stellen die gleiche Position in der Probenkammer aber erfassen unterschiedliche Spektralbereiche des Lichts Fluoreszenz dar. Der gleiche Bezeichner für Anregung und Emission werden in diesem Protokoll verwendet (z.B. "blau", "grün" und "rot").
Aufgrund experimenteller Mängel hängen die gemessenen Fluoreszenz-Intensitäten nicht nur auf die Energieübertragung, sondern auch von Fluorophor und Setup-Eigenschaften. Um die wahre Energieeffizienz der Übertragung zwischen zwei Fluorophore zu erhalten, müssen die gemessenen Intensitäten korrigiert werden. Die folgende Prozedur basiert auf Referenz9. Korrekturfaktoren für scheinbare Leckage (lk, d. h. der Nachweis von Photonen aus einem Fluorophor in einem Kanal für ein anderer Farbstoff bezeichnet) und scheinbare Gamma (ag, d. h. die Quantenausbeute der Fluoreszenz des Farbstoffes und die Nachweiseffizienz des Kanals) stammen aus Einzelmolekül-Spuren, die ein Akzeptor bleichen Veranstaltung zeigen.
Das Austreten von der Spender-Farbstoff in jeden möglichen Akzeptor-Kanal errechnet sich aus alle Datenpunkte in der aufgezeichneten Fluoreszenz Spuren wo der Akzeptor Farbstoff gebleicht aber der Spender ist noch fluoreszierende ( ):
):

Der Median des Histogramms Leckage dient als scheinbare Leckage Faktor. Nach der Korrektur auf Dichtheit richtet sich der scheinbare Gammafaktor aus den gleichen Satz von Spuren. Es wird ermittelt, indem die Änderung der Fluoreszenz in den Akzeptor-Kanal durch die Änderung der Fluoreszenz in der Geber-Kanal beim Bleichen von der Akzeptor Farbstoff:

Wobei c wieder die Erkennung Kanal für den Akzeptor mit der längsten Wellenlänge ist. Der Median der daraus resultierenden Verteilung wird als die scheinbare Korrekturfaktor verwendet.
Die korrigierten Intensitäten in jedem Kanal stammen von:

PF wird dann entsprechend berechnet:

Verschiedene Populationen können in der multi-dimensionalen Raum erstreckte sich von der PFs getrennt werden. Die Position und Breite der einzelnen Staaten wird durch Anpassung der Daten mit multi-dimensionalen Gaußsche Funktionen bestimmt. Anschließende Optimierung von einem globalen HMM basierend auf alle PF -Spuren bietet eine quantitative Beschreibung der beobachteten Kinetik. Selbst kleine Änderungen der Preise sind nachweisbar.
Hmm bieten eine Möglichkeit der Ableitung ein Zustandsmodell aus einer Sammlung von lauten Zeitspuren. Das System wird als in einem der eine Reihe von diskreten, versteckten Staaten an einem bestimmten Zeitpunkt und die tatsächliche Beobachtung (d. h. die Emission) ist eine probabilistische Funktion von diesem Status "verborgen"20. Bei TIRFM SmFRET Daten kann die Emission Wahrscheinlichkeiten bich pro Staat ich durch kontinuierliche "glockenförmig" Wahrscheinlichkeitsdichte Funktionen modelliert werden. Zu regelmäßigen Abständen diskreten Zeitpunkten können Übergänge von einem in einen anderen Staat nach dem Übergang Wahrscheinlichkeit auftreten, die Zeit-invariante und hängt nur von den aktuellen Zustand. Der Übergangsmatrix A enthält diesen Übergang Wahrscheinlichkeiten einerIj zwischen allen versteckten Staaten. Der Ausgangszustand Verteilung  gibt die bundeslandspezifischen Wahrscheinlichkeiten
gibt die bundeslandspezifischen Wahrscheinlichkeiten  für den ersten Zeitpunkt der Zeit spurlos. Mit einem Maximum-Likelihood-Ansatz, können dieser Parameter optimiert werden, um die Daten mit der vorwärts-rückwärts und Baum-Welch Algorithmen20,21am besten beschreiben. Daraus ergibt sich die maximum-Likelihood-Schätzer (MLE). Schließlich kann der Staat-Sequenz, die höchstwahrscheinlich die Flugbahn des Beobachtungen produziert mit dem Viterbi-Algorithmus abgeleitet werden. Im Gegensatz zu anderen HMM Analysen von SmFRET Daten24,25,26 verwenden wir keine HMM als eine bloße "Glättung" der Daten, sondern die kinetische Staatsmodell Extrakt aus dem Datensatz ohne die Notwendigkeit für den Einbau Verweilzeit Histogramme27. HMM die Auswertung erfolgt mit eigenen Skripten mit Igor Pro. Umsetzung des Kodex basiert auf Referenz21. Wir bieten eine Softwarekit und beispielhafte Daten auf unserer Webseite um Abschnitte 5 und 6 dieses Protokolls (https://www.singlemolecule.uni-freiburg.de/software/3d-fret) folgen. Vollständige Software ist auf Anfrage erhältlich.
für den ersten Zeitpunkt der Zeit spurlos. Mit einem Maximum-Likelihood-Ansatz, können dieser Parameter optimiert werden, um die Daten mit der vorwärts-rückwärts und Baum-Welch Algorithmen20,21am besten beschreiben. Daraus ergibt sich die maximum-Likelihood-Schätzer (MLE). Schließlich kann der Staat-Sequenz, die höchstwahrscheinlich die Flugbahn des Beobachtungen produziert mit dem Viterbi-Algorithmus abgeleitet werden. Im Gegensatz zu anderen HMM Analysen von SmFRET Daten24,25,26 verwenden wir keine HMM als eine bloße "Glättung" der Daten, sondern die kinetische Staatsmodell Extrakt aus dem Datensatz ohne die Notwendigkeit für den Einbau Verweilzeit Histogramme27. HMM die Auswertung erfolgt mit eigenen Skripten mit Igor Pro. Umsetzung des Kodex basiert auf Referenz21. Wir bieten eine Softwarekit und beispielhafte Daten auf unserer Webseite um Abschnitte 5 und 6 dieses Protokolls (https://www.singlemolecule.uni-freiburg.de/software/3d-fret) folgen. Vollständige Software ist auf Anfrage erhältlich.
Zeitpunkten in den Daten mit PF <-1 oder PF > 2 in jedem Kanal Erkennung sind die minimale Emission Wahrscheinlichkeit für alle Staaten (10-200) zugeordnet. Dies verhindert, dass künstliche Übergänge an diese Datenpunkte.
Die Passform des 3D PF Histogramms mit Gaußsche Funktionen werden die Parameter für die Emission Wahrscheinlichkeiten entnommen, wie im Schritt 5.7 beschrieben. Diese Parameter werden bei der Optimierung der HMM fest gehalten.
Des vorgestellten Ansatzes dienen der Ausgangszustand Verteilung Vektor und die Übergangsmatrix weltweit, das gesamte Ensemble der Spuren zu beschreiben. Sie werden aktualisiert, basierend auf alle N Moleküle aus dem Datensatz nach Verweis27.
Startparameter für den Anfangszustand Vertrieb aus 2D Projektionen des Histogramms PF (Schritt 5.3) ermittelt und die Übergangswahrscheinlichkeiten werden sich voraussichtlich auf 0,05 mit Ausnahme der Wahrscheinlichkeiten bleiben im gleichen Zustand, die solche ausgewählt werden dass die Wahrscheinlichkeit, einen bestimmten Zustand zu verlassen zur Einheit normalisiert.
Ein Wahrscheinlichkeit Profiler-Methode dient zur Konfidenzintervalle (CIs) für alle Übergang Preise21,22, dienen als sinnvolle Schätzungen für ihre Unsicherheit zu geben. Um die Grenzen der CI zu einem bestimmten Preis zu berechnen, ist die Wahrscheinlichkeit Übergang von Interesse auf einen anderen Wert als die MLE fixiert. Daraus ergibt sich die Test-Modell λ ". Eine Wahrscheinlichkeit-Verhältnis (LR) Test der Wahrscheinlichkeit  angesichts des Datensatzes erfolgt nach 0 :
angesichts des Datensatzes erfolgt nach 0 :

Das 95 %-Konfidenzintervall gebunden für der Parameter erreicht ist, überschreitet LR 3.841, das 95 %-Quantil der ein x2-Verteilung mit einem Freiheitsgrad22,23.
Die Macht der Methode wird unter Verwendung der Hsp90 demonstriert. Dieses reichliche Protein findet sich in Bakterien und Eukaryoten und ist Bestandteil der zellulären Stress-Reaktion-28. Es ist ein viel versprechendes Medikament Ziel in Krebs Behandlung29. Hsp90 ist ein Homodimer mit einem Nukleotid-Bindungstasche in die N-terminale Domäne von jeder Untereinheit30. Es kann Übergänge zwischen mindestens zwei weltweit unterschiedliche Konformationen, geschlossen und eine N-terminale offen, v-förmige Konformation19,31,32unterziehen. Die dimeres Natur Frage direkt die des Zusammenspiels zwischen die zwei Nukleotid-Bindungsstellen im Hsp90.
Im folgenden bieten wir eine Schritt für Schritt-Protokoll für die Datenerfassung und Analyse einer dreifarbigen SmFRET Experiment auf Hefe Hsp90 und Nukleotid. Die Konformation-abhängige Bindung von Eindringmittel beschrifteten AMP-PNP (AMP-PNP *, ein nicht-hydrolysierbare Analogon von ATP) wird analysiert. Die Anwendung des beschriebenen Verfahrens erlaubt der Studie der Nukleotid-Bindung und zur gleichen Zeit die Konformationsänderungen Hsp90 und zeigt damit die Kooperativität zwischen den zwei Nukleotid verbindliche Taschen von Hsp90.
Protokoll
1. Aufbau und Voraussetzungen
- Führen Sie die Multi-Color SmFRET Messungen auf ein Prisma-Typ TIRFM. Eine Beschreibung eines zwei-Farben-Setup wie eine Jupiter-Veröffentlichung gegeben ist,12verweisen.
- Ein Multi-Color-TIRFM zu konstruieren. Eine allgemeine Layout wird in 9erläutert.
- Verwendung umschaltbar, gepumpt Diode kontinuierliche Welle Festkörperlaser, die die Verwendung von mechanischen Rollläden in die Erregung Pfade unnötig machen.
- Beschäftigen Sie eine asymmetrische, längliche Prisma, die verhindert, dass die hintere Reflexion des Strahls Erregung von der Rückseite her das Ziel eingeben.
- Verwenden Sie 2-Zoll-achromatisch asphärische Fused-Silica-Objektive in der Erkennung-Pfade, die so viel Licht wie möglich zu sammeln und zu verhindern, dass Auto-Fluoreszenz und Verirrungen, z. B. Verzerrungen in Regionen außerhalb der Mitte des Bildes.
- Jeder Erkennung Pfad auf dem Chip der EMCCD mit einem separaten Objektiv zu konzentrieren. Dies ermöglicht optimale Fokussierung des Kanals Erkennung.
Vorsicht: Klasse 3 b Laser werden in der TIRFM verwendet. Dies bedeutet, dass sie gefährlich sind, wenn das Auge direkt ausgesetzt ist, aber diffuse Reflexionen nicht schädlich sind. Einhaltung der Laser Sicherheitsvorkehrungen gemäß den Regierungsvorschriften der lokalen bevor das System betrieben wird.
- Die Korrekturfaktoren für Setup und Fluorophor Eigenschaften vorher mit DsDNA Proben zu bestimmen.
- Verwenden Sie eine hoch-FRET DsDNA-Probe für jede Farbe in Kombination mit den Akzeptor hat die längste Wellenlänge Erregung (für die vorgestellten Setup: Atto488-Atto647N, Atto550-Atto647N Atto594-Atto647N). Stellen Sie sicher, dass die DNA mit Biotin zusätzlich geändert wird.
- Verdünnen die Probe 5 nM mit TNM Puffer (5 mM Tris pH 7.5, 5 mM NaCl, 20 mM MgCl2) und 2 mM Trolox (verwenden Sie diesen Puffer auch für Messungen).
- Die DsDNA zu immobilisieren, wie für Hsp90 2.5 und 2.7 beschrieben.
- Berechnen Sie die Korrekturfaktoren für scheinbare Leckage (lk) und scheinbare Gamma (ag) vom Einzelmolekül-Spuren, die ein Akzeptor bleichen Ereignis zeigen.
- Konstruieren Sie eine Flow-Kammer, die ein Sandwich einer PEG/Biotin-PEG passivierte Quarz Folie ist, einen dünnen Film, der Kleber auf beiden Seiten und einem Deckglas ist. Detail-Protokolle für die Reinigung des Quarzes Folien und Passivierung finden Sie in der Referenz9.
- Nutzung (3 mm) Dicke Quarz gleitet, um geometrisch die Sammlung des Laserlichts verstreut an der Quarz-Glycerin-Quarz-Schnittstelle zwischen dem Prisma und der funktionalisierten Quarz-Folie zu verhindern.
- Verwenden Sie eine dünne (40 µm) Abdichtung Film, der mit Klebstoff auf der nicht klebenden Seite gesprüht wird. Die dünne Folie reduziert den Abstand zwischen der Oberfläche befestigt Moleküle und Ziel. Erhitzen auf 80 ° C und drücken Sie auf.
- Platzieren Sie ein Deckglas auf der Oberseite. Erhitzen auf 80 ° C und drücken Sie auf. Verwenden Sie einen Tropfen des Glycerins bei der Platzierung der Strömungsraum über das Prisma.
Hinweis: Die Materialien des Prismas und die Quarz-Folien sowie dem Glycerin sind Index-abgestimmt.
- Hsp90 von Saccharomyces Cerevisiae in Form von zwei einzelnen Cystein Punkt Mutanten an den Positionen D61 oder Q385 auszudrücken. Fügen Sie eine C-terminale coiled-Coil Motiv um Dimer Dissoziation bei pikolomaren Konzentrationen zu vermeiden. Beschriften Sie der mutierten Proteine separat und tauschen Sie die Monomere um Heterodimere beschriftet mit Atto488 an Aminosäure Position 61 und Atto550 an der Aminosäure Position 3859zu erhalten.
2. Messung
- Starten Sie die Kamerasoftware und die bildgebenden Parameter wie unten angegeben:
- Stellen Sie die Temperatur des Sensors Kühlung so niedrig wie möglich (-95 ° C mit externen Wasserkühlung) um die aktuellen Dunkelrauschen zu verringern.
- Verwenden Sie Kameraeinstellungen, die optimiert sind für die Einzelmolekül-Aufnahme: 3,3 µs vertikale Verschiebung Geschwindigkeit, normale vertikale Uhr Spannung, 17 MHz 16-Bit horizontale ausgelesen, Vorverstärkung Gain 3, der Elektronen-Vervielfacher 1.000 zu gewinnen.
- Die Auslösung des Erwerbs für "Externe" und die Belichtungszeit bis 70 Ms.-Rekord-Filme mit einer Länge von 750 Erwerb Zyklen festgelegt.
Hinweis: Schalten Sie das Raumlicht, wenn die Kamera erwirbt, um Sättigung des EMCCD Sensors zu verhindern.
- Erstellen Sie einen Ordner auf der lokalen SSD für die Messung. In der Software Einstellungen zur < Auto-Save > Reiter, < automatisches Speichern > aktivieren, und wählen Sie Datei-Format "Tiff" für Film-Übernahme. Wählen Sie den Ordner auf der SSD als Auto-speichern-Standort.
- Starten Sie die Software, die Kontrollen der akusto-optischen durchstimmbare Filter (Aventurien), die Software, die steuert den Betrieb der Laser und die Trigger-Software, die die Laser, Aventurien, synchronisiert Fensterläden in der Erkennung Pfad und Kameras. Passen Sie die Leistung des Lasers mit Aventurien (ca. 3 mW vor dem Eintritt in das Prisma) und laden Sie die richtige auslösende Muster.
- Befestigen Sie die Probenhalterung mit dem Prisma und den Strömungsraum, befestigen Sie Schlauch, legen Sie den Zulauf-Schlauch in einem Microcentrifuge Cup und schließen Sie den Auslass-Schlauch an eine Spritzenpumpe. Der Saal mit ca. 150 µL Puffer spülen, richten Sie die Erregung Strahl und Fokus. Bleichen von fluoreszierenden Verunreinigungen auf der Oberfläche von langsam entlang der kompletten Erfassungsbereich der Folie mit einer Laserleistung von etwa 10 mW für alle Laser. Dies dauert etwa 1 h.
Hinweis: soweit nicht anders angegeben, sind der verwendete Puffer 40 mM HEPES pH 7.5, 150 mM KCl, 10 mM MgCl2. - Spülen Sie rund 300 µL einer NeutrAvidin Lösung (0,25 mg/mL in Puffer) in die Kammer und 1 min. Flush aus ungebundenen NeutrAvidin mit Puffer und Flush ca. 300 µL einer BSA-Lösung (0,5 mg/mL in Puffer) durch die Kammer inkubieren.
Hinweis: Dieser Block, die restliche Oberfläche Funktionalisierung Mängel durch unspezifische Adsorption der BSA an die Oberfläche. - Die Probe durch das Laden der Strömungsraum mit ca. 150 µL der biotinylierte zu immobilisieren und beschriftet Hsp90 bei steigenden Konzentrationen (verdünnt in Puffer + 0,5 mg/mL BSA) bis eine ausreichende Oberfläche Dichte erreicht wird, was in der Regel der Fall ist bei einer Konzentration von 5-22:00. Ungebundene Protein mit etwa 300 µL Puffer + 0,5 mg/mL BSA auswaschen.
- Bündig in 150 µL 25 nM AMP-PNP * Puffer + 0,5 mg/mL BSA. Lassen Sie es 5 min inkubieren und wiederholen Sie diesen Schritt einmal um korrekte Nukleotid-Konzentration zu gewährleisten.
Hinweis: Für Experimente im Beisein von zusätzlichen, unbeschriftete AMP-PNP, auch hinzufügen 250 µM AMP-PNP. - Beginnen Sie mit der Datenerfassung. Für eine angemessene Menge an Daten zu erwerben ca. 20 Filme, die dauert ca. 1,5 h.
- Verschieben Sie die Position der Probenkammer senkrecht zu der Erregung Strahl mit einem Piezo-Stepper, das Sichtfeld zu ändern.
- Stellen Sie den Fokus mit der Z-Piezo, die die Höhe des Ziels steuert, wenn nötig. Dies sollte nicht erforderlich sein, allzu oft, wenn die Messkammer ohne Neigung montiert ist.
- Bereiten Sie die Aufzeichnung der Kameras durch die Kamerasoftware < nehmen Signal > Einpressen und starten Sie die Erregung/Erwerb Zyklen mit der < Start >-Taste in der Trigger-Software zu. Dies startet den Erwerb der Fluoreszenzintensität.
- Führen Sie eine Kanalregistrierung durch erste Filmaufnahme mit fluoreszierenden Perlen, die Fluoreszenzemission im Spektralbereich aller Kanäle der Erkennung des Setups zu zeigen. Dann erkennen Sie Perle Positionen in der Kalibrierung-Film zu, indem die hellsten Stellen suchen und bestimmen Sie, dass die zentrale Lage von einer Gaußschen Intensität Profil passen. Speichern Sie die Koordinaten der Perlen, die findet man in allen Kanälen und passen sowohl den Mapping-Offset in x- und y-Richtung mit einem 2D Polynom von Grad 39.
3. Auswahl der Einzelmolekül-Spuren
- Analyse der Daten erfolgt mit eigenen Skripten mit Igor Pro. Laden Sie alle notwendigen Skripte durch "iniTIRF.ipf" öffnen und wählen Sie den richtigen Typ des Experiments.
Hinweis: im folgenden Schaltfläche "<" > gibt klickbare Elemente in das Menü oder das User-Interface. Funktionsaufrufe werden in Anführungszeichen, z.B. "Print"Hello World"" angezeigt. Diese Befehle können in der Befehlszeile von Igor Pro (ohne die umschließenden Anführungszeichen) eingefügt werden. - Stellen Sie sicher, dass die Parameter für die Erkennung-Kanal-Registrierung geladen werden.
- Drücken Sie zunächst die GUI < SmFRET neue | Analyse GUI >.
- Laden Sie die Filme (d. h. die Reihenfolge der Frames mit 512 x 512 Pixel gespeichert als 16-Bit TIFF-Stapel), die die Intensität in den jeweiligen Kanälen zu halten. Zu diesem Zweck der < Last Film >-Taste und Auswahl der Dateien aus den so genannten "master" und "Slave" Kameras einer nach dem anderen.
- Identifizieren Sie die Positionen der potenziellen Einzelmoleküle durch die Suche nach den hellsten Stellen in der Summe der ersten fünf Frames in einem bestimmten Erkennung-Kanal. Berechnen Sie die entsprechenden Positionen in anderen Erkennung-Kanäle aus der Kanalzuordnung. Um die Fluoreszenz-Intensität-Ablaufverfolgung zu erhalten, addieren Sie die Intensität der ein Pixel-Quadrat um die zentrale Position für jeden Frame. Tun Sie dies durch Drücken der Taste < finden Spuren > in der GUI.
Hinweis: Die Seitenlänge des Quadrats (in Pixel) ergibt sich aus: 2 * < Summe Pxs > + 1. - Berechnen Sie für jedes Molekül eine gemeinsame rohen Intensität Spur als die Summe von allen Spuren dieses Ortes mit der gleichen Farbe der Erregung. Bewerten Sie die Intensität Profil des Moleküls in allen Kanälen nach den folgenden Kriterien:
- Ein etwa flaches Plateau in der gemeinsamen roh Intensität und einem einzigen bleaching Schritt für alle Erregung Farben, Verhalten Verhalten in den entsprechenden Nachweis, Nachweis von rote Fluoreszenz (Angabe Bund zu einem gebundenen AMP-PNP *) mindestens einmal innerhalb der Trace und keine mehrfache Schritte in die rote Fluoreszenz, die das Vorhandensein von zwei AMP-PNP darauf hinweisen würden * ein Hsp90 Dimer verpflichtet.
- Die Fluoreszenz-Spuren des Spots zur weiteren Analyse zu speichern, wenn diese Kriterien erfüllt sind. Tun Sie dies, indem Sie die Ablaufverfolgung mit dem Cursor auswählen und drücken dann die Schaltfläche < speichern > in der "Chronik" Grafik. Manuell überprüfen Sie die Intensität Spuren in allen drei Kanälen der Erkennung nach blauen Anregung für etwa 200 Moleküle pro Film.
4. Berechnung der Spuren, die teilweise Fluoreszenz
- Angezeigt werden alle Spuren der Fluoreszenz-Intensität für eines der gespeicherten Moleküle. Verwenden Sie den Cursor in das Diagramm, um Zeitbereiche auszuwählen.
- Wählen Sie ein Zeitintervall, wo alle Fluorophore bereits gebleicht werden. Die mittlere Hintergrundintensität aus diesem Bereich berechnet und subtrahiert die Intensität Ablaufverfolgung in jedem Kanal. Tun Sie dies durch Drücken der Schaltfläche "< Hintergrund >".
- Wählen Sie den Bund Wirkungsgradbereich, wo zumindest sowohl die Farbstoffe an Hsp90 (Atto488 und Atto550) sind vorhanden. Stellen Sie sicher, Spuren ausschließen, die einen blinkenden Ereignis enthalten (siehe Abb. 3 b). Diese Veranstaltungen zeichnen sich durch ein Drop-in-Fluoreszenzintensität in einem Kanal ohne begleitende Erhöhung in einem anderen Kanal.
- Berechnen Sie die PF -Spuren. Tun Sie dies durch Drücken der Schaltfläche < PF berechnen >. Die vordefinierten Korrekturfaktoren für scheinbare Leckage (lk) und scheinbare Gamma (ag) gelten für die rohen Intensität um Foto-physische zu korrigieren und setup-Eigenschaften.
5. Bevölkerung Auswahl und 3D Histogramm Fitting
- Entfernen Sie Moleküle, die ein niedriges Signal-Rausch-Verhältnis in der PF -Spuren zeigen. Moleküle, die das Intervall [-1; 2] in PF Spur für mehr als 10 % der Rahmen überschreiten werden aus dem Datensatz entfernt. Tun Sie dies durch Ausführen von "RemoveTracesLowSNR()" aus dem Befehlsfenster.
- Gebinnten 2D Projektionen der PF Daten zu berechnen. Plot
 über
über 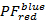 und
und 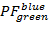 über
über 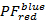 im Bereich von [-0,5; 1,5] mit einer Auflösung von 100 x 100 Lagerplätzen. Dazu führen Sie Folgendes aus:
im Bereich von [-0,5; 1,5] mit einer Auflösung von 100 x 100 Lagerplätzen. Dazu führen Sie Folgendes aus:- "HistFret2D ("R_b","R_g", BinHist = 100)"
- "HistFret2D ("R_b","G_b", BinHist = 100); MoveWindow 553.5, 42,5, 1055.25, 508,25"
- Bestimmen der relativen Bevölkerung jedes unterscheidbar Staates in den 2D Projektionen.
- Bringen Sie die entsprechende Grafik nach vorne zu und führen Sie "panelHist2DCount()".
- Drücken Sie die Schaltfläche "< Init >" und zeichnen Sie ein Polygon freie Hand rund um den Gipfel.
- Klicken Sie auf < Anzahl >. Die Anzahl der Datenpunkte im Polygon und die Gesamtzahl der Datenpunkte in der Projektion werden im Befehlsfenster gedruckt.
- Bereiten Sie ein 3D Histogramm der PF Daten durch Ausführen von "HistFret3D ("G_b","R_b","R_g")".
- Das 3D Histogramm zu einem Integral von 1 zu normalisieren. Führen Sie die folgenden:
- "NewDataFolder/S fit0"
- "Duplikat/O:: Bund: Hist3D, Hist3D"
- "Variable/g Div = sum(Hist3D)*(DimDelta(Hist3D,0)) ^ 3"
- "Hist3D / = Div; Print Div"
- Bieten Sie Ausgangsparameter für die 3D Gauß passen und bereiten die notwendigen Datenstrukturen.
- Führen Sie "Gauss3D_initParam(); Bearbeiten Sie W_coef_old."
- Fügen Sie am Ende des Vektors Parameter Zustand Populationen.
- Führen Sie "Gauss3D_prepareFit()."
Hinweis: W_coef_old ist ein Vektor, der die ursprünglichen Parameter für die Fit hält. Pro Staat dadurch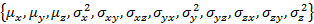 und Bevölkerung, die am Ende des Vektors verkettet ist. Stellen Sie sicher, dass die Kovarianzmatrix symmetrisch ist.
und Bevölkerung, die am Ende des Vektors verkettet ist. Stellen Sie sicher, dass die Kovarianzmatrix symmetrisch ist.
- Passen Sie die Summe S 3D Gaußsche Funktionen auf das 3D PF -Histogramm mit S ist die Zahl der unterscheidbaren Staaten.
- Führen Sie "do3D()." Dies kann eine Stunde oder länger auf einem normalen Büro-PC, abhängig von der Qualität der ursprünglichen Parameter dauern.
- Führen Sie "postprocessFitMultiGauss3D(); evalFitMultiGauss3D(); Bearbeiten Sie W_coef."
- Das Fit Ergebnis anzeigen. Verwenden Sie für jede der beiden 2D Projektionen die folgenden Befehle:
- "contourPF3D_new(0); contourPF3D_new(1); contourPF3D_new(2); contourPF3D_new(3); contourPF3D_new(4)"
- "contourPF3D_colorize()"
(6) kinetische Analyse mit 3D Ensemble HMM
- Bereiten Sie ein Ensemble HMM ausgeführt, um die kinetische Informationen extrahieren. Ein HMM ist von aller Moleküle im Dataset optimiert. Verwenden Sie die Informationen, die im vorherigen Schritt, um die Position und Breite der einzelnen Staaten im 3D-Raum PF definieren.
- HMM-User-Interface zu initialisieren (< HMM | Init HMM >), und wählen Sie die entsprechende Anzahl von Staaten (im Falle von Hsp90 Daten bedeutet dies < NumStates > = 5), Anzahl der Dimensionen des Eingangssignals (< NumDims > = 3), und die Art der Eingabe (< Eingabetyp > = "Bund 3D Bgr").
- Die Parameter des HMM zu optimieren, indem man die Software, die die Wahrscheinlichkeit, dass die HMM konvergieren (Ausführen von "prepENS_CONVERGE_gB(GetDataFolder(1),-14)") bis die Änderung in der Übergangsmatrix im Vergleich zu der vorherigen Iteration unterhalb einer Schwelle (10-14fällt für die Summe der absoluten Veränderung für jede Übergang Wahrscheinlichkeit). Daraus ergibt sich die MLE für die Übergangswahrscheinlichkeiten in etwa eine Stunde auf einem normalen Büro-PC.
- Wiederholen Sie die Bevölkerung Auswahl, Gaußsche Anpassung und die HMM-Optimierung für Teilmengen von Daten (z. B. 75 % von den vollständigen Datensatz). Wenn von verschiedenen Experimenten den vollständigen Datensatz zusammengeführt wurde, wiederholen Sie die Optimierung auch für jede der einzelnen Experimente. Analyse von Teilmengen ermöglicht, um die Unsicherheit der Bevölkerung manuelle Auswahl und die Variabilität innerhalb des Datensatzes zu schätzen.
- Berechnen Sie das CI für die Übergangswahrscheinlichkeiten, die über die Heterogenität des Datensatzes und der Präzision der HMM berichten.
- Erhalten Sie eine grobe Schätzung der CI Grenzen durch Ausführen von "cd $(Wurzel: path3Dimport +"HMM"); loop_getCI_estimate_limits()."
- Berechnen Sie die genauen Grenzen des CI, indem Sie ausführen:
- "loop_getCI_HMM_converge(1)"
- "CIresults_conv_new()"
- "cd:: HMM_CIresult; reportCI_conv()"
- "cd:: Cmp_CI_conv; CI_plot2 ("HMM", DoAppend = 0) "
- Kondensieren Sie die kinetische Informationen um die Interpretation zu erleichtern.
- Sammeln Sie Informationen über die Zeit, dass eine beschriftete Nukleotid Hsp90 verpflichtet bleibt. Zu diesem Zweck, Zusammenbruch der Staaten, die an AMP-PNP gebunden sind * (d. h. S1, S2 und S3, siehe auch Abbildung 2) und das Wohnen Zeit Histogramm zu kompilieren. Führen Sie "cd $(Wurzel: path3Dimport +"HMM"); collapse_states_get_DT({0,1,1,1,0}) ", S0 und S4 und S1, S2und S3Staaten, wohnt der verbindet.
- Der Viterbi Weg für jeden Zustand des Interesses der Verweilzeiten entziehen und das daraus resultierende wohnen Zeit Histogramm für verschiedenen experimentellen Bedingungen zu vergleichen. Führen Sie "Plot_collapsed_DT_Hist (wDTo_01110_record1)."
- Ausblenden Sie für ein detaillierteres Bild Staaten, die verschiedene PF aber funktional identisch mit die weiteren Datenanalyse, z. B.im Falle einer dreifarbigen Experiment mit beschrifteten Hsp90 und Nukleotid, Staaten S2 und S zu erleichtern 3 können reduziert werden. Führen Sie "collapse_states_get_DT({0,1,2,2,4})."
Ergebnisse
Multi-Color SmFRET Messungen ermöglichen die unmittelbare Erfassung der Korrelation zwischen zwei oder mehr ausgeprägte Interaktion Standorte. Dies macht die Technik einzigartig, Mehrkomponenten-Systeme, wie Proteinkomplexe zu untersuchen. Wir konzentrieren uns auf die Präsentation einer dreifarbigen SmFRET Experiment hier dient als ein anschauliches Beispiel.
Die allgemeine Workflow der Methode ist in Abbildung 1dargestellt. Der erste Teil umfasst die Aufnahme von Multi-Color SmFRET Daten über ein Prisma-Typ TIRF Mikroskop. Die Oberfläche Anlage-Strategie und die Schaltpläne des Setups sind in Abbildung 2Adargestellt. Eine ausführlichere Beschreibung der Installation finden Sie unter9verweisen. Der zweite Teil der vorgestellten Methode konzentriert sich auf die Datenanalyse. Beispielhafte Fluoreszenz Intensität Spuren sind in Abbildung 2 bdargestellt. Geeigneter Zeitpunkt Spuren zeigen: (i) einen deutlichen bleichen Schritt für beide Fluorophore verbunden Hsp90, (Ii) flache Intensität Hochebenen, (Iii) Anti-korreliert Verhalten in der entsprechenden Kanäle und (iv) mindestens eine verbindliche Event der AMP-PNP * (Abbildung 3). Mehr als 400 Moleküle, die die Auswahlkriterien erfüllen wurden ausgewählt, um zuverlässige Statistiken ergeben. Des untersuchten Systems können fünf Staaten durch die Fluoreszenz-Intensitäten mit vier Staaten funktionell unterschiedliche (Abbildung 2) unterschieden werden.
Spuren werden die teilweise Fluoreszenz (PF, die Verlängerung der FRET-Effizienz für Multi-Color SmFRET Experimente) berechnet aus der Fluoreszenzintensität (Abb. 4A). Die PF bezieht sich auf die Nähe der Farbstoffe. In einem dreifarbigen SmFRET Experiment umfasst die Daten einen 3D-Raum (Abb. 5 b). 2D Projektionen des 3D Histogramms haben sich als nützlich für die Trennung von Staat (Abbildung 4 b, C). In ein gelungenes Experiment sind alle Zustände, die theoretisch unter den angewandten experimentellen Bedingungen erwartet werden durch ihre PF in den 2D Projektionen.
Die relative Bevölkerung der Staaten aus der 2D Projektionen Zeichnung frei Hand Polygone bestimmt, die den Gipfel (Abbildung 5) umschließen. Dieser Ansatz erwies sich genaue und zuverlässige9. Die Emission Wahrscheinlichkeiten für die HMM ergeben sich durch den Einbau der 3D PF -Histogramm mit der Summe der Gaußschen 3D-Funktionen, wo S ist die Anzahl der unterscheidbaren Staaten (fünf im dargestellten Fall; Abbildung 5). Für diesen Sitz ordnungsgemäß konvergieren hat nur die relative Bevölkerung pich von jedem Staat ich konstant gehalten werden, während die Position und Breite der Mittelwerte kostenlos sind.
Ein Ensemble HMM ist über den vollständigen Datensatz mit der Emission Wahrscheinlichkeiten fest mit den Parametern erhalten von der Gaußschen Fit (Abbildung 6) optimiert. Um ein Maß für die Unsicherheit der extrahierten Übergangswahrscheinlichkeiten, die 95 %-CI erhalten, für jeden Übergang ist bestimmt (Abbildung 7).
Darüber hinaus die durchschnittliche Zeit, dass die beschriftete Reporter Nukleotid AMP-PNP * bleibt gebunden an Hsp90 unter verschiedenen experimentellen Bedingungen (Abb. 8A) extrahiert werden können. Dies hilft, um die Komplexität der Ergebnisse weiter zu reduzieren. Dazu, Staaten, die AMP-PNP darstellen * gebundene und ungebundene Konformationen werden in die staatlichen Bahnen bzw. reduziert. Daraus die durchschnittliche Verweilzeit für den AMP-PNP * Dissoziation werden kann (Abb. 8 b) berechnet.
Vergleicht man die Kinetik in Abwesenheit und das Vorhandensein von zusätzlichen beobachtet, kann unbeschriftete AMP-PNP, einzigartige Informationen über die Korrelation zwischen der Konformationsänderungen Hsp90 und der Nukleotid-Staat gewonnen werden. Dies macht es möglich, direkt die Kooperativität zwischen den zwei Nukleotid verbindliche Taschen von Hsp90 zu studieren. Außerdem umgeht die Notwendigkeit einer Titration Experimente, die die Bindung Website Besetzung als Funktion der Substratkonzentration (z. B. Hill Grundstücke) zu messen. Für hochdynamische Protein Systeme wie Hsp90 ist dieser Ansatz auch empfindlich auf kleine Änderungen in der Tarife-11.

Abbildung 1 : Allgemeine Workflow von der Datenerfassung und Analyse. Daten werden auf einem Multi-Color SmFRET Totalreflexion Fluoreszenzmikroskop (TIRFM) erworben. Nach Spur Auswahl und Berechnung der partiellen Fluoreszenz (PF) sind die 3D PF Histogramm und 2D Projektionen davon zusammengestellt. Mit der 2D Projektionen, kann die Bevölkerung aller unterscheidbar Staaten bestimmt werden. Diese dienen als Hemmnis für eine 3D Gauß passen auf die PF -Histogramm. Die Gaußsche Wahrscheinlichkeitsdichte Funktionen dienen als Emission Wahrscheinlichkeiten für das anschließende 3D Ensemble Hidden Markov Modellierung (HMM). Daraus ergibt sich die kinetische Beschreibung des Systems. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2 : Regelung der Datenerfassung. (A) Piktogramm des untersuchten Systems bestehend aus einem Hsp90 Dimer (gelbe ovale repräsentieren die Domänenstruktur) an der Oberfläche mit den Bezeichnungen Atto488 (blau) und Atto550 (grün) und der Reporter Nukleotid AMP-PNP befestigt * in Lösung, beschriftet mit Atto647N (rot). Daten werden auf einem Prisma-Typ TIRF Mikroskop mit abwechselnden Laseranregung (ALEX) erfasst. (B) vorbildlich Fluoreszenz Intensität (FL. Int.) Spuren nach blauen und grünen Anregung. (C) Piktogramme der unterscheidbaren Konformationsänderungen Staaten von Hsp90 (S-0, S1, S2, S3, S4) und ihre jeweiligen Bezeichner für den Funktionszustand (O, C, O *, C *) in dieser Arbeit verwendet. Fluorophor Positionen sind in blau, grün und rot gekennzeichnet. Zwei Populationen vertreten den gleichen funktionalen Zustand, nämlich offene Hsp90 mit AMP-PNP * gebunden (S2 und S3). Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3 : Auswahlkriterien. (A) A Molekül zur weiteren Analyse ausgewählt. (B, C) Moleküle nicht für die weitere Analyse ausgewählt. (B) keine flachen Hochebenen und Atto550 ist in einem dunklen Zustand ca. 30 s nach grünen Anregung (durch Pfeile angedeutet). (C) mehrere bleichen Schritte nach grünen Anregung (durch Pfeile angedeutet). FL. Int.: Fluoreszenzintensität, blau: Atto488, grün: Atto550, rote Atto647N, ex: Erregung. Bitte klicken Sie hier für eine größere Version dieser Figur.
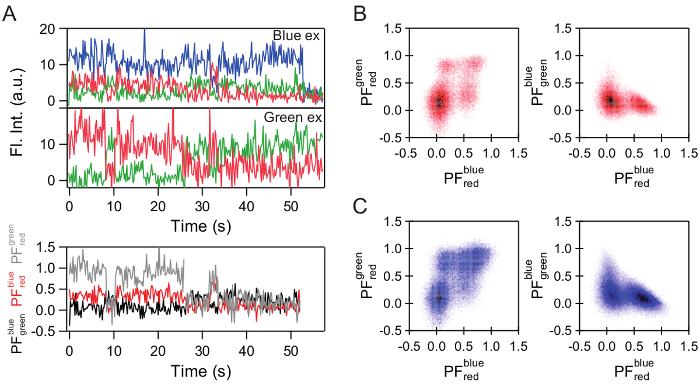
Abbildung 4 : Berechnung der PF Spuren und repräsentative Histogramme. (A) repräsentative Fluoreszenz Intensität (FL. Int.) Spuren und die entsprechenden Spuren teilweise Fluoreszenz (PF). (B) zwei 2D Projektionen des 3D PF Histogramms für die Messung in Abwesenheit von zusätzlichen, unbeschriftete Nukleotid. (C) die gleichen Projektionen für das Experiment im Beisein von zusätzlichen 250 µM AMP-PNP. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 5 : Bevölkerung Auswahlprozess. (A) die Position der fünf unterscheidbaren Populationen in den zwei 2D Projektionen. (B) Kopie der Piktogramme in Abbildung 2. (C) repräsentative 3D Streudiagramm der PF -Daten. Färbung der Datenpunkte ist nur für die Visualisierung. Der gleichen Farbcode wie in B dient. (D) Bestimmung der relativen Bevölkerung erfolgt durch die Zeichnung frei Hand Polygone um die Gipfel im 2D Histogramm. Mit einer Kombination der zwei Projektionen in Abbildung 3dargestellt, können alle fünf Populationen unterschieden werden. (E) Ergebnisse der 3D Gauß passen auf das Histogramm der PF in A. Depicted angezeigten Daten sind Isoflächen bei FWHM, die fünf verschiedenen Populationen darstellen. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 6 : Flussdiagramm der Ensemble-Optimierung für 3D HMM. Ein Modell ist für alle Moleküle aus dem Datensatz optimiert. Die Startwerte sind durch das Eingabemodell (mit einer definierten Anzahl von Staaten) gegeben. Die Wahrscheinlichkeit, dass das Modell angesichts der Daten (die 3D PF Spuren) ist mit der vorwärts-rückwärts (FB) Algorithmus bewertet. Der Baum-Welch (BW) Algorithmus ergibt eine lokale maximum-Likelihood-Schätzung (MLE) der Parameter. Die globale MLE kann dann iterativ gefunden werden. Der Viterbi-Algorithmus berechnet die am wahrscheinlichsten Zustand Flugbahn ein Modell gegeben. Bitte klicken Sie hier für eine größere Version dieser Figur.
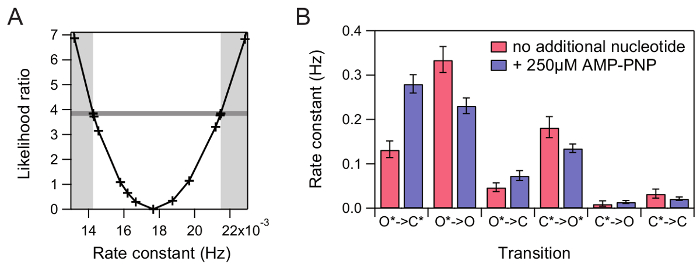
Abbildung 7 : Aussagekräftige Unsicherheit Schätzung mit CI. (A) Bestimmung der CI für eine beispielhafte Geschwindigkeitskonstante. Das Wahrscheinlichkeitsverhältnis (LR) des Testmodells gegenüber dem maximum-Likelihood-Schätzer (MLE)-Modell errechnet sich in der Region um die MLE für die Geschwindigkeitskonstante. Das 95 %-Konfidenzintervall gebunden ist erreicht, wenn die LR 3.841 (horizontale dunkle graue Linie) übersteigt. (B) die extrahierten Rate konstanten ohne zusätzliche Nukleotid (rot) und mit AMP-PNP (blau) und ihre 95 %-CI. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 8 : Die durchschnittliche wohnen Zeit der Reporter Nukleotid AMP-PNP * gebunden an Hsp90 wird in Anwesenheit von zusätzliche Nukleotid verlängert. (A) Piktogramm der beobachteten Dissoziation von beschrifteten AMP-PNP * von Hsp90-Dimer, Mittelung über alle Konformationen von Hsp90. Die Domänenstruktur des Hsp90 ist durch gelbe ovale dargestellt und die Konformationsänderungen Flexibilität wird durch die Überlagerung einer offenen und geschlossenen Dimer. (B) Durchschnitt wohnen Zeit des AMP-PNP * Hsp90 verpflichtet, ohne zusätzliche Nukleotid (rot) und in Anwesenheit von 250 µM unmarkiertem AMP-PNP (blau). Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Wir präsentieren die Versuchsdurchführung um dreifarbigen SmFRET Daten für ein komplexes Protein-System und eine schrittweise Beschreibung der Analyse dieser Messungen zu erhalten. Dieser Ansatz bietet die einzigartige Möglichkeit, die Korrelation zwischen mehreren Websites Interaktion oder Konformationsänderungen direkt zu beurteilen.
Um geeignete Multi-Color Einzelmolekül-Daten auf Proteine zu erhalten ist es wichtig, reproduzierbare Messungen mit einem niedrigen Geräuschpegel. Dies kann erreicht werden, über eine effiziente und zuverlässige Oberfläche Passivierung Protokoll für den Fluss Kammer9. Eine ausreichende Dichte der Moleküle an der Oberfläche immobilisiert sollte verwendet werden, um den Ertrag der Moleküle pro aufgezeichneten Film zu steigern. Dies bedeutet, dass Fluoreszenz Licht aus einzelnen Molekülen auf etwa 5-10 % der Pixel in den aufgenommenen Bildern fallen sollte. Gleichzeitig sollte eine Überlastung verhindert werden, weil es eine Überlappung der benachbarten Molekülen führen würde. Wann ist eine beschrifteten Arten frei in Lösung (AMP-PNP * in der vorgestellten Studie), sicherstellen, dass die Konzentration ist niedrig genug, um nicht zu stören, die Messung durch direkte Anregung von Lasern mit kürzerer Wellenlänge (d. h. in der Regel weit unterhalb eines mikromolaren (Konzentration). Stellen Sie außerdem sicher, dass die Konzentration dieser Verbindungen nicht niedriger als geplant aufgrund der unspezifischen Bindung an Schnittstellen. Darüber hinaus steht für die optimale Erregung macht einen Kompromiss zwischen der Signal-Rausch-Verhältnis und die Immunofluoreszenz die Fluorophore.
Kein Sauerstoff Aufräumvorgang System wird in den vorgestellten Protokoll eingesetzt. Die verwendeten Farbstoffe Atto-Tec zeigen keine signifikanten blinken auf der Zeitskala des Experiments (70 ms Beleuchtung pro Anregung Zyklus) und keinen Rückgang der Foto Bleichmittel Rate beobachtet durch ein Scavenger-System bestehend aus Glukose-Oxidase, Katalase und Trolox33 ,34. Abwesenheit von Sauerstoff Fänger vermeidet auch, Artefakte durch unspezifische Protein-Interaktionen, wie Aufräumvorgang Sauerstoffsysteme in der Regel Proteinkomponenten bis hin zu einem mikromolaren Konzentration35 enthalten.
Ein weiterer wichtiger Schritt ist die Spur-Auswahl. Die Größe des Bereichs für die Intensität der einzelnen Fluorophor summieren wird ausgewählt, so klein wie möglich, aber immer noch größer als der Punkt Funktion des Setups zu verbreiten. Beachten Sie, dass ein flaches Plateau in der gemeinsamen Intensität, die Spuren nur erwartet werden können, wenn die Erkennung Kanäle verfügen über ähnliche scheinbare Gamma Faktoren, da die Spuren nicht in diesem Stadium der Analyse korrigiert werden. Nur Moleküle, die klar definierte für die Fluoreszenz-Intensität-Spuren Kriterien sind in der Analyse (Abschnitt 3) enthalten. Moleküle mit einem schlechten Signal-Rausch-Verhältnis in der PF -Spuren sind von der weiteren Analyse ausgeschlossen und haben keinen Einfluss auf die Datenauswertung als Auswahl aufheben, die Kriterien verwendet werden.
Abhängig von der Qualität der Daten bieten alternative Ansätze optimalen Allokation. Eine kostenlose multi-dimensionalen Ensemble HMM basiert entweder auf der Fluoreszenz Intensität Spuren27 oder PF Spuren angewandt werden, um den zugrunde liegenden Staaten zu verteilen, durch die Optimierung der entsprechenden Emission Wahrscheinlichkeiten (z.B. Gauß PDF-Dateien) und gleichzeitig die Optimierung des kinetischen Modells. Beide Ansätze haben ihre vor- und Nachteile. PF -Spuren können ungünstige Spitzen enthalten, während Fluoreszenz Intensität von Molekül zu Molekül unterschiedlich. Die vorgestellte Protokoll löst dieses Problem durch künstliche Übergänge zu verhindern, wenn Spitzen auftreten und schätzen, dass die Emission Wahrscheinlichkeiten von 3D Gauß auf der PF -Histogramm passen und nur die Übergangswahrscheinlichkeiten in die anschließende Optimierung Ensemble HMM ausgeführt.
Der vorgestellte Ansatz ist begrenzt durch die Notwendigkeit einer beschrifteten Interaktionspartner, der mit einer vergleichsweise hohen Affinität bindet, um kompatibel mit der niedrigen Konzentrationen für konventionelle Einzelmolekül-Messungen notwendig sein. Dies kann überwunden werden durch Strategien zur Erhöhung der lokalen Konzentration (Anbindehaltung von Interaktion Partner36, Kapselung in Vesikeln37,38) oder Methoden, die aufgeregt Lautstärke (z. B. Null-Modus Wellenleiter-39). Darüber hinaus Kennzeichnung Positionen müssen mit Bedacht gewählt werden und Experimente sind notwendig, um schwere Nebenwirkungen der Fluorophore auszuschließen. Im Falle von Hsp90 ist dies in der Regel durch eine ATPase-Assay, aktiviert, die enzymatische Aktivität40beweist. Falls vorhanden, sollte Strukturinformationen aus Kristall oder NMR Struktur betrachtet werden. Bevorzugte Positionen sind in Oberfläche-exponierten Schleifen von Interaktion Schnittstellen und nicht in ein Protein Oberfläche Tasche begraben. Dies gewährleistet eine großvolumige zugänglich für den Farbstoff.
Der Rahmen eignet sich für ein Multi-Color-SmFRET-Experiment mit einer beliebigen Anzahl von Farben. Wir konzentrieren uns auf Dreifarben Messungen hier, da diese bereits die Funktion, der sie unterscheidet sich von anderen Einzelmolekül-Experimente, nämlich die Möglichkeit bieten, direkt die Korrelation zwischen zwei Prozessen beobachten macht (z. B. Bindung von einer Interaktionspartner und Konformationsänderungen). Diese Informationen kann nicht zugegriffen werden, in einem standard Zweifarben-SmFRET-Experiment aber ist von fundamentalem Interesse für das Verständnis der Funktion molekulare Maschine.
Die vorgestellten Experiment und Analyse ist in der Lage, die Charakterisierung von Protein-Systeme, die möglicherweise angezeigt werden Dynamik auf den unterschiedlichsten Zeitskalen zwischen Staaten, die hochflexible selbst3. Im Gegensatz dazu zuvor veröffentlichten Multi-Color SmFRET Experimente, die auf DNA oder RNA Modellsysteme, z. B. Holliday Junctions4, ausstellenden Übergänge zwischen (meist zwei) definierten Zuständen konzentriert. In Protein-Systeme das Signal-Rausch-Verhältnis ist niedriger und extrahieren die Gesamtheit der Übergangswahrscheinlichkeiten ist schwierig wegen der begrenzten zeitlichen Bandbreite in SmFRET Experimente durch Immunofluoreszenz. Hier zeigen wir, dass - können wenn sorgfältig und mit angemessenen Einschränkungen - angewandt die Hindernisse bei der Messung von Protein-Systeme mit Multi-Color SmFRET mit multi-dimensionalen Zustand Zuweisung und Ensemble hidden Markov Analyse9überwunden werden.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Diese Arbeit wird von der Deutschen Forschungsgemeinschaft (INST 39/969-1) und des European Research Council durch die ERC-Grant Agreement n. 681891 finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Setup | |||
| vibration-damped optical table | Newport, Irvine, CA, USA | RS2000 | |
| OBIS 473nm LX 75mW LASER | Coherent Inc, Santa Clara, CA, USA | 1185052 | |
| OBIS 532nm LS 50mW LASER | Coherent Inc, Santa Clara, CA, USA | 1261779 | |
| OBIS 594nm LS 60mW LASER | Coherent Inc, Santa Clara, CA, USA | 1233470 | |
| OBIS 637nm LX 140mW LASER | Coherent Inc, Santa Clara, CA, USA | 1196625 | |
| laser control unit | Coherent Inc, Santa Clara, CA, USA | 1234465 | Scientific Remote |
| aspheric telescope lenses | Thorlabs Inc, Newton, New Jersey, USA | d=25.4mm, f=50mm and f=100mm | |
| CF ex1 | AHF analysentechnik AG, Tübingen, Germany | ZET 473/10 | cleanup filter excitation |
| CF ex2 | AHF analysentechnik AG, Tübingen, Germany | ZET 532/10 | cleanup filter excitation |
| CF ex3 | AHF analysentechnik AG, Tübingen, Germany | ZET 594/10 | cleanup filter excitation |
| CF ex4 | Thorlabs Inc, Newton, New Jersey, USA | FL635-10 | cleanup filter excitation |
| DM ex1 | AHF analysentechnik AG, Tübingen, Germany | ZQ594RDC | dichroic mirror excitation |
| DM ex2 | AHF analysentechnik AG, Tübingen, Germany | 570DCXR | dichroic mirror excitation |
| DM ex3 | AHF analysentechnik AG, Tübingen, Germany | ZQ491RDC | dichroic mirror excitation |
| AOTFnC-Vis | AA Opto-Electronic, Orsay, France | ||
| λ/4 plate | Thorlabs Inc, Newton, New Jersey, USA | AQWP05M-600 | |
| CFI Apo TIRF 100x | Nikon Instruments Inc, Melville, NY, USA | high-NA objective | |
| piezo focus positioner MIPOS 250 CAP | piezosystem jena GmbH, Jena, Germany | Piezo Controller NV 40/1 CLE | |
| piezo stepper | Newport, Irvine, CA, USA | PZA12 | PZC200-KT NanoPZ Actuator Kit |
| achromatic aspheric lenses | Qioptiq Photonics GmbH & Co. KG, Göttingen, Germany | G322-304-000 | d=50mm, f=200mm |
| adjustable optical slit | Owis GmbH, Staufen i. Br., Germany | 27.160.1212 | max. aperture 12 x 12 mm |
| DM det1 | AHF analysentechnik AG, Tübingen, Germany | T 600 LPXR | dichroic mirror detection |
| DM det2 | AHF analysentechnik AG, Tübingen, Germany | H 560 LPXR superflat | dichroic mirror detection |
| DM det3 | AHF analysentechnik AG, Tübingen, Germany | HC BS R635 | dichroic mirror detection |
| BP det1 | AHF analysentechnik AG, Tübingen, Germany | 525/40 BrightLine HC | bandpass filter detection |
| BP det2 | AHF analysentechnik AG, Tübingen, Germany | 586/20 BrightLine HC | bandpass filter detection |
| BP det3 | AHF analysentechnik AG, Tübingen, Germany | 631/36 BrightLine HC | bandpass filter detection |
| BP det4 | AHF analysentechnik AG, Tübingen, Germany | 700/75 ET Bandpass | bandpass filter detection |
| optical shutters detection | Vincent Associates, Rochester, NY, USA | Uniblitz VS25S2T0 | |
| EMCCD iXon Ultra 897 | Andor Technology Ltd, Belfast, Northern Ireland | ||
| digital I/O card, PCIe-6535 | National Instruments, Austin, Texas, USA | ||
| syringe pump | Harvard Apparatus, Holliston, MA, USA | PHD22/2000 | |
| Name | Company | Catalog Number | Comments |
| Flow chamber | |||
| quartz slides | G. Finkenbeiner Inc, Waltham, MA, USA | Spectrosil2000, h=3mm | |
| TEGADERM film | 3M Deutschland GmbH, Neuss, Germany | 1626W | 10 x 12cm |
| spray adhesive | 3M Deutschland GmbH, Neuss, Germany | Photo Mount 050777 | |
| glycerol | Carl Zeiss AG, Oberkochen, Germany | Immersol G | |
| immersion oil | OLYMPUS EUROPA SE & CO. KG, Hamburg, Germany | IMMOIL-F30CC | |
| prism | Vogelsberger Quarzglastechnik GmbH, Hauzenberg, Germany | Suprasil1 | |
| aluminium prism holder | custom built | ||
| hollow setscrews | Thorlabs Inc, Newton, New Jersey, USA | with custom drilling | |
| Tygon S3 E-3603 tubing | neoLab Migge GmbH, Heidelberg, Germany | 2-4450 | ACF00001 |
| PTFE tubing | Bohlender GmbH, Grünsfeld, Germany | S1810-08 | |
| Name | Company | Catalog Number | Comments |
| Sample | |||
| yeast Hsp90 D61C, Q385C_biotin | UniProt ID P02829 | ||
| Maleimide derivatives of Atto488, Atto550 | ATTO-TEC GmbH, Siegen, Germany | ||
| AMP-PNP* | Jena Bioscience, Jena, Germany | γ-[(6-Aminohexyl)-imido]-AMP-PNP-Atto647N | |
| Fluospheres | Thermo Fisher Scientific, Waltham, MA, USA | F8764 | amine-modified, 0.2 μm, yellow-green fluorescent |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Andor Solis | Andor Technology Ltd, Belfast, Northern Ireland | version 4.30 | |
| LabVIEW | National Instruments, Austin, Texas, USA | version 2012, 32bit; misc. hardware control | |
| MDS control software | AA Opto-Electronic, Orsay, France | version 2.03a | |
| Coherent Connection | Coherent Inc, Santa Clara, CA, USA | version 3 | |
| Igor Pro | WaveMetrics Inc, Portland, OR, USA | version 6.37 |
Referenzen
- Nooren, I. M. A., Thornton, J. M. Diversity of protein-protein interactions. EMBO J. 22 (14), 3486-3492 (2003).
- Marsh, J. A., Teichmann, S. A. Structure, dynamics, assembly, and evolution of protein complexes. Annu Rev Biochem. 84, 551-575 (2015).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Hohng, S., Joo, C., Ha, T. Single-Molecule Three-Color FRET. Biophys J. 87 (2), 1328-1337 (2004).
- Person, B., Stein, I. H., Steinhauer, C., Vogelsang, J., Tinnefeld, P. Correlated movement and bending of nucleic acid structures visualized by multicolor single-molecule spectroscopy. ChemPhysChem. 10 (9-10), 1455-1460 (2009).
- Lee, J., Lee, S., Ragunathan, K., Joo, C., Ha, T., Hohng, S. Single-molecule four-color FRET. Angew Chem Int Ed. 49 (51), 9922-9925 (2010).
- Ratzke, C., Berkemeier, F., Hugel, T. Heat shock protein 90's mechanochemical cycle is dominated by thermal fluctuations. Proc Natl Acad Sci U S A. 109 (1), 161-166 (2012).
- Ratzke, C., Hellenkamp, B., Hugel, T. Four-colour FRET reveals directionality in the Hsp90 multicomponent machinery. Nat Commun. 5, 4192 (2014).
- Götz, M., Wortmann, P., Schmid, S., Hugel, T. A Multicolor Single-Molecule FRET Approach to Study Protein Dynamics and Interactions Simultaneously. Methods Enzymol. 581, 487-516 (2016).
- Yengo, C. M., Berger, C. L. Fluorescence anisotropy and resonance energy transfer: Powerful tools for measuring real time protein dynamics in a physiological environment. Curr Opin Pharmacol. 10 (6), 731-737 (2010).
- Wortmann,P , ., Götz M, ., Hugel T , . Cooperative Nucleotide Binding in Hsp90 and Its Regulation by Aha1. Biophys J. 113, 1711-1718 (2017).
- Dörfler, T., Eilert, T., Röcker, C., Nagy, J., Michaelis, J. Structural Information from Single-molecule FRET Experiments Using the Fast Nano-positioning System. J Vis Exp. (120), e54782 (2017).
- Stephanopoulos, N., Francis, M. B. Choosing an effective protein bioconjugation strategy. Nature chemical biology. 7 (12), 876-884 (2011).
- Sánchez-Rico, C., Voith von Voithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of Fluorophore Attachment on Protein Conformation and Dynamics Studied by spFRET and NMR Spectroscopy. Chemistry (Weinheim an der Bergstrasse, Germany). , (2017).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nat Methods. 5 (6), 507-516 (2008).
- Lee, N. K., et al. Three-color alternating-laser excitation of single molecules: monitoring multiple interactions and distances. Biophys J. 92 (1), 303-312 (2007).
- Kapanidis, A. N., Lee, N. K., Laurence, T. A., Doose, S., Margeat, E., Weiss, S. Fluorescence-aided molecule sorting: analysis of structure and interactions by alternating-laser excitation of single molecules. Proc Natl Acad Sci U S A. 101 (24), 8936-8941 (2004).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chem Soc Rev. 43 (4), 1156-1171 (2014).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nat Methods. 14, 174-180 (2017).
- Rabiner, L. R. A tutorial on hidden Markov models and selected applications in speech recognition. Proc IEEE. 77 (2), 257-286 (1989).
- Fink, G. A. . Markov Models for Pattern Recognition. , (2014).
- Giudici, P., Ryden, T., Vandekerkhove, P. Likelihood-Ratio Tests for Hidden Markov Models. Biometrics. 56 (3), 742-747 (2000).
- Visser, I., Raijmakers, M. E. J., Molenaar, P. C. M. Confidence intervals for hidden Markov model parameters. Br J Math Stat Psychol. 53 (2), 317-327 (2000).
- McKinney, S. A., Joo, C., Ha, T. Analysis of Single-Molecule FRET Trajectories Using Hidden Markov Modeling. Biophys J. 91 (5), 1941-1951 (2006).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L., Wiggins, C. H. Learning Rates and States from Biophysical Time Series: A Bayesian Approach to Model Selection and Single-Molecule FRET Data. Biophys J. 97 (12), 3196-3205 (2009).
- Greenfeld, M., Pavlichin, D. S., Mabuchi, H., Herschlag, D. Single Molecule Analysis Research Tool (SMART): an integrated approach for analyzing single molecule data. PLoS ONE. 7 (2), e30024 (2012).
- Schmid, S., Götz, M., Hugel, T. Single-Molecule Analysis beyond Dwell Times: Demonstration and Assessment in and out of Equilibrium. Biophys J. 111 (7), 1375-1384 (2016).
- Taipale, M., Jarosz, D. F., Lindquist, S. HSP90 at the hub of protein homeostasis: emerging mechanistic insights. Nat Rev Mol Cell Biol. 11 (7), 515-528 (2010).
- Trepel, J., Mollapour, M., Giaccone, G., Neckers, L. Targeting the dynamic HSP90 complex in cancer. Nat Rev Cancer. 10 (8), 537-549 (2010).
- Wayne, N., Bolon, D. N. Dimerization of Hsp90 is required for in vivo function. Design and analysis of monomers and dimers. J Biol Chem. 282 (48), 35386-35395 (2007).
- Ali, M. M. U., et al. Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex. Nature. 440 (7087), 1013-1017 (2006).
- Southworth, D. R., Agard, D. A. Species-dependent ensembles of conserved conformational states define the Hsp90 chaperone ATPase cycle. Mol Cell. 32 (5), 631-640 (2008).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proc Natl Acad Sci U S A. 101 (35), 12893-12898 (2004).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophys J. 94 (5), 1826-1835 (2008).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without pH drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Rognoni, L., Stigler, J., Pelz, B., Ylänne, J., Rief, M. Dynamic force sensing of filamin revealed in single-molecule experiments. Proc Natl Acad Sci U S A. 109 (48), 19679-19684 (2012).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in Surface-Tethered Lipid Vesicles as a New Tool for Single Biomolecule Spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Levene, M. J., Korlach, J., Turner, S. W., Foquet, M., Craighead, H. G., Webb, W. W. Zero-mode waveguides for single-molecule analysis at high concentrations. Science. 299 (5607), 682-686 (2003).
- Panaretou, B., et al. ATP binding and hydrolysis are essential to the function of the Hsp90 molecular chaperone in vivo. EMBO J. 17 (16), 4829-4836 (1998).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten