Method Article
Um método fácil para a planta polysome Profiling
Neste Artigo
Resumo
This protocol describes an easy method to extract and fractionate transcripts from plant tissues on the basis of the number of bound ribosomes. It allows a global estimate of translation activity and the determination of the translational status of specific mRNAs.
Resumo
Translation of mRNA to protein is a fundamental and highly regulated biological process. Polysome profiling is considered as a gold standard for the analysis of translational regulation. The method described here is an easy and economical way for fractionating polysomes from various plant tissues. A sucrose gradient is made without the need for a gradient maker by sequentially freezing each layer. Cytosolic extracts are then prepared in a buffer containing cycloheximide and chloramphenicol to immobilize the cytosolic and chloroplastic ribosomes to mRNA and are loaded onto the sucrose gradient. After centrifugation, six fractions are directly collected from the bottom to the top of the gradient, without piercing the ultracentrifugation tube. During collection, the absorbance at 260 nm is read continuously to generate a polysome profile that gives a snapshot of global translational activity. Fractions are then pooled to prepare three different mRNA populations: the polysomes, mRNAs bound to several ribosomes; the monosomes, mRNAs bound to one ribosome; and mRNAs that are not bound to ribosomes. mRNAs are then extracted. This protocol has been validated for different plants and tissues including Arabidopsis thaliana seedlings and adult plants, Nicotiana benthamiana, Solanum lycopersicum, and Oryza sativa leaves.
Introdução
A síntese de proteínas é um processo essencial e dispendiosa energeticamente em todas as células 1. Em primeiro lugar, as células devem investir energia na produção da maquinaria de tradução, os ribossomas. Por exemplo, uma célula de levedura dividindo ativamente produz tanto quanto 2.000 ribossomas por minuto. Essa produção exige até 60% da actividade de transcrição total e até 90% da actividade total de splicing da célula 2. Além disso, é necessária energia para a síntese de aminoácidos, aminoacil-ARNt e ligações peptídicas. Nas plantas, a adição de um aminoácido a uma cadeia peptídica custos de 4,5 a 5,9 moléculas de ATP 3. Portanto, não é surpreendente que a tradução do mRNA para a proteína é um importante local de regulação, especialmente quando se trata de lidar com a mudança de condições ambientais.
A etapa de iniciação da tradução, que é a associação de um mRNA com o ribossoma, é o principal alvo da regulamentação dosTradução 4. Como uma consequência da regulação da tradução, bem como outros passos de regulação pós-transcricional, apenas 40% das variações na concentração de proteína pode ser explicada por ARNm de abundância 5,6. Assim, o estudo de mRNA total fornece relativamente pouca informação sobre a abundância de proteínas. Por outro lado, a associação de mRNA com ribossomos dá melhor visão sobre a abundância de proteínas, dando acesso a esses mRNAs envolvidos na tradução. mRNAs activamente traduzida estão associados com vários ribossomas em estruturas chamadas polissomas. Por outro lado, mRNAs mal traduzidas será associado a apenas um ribossomo (monosome). Por conseguinte, o status translacional de um ARNm pode ser avaliado por monitorização da sua associação com ribossomas 7.
Este protocolo descreve o isolamento de polissomas a partir de seis dias de idade plântulas de Arabidopsis thaliana, o subsequente isolamento do ARN e a análise dos resultados. Polysomes monosomes e são separadas através de um gradiente de densidade de sacarose. Gradientes são coletados em seis fracções. Algumas das fracções são reunidas para obter três fracções bem separados: polissomos, monosomes ea fração leve (doravante denominado sobrenadante), que contém os 60 e 40S subunidades ribossomais livres e mRNAs que não estão associados a ribossomas. actividade de tradução global pode ser estimada através da geração de uma relação de polissoma / monosome, que é determinada por integração da área sob a curva, e por comparação dos perfis de polissomas. ARNm e as proteínas são, em seguida, extraiu-se a partir das diferentes fracções e utilizado para análise por RT-PCR, qRT-PCR, Northern blot, microarray, Western blot ou proteómica. Este protocolo foi validado para outras plantas e tecidos.
Os equipamentos necessários para realizar este protocolo são comumente encontrados na maioria dos laboratórios: Não há necessidade de uma máquina de inclinação. Congelação cada camada antes de adicionar o próximo prevenirs de qualquer mistura ou perturbação das camadas. Nenhuma perfurador tubo é utilizado para a recolha de inclinação que pode ser obtida por imersão de um tubo capilar de vidro no gradiente. Por conseguinte, os tubos de ultracentrífuga dispendiosos não ficam danificadas e podem ser re-utilizada várias vezes. Coletivamente, isso faz com que o presente protocolo de um método fácil e barato para profiling polysome.
Protocolo
1. Preparação de 20 a 50% (p / v) de sacarose Gradientes
Nota: Os gradientes são feitos de 4 camadas de sacarose (50%, 35% e 2 camadas de 20%) em 13,2 ml de um tubo de ultracentrífuga. Em nossa experiência, despejando a sacarose 20% em duas camadas separadas melhora muito a qualidade das preparações polissomas.
- Preparar as soluções de estoque. Certifique-se que todas as soluções são RNAse e DNAse livre.
- Prepare 10X Solução Salina: Tris-HCl 400 pH 8,4, KCl 200 mM e MgCl 2 100 mM.
- Prepare M de sacarose Solução 2: Para 200 ml, dissolver 137 g de sacarose em 1X solução salina.
- Dilui-se a solução de sacarose em solução salina, como descrito na Tabela 1, para preparar os gradientes. Os volumes indicados são para seis gradientes.
| Concentração de sacarose final | sacarose 2H (ml) | Sal solução 1X (ml) | Vol final. (ml) |
| 50% | 8,8 | 3.2 | 12 |
| 35% | 12,9 | 12.1 | 25 |
| 20% | 7,4 | 17,6 | 25 |
| 20% | 5.8 | 14.2 | 20 |
Tabela 1. As diluições da solução de sacarose para preparar seis gradientes.
- Despeje as camadas conforme Tabela 2.
| camada de sacarose | 50% | 35% | 20% | 20% |
| Vol. (Ml) | 1.85 | 3.65 | 3.65 | 1,35 |
Tabela 2. Volume de solução de sacarose por camada.
- Depois de verter cada camada, manter os tubos num -40 ° C ou -80 ° C até estar completa congelador de congelação antes da adição de sacarose a camada seguinte. A congelação é geralmente alcançada após 2 h a -40 ° C, mas de esperar cerca de 6 h antes de se verter a camada seguinte é recomendado. A última camada de 20% pode ser congelada ou adicionado de fresco no dia da experiência.
Nota: Congelamento cada camada antes de adicionar o próximo impede a partir de qualquer mistura ou perturbação das camadas. Os gradientes podem ser mantidos numa -40 ° C ou -80 ° C congelador durante pelo menos seis meses. - Se ainda não tiver sido feito, adicionar a última camada de 20% para os gradientes no dia da experiência. Então, deixe-os gradientes descongelar em uma sala fria ou um frigorífico.
2. Preparação de Extratos citossólicos
Nota: Nós recomendaçãacabar usando dois gradientes por amostra biológica. 300 mg é a quantidade óptima de material vegetal para preparar dois gradientes quando se trabalha com 6 dias de idade plântulas de Arabidopsis thaliana. Quando se trabalha com tecidos traducionalmente menos activos, a quantidade de material de planta pode ser aumentada até 600 mg.
- Colheita de seis dias da germinação cultivadas em ½ Murashige e Skoog 8 meio suplementado com 1% de sacarose ou mídia equivalente a-congelação rápida em azoto líquido
- material vegetal moer num almofariz e pilão pré-arrefecida com azoto líquido.
- Pesar 300 mg de material em pó em um prato de pesagem pré-arrefecido. Execute esta etapa rapidamente para evitar o descongelamento da amostra.
Nota: Mantenha as amostras congeladas por mais de uma semana em um C congelador -80 °. - Adicionar 2,4 ml de tampão pré-arrefecida polissoma (Solução Salina 4X, EGTA 5,26 mM, 0,5% (v / v), octilfenoxi poli (etilenoxi) etanol, ramificadas, 50 μg.ml -1 ciclo-heximida, 50 1 μg.mlcloranfenicol) e homogeneizar por mistura com a ponta da pipeta. Transferir 1,5 ml para dois tubos. Trabalhar rapidamente para impedir que as amostras de aquecimento.
- Centrifuga-se a 16000 xg durante 15 min a 4 ° C numa microcentrífuga para sedimentar os detritos.
Nota: Para evitar a degradação do RNA, mantenha sempre as amostras a 4 ° C, utilize RNase / DNase soluções livres e trabalhar em RNase / DNase condições livres. A heparina pode ser adicionado ao tampão de polissoma, a uma concentração final de 300 μg.ml -1, para melhorar a protecção de ARN. No entanto, uma vez que a heparina pode interferir com a análise a jusante, que terá de ser removido durante o passo de precipitação de ARN através da realização de precipitação de cloreto de lítio em vez de precipitação com etanol (nota cf. 4.7).
3. polysome Profiling
- Cuidadosamente pipeta o sobrenadante sem perturbar o sedimento. Se fragmentos de plantas foram pipetados, repita o passo 2.5. Carregar o sobrenadante no topo do gradiente por pipettin suavementeg para a parede lateral do tubo num fluxo constante. Use um gradiente para cada tubo de 1,5 ml.
- Transferência para o pré-arref ecido baldes e centrifugar a 175.000 xg durante 2 h 45 min a 4 ° C, em uma ultracentrífuga (por exemplo 32.000 rpm ao utilizar um rotor SW41).
- Configurar o sistema de recolha de gradiente, tal como descrito na Figura 1. A cuveta UV tem um percurso de 1 mm.

Figura 1. Sistema de recolha de inclinação. A cuvete de UV está ligado um tubo de cloreto de polivinilo a um tubo capilar de vidro, que desce para o fundo do gradiente. O gradiente progride através do sistema graças a uma bomba peristáltica. OD 260 é continuamente lido e 2 ml frações são coletadas. Por favor clique aqui para ver uma versão maioresta figura.
- Ajustar o colector de fracções para a TA, e definir o carrossel, a uma velocidade que permita a recolha de fracções de 2 mL. Use tubos de coleta pré-arrefecido.
- Recolher as fracções de baixo para cima utilizando um tubo capilar de vidro ligada a um tubo de cloreto de polivinilo para o sistema de recolha de gradiente. Usar um filme de parafina de plástico para assegurar a ligação entre o tubo de cloreto de polivinilo e o tubo capilar de vidro.
- Ler a absorvância a 260 nm a partir do fundo para o topo do gradiente. Quando todo o gradiente é recolhido, coloque os tubos de coleta no gelo antes de extração de RNA.
4. Extracção de ARN
- Reunir as fracções recolhidas a partir de dois gradientes, em 50 ml tapados tubos de centrifugação, como segue:
Polissomas: fracções de 1 a 3 (12 ml)
Monosomes: valor correspondente a 4 (4 ml)
Sobrenadante: fracções 5 e 6 (8 ml) - Para cada fracção, adicione 1 vol. Hidrocloreto de guanidina 8M,50 ug transportador acrílico e 1,5 vol. isopropanol.
Nota: Acryl transportador é acrilamida linear, usado como um co-precipitante para melhorar a recuperação de ácidos nucleicos durante a precipitação com álcool. - Misturar por inversão dos tubos e precipitar O / N à temperatura de -20 ° C.
- Centrifugar a 175.000 xg durante 1 h a 4 ° C, em uma ultracentrífuga (32.000 rpm num rotor SW32). Se o passo de precipitação é realizada de um tubo, o qual não é compatível com a ultracentrifugação, transferir para um tubo adequado.
- Elimine o sobrenadante e dissolve-se o sedimento em 200 de tampão TE isento de ARNase ul (Tris-HCl 10 mM, pH 7,5-1 mM de EDTA). Transferir para um novo tubo de 1,5 ml.
- Extrair ARN por adição de 1 vol. fenol saturado de água (pH 6,6) e 1 vol. clorofórmio: álcool isoamílico (24: 1). Misturar energicamente. Centrifuga-se a 15000 xg durante 20 min a 4 ° C. Transferir a fase aquosa para um novo tubo de 1,5 mL.
Nota: ácido fenol (pH 4,5) pode ser utilizado a fim de minimizar a contaminação do ADN. - Precipitar o ARN por adição de 1/10 vol. M de acetato de sódio 3 e 3 vol. 100% de etanol. Permitir a precipitação a -80 ° C durante 20 min ou O / N à temperatura de -20 ° C. Centrifuga-se a 15000 xg durante 15 min a 4 ° C.
Nota: Se estiver usando heparina no tampão polysome (nota cf. 2.5), execute um cloreto de lítio precipitação (LiCl) em vez de precipitação de etanol para remover adequadamente qualquer vestígio de heparina que possa interferir com a análise a jusante 9. Após extracção, adicionar LiCl até uma concentração final de 2,5 M. permitir a precipitação durante 30 minutos à temperatura de -20 ° C ou O / N a 4 ° C. Centrifuga-se a 15000 xg durante 15 min a 4 ° C. - Lavar o sedimento com 500 ul de etanol a 75%. Ar secar o sedimento e dissolvem-se em 30 ul de tampão TE. Avaliar a qualidade do ARN por electroforese num ARNase livre de 1,2% em gel de agarose (100V, 15 min) ou por métodos de electroforese capilar.
Análise 5. Os dados
- Exportação de dados brutos para um software gráfico e de análise de dados.
Nota: Como mostrado na Figura 2A e B, os perfis de matérias dar informação qualitativa: o número de picos polissomas que pode ser visto, a altura do pico monosome, e a presença de um ombro correspondente para as subunidades de ribossomas livres. - Mesclar perfis para permitir a comparação dos perfis entre as amostras. Utilizar a ferramenta de leitor de tela para determinar o valor de X do pico monosome para cada amostra e alinhar os picos monosome removendo pontos de dados adicionais no início das curvas. Identificar o ponto mais baixo da curva e determinar seu valor Y (utilizando a ferramenta de leitor de tela). Defina o valor mais baixo Y ponto a 0 usando a função "valor definido colunas".
- Determinar a proporção polissomas / monosomes por integração da área sob a curva de perfis em bruto (Figura 3C). Utilizar a ferramenta selector de dados para definir a fronteira da área a ser integrados. Em seguida, use a função de integrar (Análise-Matemática).
- Normalizaras curvas para o pico monosome dividindo todos os pontos de dados de uma curva pelo valor Y da cimeira do pico monosome (determinado utilizando a ferramenta de leitor de tela). Selecione a coluna, então sob análise em Matemática, selecione Normalize e escolher os "dividido por um valor especificado" métodos. Este passo permite a comparação do nível relativo de polissomas (Figura 3D).
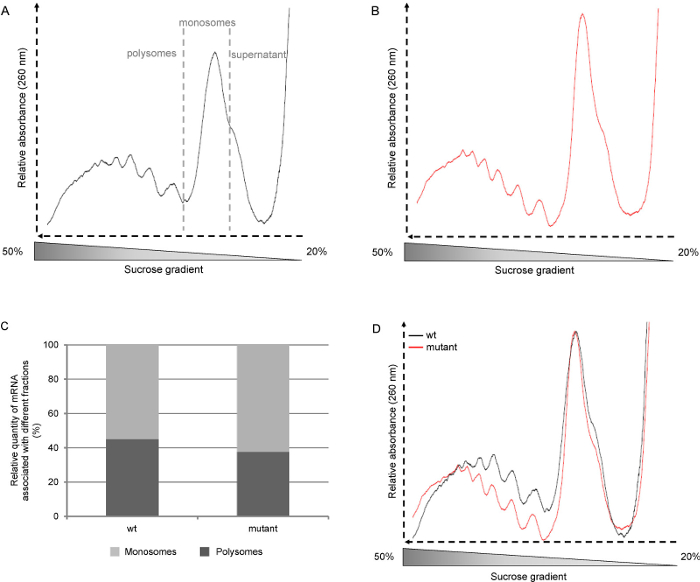
Figura 2. Perfis polissomas representativos. Arabidopsis thaliana de tipo selvagem (wt, ecotipo Col-0) e mudas mutantes foram cultivadas durante seis dias em ½ Murashige e Skoog sob fotoperíodos o dia (luz 16 horas, 8 horas escuro). A: perfis polissomas brutos de mudas wt. B: perfis polissomas brutos de mudas mutantes. C: Determinação da percentagem de polissomas e monosomes por integração da área sob a curva D: profi polissomales normalizados para o pico monosome. Por favor clique aqui para ver uma versão maior desta figura.
Resultados
Na literatura, os perfis de polissomas são mostrados frequentemente a partir da fracção de luz para a fracção pesada como um resultado da forma como os gradientes são recolhidos, ou seja, a partir do topo para o fundo. Uma vez que no protocolo aqui descrito os gradientes são recolhidos a partir da base para o topo, os perfis que mostram começar com a fração pesada (as polissomos) e vá para a fração de luz (subunidades de ribossomas livres e RNAs) (Figura 2A). Em seguida, recolher cada gradiente em seis fracções de 2 ml, mas fracções de menor dimensão pode ser recolhida se uma análise mais detalhada do conteúdo polissoma tem de ser levada a cabo.
Mesclando e normalizando as curvas para o pico monosome (Figura 2D) permite a comparação de perfis de diferentes linhas ou condições de crescimento. Este fornece informação sobre a quantidade relativa de polissomas, independentemente do nível de iniciação. Outra forma de umnalyze os perfis é para calcular a área sob a curva, assim, pode-se determinar a quantidade relativa de ARNm associado tanto com as monosomes ou os polissomas (Figura 2C). Esta relação é específico de uma planta de crescimento e condições. No entanto, esta abordagem pode não ser relevantes no caso de tecidos em translação activo fracamente.
Temos utilizado este método para A. plantas inteiras thaliana, rosetas jovens e idosos, bem como para a N. benthamiana (Figura 3C), S. lycopersicum (Figura 3E) e O. folhas de sativa (Figura 3D). A forma do perfil depende das condições de crescimento, idade da planta e os tecidos analisados. Aqui usamos A. thaliana seis dias mudas de idade. Nesta fase, a actividade de translação é alta, e o perfil mostra picos bem perfilada (Figura 2A e B). Este é também o casoquando A. thaliana flores são usadas (Figura 3A). Ao usar 4 semanas de idade A. thaliana rosetas (Figura 3B), as amostras contêm principalmente deixa adulto totalmente desenvolvido, onde as células não se dividem. Assim, o montante global de polissomos e monosomes é menor. O perfil mostra menos, mas ainda bem em forma de picos polissomas. Próximo ao pico monosome, o ombro mostra a grande quantidade de 60S livres subunidades ribossomais. Com outro tipo de plantas ou tecidos, os picos polissomas pode ser pouco visível. Este é o caso quando se usa 30 dias de idade O. folhas de sativa (Figura 3D). Mesmo quando a quantidade de ARNm envolvidos na tradução é muito baixo (nenhum pico polissoma pode ser visto em perfil), a presença do pico monosome indica que o fraccionamento foi adequadamente realizado e que os ARNm pode ser, posteriormente, extraída a partir da fracção para análise posterior . A qualidade da qualidade do RNA extraído é avaliada por electroforese em gel de agarose ( ong> Figura 4). O 25S e 18S RNA ribossomal citosólica deve ser claramente visível no gel. Bandas inferior correspondente ao RNA ribossomal cloroplasmático deve também ser visível quando a ARN são extraídos a partir de tecidos verdes 10.
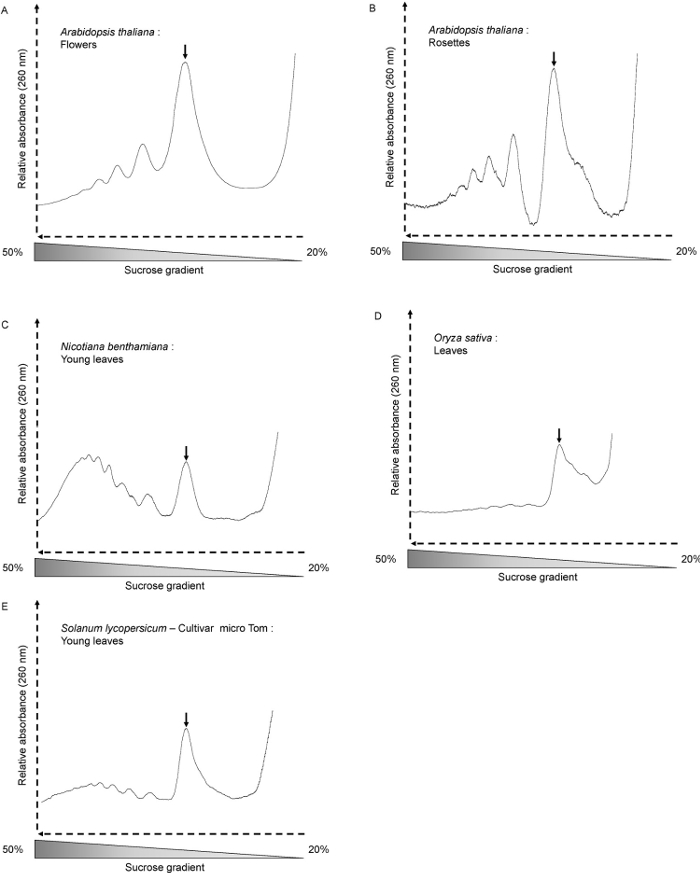
Figura 3. perfis polysome de diferentes materiais e espécies vegetais. (A) flores Arabidopsis thaliana (300 mg), (B) de Arabidopsis thaliana 4 semanas de idade rosetas (600mg), benthamiana (C) Nicotiana (folhas jovens de 40 dias as plantas cultivadas-dia curto de idade - 300mg), (D) Oryza sativa ( folhas de plantas de 30 dias de idade - 300mg), (E) Solanum lycopersicum (folhas jovens de 35 dias as plantas cultivadas-dia curto de idade - 300mg). Os picos monosome são indicados por setas.iles / ftp_upload / 54231 / 54231fig3large.jpg "target =" _ blank "> Clique aqui para ver uma versão maior desta figura.
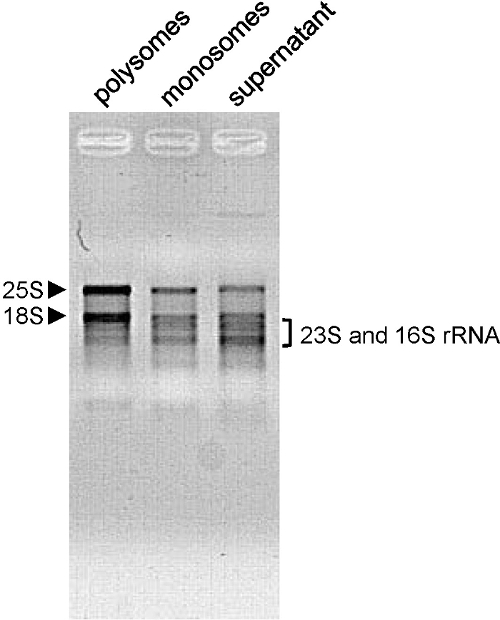
. Figura 4. Avaliação da qualidade do ARN ARN (500 ng extraída a partir de rebentos de 6 dias de idade plântulas de Arabidopsis thaliana) a partir das fracções indicadas foram aplicadas num gel de agarose a 1,2% e separados por electroforese (100V - 15 min). Citosólicas (25S e 18S) rRNA são indicados por setas e cloroplasmático (23S e 16S) rRNA por um suporte. Por favor clique aqui para ver uma versão maior desta figura.
Discussão
The protocol we present here is an easy and cheap method for generating polysome profiles and isolating mRNAs associated with polysomes, single ribosomes or free of ribosomes. A wide range of different polysome fractionation methods is described in the literature. The method we have described here has been optimized to keep only the necessary compounds and has been adapted for plant material. In particular, we reduced the amount of detergent11 and added chloramphenicol to the buffer to fix the chloroplastic ribosomes to the mRNA (as cycloheximide does for the cytosolic ribosomes)12 . We have also reduced the total ultracentrifugation time7.
To obtain high quality polysome profiles, it is essential to use freshly collected plant material and to perform all steps at 4°C. When using tissues that are poorly translationally active, more plant material can be loaded on the gradient (up to 600 mg).
Analysis of polysome profiles can provide insights into both the overall translational status of cells13 and the translational status of a specific mRNA. We have used RNAs isolated by this method for different applications. Using the RNAs for microarrays allowed us to identify a class of cadmium stress response genes for which transcription and translation are uncoupled 14. We also used this method to identify small RNAs associated with polysomal fractions. This identification was made by northern blotting15 of RNA extracted from polysomal fractions, and provided biochemical evidence for a translational component in the miRNA pathway in plants. In another study, a cis-NAT RNA was identified by quantitative RT-PCR. This cis-NAT is associated to the phosphate homeostasis and promote translation of the PHO1;2 transcript 16.
The main limits of the polysome profiling approach are the lack of information concerning both the position of the ribosome on the mRNA and its progression along the mRNA. Ribosome profiling has emerged to address these limitations 17 and has been successfully used on plant tissues18. Ribosome profiling provides a global measurement of translation by taking advantage of the advances in sequencing technology. Nevertheless, as for any sequencing based assays, the quality of the results depends on the mapping of the sequences to the genome, therefore focusing on the small ribosome-protected fragments makes it difficult to deconvolute repetitive sequences. Moreover, the digestion of RNA not protected by the ribosomes leads to the loss of regulatory information contained in the 3' and 5' UTRs. It is then impossible to distinguish transcript variants that have different 3' or 5' UTRs and show different levels of translation19. The polysome profiling method described here is rapid and does not require specific technical skills. Altogether, these two methods represent complementary approaches to study translation regulation.
Divulgações
The authors have nothing to disclose
Agradecimentos
Este trabalho foi apoiado pela Agência Nacional de Investigação francesa (ANR-14-CE02-0010). Agradecemos Dr. Benjamin Campo e Dr. Elodie Lanet para a leitura crítica do manuscrito. Agradecemos ao Sr. Michel Terese por sua ajuda com edição de vídeo.
Materiais
| Name | Company | Catalog Number | Comments |
| Ultracentrifuge tube, thinwall, polyallomer - 13.2 ml | Beckman Coulter | 331372 | |
| Ultracentrifuge tube, thinwall, polyallomer - 38.5 ml | Beckman Coulter | 326823 | |
| Glass capillary tube | Drummond Scientific | 1-000-1000 | |
| Ultracentrifuge | Beckman Coulter | Optima series | |
| Ultracentrifuge Rotor SW41 | Beckman Coulter | 331362 | |
| Ultracentrifuge Rotor SW32 | Beckman Coulter | 369650 | |
| Peristaltic pump | Any | ||
| Tygon R3607 polyvinyl chloride tubing | Fisher Scientific | 070534-22 | Polyvinyl chloride tubing, 2.29 mm |
| Fraction collector Model 2110 | Bio-Rad | 731-8120 | |
| UV cuvette | Hellma | 170.700-QS | Quartz flow-through cuvette |
| UV Spectrophotometer | Varian | Cary50 | Read every 0.0125 sec |
| All chemicals | Any | Use only Molecular Biology Grade | |
| Murashige and Skoog Basal Salt Mixture (MS) | Sigma-Aldrich | M5524 | |
| Rnase-Free water | Any | ||
| Petri Dishes | Fisher Scientific | 10083251 | |
| Octylphenoxy poly(ethyleneoxy)ethanol, branched (Nonidet P40) | Euromedex | UN3500 | |
| Linear acrylamide (acryl carrier) | ThermoFischer scientific | AM9520 | RNA precipitation carrier |
| OriginPro 8 | OriginLab | Analysis software |
Referências
- Nelson, C. J., Millar, A. H. Protein turnover in plant biology. Nat Plants. 1 (3), 15017 (2015).
- Warner, J. R. The economics of ribosome biosynthesis in yeast. Trends Biochem Sci. 24 (11), 437-440 (1999).
- Amthor, J. S. The McCree-de Wit-Penning de Vries-Thornley Respiration Paradigms: 30 Years Later. Ann Bot. 86 (1), 1-20 (2000).
- Preiss, T., W Hentze, M. Starting the protein synthesis machine: eukaryotic translation initiation. BioEssays news and reviews in molecular, cellular and developmental biology. 25 (12), 1201-1211 (2003).
- Vogel, C., Marcotte, E. M. Insights into the regulation of protein abundance from proteomic and transcriptomic analyses. Nat Rev Genet. 13 (4), 227-232 (2013).
- Baerenfaller, K., et al. Genome-scale proteomics reveals Arabidopsis thaliana gene models and proteome dynamics. Science. 320 (5878), 938-941 (2008).
- Zanetti, E., Chang, I., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of Polyribosomal Complexes of Arabidopsis for Global Analysis of Gene Expression. Plant physiol. 138 (2), 624-635 (2005).
- Murashige, T., Skoog, F. A revised medium for rapid growth and bioassays with tobacco tissue cultures. Physiol plant. 15 (3), 473-497 (1962).
- del Prete, M. J., Vernal, R., Dolznig, H., Müllner, E. W., Garcia-Sanz, J. A. Isolation of polysome-bound mRNA from solid tissues amenable for RT-PCR and profiling experiments. RNA. 13 (3), 414-421 (2007).
- Salinas, J., Sanchez-Serrano, J. J. . Arabidopsis protocols. , (2006).
- Piques, M., et al. Ribosome and transcript copy numbers, polysome occupancy and enzyme dynamics in Arabidopsis. Mol syst biol. 5 (314), 314 (2009).
- Morita, M., Alain, T., Topisirovic, I., Sonenberg, N. Polysome Profiling Analysis. bio-protocol. 3 (14), 3-8 (2013).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS One. 8 (8), (2013).
- Sormani, R., et al. Sublethal cadmium intoxication in Arabidopsis thaliana impacts translation at multiple levels. Cell Physiol. 52 (2), 436-447 (2011).
- Lanet, E., et al. Biochemical evidence for translational repression by Arabidopsis microRNAs. Plant cell. 21 (6), 1762-1768 (2009).
- Jabnoune, M., Secco, D., Lecampion, C., Robaglia, C., Shu, Q., Poirier, Y. A Rice cis-Natural Antisense RNA Acts as a Translational Enhancer for Its Cognate mRNA and Contributes to Phosphate Homeostasis and Plant Fitness. Plant cell. 25 (10), 4166-4182 (2013).
- Ingolia, N. T. Genome-wide translational profiling by ribosome footprinting. Methods Enzymol. 470 (10), (2010).
- Juntawong, P., Girke, T., Bazin, J., Bailey-Serres, J. Translational dynamics revealed by genome-wide profiling of ribosome footprints in Arabidopsis. PNAS. 111 (1), 203-212 (2013).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nat Rev Genet. 15 (3), 205-213 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados