Method Article
Un método fácil para la planta de perfiles de polisomas
En este artículo
Resumen
This protocol describes an easy method to extract and fractionate transcripts from plant tissues on the basis of the number of bound ribosomes. It allows a global estimate of translation activity and the determination of the translational status of specific mRNAs.
Resumen
Translation of mRNA to protein is a fundamental and highly regulated biological process. Polysome profiling is considered as a gold standard for the analysis of translational regulation. The method described here is an easy and economical way for fractionating polysomes from various plant tissues. A sucrose gradient is made without the need for a gradient maker by sequentially freezing each layer. Cytosolic extracts are then prepared in a buffer containing cycloheximide and chloramphenicol to immobilize the cytosolic and chloroplastic ribosomes to mRNA and are loaded onto the sucrose gradient. After centrifugation, six fractions are directly collected from the bottom to the top of the gradient, without piercing the ultracentrifugation tube. During collection, the absorbance at 260 nm is read continuously to generate a polysome profile that gives a snapshot of global translational activity. Fractions are then pooled to prepare three different mRNA populations: the polysomes, mRNAs bound to several ribosomes; the monosomes, mRNAs bound to one ribosome; and mRNAs that are not bound to ribosomes. mRNAs are then extracted. This protocol has been validated for different plants and tissues including Arabidopsis thaliana seedlings and adult plants, Nicotiana benthamiana, Solanum lycopersicum, and Oryza sativa leaves.
Introducción
La síntesis de proteínas es un proceso esencial y energéticamente costoso en todas las células 1. En primer lugar, las células deben invertir energía en la producción de la maquinaria de traducción, los ribosomas. Por ejemplo, una célula de levadura dividiendo activamente produce tanto como 2000 ribosomas por minuto. Una producción de este tipo requiere de hasta 60% de la actividad transcripcional total y hasta 90% de la actividad total de empalme de la célula 2. Además, se requiere energía para la síntesis de aminoácidos, aminoacil-tRNA y enlaces peptídicos. En las plantas, la adición de un aminoácido a un costos de la cadena de péptido de 4,5 a 5,9 moléculas de ATP 3. Por lo tanto, no es sorprendente que la traducción de ARNm de la proteína es un sitio importante de la regulación, en particular cuando se trata de lidiar con el cambio de las condiciones ambientales.
La etapa de iniciación de la traducción, que es la asociación de un ARNm con el ribosoma, es el principal objetivo de la regulación deTraducción 4. Como consecuencia de la regulación de la traducción, así como otras medidas de regulación post-transcripcional, sólo el 40% de las variaciones en la concentración de proteína se puede explicar por la abundancia de ARNm 5,6. Por lo tanto, el estudio de ARNm total de da relativamente pobre información sobre la abundancia de proteínas. Por otra parte, la asociación de ARNm con ribosomas da una mejor penetración en la abundancia de proteínas, dando acceso a esos mRNAs implicados en la traducción. mRNAs traducidos de forma activa se asocian con varios ribosomas en estructuras llamadas polisomas. Por el contrario, los ARNm mal traducidas estarán asociadas con un solo ribosoma (monosome). En consecuencia, el estado de la traducción de un ARNm puede ser evaluada mediante el control de su asociación con ribosomas 7.
Este protocolo describe el aislamiento de polisomas procedentes de seis días plántulas de Arabidopsis thaliana, el posterior aislamiento de RNA, y el análisis de los resultados. Correoslysomes y monosomas se separan a través de un gradiente de densidad de sacarosa. Los gradientes se recogen en seis fracciones. Algunas de las fracciones se agrupan para obtener tres fracciones separadas así: polisomas, monosomas y la fracción ligera (de aquí en adelante llamado sobrenadante), que contiene los libres 60S y 40S ribosomal subunidades y mRNAs que no están asociados con los ribosomas. actividad de traducción Global se puede estimar mediante la generación de un / relación de polisomas monosome, que se determina por la integración del área bajo la curva, y mediante la comparación de los perfiles de polisomas. mRNAs y proteínas se extraen a partir de las diferentes fracciones y se utilizaron para el análisis por RT-PCR, QRT-PCR, transferencia de Northern, microarray, Western blot o la proteómica. Este protocolo ha sido validado para otras plantas y tejidos.
El equipo necesario para llevar a cabo este protocolo se encuentran comúnmente en la mayoría de los laboratorios: No hay necesidad de un fabricante de gradiente. Congelación de cada capa antes de añadir el siguiente prevenirs de cualquier mezcla o perturbación de las capas. No perforador tubo se utiliza para la recogida de gradiente que puede lograrse mediante la inmersión de un tubo capilar de vidrio en el gradiente. Por lo tanto, los tubos de ultracentrífuga costosos permanecen sin daños y se pueden volver a utilizar muchas veces. En conjunto, esto hace que el presente protocolo de un método sencillo y barato para los perfiles de polisomas.
Protocolo
1. Preparación de 20 a 50% (p / v) de sacarosa gradientes
Nota: Los gradientes son de 4 capas de sacarosa (50%, 35% y el 2 capas de 20%) en un tubo de ultracentrífuga 13.2 ml. En nuestra experiencia, el vertido de la sacarosa al 20% en dos capas separadas mejora en gran medida la calidad de las preparaciones polisomas.
- Preparar las soluciones madre. Asegúrese de que todas las soluciones son de RNAsa y DNAsa.
- Preparar 10X Solución Salina: mM Tris-HCl pH 8,4 400, KCl 200 mM y MgCl 100 mM 2.
- Preparar la solución 2 M de sacarosa: Por 200 ml, se disuelven 137 g de sacarosa en 1X solución salina.
- Diluir la solución de sacarosa en Salt Solution, como se describe en la Tabla 1, para preparar los gradientes. Los volúmenes indicados se refieren a seis gradientes.
| La concentración de sacarosa final | sacarosa 2M (ml) | La solución salina que 1X (ml) | Vol Final. (ml) |
| 50% | 8.8 | 3.2 | 12 |
| 35% | 12.9 | 12.1 | 25 |
| 20% | 7.4 | 17.6 | 25 |
| 20% | 5.8 | 14.2 | 20 |
Tabla 1. Las diluciones de la solución de sacarosa para preparar seis gradientes.
- Verter las capas según la Tabla 2.
| capa de sacarosa | 50% | 35% | 20% | 20% |
| Vol. (Ml) | 1.85 | 3.65 | 3.65 | 1.35 |
Tabla 2. Volumen de la solución de sacarosa por capa.
- Después de verter cada capa, mantener los tubos en un -40 ° C o -80 ° C congelador hasta congelación completa antes de añadir la siguiente capa de sacarosa. La congelación se consigue normalmente después de 2 horas a -40 ° C, pero esperando sobre 6 hr antes de verter la siguiente capa se recomienda. La última capa 20% se puede congelar o añadido recientemente en el día del experimento.
Nota: Congelación cada capa antes de añadir el siguiente impide cualquier mezcla o perturbación de las capas. Los gradientes se pueden mantener en un -40 ° C o -80 ° C congelador durante al menos seis meses. - Si ya no se ha hecho, añadir la última capa 20% a los gradientes en el día del experimento. A continuación, dejar que los gradientes se descongelan en una habitación fría o una nevera.
2. Preparación de extractos citosólicos
Nota: Se recomterminar utilizando dos gradientes por muestra biológica. 300 mg es la cantidad óptima de material vegetal para preparar dos gradientes cuando se trabaja con 6 días plántulas de Arabidopsis thaliana. Cuando se trabaja con tejidos menos translationally activos, la cantidad de material de la planta se puede incrementar hasta 600 mg.
- Cosecha de seis días de edad plántulas cultivadas en ½ Murashige y Skoog 8 medio suplementado con 1% de sacarosa o medios equivalentes por congelación rápida en nitrógeno líquido
- material vegetal molido en un mortero y una maja previamente enfriada con nitrógeno líquido.
- Pesar 300 mg de material en polvo en un plato de pesada previamente enfriado. Realice este paso rápidamente para evitar la descongelación de la muestra.
Nota: Mantenga las muestras congeladas por no más de una semana en un congelador a -80 °. - Añadir 2,4 ml de tampón de polisomas preenfriada (sal Solución 4X, EGTA 5,26 mM, 0,5% (v / v) octilfenoxi poli (etilenoxi) etanol, ramificados, 50 ìg.mL -1 cicloheximida, 50 ìg.mL 1cloranfenicol) y homogeneizar mediante la mezcla con la punta de la pipeta. Transferir 1,5 ml en dos tubos. Trabajar con rapidez para evitar que las muestras de calentamiento.
- Centrifugar a 16.000 xg durante 15 min a 4 ° C en una microcentrífuga para sedimentar los desechos.
Nota: Para evitar la degradación del ARN, mantener siempre las muestras a 4 ° C, utilizar las soluciones y de trabajo libre de RNasa / DNasa en RNasa / DNasa condiciones libres. La heparina puede ser añadido a la memoria intermedia de polisomas, a una concentración final de 300 ìg.mL -1, para mejorar la protección de ARN. Sin embargo, ya que la heparina puede interferir con análisis de aguas abajo, que tendrá que ser retirado durante la etapa de precipitación del ARN mediante la realización de la precipitación de cloruro de litio en lugar de la precipitación con etanol (cf. nota 4,7).
3. Perfiles de polisomas
- Con cuidado la pipeta el sobrenadante sin perturbar el sedimento. Si los fragmentos de plantas han sido con pipeta, repita el paso 2.5. Cargar el sobrenadante en la parte superior del gradiente por pipettin suavementeg sobre la pared lateral del tubo en una corriente constante. Utilice uno de gradiente para cada tubo de 1,5 ml.
- Transferencia a preenfriado cubos y se centrifuga a 175.000 xg durante 2 hr 45 min a 4 ° C, en una ultracentrífuga (por ejemplo 32.000 rpm utilizando un rotor SW41).
- Configurar el sistema de recolección de gradiente tal como se describe en la Figura 1. La cubeta UV tiene una longitud de trayectoria de 1 mm.

Figura 1. sistema de recogida de gradiente. La cubeta UV está conectado mediante un tubo de cloruro de polivinilo a un tubo capilar de vidrio que desciende hasta el fondo del gradiente. El gradiente avanza en el sistema gracias a una bomba peristáltica. DO 260 es leído de forma continua y se recogen fracciones de 2 ml. Por favor, haga clic aquí para ver una versión más grande deesta figura.
- Ajuste el colector de muestras hasta la temperatura ambiente, y establecer el carrusel a una velocidad que permita la recogida de fracciones de 2 ml. Use tubos pre-enfriado de recogida.
- Reunir las fracciones de abajo hacia arriba usando un tubo capilar de vidrio conectada mediante un tubo de cloruro de polivinilo al sistema de recolección de gradiente. Utilizar una película de parafina de plástico para asegurar la conexión entre el tubo de cloruro de polivinilo y el tubo capilar de vidrio.
- Leer la absorbancia a 260 nm de la parte inferior a la parte superior del gradiente. Cuando se recoge todo el degradado, colocar los tubos de recogida en el hielo antes de la extracción de RNA.
4. La extracción de RNA
- Reunir las fracciones recogidas a partir de dos gradientes, en tapón de 50 ml tubos de centrífuga, como sigue:
Polisomas: fracciones 1 a 3 (12 ml)
Monosomas: fracción 4 (4 ml)
De sobrenadante: fracciones 5 y 6 (8 ml) - Para cada fracción, se añade 1 vol. hidrocloruro de guanidina 8M,50 g de soporte acrílicas y de 1,5 vol. isopropanol.
Nota: portador Acryl es acrilamida lineal, que se utiliza como un coprecipitante de mejorar la recuperación de ácidos nucleicos durante la precipitación de alcohol. - Mezclar invirtiendo los tubos y precipitar O / N a -20 ° C.
- Centrifugar a 175.000 xg durante 1 hora a 4 ° C, en una ultracentrífuga (32.000 rpm en un rotor SW32). Si la etapa de precipitación se lleva a cabo en un tubo que no es compatible con la ultracentrifugación, transferir a un tubo adecuado.
- Desechar el sobrenadante y disolver el precipitado en 200 l de tampón TE libre de ARNasa (Tris-HCl 10 mM pH 7,5 a 1 mM de EDTA). Transferir a un nuevo tubo de 1,5 ml.
- Extracto de ARN mediante la adición de 1 vol. fenol saturado de agua (pH 6,6) y 1 vol. cloroformo: alcohol isoamílico (24: 1). Mezclar vigorosamente. Centrifugar a 15.000 xg durante 20 min a 4 ° C. Transferir la fase acuosa a un nuevo tubo de 1,5 ml.
Nota: fenol ácido (pH 4.5) se puede utilizar con el fin de minimizar la contaminación de ADN. - Precipitar el ARN mediante la adición de un décimo vol. acetato de sodio 3 M y 3 vol. 100% de etanol. Permitir la precipitación a -80 ° C durante 20 minutos o O / N a -20 ° C. Centrifugar a 15.000 xg durante 15 min a 4 ° C.
Nota: Si se utiliza heparina en el tampón de polisomas (véase la nota 2.5), realizar una precipitación de cloruro de litio (LiCl) en lugar de la precipitación con etanol para eliminar adecuadamente cualquier rastro de heparina que pueda interferir con el análisis de aguas abajo 9. Después de la extracción, añadir LiCl a una concentración final de 2,5 M. permitir la precipitación durante 30 min a -20 ° C o O / N a 4 ° C. Centrifugar a 15.000 xg durante 15 min a 4 ° C. - Lavar el precipitado con 500 l 75% de etanol. Deje secar al aire el precipitado y se disuelve en 30 l de tampón TE. Evaluar la calidad del RNA por electroforesis en un gel de agarosa libre de 1,2% de ARNasa (100 V, 15 min) o por métodos de electroforesis capilar.
Análisis 5. Datos
- Exportar datos en bruto a un software gráfico y análisis de datos.
Nota: Como se muestra en la Figura 2A y B, los perfiles primas dan información cualitativa: el número de picos polisomas que se puede ver, la altura del pico de monosome, y la presencia de un hombro correspondiente a las subunidades de ribosomas libres. - Combinar perfiles para permitir la comparación de los perfiles entre las muestras. Utilice la herramienta de lector de pantalla para determinar el valor X del pico monosome para cada muestra y alinear los picos monosome mediante la eliminación de puntos de datos adicionales en el inicio de las curvas. Identificar el punto más bajo de la curva y determinar su valor de Y (con la función de lector de pantalla). Establecer el valor de Y punto más bajo a 0 mediante el uso de la función "valor de columnas del conjunto".
- Determine la relación polisomas / monosomas integrando el área bajo la curva a partir de perfiles en bruto (Figura 3C). Utilice la herramienta de selección de datos para definir la frontera de la zona a integrarse. A continuación, utilice la función de integrar (Análisis-Matemáticas).
- Normalizarlas curvas a la cima monosome dividiendo todos los puntos de datos de una curva por el valor Y de la cumbre del pico monosome (determinaron utilizando la herramienta de lector de pantalla). Seleccione la columna, a continuación, en Análisis-Matemáticas, seleccione Normalizar y elegir las "dividido por un valor especificado" métodos. Este paso permite la comparación del nivel relativo de polisomas (Figura 3D).
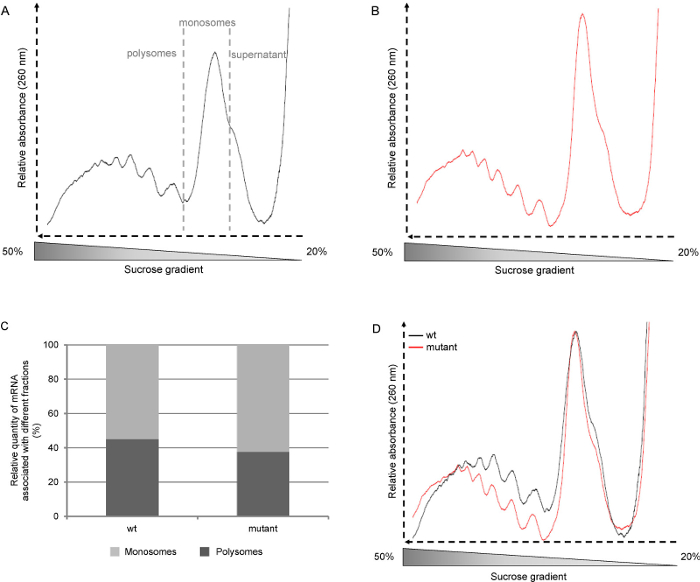
Figura 2. Perfiles de polisomas representativos. Arabidopsis thaliana de tipo silvestre (wt, ecotipo Col-0) y plántulas mutantes se cultivaron durante seis días en ½ Murashige y Skoog bajo fotoperiodos el día (16 horas de luz, 8 horas de oscuridad). R: perfiles de polisomas primas procedentes de las plantas de semillero en peso. B: Los perfiles de polisomas primas de plántulas mutantes. C: Determinación del porcentaje de polisomas y monosomas por integración de la área bajo la curva D: Profi polisomasles normalizó al pico monosome. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Resultados
En la literatura, los perfiles de polisomas a menudo se muestran a partir de la fracción ligera de la fracción pesada como resultado de la forma en que se recogen los gradientes, es decir, desde la parte superior a la parte inferior. Dado que en el protocolo descrito aquí los gradientes son recogidos desde el fondo hasta la parte superior, los perfiles mostrados comienzan con la fracción pesada (los polisomas) e ir a la fracción ligera (subunidades de ribosomas libres y ARN) (Figura 2A). A continuación recogemos cada gradiente en seis fracciones de 2 ml, pero fracciones más pequeñas se pueden recoger si un análisis más detallado del contenido de polisomas tiene que ser llevado a cabo.
La fusión y la normalización de las curvas a la cima monosome (Figura 2D) permite la comparación de los perfiles de diferentes líneas o las condiciones de crecimiento. Esto proporciona información sobre la cantidad relativa de polisomas independientemente del nivel de iniciación. Otra forma de unanalyze los perfiles es calcular el área bajo la curva, por lo tanto, se puede determinar la cantidad relativa de mRNA asociado con cualquiera de los monosomas o los polisomas (Figura 2C). Esta relación es específico de una planta y condiciones de crecimiento. Sin embargo, este enfoque puede no ser relevante en el caso de los tejidos en traslación activo poco.
Hemos utilizado este método para A. plantas enteras thaliana, rosetas jóvenes y viejos, así como para N. benthamiana (Figura 3C), S. lycopersicum (Figura 3E) y O. hojas sativa (Figura 3D). La forma del perfil depende de las condiciones de crecimiento, edad de la planta y los tejidos analizados. Aquí hemos utilizado A. thaliana seis días las plantas de semillero de edad. En esta etapa, la actividad de traducción es alta, y el perfil de muestra así picos de forma (Figura 2A y B). Este es también el casocuando A. thaliana flores se utilizan (Figura 3A). Cuando se utilizan 4 semanas de edad A. thaliana rosetas (Figura 3B), las muestras contienen principalmente adulto plenamente desarrollado hojas donde las células no se dividen. Por lo tanto, la cantidad total de polisomas y monosomas es menor. El perfil muestra un menor número, pero aún así en forma de picos polisomas. Al lado del pico monosome, el hombro muestra la gran cantidad de 60S libres subunidades ribosomales. Con otro tipo de plantas o tejidos, los picos de polisomas pueden ser apenas visible. Este es el caso cuando se utiliza 30 días de edad O. hojas sativa (Figura 3D). Incluso cuando la cantidad de ARNm implicados en la traducción es muy baja (sin pico polisomas se puede ver en el perfil), la presencia del pico monosome indica que el fraccionamiento se realiza correctamente y que los ARNm se pueden extraer más lejos de la fracción para su posterior análisis . La calidad de la calidad del ARN extraído se evaluó por electroforesis en gel de agarosa ( ong> Figura 4). El 25S y 18S ARN ribosomal citosólica debe ser claramente visible en el gel. Bandas inferiores correspondientes al ARN ribosomal cloroplástico también deben ser visibles cuando el ARN se extrae de los tejidos verdes 10.
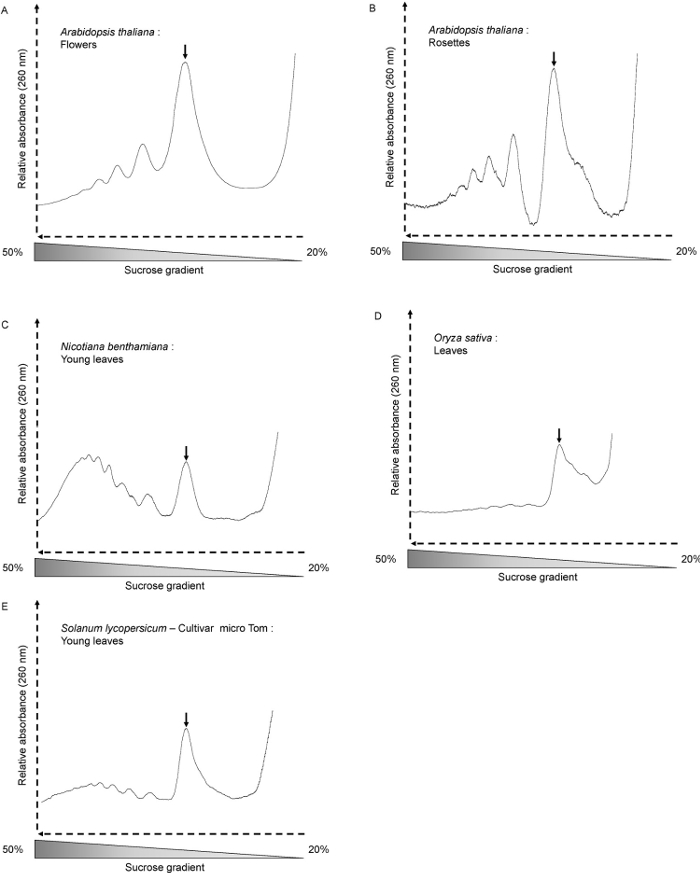
Figura 3. Los perfiles de polisomas de diferente material vegetal y especies. (A) las flores Arabidopsis thaliana (300 mg), (B) de Arabidopsis thaliana 4 semanas de edad rosetas (600 mg), benthamiana (C) Nicotiana (hojas jóvenes de 40 días de edad plantas día de cosecha cortos - 300 mg), (D) Oryza sativa ( hojas de plantas de 30 días de antigüedad - 300mg), (E) Solanum lycopersicum (hojas jóvenes de 35 días de edad plantas de día corto-crecido - 300 mg). Los picos monosome se indican mediante flechas.iles / ftp_upload / 54231 / 54231fig3large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
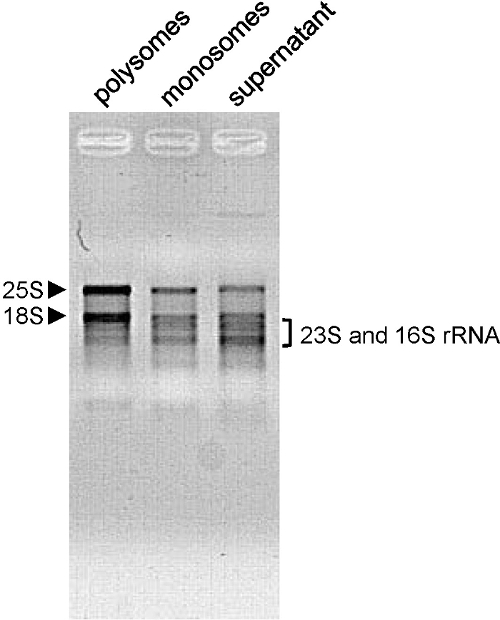
. Figura 4. Evaluación de la calidad del ARN ARN (500 ng extrajeron a partir de brotes de 6 días plántulas de Arabidopsis thaliana) de las fracciones indicadas se cargaron en un gel de agarosa al 1,2% y separadas por electroforesis (100V - 15 min). Citosólicas (25S y 18S rRNA) se indican mediante puntas de flecha y los cloroplastos (23S y 16S rRNA) por un soporte. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Discusión
The protocol we present here is an easy and cheap method for generating polysome profiles and isolating mRNAs associated with polysomes, single ribosomes or free of ribosomes. A wide range of different polysome fractionation methods is described in the literature. The method we have described here has been optimized to keep only the necessary compounds and has been adapted for plant material. In particular, we reduced the amount of detergent11 and added chloramphenicol to the buffer to fix the chloroplastic ribosomes to the mRNA (as cycloheximide does for the cytosolic ribosomes)12 . We have also reduced the total ultracentrifugation time7.
To obtain high quality polysome profiles, it is essential to use freshly collected plant material and to perform all steps at 4°C. When using tissues that are poorly translationally active, more plant material can be loaded on the gradient (up to 600 mg).
Analysis of polysome profiles can provide insights into both the overall translational status of cells13 and the translational status of a specific mRNA. We have used RNAs isolated by this method for different applications. Using the RNAs for microarrays allowed us to identify a class of cadmium stress response genes for which transcription and translation are uncoupled 14. We also used this method to identify small RNAs associated with polysomal fractions. This identification was made by northern blotting15 of RNA extracted from polysomal fractions, and provided biochemical evidence for a translational component in the miRNA pathway in plants. In another study, a cis-NAT RNA was identified by quantitative RT-PCR. This cis-NAT is associated to the phosphate homeostasis and promote translation of the PHO1;2 transcript 16.
The main limits of the polysome profiling approach are the lack of information concerning both the position of the ribosome on the mRNA and its progression along the mRNA. Ribosome profiling has emerged to address these limitations 17 and has been successfully used on plant tissues18. Ribosome profiling provides a global measurement of translation by taking advantage of the advances in sequencing technology. Nevertheless, as for any sequencing based assays, the quality of the results depends on the mapping of the sequences to the genome, therefore focusing on the small ribosome-protected fragments makes it difficult to deconvolute repetitive sequences. Moreover, the digestion of RNA not protected by the ribosomes leads to the loss of regulatory information contained in the 3' and 5' UTRs. It is then impossible to distinguish transcript variants that have different 3' or 5' UTRs and show different levels of translation19. The polysome profiling method described here is rapid and does not require specific technical skills. Altogether, these two methods represent complementary approaches to study translation regulation.
Divulgaciones
The authors have nothing to disclose
Agradecimientos
Este trabajo fue apoyado por la Agencia Nacional de Investigación (ANR-14-CE02-0010). Se agradece al Dr. Benjamin campo y el Dr. Elodie Lanet para la lectura crítica del manuscrito. Agradecemos al Sr. Michel Terese por su ayuda con la edición de vídeo.
Materiales
| Name | Company | Catalog Number | Comments |
| Ultracentrifuge tube, thinwall, polyallomer - 13.2 ml | Beckman Coulter | 331372 | |
| Ultracentrifuge tube, thinwall, polyallomer - 38.5 ml | Beckman Coulter | 326823 | |
| Glass capillary tube | Drummond Scientific | 1-000-1000 | |
| Ultracentrifuge | Beckman Coulter | Optima series | |
| Ultracentrifuge Rotor SW41 | Beckman Coulter | 331362 | |
| Ultracentrifuge Rotor SW32 | Beckman Coulter | 369650 | |
| Peristaltic pump | Any | ||
| Tygon R3607 polyvinyl chloride tubing | Fisher Scientific | 070534-22 | Polyvinyl chloride tubing, 2.29 mm |
| Fraction collector Model 2110 | Bio-Rad | 731-8120 | |
| UV cuvette | Hellma | 170.700-QS | Quartz flow-through cuvette |
| UV Spectrophotometer | Varian | Cary50 | Read every 0.0125 sec |
| All chemicals | Any | Use only Molecular Biology Grade | |
| Murashige and Skoog Basal Salt Mixture (MS) | Sigma-Aldrich | M5524 | |
| Rnase-Free water | Any | ||
| Petri Dishes | Fisher Scientific | 10083251 | |
| Octylphenoxy poly(ethyleneoxy)ethanol, branched (Nonidet P40) | Euromedex | UN3500 | |
| Linear acrylamide (acryl carrier) | ThermoFischer scientific | AM9520 | RNA precipitation carrier |
| OriginPro 8 | OriginLab | Analysis software |
Referencias
- Nelson, C. J., Millar, A. H. Protein turnover in plant biology. Nat Plants. 1 (3), 15017 (2015).
- Warner, J. R. The economics of ribosome biosynthesis in yeast. Trends Biochem Sci. 24 (11), 437-440 (1999).
- Amthor, J. S. The McCree-de Wit-Penning de Vries-Thornley Respiration Paradigms: 30 Years Later. Ann Bot. 86 (1), 1-20 (2000).
- Preiss, T., W Hentze, M. Starting the protein synthesis machine: eukaryotic translation initiation. BioEssays news and reviews in molecular, cellular and developmental biology. 25 (12), 1201-1211 (2003).
- Vogel, C., Marcotte, E. M. Insights into the regulation of protein abundance from proteomic and transcriptomic analyses. Nat Rev Genet. 13 (4), 227-232 (2013).
- Baerenfaller, K., et al. Genome-scale proteomics reveals Arabidopsis thaliana gene models and proteome dynamics. Science. 320 (5878), 938-941 (2008).
- Zanetti, E., Chang, I., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of Polyribosomal Complexes of Arabidopsis for Global Analysis of Gene Expression. Plant physiol. 138 (2), 624-635 (2005).
- Murashige, T., Skoog, F. A revised medium for rapid growth and bioassays with tobacco tissue cultures. Physiol plant. 15 (3), 473-497 (1962).
- del Prete, M. J., Vernal, R., Dolznig, H., Müllner, E. W., Garcia-Sanz, J. A. Isolation of polysome-bound mRNA from solid tissues amenable for RT-PCR and profiling experiments. RNA. 13 (3), 414-421 (2007).
- Salinas, J., Sanchez-Serrano, J. J. . Arabidopsis protocols. , (2006).
- Piques, M., et al. Ribosome and transcript copy numbers, polysome occupancy and enzyme dynamics in Arabidopsis. Mol syst biol. 5 (314), 314 (2009).
- Morita, M., Alain, T., Topisirovic, I., Sonenberg, N. Polysome Profiling Analysis. bio-protocol. 3 (14), 3-8 (2013).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS One. 8 (8), (2013).
- Sormani, R., et al. Sublethal cadmium intoxication in Arabidopsis thaliana impacts translation at multiple levels. Cell Physiol. 52 (2), 436-447 (2011).
- Lanet, E., et al. Biochemical evidence for translational repression by Arabidopsis microRNAs. Plant cell. 21 (6), 1762-1768 (2009).
- Jabnoune, M., Secco, D., Lecampion, C., Robaglia, C., Shu, Q., Poirier, Y. A Rice cis-Natural Antisense RNA Acts as a Translational Enhancer for Its Cognate mRNA and Contributes to Phosphate Homeostasis and Plant Fitness. Plant cell. 25 (10), 4166-4182 (2013).
- Ingolia, N. T. Genome-wide translational profiling by ribosome footprinting. Methods Enzymol. 470 (10), (2010).
- Juntawong, P., Girke, T., Bazin, J., Bailey-Serres, J. Translational dynamics revealed by genome-wide profiling of ribosome footprints in Arabidopsis. PNAS. 111 (1), 203-212 (2013).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nat Rev Genet. 15 (3), 205-213 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados