Method Article
שיטה קלה הצמח Polysome Profiling
In This Article
Summary
This protocol describes an easy method to extract and fractionate transcripts from plant tissues on the basis of the number of bound ribosomes. It allows a global estimate of translation activity and the determination of the translational status of specific mRNAs.
Abstract
Translation of mRNA to protein is a fundamental and highly regulated biological process. Polysome profiling is considered as a gold standard for the analysis of translational regulation. The method described here is an easy and economical way for fractionating polysomes from various plant tissues. A sucrose gradient is made without the need for a gradient maker by sequentially freezing each layer. Cytosolic extracts are then prepared in a buffer containing cycloheximide and chloramphenicol to immobilize the cytosolic and chloroplastic ribosomes to mRNA and are loaded onto the sucrose gradient. After centrifugation, six fractions are directly collected from the bottom to the top of the gradient, without piercing the ultracentrifugation tube. During collection, the absorbance at 260 nm is read continuously to generate a polysome profile that gives a snapshot of global translational activity. Fractions are then pooled to prepare three different mRNA populations: the polysomes, mRNAs bound to several ribosomes; the monosomes, mRNAs bound to one ribosome; and mRNAs that are not bound to ribosomes. mRNAs are then extracted. This protocol has been validated for different plants and tissues including Arabidopsis thaliana seedlings and adult plants, Nicotiana benthamiana, Solanum lycopersicum, and Oryza sativa leaves.
Introduction
סינתזת חלבון היא תהליך חיוני ויקר במרץ בכל התאים 1. קודם כל, התאים חייבים להשקיע אנרגיה בייצור של מכונות תרגום, הריבוזומים. לדוגמא תא שמרים שעדיין מתחלק מייצר ככל 2,000 ריבוזומים לדקה. כזה הייצור דורש עד 60% של פעילות תעתיק הכולל עד 90% מהפעילות שחבור הכולל של התא 2. בנוסף, אנרגיה נדרשת לסינתזה של חומצות אמינו, aminoacyl-tRNA ו קשרים פפטידיים. בצמחים, הוספת חומצה אמינית אחת על עלויות שרשרת הפפטיד מן 4.5 ל 5.9 מולקולות של ATP 3. לכן, אין זה מפתיע כי התרגום של mRNA לחלבון הוא אתר עיקרי של רגולציה, במיוחד כאשר מדובר בהתמודדות עם תנאי סביבה משתנה.
צעד החניכה של תרגום, כלומר את שיוכה של mRNA עם הריבוזום, הוא המטרה העיקרית של ההסדרהתרגום 4. כתוצאה מכך של הסדרת התרגום וכן צעדים רגולטוריים שלאחר תעתיק אחרים, רק 40% של וריאציות בריכוז החלבון יכול להיות מוסבר על ידי mRNA שפע 5,6. לכן, המחקר של mRNA הכולל נותן מידע עני יחסית על שפע חלבון. מצד שני, העמותה של mRNA עם ריבוזומים נותן תובנות טובות יותר שפע החלבון על ידי מתן גישה mRNAs המעורבים התרגום. mRNAs המתורגם באופן פעיל משויך כמה ריבוזומים במבנים שנקראו polysomes. לעומת זאת, mRNAs מתורגם גרוע ישויך רק אחד הריבוזום (monosome). כתוצאה מכך, מעמדם translational של mRNA יכול להיות מוערך על ידי מעקב אחר הקשר שלה עם ריבוזומים 7.
פרוטוקול זה מתאר את הבידוד של polysomes מן שתילי thaliana ארבידופסיס בת שישה ימים, הבידוד הבא של RNA, ואת ניתוח התוצאות. Polysomes ו monosomes מופרדים באמצעות שיפוע צפיפות סוכרוז. הדרגתיים נאספים לשישה שברים. חלק מן השברים הם אספו להשיג שלושה שברים מופרדים היטב: polysomes, monosomes ואת שבריר אור (הנקרא להלן supernatant), אשר מכיל את יחידות משנה ריבוזומלי 60S ו 40S בחינם mRNAs שאינם משויכים עם ריבוזומים. ניתן לאמוד פעילות תרגום עולמית על ידי יצירת יחס polysome / monosome, אשר נקבע על ידי שילוב של השטח מתחת לעקומה, ועל ידי השוואת פרופילי polysomes. mRNAs וחלבונים אז המחולצים שברים שונים ומשמש לניתוח על ידי RT-PCR, qRT-PCR, כתם הצפון, microarray, כתם או פרוטאומיקה המערבי. פרוטוקול זה יאומת לצמחים ורקמות אחרים.
הציוד נדרש לבצע פרוטוקול זה מצוי לרוב ברוב המעבדות: אין צורך לעושה שיפוע. הקפאת כל שכבה לפני הוספת הבאה למנועs מכל תערובת או הפרעה של השכבות. לא דוקר צינור משמש לאיסוף שיפוע אשר יכול להיות מושגת על ידי טבילה של צינור נימי זכוכית של ההדרגה. לכן, צינורות ultracentrifuge יקר להישאר ניזוק וניתן להשתמש בהן שוב ושוב פעמים רבות. ביחד, זה הופך את הפרוטוקול הנוכחי שיטה קלה וזולה עבור פרופיל polysome.
Protocol
1. הכנה של 20 עד 50% (w / v) מעברי צבע סוכרוז
הערה: הדרגתיים עשויים 4 שכבות של סוכרוז (50%, 35% ו -2 שכבות של 20%) בצינור ultracentrifuge 13.2 מ"ל. מניסיוננו, מזיגת סוכרוז 20% בשתי שכבות נפרדות משפרת באופן משמעותי את איכות הכנות polysome.
- הכן את פתרונות המניות. ודא כי כל הפתרונות הם RNAse ו DNAse בחינם.
- הכן תמיסת מלח 10X: 400 mM Tris-HCl pH 8.4, 200 מ"מ KCl ו -100 מ"מ MgCl 2.
- הכן 2 פתרון M סוכרוז: עבור 200 מ"ל, לפזר 137 גרם של סוכרוז פתרון 1X מלח.
- לדלל את הפתרון סוכרוז בסולט פתרון, כמתואר בטבלה 1, להכין הדרגתיים. הכרכים הנתונים הם שש הדרגתיים.
| ריכוז סוכרוז סופי | סוכרוז 2M (מ"ל) | פתרון מלח 1X (מ"ל) | כרך סופי. (מ"ל) |
| 50% | 8.8 | 3.2 | 12 |
| 35% | 12.9 | 12.1 | 25 |
| 20% | 7.4 | 17.6 | 25 |
| 20% | 5.8 | 14.2 | 20 |
טבלה 1. דילולים של תמיסת סוכרוז להכין שש הדרגתיים.
- יוצקים שכבות לפי טבלה 2.
| שכבת סוכרוז | 50% | 35% | 20% | 20% |
| Vol. (מ"ל) | 1.85 | 3.65 | 3.65 | 1.35 |
נפח טבלה 2. של תמיסת סוכרוז לכל שכבה.
- לאחר שמזג כל שכבה, לשמור על הצינורות בתוך C -40 ° או מקפיאה -80 מעלות צלזיוס עד הקפאה מוחלטת לפני הוספת ההשכבה סוכרוז הבאה. מקפיא מושגת בדרך כלל לאחר 2 שעות ב -40 מעלות צלזיוס, אבל מחכה כ -6 שעות לפני לשפוך את השכבה הבאה מומלץ. שכבת 20% האחרונה אפשר להקפיא או הוסיפה טרי ביום הניסוי.
הערה: הקפאת כל שכבה לפני הוספת הבאה מונעת מכל ערבוב או הפרעה של השכבות. הדרגתיים יכולים להישמר C -40 ° או -80 ° C במקפיא במשך שישה חודשים לפחות. - אם זה כבר לא נעשה, להוסיף את שכבת 20% האחרונים ההדרגתיים ביום הניסוי. לאחר מכן, תן ההדרגתי להפשיר בחדר קר או מקרר.
2. הכנת תמציות Cytosolic
הערה: אנו recommבסופו של שימוש בשני מילויים לדגימה ביולוגית. 300 מ"ג הוא הכמות האופטימלית של חומר צמחי להכין שני מילויים כשעובדים עם 6 ימים שתילי thaliana ארבידופסיס ישנים. כשעובד עם פחות רקמות translationally פעילות, כמות חומר צמחי ניתן להגדיל עד 600 מ"ג.
- שתילים ישנים קציר ששת ימים גדלו על ½ Murashige ו Skoog 8 בינוניים בתוספת 1% סוכרוז או מדיה מקבילה ידי מהיר הקפאה בחנקן נוזלי
- חומר צמחי טוחנים במכתש ועלי precooled עם חנקן נוזלי.
- לשקול 300 מ"ג של חומר אבקת בצלחת במשקל precooled. בצע פעולה זו במהירות כדי למנוע הפשרת המדגם.
הערה: שמור על דגימות קפואות לתקופה שלא תעלה על שבוע אחד במקפיא -80 מעלות צלזיוס. - להוסיף 2.4 מ"ל של חיץ polysome precooled (4X תמיסת מלח, 5.26 מ"מ EGTA, 0.5% (v / v) פולי Octylphenoxy (ethyleneoxy אתנול), מסועף, 50 μg.ml -1 cycloheximide, 50 μg.ml 1chloramphenicol) ו homogenize ידי ערבוב עם קצה פיפטה. העבר לשתי צינורות 1.5 מ"ל. לעבוד במהירות כדי למנוע את הדגימות מן ההתחממות.
- צנטריפוגה ב XG 16,000 במשך 15 דקות ב 4 ° C ב microcentrifuge כדי גלולה פסולת.
הערה: כדי למנוע השפלה RNA, תמיד לשמור על דגימות ב 4 ° C, השתמש פתרונות חינם RNase / DNase ולעבוד בתנאים חינם RNase / DNase. הפרין ניתן להוסיף למאגר polysome, לריכוז סופי של 300 μg.ml -1, כדי לשפר את ההגנה RNA. עם זאת, מאז הפרין יכול להפריע ניתוח במורד זרם, זה יהיה חייב להיות מוסר במהלך שלב משקעי RNA על ידי ביצוע משקע ליתיום כלוריד במקום משקעי אתנול (הערה השווה 4.7).
3. פרופיל Polysome
- בזהירות פיפטה את supernatant מבלי להפריע גלולה. אם שברי צמח כבר pipetted, חזור על שלב 2.5. טען את supernatant על גבי שיפוע ידי בעדינות pipetting על דפנות הצינור בזרם קבוע. השתמש שיפוע אחד לכל צינור 1.5 מ"ל.
- מעבירים precooled דליים צנטריפוגות ב 175,000 XG במשך שעה 2. 45 דקות ב 4 ° C, בתוך ultracentrifuge (למשל 32,000 סל"ד כשמשתמשים הרוטור SW41).
- הגדר את מערכת איסוף שיפוע כמתואר באיור 1. קובט UV יש pathlength 1 מ"מ.

איור 1. מערכת איסוף Gradient. קובט UV מחובר על ידי צינורות פוליוויניל כלוריד צינור נימי זכוכית יורד לתחתית השיפוע. שיפוע התקדמות דרך בזכות מערכת משאבה peristaltic. OD 260 קורא ברציפות ו -2 מ"ל שברים נאספים. אנא לחץ כאן כדי להציג גרסה גדולה יותר שלנתון זה.
- התאם את אספן חלק כדי RT, ולהגדיר את הקרוסלה במהירות שתאפשר גביית 2 מ"ל שברים. השתמש צינורות אוסף מראש מקורר.
- אסוף את השברים מלמטה למעלה באמצעות צינור נימי זכוכית המחוברים באמצעות צינורות פוליוויניל כלוריד למערכת אוסף השיפוע. השתמש סרט פרפין פלסטיק כדי לאבטח את החיבור בין צינורות פוליוויניל כלוריד ואת צינור נימי זכוכית.
- קראו את הספיגה ב 260 ננומטר מלמטה העליון של השיפוע. כאשר השיפוע כולו נאסף, ומקום הצינורות האוספים על קרח לפני מיצוי RNA.
4. RNA הפקה
- פינת השברים שנאספו משני הדרגתיים, ב 50 מ"ל כתרי צינורות צנטריפוגה, כדלקמן:
Polysomes: שברים 1 עד 3 (12 מ"ל)
Monosomes: חלק 4 (4 מ"ל)
Supernatant: שברים 5 ו -6 (8 מ"ל) - לכל חלק, להוסיף 1 כרך. hydrochloride guanidine 8M,50 מיקרוגרם מובילים אקריליק ו -1.5 vol. isopropanol.
הערה: מנשא אקריליק הוא acrylamide ליניארי, משמש coprecipitant כדי לשפר את ההתאוששות של חומצות גרעין במהלך משקעי אלכוהול. - מערבבים על ידי היפוך הצינורות המשקע O / N ב -20 ° C.
- צנטריפוגה ב 175,000 XG במשך שעה 1 ב 4 ° C, בתוך ultracentrifuge (32,000 סל"ד ב הרוטור SW32). אם השלב משקעים מתבצע בתוך שפופרת אשר אינה עולה בקנה אחד עם ultracentrifugation, והעברת צינור מתאים.
- בטל supernatant ו לפזר את גלולה ב 200 חיץ TE μl RNAse ללא (10 mM Tris-HCl pH 7.5 - 1 מ"מ EDTA). מעביר צינור 1.5 מיליליטר טרי.
- חלץ RNA על ידי הוספת 1 כרך. מים פנול רווי (pH 6.6) ו 1 כרך. כלורופורם: אלכוהול isoamyl (24: 1). מערבבים נמרצות. צנטריפוגה ב 15,000 XG במשך 20 דקות ב 4 °. מעביר את השלב המימי לצינור 1.5 מיליליטר חדש.
הערה: פנול חומצה (pH 4.5) ניתן להשתמש כדי למזער זיהום הדנ"א. - המשקע RNA על ידי הוספת 1/10 vol. 3 נתרן אצטט M ו- 3 vol. 100% אתנול. אפשר משקעים ב -80 מעלות צלזיוס במשך 20 דקות או O / N ב -20 ° C. צנטריפוגה ב 15,000 XG במשך 15 דקות ב 4 ° C..
הערה: אם אתה משתמש הפרין למאגר polysome (הערה השוו 2.5), לבצע ליתיום כלוריד (LiCl) ממטרים במקום משקעים אתנול כדי להסיר כל שמץ של הפרין כראוי שעלולה להפריע ניתוח במורד הזרם 9. לאחר החילוץ, להוסיף LiCl לריכוז סופי של 2.5 מ 'אפשר משקעים במשך 30 דקות ב -20 ° C או O / N ב 4 ° C. צנטריפוגה ב 15,000 XG במשך 15 דקות ב 4 ° C.. - שטפו את הכדור עם אתנול 500 μl 75%. האוויר היבש גלולה ו להתמוסס 30 μl חיץ TE. הערכת איכות RNA על ידי אלקטרופורזה על ג'ל agarose 1.2% חינם RNAse (100V, 15 דקות) או על ידי שיטות אלקטרופורזה נימי.
ניתוח 5. נתונים
- נתונים גולמיים ייצא תוכנת ניתוח גרפית ונתונים.
הערה: כפי שניתן לראות בתרשים 2A ו- B, את הפרופילים גלם לתת מידע איכותי: מספר פסגות polysome כי ניתן לראות, את גובה שיא monosome, ואת הנוכחות של הכתף המתאים יחידות משנה הריבוזום בחינם. - מיזוג פרופילים על מנת לאפשר השוואה של פרופילים בין דגימות. השתמש בכלי קורא מסך כדי לקבוע את ערך X של שיא monosome עבור כל דגימה וליישר את פסגות monosome ידי הסרת נקודות נתונים נוספות בתחילת העקומות. זהה את הנקודה הנמוכה ביותר של העקומה ולקבוע ערך Y שלה (שימוש באפשרות קורא המסך). הגדר את הערך Y הנקודה הנמוכה ביותר ל -0 באמצעות הפונקציה "סט עמודות ערך".
- קבע את יחס polysomes / monosomes ידי שילוב השטח מתחת לעקומה מפרופילי גלם (איור 3 ג). השתמש בכלי בורר נתונים להגדרת הגבול של האזור כדי להיות משולב. ואז להשתמש בפונקציה לשלב (ניתוח-מתמטיקה).
- נרמלהעקומות אל פסגת monosome ידי חלוקת כל נקודות הנתונים של עקומה ידי ערך Y של הפסגה של פסגת monosome (נקבעו באמצעות כלי קורא המסך). בחר את העמודה, ולאחר מכן תחת-מתמטיקה ניתוח, בחר לנרמל ולבחור את "מחולק לסכום מסוים" שיטות. שלב זה מאפשר השוואה של הרמה היחסית של polysomes (איור 3D).
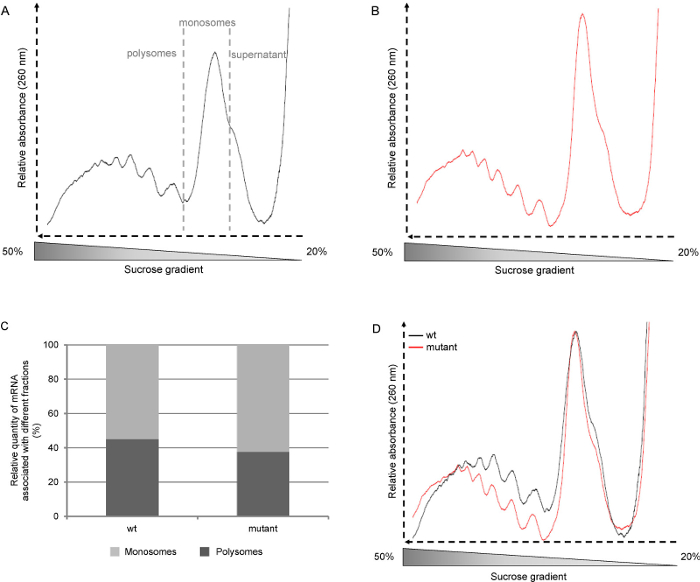
איור 2. פרופילים polysome נציג. ארבידופסיס thaliana wild-type (wt, אקוטיפ Col-0) ושתילים מוטציה גדלו במשך שישה ימים על ½ Murashige ובינוניים Skoog תחת photoperiods יום ארוך (16 שעות אור, 8 שעות חושך). ת: פרופילי polysome גלם מן השתילים WT. B: פרופילי polysome גלם מן שתילי מוטציה. C: קביעת אחוז polysomes ו monosomes ידי שילוב של השטח מתחת לעקומה D: Profi Polysomeles מנורמל שיא monosome. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
תוצאות
בספרות, פרופילי polysome לעתים קרובות מוצגים מתוך שבריר האור לשבר כבד כתוצאת הדרך ההדרגתית נאספת, כלומר מלמעלה עד למטה. מאז בפרוטוקול המתואר כאן ההדרגתיים נאספים מלמטה למעלה, את הפרופילים אנו מראים להתחיל עם השבר העבה (polysomes) וללכת שבר האור (יחידות מהשנה הריבוזום בחינם RNAs) (איור 2 א). לאחר מכן, אנו אוספים כל שיפוע בשישה 2 מ"ל שברים, אבל שברים קטנים ניתן לאסוף אם ניתוח מפורט יותר של תוכן polysome צריך להתבצע.
מיזוג ו הנרמול העקום אל פסגת monosome (איור 2 ד) מאפשר השוואה של פרופילי קווים שונים או תנאי גידול. זה מספק מידע על הכמות היחסית של polysomes העצמאי של רמת חניכה. דרך נוספת אלnalyze הפרופילים הוא לחשב את השטח מתחת לעקומה, ובכך, אפשר לקבוע את הכמות היחסית של mRNA הקשורים גם את monosomes או polysomes (איור 2 ג). יחס זה הוא ספציפי תנאי צמח וצמיחה. עם זאת, גישה זו לא יכולה להיות רלוונטית במקרה של רקמות גרועות translationally פעילים.
השתמשנו בשיטה זו עבור A. שתילים כולו thaliana, רוזטות צעירות ומבוגרות, כמו גם עבור נ benthamiana (איור 3 ג), ס lycopersicum (3E איור) וע ' עלי sativa (איור 3D). צורת פרופיל תלויה בתנאי הגידול, גיל צמח ואת הרקמות מנותחות. כאן השתמשנו א thaliana שישה ימי שתילים ישנים. בשלב זה, פעילות translational היא גבוהה, ופרופיל תערוכות היטב פסגות בצורה (איור 2 א 'וב'). זהו גם המקרהכאשר א פרחי thaliana משמשים (איור 3 א). בעת שימוש 4 שבועות א רוזטות thaliana (איור 3 ב), הדגימות בעיקר מכילות משאיר מבוגר מפותח שבו תאים לא מחלקים. לפיכך, הסכום הכולל של polysomes ו monosomes נמוך. פרופיל תערוכות פחות, אבל עדיין נאה צורת פסגות polysome. ליד השיא monosome, הכתף מראה את כמות גדולה של 60S חינם יחידות משנה ריבוזומלי. עם סוג אחר של צמחים או רקמות, פסגות polysome עשויות להיות בקושי נראית. זהו המקרה כאשר משתמשים 30 בימים ההם O. עלי sativa (איור 3D). גם כאשר כמות mRNAs המעורבת תרגום היא נמוכה מאוד (לא שיא polysome ניתן לראות על הפרופיל), בנוכחות שיא monosome מציינת כי החלוקה נעשתה כראוי וכי mRNAs ניתן לחלץ רחוק יותר מן השבר לניתוח נוסף . איכות איכות RNA חילוץ נבחנת על ידי ג'ל אלקטרופורזה agarose ( אונג> איור 4). 25S ו 18S cytosolic RNA ריבוזומלי צריך להיות בבירור על הג'ל. להקות תחתונות המתאימות RNA ריבוזומלי chloroplastic צריכות להיות גלויות לעין כאשר RNA המחולצים מפיות ירוקות 10.
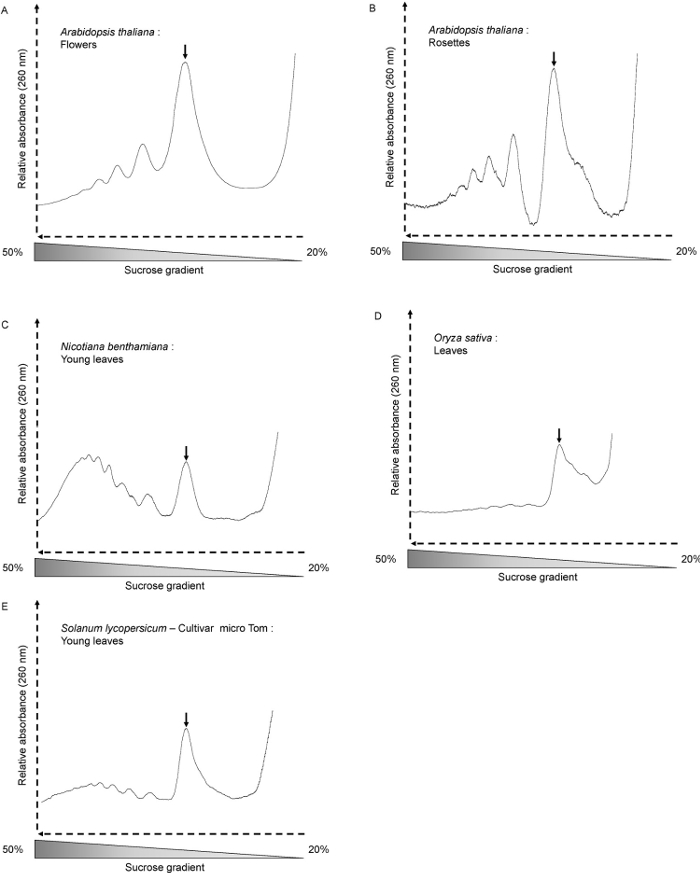
איור 3. פרופילי Polysome מחומר ו מיני צמחים שונים. (א) פרחי ארבידופסיס thaliana (300mg), (ב) ארבידופסיס thaliana 4 שבועות רוזטות ישן (600mg), (ג) ניקוטיאנה benthamiana (עלים צעירים של 40 ימים צמחים קצרים בן-גדל היום - 300mg), (ד) סאטיבה Oryza ( עלים של 30 ימים צמחים ישנים - 300 מ"ג), (ה) Solanum lycopersicum (עלים צעירים של 35 ימים צמחי יום-גדל קצרים ישנים - 300 מ"ג). פסגות monosome מסומנות על ידי חצים.Iles / ftp_upload / 54,231 / 54231fig3large.jpg "target =" _ blank "> לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
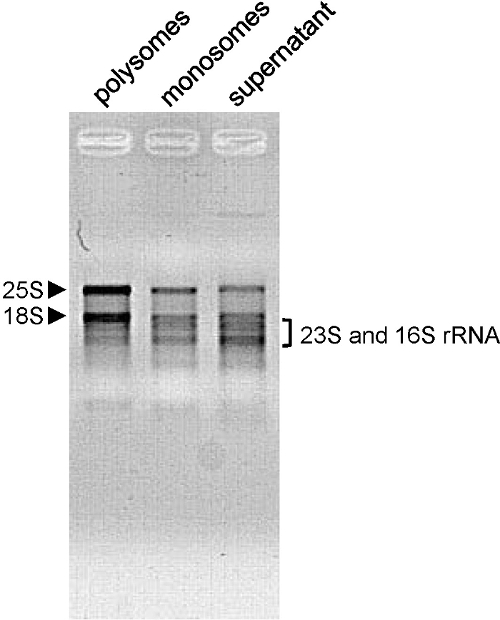
. איור 4. הערכת איכות RNA RNA (500 NG מופק יורה של 6 ימי שתילי thaliana ארבידופסיס ישנים) מן השברים הצביעו הועמסו על ג'ל 1.2% agarose והופרד אלקטרופורזה (100V - 15 דק '). Cytosolic (25S ו 18S) rRNA מסומנים על ידי ראשי חץ chloroplastic (23S ו 16S) rRNA על ידי סוגר. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
Discussion
The protocol we present here is an easy and cheap method for generating polysome profiles and isolating mRNAs associated with polysomes, single ribosomes or free of ribosomes. A wide range of different polysome fractionation methods is described in the literature. The method we have described here has been optimized to keep only the necessary compounds and has been adapted for plant material. In particular, we reduced the amount of detergent11 and added chloramphenicol to the buffer to fix the chloroplastic ribosomes to the mRNA (as cycloheximide does for the cytosolic ribosomes)12 . We have also reduced the total ultracentrifugation time7.
To obtain high quality polysome profiles, it is essential to use freshly collected plant material and to perform all steps at 4°C. When using tissues that are poorly translationally active, more plant material can be loaded on the gradient (up to 600 mg).
Analysis of polysome profiles can provide insights into both the overall translational status of cells13 and the translational status of a specific mRNA. We have used RNAs isolated by this method for different applications. Using the RNAs for microarrays allowed us to identify a class of cadmium stress response genes for which transcription and translation are uncoupled 14. We also used this method to identify small RNAs associated with polysomal fractions. This identification was made by northern blotting15 of RNA extracted from polysomal fractions, and provided biochemical evidence for a translational component in the miRNA pathway in plants. In another study, a cis-NAT RNA was identified by quantitative RT-PCR. This cis-NAT is associated to the phosphate homeostasis and promote translation of the PHO1;2 transcript 16.
The main limits of the polysome profiling approach are the lack of information concerning both the position of the ribosome on the mRNA and its progression along the mRNA. Ribosome profiling has emerged to address these limitations 17 and has been successfully used on plant tissues18. Ribosome profiling provides a global measurement of translation by taking advantage of the advances in sequencing technology. Nevertheless, as for any sequencing based assays, the quality of the results depends on the mapping of the sequences to the genome, therefore focusing on the small ribosome-protected fragments makes it difficult to deconvolute repetitive sequences. Moreover, the digestion of RNA not protected by the ribosomes leads to the loss of regulatory information contained in the 3' and 5' UTRs. It is then impossible to distinguish transcript variants that have different 3' or 5' UTRs and show different levels of translation19. The polysome profiling method described here is rapid and does not require specific technical skills. Altogether, these two methods represent complementary approaches to study translation regulation.
Disclosures
The authors have nothing to disclose
Acknowledgements
עבודה זו נתמכה על ידי הסוכנות למחקר הלאומית הצרפתית (ANR-14-CE02-0010). אנו מודים ד"ר בנימין שדה וד"ר אלודי Lanet לקריאה ביקורתית של כתב היד. אנו מודים למר מישל תרז על עזרתו עם עריכת וידאו.
Materials
| Name | Company | Catalog Number | Comments |
| Ultracentrifuge tube, thinwall, polyallomer - 13.2 ml | Beckman Coulter | 331372 | |
| Ultracentrifuge tube, thinwall, polyallomer - 38.5 ml | Beckman Coulter | 326823 | |
| Glass capillary tube | Drummond Scientific | 1-000-1000 | |
| Ultracentrifuge | Beckman Coulter | Optima series | |
| Ultracentrifuge Rotor SW41 | Beckman Coulter | 331362 | |
| Ultracentrifuge Rotor SW32 | Beckman Coulter | 369650 | |
| Peristaltic pump | Any | ||
| Tygon R3607 polyvinyl chloride tubing | Fisher Scientific | 070534-22 | Polyvinyl chloride tubing, 2.29 mm |
| Fraction collector Model 2110 | Bio-Rad | 731-8120 | |
| UV cuvette | Hellma | 170.700-QS | Quartz flow-through cuvette |
| UV Spectrophotometer | Varian | Cary50 | Read every 0.0125 sec |
| All chemicals | Any | Use only Molecular Biology Grade | |
| Murashige and Skoog Basal Salt Mixture (MS) | Sigma-Aldrich | M5524 | |
| Rnase-Free water | Any | ||
| Petri Dishes | Fisher Scientific | 10083251 | |
| Octylphenoxy poly(ethyleneoxy)ethanol, branched (Nonidet P40) | Euromedex | UN3500 | |
| Linear acrylamide (acryl carrier) | ThermoFischer scientific | AM9520 | RNA precipitation carrier |
| OriginPro 8 | OriginLab | Analysis software |
References
- Nelson, C. J., Millar, A. H. Protein turnover in plant biology. Nat Plants. 1 (3), 15017(2015).
- Warner, J. R. The economics of ribosome biosynthesis in yeast. Trends Biochem Sci. 24 (11), 437-440 (1999).
- Amthor, J. S. The McCree-de Wit-Penning de Vries-Thornley Respiration Paradigms: 30 Years Later. Ann Bot. 86 (1), 1-20 (2000).
- Preiss, T., W Hentze, M. Starting the protein synthesis machine: eukaryotic translation initiation. BioEssays news and reviews in molecular, cellular and developmental biology. 25 (12), 1201-1211 (2003).
- Vogel, C., Marcotte, E. M. Insights into the regulation of protein abundance from proteomic and transcriptomic analyses. Nat Rev Genet. 13 (4), 227-232 (2013).
- Baerenfaller, K., et al. Genome-scale proteomics reveals Arabidopsis thaliana gene models and proteome dynamics. Science. 320 (5878), 938-941 (2008).
- Zanetti, E., Chang, I., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of Polyribosomal Complexes of Arabidopsis for Global Analysis of Gene Expression. Plant physiol. 138 (2), 624-635 (2005).
- Murashige, T., Skoog, F. A revised medium for rapid growth and bioassays with tobacco tissue cultures. Physiol plant. 15 (3), 473-497 (1962).
- del Prete, M. J., Vernal, R., Dolznig, H., Müllner, E. W., Garcia-Sanz, J. A. Isolation of polysome-bound mRNA from solid tissues amenable for RT-PCR and profiling experiments. RNA. 13 (3), 414-421 (2007).
- Arabidopsis protocols. Salinas, J., Sanchez-Serrano, J. J. , Methods in molecular biology; 323. Clifton, N.J. (2006).
- Piques, M., et al. Ribosome and transcript copy numbers, polysome occupancy and enzyme dynamics in Arabidopsis. Mol syst biol. 5 (314), 314(2009).
- Morita, M., Alain, T., Topisirovic, I., Sonenberg, N. Polysome Profiling Analysis. bio-protocol. 3 (14), 3-8 (2013).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS One. 8 (8), (2013).
- Sormani, R., et al. Sublethal cadmium intoxication in Arabidopsis thaliana impacts translation at multiple levels. Cell Physiol. 52 (2), 436-447 (2011).
- Lanet, E., et al. Biochemical evidence for translational repression by Arabidopsis microRNAs. Plant cell. 21 (6), 1762-1768 (2009).
- Jabnoune, M., Secco, D., Lecampion, C., Robaglia, C., Shu, Q., Poirier, Y. A Rice cis-Natural Antisense RNA Acts as a Translational Enhancer for Its Cognate mRNA and Contributes to Phosphate Homeostasis and Plant Fitness. Plant cell. 25 (10), 4166-4182 (2013).
- Ingolia, N. T. Genome-wide translational profiling by ribosome footprinting. Methods Enzymol. 470 (10), Elsevier Inc. (2010).
- Juntawong, P., Girke, T., Bazin, J., Bailey-Serres, J. Translational dynamics revealed by genome-wide profiling of ribosome footprints in Arabidopsis. PNAS. 111 (1), 203-212 (2013).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nat Rev Genet. 15 (3), 205-213 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved