Method Article
공장 Polysome 프로파일 링을위한 쉬운 방법
요약
This protocol describes an easy method to extract and fractionate transcripts from plant tissues on the basis of the number of bound ribosomes. It allows a global estimate of translation activity and the determination of the translational status of specific mRNAs.
초록
Translation of mRNA to protein is a fundamental and highly regulated biological process. Polysome profiling is considered as a gold standard for the analysis of translational regulation. The method described here is an easy and economical way for fractionating polysomes from various plant tissues. A sucrose gradient is made without the need for a gradient maker by sequentially freezing each layer. Cytosolic extracts are then prepared in a buffer containing cycloheximide and chloramphenicol to immobilize the cytosolic and chloroplastic ribosomes to mRNA and are loaded onto the sucrose gradient. After centrifugation, six fractions are directly collected from the bottom to the top of the gradient, without piercing the ultracentrifugation tube. During collection, the absorbance at 260 nm is read continuously to generate a polysome profile that gives a snapshot of global translational activity. Fractions are then pooled to prepare three different mRNA populations: the polysomes, mRNAs bound to several ribosomes; the monosomes, mRNAs bound to one ribosome; and mRNAs that are not bound to ribosomes. mRNAs are then extracted. This protocol has been validated for different plants and tissues including Arabidopsis thaliana seedlings and adult plants, Nicotiana benthamiana, Solanum lycopersicum, and Oryza sativa leaves.
서문
단백질 합성은 모든 세포 하나에 필수적이고 정력적으로 비용이 많이 드는 과정이다. 우선, 세포 변환 기계, 리보솜의 생산 에너지 투자해야한다. 예를 들어 적극적으로 분할 효모 세포는 분당 많은 2,000 리보솜을 생산하고 있습니다. 이러한 생산량은 전체 전사 활성의 60 %로하고, 전지 (2)의 전체 접합 활성의 90 %까지 필요하다. 또한, 에너지는 아미노산, 아미노 아실 tRNA의 결합 펩티드의 합성에 필요하다. 식물, ATP 3 4.5 5.9 분자 펩티드 사슬 비용으로 하나의 아미노산을 첨가. 따라서,이 단백질의 mRNA의 번역은 변화하는 환경 조건을 처리에 관해서 특히, 규정의 주요 사이트 것은 놀라운 일이 아니다.
리보솜을 가진 mRNA의 협회 인 번역의 개시 단계는,의 조절의 주요 표적이다번역 4. 번역의 조정의 결과뿐만 아니라 다른 후 - 전사 조절 단계로서, 단백질 농도의 변화의 40 %의 mRNA의 풍부 5,6- 의해 설명 될 수있다. 따라서, 전체의 mRNA의 연구는 단백질 풍부에 대한 상대적으로 빈약 한 정보를 제공합니다. 반면에, 리보솜과의 mRNA의 협회는 번역에 관련된 사람들의 mRNA에 대한 액세스를 제공함으로써 단백질 풍부에 대한 정보를 얻을 수 있습니다. 적극적 번역의 mRNA는 polysomes라는 구조에서 여러 가지 변이와 연관됩니다. 반대로, 제대로 번역의 mRNA는 하나의 리보솜 (monosome)와 연결됩니다. 결과적으로, mRNA의 번역의 리보솜 상태 (7)의 연결을 모니터링함으로써 평가 될 수있다.
이 프로토콜 여섯 일 이전 애기 장대 모종, RNA의 연속적인 분리에서 polysomes의 분리 및 결과의 분석을 설명한다. 포lysomes 및 monosomes은 자당 밀도 구배를 통해 분리된다. 그라디언트 여섯 분획으로 수집됩니다. polysomes, monosomes 및 변이와 연관되지 않은 자유 60S와 40S 리보솜 서브 유닛과의 mRNA를 포함하는 가벼운 부분 (이하라고 뜨는) : 분획물 중 일부는 세 가지 잘 분리 분획을 얻기 위해 풀링된다. 글로벌 번역 활성 및 polysomes 프로파일을 비교하여 곡선 아래의 면적의 적분에 의해 결정된다 polysome / monosome 비율을 생성함으로써 추정 될 수있다. 의 mRNA와 단백질은 다른 분수에서 추출 및 RT-PCR, QRT-PCR, 노던 블롯, 마이크로 어레이, 웨스턴 블롯 또는 단백질 체학에 의한 분석에 사용됩니다. 이 프로토콜은 다른 식물과 조직에 대한 검증되었습니다.
이 프로토콜을 수행하는 데 필요한 장비는 일반적으로 대부분의 실험실에서 발견된다 : 구배 머신은 필요 없다. 다음 하나가되지 않도록 추가하기 전에 각 층을 동결층들 중 하나 또는 혼합 교란들. 어떠한 튜브 뚫음은 구배로 유리 모세관의 침지에 의해 달성 될 수 구배 컬렉션에 사용되지 않는다. 따라서, 고가의 초 원심 분리 튜브 손상을 유지하고 여러 번 재사용 될 수있다. 종합적으로,이 의정서 polysome 프로파일에 대한 쉽고 저렴한 방법합니다.
프로토콜
20 ~ 50 % (w / v)의 자당 그라디언트 1. 준비
주 : 그래디언트 13.2 mL를 초 원심 분리 튜브에 자당의 4 층 (50 %, 35 % 및 20 %의 2 층)으로 만들어진다. 우리의 경험에 의하면, 두 개의 분리 된 층에서 20 %의 자당을 붓는 크게 polysome 제제의 품질을 향상시킵니다.
- 주식 솔루션을 준비합니다. 전체 솔루션의 RNAse와 DNase를 무료 있는지 확인합니다.
- 400 mM 트리스 - 염산의 pH 8.4, 200 mM의 KCl을 100 밀리미터의 MgCl 2 : 10X 소금 솔루션을 준비합니다.
- 200 ml를 들어 1X 소금 솔루션에서 자당 137g을 용해 2 M 자당 솔루션을 준비합니다.
- 기울기를 제조 표 1에 기재된 바와 같이, 염 용액에 자당 용액을 희석. 주어진 볼륨은 여섯 그라디언트위한 것입니다.
| 최종 자당 농도 | 자당 2M (㎖) | 소금 용액 1X (㎖) | 최종 권. (㎖) |
| 50 % | 8.8 | 3.2 | (12) |
| 35 % | 12.9 | 12.1 | (25) |
| 20 % | 7.4 | 17.6 | (25) |
| 20 % | 5.8 | 14.2 | (20) |
표 자당 솔루션 1. 희석 여섯 그라디언트를 준비합니다.
- 표 2에 따라 레이어를 따르십시오.
| 자당 층 | 50 % | 35 % | 20 % | 20 % |
| 집. (㎖) | 1.85 | 3.65 | 3.6(5) | 1.35 |
레이어 당 자당 용액의 표 2 권.
- 각 층을 붓는 후, -40 ° C에서 튜브를 유지하거나 -80 C의 냉동고 ° 완전 동결 될 때까지 다음 크로스 레이어를 추가하기 전에. 동결 통상 -40 ° C에서 2 시간 후에 달성되지만, 다음 층을 주입하기 전에 약 6 시간을 기다리고 권장한다. 마지막으로 20 %의 층을 동결하거나 실험 일에 새로 첨가 할 수있다.
참고 : 다음 중 하나를 추가하기 전에 각 레이어를 동결하면 레이어의 혼합 또는 방해에서 방지 할 수 있습니다. 그라디언트는 -40 ° C에 보관하거나 할 수 있습니다 -80 C의 냉동고 ° 최소 6 개월. - 아직 완료되지 않은 경우, 실험 당일 기울기로 최종 20 %의 층을 추가한다. 그 후, 그라디언트가 추운 방이나 냉장고에서 해동 할 수 있습니다.
세포질 추출물 2. 준비
참고 : 우리는 권장 합생물학적 샘플 당 두 그라디언트를 사용하여 종료. 300 mg을 6 일 이전 애기 장대 모종 작업 할 때 두 그라디언트를 준비하는 식물 재료의 최적의 양이다. 이하 병진 활성 조직 작업시 식물 재료의 양은 600 mg을까지 증가 될 수있다.
- ½ 무라 시게 및 스쿡에서 재배 수확 육일 된 모종은 8 매체는 액체 질소로 급속 냉동하여 1 % 자당 또는 이에 상응하는 미디어로 보충
- 액체 질소와 미리 냉각 박격포와 유 봉에서 분쇄 식물 재료.
- 예냉 무게 접시에 가루 물질 300 mg의 무게. 샘플의 해동을 피하기 위해 빨리이 단계를 수행합니다.
주 : -80 ° C의 냉동고에 더 일주일 이상 냉동 샘플을 보관하십시오. - 분기 미리 냉각 polysome 버퍼의 2.4 ㎖ (소금 솔루션 4 배, 5.26 mM의 EGTA, 0.5 % (v / v)의 옥틸 페녹시 폴리 (에틸렌 옥시) 에탄올, 50 μg.ml -1 사이클로 헥시 미드를 추가, 50 μg.ml 1클로람페니콜) 및 피펫 팁과 혼합하여 균질화. 이 1.5 ml의 튜브에 전송합니다. 온난화에서 샘플을 방지하기 위해 신속하게 작업 할 수 있습니다.
- 미세 원심에서 4 ° C에서 15 분 동안 16,000 XG에 원심 분리기 파편 펠렛합니다.
참고 : 항상 RNA 저하를 방지 4 ° C에서 샘플을 유지하기 위해,의 RNase / DNase의 무료 조건에서의 RNase / DNase의 무료 솔루션 및 작업을 사용합니다. 헤파린은 RNA 보호를 강화하기 위해, 300 μg.ml -1의 최종 농도는 polysome 완충액에 첨가 할 수있다. 그러나, 하류 분석을 방해 할 수 헤파린 때문에, 염화 리튬 석출 대신 에탄올 침전 (참조, 참고 4.7)을 수행하여 RNA를 침전 단계에서 제거 될 것이다.
3. Polysome 프로파일
- 조심스럽게 펠렛을 방해하지 않고 뜨는을 피펫. 식물 조각이 피펫 된 경우, 단계 2.5를 반복합니다. 부드럽게 pipettin에 의해 그라디언트의 상단에 뜨는로드일정한 스트림 튜브의 측벽 상 g. 각 1.5 ML 튜브에 대해 하나의 그라데이션을 사용합니다.
- (A SW41 로터를 사용하는 경우 예를 들어, 32,000 rpm으로) 초 원심 분리기에서 4 ° C에서 2 시간 45 분 동안 175,000 XG에 양동이와 원심 분리기를 예비 냉각에 전송합니다.
- 도 1에 기재된 바와 같이 기울기 수집 시스템을 설정한다. 자외선 큐벳은 1mm의 경로 길이를 갖는다.

도 1 그라데이션 수집 시스템. 자외선 큐벳 경사 아래쪽으로 하강 유리 모세관에 폴리 염화 비닐 튜브에 의해 연결된다. 구배 연동 펌프 시스템 덕분으로 진행한다. OD 260 계속 읽기 2 ml의 분수가 수집됩니다. 의 더 큰 버전을 보려면 여기를 클릭하십시오이 그림.
- RT에 분수 콜렉터를 조정하고 2 ml의 분수의 수집을 허용 속도로 회전 목마를 설정합니다. 사전 냉각 수집 튜브를 사용합니다.
- 그래디언트 수집 시스템에 폴리 염화 비닐 튜브에 의해 접속 된 유리 모세관을 이용하여 아래에서 위로 분획을 수집한다. 폴리 염화 비닐 튜브, 유리 모세관 튜브 사이의 연결을 고정 파라핀 플라스틱 필름을 사용한다.
- 그라디언트의 상단에 바닥에서 260 nm에서의 흡광도를 참조하십시오. 전체 기울기가 수집되면, RNA 추출 전에 얼음에 수집 튜브를 배치합니다.
4. RNA 추출
- 다음과 같이 원심 분리기 튜브 출장 50 ㎖에 두 그라디언트에서 수집 분수, 수영장 :
Polysomes : 분획 1 내지 3 (12 mL) 중
Monosomes : 분수 4 (4 ㎖)
상청액 : 분획 5, 6 (8 mL) 중 - 각 부분에, 1 권을 추가합니다. 8 M 구아니딘 히드로 클로라이드,50 μg의 아크릴 캐리어 1.5 VOL. 이소프로판올.
주 : 아크릴 담체는 알코올 침전 중 핵산의 회수를 향상시키기 coprecipitant로서 사용 선형 아크릴 아미드이다. - 튜브를 반전하여 혼합 및 -20 ° C에서 O / N을 침전.
- 초 원심 분리기에서 4 ° C에서 1 시간,의 (a SW32 로터에서 32,000 rpm으로)에 대한 175,000 XG에 원심 분리기. 침전 공정은 초 원심 분리와 호환되지 튜브에서 수행되는 경우, 적합한 관에 옮긴다.
- 뜨는을 취소하고 200 μL의 RNA 분해 효소 무료 TE 완충액 (10 mM 트리스 - 염산의 pH 7.5-1 mM의 EDTA)에서 펠렛을 녹인다. 신선한 1.5 ML 튜브로 전송합니다.
- 1 권을 추가하여 RNA를 추출합니다. 물 포화 페놀 (PH 6.6) 1 권. 클로로포름 : 이소 아밀 알코올 (24 : 1). 적극적으로 섞는다. 4 ℃에서 20 분 동안 15,000 XG에 원심 분리기. 새로운 1.5 ML 튜브에 수성 단계를 전송합니다.
주 : 페놀 산 (PH 4.5) DNA의 오염을 최소화하기 위해 사용될 수있다. - 1/10 부피를 첨가하여 RNA를 침전. 3 M 아세트산 나트륨 3 VOL. 100 % 에탄올. -20 ° C에서 20 분 또는 O / N에 대한 -80 ° C에서 침전을 허용합니다. 4 ℃에서 15 분 동안 15,000 XG에 원심 분리기.
참고 : polysome 버퍼 (참조, 참고 2.5)에 헤파린을 사용하여 제대로 다운 스트림 분석 (9)을 방해 할 수 헤파린의 흔적을 제거하기 위해 에탄올 침전 대신에 염화 리튬 (LiCl을) 침전을 수행합니다. 추출 후, 4 ° C에서 -20 ° C 또는 O / N에서 30 분 동안 침전을 허용 2.5 M.의 최종 농도로하여 LiCl을 추가합니다. 4 ℃에서 15 분 동안 15,000 XG에 원심 분리기. - 500 ㎕를 75 % 에탄올로 펠렛을 씻으십시오. 공기는 건조 된 펠릿을 30 ㎕의 TE 완충액에 용해. RN 아제 (RNase) 자유 1.2 % 아가 로스 겔 (100V, 15 분) 또는 모세관 전기 영동 방법에서 전기 영동에 의해 RNA의 품질을 평가한다.
5. 데이터 분석
- 그래픽 및 데이터 분석 소프트웨어를 수출 원시 데이터.
노트:도 2A 및 B에 도시 된 바와 같이, 원시 정보는 질적 정보를 제공 : 알 수 polysome 피크의 수, monosome 피크의 높이와 자유 리보좀 서브 유닛에 대응하는 어깨의 존재. - 샘플 사이의 프로파일의 비교를 할 수 있도록 프로필을 병합합니다. 각 샘플에 대한 monosome 피크의 X 값을 결정하기 위해 스크린 리더 도구를 사용하여 곡선의 시작 부분에 추가 데이터 포인트를 제거하여 monosome 피크를 정렬. 곡선의 가장 낮은 지점을 확인하고 Y 값 (화면 판독기 도구를 사용)를 결정합니다. 은 "세트 열 값"기능을 사용하여 0에 가장 낮은 지점의 Y 값을 설정합니다.
- 원시 정보 (도 3c)의 곡선 아래의 면적을 적분하여 polysomes / monosomes 비율을 결정한다. 지역의 경계가 통합 될 정의 할 데이터 선택 도구를 사용합니다. 그런 다음 통합 기능 (분석 - 수학)를 사용합니다.
- 정규화monosome 피크의 정상 회담의 Y 값에 의한 곡선의 모든 데이터 포인트를 분할하여 monosome 피크에 곡선 (화면 판독기 도구를 사용하여 결정). 열을 선택하고 분석 - 수학에서, 표준화를 선택하고 "특정 값으로 나눈"방법을 선택합니다. 이 단계는 polysomes의 상대적 수준 (그림 3D)의 비교를 할 수 있습니다.
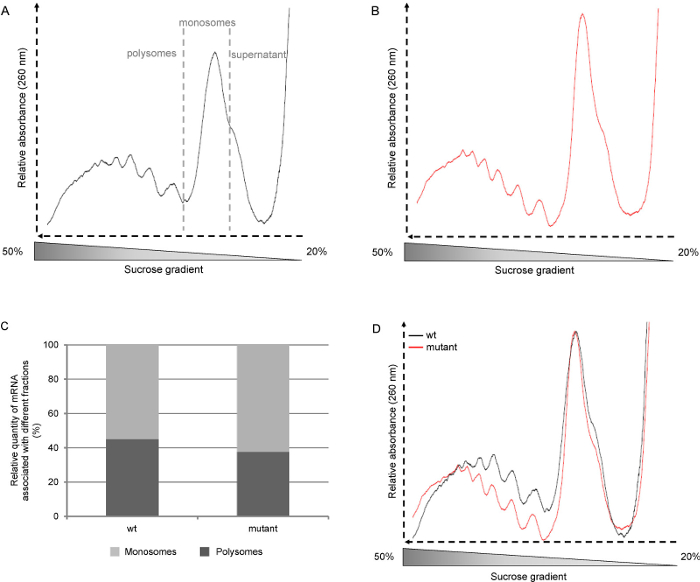
그림 2. 대표 polysome 프로필. 애기 장대 야생형이 (중량, 생태형의 골-0) 및 돌연변이 모종 긴 하루 photoperiods (16 시간 빛, 8 시간의 어두운)에서 ½ 무라 시게 및 스쿡 매체에 육일 동안 성장했다. A : 중량 모종 원시 polysome 프로필. B : 돌연변이 모종 원시 polysome 프로필. C : 곡선 D에서 영역의 통합에 의해 polysomes 및 monosomes의 비율의 결정 : Polysome PROFI레는 monosome 피크에 정규화. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
문헌에서, 정보는 종종 polysome 즉, 위에서 아래로, 기울기가 수집되는 방식의 결과로서 중량 분율로 가벼운 부분에서 도시된다. 여기에 설명 된 프로토콜에 그라디언트 가기 바닥에서 수집되기 때문에, 우리가 보여 프로파일은 무거운 부분합니다 (polysomes)로 시작하고 가벼운 부분 (무료 리보솜 서브 유닛 및 RNA를) (그림 2A)로 이동합니다. 그런 다음 여섯 2ml를 구배 각 분획을 수집하지만 polysome 함량의보다 상세한 분석이 수행되어야한다면 작은 분획을 회수 할 수있다.
병합 및 monosome 피크 (그림 2D)에 곡선을 정상화하는 것은 다른 선이나 성장 조건에서 프로파일의 비교를 할 수 있습니다. 이는 독립적 개시의 수준 polysomes의 상대적인 양에 대한 정보를 제공한다. A와 또 다른 방법프로파일을 nalyze하여 곡선 아래의 면적을 계산하기 때문에, 하나의 monosomes 또는 polysomes (도 2c) 중 하나와 관련된 mRNA의 상대적인 양을 결정할 수있다. 이 비율은 식물 성장 조건에 따라 다릅니다. 그러나,이 방법은 활성이 저조한 병진 조직의 경우에 해당되지 않을 수있다.
우리는 A.이 방법을 사용했다 장대 전체 모종, 젊은이와 노인 근엽뿐만 아니라 N. 용으로 benthamiana (그림 3C), S. lycopersicum (그림 3E) 및 O. 사티 잎 (그림 3D). 프로파일 형상은 성장 조건, 식물 연령과 분석 조직에 따라 달라집니다. 여기에서 우리는 A를 사용 육일 오래된 모종 장대. 이 단계에서, 번역 활성이 높고, 프로필 잘 모양의 피크 (도 2A와 B)을 나타낸다. 이것은 또한 사건때 A. 장대 꽃 (그림 3A)를 사용한다. 4 주 오래 A를 사용하는 경우 장대 로제트 (그림 3B)는 샘플은 주로 세포가 분열하지 않는 경우 완전히 개발 된 성인 잎이 포함되어 있습니다. 따라서 polysomes 및 monosomes의 전체 량은 낮다. 프로파일은 적은,하지만 여전히 잘 모양의 polysome 봉우리를 보여줍니다. monosome 피크 옆, 어깨 무료 60S 리보솜 서브 유닛의 많은 양을 보여줍니다. 식물이나 조직의 다른 종류로, polysome 봉우리는 겨우 볼 수 있습니다. 삼십일 오래 O.를 사용할 때의 경우 사티 잎 (그림 3D). 번역에 관여 된 mRNA의 양 (단 polysome 피크는 프로파일에서 볼 수 없다) 매우 낮은 경우에도, monosome 피크의 존재는 분별이 제대로 이루어과의 mRNA는 상기 추가 분석을 위해 분획으로부터 추출 될 수 있다는 것을 나타낸다 . 추출한 RNA 품질의 품질은 아가 로스 겔 전기 영동으로 평가된다 ( 옹> 그림 4). 25S와 18S 세포질 리보솜 RNA는 겔에 명확하게 볼 수 있어야합니다. RNA는 녹색 조직 (10)로부터 추출 될 때 엽록체 리보솜 RNA에 해당하는 낮은 밴드도 볼 수 있어야합니다.
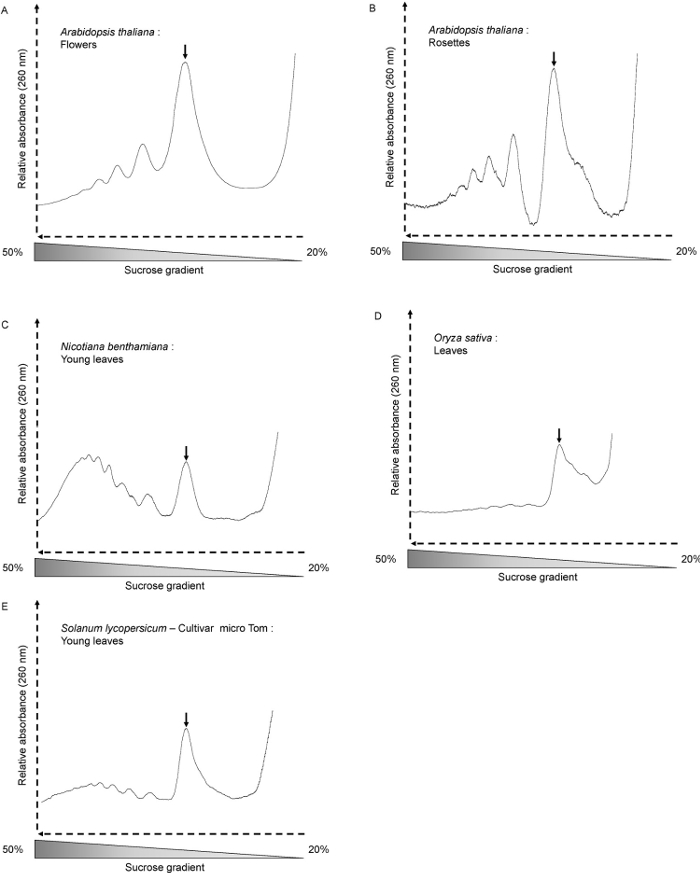
그림 다른 식물 재료와 종에서 3 Polysome 프로필. (A) 애기 장대의 꽃 (300 ㎎), (B) 애기 장대 4 주 오래 된 로제트 (600 ㎎), (C) 담배 속에서 benthamiana (사십일 된 짧은 하루 성장 식물의 어린 잎 - 300 ㎎), (D) 벼 ( 삼십일 된 식물의 잎 - 300mg의), (E) 까 lycopersicum (삼십오일 된 짧은 하루 성장 식물의 어린 잎 - 300 ㎎). monosome 피크는 화살표로 표시됩니다.세틸 / ftp_upload / 54231 / 54231fig3large.jpg "대상 ="_ 빈 ">이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
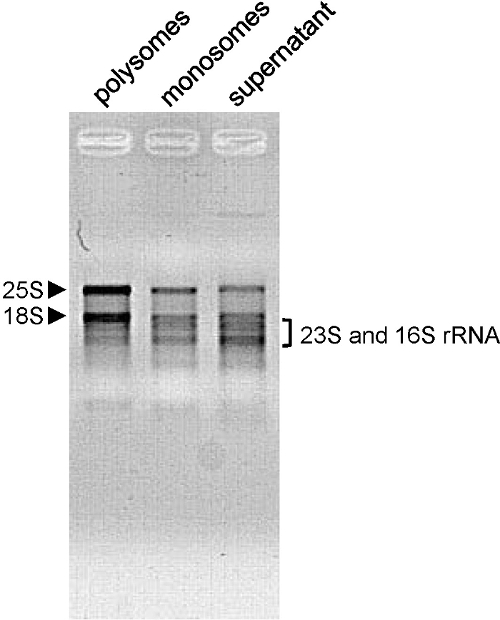
. (- 15 분 100V)도 RNA 품질 4. 평가 RNA 표시된 분획에서 1.2 % 아가 로스 겔에 로딩 및 전기 영동에 의해 분리 하였다 (500 6 일 이전 애기 장대 모종 촬영 추출 NG). 세포질 (25S와 18S) rRNA의이 화살촉과 엽록체 브라켓에 의해 (23S와 16S) rRNA의로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
The protocol we present here is an easy and cheap method for generating polysome profiles and isolating mRNAs associated with polysomes, single ribosomes or free of ribosomes. A wide range of different polysome fractionation methods is described in the literature. The method we have described here has been optimized to keep only the necessary compounds and has been adapted for plant material. In particular, we reduced the amount of detergent11 and added chloramphenicol to the buffer to fix the chloroplastic ribosomes to the mRNA (as cycloheximide does for the cytosolic ribosomes)12 . We have also reduced the total ultracentrifugation time7.
To obtain high quality polysome profiles, it is essential to use freshly collected plant material and to perform all steps at 4°C. When using tissues that are poorly translationally active, more plant material can be loaded on the gradient (up to 600 mg).
Analysis of polysome profiles can provide insights into both the overall translational status of cells13 and the translational status of a specific mRNA. We have used RNAs isolated by this method for different applications. Using the RNAs for microarrays allowed us to identify a class of cadmium stress response genes for which transcription and translation are uncoupled 14. We also used this method to identify small RNAs associated with polysomal fractions. This identification was made by northern blotting15 of RNA extracted from polysomal fractions, and provided biochemical evidence for a translational component in the miRNA pathway in plants. In another study, a cis-NAT RNA was identified by quantitative RT-PCR. This cis-NAT is associated to the phosphate homeostasis and promote translation of the PHO1;2 transcript 16.
The main limits of the polysome profiling approach are the lack of information concerning both the position of the ribosome on the mRNA and its progression along the mRNA. Ribosome profiling has emerged to address these limitations 17 and has been successfully used on plant tissues18. Ribosome profiling provides a global measurement of translation by taking advantage of the advances in sequencing technology. Nevertheless, as for any sequencing based assays, the quality of the results depends on the mapping of the sequences to the genome, therefore focusing on the small ribosome-protected fragments makes it difficult to deconvolute repetitive sequences. Moreover, the digestion of RNA not protected by the ribosomes leads to the loss of regulatory information contained in the 3' and 5' UTRs. It is then impossible to distinguish transcript variants that have different 3' or 5' UTRs and show different levels of translation19. The polysome profiling method described here is rapid and does not require specific technical skills. Altogether, these two methods represent complementary approaches to study translation regulation.
공개
The authors have nothing to disclose
감사의 말
이 작품은 프랑스 국립 연구 기관 (ANR-14 CE02-0010)에 의해 지원되었다. 우리는 원고의 중요한 읽기 박사 벤자민 필드 박사 엘로 디 Lanet 감사합니다. 우리는 비디오 편집과 그의 도움 씨 미셸 테 레스 감사합니다.
자료
| Name | Company | Catalog Number | Comments |
| Ultracentrifuge tube, thinwall, polyallomer - 13.2 ml | Beckman Coulter | 331372 | |
| Ultracentrifuge tube, thinwall, polyallomer - 38.5 ml | Beckman Coulter | 326823 | |
| Glass capillary tube | Drummond Scientific | 1-000-1000 | |
| Ultracentrifuge | Beckman Coulter | Optima series | |
| Ultracentrifuge Rotor SW41 | Beckman Coulter | 331362 | |
| Ultracentrifuge Rotor SW32 | Beckman Coulter | 369650 | |
| Peristaltic pump | Any | ||
| Tygon R3607 polyvinyl chloride tubing | Fisher Scientific | 070534-22 | Polyvinyl chloride tubing, 2.29 mm |
| Fraction collector Model 2110 | Bio-Rad | 731-8120 | |
| UV cuvette | Hellma | 170.700-QS | Quartz flow-through cuvette |
| UV Spectrophotometer | Varian | Cary50 | Read every 0.0125 sec |
| All chemicals | Any | Use only Molecular Biology Grade | |
| Murashige and Skoog Basal Salt Mixture (MS) | Sigma-Aldrich | M5524 | |
| Rnase-Free water | Any | ||
| Petri Dishes | Fisher Scientific | 10083251 | |
| Octylphenoxy poly(ethyleneoxy)ethanol, branched (Nonidet P40) | Euromedex | UN3500 | |
| Linear acrylamide (acryl carrier) | ThermoFischer scientific | AM9520 | RNA precipitation carrier |
| OriginPro 8 | OriginLab | Analysis software |
참고문헌
- Nelson, C. J., Millar, A. H. Protein turnover in plant biology. Nat Plants. 1 (3), 15017(2015).
- Warner, J. R. The economics of ribosome biosynthesis in yeast. Trends Biochem Sci. 24 (11), 437-440 (1999).
- Amthor, J. S. The McCree-de Wit-Penning de Vries-Thornley Respiration Paradigms: 30 Years Later. Ann Bot. 86 (1), 1-20 (2000).
- Preiss, T., W Hentze, M. Starting the protein synthesis machine: eukaryotic translation initiation. BioEssays news and reviews in molecular, cellular and developmental biology. 25 (12), 1201-1211 (2003).
- Vogel, C., Marcotte, E. M. Insights into the regulation of protein abundance from proteomic and transcriptomic analyses. Nat Rev Genet. 13 (4), 227-232 (2013).
- Baerenfaller, K., et al. Genome-scale proteomics reveals Arabidopsis thaliana gene models and proteome dynamics. Science. 320 (5878), 938-941 (2008).
- Zanetti, E., Chang, I., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of Polyribosomal Complexes of Arabidopsis for Global Analysis of Gene Expression. Plant physiol. 138 (2), 624-635 (2005).
- Murashige, T., Skoog, F. A revised medium for rapid growth and bioassays with tobacco tissue cultures. Physiol plant. 15 (3), 473-497 (1962).
- del Prete, M. J., Vernal, R., Dolznig, H., Müllner, E. W., Garcia-Sanz, J. A. Isolation of polysome-bound mRNA from solid tissues amenable for RT-PCR and profiling experiments. RNA. 13 (3), 414-421 (2007).
- Arabidopsis protocols. Salinas, J., Sanchez-Serrano, J. J. , Methods in molecular biology; 323. Clifton, N.J. (2006).
- Piques, M., et al. Ribosome and transcript copy numbers, polysome occupancy and enzyme dynamics in Arabidopsis. Mol syst biol. 5 (314), 314(2009).
- Morita, M., Alain, T., Topisirovic, I., Sonenberg, N. Polysome Profiling Analysis. bio-protocol. 3 (14), 3-8 (2013).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS One. 8 (8), (2013).
- Sormani, R., et al. Sublethal cadmium intoxication in Arabidopsis thaliana impacts translation at multiple levels. Cell Physiol. 52 (2), 436-447 (2011).
- Lanet, E., et al. Biochemical evidence for translational repression by Arabidopsis microRNAs. Plant cell. 21 (6), 1762-1768 (2009).
- Jabnoune, M., Secco, D., Lecampion, C., Robaglia, C., Shu, Q., Poirier, Y. A Rice cis-Natural Antisense RNA Acts as a Translational Enhancer for Its Cognate mRNA and Contributes to Phosphate Homeostasis and Plant Fitness. Plant cell. 25 (10), 4166-4182 (2013).
- Ingolia, N. T. Genome-wide translational profiling by ribosome footprinting. Methods Enzymol. 470 (10), Elsevier Inc. (2010).
- Juntawong, P., Girke, T., Bazin, J., Bailey-Serres, J. Translational dynamics revealed by genome-wide profiling of ribosome footprints in Arabidopsis. PNAS. 111 (1), 203-212 (2013).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nat Rev Genet. 15 (3), 205-213 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유