Method Article
Triagem géneros alimentícios destinados a uma classe integrons e Gene Cassetes
Neste Artigo
Resumo
This protocol describes the detection of class 1 integrons and their associated gene cassettes in foodstuffs.
Resumo
Antibiotic resistance is one of the greatest threats to health in the 21st century. Acquisition of resistance genes via lateral gene transfer is a major factor in the spread of diverse resistance mechanisms. Amongst the DNA elements facilitating lateral transfer, the class 1 integrons have largely been responsible for spreading antibiotic resistance determinants amongst Gram negative pathogens. In total, these integrons have acquired and disseminated over 130 different antibiotic resistance genes. With continued antibiotic use, class 1 integrons have become ubiquitous in commensals and pathogens of humans and their domesticated animals. As a consequence, they can now be found in all human waste streams, where they continue to acquire new genes, and have the potential to cycle back into humans via the food chain. This protocol details a streamlined approach for detecting class 1 integrons and their associated resistance gene cassettes in foodstuffs, using culturing and PCR. Using this protocol, researchers should be able to: collect and prepare samples to make enriched cultures and screen for class 1 integrons; isolate single bacterial colonies to identify integron-positive isolates; identify bacterial species that contain class 1 integrons; and characterize these integrons and their associated gene cassettes.
Introdução
A descoberta dos antibióticos foi uma das maiores conquistas científicas do século 20. No entanto, o uso e abuso de antibióticos levou à rápida evolução de bactérias resistentes aos antibióticos, e estes agora representam uma séria ameaça para a saúde pública no século 21. O surgimento de cepas bacterianas resistentes à maioria das opções de tratamento levanta a possibilidade de que estamos entrando em uma era onde as drogas antimicrobianas não são mais eficazes 1,2.
A maquinaria genética que confere resistência aos antibióticos é um sistema antigo, anterior a seres humanos e pressões de seleção de antibióticos por milhões de anos 3. Elementos genéticos móveis, tais como plasmídeos, transposões, ilhas genômicas, elementos conjugativos integrativas e integrons podem disseminar genes de resistência a antibióticos (ARG) tanto dentro como entre espécies bacterianas 4. Destes, integrões têm desempenhado um papel central na propagação de Arg, apesar dafato de que eles dependem de plasmídeos e transposons de mobilização e de inserção no genoma bacteriano 5. Integrons capturar cassetes de genes que utilizam um íntegron-integrase, e, em seguida, expressar as cassetes utilizando um promotor codificado íntegron 6,7 (Figura 1). Integron gene cassetes são pequenos elementos móveis consistindo de quadros individuais de leitura abertas (ORF), cujos produtos podem conferir resistência a antibióticos ou desinfectantes 8. Classe 1 integrons são os integrons mais comumente recuperados a partir de isolados clínicos 5, onde têm adquiriu coletivamente mais de 130 diferentes cassetes de genes de resistência a antibióticos 9.
A propagação da classe 1 integrons em comensais e patogênicas de bactérias associadas a humanos gera fluxos de resíduos humanos que contêm um grande número destes elementos genéticos 10. Estima-se que 10 19 bactérias que contêm uma classe integrons são liberados via lodo de esgoto cada ano in o Reino Unido 11. Portanto, não é surpreendente que uma classe integrons conferem resistências a antibióticos estão agora a ser detectado em microbiota das aves selvagens, peixes e outros animais selvagens nativos 12-14. Soltar integrons volta para o ambiente constitui uma ameaça grave de saúde pública, desde a aquisição de novos cassetes de genes e rearranjos complexos com outros elementos móveis continua a ocorrer, particularmente em estações de tratamento de esgoto e outros corpos d'água 15-18. O ambiente natural, em seguida, torna-se um terreno fértil para o recrutamento de novos determinantes de resistência e patógenos oportunistas 19,20. Bactéria contendo integron novos e novos ARGs pode circular de volta para a comunidade humana através de água e alimentos contaminados 21,22. Vigilância de ARGs ambientais é uma estratégia fundamental para a compreensão e manejo da resistência aos antibióticos no futuro 23. Em particular, deve ser dada atenção aos géneros alimentícios que são comidos crus oulevemente cozidos, uma vez que estes apresentam a maior ameaça para a transmissão de novos elementos celulares e patógenos.
Neste protocolo, uma abordagem simplificada para a detecção, identificação e caracterização de classe 1 integrons e suas cassetes de genes associados nos alimentos são descritas (Figura 2). Usando uma combinação de reacção cultura e polimerase em cadeia (PCR), integrões pode ser rapidamente detectada em comunidades bacterianas e complexos isolados individuais. Métodos de identificação das espécies de bactérias e a conformação ea identidade das cassetes de genes associados a integron são dadas. O método é adequado para uma ampla variedade de alimentos de origem vegetal e animal, e exemplos de fluxos típicos são dados para cada um destes tipos de alimentos.
Protocolo
Os géneros alimentícios que são comidos crus ou levemente cozidos são de maior preocupação para a saúde humana. Exemplos incluem hortaliças, frutas, mariscos e crustáceos.
Coleção 1. Amostra
- Recolha de amostras sob condições que minimizam a contaminação, e armazenadas em sacos separados, limpas durante o transporte. Uma vez recolhidas, as amostras devem ser armazenadas a 4 ° C e processados dentro de 24 horas.
2. Enriquecido Cultura Preparação
- Frutas e vegetais:
- Colocar aproximadamente 10 g de material em um saco de plástico durável. Se a transformação de matérias maior, concentrar-se na pele do fruto ou legume. Adicionar 90 ml de tampão 0,1 M de fosfato de sódio, pH 7,0 e processo num misturador de pás durante 30 s. Em alternativa, o material pode ser agitada manualmente, se um misturador de pás não está disponível.
- Recolhe-se o líquido em dois tubos Falcon de 50 ml. Tubos de centrifugação a 4.000 xg por 7 min de bactérias de pelotização.
- Remova cuidadosamente o sobrenadante e suspender as bactérias peletizadas em 30 ml de Luria estéril Bertani (LB).
- Incubar num agitador de O / N, que prende um tubo a 25 ° C e o outro a 37 ° C. As culturas deverão ser agitados a 200 oscilações por minuto (OPM). Armazenar as culturas a 4 ° C até serem necessárias.
- Seafood:
- Dissecar o estômago (ostras) ou do tracto digestivo (camarão), utilizando fórceps e pinças estéreis e colocar num tubo de 1,5 ml esterilizado, contendo 200 ul de tampão de fosfato de sódio 0,1 M, pH 7,0. Macerar o exemplo para criar um homogeneizado.
- Pipetar 5 ml de caldo LB estéril em dois tubos de 5 ml, e inocular cada um com o homogeneizado de marisco.
- Incubar num agitador de O / N, que prende um tubo a 25 ° C e o outro a 37 ° C. As culturas deverão ser agitados a 200 opm. Armazenar as culturas a 4 ° C até serem necessárias.
3. As culturas de rastreio para integrons
NOTA: Os protocolos de PCR convencionais são utilizados ao longo desta metodologia, utilizando tampões fornecidos com a enzima, e uma concentração final de MgCl2 de 2,5 mM. Se necessário, Lorenz (2011) 24 delineou PCR otimização e solução de problemas métodos em uma edição anterior desta revista.
- Preparação de ADN fervido:
- Dispensar 100 l de cultura enriquecido em um estéril 0,5 ml tubo de PCR. Aquece-se a amostra a 99 ° C durante 10 min, utilizando um banho-maria ou bloco de aquecimento. Encaixe frio em gelo durante 2 min.
- Microcentrífuga a 14000 xg durante 5 min para sedimentar os detritos celulares. Retornar ao gelo. As amostras de ADN podem ser congeladas a -20 ° C até serem necessárias, mas deve ser descongelada em gelo.
- Rastreio de classe 1 integrons usando PCR:
NOTA: culturas mistas do O / N incubação são selecionados para a classe 1 integrons usando o protocolo HS463a / HS464 PCR (Tabela 1).- Descongelar todos os reagentes de PCR em gelo, longe de qualquer potencial de souRCEs de ADN contaminante. Compõem 48 ul PCR mastermix por amostra, incluindo controlos negativos (Tabela 2). Dispense 48 ul de PCR master mix para cada tubo, usando dicas de barreira.
- Adicionar 2 mL de ADN descongeladas para tubos de PCR individuais, utilizando pontas de barreira. Executar programa adequado no termociclador de PCR.
- Para avaliar os resultados da amplificação, electroforese 7 uL de produto de PCR num gel de agarose a 2% e executar vertida em tampão TBE (90 mM Tris-borato, 2 mM de EDTA). Incluem um marcador de peso molecular, tais como uma escada de 100 pb.
- Adicione mancha do gel com um corante de ADN, e visualizar utilizando um transiluminador de UV. Verificar que não houve nenhuma amplificação no controlo negativo. A banda forte em torno de 470 pb para qualquer determinada amostra indica que a cultura mista contém bactérias que carregam uma classe 1 integron. Colónias puras vai agora ser isolado a partir de qualquer cultura mista que devolvido um PCR positivo.
4. Triagem de colônias Individual para Classe 1 integrons
- Diluições em série e placas espalhadas:
NOTA: Para isolar colónias simples a partir das culturas mistas de PCR-positivas, a cultura mista é diluída em série de 10 vezes em tampão de fosfato de sódio, e depois plaqueadas para recuperar as colónias individuais.- Adicionar 1 ml de cultura mista a 9 ml de tampão de fosfato de sódio 0,1 M, pH 7,0 e misturar por inversão. Adicionar 1 ml da diluição para uma mais 9 ml de tampão de fosfato, e repetir até uma diluição em série de 10 -8 é atingido.
- Espalhe 100 ul dos 10 -4 a 10 -8 diluições em agar LB, em duplicado. Incubar as placas de O / N à mesma temperatura utilizada para a cultura mista inicial. As placas podem então ser armazenadas a 4 ° C até ser necessário, no entanto, é melhor para processar as colónias individuais relativamente prontamente.
- Selecção de colónias únicas e o ADN fervido preparação:
- Escolha colónias individuais das placas de diluição em série, SElecting como muitos tipos diferentes de colónias possível, usando critérios como tamanho da colônia, forma e cor. Use palitos esterilizados para selecionar colônias individuais da placa propagação.
- Toque no palito para a colônia, e depois transferir para um tubo de PCR contendo 100 ml de água estéril. Se um grande número de colónias estão a ser pesquisadas, em seguida, as colónias podem ser preparados num tabuleiro de microtitulação. Girar o palito entre os dedos para deslocar algumas das células na água.
- Usando o mesmo palito, inocular uma placa de LB com uma raia da colônia. Um grande número de colónias pode ser armazenada em placas marcadas se cada raia é de cerca de 1 cm de comprimento. Repita os passos 1 a 3 até que você tenha selecionado um número apropriado de isolados.
- Incubar a placas LB O / N, à mesma temperatura que o utilizado para a cultura enriquecido inicial. As culturas podem depois ser armazenado a 4 ° C até serem necessárias.
- Para a preparação de DNA a partir das suspensões bacterianas, siga os passos como UOtlined na secção 3.1. Para o rastreio de lisados de cultura pura, execute o HS463a / HS464 PCR, conforme descrito no ponto 3.2. Os isolados que retornar um PCR positivo será utilizado para a preparação de ADN genómico (seção 5), e para análises posteriores.
5. As colónias individuais Genomic DNA Extração de Integron-positivos Usando Bead Batendo 25
- Inocular 5 ml de caldo de LB com uma amostra de cultura pura isolada na secção 4.2. Incubar S / N, agitando à mesma temperatura que foi utilizado para a cultura enriquecido inicial.
- Células de pelotas em 4000 g durante 7 min, em seguida, remover o sobrenadante. Volte a suspender as células sedimentadas em 1 ml de solução CLS-TC em um Lysing Matrix E tubo de preparação rápida. Usando uma máquina de talão de espancamento, processar a amostra em 5,5 m / s por 30 s.
- Tubo de centrifugação a 14000 xg durante 5 min e recuperar 700 ul de sobrenadante para um tubo esterilizado de 1,5 mL.
- Adicionar 700 ul de matriz obrigatório diluído 1: 5 com 6 M guanidinium tiocianato e misturar à temperatura ambiente durante 5 min. Centrifugar a 14.000 xg durante 1 min, o sobrenadante de descarte.
- Adicionar 800 ul de lavagem com etanol sal (etanol a 70%, acetato de sódio 0,1 M) e agitar com vortex até que o sedimento é ressuspenso completamente. Lavar durante 5 min à temperatura ambiente, centrifugar a 14.000 xg durante 1 min e descartar o sobrenadante. Assegurar-se que o sobrenadante foi removido, utilizando uma micropipeta, se necessário, em seguida deixar secar ao ar durante 5 min.
- Ressuspender o sedimento em matriz de 200 ul de TE (10 mM Tris-HCl, 1 mM de EDTA, pH 7,6), pipetando para cima e para baixo. Incubar à temperatura ambiente durante 3 min. Centrifugar a 14.000 xg durante 3 min e a transferência de 160 uL do ADN eluído em um tubo estéril. Executar uma alíquota da extracção do ADN genómico em um 2% w / v de gel de agarose para verificar a integridade e rendimento da extracção do ADN. O ADN genómico purificado é armazenado a -20 ° C e descongelados em gelo, quando necessário para o teste de PCR.
6. PCRs de Diagnóstico e seqüenciamento de DNA
NOTA: ODNA genómico preparado na seção 5 será usado para todos os PCRs de diagnóstico, e para confirmar o teste positivo para a classe 1 integrons.
- Descongele DNA genômico em gelo, vortex brevemente a amostra de DNA e pulso rotação a 14.000 xg por 20 s. Repetir a PCR para o gene da integrase íntegron-classe 1 usando HS463a / HS464 (Tabela 1), confirmando que o isolado é positivo para o amplicão 471 pb (Secção 3.2).
- Identificação de culturas positivas ao nível de espécie é conseguida por análise da pequena subunidade do gene de rRNA (16S ADNr). Amplificar o gene 16S utilizando os iniciadores F27 / r1492 (Tabela 1). Confira os produtos de PCR utilizando eletroforese. Todos os alvos bacterianos deve gerar um 16S amplicon de cerca de 1450 pb.
- Sequência do amplicão do gene 16S rRNA para determinar a identidade das espécies, mas uma vez que não é provável que sejam múltiplos PCR positivos a partir do passo 6.2, e muitos destes positivos serão as mesmas espécies bacterianas, vamos primeiro distinguir as diferentes espécies using digestão de restrição do produto de PCR 16S.
- Digerir uma aliquota do PCR 16S com a enzima de restrição Hinf 1. Qualquer enzima que reconhece sítios de clivagem 4 pb vai fazer, mas Hinf 1 gera boa diversidade de alvos 16S, e é uma enzima confiável e barata.
- Configurar uma restrição de 30 ul digerir mastermix (Tabela 2) em um tubo estéril e adicionar 20 l de produto de PCR 16S purfied-un. Incubar em um banho de água a 37 ° C O / N. Verificar o produto da digestão num gel de agarose a 2%.
- Pontuação dos padrões de digestão produzidos por Hinf 1. Os isolados com o mesmo perfil de restrição 16S são susceptíveis de ser da mesma espécie. Em vez de seqüenciar todos os produtos de PCR 16S, seqüência, no máximo, três representantes de cada padrão de restrição Hinf 1.
- Purifica-se os produtos de PCR 16S utilizando um kit comercial. Um único conjunto de exonuclease de cadeia pode ser usado, mas com base em coluna ou métodos de precipitação também funciona bem para remover unincprimers orporated e ADN de cadeia simples. Sequência os amplicons 16S usando R910 iniciador (Tabela 1), que estabelecem as reações como especificado pelo serviço de sequenciação.
- Use a sequência de DNA resultante para interrogar o banco de dados de DNA NCBI com a função blastn ( http://blast.ncbi.nlm.nih.gov/Blast.cgi ). Mais do que 97% de identidade de nucleótidos geralmente é tomado como a identidade das espécies.
7. Mapeamento e Caracterização de integrons e cassete Arrays
NOTA: Classe 1 integrons que emanam de ecossistemas dominados pelo homem tendem a ser os integrons mais comuns em todas as suas amostras. Estes integrons todos têm uma origem única recente e, portanto, têm uma sequência de DNA altamente conservado 5.
- Diferenciar entre uma classe 1 integrões que emanam a partir de fontes humanas e as que ocorrem naturalmente em amostras ambientais através da realização de umPCR com iniciadores definido intI1F165 / intI1R476 10 (Tabela 1).
NOTA: Classe 1 integrons de bactérias comensais clínicos ou irá gerar um produto de ~ 300 pb, enquanto classe ambiental 1 integrons não irá gerar qualquer produto. - Caracterizar os integrons clínicos, amplificando a matriz cassete com conjunto de primers HS458 / HS459. Esta PCR amplifica a região entre intI1 e segmento conservado a 3 'no terminal da maior parte das matrizes de cassetes. Uma vez que o número e identidade de cassetes é variável, o tamanho do produto de PCR também irá variar.
- Purifica-se produtos de amplificação e a sequência de ADN utilizando cada um dos iniciadores de amplificação. Use seqüências para interrogar bases de dados de ADN utilizando a função blastn ( http://blast.ncbi.nlm.nih.gov/Blast.cgi ).
- Certifique-se de nomear de cassetes siga nomenclatura padronizada 9, uma vez que muitos depoimentos dados para cassete gene integrons usar nomes inconsistentes ou ambíguas. Dê especial atenção às cassetes de genes novos, uma vez que estes podem codificar novos fenótipos, incluindo genes que conferem aumento transmissibilidade, patogenicidade ou virulência 8,21.
NOTA: pré-clínica formas da classe 1 integron ainda circulam em ambientes naturais. Em particular, aqueles que estão ligados a ativos Tn 402 transposons são de maior interesse 26. As matrizes de cassetes dessas integrons não vai amplificar com conjunto de primers HS458 / HS459, mas pode ser amplificado com o MRG284 / MRG285 primário conjunto (Tabela 1), gerando amplicons de tamanho variável dependente conteúdo cassete. Os produtos de PCR deve ser purificado e o ADN sequenciado conforme descrito em 7.2.
Resultados
Triagem de culturas mistas e isolados bacterianos para intI1
O conjunto iniciador HS463a / HS464 PCR pode ser usado para detectar a presença do gene da integrase íntegron-classe 1, intI1 (Figura 1). Este conjunto de iniciadores para a detecção trabalha bem intI1 em culturas mistas, e também é utilizado para rastrear colónias bacterianas colhidas a partir de placas de propagação (Figura 2). Isolados positivos devem gerar uma única banda forte em 471 pb usando este conjunto de primers (Figura 3A). A maioria dos isolados positivos vai levar intI1 que tem originado a partir de seres humanos ou os animais agrícolas e domésticas. Estes isolados também será positivo numa PCR utilizando os iniciadores F165 / R476, que deverá gerar um amplicão de 311 pb (Figura 1). Fontes ambientais de intI1 geralmente não será positivo neste segundo ensaio.
Caracterização das matrizes cassete integron
O DNA genômico de isolados puros é utilizado para caracterização de matrizes de cassetes integron. As matrizes de cassetes de 402 Tn classe -associated um integrons pode ser amplificada utilizando iniciadores HS458 / HS459. Estes iniciadores alvo, respectivamente, o local de recombinação íntegron, e a extremidade 3 'da cassete de matriz, que normalmente termina em fusão de genes a qacEΔ sul1 (Figura 1). O tamanho dos produtos de PCR gerados neste ensaio varia de acordo com o número e identidade dos cassetes na matriz (Figura 3B). Classe ambiental 1 integrões são frequentemente incorporados nos cromossomas bacterianos, e suas matrizes de cassete pode ser amplificado por iniciadores que têm como alvo as extremidades proximal e a maioria dos locais de recombinação distais (Figura 1). Os iniciadores MRG284 / 285 são concebidos para amplificar esta região, e de novo, porque o conteúdo cassete varia, o tamanho dos produtos de amplificação também varia (Figura 3C). A sequenciação do HS458 / 459 PCRprodutos serão normalmente recuperar conhecidos determinantes de resistência a antibióticos, enquanto sequenciação MRG284 / 285 produtos de PCR serão geralmente recuperar cassetes de genes que codificam polipéptidos de função desconhecida.
Identificação de espécies bacterianas
As bactérias são identificadas usando 16S rDNA e banco de dados comparações. O ADN genómico é utilizado como um molde para a amplificação dos genes de ARNr 16S subunidade pequena. Utilizando os iniciadores sugerido, este deve gerar um amplicão de cerca de 1450 pb. Porque um grande número de colónias pode ser exibido em qualquer momento, é empregue um método de rastreio hierárquica. 16S produtos de PCR amplificados foram digeridos com a enzima de restrição Hinf 1, e estas digestões são separados em géis de agarose. Espécies individuais vai gerar padrões distintos após digerir, de tal modo que os isolados da mesma espécie podem ser facilmente identificados (Figura 3D). A sequenciação dos produtos de PCR do gene de ARNr 16S a partir de um máximo de trêsisola representando qualquer padrão de uma restrição permite a identificação eficiente de todas as espécies susceptíveis em uma coleção de integron isolados positivos.
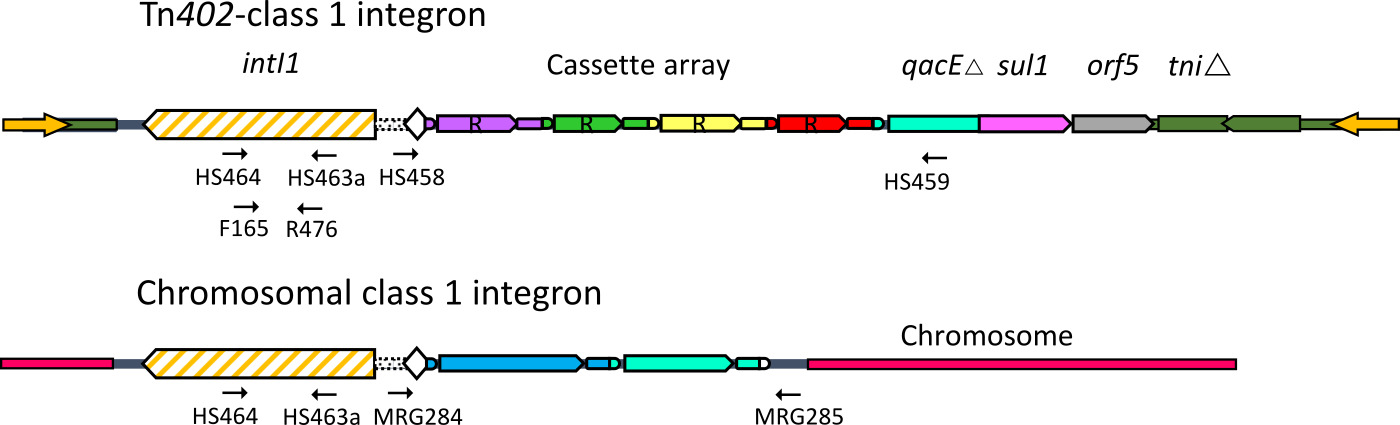
Figura 1. Estrutura de classe 1 integrons. Diagramas esquemáticos de classe clínica e ambiental, mostrando uma integrons PCR locais de ligação primário que se refere o manuscrito. Classe 1 integrões consistir de um gene íntegron-integrase (intI1) que catalisa a captação e a expressão de cassetes de genes de modo a formar uma matriz de cassete. Antes do advento do uso de antibióticos, a maioria de classe 1 integrons foram cromossômica, e levou cassetes de genes cujas funções são ainda a ser determinado. Seleção forte via uso de antibióticos aumentou muito a abundância de uma variante de sequência de classe 1 integron associado ao Tn 402 transposon.Estes integrons 'clínicas' adquiriram matrizes de cassetes que codificam a resistência aos antibióticos. É estes integrons que estão atualmente poluentes ambientes naturais e da cadeia de produção de alimentos. Por favor clique aqui para ver uma versão maior desta figura.

Figura 2. fluxograma esquemático para a detecção de uma classe integrões em produtos alimentares. Exemplos de alimentos são usados para inocular meios e gerar uma cultura bacteriana mista. Estas culturas mistas são rastreados quanto à presença de uma classe integrões, e culturas positivas utilizado para preparar placas recobertas. As colónias individuais das placas de propagação são novamente pesquisadas para integrons. As culturas positivas são purificados, o DNA extraído e caracterizado quanto ao teor de ADN por si só casseteQuencing. Seqüenciamento do gene 16S rRNA é usado para identificação de espécies. Por favor clique aqui para ver uma versão maior desta figura.

Figura 3. análise electroforética representativas do processo de triagem. (A) Rastreio colónias individuais utilizando os iniciadores intI1 HS463a / 464. As colónias positivas gerar uma forte banda única em 471 pb. (B) Amplificação de matrizes de cassetes de Tn 402 integrons utilizando iniciadores HS458 / 459. Este produto de PCR gera tamanhos variável dependente da identidade e tamanho das cassetes de componentes da matriz. A identificação destas cassetes exige sequenciação de ADN. (C) Amplificação de matrizes de cassetes de classe ambiental 1 integrons utilizando iniciadores MRG284 / 285. Este produto de PCR também gera tamanhos variável dependente da identidade e tamanho das cassetes de componentes na matriz, no entanto cassetes em matrizes ambientais é improvável que codificam a resistência aos antibióticos. (D) Rastreio dos produtos de PCR 16S por digestão com Hinf 1. Os isolados com padrões de restrição idênticos é provável que sejam da mesma espécie. Se um grande número de um único tipo de restrição são recuperadas, apenas alguns deles precisam ser sequenciado. Por favor clique aqui para ver uma versão maior desta figura.
| PCR | Gene alvo | Nome do iniciador | Direção | Sequência de 5 '- 3' | Condições Ciclismo | REFERÊnce |
| HS463a / HS464 | Classe 1 Integron | HS464 | Para a frente | ACATGCGTGTAAATCATCGTCG | 94 ° C 3 min; 35 ciclos 94 ° C 30 seg, 60 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 minutos | Stokes et al., 2006 28 |
| HS463a | Reversa | CTGGATTTCGATCACGGCACG | ||||
| F27 / r1492 | 16S rRNA | F27 | Para a frente | AGAGTTTGATCMTGGCTCAG | 94 ° C 3 min; 35 ciclos 94 ° C 30 seg, 60 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 minutos | Lane, 1991 29 |
| r1492 | Reversa | TACGGYTACCTTGTTACGACTT | ||||
| intI1F165 / IntI1R476 | Classe clínica 1 integron | Inti1F165 | Para a frente | CGAACGAGTGGCGGAGGGTG | 94 ° C 3 min; 35 ciclos 94 ° C 30 seg, 55 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 minutos | Gillings de 2014 10 |
| intI1R476 | Reversa | TACCCGAGAGCTTGGCACCCA | ||||
| HS549 / HS550 | 3'CS Clínica Integron | HS549 | Para a frente | ACTAAGCTTGCCCCTTCCGC | 94 ° C 3 min; 35 ciclos 94 ° C 30 seg, 65 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 minutos | Stokes et al., 2006 28 |
| HS550 | Reversa | CTAGGCATGATCTAACCCTCGG | ||||
| HS458 / HS459 | Cassete matriz clínica | HS458 | Para a frente | GCAAAAAGGCAGCAATTATGAGCC | 946; C 3 min; 35 ciclos 94 ° C 30 seg, 55 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 minutos | Holmes et al., 2003 30 |
| HS459 | Reversa | GTTTGATGTTATGGAGCAGCAACG | ||||
| MRG284 / MRG285 | Cassete Ambiental | MRG284 | Para a frente | GTTACGCCGTGGGTCGATG | 94 ° C 3 min; 35 ciclos 94 ° C 30 seg, 55 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 minutos | Gillings et al., 2009 21 |
| MRG285 | Reversa | CCAGAGCAGCCGTAGAGC |
Tabela 1. As sequências dos iniciadores e condições de PCR utilizados para a identificação de uma classe integrões e as suas cassetes de genes associados.
| Os reagentes de PCR | Volume (uL) | Restrição reagentes Digest | Volume (uL) |
| Água estéril | 21,5 | Água estéril | 23 |
| 10x GoTaq Branco | 25 | Tampão B | 5 |
| Com ARNase A [1 mg / ml] | 0,5 | Albumina de soro bovino [1 mg / ml] | 1 |
| Adiante Primer [50? M] | 0,5 | HinfI [10 unidades / ul] | 1 |
| Reverter Primer [50 mM] | 0,5 | ||
| Volume de Mastermix | 48 | Volume de Mastermix | 30 |
| ADN molde | 2 | Template PCR | 20 |
| O volume final de PCR | 50 | Restrição último volume digest | 50 |
Tabela 2. PCR e restrição digerir Mastermix constituintes.
Discussão
A identificação de integrons e suas cassetes de genes associados é potencialmente um passo fundamental para prever o surgimento de novos patógenos oportunistas, acompanhamento de percursos para patógenos na cadeia alimentar humana e identificando nova resistência e virulência determinantes 8,21,26. O objetivo deste trabalho foi descrever uma abordagem simplificada para a triagem de amostras para a classe 1 integrons, caracterizando suas matrizes cassete e identificar as espécies de bactérias em que residem. Etapas críticas do protocolo envolvem boa prática de microbiologia e prevenção da contaminação PCR que iria gerar falsos positivos.
O protocolo descrito aqui pode ser facilmente modificado para detectar outros integrons clinicamente relevantes, incluindo a classe 2 e classe 3 integrons que também são encontrados em patógenos humanos. Ele também pode ser modificado para detectar integrões em comunidades microbianas a partir de água, os biofilmes, do solo ou de sedimento. Existem alguns limitations a esta técnica, que surgem a partir de uma dependência de cultura de células bacterianas. Muitas bactérias ambientais não são facilmente cultivável, e o protocolo descrito aqui não iria detectar estas espécies. A gama de espécies que são recuperados poderia ser expandida por meio de diferentes formulações de meios de bactérias e os tempos de incubação mais longos. No entanto, a maioria das espécies de interesse para a saúde humana são susceptíveis de crescer nas condições descritas aqui.
Este protocolo tem algumas vantagens sobre as técnicas que utilizam plaqueamento em meios seletivos. Não precisam ser feitas suposições sobre a identidade de determinantes de resistência realizadas por isolados individuais, e genes de resistência a novos podem ser recuperados e caracterizados. Em termos mais gerais, o fluxo de trabalho também pode ser adaptado para detectar qualquer elemento do mobilome resistome 27 ou que possam ser de interesse 4. Tais ensaios são importantes para a compreensão da dinâmica dos vários elementos de DNA envolvidas em umtibiotic resistência e crítica para a conservação do arsenal cada vez menor de compostos antimicrobianos.
Divulgações
Os autores não têm nada de relevante a divulgar.
Agradecimentos
Graças a Michaela Hall, Larissa Bispo e Gustavo Tavares de Assistência Técnica.
Materiais
| Name | Company | Catalog Number | Comments |
| GoTaq Colourless Mastermix | Promega | M7132 | Used in all PCRs |
| RNAse (Ribonuclease A from bovine pancreas) | Sigma | R6513-10MG | Used in all PCRs |
| HinFI restriction enzyme | Promega | R6201 | Used to digest 16S rDNA PCR poducts. Enzyme comes with optimal buffer and BSA |
| 100 bp ladder | GE Healthcare | 27400701 | Used as a size standard on all agarose gels |
| GelRed DNA stain | Biotium | 41003 | CAUTION: Personal protection must be worn when handling this material |
| Guanidinium thiocyanate | Life Technologies | AM9422 | CAUTION: Personal protection must be worn when handling this material |
| CLS-TC Solution | MP Biomedicals | 6540409 | Resuspension solution used at the begining of the genomic DNA extraction |
| Lysing Matrix E FastPrep tubes | MP Biomedicals | 116914500 | Tube required for mechanical disruption of bacterial cell walls. This code is used for packs of 500 tubes, smaller quantities are available. |
| Binding matrix | MP Biomedicals | 116540408 | Diluted 1:5 with 6 M guanidinium thiocyanate and used in the genomic DNA extraction method. |
| Fast Prep machine | MP Biomedicals | Number of options available | MP Biomedicals has a number of FastPrep machines available to purchase. Visit http://www.mpbio.com for more information |
Referências
- Bush, K., et al. Tackling antibiotic resistance. Nature Rev. Microbiol. 9, 894-896 (2011).
- Davies, J., Davies, D. Origins and evolution of antibiotic resistance. Microbiol. Mol. Biol. Rev. 74 (3), 417-433 (2010).
- Costa, V. M., et al. Antibiotic resistance is ancient. Nature. 477 (7365), 457-461 (2011).
- Gillings, M. R. Evolutionary consequences of antibiotic use for the resistome, mobilome and microbial pangenome. Front. Microbiol. 4, 4(2013).
- Gillings, M., et al. The evolution of class 1 integrons and the rise of antibiotic resistance. J. Bacteriol. 190 (14), 5095-5100 (2008).
- Brassard, S., Lapointe, J., Roy, P. H. Diversity and relative strength of tandem promoters for the antibiotic-resistance genes of several integrons. Gene. 142 (1), 49-54 (1994).
- Partridge, S. R., et al. Definition of the attI1 site of class 1 integrons. Microbiol. 146 (11), 2855-2864 (2000).
- Gillings, M. R. Integrons: Past, Present, and Future. Microbiol. Mol. Biol. Rev. 78 (2), 257-277 (2014).
- Partridge, S. R., Tsafnat, G., Coiera, E., Iredell, J. R. Gene cassettes and cassette arrays in mobile resistance integrons. FEMS Microbiol. Rev. 33 (4), 757-784 (2009).
- Gillings, M. R., et al. Using the class 1 integron-integrase gene as a proxy for anthropogenic pollution. ISME J. In press, (2014).
- Gaze, W. H., et al. Impacts of anthropogenic activity on the ecology of class 1 integrons and integron-associated genes in the environment. ISME J. 5, 1253-1261 (2011).
- Gerzova, L., et al. Characterization of microbiota composition and presence of selected antibiotic resistance genes in carriage water of ornamental fish. PLoS ONE. 9, e103865(2014).
- Power, M., Emery, S., Gillings, M. Into the wild: dissemination of antibiotic resistance determinants via a species recovery program. PLoS ONE. 8, e63017(2013).
- Stokes, H. W., Gillings, M. R. Gene flow, mobile genetic elements and the recruitment of antibiotic resistance genes into Gram-negative pathogens. FEMS Microbiol. Rev. 35 (5), 790-819 (2011).
- Moura, A., Oliveira, C., Henriques, I., Smalla, K., Correia, A. Broad diversity of conjugative plasmids in integron-carrying bacteria from wastewater environments. FEMS Microbiol. Lett. 330 (2), 157-164 (2012).
- Schlüter, A., Krause, L., Szczepanowski, R., Goesmann, A., Pühler, A. Genetic diversity and composition of a plasmid metagenome from a wastewater treatment plant. J. Biotech. 136 (1-2), 65-76 (2008).
- Taylor, N. G. H., Verner-Jeffreys, D. W., Baker-Austin, C. Aquatic systems: maintaining, mixing and mobilising antimicrobial resistance. TREE. 26 (6), 278-284 (2011).
- Stalder, T., et al. Quantitative and qualitative impact of hospital effluent on dissemination of the integron pool. ISME J. 8, 768-777 (2014).
- Allen, H. K., et al. Call of the wild: antibiotic resistance genes in natural environments. Nature Rev. Microbiol. 8, 251-259 (2010).
- Wellington, E. M., et al. The role of the natural environment in the emergence of antibiotic resistance in Gram-negative bacteria. Lancet Infect. Dis. 13 (2), 155-165 (2013).
- Gillings, M. R., et al. Mobilization of a Tn402-like class 1 integron with a novel cassette array via flanking miniature inverted-repeat transposable element-like structures. Appl. Env. Microbiol. 75 (18), 6002-6004 (2009).
- Graham, D. W., Collignon, P., Davies, J., Larsson, D. J., Snape, J. Underappreciated role of regionally poor water quality on globally increasing antibiotic resistance. Env. Sci. Technol. 48 (20), 11746-11747 (2014).
- Perry, J. A., Westman, E. L., Wright, G. D. The antibiotic resistome: what's new. Curr. Opinion Microbiol. 21, 45-50 (2014).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. 63, e3998-e3998 (2011).
- Gillings, M. R. Rapid Extraction of PCR-Competent DNA from Recalcitrant Environmental Samples. Env. Microbiol. 1096, 17-23 (2014).
- Sajjad, A., Holley, M. P., Labbate, M., Stokes, H., Gillings, M. R. Preclinical class 1 integron with a complete Tn402-like transposition module. Appl. Env. Microbiol. 77 (1), 335-337 (2011).
- Wright, G. D. Antibiotic resistance in the environment: A link to the clinic. Curr. Opinion Microbiol. 13 (5), 589-594 (2010).
- Stokes, H., Nesbo, C., Holley, M., Bahl, M., Gillings, M., Boucher, Y. Class 1 integrons predating the association with Tn402.-like transposition genes are present in a sediment microbial community. Journal of Bacteriology. 188, 5722-5730 (2006).
- Lane, D. J. Nucleic Acid Techniques in Bacterial Systematics. , John Wiley &Sons, Inc. 115-175 (1991).
- Holmes, A. J., et al. Recombination activity of a distinctive integron-gene cassette system associated with stutzeri. populations in soil. Journal of Bacteriology. 185, 918-928 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados