Method Article
Screening prodotti alimentari per la Classe 1 integroni e Gene cassette
In questo articolo
Riepilogo
This protocol describes the detection of class 1 integrons and their associated gene cassettes in foodstuffs.
Abstract
Antibiotic resistance is one of the greatest threats to health in the 21st century. Acquisition of resistance genes via lateral gene transfer is a major factor in the spread of diverse resistance mechanisms. Amongst the DNA elements facilitating lateral transfer, the class 1 integrons have largely been responsible for spreading antibiotic resistance determinants amongst Gram negative pathogens. In total, these integrons have acquired and disseminated over 130 different antibiotic resistance genes. With continued antibiotic use, class 1 integrons have become ubiquitous in commensals and pathogens of humans and their domesticated animals. As a consequence, they can now be found in all human waste streams, where they continue to acquire new genes, and have the potential to cycle back into humans via the food chain. This protocol details a streamlined approach for detecting class 1 integrons and their associated resistance gene cassettes in foodstuffs, using culturing and PCR. Using this protocol, researchers should be able to: collect and prepare samples to make enriched cultures and screen for class 1 integrons; isolate single bacterial colonies to identify integron-positive isolates; identify bacterial species that contain class 1 integrons; and characterize these integrons and their associated gene cassettes.
Introduzione
La scoperta degli antibiotici è stata una delle più grandi conquiste scientifiche del 20 ° secolo. Tuttavia, l'uso e l'abuso di antibiotici ha portato alla rapida evoluzione dei batteri resistenti agli antibiotici, e questi ora rappresentare una seria minaccia per la salute pubblica nel 21 ° secolo. L'aumento di ceppi batterici resistenti alla maggior parte delle opzioni di trattamento solleva la possibilità che stiamo entrando in un'era in cui i farmaci antimicrobici non sono più efficaci 1,2.
Il macchinario genetico che conferisce resistenza agli antibiotici è un antico sistema, anticipando gli esseri umani e le pressioni di selezione antibiotici da milioni di anni 3. Elementi genetici mobili, come i plasmidi, trasposoni, isole genomiche, elementi di coniugazione integrativi e integroni possono diffondere geni resistenti agli antibiotici (ARG), sia all'interno e tra specie batteriche 4. Di questi, integroni hanno svolto un ruolo centrale nella diffusione di ARG, nonostante lafatto che si basano su plasmidi e trasposoni per la mobilitazione e l'inserimento in genomi batterici 5. Integroni catturano cassette geniche che utilizzano un integrone-integrasi, e quindi esprimono cassette utilizzando un integrone codificato promotore 6,7 (Figura 1). Integron gene cassette sono piccoli elementi mobili costituiti da singole fasi di lettura aperti (ORF) i cui prodotti possono conferire resistenza agli antibiotici e disinfettanti 8. Classe 1 integroni sono i integroni più comunemente recuperati da isolati clinici 5, dove hanno complessivamente acquisito oltre 130 diverse cassette antibiotico gene di resistenza 9.
La diffusione di classe 1 integroni in commensali e batteri patogeni umani associata genera flussi di rifiuti umani che contengono un gran numero di questi elementi genetici 10. Si stima che 10 19 batteri che contengono classe 1 integroni vengono rilasciati tramite fanghi di depurazione ogni anno in il Regno Unito 11. Non è quindi sorprendente che classe 1 integroni che conferiscono resistenza agli antibiotici sono ora rilevati nel microbiota degli uccelli selvatici, pesci e altri fauna selvatica 12-14. Rilasciando integroni indietro nell'ambiente costituisce una significativa minaccia per la salute pubblica, dal momento che l'acquisizione di nuove cassette geniche e riarrangiamenti complessi con altri elementi mobili continua a verificarsi, soprattutto negli impianti di trattamento delle acque reflue e di altri corpi idrici 15-18. L'ambiente naturale diventa un terreno di reclutamento fertile per i nuovi determinanti di resistenza e patogeni opportunisti 19,20. Batteri integrone contenente Nuovi e nuovi ARG possono fare il giro nella comunità umana attraverso acqua e cibo contaminati 21,22. Sorveglianza di ARG ambientali è una strategia chiave per la comprensione e la gestione di resistenza agli antibiotici in futuro 23. In particolare, occorre prestare attenzione agli alimenti che vengono consumati crudi opoco cotti, dal momento che questi rappresentano la più grande minaccia per la trasmissione di nuovi elementi mobili e gli agenti patogeni.
In questo protocollo, un approccio semplificato per il rilevamento, l'identificazione e la caratterizzazione di classe 1 integroni e le loro cassette geniche associate nei prodotti alimentari sono delineati (Figura 2). Utilizzando una combinazione di reazione coltura e della polimerasi a catena (PCR), integroni possono essere rapidamente individuati nelle comunità batteriche complesse e singoli isolati. I metodi per identificare le specie di batteri e la conformazione e l'identità delle cassette geniche Integron associata sono dati. Il metodo è adatto per una vasta gamma di alimenti vegetali e animali, ed esempi di workflow tipici sono fornite per ciascuno di questi tipi di alimenti.
Protocollo
I prodotti alimentari che vengono consumati crudi o poco cotti sono più preoccupazione per la salute umana. Gli esempi includono insalata di verdure, frutta, frutti di mare e crostacei.
1. Raccolta dei campioni
- Raccogliere i campioni in condizioni che riducono al minimo la contaminazione, e conservati in sacchetti puliti, separati durante il trasporto. Una volta raccolti, i campioni devono essere conservati a 4 ° C e trattati entro 24 ore.
2. Enriched Cultura Preparazione
- Frutta e verdura:
- Posizionare circa 10 g di materiale in un sacchetto di plastica resistente. Se l'elaborazione di materiale più grande, concentrarsi sulla pelle della frutta o verdura. Aggiungere 90 ml di 0,1 M tampone fosfato di sodio, pH 7,0 e di processo in un miscelatore a pale per 30 sec. In alternativa, il materiale può essere agitato manualmente se un miscelatore a pale non è disponibile.
- Raccogliere il liquido in due provette da 50 ml Falcon. Provette per centrifuga a 4.000 xg per 7 minuti a batteri pellet.
- Rimuovere con attenzione il surnatante e sospendere i batteri pellet in 30 ml di sterile Luria Bertani (LB) brodo.
- Incubare su un agitatore O / N, tenendo un tubo a 25 ° C e l'altro a 37 ° C. Le colture devono essere agitati a 200 oscillazioni al minuto (OPM). Conservare le culture a 4 ° C fino a quando necessario.
- Frutti di mare:
- Sezionare lo stomaco (ostriche) o del tratto digestivo (gamberi) con pinza sterile e pinzette e posto in una provetta sterile da 1,5 ml contiene 200 ml di tampone fosfato 0,1 M di sodio, pH 7,0. Macerare il campione per creare un omogenato.
- Dispensare 5 ml di brodo LB in due sterili provette da 5 ml, e inoculare ciascuna con l'omogeneizzato frutti di mare.
- Incubare su un agitatore O / N, tenendo un tubo a 25 ° C e l'altro 37 ° C. Le colture devono essere agitati a 200 opm. Conservare le culture a 4 ° C fino a quando necessario.
3. Culture screening per integroni
NOTA: protocolli di PCR standard sono utilizzati in tutto questa metodologia, utilizzando tamponi forniti con l'enzima, e una concentrazione finale MgCl 2 di 2,5 mm. Se necessario, Lorenz (2011) 24 ha delineato l'ottimizzazione e la risoluzione dei problemi di PCR metodi in un numero precedente di questa rivista.
- La preparazione del DNA Bollito:
- Dispensare 100 l di cultura arricchito in una sterile 0,5 ml provetta PCR. Riscaldare il campione a 99 ° C per 10 minuti, utilizzando un bagnomaria o calore. Snap freddo in ghiaccio per 2 minuti.
- Microcentrifuga a 14000 g per 5 minuti per far sedimentare i detriti cellulari. Ritorno a ghiaccio. I campioni di DNA possono essere congelati a -20 ° C fino al momento, ma devono essere scongelati in ghiaccio.
- Screening per la classe 1 integroni utilizzando PCR:
NOTA: culture miste dal / N incubazione O sono proiettati per la classe 1 integroni che utilizzano il protocollo HS463a / HS464 PCR (Tabella 1).- Scongelare tutti i reagenti PCR su ghiaccio, lontano da qualsiasi potenziale souRCE di contaminazione del DNA. Make up 48 microlitri PCR mastermix per campione, inclusi i controlli negativi (Tabella 2). Dispensare 48 ml di PCR master mix in ogni provetta, con punte di barriera.
- Aggiungere 2 ml di DNA scongelata per provette PCR singole con puntali. Eseguire apposito programma nel termociclatore PCR.
- Per valutare i risultati della amplificazione, elettroforesi 7 ml di prodotto di PCR su un gel di agarosio al 2% versato ed eseguire in tampone TBE (90 mM TRIS-borato, 2 mM EDTA). Include un marcatore di peso molecolare, come ad esempio una scala di 100 bp.
- Posta macchia il gel con una macchia DNA, e visualizzare utilizzando un transilluminatore UV. Verificare che non vi è stata alcuna amplificazione nel controllo negativo. Una forte banda a circa 470 bp per qualsiasi campione particolare indica che la cultura mista contiene batteri che portano una classe 1 integrone. Colonie Pure verranno isolate da qualsiasi cultura mista che ha restituito una PCR positiva.
4. Proiezione di singole colonie per Classe 1 integroni
- Diluizioni seriali e piastre spread:
NOTA: Per isolare singole colonie da colture miste PCR-positivi, la coltura mista è serialmente diluiti 10 volte in tampone fosfato di sodio, e poi placcato recuperare singole colonie.- Aggiungere 1 ml di coltura mista a 9 ml di tampone fosfato 0,1 M di sodio, pH 7,0 e mescolare per inversione. Aggiungere 1 ml della diluizione ad un ulteriore 9 ml di tampone fosfato, e ripetere fino una diluizione seriale di 10 -8 è raggiunto.
- Stendere 100 ml di i 10 -4 a 10 -8 diluizioni su agar LB, in duplice copia. Incubare le piastre O / N alla stessa temperatura utilizzata per la coltura mista iniziale. Le piastre possono quindi essere conservati a 4 ° C fino al momento, ma è meglio elaborare le singole colonie abbastanza rapidamente.
- Selezione colonia unico e la preparazione del DNA bollito:
- Scegli singole colonie dalle piastre di diluizione seriali, SEnando come molti tipi differenti di colonie possibile, utilizzando criteri come le dimensioni delle colonie, forma e colore. Usa stuzzicadenti sterile per selezionare singole colonie dalla piastra diffusione.
- Toccare lo stuzzicadenti alla colonia, e poi trasferire in un tubo di PCR contenente 100 ml di acqua sterile. Se un gran numero di colonie devono essere sottoposti a screening, quindi le colonie possono essere preparati in un vassoio di microtitolazione. Spin stuzzicadenti tra le dita per rimuovere alcune delle cellule in acqua.
- Utilizzando lo stesso stecchino, inoculare una piastra LB con una striscia della colonia. Un gran numero di colonie possono essere memorizzati su piastre etichettati se ogni striscia è lungo circa 1 cm. Ripetere i passaggi da 1 a 3 fino a selezionare un numero adeguato di isolati.
- Incubare la piastre LB O / N, alla stessa temperatura di quello usato per la coltura arricchita iniziale. Le culture possono quindi essere conservati a 4 ° C fino all'utilizzo.
- Per la preparazione del DNA dalle sospensioni batteriche, seguire la procedura come outlined nella sezione 3.1. Per lo screening dei lisati coltura pura, eseguire la HS463a / HS464 PCR come indicato nella sezione 3.2. Gli isolati che restituiscono un PCR positivo sarà utilizzato per la preparazione di DNA genomico (sezione 5), e per ulteriori analisi.
5. Le colonie singole genomico Estrazione del DNA di Integron positivi Uso Bead Beating 25
- Inoculare 5 ml di brodo LB con un campione della coltura pura isolato nella sezione 4.2. Incubare O / N, agitando alla stessa temperatura che è stato utilizzato per la coltura arricchita iniziale.
- Cellule pellet a 4.000 xg per 7 minuti, quindi rimuovere il surnatante. Risospendere le cellule pellet in 1 ml di soluzione CLS-TC in un tubo di preparazione veloce di Lisi Matrix E. Utilizzando una macchina tallone pestaggio, elaborare il campione a 5,5 m / sec per 30 sec.
- Provetta da centrifuga a 14000 xg per 5 min e recuperare 700 pl di surnatante in una provetta sterile da 1.5 ml.
- Aggiungere 700 ml di matrice legante diluito 1: 5 con 6 M guanidinium tiocianato e mescolare a temperatura ambiente per 5 minuti. Centrifugare a 14000 g per 1 minuto, il surnatante scarto.
- Aggiungere 800 microlitri etanolo di lavaggio sale (70% di etanolo, 0,1 M di acetato di sodio) e vortex fino a quando il pellet è completamente risospeso. Lavare per 5 minuti a RT, centrifugare a 14.000 xg per 1 minuto e scartare il surnatante. Assicurarsi tutte surnatante è stato rimosso, usando una micropipetta se necessario, quindi lasciare asciugare all'aria per 5 min.
- Risospendere la matrice pellet in 200 microlitri TE (10 mM Tris-HCl, 1 mM EDTA, pH 7,6) pipettando su e giù. Incubare a temperatura ambiente per 3 min. Centrifugare a 14.000 xg per 3 min e trasferimento 160 microlitri della DNA eluito in una provetta sterile. Eseguire una aliquota della estrazione del DNA genomico su 2% w / v gel di agarosio per verificare la resa e l'integrità della estrazione del DNA. Il DNA genomico purificato è conservato a -20 ° C e scongelato in ghiaccio quando richiesto per il test PCR.
6. PCR diagnostici e sequenziamento del DNA
NOTA: IlDNA genomico preparata al punto 5 sarà utilizzato per tutte le PCR diagnostici, e per confermare il test positivo per la classe 1 integroni.
- Scongelare il DNA genomico sul ghiaccio, vortice brevemente il campione di DNA e di spin impulso a 14000 g per 20 sec. Ripetere la PCR per la classe 1 integrone-integrasi gene utilizzando HS463a / HS464 (Tabella 1), a conferma che l'isolato è positivo per l'amplicone 471 bp (Sezione 3.2).
- Identificazione delle colture positive a livello di specie è ottenuta mediante analisi della piccola subunità gene rRNA (16S rDNA). Amplificare il gene 16S con primer f27 / r1492 (Tabella 1). Controllare i prodotti PCR mediante elettroforesi. Tutti gli obiettivi batterici dovrebbero generare un 16S amplicone di circa 1450 bp.
- Sequenza gene amplicone 16S rRNA per determinare l'identità delle specie, ma dal momento che ci sono probabilmente più PCR positivi dal punto 6.2, e molti di questi aspetti positivi saranno le stesse specie di batteri, abbiamo in primo luogo distinguere diverse specie using restrizione digestione del prodotto di PCR 16S.
- Digest un'aliquota del 16S PCR con l'enzima di restrizione Hinf 1. Ogni enzima che riconosce siti di rottura 4 bp farà, ma Hinf 1 genera buona diversità dagli obiettivi 16S, ed è un affidabile, enzima poco costoso.
- Impostare una limitazione di 30 microlitri digerire mastermix (Tabella 2) in una provetta sterile e aggiungere 20 ml di un-purfied prodotto 16S PCR. Incubare a 37 ° C bagnomaria O / N. Controllare il digest su un gel di agarosio al 2%.
- Nota i modelli prodotti dalla digestione Hinf 1. Isolati con la stessa 16S profilo di restrizione saranno probabilmente della stessa specie. Piuttosto che sequenziare ogni prodotto 16S PCR, sequenza al massimo tre rappresentanti di ogni Hinf 1 restrizione modello.
- Purificare i prodotti 16S PCR utilizzando un kit commerciale. Un unico kit exonuclease filamento può essere utilizzato, ma sulla base di colonna o metodi di precipitazione anche lavorare bene per rimuovere unincprimer orporated e DNA cavo rigido. Sequenza 16S amplificati usando Primer R910 (Tabella 1), che istituiscono le reazioni, come specificato dalla struttura sequenziamento.
- Utilizzare la sequenza di DNA derivati di interrogare la banca dati del DNA NCBI con la funzione BLASTN ( http://blast.ncbi.nlm.nih.gov/Blast.cgi ). Superiore al 97% nucleotide identità è solitamente considerato come l'identità di specie.
7. mappatura e caratterizzazione di integroni e cassette Array
NOTA: Classe 1 integroni provenienti da ecosistemi umani dominati sono probabilmente i integroni più comuni in tutti i campioni. Questi integroni tutti hanno un recente un'unica origine, e quindi hanno una sequenza di DNA altamente conservata 5.
- Distinguere tra una classe 1 integroni provenienti da fonti umane e quelle che si trovano in natura in campioni ambientali eseguendo unaPCR con primer set intI1F165 / intI1R476 10 (Tabella 1).
NOTA: Classe 1 integroni da batteri clinici o commensali genererà un prodotto ~ 300 pb, mentre la classe ambientale 1 integroni non genereranno alcun prodotto. - Caratterizzare le integroni clinici amplificando matrice cassetta con primer HS458 / HS459. Questo PCR amplifica la regione tra intI1 e segmento conservato il 3 'al capolinea della maggior parte degli array di cassette. Poiché il numero e l'identità di cassette è variabile, le dimensioni del prodotto di PCR anche variare.
- Purificare ampliconi e sequenziare il DNA utilizzando ciascuno dei primer di amplificazione. Utilizzare sequenze di interrogare banche dati del DNA utilizzando la funzione BLASTN ( http://blast.ncbi.nlm.nih.gov/Blast.cgi ).
- Garantire la denominazione di cassette seguire nomenclatura standardizzata 9, dal momento che molte deposizioni di dati per cassette gene integrones usano nomi incoerenti o ambigui. Dare particolare attenzione alle cassette geniche nuovi, dal momento che questi possono codificare nuovi fenotipi, compresi i geni che conferiscono aumentata trasmissibilità, patogenicità o la virulenza 8,21.
NOTA: preclinici forme della classe 1 integrone circolano ancora in ambienti naturali. In particolare, coloro che sono legati alla attive Tn 402 trasposoni sono di maggior interesse 26. Gli array di cassette di questi integroni non amplificare con primer HS458 / HS459, ma possono essere amplificati con la MRG284 / MRG285 Primer set (Tabella 1), generando ampliconi di grandezza variabile dipendente sui contenuti cassetta. I prodotti di PCR devono essere purificati e DNA sequenziati come indicato al punto 7.2.
Risultati
Proiezione di colture miste e isolati batterici per intI1
Primer HS463a / HS464 PCR può essere utilizzato per rilevare la presenza della classe 1 integrone-integrasi gene, intI1 (Figura 1). Questo set di primer funziona bene per la rilevazione intI1 in colture miste, ed è anche utilizzato per lo screening colonie batteriche raccolte da dispositivo di spargimento (Figura 2). Isolati positivi dovrebbero generare una singola banda forte a 471 bp con questo set di primer (Figura 3A). La maggior parte degli isolati positivi porterà intI1 che si è originato da esseri umani o dei loro animali agricoli e domestici. Questi isolati saranno anche positivo in un PCR utilizzando primer F165 / R476, che dovrebbe generare un amplicone 311 bp (Figura 1). Fonti ambientali di intI1 non saranno generalmente positiva in questo secondo test.
Caratterizzazione di matrici cassette integrone
Il DNA genomico da isolati pure viene utilizzato per la caratterizzazione di matrici cassette integrone. Gli array di cassette di Tn 402 classe -associated 1 integroni possono essere amplificati utilizzando primer HS458 / HS459. Questi primer rispettivamente bersaglio la ricombinazione sito integrone, e all'estremità 3 'della matrice cassetta, che termina normalmente nella qacEΔ sul1 gene di fusione (Figura 1). La dimensione dei prodotti di PCR generati in questo dosaggio varia a seconda del numero e l'identità delle cassette nella matrice (Figura 3B). Classe ambientale 1 integroni sono spesso incorporati nei cromosomi batterici, e le loro matrici cassette possono essere amplificati da primer che colpiscono la maggior parte dei siti prossimali e distali ricombinazione (Figura 1). Primers MRG284 / 285 sono progettati per amplificare questa regione, e di nuovo, perché il contenuto della cassetta varia, la dimensione degli amplificati varia anche (Figura 3C). Il sequenziamento del HS458 / 459 PCRprodotti normalmente recuperare noti determinanti di resistenza agli antibiotici, mentre il sequenziamento MRG284 / 285 PCR prodotti generalmente recuperare cassette geniche che codificano polipeptidi di funzione sconosciuta.
Identificare le specie batteriche
I batteri sono identificati mediante sequenziamento del 16S rDNA e database confronti. Il DNA genomico è usato come modello per l'amplificazione dei 16S piccole subunità gene rRNA. Utilizzando i primers suggerito, questo dovrebbe generare un amplicone di circa 1450 bp. Poiché un gran numero di colonie possono essere proiettati contemporaneamente, un metodo di screening gerarchico è impiegato. Amplificati prodotti 16S PCR sono digeriti con l'enzima di restrizione Hinf 1, e questi digest sono separati su gel di agarosio. Specie individuali genereranno modelli distintivo dopo digerire, in modo tale che gli isolati della stessa specie possono essere facilmente identificabili (Figura 3D). Sequenziamento dei prodotti di PCR gene 16S rRNA da un massimo di treisola che rappresenta tutto il modello una restrizione consente l'identificazione efficiente di tutte le specie che possono in una collezione di integrone isolati positivi.
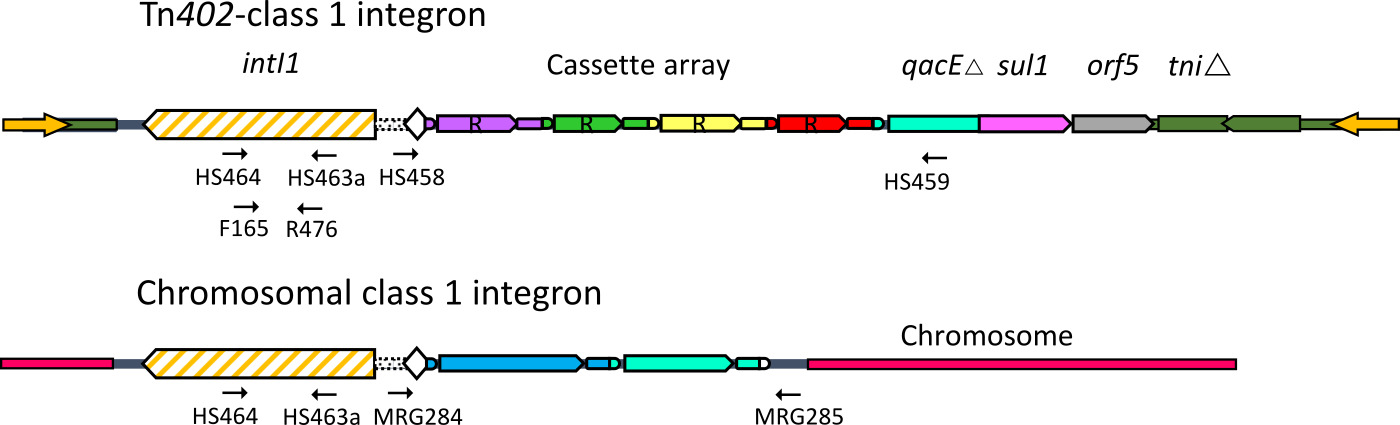
Figura 1. Struttura di classe 1 integroni. Diagrammi di classe clinica e ambientale 1 integroni, mostrando PCR Primer siti di legame di cui al manoscritto. Classe 1 integroni sono costituiti da un gene integrone-integrasi (intI1) che catalizza la cattura e l'espressione di cassette geniche per formare una matrice di cassetta. Prima dell'avvento di uso di antibiotici, la maggior parte di classe 1 integroni erano cromosomico, e portava cassette geniche le cui funzioni sono ancora da determinare. Forte selezione tramite l'uso di antibiotici è notevolmente aumentato l'abbondanza di una variante sequenza di classe 1 integrone associata al Tn 402 trasposoni.Questi integroni 'cliniche' hanno acquisito matrici di cassette che codificano la resistenza agli antibiotici. Sono questi integroni che attualmente inquinano ambienti naturali e della catena di produzione alimentare. Clicca qui per vedere una versione più grande di questa figura.

Figura 2. Schema diagramma di flusso per la rilevazione di classe 1 integroni negli alimenti. I campioni dei prodotti alimentari sono utilizzati per inoculare i terreni e generare una coltura batterica mista. Queste culture miste sono sottoposti a screening per la presenza di classe 1 integroni, e colture positive utilizzati per preparare piatti diffusione. Colonie individuali dalle piastre spread sono nuovo controllo per integroni. Colture positive sono purificati, DNA estratto e caratterizzata per il contenuto della cassetta dal DNA sequencing. Il sequenziamento del gene 16S rRNA viene utilizzato per l'identificazione delle specie. Clicca qui per vedere una versione più grande di questa figura.

Figura 3. elettroforetico rappresentante analizza dal processo di screening. (A) Screening singole colonie utilizzando intI1 primers HS463a / 464. Colonie positivi generano una forte banda di singolo a 471 bp. (B) Amplificazione di array di cassette da TN 402 integroni utilizzando primer HS458 / 459. Questo prodotto di PCR genera variabile dimensioni dipende dall'identità e dimensioni delle cassette componenti nella matrice. L'identificazione di queste cassette richiede il sequenziamento del DNA. (C) Amplificazione di array cassetta dalla classe ambientale 1 integrons utilizzando primer MRG284 / 285. Questa PCR genera anche prodotto variabile dimensioni dipende dall'identità e dimensioni delle cassette componenti nella matrice, tuttavia cassette in matrici ambientali difficilmente codificare resistenza agli antibiotici. (D) Screening di prodotti 16S PCR mediante digestione con Hinf 1. Gli isolati con profili di restrizione identici possono essere della stessa specie. Se si recuperano grandi quantità di un singolo tipo di restrizione, solo alcuni di questi devono essere sequenziato. Fate clic qui per visualizzare una versione più grande di questa figura.
| PCR | Bersaglio Gene | Nome Primer | Direzione | Sequenza 5 '- 3' | Condizioni ciclismo | Reference |
| HS463a / HS464 | Classe 1 Integron | HS464 | Avanti | ACATGCGTGTAAATCATCGTCG | 94 ° C 3 min; 35 cicli di 94 ° C 30 sec, 60 ° C 30 sec, 72 ° C 1 min 30 sec; 72 ° C 5 min | Stokes et al., 2006 28 |
| HS463a | Reverse | CTGGATTTCGATCACGGCACG | ||||
| f27 / r1492 | 16S rRNA | f27 | Avanti | AGAGTTTGATCMTGGCTCAG | 94 ° C 3 min; 35 cicli di 94 ° C 30 sec, 60 ° C 30 sec, 72 ° C 1 min 30 sec; 72 ° C 5 min | Lane, 1991 29 |
| r1492 | Reverse | TACGGYTACCTTGTTACGACTT | ||||
| intI1F165 / IntI1R476 | Classe clinico 1 integrone | intI1F165 | Avanti | CGAACGAGTGGCGGAGGGTG | 94 ° C 3 min; 35 cicli di 94 ° C 30 sec, 55 ° C 30 sec, 72 ° C 1 min 30 sec; 72 ° C 5 min | Gillings 2014 10 |
| intI1R476 | Reverse | TACCCGAGAGCTTGGCACCCA | ||||
| HS549 / HS550 | 3'CS Clinical Integron | HS549 | Avanti | ACTAAGCTTGCCCCTTCCGC | 94 ° C 3 min; 35 cicli di 94 ° C 30 sec, 65 ° C 30 sec, 72 ° C 1 min 30 sec; 72 ° C 5 min | Stokes et al., 2006 28 |
| HS550 | Reverse | CTAGGCATGATCTAACCCTCGG | ||||
| HS458 / HS459 | Matrice cassette clinica | HS458 | Avanti | GCAAAAAGGCAGCAATTATGAGCC | 946; C 3 min; 35 cicli di 94 ° C 30 sec, 55 ° C 30 sec, 72 ° C 1 min 30 sec; 72 ° C 5 min | Holmes et al., 2003 30 |
| HS459 | Reverse | GTTTGATGTTATGGAGCAGCAACG | ||||
| MRG284 / MRG285 | Cassette ambientale | MRG284 | Avanti | GTTACGCCGTGGGTCGATG | 94 ° C 3 min; 35 cicli di 94 ° C 30 sec, 55 ° C 30 sec, 72 ° C 1 min 30 sec; 72 ° C 5 min | 2009 21 Gillings et al. |
| MRG285 | Reverse | CCAGAGCAGCCGTAGAGC |
Tabella 1. sequenze primer e le condizioni di PCR utilizzati per l'identificazione di Classe 1 integroni e le loro cassette geniche associate.
| Reagenti PCR | Volume (ml) | Restrizione reagenti digest | Volume (ml) |
| Acqua sterile | 21.5 | Acqua sterile | 23 |
| 10x GoTaq Bianco | 25 | Buffer B | 5 |
| RNAseA [1 mg / ml] | 0.5 | Bovine Serum Albumin [1 mg / ml] | 1 |
| Forward Primer [50 micron] | 0.5 | HinfI [10 unità / ml] | 1 |
| Reverse Primer [50 micron] | 0.5 | ||
| Volume Mastermix | 48 | Volume Mastermix | 30 |
| Modello di DNA | 2 | PCR Template | 20 |
| Volume PCR finale | 50 | Restrizione finale del volume digest | 50 |
Tabella 2. PCR e restrizione digerire Mastermix costituenti.
Discussione
L'identificazione di integroni e le loro cassette geniche associate è potenzialmente un passo fondamentale nel predire l'emergere di nuovi patogeni opportunisti, tracking percorsi per gli agenti patogeni nella catena alimentare umana, e identificando nuova resistenza e la virulenza determinanti 8,21,26. Lo scopo di questo lavoro è stato quello di descrivere un approccio semplificato per lo screening dei campioni per la classe 1 integroni, che caratterizzano le loro matrici cassette e identificare le specie batteriche in cui risiedono. Passaggi critici nel protocollo riguardano buona prassi microbiologica, e prevenire la contaminazione della PCR che genererebbe falsi positivi.
Il protocollo qui descritto può essere facilmente modificato per rilevare altri integroni clinicamente rilevanti, compresa la classe 2 e classe 3 integroni che si trovano anche in agenti patogeni umani. Può anche essere modificato per rilevare integroni in comunità microbiche dall'acqua, biofilm, del suolo o sedimenti. Ci sono alcuni limitations a questa tecnica, che derivano da una dipendenza da coltura di cellule batteriche. Molti batteri ambientali non sono facilmente coltivabili, e il protocollo qui descritto non rileverebbero queste specie. La gamma di specie che sono state recuperate potrebbe essere ampliata utilizzando diverse formulazioni dei media batterica e tempi di incubazione più lunghi. Tuttavia, la maggior parte delle specie di interesse per la salute umana sono suscettibili di crescere sotto le condizioni qui descritte.
Questo protocollo presenta alcuni vantaggi rispetto alle tecniche che utilizzano placcatura su terreni selettivi. Nessuna ipotesi devono essere fatte sull'identità di determinanti di resistenza condotti da singoli isolati, e di nuovi geni di resistenza possono essere recuperati e caratterizzati. In termini più generali, il flusso di lavoro può essere atto a rilevare qualsiasi elemento del mobilome resistome 27 o che potrebbero essere di interesse 4. Tali test sono importanti per la comprensione della dinamica dei vari elementi di DNA coinvolti in unla resistenza antibiotica, e fondamentale per conservare l'armeria diminuzione dei composti antimicrobici.
Divulgazioni
Gli autori non hanno niente di rilevante da rivelare.
Riconoscimenti
Grazie a Michaela Hall, Larissa Bispo e Gustavo Tavares per l'assistenza tecnica.
Materiali
| Name | Company | Catalog Number | Comments |
| GoTaq Colourless Mastermix | Promega | M7132 | Used in all PCRs |
| RNAse (Ribonuclease A from bovine pancreas) | Sigma | R6513-10MG | Used in all PCRs |
| HinFI restriction enzyme | Promega | R6201 | Used to digest 16S rDNA PCR poducts. Enzyme comes with optimal buffer and BSA |
| 100 bp ladder | GE Healthcare | 27400701 | Used as a size standard on all agarose gels |
| GelRed DNA stain | Biotium | 41003 | CAUTION: Personal protection must be worn when handling this material |
| Guanidinium thiocyanate | Life Technologies | AM9422 | CAUTION: Personal protection must be worn when handling this material |
| CLS-TC Solution | MP Biomedicals | 6540409 | Resuspension solution used at the begining of the genomic DNA extraction |
| Lysing Matrix E FastPrep tubes | MP Biomedicals | 116914500 | Tube required for mechanical disruption of bacterial cell walls. This code is used for packs of 500 tubes, smaller quantities are available. |
| Binding matrix | MP Biomedicals | 116540408 | Diluted 1:5 with 6 M guanidinium thiocyanate and used in the genomic DNA extraction method. |
| Fast Prep machine | MP Biomedicals | Number of options available | MP Biomedicals has a number of FastPrep machines available to purchase. Visit http://www.mpbio.com for more information |
Riferimenti
- Bush, K., et al. Tackling antibiotic resistance. Nature Rev. Microbiol. 9, 894-896 (2011).
- Davies, J., Davies, D. Origins and evolution of antibiotic resistance. Microbiol. Mol. Biol. Rev. 74 (3), 417-433 (2010).
- Costa, V. M., et al. Antibiotic resistance is ancient. Nature. 477 (7365), 457-461 (2011).
- Gillings, M. R. Evolutionary consequences of antibiotic use for the resistome, mobilome and microbial pangenome. Front. Microbiol. 4, 4(2013).
- Gillings, M., et al. The evolution of class 1 integrons and the rise of antibiotic resistance. J. Bacteriol. 190 (14), 5095-5100 (2008).
- Brassard, S., Lapointe, J., Roy, P. H. Diversity and relative strength of tandem promoters for the antibiotic-resistance genes of several integrons. Gene. 142 (1), 49-54 (1994).
- Partridge, S. R., et al. Definition of the attI1 site of class 1 integrons. Microbiol. 146 (11), 2855-2864 (2000).
- Gillings, M. R. Integrons: Past, Present, and Future. Microbiol. Mol. Biol. Rev. 78 (2), 257-277 (2014).
- Partridge, S. R., Tsafnat, G., Coiera, E., Iredell, J. R. Gene cassettes and cassette arrays in mobile resistance integrons. FEMS Microbiol. Rev. 33 (4), 757-784 (2009).
- Gillings, M. R., et al. Using the class 1 integron-integrase gene as a proxy for anthropogenic pollution. ISME J. In press, (2014).
- Gaze, W. H., et al. Impacts of anthropogenic activity on the ecology of class 1 integrons and integron-associated genes in the environment. ISME J. 5, 1253-1261 (2011).
- Gerzova, L., et al. Characterization of microbiota composition and presence of selected antibiotic resistance genes in carriage water of ornamental fish. PLoS ONE. 9, e103865(2014).
- Power, M., Emery, S., Gillings, M. Into the wild: dissemination of antibiotic resistance determinants via a species recovery program. PLoS ONE. 8, e63017(2013).
- Stokes, H. W., Gillings, M. R. Gene flow, mobile genetic elements and the recruitment of antibiotic resistance genes into Gram-negative pathogens. FEMS Microbiol. Rev. 35 (5), 790-819 (2011).
- Moura, A., Oliveira, C., Henriques, I., Smalla, K., Correia, A. Broad diversity of conjugative plasmids in integron-carrying bacteria from wastewater environments. FEMS Microbiol. Lett. 330 (2), 157-164 (2012).
- Schlüter, A., Krause, L., Szczepanowski, R., Goesmann, A., Pühler, A. Genetic diversity and composition of a plasmid metagenome from a wastewater treatment plant. J. Biotech. 136 (1-2), 65-76 (2008).
- Taylor, N. G. H., Verner-Jeffreys, D. W., Baker-Austin, C. Aquatic systems: maintaining, mixing and mobilising antimicrobial resistance. TREE. 26 (6), 278-284 (2011).
- Stalder, T., et al. Quantitative and qualitative impact of hospital effluent on dissemination of the integron pool. ISME J. 8, 768-777 (2014).
- Allen, H. K., et al. Call of the wild: antibiotic resistance genes in natural environments. Nature Rev. Microbiol. 8, 251-259 (2010).
- Wellington, E. M., et al. The role of the natural environment in the emergence of antibiotic resistance in Gram-negative bacteria. Lancet Infect. Dis. 13 (2), 155-165 (2013).
- Gillings, M. R., et al. Mobilization of a Tn402-like class 1 integron with a novel cassette array via flanking miniature inverted-repeat transposable element-like structures. Appl. Env. Microbiol. 75 (18), 6002-6004 (2009).
- Graham, D. W., Collignon, P., Davies, J., Larsson, D. J., Snape, J. Underappreciated role of regionally poor water quality on globally increasing antibiotic resistance. Env. Sci. Technol. 48 (20), 11746-11747 (2014).
- Perry, J. A., Westman, E. L., Wright, G. D. The antibiotic resistome: what's new. Curr. Opinion Microbiol. 21, 45-50 (2014).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. 63, e3998-e3998 (2011).
- Gillings, M. R. Rapid Extraction of PCR-Competent DNA from Recalcitrant Environmental Samples. Env. Microbiol. 1096, 17-23 (2014).
- Sajjad, A., Holley, M. P., Labbate, M., Stokes, H., Gillings, M. R. Preclinical class 1 integron with a complete Tn402-like transposition module. Appl. Env. Microbiol. 77 (1), 335-337 (2011).
- Wright, G. D. Antibiotic resistance in the environment: A link to the clinic. Curr. Opinion Microbiol. 13 (5), 589-594 (2010).
- Stokes, H., Nesbo, C., Holley, M., Bahl, M., Gillings, M., Boucher, Y. Class 1 integrons predating the association with Tn402.-like transposition genes are present in a sediment microbial community. Journal of Bacteriology. 188, 5722-5730 (2006).
- Lane, D. J. Nucleic Acid Techniques in Bacterial Systematics. , John Wiley &Sons, Inc. 115-175 (1991).
- Holmes, A. J., et al. Recombination activity of a distinctive integron-gene cassette system associated with stutzeri. populations in soil. Journal of Bacteriology. 185, 918-928 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon