Method Article
Screening productos alimenticios para la Clase 1 integrones y Gene Cassettes
En este artículo
Resumen
This protocol describes the detection of class 1 integrons and their associated gene cassettes in foodstuffs.
Resumen
Antibiotic resistance is one of the greatest threats to health in the 21st century. Acquisition of resistance genes via lateral gene transfer is a major factor in the spread of diverse resistance mechanisms. Amongst the DNA elements facilitating lateral transfer, the class 1 integrons have largely been responsible for spreading antibiotic resistance determinants amongst Gram negative pathogens. In total, these integrons have acquired and disseminated over 130 different antibiotic resistance genes. With continued antibiotic use, class 1 integrons have become ubiquitous in commensals and pathogens of humans and their domesticated animals. As a consequence, they can now be found in all human waste streams, where they continue to acquire new genes, and have the potential to cycle back into humans via the food chain. This protocol details a streamlined approach for detecting class 1 integrons and their associated resistance gene cassettes in foodstuffs, using culturing and PCR. Using this protocol, researchers should be able to: collect and prepare samples to make enriched cultures and screen for class 1 integrons; isolate single bacterial colonies to identify integron-positive isolates; identify bacterial species that contain class 1 integrons; and characterize these integrons and their associated gene cassettes.
Introducción
El descubrimiento de los antibióticos fue uno de los mayores logros científicos del siglo 20. Sin embargo, el uso y abuso de antibióticos ha llevado a la rápida evolución de las bacterias resistentes a los antibióticos, y estos ahora representan una seria amenaza para la salud pública en el siglo 21. El surgimiento de cepas bacterianas resistentes a la mayoría de las opciones de tratamiento plantea la posibilidad de que estamos entrando en una era en que los medicamentos antimicrobianos ya no son eficaces 1,2.
La maquinaria genética que confiere resistencia a los antibióticos es un sistema antiguo, anterior a los seres humanos y las presiones de selección de antibióticos por millones de años 3. Elementos genéticos móviles, tales como plásmidos, transposones, islas genómicas, elementos y integrones conjugativos integradores pueden difundir los genes de resistencia a antibióticos (ARG), tanto dentro como entre las especies bacterianas 4. De éstos, los integrones han jugado un papel central en la propagación de ARG, a pesar de lahecho de que se basan en plásmidos y transposones para la movilización y la inserción en los genomas bacterianos 5. Los integrones capturan casetes de genes usando un integrón-integrasa, y luego expresan casetes utilizando un integrón codificado promotor 6,7 (Figura 1). Integrón gen casetes son pequeños elementos móviles que consisten en cuadros individuales abiertos de lectura (ORF), cuyos productos pueden conferir resistencia a los antibióticos o desinfectantes 8. 1 integrones de clase son los integrones más comúnmente recuperados de aislados clínicos 5, donde han adquirido colectivamente más de 130 diferentes casetes de genes de resistencia a antibióticos 9.
La difusión de la clase 1 integrones en comensales y patógenos bacterias humanas asociadas genera corrientes de desechos humanos que contienen un gran número de estos elementos genéticos 10. Se estima que 10 19 bacterias que contienen integrones de clase 1 se liberan a través de los lodos de depuradora cada año in el Reino Unido 11. Por lo tanto, no es sorprendente que la clase 1 integrones que confieren resistencias a antibióticos están siendo detectados en la microbiota de las aves silvestres, peces y otra fauna nativa 12-14. Liberar integrones de nuevo en el medio ambiente plantea un importante problema de salud pública, ya que la adquisición de nuevos cassettes genéticos y reordenamientos complejos con otros elementos móviles persiste, especialmente en las plantas de tratamiento de aguas residuales y otros cuerpos de agua 15-18. El entorno natural, entonces se convierte en un campo de reclutamiento fértil para nuevos determinantes de resistencia y patógenos oportunistas 19,20. Bacterias que contiene integrón-Novel y nuevas ARGs pueden rodear de nuevo en la comunidad humana a través del agua y alimentos contaminados 21,22. Vigilancia de ARGs ambientales es una estrategia clave para la comprensión y el manejo de la resistencia a los antibióticos en el futuro 23. En particular, se debe prestar atención a los alimentos que se comen crudos oligeramente cocidos, ya que estos presentan la mayor amenaza para la transmisión de nuevos elementos móviles y agentes patógenos.
En este protocolo, un enfoque simplificado para la detección, identificación y caracterización de integrones de clase 1 y de sus cassettes genéticos asociados en los productos alimenticios se indica (Figura 2). Usando una combinación de cultivo y reacción en cadena de polimerasa (PCR), integrones se pueden detectar rápidamente en comunidades bacterianas complejas y aislamientos individuales. Se dan métodos para la identificación de las especies de bacterias y la conformación y la identidad de los casetes de genes integrón-asociado. El método es adecuado para una amplia variedad de alimentos vegetales y animales, y los ejemplos de los flujos de trabajo típicos se indican para cada uno de estos tipos de alimentos.
Protocolo
Los productos alimenticios que se consumen crudos o ligeramente cocidos son de mayor preocupación para la salud humana. Los ejemplos incluyen ensalada de verduras, frutas, mariscos y crustáceos.
1. Recogida de muestras
- Recoger las muestras en condiciones que reduzcan al mínimo la contaminación, y se almacena en sacos limpios, separados durante el transporte. Una vez recogidos, las muestras deben almacenarse a 4 ° C y procesadas dentro de 24 horas.
2. Preparación Enriquecido Cultura
- Frutas y vegetales:
- Poner unos 10 g de material en una bolsa de plástico resistente. Si el procesamiento de material más grande, se concentran en la piel de la fruta o verdura. Añadir 90 ml 0,1 M tampón fosfato de sodio, pH 7,0 y el proceso en un mezclador de paletas durante 30 seg. Alternativamente, el material se puede agitar manualmente si un mezclador de paleta no está disponible.
- Recoger el líquido en dos 50 ml tubos Falcon. Tubos centrífuga a 4.000 xg durante 7 minutos a las bacterias de pellets.
- Retirar con cuidado el sobrenadante y suspender las bacterias sedimentadas en 30 ml de caldo estéril Luria Bertani (LB).
- Incubar en un agitador O / N, la celebración de un tubo a 25 ° C y el otro a 37 ° C. Los cultivos deben agitaron a 200 oscilaciones por minuto (OPM). Culturas Almacenar a 4 ° C hasta su utilización.
- Mariscos:
- Diseccionar el estómago (ostras) o del tracto digestivo (gambas) con unas pinzas y pinzas estériles y colocarlos en un tubo de 1,5 ml estéril que contiene 200 l de tampón de fosfato de sodio 0,1 M, pH 7,0. Macerar la muestra para crear un homogeneizado.
- Dispense 5 ml de caldo LB en dos tubos de 5 ml estériles, e inocular cada una con el homogeneizado de mariscos.
- Incubar en un agitador O / N, la celebración de un tubo a 25 ° C y el otro 37 ° C. Los cultivos deben agitaron a 200 opm. Culturas Almacenar a 4 ° C hasta su utilización.
3. Los cultivos de cribado para integrones
NOTA: protocolos de PCR estándar se utilizan a lo largo de esta metodología, utilizando tampones suministrados con la enzima, y una concentración final de MgCl2 de 2,5 mM. Si es necesario, Lorenz (2011) 24 ha descrito métodos de optimización y solución de problemas de la PCR en un número anterior de esta revista.
- Preparación de ADN Hervido:
- Prescindir de 100 l de cultivo enriquecido en un 0,5 ml tubo de PCR estériles. Calentar la muestra a 99 ° C durante 10 min, utilizando un baño de agua o bloque de calor. Encajar frialdad en hielo durante 2 min.
- Microcentrífuga a 14.000 xg durante 5 min para sedimentar los restos celulares. Regresar a hielo. Las muestras de ADN pueden ser congeladas a -20 ° C hasta que sea necesario, pero deben ser descongeladas en hielo.
- La detección de la clase 1 integrones utilizando PCR:
NOTA: Los cultivos mixtos de la / N incubación O son examinados para detectar la clase 1 integrones utilizando el protocolo HS463a / HS464 PCR (Tabla 1).- Descongele todos los reactivos de PCR en hielo, lejos de cualquier sou potencialRCE de contaminación de ADN. Conforman 48 l PCR Mastermix por muestra, incluidos los controles negativos (Tabla 2). Dispensar 48 l de mezcla maestra de PCR a cada tubo, usando puntas de barrera.
- Añadir 2 l de ADN descongelado para tubos de PCR individuales utilizando puntas de barrera. Ejecutar programa apropiado en el termociclador de PCR.
- Para evaluar los resultados de la amplificación, a electroforesis 7 l de producto de PCR en un gel de agarosa al 2% se vertió y se ejecutan en tampón TBE (90 mM TRIS-borato, EDTA 2 mM). Incluir un marcador de peso molecular, tal como una escalera de 100 pb.
- Publicar mancha el gel con una mancha de ADN, y visualizar utilizando un transiluminador UV. Compruebe que no ha habido ninguna amplificación en el control negativo. Un fuerte banda en torno a 470 pb para cualquier muestra particular indica que el cultivo mixto contiene bacterias que llevan un integrón de clase 1. Colonias puras ahora se aíslan de cualquier cultura mixta que regresó una PCR positiva.
4. Proyección del Individual colonias de integrones de clase 1
- Las diluciones en serie y placas de cálculo:
NOTA: Para aislar colonias individuales a partir de los cultivos mixtos PCR-positivos, el cultivo mixto se diluyó en serie 10 veces en tampón de fosfato de sodio, y luego plateado para recuperar colonias individuales.- Añadir 1 ml de cultivo mixto a 9 ml de tampón de fosfato de sodio 0,1 M, pH 7,0 y mezclar por inversión. Añadir 1 ml de la dilución a una más 9 ml de tampón de fosfato, y repetir hasta una dilución en serie de 10 -8 se alcanza.
- Corre 100 l de los 10 -4 a 10 -8 diluciones en agar LB, por duplicado. Incubar las placas de O / N a la misma temperatura usada para el cultivo mixto inicial. Las placas pueden entonces ser almacenados a 4 ° C hasta que sea necesario, sin embargo es mejor para procesar las colonias individuales bastante rápidamente.
- Selección colonia individual y preparación de ADN hervida:
- Escoja colonias individuales de las placas de dilución de serie, sínando tantos tipos de colonias diferentes como sea posible, utilizando criterios como tamaño de la colonia, forma y color. Use palillos de dientes estériles para seleccionar colonias individuales de la placa de expansión.
- Toque el palillo de dientes a la colonia, y luego transferir a un tubo de PCR que contiene 100 l de agua estéril. Si un gran número de colonias son de ser controlados, a continuación, las colonias se pueden preparar en una bandeja de microtitulación. Girar el palillo de dientes entre los dedos para desalojar algunas de las células en el agua.
- Utilizando el mismo palillo de dientes, inocular una placa de LB con una raya de la colonia. Un gran número de colonias se pueden almacenar en placas marcadas si cada racha es de aproximadamente 1 cm de largo. Repita los pasos 1 a 3 hasta que haya seleccionado un número adecuado de aislamientos.
- Incubar el LB placas O / N, a la misma temperatura que la utilizada para el cultivo enriquecido inicial. Las culturas pueden entonces ser almacenados a 4 ° C hasta su utilización.
- Para la preparación de ADN a partir de las suspensiones bacterianas, siga los pasos como outlined en el apartado 3.1. Para la selección de los lisados de cultivos puros, realice el HS463a / HS464 PCR como se indica en el apartado 3.2. Aislamientos que volver una PCR positiva será utilizada para la preparación de ADN genómico (artículo 5), y para otros análisis.
5. Las colonias individuales de ADN genómico La extracción de integrón positivos Uso Bead Vencer a 25
- Se inoculan 5 ml de caldo LB con una muestra del cultivo puro aislado en el apartado 4.2. Incubar O / N, agitando a la misma temperatura que se utilizó para el cultivo enriquecido inicial.
- Células de pellets a 4.000 xg durante 7 minutos, a continuación, eliminar el sobrenadante. Resuspender las células sedimentadas en 1 ml de solución CLS-TC en un tubo de preparación rápida lisis Matrix E. El uso de una máquina de golpes de talón, procesar la muestra a 5,5 m / s durante 30 s.
- Tubo de centrífuga a 14.000 xg durante 5 min y se recuperan 700 l de sobrenadante en un tubo de 1,5 ml estéril.
- Añadir 700 l de matriz de unión diluido 1: 5 con 6 M guanidinium tiocianato y mezclar a temperatura ambiente durante 5 min. Centrifugar a 14.000 xg durante 1 min, el sobrenadante de descarte.
- Añadir 800 l de etanol de lavado de sal (70% de etanol, acetato de sodio 0,1 M) y agitar hasta que el sedimento se resuspendió completamente. Lavar durante 5 min a RT, se centrifuga a 14.000 xg durante 1 min y desechar el sobrenadante. Asegúrese de que todos sobrenadante se ha eliminado, utilizando una micropipeta si es necesario, a continuación, dejar secar al aire durante 5 minutos.
- Resuspender el pellet matriz en 200 l de TE (Tris-HCl 10, 1 mM EDTA, pH 7,6) pipeteando arriba y abajo. Incubar a temperatura ambiente durante 3 min. Centrifugar a 14.000 xg durante 3 min y la transferencia de 160 l de la ADN eluido en un tubo estéril. Ejecutar una alícuota de la extracción de ADN genómico en un / gel de agarosa al 2% w v para comprobar el rendimiento y la integridad de la extracción de ADN. El ADN genómico purificado se almacena a -20 ° C y descongelado en hielo cuando sea necesario para las pruebas de PCR.
6. PCR de diagnóstico y secuenciación de ADN
NOTA: LaADN genómico preparado en la sección 5 se utiliza para todas las PCR de diagnóstico, y para confirmar el resultado positivo de la clase 1 integrones.
- Descongelar el ADN genómico en hielo, vórtice brevemente la muestra de ADN y el giro pulso en 14.000 xg durante 20 seg. Repita la PCR para el gen de la clase 1-integrón integrasa usando HS463a / HS464 (Tabla 1), lo que confirma que el aislado es positivo para la amplificación de 471 pb (Sección 3.2).
- Identificación de cultivos positivos a nivel de especie se logra mediante el análisis de la pequeña subunidad del gen rRNA (16S ADNr). Amplificar el gen 16S usando los cebadores F27 / R1492 (Tabla 1). Compruebe los productos de PCR utilizando electroforesis. Todos los objetivos bacterianas deben generar un amplicón 16S de aproximadamente 1.450 pb.
- Secuencia del gen amplicón ARNr 16S para determinar identidad de la especie, pero como no es probable que sean múltiples PCR positivos desde el paso 6.2, y muchos de estos positivos serán las mismas especies bacterianas, nosotros primero distinguir las diferentes especies using digestión de restricción del producto de PCR 16S.
- Digerir una alícuota de la PCR 16S con la enzima de restricción Hinf 1. Cualquier enzima que reconoce los sitios de escisión 4 pb harán, pero Hinf 1 genera buena diversidad de 16S objetivos, y es una enzima barata fiable.
- Configure una restricción 30 l digerir mezcla maestra (Tabla 2) en un tubo estéril y añadir 20 l de producto de PCR 16S un-una vez purificada. Incubar en un baño de agua 37 ° C O / N. Revise el resumen en un gel de agarosa al 2%.
- Puntuación de los patrones de digestión producidos por Hinf 1. Los aislados con el mismo perfil de restricción 16S es probable que sean la misma especie. En vez de secuenciar cada producto PCR 16S, secuencia en la mayoría de los tres representantes de cada patrón de restricción Hinf 1.
- Purificar los productos de PCR 16S utilizando un kit comercial. Un kit de exonucleasa de cadena sencilla se puede utilizar, pero basa columna o métodos de precipitación también funciona bien para eliminar uninccebadores orporated y los ADN de cadena sencilla. Secuencia de los amplicones 16S utilizando r910 imprimación (Tabla 1), la creación de las reacciones según lo especificado por la instalación de secuenciación.
- Utilice la secuencia de ADN resultante para interrogar a la base de datos de ADN del NCBI con la función blastn ( http://blast.ncbi.nlm.nih.gov/Blast.cgi ). Más del 97% de identidad de nucleótidos se suele tomar como identidad de la especie.
7. Mapeo y caracterización de integrones y cassettes Arrays
NOTA: Clase 1 integrones que emanan de los ecosistemas dominados por el hombre es probable que sean los integrones más comunes en todas sus muestras. Estos integrones todos tienen un solo origen reciente, por lo que tienen una secuencia de ADN altamente conservadas 5.
- Diferenciar entre una clase 1 integrones que emanan de fuentes humanas y los que se encuentran naturalmente en muestras ambientales mediante la realización de unPCR con el cebador establece intI1F165 / intI1R476 10 (Tabla 1).
NOTA: Clase 1 integrones de bacterias comensales clínicos o generarán un producto ~ 300 pb, mientras que la clase del medio ambiente 1 integrones no generarán ningún producto. - Caracterizar los integrones clínicos mediante la amplificación de la matriz de cassette con el juego de primers HS458 / HS459. Esta PCR se amplificar la región entre intI1 y el segmento conservado el 3 'en el extremo de la mayoría de casete arrays. Debido a que el número y la identidad de cassettes es variable, el tamaño del producto de PCR también variará.
- Purificar amplicones y secuenciar el ADN utilizando cada uno de los cebadores de amplificación. Utilice secuencias para interrogar las bases de datos de ADN utilizando la función blastn ( http://blast.ncbi.nlm.nih.gov/Blast.cgi ).
- Asegúrese de nombrar de casetes siga nomenclatura estandarizada 9, ya que muchas deposiciones de datos para casete génico integróns usan nombres inconsistentes o ambiguas. Preste especial atención a los nuevos casetes de genes, ya que estos pueden codificar nuevos fenotipos, incluyendo genes que confieren un aumento transmisibilidad, patogenicidad o virulencia 8,21.
Todavía circulan preclínicos sobre las formas de la clase 1 integrón en ambientes naturales: NOTA. En particular, aquellos que están vinculados a activos Tn 402 transposones son de mayor interés 26. Las matrices de cassette de estos integrones no amplificarán con el juego de primers HS458 / HS459, pero pueden ser amplificado con la MRG284 / MRG285 imprimación conjunto (Tabla 1), la generación de amplicones de dependiente en el contenido de cassette de tamaño variable. Productos de PCR deben ser purificadas y ADN secuenciado como se indica en 7.2.
Resultados
Proyección de cultivos mixtos y aislamientos bacterianos para intI1
Conjunto de cebadores HS463a / HS464 PCR se puede utilizar para detectar la presencia del gen de la integrasa integrón de clase 1, intI1 (Figura 1). Este conjunto de cebadores funciona bien para detectar intI1 en cultivos mixtos, y también se utiliza para detectar colonias de bacterias recogidas de placas separadas (Figura 2). Aislamientos positivos deberían generar una sola banda fuerte a 471 pb utilizando este conjunto de cebadores (Figura 3A). La mayoría de los aislamientos positivos llevará intI1 que tiene su origen los seres humanos o los animales agrícolas y domésticos. Estas cepas también serán positivos en una PCR utilizando cebadores F165 / R476, que debe generar un amplicón de 311 pb (Figura 1). Fuentes ambientales de intI1 generalmente no serán positivos en este segundo ensayo.
Caracterización de cassette arrays integrón
ADN genómico de cepas puras se utiliza para la caracterización de cassette arrays integrón. Las matrices de cassette de 402 Tn clase -Associated 1 integrones pueden amplificarse usando cebadores HS458 / HS459. Estos cebadores se dirigen, respectivamente, el sitio de recombinación integron, y el extremo 3 'de la matriz de casete, que normalmente termina en la qacEΔ sul1 gen de fusión (Figura 1). El tamaño de los productos PCR generados en este ensayo varía de acuerdo con el número y la identidad de cassettes en la matriz (Figura 3B). Clase ambiental 1 integrones son a menudo incorporados en los cromosomas bacterianos, y sus casete arrays pueden ser amplificados por cebadores que se dirigen a los extremos proximal y la mayoría de los sitios de recombinación distales (Figura 1). Primers MRG284 / 285 están diseñadas para amplificar esta región, y de nuevo, porque el contenido de casete varía, el tamaño de los amplicones también varía (Figura 3C). La secuenciación de HS458 / 459 PCRproductos normalmente se recuperan determinantes de resistencia a los antibióticos conocidos, mientras que la secuenciación MRG284 / 285 productos de PCR generalmente recuperar casetes de genes que codifican polipéptidos de función desconocida.
La identificación de especies bacterianas
Las bacterias se identificaron utilizando 16S rDNA de secuenciación de bases de datos y las comparaciones. El ADN genómico se usa como una plantilla para la amplificación de los genes 16S rRNA subunidad pequeña. Usando los cebadores sugirieron, esto debería generar un amplicón de aproximadamente 1450 pb. Debido a que un gran número de colonias pueden ser examinados en un momento dado, se emplea un método de cribado jerárquica. Productos 16S amplificadas por PCR se digirieron con la enzima de restricción Hinf 1, y estas digestiones se separaron en geles de agarosa. Las especies individuales generarán patrones distintivos después de digestión, de modo que los aislamientos de la misma especie pueden ser fácilmente identificados (Figura 3D). La secuenciación de los productos de PCR del gen 16S rRNA de un máximo de tresaislamientos que representa patrón de cualquier restricción permite la identificación eficiente de todas las especies susceptibles en una colección de integrón aislamientos positivos.
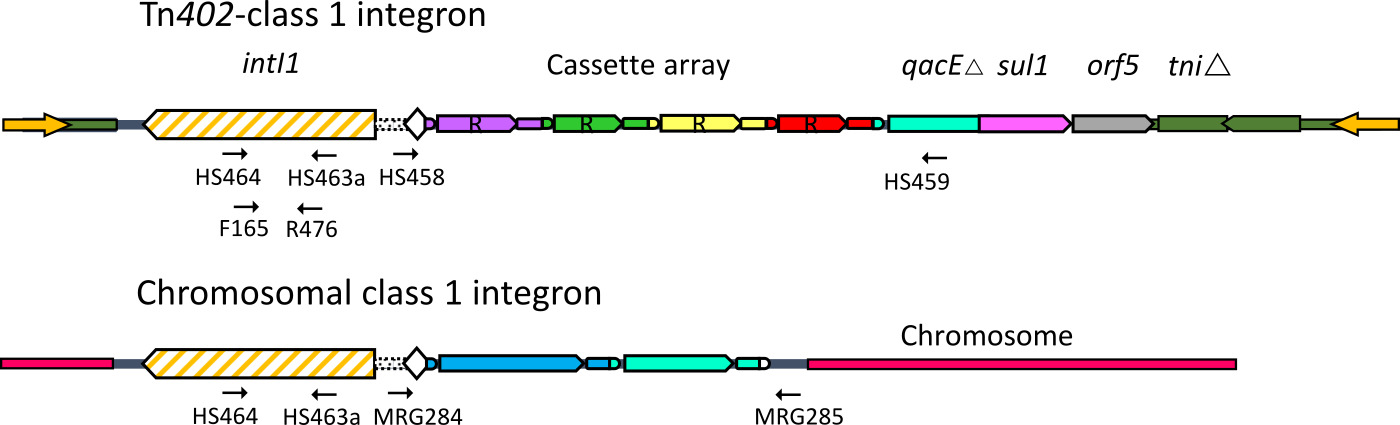
Figura 1. Estructura de la clase 1 integrones. Diagramas esquemáticos de clase clínica y ambiental 1 integrones, mostrando PCR primer sitios de unión se hace referencia en el manuscrito. 1 integrones clase consisten en un gen integrón-integrasa (intI1) que cataliza la captura y la expresión de casetes de genes para formar una matriz de cassette. Antes del advenimiento del uso de antibióticos, la mayoría de la clase 1 integrones eran cromosómica, y llevó a los casetes de genes cuyas funciones están aún por determinar. Selección fuerte a través del uso de antibióticos ha aumentado enormemente la abundancia de una variante de la secuencia de la clase 1 integrón asociado con el transposón Tn 402.Estos integrones 'clínicas' han adquirido matrices de casetes que codifican la resistencia a antibióticos. Son estos integrones que actualmente están contaminando los ambientes naturales y la cadena de producción de alimentos. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 2. Diagrama de flujo esquemático para la detección de la clase 1 integrones en los productos alimenticios. Las muestras de los productos alimenticios se utilizaron para inocular medios de comunicación y generar un cultivo bacteriano mixto. Estos cultivos mixtos son examinados para detectar la presencia de integrones de clase 1 y cultivos positivos utilizados para preparar placas separadas. Las colonias individuales de las placas de propagación son reevaluados para integrones. Los cultivos positivos se purifican, ADN extraído y caracterizado para el contenido de cassette por el ADN sesecuenciación. La secuenciación del gen 16S rRNA se utiliza para la identificación de especies. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Los análisis electroforético Representante del proceso de selección. (A) Screening colonias individuales utilizando intI1 cebadores HS463a / 464. Las colonias positivas generan una sola banda fuerte a 471 pb. (B) La amplificación de cassette arrays de 402 Tn integrones utilizando cebadores HS458 / 459. Esta PCR genera tamaños de los productos variable dependiente sobre la identidad y tamaño de los cassettes de componentes de la matriz. La identificación de estos cassettes requiere la secuenciación del ADN. (C) La amplificación de cassette arrays de la clase ambiental 1 integrons utilizando cebadores MRG284 / 285. Esta PCR también genera tamaños de los productos variable dependiente sobre la identidad y tamaño de los cassettes de componentes de la matriz, sin embargo en casetes son poco probable para codificar resistencia a los antibióticos matrices ambientales. (D) La selección de productos de PCR 16S por digestión con Hinf 1. Los aislamientos con patrones de restricción idénticos es probable que sean la misma especie. Si se recuperan grandes cantidades de un solo tipo de restricción, sólo unos pocos de ellos necesitan ser secuenciado. Por favor, haga clic aquí para ver una versión más grande de esta figura.
| PCR | Gen diana | Primer nombre | Dirección | Secuencia 5 '- 3' | Ciclismo condiciones | Reference |
| HS463a / HS464 | Integrón de clase 1 | HS464 | Adelante | ACATGCGTGTAAATCATCGTCG | 94 ° C 3 min; 35 ciclos de 94 ° C 30 seg, 60 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 min | Stokes et al., 2006 28 |
| HS463a | Reverse | CTGGATTTCGATCACGGCACG | ||||
| f27 / R1492 | 16S rRNA | f27 | Adelante | AGAGTTTGATCMTGGCTCAG | 94 ° C 3 min; 35 ciclos de 94 ° C 30 seg, 60 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 min | Lane, 1991 29 |
| R1492 | Reverse | TACGGYTACCTTGTTACGACTT | ||||
| intI1F165 / IntI1R476 | Clase Clínica 1 integrón | intI1F165 | Adelante | CGAACGAGTGGCGGAGGGTG | 94 ° C 3 min; 35 ciclos de 94 ° C 30 seg, 55 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 min | Gillings, 2014 10 |
| intI1R476 | Reverse | TACCCGAGAGCTTGGCACCCA | ||||
| HS549 / HS550 | 3'CS Clínica integrón | HS549 | Adelante | ACTAAGCTTGCCCCTTCCGC | 94 ° C 3 min; 35 ciclos de 94 ° C 30 seg, 65 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 min | Stokes et al., 2006 28 |
| HS550 | Reverse | CTAGGCATGATCTAACCCTCGG | ||||
| HS458 / HS459 | Cassette gama Clínico | HS458 | Adelante | GCAAAAAGGCAGCAATTATGAGCC | 946; C 3 min; 35 ciclos de 94 ° C 30 seg, 55 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 min | Holmes et al., 2003 30 |
| HS459 | Reverse | GTTTGATGTTATGGAGCAGCAACG | ||||
| MRG284 / MRG285 | Casete Ambiental | MRG284 | Adelante | GTTACGCCGTGGGTCGATG | 94 ° C 3 min; 35 ciclos de 94 ° C 30 seg, 55 ° C 30 seg, 72 ° C 1 min 30 seg; 72 ° C 5 min | Gillings et al., 2009 21 |
| MRG285 | Reverse | CCAGAGCAGCCGTAGAGC |
Tabla 1. Primer secuencias y condiciones de PCR utilizados para la identificación de la clase 1 integrones y sus cassettes genéticos asociados.
| Reactivos de PCR | Volumen (l) | Restricción reactivos digerir | Volumen (l) |
| Agua estéril | 21.5 | Agua estéril | 23 |
| 10x GoTaq Blanca | 25 | Tampón B | 5 |
| RNaseA [1 mg / ml] | 0.5 | Albúmina de suero bovino [1 mg / ml] | 1 |
| Cebador [50 mM] | 0.5 | HinfI [10 unidades / l] | 1 |
| Cebador inverso [50 mM] | 0.5 | ||
| Volumen Mastermix | 48 | Volumen Mastermix | 30 |
| Molde de ADN | 2 | Plantilla de PCR | 20 |
| El volumen final de PCR | 50 | Restricción final volumen digest | 50 |
Tabla 2. PCR y restricción de digerir Mastermix constituyentes.
Discusión
La identificación de los integrones y sus cassettes genéticos asociados es potencialmente un paso clave en la predicción de la aparición de nuevos agentes patógenos oportunistas, el seguimiento de las vías para los patógenos en la cadena alimentaria humana, y la identificación de una nueva resistencia y virulencia determinantes 8,21,26. El objetivo de este trabajo fue describir un enfoque simplificado para el cribado de muestras para la clase 1 integrones, caracterizando sus matrices de cassette y la identificación de las especies bacterianas en las que residen. Los pasos críticos en el protocolo implican buenas prácticas microbiológicas, y la prevención de la contaminación de PCR que generarían falsos positivos.
El protocolo descrito aquí se puede modificar fácilmente para detectar otros integrones clínicamente relevantes, incluyendo los de clase 2 y clase 3 integrones que también se encuentran en los patógenos humanos. También puede ser modificado para detectar integrones en comunidades microbianas de agua, biofilms, suelo o sedimento. Hay algunos limitations a esta técnica, que surgen de una dependencia de cultivo de células bacterianas. Muchas bacterias ambientales no son fácilmente cultivables, y el protocolo descrito aquí no detectar estas especies. La gama de especies que se recuperan podría ampliarse mediante el uso de diferentes formulaciones de medios bacteriana y los tiempos de incubación más largos. Sin embargo, la mayoría de las especies de interés para la salud humana es probable que crezca en las condiciones descritas aquí.
Este protocolo tiene algunas ventajas sobre las técnicas que utilizan enchapado en medios selectivos. No hay supuestos necesitan ser hechas sobre la identidad de los determinantes de resistencia realizadas por los aislados individuales, y los nuevos genes de resistencia se puede recuperar y caracterizado. En términos más generales, el flujo de trabajo también puede ser adaptado para detectar cualquier elemento del mobilome resistome 27 o que podrían ser de interés 4. Dichos ensayos son importantes para la comprensión de la dinámica de los diversos elementos de ADN involucradas en unresistencia antibiótica, y crítica para la conservación de la armería de disminución de compuestos antimicrobianos.
Divulgaciones
Los autores no tienen nada relevante a revelar.
Agradecimientos
Gracias a Michaela Hall, Larissa Bispo y Gustavo Tavares para la asistencia técnica.
Materiales
| Name | Company | Catalog Number | Comments |
| GoTaq Colourless Mastermix | Promega | M7132 | Used in all PCRs |
| RNAse (Ribonuclease A from bovine pancreas) | Sigma | R6513-10MG | Used in all PCRs |
| HinFI restriction enzyme | Promega | R6201 | Used to digest 16S rDNA PCR poducts. Enzyme comes with optimal buffer and BSA |
| 100 bp ladder | GE Healthcare | 27400701 | Used as a size standard on all agarose gels |
| GelRed DNA stain | Biotium | 41003 | CAUTION: Personal protection must be worn when handling this material |
| Guanidinium thiocyanate | Life Technologies | AM9422 | CAUTION: Personal protection must be worn when handling this material |
| CLS-TC Solution | MP Biomedicals | 6540409 | Resuspension solution used at the begining of the genomic DNA extraction |
| Lysing Matrix E FastPrep tubes | MP Biomedicals | 116914500 | Tube required for mechanical disruption of bacterial cell walls. This code is used for packs of 500 tubes, smaller quantities are available. |
| Binding matrix | MP Biomedicals | 116540408 | Diluted 1:5 with 6 M guanidinium thiocyanate and used in the genomic DNA extraction method. |
| Fast Prep machine | MP Biomedicals | Number of options available | MP Biomedicals has a number of FastPrep machines available to purchase. Visit http://www.mpbio.com for more information |
Referencias
- Bush, K., et al. Tackling antibiotic resistance. Nature Rev. Microbiol. 9, 894-896 (2011).
- Davies, J., Davies, D. Origins and evolution of antibiotic resistance. Microbiol. Mol. Biol. Rev. 74 (3), 417-433 (2010).
- Costa, V. M., et al. Antibiotic resistance is ancient. Nature. 477 (7365), 457-461 (2011).
- Gillings, M. R. Evolutionary consequences of antibiotic use for the resistome, mobilome and microbial pangenome. Front. Microbiol. 4, 4(2013).
- Gillings, M., et al. The evolution of class 1 integrons and the rise of antibiotic resistance. J. Bacteriol. 190 (14), 5095-5100 (2008).
- Brassard, S., Lapointe, J., Roy, P. H. Diversity and relative strength of tandem promoters for the antibiotic-resistance genes of several integrons. Gene. 142 (1), 49-54 (1994).
- Partridge, S. R., et al. Definition of the attI1 site of class 1 integrons. Microbiol. 146 (11), 2855-2864 (2000).
- Gillings, M. R. Integrons: Past, Present, and Future. Microbiol. Mol. Biol. Rev. 78 (2), 257-277 (2014).
- Partridge, S. R., Tsafnat, G., Coiera, E., Iredell, J. R. Gene cassettes and cassette arrays in mobile resistance integrons. FEMS Microbiol. Rev. 33 (4), 757-784 (2009).
- Gillings, M. R., et al. Using the class 1 integron-integrase gene as a proxy for anthropogenic pollution. ISME J. In press, (2014).
- Gaze, W. H., et al. Impacts of anthropogenic activity on the ecology of class 1 integrons and integron-associated genes in the environment. ISME J. 5, 1253-1261 (2011).
- Gerzova, L., et al. Characterization of microbiota composition and presence of selected antibiotic resistance genes in carriage water of ornamental fish. PLoS ONE. 9, e103865(2014).
- Power, M., Emery, S., Gillings, M. Into the wild: dissemination of antibiotic resistance determinants via a species recovery program. PLoS ONE. 8, e63017(2013).
- Stokes, H. W., Gillings, M. R. Gene flow, mobile genetic elements and the recruitment of antibiotic resistance genes into Gram-negative pathogens. FEMS Microbiol. Rev. 35 (5), 790-819 (2011).
- Moura, A., Oliveira, C., Henriques, I., Smalla, K., Correia, A. Broad diversity of conjugative plasmids in integron-carrying bacteria from wastewater environments. FEMS Microbiol. Lett. 330 (2), 157-164 (2012).
- Schlüter, A., Krause, L., Szczepanowski, R., Goesmann, A., Pühler, A. Genetic diversity and composition of a plasmid metagenome from a wastewater treatment plant. J. Biotech. 136 (1-2), 65-76 (2008).
- Taylor, N. G. H., Verner-Jeffreys, D. W., Baker-Austin, C. Aquatic systems: maintaining, mixing and mobilising antimicrobial resistance. TREE. 26 (6), 278-284 (2011).
- Stalder, T., et al. Quantitative and qualitative impact of hospital effluent on dissemination of the integron pool. ISME J. 8, 768-777 (2014).
- Allen, H. K., et al. Call of the wild: antibiotic resistance genes in natural environments. Nature Rev. Microbiol. 8, 251-259 (2010).
- Wellington, E. M., et al. The role of the natural environment in the emergence of antibiotic resistance in Gram-negative bacteria. Lancet Infect. Dis. 13 (2), 155-165 (2013).
- Gillings, M. R., et al. Mobilization of a Tn402-like class 1 integron with a novel cassette array via flanking miniature inverted-repeat transposable element-like structures. Appl. Env. Microbiol. 75 (18), 6002-6004 (2009).
- Graham, D. W., Collignon, P., Davies, J., Larsson, D. J., Snape, J. Underappreciated role of regionally poor water quality on globally increasing antibiotic resistance. Env. Sci. Technol. 48 (20), 11746-11747 (2014).
- Perry, J. A., Westman, E. L., Wright, G. D. The antibiotic resistome: what's new. Curr. Opinion Microbiol. 21, 45-50 (2014).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. 63, e3998-e3998 (2011).
- Gillings, M. R. Rapid Extraction of PCR-Competent DNA from Recalcitrant Environmental Samples. Env. Microbiol. 1096, 17-23 (2014).
- Sajjad, A., Holley, M. P., Labbate, M., Stokes, H., Gillings, M. R. Preclinical class 1 integron with a complete Tn402-like transposition module. Appl. Env. Microbiol. 77 (1), 335-337 (2011).
- Wright, G. D. Antibiotic resistance in the environment: A link to the clinic. Curr. Opinion Microbiol. 13 (5), 589-594 (2010).
- Stokes, H., Nesbo, C., Holley, M., Bahl, M., Gillings, M., Boucher, Y. Class 1 integrons predating the association with Tn402.-like transposition genes are present in a sediment microbial community. Journal of Bacteriology. 188, 5722-5730 (2006).
- Lane, D. J. Nucleic Acid Techniques in Bacterial Systematics. , John Wiley &Sons, Inc. 115-175 (1991).
- Holmes, A. J., et al. Recombination activity of a distinctive integron-gene cassette system associated with stutzeri. populations in soil. Journal of Bacteriology. 185, 918-928 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados