Method Article
Transfection, seleção e Colony-picking de induzidas pelo homem pluripotentes Células-Tronco TALEN-alvo com um gene GFP no AAVS1 Safe Harbor
Neste Artigo
Resumo
TALEN-mediated gene editing at the safe harbor AAVS1 locus enables high-efficiency transgene addition in human iPSCs. This protocol describes the procedures for preparing iPSCs for TALEN and donor vector delivery, transfecting iPSCs, and selecting and isolating iPSC clones to achieve targeted integration of a GFP gene to generate reporter lines.
Resumo
Targeted transgene addition can provide persistent gene expression while circumventing the gene silencing and insertional mutagenesis caused by viral vector mediated random integration. This protocol describes a universal and efficient transgene targeted addition platform in human iPSCs based on utilization of validated open-source TALENs and a gene-trap-like donor to deliver transgenes into a safe harbor locus. Importantly, effective gene editing is rate-limited by the delivery efficiency of gene editing vectors. Therefore, this protocol first focuses on preparation of iPSCs for transfection to achieve high nuclear delivery efficiency. When iPSCs are dissociated into single cells using a gentle-cell dissociation reagent and transfected using an optimized program, >50% cells can be induced to take up the large gene editing vectors. Because the AAVS1 locus is located in the intron of an active gene (PPP1R12C), a splicing acceptor (SA)-linked puromycin resistant gene (PAC) was used to select targeted iPSCs while excluding random integration-only and untransfected cells. This strategy greatly increases the chance of obtaining targeted clones, and can be used in other active gene targeting experiments as well. Two weeks after puromycin selection at the dose adjusted for the specific iPSC line, clones are ready to be picked by manual dissection of large, isolated colonies into smaller pieces that are transferred to fresh medium in a smaller well for further expansion and genetic and functional screening. One can follow this protocol to readily obtain multiple GFP reporter iPSC lines that are useful for in vivo and in vitro imaging and cell isolation.
Introdução
A capacidade de reprogramar células somáticas humanas em células-tronco pluripotentes induzidas semelhantes a células-tronco embrionárias (iPSCs) foi descoberto pela primeira vez por Takahashi et al., Em 2007 1. Fibroblastos dérmicos humanos transduzidas com retrovírus expressando quatro fatores de transcrição (The Yamanaka assim apelidado fatores OCT3 / 4, Sox2, foram mostradas c-Myc, e KLF4) ser altamente semelhante às células estaminais embrionárias humanas (hESCs) com base na morfologia, a proliferação, a expressão do gene, e estado epigenética; crucialmente, iPSCs também são capazes de se diferenciarem em células de todas as três camadas germinais 1. A capacidade potencial e diferenciação proliferativa das iPSCs torna ferramentas muito atraentes; reprogramando as células de pacientes que sofrem de doenças específicas, iPSCs pode ser utilizado tanto como em sistemas modelo in vitro da doença e como potenciais agentes terapêuticos.
Para este último efeito, várias questões devem ser abordadas antes de todo o potencial das iPSCsem um ambiente clínico pode ser realizado; o potencial tumorigénico de hESCs cultivadas in vitro e iPSCs, o uso de derivados de xenogénicas durante reprogramação e manutenção de células, e a necessidade de rastrear células transplantadas in vivo de todos os obstáculos cruciais para a aplicação clínica de células estaminais pluripotentes (Avaliado por Hentze et al. 2). Uma solução ideal para a necessidade de rastrear células diferenciadas pós-transplante envolveria um marcador visualmente detectável que resiste ao silenciamento e variegação, independentemente da aplicação. Expressão robusta e sustentada de transgenes integrados é o mais prontamente possível quando DNA exógeno é introduzido no porto seguro loci; isto é, locais genómicos que permitem a transcrição de um vector suficiente integrado, enquanto ao mesmo tempo atenuar as perturbações de expressão de genes vizinhos 3. Um tal local que tem sido muito bem caracterizada, desde a sua descoberta, é a integr vírus adeno-associadoção local 1 (AAVS1), no primeiro intrão 1 do regulador 12C subunidade (PPP1R12C) do gene da proteína fosfatase. Este local foi mostrado não só para permitir sustentada e expressão robusta de transgenes integrados através do tempo estendido na cultura e na diferenciação vitro 3, mas também para proteger circundante genes de perturbação da transcrição 4; ambas as características são provavelmente devido à presença de elementos isoladores de cromatina endógenas que flanqueiam o local AAVS1 5.
Os avanços na engenharia ferramentas genoma durante apenas a última década, têm muito facilitada a facilidade e eficiência com que as manipulações genéticas em qualquer tipo de célula pode ser conseguido. Embora experiências bem sucedidas iniciais invocado extremamente baixos níveis de recombinação homóloga endógena (RH) com um doador introduzidas para se abordagem selectiva de genes em 6,7 CES, o uso de nucleases específicas do local, tais como as nucleases de dedos de zinco (ZFNs), que signivamente induzir a recombinação homóloga através da geração de uma quebra de cadeia dupla de DNA tem aumentado consideravelmente a eficiência de tais experiências 8,9. O reaproveitamento de ambos os ativadores de transcrição-como efetoras (contos) da planta patogênica xanthomonas gêneros e os procariotas cluster regularmente interspaced repete palindrômicas curtas (CRISPR) system / Cas9 em eficientes nucleases grife específicas do local fez gene alvo em células-tronco pluripotentes uma acessível e metodologia praticável 10-13.
Um artigo recente descreveu um método eficiente para a integração estável de uma cassete repórter fluorescente no AAVS1 porto seguro locus iPSCs humanos utilizando nucleases Tale (TALENS) 14. Essas iPSCs direcionados mantiveram sua fluorescência mesmo após a diferenciação direcionado para cardiomiócitos e transplante em um modelo de rato de infarto do miocárdio (MI), proporcionando uma forte evidência para a utilidade de talestavelmente células estaminais pluripotentes fluorescente 14. Para obter colónias específicas, um método gene armadilha foi utilizado em que um aceitador de splicing (SA), 2A sequência do péptido de auto-clivagem coloca a puromicina N-acetil-transferase gene (PAC) sob o controlo do promotor endógeno PPP1R12C; assim, apenas iPSCs que tenham incorporado o ADN dador no locus AAVS1 expressar PAC, tornando-os selecionáveis baseado em puromicina-resistência; (Figura 1, 15). Este protocolo detalha os procedimentos de geração de AAVS1-GFP iPSCs relatado no recente documento 14, incluindo o processo de transfecção de iPSCs com TALENS e um doador de 9,8 kb para integrar um fragmento de DNA 4.2kb no locus AAVS1 porto seguro, a seleção de iPSCs com base em picking colônias para a expansão clonal �puromicina-resistência, e. As técnicas aqui descritas podem ser aplicadas a diversos experimentos de engenharia genoma.
Protocolo
1. Preparação da Membrana Basal Matrix e Revestimento de Plasticware
- Colocar a membrana basal estoque matriz congelada de -20 ° C em gelo e descongelar durante a noite a 4 ° C.
- Após o descongelamento, pipeta 2 mg alíquotas de matriz da membrana basal em tubos Eppendorf de pré-refrigerados. Armazenar estes a -20 ° C até ser necessário.
- Para preparar placas revestidas com matriz da membrana basal, descongelar uma alíquota sobre o gelo até o último pedaço de gelo nas desaparece tubo eppendorf (geralmente dentro de ~ 2 horas).
- Após a descongelação, adicionar matriz da membrana basal para 12 ml de frio (4 ° C) DMEM / F12 para fazer a solução de revestimento de matriz de membrana basal.
- Adicionar solução de matriz da membrana basal para o recipiente de cultura apropriado. Para uma placa de 6 poços, dispensar 1 ml por poço. Agitar a placa para garantir que a solução de matriz membrana basal cobre completamente cada poço.
- Parafilm selar a membrana basal revestida de matriz placa / prato, e incubar a room temperatura durante 1 hora antes da utilização. Alternativamente, placas revestidas com matriz da membrana basal store / pratos a 4 ° C e usar dentro de 2 semanas de revestimento.
NOTA: Adicionar DMEM adicional / F12 para a membrana basal revestida de matriz placa / recipiente para evitar a secagem. Antes de usar 4 ° C membrana basal armazenado revestido de matriz placa / prato, coloque-o em uma cabine de segurança biológica e deixe-o voltar à temperatura ambiente por pelo menos 30 min. - Aspirar matriz da membrana basal completamente antes da adição de meio e células.
2. Preparação de meio E8
- Prepare E8 meio de cultura por degelo suplemento E8 durante a noite a 4 ° C.
- Retirar 10 ml de E8 meio basal do estoque de 500 ml e descarte.
- Pipeta todo o frasco de 10 ml de suplemento E8 diretamente em 490 ml de E8 meio basal. Do meio E8 completa não quente em um banho de água a 37 ° C, como flutuações de temperatura repetidas pode degradar a bFGF no complemédio te E8.
- Use meio E8 completa dentro de 2 semanas de adição do suplemento.
3. O descongelamento de iPSCs
- Retire um frasco de iPSCs congelados de nitrogênio líquido e colocar no gelo seco.
- Descongelar rapidamente o frasco num banho de água a 37 ° C; agite o frasco em banho-maria até que apenas um pequeno fragmento de restos de gelo.
- Pulverizar o frasco com etanol 70% e transferência para uma cabine de segurança biológica.
- Adicionar 1 ml da temperatura ambiente, gota a gota E8 forma directamente para o frasco.
- Usando uma pipeta de 2 ml, gota a gota, transferir a suspensão de células em 9 ml de meio E8 em um tubo de 15 ml. Agitar o tubo de frequência para garantir que as células e o meio de misturar bem rapidamente.
- Centrifuga-se as células a 200 xg durante 5 min.
- Aspirar o sobrenadante e ressuspender o sedimento de células em um volume apropriado de E8 suplementado com 10 uM Y-27632.
- Adicionar as células a um número apropriado de porão membranpoços e revestido matriz, e coloque em uma temperatura de 37 ° C, 5% de CO2 durante a noite. Recomenda-se para a chapa, pelo menos, 0,2 x 10 6 iPSC por poço de uma placa de 6 poços para permitir a recuperação rápida após a descongelação.
4. Manutenção e Passaging Rotina de iPSCs
- Refresque médio E8 diária.
- Monitorar a morfologia e de confluência de células com um microscópio invertido. iPSCs de alta qualidade crescer em colónias planas com aréolas distintas; colónias individuais possuem uma aparência de "paralelepípedos-like".
- Passagem as células IPSCs quando atingem ~ 70% de confluência.
- Prepara-se uma solução de EDTA passaging adicionando 0,9 g de NaCl e 500 ul de EDTA 0,5 M a 500 ml de DPBS. Misture bem para dissolver de NaCl, e filtra-se sob vácuo para esterilizar. Aquecer-se uma alíquota da solução em uma passagem em 37 ° C num banho de água, antes da passagem em.
- Para passagem, aspirado de meio de cultura gasto e lave as células uma vez com um volume igual de pas quentessolução Saging. Aspirar, e pipeta de EDTA suficiente de solução para revestir as passagens em células (1 ml por poço de uma placa de 6 poços).
- Coloque as células em um microscópio invertido e observar as colônias da IPSC. O aparecimento de buracos dentro de colônias e bordas elevadas devem se manifestar dentro de 2 a 5 min.
- Aspirar cuidadosamente a solução EDTA passaging.
- Utilizando uma pipeta de 10 ml, dispensar 4 ml de meio E8 (se usar uma placa de 6 poços), sob alta pressão directamente em cada cavidade a ser passadas.
- Recolher os aglomerados IPSC, e dividida em um número adequado de poços, dependendo da razão de divisão de 1: 8 a 1:12. Não excesso de pipeta, como a desagregação de aglomerados de células resultará na viabilidade pobres.
- Coloque a placa em uma incubadora, e balance a placa de volta-e-vem e lado-a-lado várias vezes para dispersar as células.
5. Preparação de MEFs e iPSCs para Tansfection
- 48 horas antes da transfection, iPSCs passagem em ~ 1: 6 em quatro ou mais poços de uma membrana basal revestida de matriz placa de 6 poços, de modo que será de 70% confluentes, portanto, dois dias.
- No dia seguinte, descongelar DR4 MEFs em meio MEF consistindo de DMEM (alto teor de glucose) suplementado com 10% de FBS e 1x MEM-NEAA.
- Placa DR4 MEFs em duas placas de 10 cm a 2 x 10 ~ 4 células / cm2 e incubar durante a noite a 37 ° C.
- No dia da transfecção, alterar forma MEF a E8 suplementado com 10 uM Y-27632 30 min antes de efectuar a transfecção em iPSCs.
- Opcional: Se a análise de citometria de fluxo de iPSCs pós-transfecção é desejado, remover uma placa revestida com matriz de membrana basal a partir de 4 ° C e coloca à temperatura ambiente.
- Opcional: 4 horas antes da transfecção, complementar pré-cultura iPSC transfecção com Y-27632 na concentração final de 10 uM.
6. delicado de células dissociação Reagente um tratamentond transfecção de iPSCs usando um sistema de electroporação
- Remoção da solução primária P3 transfecção de células a partir de 4 ° C e deixar que esta atinja a temperatura ambiente durante ~ 30 min. Adicionar todo o suplemento de 100 ul para a solução de transfecção antes da utilização.
- Reagente dissociação Warm-célula suave em um banho de água a 37 ° C.
- Obter AAVS1 TALENS (PZT-AAVS1-L1 e PZT-AAVS1-R1) e AAVS1-CAG-EGFP doador de -20 ° C.
- Remover iPSCs da incubadora e lavar uma vez com DPBS.
- Adicionar 1 ml de reagente de dissociação de células por poço suave, e incubar iPSCs a 37 ° C durante 5 minutos, ou até maior do que 50% das células foram dissociadas a partir do vaso de cultura.
- Pipeta as células para cima e para baixo algumas vezes usando uma pipeta P1000 dissociar as células restantes a partir do vaso de cultura, e para quebrar aglomerados da IPSC.
- Adicionar 2 ml de meio a cada poço E8, e pipeta cima e para baixo várias vezes, utilizando uma pipeta de 10 ml de Further desagregar aglomerados de células em células individuais
NOTA: A eficiência de transfecção declina significativamente se aglomerados de células não são suficientemente desagregada. - Recolhe iPSCs em um tubo cónico de 15 ml e centrifugar a 100 xg durante 3 min.
- Aspirar o sobrenadante e ressuspender as células em uma quantidade mínima de meio E8.
- Contar as células utilizando um hemocitómetro, após aplicação de um corante vital, tal como 0,4% de azul de Tripano. Certifique-se de que as células são suficientemente dissociado durante a contagem (1-3 células por "moita").
- Dispensar 3 x 10 6 células em cada um de dois tubos de 15 mL cónico, e girar para baixo outra vez a 100 xg durante 3 min.
NOTA: centrifugação a baixa velocidade reduz o estresse celular e permite fácil re-suspensão de iPSCs antes eletroporação. - Ajuste o sistema de eletroporação para o programa específico para o tipo de célula para a linha de células-tronco embrionárias humanas H9 (Programa CB-150).
- Após a centrifugação, aspirar o sobrenadante do cell peletes. Para uma pastilha, adicionam-se 10 ug do doador RH como amostra de controlo. Para o outro sedimento adicionar 10 ug do doador HR, juntamente com 5 ug de cada TALEN (PZT-AAVS1-L1 e PZT-AAVS1-R1) como amostra experimental.
- Ressuspender cada pelete de células em 100 ul de solução de transfecção de células primárias P3, e transferir para uma cuvete.
- Realize a transfecção e imediatamente adicionar 500 mL de meio E8 temperatura ambiente em cada cubeta.
- Transfira o iPSCs gota a gota a uma transf 10 cm prato contendo MEFs DR4 preparados no passo 5.4.
- Opcional: Adicionar várias gotas de cada experiência para um poço de uma membrana basal revestida de matriz placa de 6 poços se citometria de fluxo anaylsis de eficiência de transfecção é desejado.
- Repita os passos de 6,15-6,17 para terminar ambas as amostras. No dia seguinte, lavar as células duas vezes com DPBS e alternar o meio de cultura para NutriStem, que parece apoiar cultura iPSC em alimentadores melhor do que E8.
- Trpicos de expressão EGFP Ansient em 48-72 horas pós-transfecção; avaliar a eficiência de transfecção quanto desejado.
7. A puromicina Selecção de iPSCs alvejados
- Comece a selecção de puromicina 72 h após a transfecção, quando as iPSCs alcançar 70% de confluência.
- Para NCRM5 iPSCs, começam selecção puromicina a 0,5 ug / ml de puromicina (1/2 da dose completa) diluída em meio de cultura NutriStem. A concentração óptima pode variar puromicina 0,25-1 ug / MLL para algumas linhas de IPSC.
- IPSCs Cultura em NutriStem + 0,5 ug / ml �puromicina durante 3 dias, refrescando o meio dia.
- Após 3 dias, aumentar a concentração de puromicina a 1 ug / ml.
- IPSCs Cultura nos termos de 1 mg / ml seleção �puromicina por mais 7-8 dias, ou até colônias distintas aparecem grande o suficiente para colônia de picking.
8. Colony Picking e Expansão das iPSCs Targeted
- Membrana basal Usematriz para revestir uma placa de 96 poços por dispensar 50 ul de solução de revestimento de matriz de membrana basal (2 mg de matriz de membrana basal diluídos em 12 ml de DMEM / F12) para cada poço. Armazenar a placa a 4 ° C durante a noite antes da utilização.
- Depois de se deixar a placa revestida com matriz de membrana basal para voltar à temperatura ambiente, aspirar a matriz da membrana basal e dispensar 100 ul E8 suplementado com 10 uM Y-27632 para cada poço.
- Coloque um microscópio invertido em uma cabine de segurança biológica, e pulverizar levemente com etanol 70% para esterilizar.
- Retire uma pipeta de Pasteur de vidro ao longo de um bico de Bunsen para obter uma ferramenta ideal colónia-colheita que tem um pequeno "gancho" na ponta. Esterilizar com etanol 70%, e coloque na capa para secar.
- Remova a placa contendo as colónias IPSC orientadas a partir do incubador, e colocar dentro do capuz sob o microscópio.
- Escolha iPSCs traçando um círculo em torno da fronteira colônia com a ferramenta de colheita colônia.
- Usar o "gancho" da ferramenta colónia-colheita para raspar suavemente e remover a colónia iPSC da superfície da placa. Para grandes colônias, desenhando um X com a ferramenta de colheita colônia de trimestre a colônia irá facilitar o crescimento das células após a re-regulamentação.
- Uma vez que aglomerados de células são separadas e livremente flutuante, use uma pipeta p200 definido para ~ 30 ul para recolher as iPSCs. Células directamente para a placa de 96 poços de placa.
- Opcional: utilizar uma pipeta de p20 definida como ~ 5 mL para recolher as iPSCs para um tubo eppendorf, em seguida, adicionar 50 ul de reagente de dissociação de células suave para digerir durante 5 min a 37 ° C. Após a digestão, adicionar 500 ul meio E8 para dentro do tubo eppendorf para diluir o reagente de dissociação de célula suave e girar para baixo as células a 200 xg numa microcentrífuga de bancada padrão durante 5 min. Em seguida, aspirar a maior parte do meio e deixar ~ 30 ul para ressuspender as células e transferir para a placa de 96 poços.
NOTA: Esteabordagem facilitará a dissociação do aglomerado de células e rápida expansão da colônia colhidos. - Continuar colónia colheita até que um número suficiente de colónias IPSC foram recolhidos (aproximadamente 20-30 colónias por experimento é recomendado).
- Após a colheita de colónias, colocar a placa de 96 poços numa incubadora durante a noite. Refresque-se com meio completo E8 no dia seguinte, e da cultura até ~ 70% confluentes.
- Células de passagem de 96 poços em um de 24 poços, em seguida, em um de 6 poços, como descrito nos passos 4,5-4,10.
NOTA: os volumes de solução de EDTA e meio E8 deve ser proporcionalmente reduzida ao utilizar poços mais pequenos do que uma placa de 6 poços. - Avaliar segmentação sucesso por junção PCR e Southern blot para confirmar a integração cassete alvejado e ausência de vetores inseridas aleatoriamente 16.
Resultados
A visualização do protocolo é fornecido na Figura 2, com períodos em que iPSCs são cultivados em meio diferente destaque por qualquer verde para E8 ou azul para NutriStem. É importante para transfectar apenas iPSCs de alta qualidade; examinar placas de cultura em toda a manutenção de rotina e verificar que as culturas IPSC conter colônias principalmente distintos portadores de uma morfologia de paralelepípedos-like (Figura 3A); células diferenciadas não deve ocupar mais do que 10% da cultura. Transfectabilidade do iPSCs é avaliada e optimizada utilizando o vector de pequeno Pmax-GFP (Figura 4A-B), tal como transfecção com Pmax-GFP tipicamente representa a eficiência máxima realizável devido ao seu pequeno tamanho. Por exemplo, obtivemos 68,6% de eficiência de transfecção com Pmax-GFP (Figura 4B). Para direcionar o AAVS1 porto seguro, iPSCs são passadas usando um reagente dissociação de uma única célula gentil e são transfectadas com AAVS1-TALENS e AAVS1-CAG-EGFP (plasmídeos representados na Figura 1A). IPSCs transfectadas são então plaqueadas numa densidade adequada de DR4 MEFs (Figura 3B). A expressão transiente de picos AAVS1-CAG-EGFP em 48 a 72 horas após a transfecção (Figura 3C); se desejado, uma pequena porção de iPSCs transfectadas podem ser analisados por SCAF. NCRM5 iPSCs podem ser transfectadas com o rendimento de 60,9% utilizando AAVS1-CAG-EGFP doador (Figura 4C). A eficiência de transfecção pode variar muito; para experimentos em que a GFP + fração é ainda um preço tão baixo quanto 10%, continuam a seleção puromicina. Depois de realizar a seleção �puromicina, clones individuais exibindo fluorescência uniforme deve ser grande o suficiente para colônia de picking (Figura 3D). Para escolher clones, uma colônia adequado está localizado em pequeno aumento (Figura 3E) e é isolado por primeiro traçado e esquartejando-lo (Figuras 3F-G). As colônias esquartejado são então rasped a partir do recipiente de cultura (Figura 3H). Usando uma pipeta de p200, aglomerados de células são então transferidos para um poço de uma membrana basal revestida de matriz placa de 96 poços. iPSCs deve anexar dentro de 2-4 horas de revestimento (Figura 3I). Se o apego não é observado dentro de 24 horas, Basement membrana matriz de revestimento ou Y-27632 tratamento foram provavelmente sub-óptima; Basement membrana matriz-coat uma nova placa de 96 poços e preparar fresco E8 + 10? M Y-27632 antes de pegar mais nenhuma colônias. Uma vez que num formato de 96 poços, orientada IPSC clones são expandidos e deve exibir uma expressão estável e uniforme de GFP (Figuras 4D-E).

Figura 1: Gene segmentação vetores e segmentação esquemática. (A) Representação da AAVS1-CAG-EGFP, e os plasmídeos de PZT-AAVS1-L1 / R1 TALENS utilizados neste estudo. O SA-2A elemento no plasmídeo AAVS1-CAG-EGFP representa o aceitador de splice-2A e auto-clivagem de péptido utilizado para restringir puromicina N-acetil-transferase (PAC) para a expressão do gene alvo eventos de integração. A globina frango β-actina (CAG) promotor é usado para conduzir a expressão de EGFP. (B) O AAVS1 porto seguro, contidos Intron 1 do gene PPP1R12C, é alvo usando TALENS para gerar uma quebra de cadeia dupla de DNA. Isso ativa a recombinação homóloga (HR) máquinas de reparação, que, em seguida, usa os (braços de homologia ostentam flanqueando o local do corte) AAVS1-CAG-EGFP doador como um substrato para o reparo. A cassete é integrado o gene PAC e é colocado sob o controlo do promotor PPP1R12C endógeno.
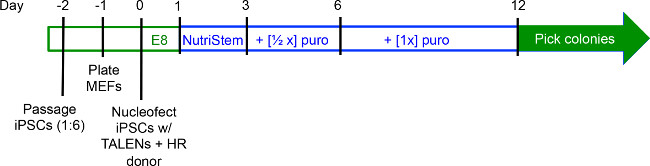
Figura 2. Timeline do gene experimento segmentação. IPSCs são cultivadasa 70% de confluência e passadas ~ 1: 6 no dia -2 (d-2). MEFs são plaqueadas em d-1, e iPSCs são recolhidos e transfectados em d0. No d1, o meio é mudado para NutriStem, e iPSCs são cultivadas durante mais dois dias antes de puromicina é adicionado ao meio em d3. Colônias são tipicamente pronto para a colheita por d12. Períodos durante os quais iPSCs são cultivadas em meio E8 são destacadas verde, enquanto que os períodos de cultura NutriStem estão em azul.

Figura 3: gene iPSC segmentação imagens representativas (A) imagem de iPSCs de alta qualidade Fase.. Note-se a fronteira de fase brilhante e morfologia paralelepípedo semelhante. (B) DR4 MEFs plaqueadas a 2 x 10 4 células / cm2 24 horas após a descongelação. (C) Setenta e duas horas após a transfecção, as células EGFP + são claramente aparente quando visto sob um micr fluorescênciaoscope. (D) Após a seleção baseada em puromicina para ~ 14 dias, as colônias que exibem fluorescência da GFP uniforme são grandes o suficiente para ser escolhido. (EH) Imagens representativas do processo de picking colônia. As colónias são colhidas utilizando uma pipeta de Pasteur puxado, em primeiro lugar, ao apontar a colónia, e, em seguida, pelo método dos quartos para se obter aglomerados de células mais pequenas. (I) escolheu colónias IPSC anexar à superfície revestida de matriz de membrana basal da placa de 96 poços no prazo de 2 a 4 horas. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 4: análise de FACS de eficiência de transfecção e iPSCs clonais orientadas. (A) Controlo NCRM5 iPSCs sem qualquer transfecção de plasmídeo. (B) NCRM5iPSCs transfectadas com o vector de pequeno teste Pmax-GFP são analisadas por FACS para a expressão de GFP. (C) A expressão transitória do plasmídeo AAVS1-CAG-EGFP é avaliada por FACS. (D) Uma NCRM5-AAVS1-CAG-EGFP clone exibe uniformemente expressão EGFP positivo, tal como testado por FACS. Observe o agrupamento apertado de iPSCs analisados em comparação com C). (Imagem E) Fluorescência de um NCRM5-AAVS1-CAG-EGFP clone alvejado expandida.
Discussão
Os passos mais críticos para a geração bem sucedida de AAVS1 porto seguro alvejado iPSCs humanos são: (1) a entrega eficiente TALEN e doadores plasmídeos em iPSCs por transfecção; (2) optimização de dissociação iPSCs em células individuais antes da transfecção e chapeamento densidade após transfecção; (3) a optimização da dose e do tempo de selecção de droga com base no crescimento da linha iPSC; (4) cuidadosamente dissecar e escolhendo colônias direcionados e transferindo a nova placa / poço. Em comparação com métodos similares utilizados no papel de Hockemeyer 10, este protocolo usado um par de código-fonte aberto AAVS1-TALENS, diferente reagente dissociação iPSC, e dispositivo de transfecção diferente para entregar TALENS e doadores vetores, o que ajudou a reduzir o número de iPSCs utilizado na experiência ao conseguir alta transfecção e visando ganhos de eficiência.
Para alcançar edição gene de alta eficiência em iPSCs humanos, é essencial para optimizar a primeira entrega de edi genereagentes ting (DNA / RNA), enquanto que o equilíbrio da morte celular aguda do método de entrega e reagentes para causar iPSCs. O objectivo é o de maximizar a eficiência de entrega, mantendo um nível aceitavelmente baixo de morte celular. Uma vez que é muito fácil para preparar grandes quantidades de plasmídeos AAVS1-TALEN que pode atingir> 50% de eficiência de RH com a ajuda de-selecção de drogas em iPSCs humanos, não é necessário fazer mRNAs a partir dos plasmídeos TALEN. O desafio de entrega vem de grandes plasmídeos dadores, que neste caso é ~ 10 kb. Recomenda-se usar o doador AAVS1-CAG-EGFP para testar a eficiência de entrega em linhas de IPSC humanos específicos ao invés de usar pMaxGFP incluído no kit de transfecção, porque pMaxGFP é um ~ 3,5 pequeno plasmídeo kb e muito fácil para entregar em todas as células. AAVS1-CAG-EGFP dador pode ser modificado utilizando enzimas de restrição mostradas na Figura 1A para atingir diferentes transgenes no locus de AAVS1. Outro doador de 12 kb, que contém uma cassete de 6,4 kb para substituir CAG-EGFP, tem seren utilizados com sucesso para alcançar eficiência de transfecção e segmentação semelhante (dados não mostrados). Em geral, iPSCs humanos estão entre os tipos de células de difícil transfecção e a eficiência de entrega para grandes plasmídeos pode ser tão baixa como 5-10%, conforme medido por análise de citometria de fluxo de células GFP +. Uma posterior optimização da eficiência de transfecção é dependente da dissociação apropriada de iPSCs humanos em células individuais, aglomerados de células, porque reduzirá a possibilidade de entregar edição gene reagentes em cada célula. Este protocolo usa um reagente suave e dissociação celular fast-trabalho, mas o tratamento iPSCs por mais de 10 minutos não é recomendado. Normalmente, a cultura iPSC é inferior a 80% confluente se segue passo 5.1, fazendo uma incubação de 5 minutos com o reagente pré-aquecido a dissociação de células suave geralmente suficiente para dissociar os iPSCs em células individuais com pipetagem suave. Se nem todas as células podem ser dissociadas em células isoladas após 10 min de tratamento, porque a cultura de células é sobre-confluente ou as colônias tornaram-se muito compacto, simplesmente recolher o número recomendado de células individuais de mais poços / placas e deixar as células firmemente ligados trás. Pipetagem pesado não é recomendado porque corte mecânico é mais prejudicial para as células do que dissociação enzimática. Ao trabalhar com iPSCs-passagem precoce (antes da passagem # 15), a prática de dissociação suave é muito importante para a sobrevivência da célula, porque as células já são mais sensíveis ao estresse transfecção de iPSCs passagem final. Se é difícil de conseguir de alta eficiência de transfecção em ambos e a sobrevivência de células, a escolha prática que dá a mais alta eficiência de transfecção. Após a transfecção, as células devem ser plaqueadas a uma densidade que vai chegar a 50-80% de confluência no dia 3, quando inicia-selecção de drogas. Uma vez que cada linha iPSC cresce de maneira diferente, a densidade de revestimento de pós-transfecção pode precisar ser otimizado para cada linha celular. Uma elevada densidade de chapeamento pode levar a confluência ao longo de-que reduz a eficácia da Drug-seleção, enquanto um extremo baixo densidade de revestimento pode causar atraso na recuperação e crescimento de colônias resistentes aos medicamentos. Normalmente 3 milhões iPSCs de partida são suficientes para ser plaqueada em ½ a 2 placas de 10 cm, dependendo da sobrevivência e crescimento de iPSCs específicos. Da mesma forma, se estiver usando uma linha iPSC recém-gerado ou ainda não testado, gerando uma curva �puromicina kill para estabelecer a menor dose eficaz em células untransfected julgar necessárias; linhas de IPSC podem variar consideravelmente em sua sensibilidade à seleção puromicina. Os MEFs foram utilizados para a seleção porque eles aparecem para apoiar o crescimento iPSC melhor do que algumas matrizes extracelulares durante a seleção de drogas. Como uma regra geral, um mínimo de 5 dias de puromicina nos dias 1 ug / ml ou 7 a 0,5 ug / ml, são necessários para completar a selecção. Finalmente, técnica de colheita colônia é crucial para preservar todas as colônias alvo após a seleção de drogas. Pipetagem suave ou tratamento reagente dissociação ajuda a quebrar as colônias em vários pedaços uniformementedistribuído para o novo bem, e, portanto, pode acelerar a expansão colónia. Se estiver usando um reagente dissociação de desagregar as colônias escolhidas antes de chapeamento, verifique se ele está suficientemente diluído com> 10x meio após o tratamento, porque o reagente dissociação residual poderia matar as células transferidas se deixado ligado durante a noite. Não importa qual a técnica é utilizada, praticando colônia escolhendo antes das experiências reais é altamente incentivado.
À medida que a estratégia descrita utiliza um método gene-armadilha para conduzir a expressão do gene PAC fora do promotor endógeno PPP1R2C, separação significativamente mais de 30 colónias não for considerada necessária. O SA-2A ligada selecção PAC teoricamente apenas elimina células-integração aleatória, mas a integração aleatória adicional (s) pode ocorrer em iPSCs rolamento integrações alvejados com sucesso. Na maioria dos casos, quase 100% dos clones selecionados por drogas têm como alvo as integrações e ~ 10-40% deles têm Integrati aleatória adicionalons. A maioria dos clones orientados tendem a ter direccionamento de um único alelo, embora experiências em que> 50% direccionamento duplo alelo ocorre. A frequência variável de integrações aleatórias adicionais, bem como a razão da única contra a segmentação de duplo alelo, é difícil de controlar. Portanto, escolhendo 20 ~ colónias é recomendado para assegurar simples ou dupla alelo direccionamento somente os clones podem ser obtidos. À luz das experiências mal sucedidas de segmentação, re-avaliação das taxas de transfectabilidade, sobrevivência e crescimento das células alvo ', a eficácia de nucleases na linha de célula específica, e a qualidade das preparações de doadores e nuclease é altamente recomendado antes de voltar a tentar a experiência na sua totalidade.
Deve notar-se que as estratégias semelhantes doadores gene armadilha pode ser utilizada para direccionar outros genes activos, mas não é adequado para direccionamento do gene silencioso que requer um promotor independente para conduzir a expressão do gene seleccionável. Além disso, enquanto the AAVS1 porto seguro é considerado como tendo a estrutura da cromatina aberta, isso não garante que qualquer transgene pode ser expressa fortemente neste locus. Na verdade, o nosso relatório recente mostrou que vários promotores fracos eram incapazes de conduzir a expressão do gene repórter fluorescente detectável após a integração orientada para AAVS1 lócus 14.
Este protocolo se concentra no uso TALENS bem validadas para atingir um locus porto seguro bem estudado no genoma humano. As técnicas são altamente reprodutível e facilmente adaptável a muitas aplicações que envolvam qualquer transgene. Comparado com os métodos de engenharia do genoma utilizando integrações aleatórias, AAVS1-TALEN genes mediada direccionamento é altamente eficiente e específico, e, no caso de iPSCs, resultou em células fluorescentes de forma estável que têm o potencial para se expandir e se diferenciar em qualquer tipo de célula humana. Embora este protocolo não descrever a concepção e validação de nucleases de grife ou a avaliação de colônias IPSC direcionados para pintegração cassete Roper e análise off-alvo, vários protocolos excelentes têm descrito esses aspectos da metodologia em detalhe 16-18. IPSC cultura e Passaging técnicas livre-de alimentação utilizando meio E8 também são descritos em detalhes na publicação anterior 19. O protocolo anterior, enquanto optimizado para a adição de genes para o AAVS1 de limiar de segurança de iPSCs humanos, podem servir como um modelo geral para experiências que envolvem específica do local mediada por nuclease gene edição / adição utilizando um dador de recombinação homóloga em qualquer tipo de célula.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
This research was supported by the NIH Common Fund and Intramural Research Program of the National Institute of Arthritis, Musculoskeletal, and Skin Diseases.
Materiais
| Name | Company | Catalog Number | Comments |
| Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix, *LDEV-Free, 10 ml | Corning | 354230 | Store at -20 °C. |
| DMEM/F-12 | Life Technologies | 11320-033 | Store at 4 °C. |
| Costar 6 Well Clear TC-Treated Multiple Well Plates | Corning | 3506 | |
| Essential 8 Medium | Life Technologies | A1517001 | Store basal medium at 4 °C. Store supplement at -20 °C. |
| Y-27632 dihydrochloride | Tocris | 1254 | Store at room temp. Once dissolved in H2O, store at -20 °C. |
| Sodium Chloride | Sigma | S5886-500G | |
| UltraPure 0.5 M EDTA, pH 8.0 | Life Technologies | 15575-020 | |
| DPBS, no calcium, no magnesium | Life Technologies | 14190-250 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| DR4 MEF 2M IRR - Academic | GlobalStem | GSC-6204G | Store in liquid Nitrogen. |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-040 | Store at 4 °C. |
| Defined Fetal Bovine Serum, US Origin | HyClone | SH30070.03 | Store at -20 °C. Thaw at 4 °C overnight and aliquot. Store aliquots at -20 °C until needed. |
| MEM Non-Essential Amino Acids Solution (100X) | Life Technologies | 11140-050 | Store at 4 °C. |
| 4D-Nucleofector Core unit | Lonza | AAF-1001B | part of the electroporation system |
| 4D-Nucleofector X unit | Lonza | AAF-1001X | part of the electroporation system |
| P3 Primary Cell 4D-Nucleofector X Kit L (24 RCT) | Lonza | V4XP-3024 | Upon arrival, remove Primary Cell Solution and supplement and store at 4 °C. |
| StemPro Accutase Cell Dissociation Reagent | Life Technologies | A1110501 | Store at -20 °C. Thaw overnight at 4°C and warm an aliquot in a 37 °C water bath before use. |
| NutriStem XF/FF Culture Medium | Stemgent | 01-0005 | Store at -20 °C. Thaw overnight at 4 °C |
| AAVS1 TALENs (pZT-AAVS1-L1 and pZT-AAVS1-R1) | Addgene | 52637 and 52638 | |
| [header] | |||
| AAVS1-CAG-EGFP Homologous Recombination donor | Addgene | 22212 | |
| Puromycin Dihydrochloride | Life Technologies | A11138-03 | Store at -20 °C. Prepare working aliquots of 1 mg/ml in ddH2O. |
| Disposable Borosilicate Glass Pasteur Pipets | Fisher Scientific | 13-678-20A | |
| Sorvall Legend XTR (Refrigerated), 120 V 60 Hz | Thermo Scientific | 75-004-521 | |
| TX-750 4 × 750 ml Swinging Bucket Rotor | Thermo Scientific | 75003607 | |
| Trypan Blue Solution, 0.4% | Life Technologies | 15250-061 | |
Referências
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Hentze, H., Graichen, R., Colman, A. Cell therapy and the safety of embryonic stem cell-derived grafts. Trends Biotechnol. 25 (1), 24-32 (2007).
- Smith, J. R., et al. Robust, persistent transgene expression in human embryonic stem cells is achieved with AAVS1-targeted integration. Stem Cells. 26 (2), 496-504 (2008).
- Lombardo, A., et al. Site-specific integration and tailoring of cassette design for sustainable gene transfer. Nat Methods. 8 (10), 861-869 (2011).
- Ogata, T., Kozuka, T., Kanda, T. Identification of an insulator in AAVS1, a preferred region for integration of adeno-associated virus DNA. J Virol. 77 (16), 9000-9007 (2003).
- Zwaka, T. P., Thomson, J. A. Homologous recombination in human embryonic stem cells. Nat Biotechnol. 21 (3), 319-321 (2003).
- Urbach, A., Schuldiner, M., Benvenisty, N. Modeling for Lesch-Nyhan disease by gene targeting in human embryonic stem cells. Stem Cells. 22 (4), 635-641 (2004).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nature biotechnology. 27 (9), 851-857 (2009).
- Zou, J., et al. Gene targeting of a disease-related gene in human induced pluripotent stem and embryonic stem cells. Cell stem cell. 5 (1), 97-110 (2009).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nature. 29 (8), 731-734 (2011).
- Sanjana, N. E., et al. A transcription activator-like effector toolbox for genome engineering. Nature protocols. 7 (1), 171-192 (2012).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Ding, Q., et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell stem cell. 12 (4), 393-394 (2013).
- Luo, Y., et al. Stable Enhanced Green Fluorescent Protein Expression After Differentiation and Transplantation of Reporter Human. Induced Pluripotent Stem Cells Generated by AAVS1 Transcription Activator-Like Effector Nucleases. Stem Cells Transl Med. 3 (7), 821-835 (2014).
- Zou, J., et al. Oxidase-deficient neutrophils from X-linked chronic granulomatous disease iPS cells: functional correction by zinc finger nuclease-mediated safe harbor targeting. Blood. 117 (21), 5561-5572 (2011).
- Luo, Y., Rao, M., Zou, J. Generation of GFP Reporter Human Induced Pluripotent Stem Cells Using AAVS1 Safe Harbor Transcription Activator-Like Effector Nuclease. Curr Protoc Stem Cell Biol. 29, 5A.7.1-5A.7.18 (2014).
- Peters, D. T., Cowan, C. A., Musunuru, K. Genome editing in human pluripotent stem cells. StemBook. , Harvard Stem Cell Institute. Cambridge, MA. Available from: http://www.stembook.org/node/1438 (2008-2013).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Beers, J., et al. Passaging and colony expansion of human pluripotent stem cells by enzyme-free dissociation in chemically defined culture conditions. Nat Protoc. 7, 2029-2040 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados