Method Article
Transfection, selezione, e Colonia-picking di cellule staminali umane pluripotenti indotte TALEN-mirati con Gene GFP nel AAVS1 Safe Harbor
In questo articolo
Riepilogo
TALEN-mediated gene editing at the safe harbor AAVS1 locus enables high-efficiency transgene addition in human iPSCs. This protocol describes the procedures for preparing iPSCs for TALEN and donor vector delivery, transfecting iPSCs, and selecting and isolating iPSC clones to achieve targeted integration of a GFP gene to generate reporter lines.
Abstract
Targeted transgene addition can provide persistent gene expression while circumventing the gene silencing and insertional mutagenesis caused by viral vector mediated random integration. This protocol describes a universal and efficient transgene targeted addition platform in human iPSCs based on utilization of validated open-source TALENs and a gene-trap-like donor to deliver transgenes into a safe harbor locus. Importantly, effective gene editing is rate-limited by the delivery efficiency of gene editing vectors. Therefore, this protocol first focuses on preparation of iPSCs for transfection to achieve high nuclear delivery efficiency. When iPSCs are dissociated into single cells using a gentle-cell dissociation reagent and transfected using an optimized program, >50% cells can be induced to take up the large gene editing vectors. Because the AAVS1 locus is located in the intron of an active gene (PPP1R12C), a splicing acceptor (SA)-linked puromycin resistant gene (PAC) was used to select targeted iPSCs while excluding random integration-only and untransfected cells. This strategy greatly increases the chance of obtaining targeted clones, and can be used in other active gene targeting experiments as well. Two weeks after puromycin selection at the dose adjusted for the specific iPSC line, clones are ready to be picked by manual dissection of large, isolated colonies into smaller pieces that are transferred to fresh medium in a smaller well for further expansion and genetic and functional screening. One can follow this protocol to readily obtain multiple GFP reporter iPSC lines that are useful for in vivo and in vitro imaging and cell isolation.
Introduzione
La possibilità di riprogrammare le cellule somatiche umane in cellule staminali pluripotenti indotte cellule simil-staminali embrionali (iPSCs) fu scoperto da Takahashi et al., Nel 2007 1. Fibroblasti dermici umani trasdotte con retrovirus che esprime quattro fattori di trascrizione (Il Yamanaka così soprannominato Fattori Oct3 / 4, Sox2, c-Myc e Klf4) hanno mostrato di essere molto simili alle cellule staminali embrionali umane (hESC) basati sulla morfologia, la proliferazione, l'espressione genica, e lo stato epigenetico; cruciale, iPSCs sono anche in grado di differenziarsi in cellule di tutti e tre i foglietti embrionali 1. Il potenziale proliferativo e la differenziazione della capacità di iPSCs li rende strumenti molto interessanti; riprogrammando cellule di pazienti affetti da malattie specifiche, iPSCs possono essere utilizzati sia come sistemi modello in vitro di malattia e come potenziali terapie.
Per quest'ultimo scopo, diverse questioni devono essere affrontate prima che il pieno potenziale di iPSCsin un ambiente clinico può essere realizzato; il potenziale oncogeno di hESC coltivate in vitro e iPSCs, l'uso di derivati xenogeniche durante la riprogrammazione e la manutenzione delle cellule, e la necessità di tenere traccia di cellule trapiantate in vivo sono tutti ostacoli cruciali per l'applicazione clinica delle cellule staminali pluripotenti (Inviato da Hentze et al. 2). Una soluzione ideale per la necessità di monitoraggio cellule differenziate post-trapianto comporterebbe un marcatore visivamente rilevabile che resiste tacere e variegatura indipendentemente dall'applicazione. Espressione robusta e sostenuta di transgeni integrati è più facilmente realizzabile quando il DNA esogeno viene introdotto in porto sicuro loci; cioè, i siti genomici che consentono sufficienti trascrizione di un vettore integrato mentre allo stesso tempo mitigare perturbazioni di espressione in geni vicini 3. Uno di questi siti che è stato molto ben caratterizzati dalla sua scoperta è il virus integr adeno-associatosito zione 1 (AAVS1), nel primo introne della proteina fosfatasi 1 subunità 12C regolamentazione (PPP1R12C) gene. Questo locus è stato dimostrato non solo di permettere sostenuta e robusta espressione dei transgeni integrati attraverso il tempo prolungato nella cultura e nella differenziazione vitro 3, ma anche per proteggere i geni di perturbazione trascrizionale 4 circostante; entrambe le funzioni si pensa sia dovuto alla presenza di elementi endogeni isolante cromatina che fiancheggiano il sito AAVS1 5.
Advances in strumenti di ingegneria del genoma sul proprio nell'ultimo decennio hanno notevolmente facilitato la facilità e l'efficienza con cui le manipolazioni genetiche in qualsiasi tipo di cellula possono essere raggiunti. Mentre i primi esperimenti riusciti invocati estremamente bassi livelli di endogena ricombinazione omologa (HR) con un donatore introdotte per ottenere gene targeting in CES 6,7, l'uso di nucleasi sito-specifiche, quali nucleasi dita di zinco (ZFNs), che signi, significativamente indurre ricombinazione omologa attraverso la generazione di un doppio filamento rottura del DNA ha notevolmente aumentato l'efficienza di tali esperimenti 8,9. La riproposizione di entrambi trascrizione attivatore come effettori (racconti) di pianta patogeno xanthomonas generi e le procariote cluster brevi ripetizioni palindromiche regolarmente intervallati (CRISPR) Sistema / Cas9 in efficienti nucleasi progettista site specific ha fatto gene targeting nelle cellule staminali pluripotenti un accessibile e metodologia possibile 10-13.
Un recente ha descritto un metodo efficace per l'integrazione stabile di una cassetta reporter fluorescente verde nel AAVS1 porto sicuro locus in iPSCs umani con nucleasi Tale (Talens) 14. Questi iPSCs mirati hanno mantenuto la loro fluorescenza, anche dopo la differenziazione diretta a cardiomiociti e il trapianto in un modello murino di infarto del miocardio (MI), che fornisce una forte evidenza per l'utilità di talestabilmente pluripotenti fluorescente cellule staminali 14. Per ottenere colonie mirati, un metodo gene-trap è stato usato cui un splicing-accettore (SA), 2A sequenza peptidica auto-scissione colloca la puromicina N-acetil-transferasi (PAC) gene sotto il controllo del promotore PPP1R12C endogena; quindi, solo iPSCs che hanno incorporato il donatore DNA a livello del locus AAVS1 esprimono PAC, rendendoli selezionabili in base puromicina-resistenza; (Figura 1, 15). Dettagli Questo protocollo le procedure di generare AAVS1-GFP iPSCs segnalato nel recente documento 14, tra cui il processo di trasfettando iPSCs con Talens e 9,8 kb donatori di integrare un frammento di 4.2kb DNA nel locus AAVS1 porto sicuro, selezionando iPSCs basati su puromicina-resistenza, e le colonie di prelievo per l'espansione clonale. Le tecniche qui descritte possono essere applicate a vari esperimenti di ingegneria genoma.
Protocollo
1. Preparazione della matrice di membrana basale e rivestimento dei Plasticware
- Posizionare il congelato membrana basale matrice magazzino da -20 ° C su ghiaccio e scongelare una notte a 4 ° C.
- Dopo lo scongelamento, pipetta 2 mg aliquote di matrice membrana basale in tubi Eppendorf pre-refrigerati. Conservare questi a -20 ° C fino al momento dell'uso.
- Per preparare piatti matrice rivestite membrana basale, scongelare un'aliquota sul ghiaccio fino a quando l'ultimo pezzo di ghiaccio nei scompare provetta Eppendorf (di solito entro ~ 2 ore).
- Dopo lo scongelamento, aggiungere matrice membrana basale a 12 ml di freddo (4 ° C) DMEM / F12 per rendere membrana basale soluzione di rivestimento matrice.
- Aggiungere la soluzione di matrice membrana basale alla nave cultura appropriata. Per un 6-pozzetti, dispensare 1 ml per pozzetto. Agitare la piastra per assicurarsi che la soluzione di matrice membrana basale copre completamente ogni pozzetto.
- Parafilm sigillare la membrana basale matrice rivestite piatto / piatto, e incubare a room temperatura per 1 ora prima dell'uso. In alternativa, le piastre matrice rivestite negozio membrana basale / piatti a 4 ° C e utilizzare entro 2 settimane di rivestimento.
NOTA: Aggiungere ulteriore DMEM / F12 alla membrana basale matrice rivestite piatto / piatto per evitare l'essiccazione. Prima di utilizzare 4 ° C membrana basale memorizzato matrice rivestite piatto / piatto, mettetelo in una cappa di sicurezza biologica e lasciare che raggiunga la temperatura ambiente per almeno 30 minuti. - Aspirare matrice membrana basale completamente prima della aggiunta del mezzo e cellule.
2. Preparazione di E8 media
- Preparare E8 terreno di coltura da scongelamento E8 supplemento notte a 4 ° C.
- Rimuovere 10 ml di E8 media basale del magazzino da 500 ml e scartare.
- Pipettare l'intero flaconcino da 10 ml di E8 supplemento direttamente in 490 ml di E8 medio basale. Non caldo terreno completo E8 in un bagno d'acqua a 37 ° C, come sbalzi di temperatura ripetuti possono degradare il bFGF nel complete E8 media.
- Utilizzare mezzo completo E8 entro 2 settimane di aggiunta del supplemento.
3. scongelamento dei iPSCs
- Rimuovere una fiala di iPSCs congelati da azoto liquido e posto in ghiaccio secco.
- Scongelare rapidamente la provetta in un bagno d'acqua a 37 ° C; agitare il flacone a bagnomaria fino a quando solo un piccolo frammento di resti di ghiaccio.
- Spruzzare il flaconcino con il 70% di etanolo e trasferimento in una cappa di sicurezza biologica.
- Aggiungere 1 ml di temperatura ambiente E8 medio a gocce direttamente al flacone.
- Con una pipetta 2 ml, trasferire la sospensione cellulare a gocce in 9 ml di E8 media in un tubo da 15 ml. Agitare il tubo frequentemente per assicurare che le cellule e medie mescolare bene rapidamente.
- Centrifugare le cellule a 200 xg per 5 min.
- Aspirare il surnatante e risospendere il pellet cellulare in un volume adeguato di E8 supplementato con 10 micron Y-27632.
- Aggiungere le cellule ad un numero adeguato di seminterrato membrane pozzi matrix-rivestito, e posto a 37 ° C, 5% CO 2 incubatore durante la notte. Si raccomanda al piatto almeno 0,2 x 10 6 iPSC per pozzetto di un 6-pozzetti per consentire rapido recupero dopo lo scongelamento.
4. Manutenzione e Passaging routine di iPSCs
- Aggiorna E8 media giornaliera.
- Monitorare la morfologia e la confluenza di cellule con un microscopio invertito. iPSCs di alta qualità crescono in colonie piatte con bordi distinti; singole colonie in possesso di un aspetto "acciottolato-like".
- Passage le cellule iPSCs quando raggiungono ~ 70% di confluenza.
- Preparare una soluzione EDTA passaging aggiungendo 0,9 g NaCl e 500 microlitri 0,5 M EDTA a 500 ml DPBS. Mescolare bene per sciogliere NaCl, e filtro di vuoto per sterilizzare. Scaldare una aliquota della soluzione passaging in un bagno d'acqua a 37 ° prima passaging.
- Per il passaggio, aspirare speso terreno di coltura e lavare le cellule una volta con un uguale volume di pas caldiSaging soluzione. Aspirare, e pipetta abbastanza EDTA soluzione passaging per rivestire le celle (1 ml per pozzetto di un 6-pozzetti).
- Mettere le cellule al microscopio invertito e osservare le colonie IPSC. La comparsa di fori all'interno colonie e bordi rialzati dovrebbe diventare evidente entro 2 a 5 min.
- Aspirare accuratamente la soluzione di EDTA passaging.
- Con una pipetta 10 ml, a meno di 4 ml di E8 medio (se si utilizza un 6-pozzetti) ad alta pressione direttamente in ogni pozzetto da diversi passaggi.
- Raccogliere le macchie IPSC, e suddiviso in un numero adeguato di pozzi in funzione del rapporto di divisione da 1: 8 a 1:12. Non over-pipetta, come disaggregazione dei grumi di cellule si provocherebbe un calo della redditività.
- Porre la piastra in un incubatore, e oscillare la piastra posteriore e indietro e da lato a lato diverse volte per disperdere le cellule.
5. Preparazione di MEF e iPSCs per Tansfection
- 48 ore prima transfection, iPSCs passaggio a ~ 1: 6 in quattro o più pozzi di un 6-pozzetti membrana basale matrice rivestite, in modo che siano il 70% confluenti due giorni da qui.
- Il giorno successivo, scongelare DR4 MEF in media MEF costituito da DMEM (glucosio) supplementato con 10% FBS e 1x MEM-NEAA.
- Piatto DR4 MEF in due piatti da 10 cm a ~ 2 x 10 4 cellule / cm 2 e incubare una notte a 37 ° C.
- Il giorno della trasfezione, modificare medio MEF a E8 supplementato con 10 mM Y-27632 30 min prima di eseguire trasfezione su iPSCs.
- Opzionale: Se si desidera citometria a flusso di iPSCs post-trasfezione, rimuovere una piastra a matrice rivestita membrana basale tra 4 ° C e posto a temperatura ambiente.
- Optional: 4 ore prima della trasfezione, completare pre-trasfezione cultura iPSC con Y-27632 alla concentrazione finale di 10 micron.
6. Gentle cellule dissociazione reagente Treatment unnd Transfection di iPSCs tramite un sistema di elettroporazione
- Rimuovere P3 soluzione trasfezione cellula primaria di 4 ° C e lasciare che raggiunga la temperatura ambiente per ~ 30 min. Aggiungere l'intero complemento 100 microlitri della soluzione di trasfezione prima dell'uso.
- Warm dolce-cell reagente dissociazione in un bagno di 37 ° C dell'acqua.
- Ottenere AAVS1 Talens (PZT-AAVS1-L1 e PZT-AAVS1-R1) e AAVS1-CAG-EGFP donatore da -20 ° C.
- Rimuovere iPSCs dal termostato e lavare una volta con DPBS.
- Aggiungere 1 ml di delicata cellule dissociazione reagente per pozzetto e incubare iPSCs a 37 ° C per 5 minuti, o fino superiore al 50% delle cellule sono dissociato dal recipiente di coltura.
- Pipettare le cellule su e giù per un paio di volte con una pipetta P1000 a dissociarsi tutte le rimanenti cellule dalla nave in cultura, e per rompere grumi IPSC.
- Aggiungere 2 ml di E8 media ad ogni bene, e pipetta su e giù più volte con una pipetta 10 ml a Fürther disaggregare grumi di cellule in cellule singole
NOTA: l'efficienza Transfection declina in modo significativo se grumi di cellule non sono sufficientemente disaggregati. - Raccogliere iPSCs in un tubo da 15 ml e centrifugare a 100 xg per 3 min.
- Aspirare il surnatante e risospendere le cellule in una quantità minima di E8 medie.
- Contare le cellule utilizzando un emocitometro dopo l'applicazione di una macchia vitali come 0,4% Trypan blu. Assicurarsi che le cellule siano sufficientemente dissociati, mentre il conteggio (1-3 cellule per "macchia").
- Dispensare 3 x 10 6 cellule in ciascuno dei due 15 ml conici, e far girare di nuovo a 100 xg per 3 min.
NOTA: bassa centrifugazione velocità riduce lo stress cellulare e permette un facile risospensione di iPSCs prima elettroporazione. - Impostare il sistema di elettroporazione per il programma specifico di cellule-tipo per la linea di cellule staminali embrionali umane H9 (programma CB-150).
- Dopo centrifugazione, aspirare il supernatante dal celpellets l. Per un pellet, aggiungere 10 mg del donatore HR come campione di controllo. Per l'altra pellet aggiungere 10 mg del donatore HR, insieme a 5 mg di ogni TALEN (PZT-AAVS1-L1 e PZT-AAVS1-R1) come campione sperimentale.
- Risospendere ogni pellet cellulare in 100 ml di soluzione di trasfezione cellulare primaria P3, e trasferire in una provetta.
- Eseguire la trasfezione e aggiungere immediatamente 500 ml di temperatura ambiente medio E8 ad ogni provetta.
- Trasferire il iPSCs gocce transfettate per un piatto di 10 cm contenente MEF DR4 preparati in fase 5.4.
- Optional: Aggiungere qualche goccia di ciascun esperimento in un pozzetto di un 6-pozzetti membrana basale matrice rivestite se citometria a flusso anaylsis di efficienza di trasfezione è desiderato.
- Ripetere i passaggi da 6,15-6,17 a finire entrambi i campioni. Il giorno seguente, lavare le cellule con DPBS due volte e passare terreno di coltura per NutriStem, che sembra sostenere la cultura iPSC su alimentatori meglio di E8.
- TrAnsient picchi di espressione EGFP da 48 a 72 ore dopo la trasfezione; valutare l'efficienza di trasfezione, se lo desideri.
7. puromicina Selezione iPSCs mirati
- Inizio selezione puromicina 72 ore dopo la trasfezione quando i iPSCs raggiungono il 70% di confluenza.
- Per NCRM5 iPSCs, iniziare selezione puromicina a 0,5 mg / ml puromicina (1/2 la dose completa) diluito in NutriStem terreno di coltura. La concentrazione ottimale può variare puromicina 0,25-1 mg / MLL per alcune linee IPSC.
- IPSCs cultura in NutriStem + 0,5 mg / ml puromicina per 3 giorni, rinfrescanti medio quotidiano.
- Dopo 3 giorni, aumentare la concentrazione di puromicina a 1 ug / ml.
- IPSCs Cultura inferiore a 1 mg / ml puromicina selezione per altri 7 a 8 giorni, o fino a quando le colonie distinte appaiono abbastanza grande per colonia picking.
8. Colony Picking ed espansione di iPSCs mirate
- Usa membrana basaleMatrice per rivestire una piastra a 96 pozzetti dispensando 50 ml di soluzione di rivestimento matrice membrana basale (2 mg matrice membrana basale diluito in 12 ml DMEM / F12) in ogni pozzetto. Conservare la piastra a 4 ° C per una notte prima dell'uso.
- Dopo aver lasciato la piastra a matrice rivestita membrana basale a venire a temperatura ambiente, aspirare la matrice membrana basale e versare 100 ml E8 supplementato con 10 micron Y-27632 in ogni pozzetto.
- Inserire un microscopio invertito in una cappa di sicurezza biologica, e inumidire con il 70% EtOH per sterilizzare.
- Estrarre una pipetta Pasteur di vetro su un becco Bunsen per ottenere uno strumento colonia picking ideale che ha un piccolo "gancio" sulla punta. Sterilizzare con il 70% EtOH, e mettere nella cappa ad asciugare.
- Rimuovere il piatto contenente colonie IPSC mirati dal termostato, e collocarle nel cofano sotto il microscopio.
- Scegli iPSCs tracciando un cerchio attorno al confine colonia con lo strumento della colonia di raccolta.
- Utilizzare il "gancio" dell'utensile colonia-picking per raschiare delicatamente e rimuovere la colonia COPSI dalla superficie della piastra. Per le colonie più grandi, disegnando una X con lo strumento della colonia di raccolta al quarto la colonia faciliterà la crescita delle cellule dopo la ri-placcatura.
- Una volta grumi di cellule si staccano e liberamente fluttuante, usare una pipetta P200 impostato a ~ 30 microlitri per raccogliere i iPSCs. Cellule piastra direttamente nella piastra a 96 pozzetti.
- Optional: usare una pipetta P20 impostato ~ 5 ml di raccogliere i iPSCs in un tubo eppendorf, quindi aggiungere 50 ml delicato cellule dissociazione reagente da digerire per 5 minuti a 37 ° C. Dopo la digestione, aggiungere 500 microlitri E8 medie nel tubo eppendorf per diluire la delicata cellule dissociazione reagente e centrifugare le cellule a 200 xg in una microcentrifuga da tavolo standard per 5 min. Poi Aspirare la maggior parte dei media e lasciare ~ 30 microlitri di riattivare e trasferire le cellule nella piastra a 96 pozzetti.
NOTA: questoapproccio faciliterà la dissociazione del gruppo di cellule e rapida espansione della colonia raccolti. - Continuare colonia prelievo prima di aver raccolto un numero sufficiente di colonie IPSC (circa 20-30 colonie per esperimento è raccomandato).
- Dopo colonia picking, posizionare la piastra a 96 pozzetti in un incubatore di notte. Refresh con terreno completo E8 il giorno seguente, e la cultura fino ~ 70% confluenti.
- Cellule passaggio dal 96 pozzetti in un 24-ben, poi in un 6-bene, come descritto nei passaggi 4,5-4,10.
NOTA: i volumi di soluzione EDTA e E8 medie dovrebbero essere proporzionalmente ridotti usando dei pozzi più piccoli di un 6-pozzetti. - Valutare il targeting successo giunzione PCR e Southern blot per confermare l'integrazione cassette mirata e l'assenza di vettori inseriti in modo casuale 16.
Risultati
Una visualizzazione del protocollo è fornita in figura 2, con i periodi durante i quali iPSCs sono coltivate in mezzo diverso evidenziata da una verde per E8 o blu per NutriStem. E 'importante per trasfettare solo iPSCs di alta qualità; esaminare piatti della cultura in tutta la manutenzione ordinaria e verificare che le culture IPSC contengono colonie principalmente distinti recanti una morfologia ciottoli-simile (Figura 3A); cellule differenziate non dovrebbe occupare più del 10% della coltura. Transfectability di iPSCs viene valutata e ottimizzata utilizzando il piccolo vettore PMAX-GFP (Figura 4A-B), come trasfezione con PMAX-GFP rappresenta in genere la massima efficienza ottenibile grazie alle sue piccole dimensioni. Ad esempio, abbiamo raggiunto il 68,6% di efficienza trasfezione con PMAX-GFP (Figura 4B). Per indirizzare il AAVS1 porto sicuro, iPSCs sono diversi passaggi con un dolce reattivo dissociazione singola cellula e sono trasfettate con AAVS1-Talens e AAVS1-CAG-EGFP (plasmidi illustrati nella Figura 1A). IPSCs transfettati vengono poi placcati su una densità adeguata di DR4 MEF (Figura 3B). Espressione transitoria dei picchi AAVS1-CAG-EGFP a 48 a 72 ore dopo la trasfezione (Figura 3C); se desiderato, una piccola porzione di iPSCs trasfettate può essere analizzata FACS. NCRM5 iPSCs può essere modificate con efficienza del 60,9% utilizzando AAVS1-CAG-EGFP donatore (Figura 4C). Efficienza di trasfezione può variare notevolmente; per gli esperimenti in cui la GFP + frazione è ancora a partire da 10%, continua a selezione puromicina. Dopo aver eseguito la selezione puromicina, singoli cloni che mostrano fluorescenza uniforme dovrebbe essere abbastanza grande per colonia raccolta (Figura 3D). Per scegliere cloni, una colonia adatto si trova sotto basso ingrandimento (Figura 3E) ed è isolato dal primo tracciato e squartando esso (Figure 3F-G). Le colonie vengono poi squartato raschiare delicatamented dal recipiente di coltura (Figura 3H). Usando una pipetta p200, grumi di cellule vengono poi trasferiti in un pozzetto di una piastra a 96 pozzetti rivestite con matrice membrana basale. iPSCs dovrebbero collegare entro 2-4 ore di placcatura (Figura 3I). Se allegato non si osserva entro 24 ore, matrice di membrana basale-coating o Y-27632 trattamento sono stati probabilmente non ottimale; Membrana basale matrix-coat una nuova piastra a 96 pozzetti e preparare fresco E8 + 10 micron Y-27632 prima di prendere qualsiasi più colonie. Una volta in un formato a 96 pozzetti, mirati cloni IPSC sono ampliati e dovrebbero mostrare espressione stabile e uniforme di GFP (Figure 4D-E).

Figura 1: Gene targeting per vettori e il targeting schematico. (A) Rappresentanze della AAVS1-CAG-EGFP e plasmidi PZT-AAVS1-L1 / R1 Talens utilizzati in questo studio. Il SA-Elemento 2A nel plasmide AAVS1-CAG-EGFP rappresenta il splice-accettore e 2A auto-scissione peptide utilizzato per limitare puromicina N-acetil-transferasi (PAC) espressione genica di eventi mirati di integrazione. La globina pollo β-actina (CAG) promotore è usato per guidare l'espressione di EGFP. (B) Il AAVS1 porto sicuro, contenuto all'interno Intron 1 del gene PPP1R12C, si rivolge utilizzando Talens per generare un doppio filamento rottura del DNA. Questo attiva il ricombinazione omologa (HR) macchine di riparazione, che utilizza le AAVS1-CAG-EGFP donatore (recanti le braccia di omologia che fiancheggiano il sito taglio) come substrato per la riparazione. La cassetta è integrato e il gene PAC è posto sotto il controllo del promotore PPP1R12C endogena.
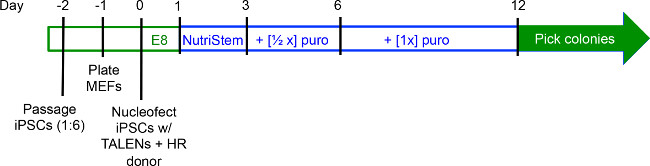
Figura 2. Timeline di gene targeting esperimento. IPSCs sono coltivateal 70% di confluenza e diversi passaggi ~ 1: 6 giorno -2 (d-2). MEF sono placcati in d-1, e iPSCs sono raccolti e trasfettate in d0. A d1, supporto viene commutato NutriStem e iPSCs sono coltivate per altri due giorni prima puromicina viene aggiunto al mezzo a d3. Le colonie sono in genere pronti per la raccolta da d12. Periodi nei quali iPSCs sono coltivate in E8 medio sono evidenziati in verde, mentre i periodi di cultura NutriStem sono in blu.

Figura 3: IPSC gene targeting immagini rappresentative (A) immagine Fase di iPSCs di alta qualità.. Notare il confine fase luminosa e ciottoli come la morfologia. (B) DR4 MEF placcato a 2 x 10 4 cellule / cm 2 24 ore dopo lo scongelamento. (C) Settantadue ore dopo la trasfezione, + cellule EGFP sono chiaramente se visto sotto un micr fluorescenzaoscope. (D) Dopo la selezione basata puromicina-per ~ 14 giorni colonie visualizzazione uniforme GFP fluorescenza sono abbastanza grandi per essere raccolti. (EH) Immagini rappresentative del processo di raccolta colonia. Le colonie vengono prelevati con una pipetta Pasteur tirato, prima delineando la colonia, e quindi del metodo dei quarti è di ottenere grumi di cellule più piccole. (I) raccolse colonie IPSC attaccano alla superficie della matrice rivestita membrana basale della piastra a 96 pozzetti entro 2 a 4 ore. Cliccate qui per vedere una versione più grande di questa figura.

Figura 4: analisi FACS di efficienza di trasfezione e iPSCs mirati clonali. (A) controllo NCRM5 iPSCs senza trasfezione di qualsiasi plasmide. (B) NCRM5iPSCs trasfettate con il test piccolo vettore PMAX-GFP sono FACS analizzati per l'espressione GFP. (C) espressione transiente del plasmide AAVS1-CAG-EGFP è valutato da FACS. (D) Un NCRM5-AAVS1-CAG-EGFP clone espressione viene visualizzata EGFP uniformemente positiva analizzati da FACS. Si noti la stretta raggruppamento di iPSCs analizzati rispetto a C). (E) fluorescenza immagine di un NCRM5-AAVS1-CAG-EGFP clone mirati espanso.
Discussione
Le fasi più critiche per il successo generazione di AAVS1 esente mirati iPSCs umani sono: (1) efficiente di Talen e donatori plasmidi in iPSCs by trasfezione; (2) ottimizzare dissociazione di iPSCs nelle singole cellule prima trasfezione e placcatura densità dopo trasfezione; (3) ottimizzare dose ed il tempo di farmaco-selezione basata sulla crescita della linea COPSI; (4) dissezionare con cura e raccolta colonie mirate e trasferimento alla nuova piastra / bene. Rispetto ai metodi simili impiegati per la carta di Hockemeyer 10, questo protocollo usato un paio di open-source AAVS1-Talens, diversi reagenti dissociazione IPSC, e dispositivo diverso trasfezione per fornire Talens e donatori vettori, che ha contribuito a ridurre il numero di iPSCs utilizzato nel esperimento, ottenendo alta transfezione e il targeting efficienza.
Per ottenere un'elevata efficienza editing gene in iPSCs umani, è essenziale ottimizzare prima della consegna del gene edireagenti Ting (DNA / RNA), mentre il bilanciamento della morte cellulare acuta il metodo di consegna e reagenti provocano al iPSCs. L'obiettivo è di massimizzare l'efficienza di consegna, pur mantenendo una discretamente basso livello di morte cellulare. Dal momento che è molto facile da preparare grandi quantità di plasmidi AAVS1-TALEN che possono raggiungere> 50% dell'efficienza HR con l'aiuto di farmaco-selezione iPSCs umani, non è necessario fare mRNA da plasmidi Talen. La sfida di consegna proviene dai grandi plasmidi donatori, che in questo caso è ~ 10 kb. Si consiglia di usare il donatore AAVS1-CAG-EGFP per testare l'efficienza di consegna in specifiche linee IPSC umani, piuttosto che utilizzare pMaxGFP incluso nel kit transfezione, perché pMaxGFP è un ~ 3.5 kb piccolo plasmidi e molto facile da trasportare in tutte le cellule. AAVS1-CAG-EGFP donatore può essere modificato con enzimi di restrizione illustrati nella Figura 1A per indirizzare diversi transgeni in AAVS1 locus. Un'altra donatore 12 kb, che contiene una cassetta 6,4 kb per sostituire CAG-EGFP, ha essereen utilizzato con successo per ottenere trasfezione simili e rivolti efficienza (dati non mostrati). In generale, iPSCs umani sono tra i tipi di cellule difficili da trasfezione e l'efficienza di consegna per grandi plasmidi possono essere partire da 5-10% misurata mediante analisi citofluorimetrica delle cellule GFP +. Ulteriore ottimizzazione dell'efficienza di transfezione dipende il corretto dissociazione iPSCs umani in singole cellule, perché grumi di cellule riducono la possibilità di fornire reagenti editing gene in ogni cella. Questo protocollo utilizza un reagente dolce e dissociazione delle cellule in rapida lavoro, ma trattando iPSCs per più di 10 minuti non è raccomandato. Di solito la cultura IPSC è inferiore all'80% confluenti se passo successivo 5.1, facendo un 5 minuti di incubazione con pre-riscaldato dolce-cellule dissociazione reagente genere sufficiente dissociare le iPSCs in celle singole con gentile pipettaggio. Se non tutte le cellule possono essere dissociate in singole cellule dopo 10 min di trattamento perché coltura cellulare è over-confluente o le colonie diventano molto compatta, semplicemente raccogliere il numero consigliato di singole cellule da più pozzetti / piastre e lasciare le cellule saldamente attaccati alle spalle. Pipettaggio pesante non è raccomandato perché tosatura meccanica è più dannoso per le cellule di dissociazione enzimatica. Quando si lavora con iPSCs precoce passaggio (prima del passaggio # 15), pratica la dissociazione dolce è molto importante per la sopravvivenza delle cellule perché le cellule sono già più sensibili allo stress trasfezione di iPSCs fine del passaggio. Se è difficile ottenere un'elevata efficienza sia trasfezione e cella di sopravvivenza, scegliere la pratica che dà massima efficienza di trasfezione. Dopo la trasfezione, le cellule devono essere placcato alla densità che raggiungerà il 50-80% di confluenza al giorno 3 quando la droga-selezione inizia. Dal momento che ogni riga iPSC cresce in modo diverso, la densità di placcatura post-trasfezione potrebbe essere necessario ottimizzare per ciascuna linea cellulare. Una alta densità di placcatura può portare a confluenza eccessiva che riduce l'efficacia di drug-selezione, mentre un a bassa densità di placcatura estremo può causare ripresa ritardata e la crescita di colonie resistenti ai farmaci. Di solito 3 milioni iPSCs partenza sono sufficienti per essere placcato in ½ a 2 10 centimetri piatti a seconda della sopravvivenza e la crescita di iPSCs specifici. Allo stesso modo, se si utilizza una linea iPSC appena generata o non ancora testato, generando una curva uccidere puromicina per stabilire la dose minima efficace in cellule untransfected eventualmente necessarie; linee IPSC possono variare notevolmente nella loro sensibilità alla selezione puromicina. Il MEF sono stati utilizzati per la selezione perché sembrano sostenere la crescita iPSC meglio di alcune matrici extracellulari durante la selezione della droga. Come regola generale, un minimo di 5 giorni di puromicina a giorni 1 mg / ml o 7 a 0.5 mg / ml sono necessari per completare la selezione. Infine, tecnica-colonia raccolta è fondamentale per conservare tutte le colonie target dopo la selezione della droga. Pipettaggio Gentle o trattamento reattivo dissociazione aiuta a rompere le colonie in più parti in modo uniformedistribuito nel nuovo pozzo, e quindi può accelerare espansione colonia. Se si utilizza un reagente dissociazione di disaggregare le colonie raccolte prima della placcatura, assicurarsi che sia sufficientemente diluito con> 10x medio dopo il trattamento, perché il reagente di dissociazione residuo potrebbe uccidere le cellule trasferite se lasciato acceso durante la notte. Non importa quale sia la tecnica utilizzata, la pratica colonia raccogliendo prima degli esperimenti reali è fortemente incoraggiata.
Come la strategia descritta utilizza un metodo gene-trap per guidare l'espressione del gene PAC fuori del promotore PPP1R2C endogena, raccogliendo significativamente più di 30 colonie non dovrebbero risultare necessario. La SA-2A legata selezione PAC elimina teoricamente cellule solo di integrazione-casuali, ma l'integrazione casuale aggiuntivo (s) può verificarsi in iPSCs recanti integrazioni mirate successo. Nella maggior parte dei casi, quasi il 100% dei cloni-droga selezionati hanno preso di mira le integrazioni e ~ 10-40% di loro hanno aggiuntivo integrati casualeons. La maggior parte dei cloni mirate tendono ad avere il targeting singolo allele, anche se gli esperimenti in cui> 50% di targeting doppio allele si verifica. La frequenza variabile di ulteriori integrazioni casuali, nonché il rapporto di singolo doppio rispetto allele targeting, è difficile da controllare. Pertanto, raccogliendo ~ 20 colonie si raccomanda di assicurare una o due allele di targeting solo cloni possono essere ottenuti. Alla luce di esperimenti falliti di targeting, ri-valutazione dei tassi di transfectability, di sopravvivenza, e la crescita delle cellule bersaglio ', l'efficacia della nucleasi nella linea cellulare specifica, e la qualità dei preparati donatori e nucleasi è altamente raccomandato prima di ri-tentare l'esperimento nella sua interezza.
Va notato che le strategie donatori gene-trap simili possono essere utilizzati per il targeting altri geni attivi, ma non è adatto per il targeting gene silenziosa che richiede un promotore indipendente per guidare l'espressione del gene selezionabile. Inoltre, mentre the AAVS1 Safe Harbor è considerato di avere la struttura della cromatina aperta, questo non garantisce che qualsiasi transgene può essere espresso con forza a questo locus. In effetti, il nostro recente rapporto ha mostrato che diversi promotori deboli sono stati in grado di guidare l'espressione del gene reporter fluorescente rilevabile dopo l'integrazione mirata a AAVS1 locus 14.
Questo protocollo è incentrato sull'utilizzo Talens ben convalidati, per indirizzare un ben studiato locus porto sicuro nel genoma umano. Le tecniche sono altamente riproducibili e facilmente adattabile a molte applicazioni che implicano un transgene. Rispetto ai metodi di ingegneria del genoma utilizzando integrazioni casuali, AAVS1-TALEN gene mediato rivolti è altamente efficace e specifico, e nel caso di iPSCs, risultati in cellule stabilmente fluorescenti che hanno il potenziale di espandersi e differenziarsi in qualsiasi tipo di cellula umana. Anche se questo protocollo non descrive la progettazione e la validazione di nucleasi di design o la valutazione di colonie IPSC mirati per pintegrazione cassette Roper e analisi off-bersaglio, diversi protocolli eccellenti hanno descritto questi aspetti della metodologia in dettaglio 16-18. I IPSC cultura e Passaging tecniche senza alimentatore utilizzando E8 media sono anche descritte in dettaglio nella pubblicazione precedente 19. Il protocollo di cui sopra, mentre ottimizzato per l'aggiunta del gene nel AAVS1 di sicurezza di iPSCs umani, può servire come modello generale per gli esperimenti che coinvolgono site-specific nucleasi-mediata gene editing / aggiunta utilizzando una ricombinazione omologa donatore in qualsiasi tipo di cellula.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari in competizione.
Riconoscimenti
This research was supported by the NIH Common Fund and Intramural Research Program of the National Institute of Arthritis, Musculoskeletal, and Skin Diseases.
Materiali
| Name | Company | Catalog Number | Comments |
| Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix, *LDEV-Free, 10 ml | Corning | 354230 | Store at -20 °C. |
| DMEM/F-12 | Life Technologies | 11320-033 | Store at 4 °C. |
| Costar 6 Well Clear TC-Treated Multiple Well Plates | Corning | 3506 | |
| Essential 8 Medium | Life Technologies | A1517001 | Store basal medium at 4 °C. Store supplement at -20 °C. |
| Y-27632 dihydrochloride | Tocris | 1254 | Store at room temp. Once dissolved in H2O, store at -20 °C. |
| Sodium Chloride | Sigma | S5886-500G | |
| UltraPure 0.5 M EDTA, pH 8.0 | Life Technologies | 15575-020 | |
| DPBS, no calcium, no magnesium | Life Technologies | 14190-250 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| DR4 MEF 2M IRR - Academic | GlobalStem | GSC-6204G | Store in liquid Nitrogen. |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-040 | Store at 4 °C. |
| Defined Fetal Bovine Serum, US Origin | HyClone | SH30070.03 | Store at -20 °C. Thaw at 4 °C overnight and aliquot. Store aliquots at -20 °C until needed. |
| MEM Non-Essential Amino Acids Solution (100X) | Life Technologies | 11140-050 | Store at 4 °C. |
| 4D-Nucleofector Core unit | Lonza | AAF-1001B | part of the electroporation system |
| 4D-Nucleofector X unit | Lonza | AAF-1001X | part of the electroporation system |
| P3 Primary Cell 4D-Nucleofector X Kit L (24 RCT) | Lonza | V4XP-3024 | Upon arrival, remove Primary Cell Solution and supplement and store at 4 °C. |
| StemPro Accutase Cell Dissociation Reagent | Life Technologies | A1110501 | Store at -20 °C. Thaw overnight at 4°C and warm an aliquot in a 37 °C water bath before use. |
| NutriStem XF/FF Culture Medium | Stemgent | 01-0005 | Store at -20 °C. Thaw overnight at 4 °C |
| AAVS1 TALENs (pZT-AAVS1-L1 and pZT-AAVS1-R1) | Addgene | 52637 and 52638 | |
| [header] | |||
| AAVS1-CAG-EGFP Homologous Recombination donor | Addgene | 22212 | |
| Puromycin Dihydrochloride | Life Technologies | A11138-03 | Store at -20 °C. Prepare working aliquots of 1 mg/ml in ddH2O. |
| Disposable Borosilicate Glass Pasteur Pipets | Fisher Scientific | 13-678-20A | |
| Sorvall Legend XTR (Refrigerated), 120 V 60 Hz | Thermo Scientific | 75-004-521 | |
| TX-750 4 × 750 ml Swinging Bucket Rotor | Thermo Scientific | 75003607 | |
| Trypan Blue Solution, 0.4% | Life Technologies | 15250-061 | |
Riferimenti
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Hentze, H., Graichen, R., Colman, A. Cell therapy and the safety of embryonic stem cell-derived grafts. Trends Biotechnol. 25 (1), 24-32 (2007).
- Smith, J. R., et al. Robust, persistent transgene expression in human embryonic stem cells is achieved with AAVS1-targeted integration. Stem Cells. 26 (2), 496-504 (2008).
- Lombardo, A., et al. Site-specific integration and tailoring of cassette design for sustainable gene transfer. Nat Methods. 8 (10), 861-869 (2011).
- Ogata, T., Kozuka, T., Kanda, T. Identification of an insulator in AAVS1, a preferred region for integration of adeno-associated virus DNA. J Virol. 77 (16), 9000-9007 (2003).
- Zwaka, T. P., Thomson, J. A. Homologous recombination in human embryonic stem cells. Nat Biotechnol. 21 (3), 319-321 (2003).
- Urbach, A., Schuldiner, M., Benvenisty, N. Modeling for Lesch-Nyhan disease by gene targeting in human embryonic stem cells. Stem Cells. 22 (4), 635-641 (2004).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nature biotechnology. 27 (9), 851-857 (2009).
- Zou, J., et al. Gene targeting of a disease-related gene in human induced pluripotent stem and embryonic stem cells. Cell stem cell. 5 (1), 97-110 (2009).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nature. 29 (8), 731-734 (2011).
- Sanjana, N. E., et al. A transcription activator-like effector toolbox for genome engineering. Nature protocols. 7 (1), 171-192 (2012).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Ding, Q., et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell stem cell. 12 (4), 393-394 (2013).
- Luo, Y., et al. Stable Enhanced Green Fluorescent Protein Expression After Differentiation and Transplantation of Reporter Human. Induced Pluripotent Stem Cells Generated by AAVS1 Transcription Activator-Like Effector Nucleases. Stem Cells Transl Med. 3 (7), 821-835 (2014).
- Zou, J., et al. Oxidase-deficient neutrophils from X-linked chronic granulomatous disease iPS cells: functional correction by zinc finger nuclease-mediated safe harbor targeting. Blood. 117 (21), 5561-5572 (2011).
- Luo, Y., Rao, M., Zou, J. Generation of GFP Reporter Human Induced Pluripotent Stem Cells Using AAVS1 Safe Harbor Transcription Activator-Like Effector Nuclease. Curr Protoc Stem Cell Biol. 29, 5A.7.1-5A.7.18 (2014).
- Peters, D. T., Cowan, C. A., Musunuru, K. Genome editing in human pluripotent stem cells. StemBook. , Harvard Stem Cell Institute. Cambridge, MA. Available from: http://www.stembook.org/node/1438 (2008-2013).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Beers, J., et al. Passaging and colony expansion of human pluripotent stem cells by enzyme-free dissociation in chemically defined culture conditions. Nat Protoc. 7, 2029-2040 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon