Method Article
La transfección, selección y Colonia-cosecha de células madre humanas pluripotentes inducidas TALEN dirigidos con un gen GFP en el puerto seguro AAVS1
En este artículo
Resumen
TALEN-mediated gene editing at the safe harbor AAVS1 locus enables high-efficiency transgene addition in human iPSCs. This protocol describes the procedures for preparing iPSCs for TALEN and donor vector delivery, transfecting iPSCs, and selecting and isolating iPSC clones to achieve targeted integration of a GFP gene to generate reporter lines.
Resumen
Targeted transgene addition can provide persistent gene expression while circumventing the gene silencing and insertional mutagenesis caused by viral vector mediated random integration. This protocol describes a universal and efficient transgene targeted addition platform in human iPSCs based on utilization of validated open-source TALENs and a gene-trap-like donor to deliver transgenes into a safe harbor locus. Importantly, effective gene editing is rate-limited by the delivery efficiency of gene editing vectors. Therefore, this protocol first focuses on preparation of iPSCs for transfection to achieve high nuclear delivery efficiency. When iPSCs are dissociated into single cells using a gentle-cell dissociation reagent and transfected using an optimized program, >50% cells can be induced to take up the large gene editing vectors. Because the AAVS1 locus is located in the intron of an active gene (PPP1R12C), a splicing acceptor (SA)-linked puromycin resistant gene (PAC) was used to select targeted iPSCs while excluding random integration-only and untransfected cells. This strategy greatly increases the chance of obtaining targeted clones, and can be used in other active gene targeting experiments as well. Two weeks after puromycin selection at the dose adjusted for the specific iPSC line, clones are ready to be picked by manual dissection of large, isolated colonies into smaller pieces that are transferred to fresh medium in a smaller well for further expansion and genetic and functional screening. One can follow this protocol to readily obtain multiple GFP reporter iPSC lines that are useful for in vivo and in vitro imaging and cell isolation.
Introducción
La capacidad de reprogramar células somáticas humanas en células madre pluripotentes inducidas similares a células madre embrionarias (células iPS) fue descubierto por primera vez por Takahashi et al. En 2007 1. Fibroblastos dérmicos humanos transducidas con retrovirus que expresa cuatro factores de transcripción (El Yamanaka así apodado-Factores Oct3 / 4, Sox2, se muestra c-Myc, y Klf4) para ser muy similares a las células madre embrionarias humanas (hESCs) basándose en la morfología, proliferación, expresión génica, y el estado epigenética; crucialmente, iPSCs también son capaces de diferenciarse en células de las tres capas germinales 1. La capacidad potencial y la diferenciación de proliferación de células iPS los hace herramientas muy atractivos; mediante la reprogramación de células de pacientes que sufren de enfermedades específicas, iPSCs se pueden utilizar tanto como sistemas modelo in vitro de la enfermedad y como agentes terapéuticos potenciales.
Para este último propósito, varios asuntos deben ser abordados antes de que el pleno potencial de iPSCsen un entorno clínico puede realizarse; el potencial tumorigénico de hESCs cultivadas in vitro y CMPI, el uso de derivados xenogénicos durante la reprogramación celular y el mantenimiento, y la necesidad de realizar un seguimiento de las células trasplantadas in vivo son todos los obstáculos cruciales para la aplicación clínica de células madre pluripotentes (Revisado por Hentze et al. 2). Una solución ideal a la necesidad para el seguimiento de las células diferenciadas post-trasplante implicaría un marcador visualmente detectable que se resiste a silenciar y variegación independientemente de la aplicación. Expresión robusta y sostenida de transgenes integrados es más fácilmente alcanzable cuando se introduce ADN exógeno en loci puerto seguro; es decir, sitios genómicos que permiten la transcripción suficiente de un vector integrado, mientras que al mismo tiempo mitigar perturbaciones de expresión en genes vecinos 3. Uno de esos sitios que ha sido muy bien caracterizado desde su descubrimiento es la integr virus adeno-asociadoEl sitio ación 1 (AAVS1), en el primer intrón de la 1 12C subunidad reguladora de genes (PPP1R12C) la proteína fosfatasa. Este locus ha sido demostrado no sólo para permitir la expresión robusta de transgenes integrados a través del tiempo extendido en la cultura y en la diferenciación in vitro sostenido y 3, sino también para proteger rodea genes de perturbación transcripcional 4; ambas características se cree que es debido a la presencia de elementos aislantes de la cromatina endógenos que flanquean el sitio AAVS1 5.
Los avances en las herramientas de ingeniería del genoma más sólo la última década han facilitado en gran medida la facilidad y eficiencia con que las manipulaciones genéticas en cualquier tipo de célula se puede lograr. Mientras que los experimentos exitosos primeros basaron en extremadamente bajos niveles de recombinación homóloga endógena (HR) con un donante introducido para lograr la orientación de genes en los CES 6,7, el uso de nucleasas específicas de sitio, tales como nucleasas de dedos de zinc (ZFNs), que signinificativamente inducir la recombinación homóloga a través de la generación de una rotura del DNA de doble cadena ha aumentado en gran medida la eficiencia de tales experimentos 8,9. La reutilización de ambos tipo activador de transcripción efectores (cuentos) de planta patógeno Xanthomonas géneros y los agrupados interspaced regularmente repeticiones palindrómicas cortas procariotas (CRISPR) / sistema Cas9 en nucleasas de diseño específicos del sitio eficientes ha hecho la orientación de genes en células madre pluripotentes un accesible y metodología practicable 10-13.
Un reciente documento describe un método eficaz para la integración estable de un reportero de cassette verde fluorescente en el AAVS1 locus puerto seguro en iPS humanas utilizando nucleasas TALE (Talens) 14. Estos iPSCs dirigidos mantuvieron su fluorescencia incluso después de la diferenciación dirigida a cardiomiocitos y trasplante en un modelo de ratón de infarto de miocardio (MI), proporcionando una fuerte evidencia de la utilidad de talesestablemente células madre pluripotentes fluorescente 14. Para obtener colonias específicas, se utilizó un método para atrapar genes en el que un corte y empalme-aceptor (SA), secuencia de péptido autoescindible 2A coloca el puromicina N-acetil-transferasa gen (PAC) bajo el control del promotor endógeno PPP1R12C; Por lo tanto, sólo iPSCs que han incorporado el donante de ADN en el locus AAVS1 expresan PAC, haciéndolos seleccionable basado en puromicina-resistencia; (Figura 1, 15). Este protocolo detalla los procedimientos de generación de AAVS1-GFP iPSCs informó en el documento reciente 14, incluido el proceso de transfección de células iPS con Talens y un donante de 9,8 kb para integrar un fragmento de ADN de 4,2 kb en el locus AAVS1 puerto seguro, la selección de iPSCs basados en puromicina-resistencia, y las colonias de la cosecha por la expansión clonal. Las técnicas descritas en el presente documento pueden aplicarse a muchos experimentos de ingeniería del genoma.
Protocolo
1. Preparación del Sótano matriz de la membrana y de revestimiento de Material de plástico
- Coloque la matriz de la membrana basal madre congelada de -20 ° C en hielo y descongelar la noche a 4 ° C.
- Después de la descongelación, la pipeta 2 mg alícuotas de matriz de membrana basal en tubos Eppendorf pre-refrigerados. Almacenar estos en -20 ° C hasta que se necesite.
- Para preparar las placas de matriz recubierto de la membrana basal, descongelar una alícuota sobre hielo hasta el último trozo de hielo en la que desaparezca tubo Eppendorf (normalmente dentro de ~ 2 h).
- Después de la descongelación, añadir matriz de membrana basal a 12 ml de frío (4 ° C) DMEM / F12 para obtener una solución de revestimiento de matriz de la membrana basal.
- Añadir solución de matriz de membrana basal al recipiente de cultivo apropiado. Para una placa de 6 pocillos, prescindir de 1 ml por pocillo. Agite la placa para asegurarse de que la solución de la matriz de la membrana basal cubre completamente cada pocillo.
- Parafilm sellar la membrana basal de la matriz recubierta de placa / placa, y se incuba a Room temperatura durante 1 hora antes de su uso. Alternativamente, las placas de matriz recubiertos tienda membrana basal / platos a 4 ° C y utilizar dentro de 2 semanas de recubrimiento.
NOTA: Añadir DMEM extra / F12 a la membrana basal de la matriz recubierta de placa / placa para evitar el secado. Antes de usar 4 ° C membrana basal almacenado matriz recubiertos plato / plato, colóquelo en una cabina de seguridad biológica y deje que llegue a temperatura ambiente durante al menos 30 minutos. - Aspirar matriz de membrana basal completamente antes de la adición de células y medio.
2. Preparación del medio E8
- Preparar medio de cultivo E8 descongelando suplemento E8 la noche a 4 ° C.
- Extraiga 10 ml de medio basal E8 de la población de 500 ml y desechar.
- Pipetear todo el vial de 10 ml de suplemento E8 directamente en 490 ml de medio basal E8. No entibie medio completo E8 en un baño de agua a 37 ° C, como las fluctuaciones de temperatura repetidos pueden degradar el bFGF en el complemedio te E8.
- Utilice un medio completo E8 dentro de 2 semanas de la adición del suplemento.
3. La descongelación de iPSCs
- Retirar un vial de iPSCs congelados de nitrógeno líquido y colocar en hielo seco.
- Rápidamente descongelar el vial en un baño de agua a 37 ° C; agitar el vial en el baño de agua hasta que sólo un pequeño fragmento de los restos de hielo.
- Rocíe el vial con etanol y la transferencia del 70% de una cabina de seguridad biológica.
- Añadir 1 ml de la temperatura ambiente E8 medio gota a gota directamente al vial.
- Usando una pipeta de 2 ml, transferir la suspensión celular en gota a gota 9 ml de medio de E8 en un tubo cónico de 15 ml. Agitar el tubo de frecuencia para asegurarse de que las células y el medio se mezclan bien rápidamente.
- Centrifugar las células a 200 xg durante 5 min.
- Aspirar el sobrenadante y resuspender el sedimento celular en un volumen apropiado de E8 suplementado con 10 mM Y-27632.
- Añadir las células a un número adecuado de sótano membrane matriz recubiertos pozos, y colóquelo en un 37 ° C, 5% de CO2 durante la noche. Se recomienda a la placa al menos 0,2 x 10 6 IPSC por pocillo de una placa de 6 pocillos para permitir una rápida recuperación después de la descongelación.
4. Mantenimiento y pases de rutina de iPSCs
- Actualizar medio E8 diaria.
- Monitorear la morfología y la confluencia de las células con un microscopio invertido. iPSCs de alta calidad crecen en colonias planas con bordes bien definidos; colonias individuales poseen una apariencia "empedrado".
- Passage las células iPSCs cuando llegan a ~ 70% de confluencia.
- Preparar una solución pases EDTA mediante la adición de 0,9 g de NaCl y 500 l 0,5 M EDTA a 500 ml de DPBS. Mezclar bien para disolver NaCl, y el filtro de vacío para esterilizar. Calentar una parte alícuota de la solución de los pases en un 37 ° C baño de agua antes de pases.
- Para pasaje, aspirado pasó medio de cultivo y lavar las células una vez con un volumen igual de pas calientessaging solución. Aspirar, y pipetear suficiente de solución EDTA pases para recubrir las células (1 ml por pocillo de una placa de 6 pocillos).
- Coloque las células bajo un microscopio invertido y observar las colonias de IPSC. La aparición de agujeros dentro de las colonias y bordes elevados debe ser evidentes dentro de 2 a 5 min.
- Aspirar con cuidado la solución passaging EDTA.
- Usando una pipeta de 10 ml, dispensar 4 ml de medio E8 (si se utiliza una placa de 6 pocillos) a alta presión directamente en cada pocillo para ser pasado.
- Recoge los grupos IPSC, y dividido en un número apropiado de pocillos en función de la relación de división de 1: 8 a 1:12. No sobre-pipeta, como desagregación de los grupos de células que puede reducirse el viabilidad.
- Colocar la placa en una incubadora, y el rock de la placa posterior y hacia adelante y de lado a lado varias veces para dispersar las células.
5. Preparación de MEFs y iPSCs para Tansfection
- 48 horas antes de la transfection, iPSCs paso a ~ 1: 6 en cuatro o más pocillos de una placa de 6 pocillos de la membrana basal de la matriz recubiertos, por lo que serán 70% confluentes aquí a dos días.
- El día siguiente, se descongele DR4 MEFs en el medio de MEF que consiste en DMEM (alta glucosa) suplementado con 10% de FBS y 1x MEM-NEAA.
- Placa DR4 MEFs en dos placas de 10 cm a ~ 2 x 10 4 células / cm 2 y se incuba durante la noche a 37 ° C.
- En el día de la transfección, cambio de medio MEF a E8 suplementado con 10 mM Y-27632 30 min antes de realizar la transfección en células iPS.
- Opcional: Si se desea análisis de citometría de flujo de iPSCs después de la transfección, quitar una placa de matriz recubiertos con membrana basal de 4 ° C y el lugar a temperatura ambiente.
- Opcional: 4 h antes de la transfección, suplemento pre-transfección cultura IPSC con Y-27632 en la concentración final de 10 mM.
6.-La disociación celular Gentle reactivo de tratamiento unand La transfección de células iPS usando un sistema de electroporación
- Retire la solución de transfección de células primarias P3 de 4 ° C y deje que llegue a temperatura ambiente durante ~ 30 min. Añadir todo el suplemento de 100 l a la solución de transfección antes de su uso.
- Calentamiento celular suave reactivo de disociación en un baño a 37 ° C.
- Obtener AAVS1 Talens (pZT-AAVS1-L1 y pZT-AAVS1-R1) y AAVS1-CAG-EGFP donante de -20 ° C.
- Retire iPSCs del incubador y lavar una vez con DPBS.
- Añadir 1 ml de reactivo de disociación de células suave por pocillo, y se incuba iPSCs a 37 ° C durante 5 min, o hasta mayor que 50% de las células se han disociado del recipiente de cultivo.
- Pipetear las células arriba y abajo un par de veces con una pipeta p1000 disociar las células restantes del recipiente de cultivo, y para romper los grumos IPSC.
- Añadir 2 ml de medio E8 a cada pocillo, y pipeta hacia arriba y abajo varias veces con una pipeta de 10 ml a Fürther deshacer los agregados celulares en células individuales
NOTA: La eficacia de transfección disminuye significativamente si los agregados celulares no son suficientemente desagregada. - Recoger iPSCs en un tubo cónico de 15 ml y centrifugar a 100 xg durante 3 min.
- Aspirar el sobrenadante y volver a suspender las células en una cantidad mínima de medio de E8.
- Contar las células utilizando un hemocitómetro después de la aplicación de un colorante vital, tales como 0,4% de azul de tripano. Asegúrese de que las células están suficientemente disociados mientras cuenta (1-3 células por "grupo").
- Prescindir de 3 x 10 6 células en cada uno de dos tubos de 15 ml cónicos, y girar de nuevo a 100 g durante 3 min.
NOTA: centrifugación a baja velocidad reduce el estrés celular y permite un fácil resuspensión de iPSCs antes de la electroporación. - Ajuste el sistema de electroporación para el programa específico de tipo celular de la línea de células madre embrionarias humanas H9 (Programa CB-150).
- Después de la centrifugación, aspirar el sobrenadante de la cell pellets. Para una pastilla, añadir 10 g de la donante de recursos humanos como muestra de control. Para el otro pellet añadir 10 g de la donante HR, junto con 5 g de cada uno TALEN (PZT-AAVS1-L1 y PZT-AAVS1-R1) como muestra experimental.
- Resuspender cada sedimento celular en 100 l de solución de transfección de células primarias P3, y transferir a una cubeta.
- Realice la transfección y añadir inmediatamente 500 l de medio E8 temperatura ambiente a cada cubeta.
- Transferir el iPSCs gota a gota transfectadas a uno de 10 cm de plato que contiene MEFs DR4 preparados en la etapa 5.4.
- Opcional: Añadir varias gotas de cada experimento en un pocillo de una placa de 6 pocillos de la membrana basal de la matriz recubierto si se desea citometría de flujo anaylsis de la eficiencia de la transfección.
- Repita los pasos 06.15 a 06.17 para terminar ambas muestras. El día siguiente, lavar las células con DPBS dos veces y cambiar el medio de cultivo a NutriStem, que parece apoyar la cultura IPSC en alimentadores mejor que E8.
- Trpicos de expresión EGFP ansient en de 48 a 72 horas después de la transfección; evaluar la eficiencia de transfección como se desee.
7. Puromicina Selección de iPSCs Targeted
- Comience selección puromicina 72 horas después de la transfección cuando las iPSCs alcanzan el 70% de confluencia.
- Para NCRM5 iPSCs, comenzará la selección puromicina a 0,5 mg / ml de puromicina (1/2 de la dosis completa) diluido en medio de cultivo NutriStem. La concentración óptima puromicina puede variar desde 0,25 hasta 1 mg / mIL para algunas líneas de IPSC.
- IPSCs Cultura en NutriStem + 0,5 mg / ml de puromicina durante 3 días, refrescantes del medio día.
- Después de 3 días, aumentar la concentración de puromicina a 1 mg / ml.
- IPSCs Cultura menores de 1 mg / ml puromycin selección durante otros 7 a 8 días, o hasta que las colonias distintas parecen lo suficientemente grande para picking colonia.
8. Colonia Picking y ampliación de iPS específicas
- Uso de la membrana basalmatriz para recubrir una placa de 96 pocillos mediante la supresión de 50 l de solución de revestimiento de matriz de la membrana basal (2 mg matriz de membrana basal diluido en 12 ml de DMEM / F12) en cada pocillo. Guarde la placa a 4 ° C durante la noche antes de su uso.
- Después de permitir que la placa de matriz recubiertos con membrana basal para llegar a la temperatura ambiente, aspirar la matriz de la membrana basal y dispensar 100 l E8 suplementado con 10 mM Y-27632 en cada pocillo.
- Publicar un microscopio invertido en una cabina de seguridad biológica, y rociar ligeramente con EtOH al 70% para esterilizar.
- Saque una pipeta Pasteur de vidrio sobre un mechero Bunsen para obtener una herramienta de colonias de picking ideal que tiene un "gancho" minúscula en la punta. Esterilizar con EtOH al 70%, y el lugar en la campana para secar.
- Retirar la cápsula que contiene colonias IPSC dirigidos de la incubadora, y colocar en el capó bajo el microscopio.
- Escoja iPSCs trazando un círculo alrededor de la frontera colonia con la herramienta de colonias de picking.
- Use el "gancho" de la herramienta de colonias de picking para raspar suavemente y quitar la colonia IPSC de la superficie de la placa. Para las colonias más grandes, dibujando una X con la herramienta de colonias de recoger a trimestre la colonia facilitará el crecimiento celular después de la re-enchapado.
- Una vez que grupos de células se separan y libremente flotante, utilice una pipeta p200 ajustado a ~ 30 l para recoger las células iPS. Células de la placa directamente en la placa de 96 pocillos.
- Opcional: utilizar una pipeta p20 ajustado a ~ 5 l para recoger las células iPS en un tubo Eppendorf, a continuación, añadir 50 l de reactivo de disociación de células suave de digerir durante 5 minutos a 37 ° C. Después de la digestión, añadir 500 l medio E8 en el tubo eppendorf para diluir el reactivo de disociación de células suave y centrifugar las células a 200 xg en una microcentrífuga de sobremesa estándar para 5 min. Entonces aspirar la mayor parte del medio y dejar ~ 30 l de volver a suspender y transferir las células en la placa de 96 pocillos.
NOTA: Esteenfoque facilitará la disociación del grupo celular y la rápida expansión de la colonia recogido. - Continuar picking colonia hasta que se hayan recogido un número suficiente de colonias IPSC (más o menos se recomienda 20 a 30 colonias por experimento).
- Después de la recolección de colonias, coloque la placa de 96 pocillos en una incubadora durante la noche. Actualizar con medio completo E8 el día siguiente, y la cultura hasta ~ 70% de confluencia.
- Células pasaje de los 96 pozos en un 24 y, a continuación, en un 6 pocillos, como se describe en los pasos 04.05 a 04.10.
NOTA: los volúmenes de solución de EDTA y medio E8 deben reducirse proporcionalmente cuando se utiliza pozos más pequeños que una placa de 6 pocillos. - Evaluar la orientación éxito por la unión de PCR y análisis de transferencia de Southern para confirmar la integración casete específica y ausencia de vectores insertados al azar 16.
Resultados
Una visualización del protocolo se proporciona en la Figura 2, con períodos durante los cuales iPSCs se cultivan en medio diferente puesto de manifiesto por cualquiera de verde para E8 o azul para NutriStem. Es importante para transfectar sólo iPSCs de alta calidad; examinar placas de cultivo de todo el mantenimiento de rutina y verificar que las culturas IPSC contienen colonias principalmente distintas que llevan una morfología de adoquines como (Figura 3A); células diferenciadas no deben ocupar más de 10% del cultivo. Transfectability de iPSCs se evalúa y optimiza utilizando el vector pequeño Pmax-GFP (Figura 4A-B), como la transfección con Pmax-GFP normalmente representa la máxima eficiencia alcanzable debido a su pequeño tamaño. Por ejemplo, hemos logrado un 68,6% de eficiencia de transfección con Pmax-GFP (Figura 4B). Para orientar la AAVS1 puerto seguro, iPSCs se pasan usando un reactivo de disociación de una sola célula suave y se transfectaron con AAVS1-Talens y AAVS1-CAG-EGFP (plásmidos representados en la Figura 1A). CMPI transfectadas se sembraron en una densidad adecuada de DR4 MEFs (Figura 3B). La expresión transitoria de los picos de AAVS1-CAG-EGFP en 48 a 72 horas después de la transfección (Figura 3C); si se desea, una pequeña porción de iPSCs transfectadas se puede FACS analizó. NCRM5 iPSCs se puede transfectar con la eficiencia de 60,9% utilizando AAVS1-CAG-EGFP donante (Figura 4C). La eficacia de transfección puede variar mucho; para los experimentos en el que la GFP + fracción es incluso tan bajo como 10%, continúe a la selección de puromicina. Después de realizar la selección de puromicina, clones individuales que muestran fluorescencia uniforme deben ser lo suficientemente grande para colonia de picking (Figura 3D). Para recoger los clones, una colonia adecuada se encuentra en condiciones de baja magnificación (Figura 3E) y se aísla por primera trazado de popa o ella (Figuras 3F-G). Las colonias están acuartelados luego raspar suavemented del recipiente de cultivo (Figura 3H). Usando una pipeta de p200, grupos de células se transfieren a continuación a un pocillo de una placa de 96 pocillos recubiertos con matriz de membrana basal. iPS deben adjuntar dentro de 2-4 horas de chapado (Figura 3I). Si el accesorio no se observa en 24 horas, Sótano membrana matriz de recubrimiento o Y-27632 tratamiento probablemente subóptimo; La membrana basal de la matriz-coat una nueva placa de 96 pocillos y preparar fresco E8 + 10 mM Y-27632 antes de recoger cualquier más colonias. Una vez en un formato de 96 pocillos, dirigidos clones IPSC se expanden y se deben exhibir la expresión estable y uniforme de las buenas prácticas agrarias (Figuras 4D-E).

Figura 1: Gene vectores de abordaje y la orientación esquemática. (A) Representaciones de la AAVS1-CAG-EGFP, y plásmidos PZT-AAVS1-L1 / R1 Talens utilizados en este estudio. La SA-Elemento 2A en el plásmido AAVS1-CAG-EGFP representa la auto-escisión de péptido de empalme-aceptor y 2A utilizado para restringir la puromicina N-acetil-transferasa (PAC) la expresión de genes a los eventos de integración dirigida. La globina de pollo β-actina (CAG) promotor se utiliza para controlar la expresión de EGFP. (B) El AAVS1 puerto seguro, contenida dentro del intrón 1 del gen PPP1R12C, está dirigido usando Talens para generar una ruptura de ADN de doble cadena. Esto activa la recombinación homóloga (HR) la maquinaria de reparación, que entonces utiliza los AAVS1-CAG-EGFP donante (que llevan brazos de homología que flanquean el sitio de corte) como un sustrato para la reparación. El casete está integrada y el gen PAC se coloca bajo el control del promotor PPP1R12C endógeno.
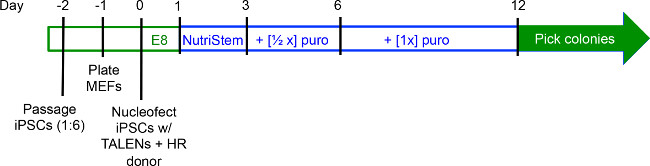
Figura 2. Cronología de gen experimento focalización. IPSCs se cultivana 70% de confluencia y se pasaron ~ 1: 6 en el día -2 (d-2). MEFs están chapados en d-1, y iPSCs se recogen y se transfectaron en d0. En d1, los medios de comunicación se cambia a NutriStem, y CMPI se cultivan durante dos días más antes de añadir la puromicina al medio a d3. Las colonias son típicamente listo para su recolección por d12. Períodos durante los cuales iPSCs se cultivan en medio E8 se resaltan en color verde, mientras que los periodos de la cultura NutriStem están en azul.

Figura 3: gen IPSC focalización imágenes representativas (A) Imagen de la Fase de iPSCs de alta calidad.. Nota de la frontera de fase brillante y adoquines morfología similar. (B) DR4 MEFs en placas a 2 x 10 4 células / cm 2 veinticuatro horas después de la descongelación. (C) Setenta y dos horas después de la transfección, las células EGFP + son claramente evidentes cuando se observa bajo un micr fluorescenciaoscope. (D) Después de la selección basada en la puromicina para ~ 14 días, las colonias que presentan fluorescencia de GFP uniforme son lo suficientemente grandes para ser recogidos. (EH) Imágenes representativas del proceso de selección de colonia. Las colonias se recogió con una pipeta Pasteur tirado, primero esbozando la colonia, y luego por cuarteo para obtener grupos de células pequeñas. (I) se llevó colonias IPSC se adhieren a la superficie de la matriz recubiertos con membrana basal de la placa de 96 pocillos dentro de 2 a 4 hr. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: análisis FACS de la eficacia de transfección y iPSCs dirigidos clonales. (A) Control NCRM5 iPS sin transfección de cualquier plásmido. (B) NCRM5CMPI transfectadas con el vector de prueba pequeña Pmax-GFP son FACS analizó para la expresión de GFP. (C) La expresión transitoria del plásmido AAVS1-CAG-EGFP se evalúa mediante FACS. (D) Un NCRM5-AAVS1-CAG-EGFP clones pantallas uniformemente expresión EGFP positivas como ensayada por FACS. Tenga en cuenta la agrupación apretada de iPSCs analizados en comparación con C). (Imagen E) Fluorescencia de un NCRM5-AAVS1-CAG-EGFP clon específico ampliado.
Discusión
The most critical steps for the successful generation of AAVS1 safe-harbor targeted human iPSCs are: (1) efficiently delivering TALEN and donor plasmids into iPSCs by transfection; (2) optimizing dissociation of iPSCs into single cells before transfection and plating density after transfection; (3) optimizing dose and time of drug-selection based on the growth of iPSC line; (4) carefully dissecting and picking targeted colonies and transferring to new plate/well. Compared to similar methods used in Hockemeyer’s paper10, this protocol used a pair of open-source AAVS1-TALENs, different iPSC dissociation reagent, and different transfection device to deliver TALENs and donor vectors, which helped to reduce the number of iPSCs used in the experiment while achieving high transfection and targeting efficiencies.
To achieve high-efficiency gene editing in human iPSCs, it is essential to first optimize the delivery of gene editing reagents (DNA/RNA), while balancing the acute cell death the delivery method and reagents cause to iPSCs. The goal is to maximize the delivery efficiency while maintaining a tolerably low level of cell death. Since it is very easy to prepare large quantities of AAVS1-TALEN plasmids that can achieve >50% HR efficiency with the help of drug-selection in human iPSCs, it is not necessary to make mRNAs from TALEN plasmids. The challenge of delivery comes from large donor plasmids, which in this case is ~10 kb. It is recommended to use the AAVS1-CAG-EGFP donor to test delivery efficiency in specific human iPSC lines rather than using pMaxGFP included in the transfection kit, because pMaxGFP is a ~3.5 kb small plasmid and very easy to deliver into any cells. AAVS1-CAG-EGFP donor can be modified using restriction enzymes shown in Figure 1A to target different transgenes into AAVS1 locus. Another 12 kb donor, which contains a 6.4 kb cassette to replace CAG-EGFP, has been used successfully to achieve similar transfection and targeting efficiency (data not shown). In general, human iPSCs are among the difficult-to-transfect cell types and the delivery efficiency for large plasmids can be as low as 5-10% as measured by flow cytometry analysis of GFP+ cells. Further optimization of transfection efficiency is dependent on the proper dissociation of human iPSCs into single cells, because cell clumps will reduce the chance of delivering gene editing reagents into each cell. This protocol uses a gentle and fast-working cell dissociation reagent, but treating iPSCs for more than 10 minutes is not recommended. Usually the iPSC culture is less than 80% confluent if following step 5.1, making a 5 minute incubation with pre-warmed gentle-cell dissociation reagent typically sufficient to dissociate the iPSCs into single cells with gentle pipetting. If not all the cells can be dissociated into single cells after 10 min of treatment because cell culture is over-confluent or the colonies become very compact, simply collect the recommended number of single cells from more wells/plates and leave the tightly attached cells behind. Heavy pipetting is not recommended because mechanical shearing is more detrimental to the cells than enzymatic dissociation. When working with early-passage iPSCs (before passage #15), gentle dissociation practice is very important for cell survival because the cells are already more sensitive to transfection stress than late passage iPSCs. If it is difficult to achieve high-efficiency in both transfection and cell survival, choose the practice that gives highest transfection efficiency. After transfection, the cells should be plated at the density that will reach 50-80% confluency at day 3 when drug-selection starts. Since each iPSC line grows differently, the post-transfection plating density might need to be optimized for each cell line. A high plating density may lead to over-confluency that reduces the efficacy of drug-selection, while an extreme low plating density may cause delayed recovery and growth of drug-resistant colonies. Usually 3 million starting iPSCs are enough to be plated into ½ to 2 10 cm dishes depending on the survival and growth of specific iPSCs. Similarly, if using a newly generated or as-yet untested iPSC line, generating a puromycin kill curve to establish the lowest effective dose in untransfected cells may be necessary; iPSC lines can vary considerably in their sensitivity to puromycin selection. The MEFs were used for selection because they appear to support iPSC growth better than some extracellular matrices during drug selection. As a rule of thumb, a minimum of 5 days of puromycin at 1 μg/ml or 7 days at 0.5 μg/ml are needed to complete selection. Finally, colony-picking technique is crucial to preserve all the targeted colonies after drug selection. Gentle pipetting or dissociation reagent treatment helps to break the colonies into multiple pieces evenly distributed into the new well, and therefore can speed up colony expansion. If using a dissociation reagent to disaggregate the picked colonies prior to plating, make sure it is sufficiently diluted with >10x medium after treatment, because the residual dissociation reagent could kill the transferred cells if left on overnight. No matter which technique is used, practicing colony picking before the real experiments is highly encouraged.
As the described strategy utilizes a gene-trap method to drive expression of the PAC gene off of the endogenous PPP1R2C promoter, picking significantly more than 30 colonies should not prove necessary. The SA-2A linked PAC selection theoretically eliminates random integration-only cells, but additional random integration(s) can occur in iPSCs bearing successful targeted integrations. In most cases, nearly 100% of drug-selected clones have targeted integrations and ~10-40% of them have additional random integrations. The majority of targeted clones tend to have single-allele targeting, although experiments wherein >50% double-allele targeting does occur. The variable frequency of additional random integrations, as well as the ratio of single- versus double-allele targeting, is difficult to control. Therefore, picking ~20 colonies is recommended to ensure single- or double-allele targeting-only clones can be obtained. In light of unsuccessful targeting experiments, re-assessment of the target cells’ transfectability, survival, and growth rates, the efficacy of nucleases in the specific cell line, and the quality of donor and nuclease preparations is highly recommended before re-attempting the experiment in its entirety.
It should be noted that similar gene-trap donor strategies can be used for targeting other active genes, but is not suitable for silent gene targeting which requires an independent promoter to drive selectable gene expression. Also, while the AAVS1 safe harbor is considered to have open chromatin structure, this does not guarantee that any transgene can be expressed strongly at this locus. Indeed, our recent report showed that several weak promoters were unable to drive detectable fluorescent reporter gene expression after targeted integration at AAVS1 locus14.
This protocol focuses on using well-validated TALENs to target a well-studied safe harbor locus in the human genome. The techniques are highly reproducible and easily adaptable to many applications involving any transgene. Compared to genome engineering methods using random integrations, AAVS1-TALEN mediated gene targeting is highly efficient and specific, and in the case of iPSCs, results in stably fluorescent cells that have the potential to expand and differentiate into any human cell type. While this protocol does not describe the design and validation of designer nucleases or the assessment of targeted iPSC colonies for proper cassette integration and off-target analysis, several excellent protocols have described these aspects of the methodology in detail16-18. The feeder-free iPSC culture and passaging techniques using E8 medium are also described in details in previous publication19. The above protocol, while optimized for gene addition into the AAVS1 safe-harbor of human iPSCs, can serve as a general template for experiments involving site-specific nuclease-mediated gene editing/addition using a homologous recombination donor in any cell type.
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
This research was supported by the NIH Common Fund and Intramural Research Program of the National Institute of Arthritis, Musculoskeletal, and Skin Diseases.
Materiales
| Name | Company | Catalog Number | Comments |
| Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix, *LDEV-Free, 10 ml | Corning | 354230 | Store at -20 °C. |
| DMEM/F-12 | Life Technologies | 11320-033 | Store at 4 °C. |
| Costar 6 Well Clear TC-Treated Multiple Well Plates | Corning | 3506 | |
| Essential 8 Medium | Life Technologies | A1517001 | Store basal medium at 4 °C. Store supplement at -20 °C. |
| Y-27632 dihydrochloride | Tocris | 1254 | Store at room temp. Once dissolved in H2O, store at -20 °C. |
| Sodium Chloride | Sigma | S5886-500G | |
| UltraPure 0.5 M EDTA, pH 8.0 | Life Technologies | 15575-020 | |
| DPBS, no calcium, no magnesium | Life Technologies | 14190-250 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| DR4 MEF 2M IRR - Academic | GlobalStem | GSC-6204G | Store in liquid Nitrogen. |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-040 | Store at 4 °C. |
| Defined Fetal Bovine Serum, US Origin | HyClone | SH30070.03 | Store at -20 °C. Thaw at 4 °C overnight and aliquot. Store aliquots at -20 °C until needed. |
| MEM Non-Essential Amino Acids Solution (100X) | Life Technologies | 11140-050 | Store at 4 °C. |
| 4D-Nucleofector Core unit | Lonza | AAF-1001B | part of the electroporation system |
| 4D-Nucleofector X unit | Lonza | AAF-1001X | part of the electroporation system |
| P3 Primary Cell 4D-Nucleofector X Kit L (24 RCT) | Lonza | V4XP-3024 | Upon arrival, remove Primary Cell Solution and supplement and store at 4 °C. |
| StemPro Accutase Cell Dissociation Reagent | Life Technologies | A1110501 | Store at -20 °C. Thaw overnight at 4°C and warm an aliquot in a 37 °C water bath before use. |
| NutriStem XF/FF Culture Medium | Stemgent | 01-0005 | Store at -20 °C. Thaw overnight at 4 °C |
| AAVS1 TALENs (pZT-AAVS1-L1 and pZT-AAVS1-R1) | Addgene | 52637 and 52638 | |
| [header] | |||
| AAVS1-CAG-EGFP Homologous Recombination donor | Addgene | 22212 | |
| Puromycin Dihydrochloride | Life Technologies | A11138-03 | Store at -20 °C. Prepare working aliquots of 1 mg/ml in ddH2O. |
| Disposable Borosilicate Glass Pasteur Pipets | Fisher Scientific | 13-678-20A | |
| Sorvall Legend XTR (Refrigerated), 120 V 60 Hz | Thermo Scientific | 75-004-521 | |
| TX-750 4 × 750 ml Swinging Bucket Rotor | Thermo Scientific | 75003607 | |
| Trypan Blue Solution, 0.4% | Life Technologies | 15250-061 | |
Referencias
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Hentze, H., Graichen, R., Colman, A. Cell therapy and the safety of embryonic stem cell-derived grafts. Trends Biotechnol. 25 (1), 24-32 (2007).
- Smith, J. R., et al. Robust, persistent transgene expression in human embryonic stem cells is achieved with AAVS1-targeted integration. Stem Cells. 26 (2), 496-504 (2008).
- Lombardo, A., et al. Site-specific integration and tailoring of cassette design for sustainable gene transfer. Nat Methods. 8 (10), 861-869 (2011).
- Ogata, T., Kozuka, T., Kanda, T. Identification of an insulator in AAVS1, a preferred region for integration of adeno-associated virus DNA. J Virol. 77 (16), 9000-9007 (2003).
- Zwaka, T. P., Thomson, J. A. Homologous recombination in human embryonic stem cells. Nat Biotechnol. 21 (3), 319-321 (2003).
- Urbach, A., Schuldiner, M., Benvenisty, N. Modeling for Lesch-Nyhan disease by gene targeting in human embryonic stem cells. Stem Cells. 22 (4), 635-641 (2004).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nature biotechnology. 27 (9), 851-857 (2009).
- Zou, J., et al. Gene targeting of a disease-related gene in human induced pluripotent stem and embryonic stem cells. Cell stem cell. 5 (1), 97-110 (2009).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nature. 29 (8), 731-734 (2011).
- Sanjana, N. E., et al. A transcription activator-like effector toolbox for genome engineering. Nature protocols. 7 (1), 171-192 (2012).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Ding, Q., et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell stem cell. 12 (4), 393-394 (2013).
- Luo, Y., et al. Stable Enhanced Green Fluorescent Protein Expression After Differentiation and Transplantation of Reporter Human. Induced Pluripotent Stem Cells Generated by AAVS1 Transcription Activator-Like Effector Nucleases. Stem Cells Transl Med. 3 (7), 821-835 (2014).
- Zou, J., et al. Oxidase-deficient neutrophils from X-linked chronic granulomatous disease iPS cells: functional correction by zinc finger nuclease-mediated safe harbor targeting. Blood. 117 (21), 5561-5572 (2011).
- Luo, Y., Rao, M., Zou, J. Generation of GFP Reporter Human Induced Pluripotent Stem Cells Using AAVS1 Safe Harbor Transcription Activator-Like Effector Nuclease. Curr Protoc Stem Cell Biol. 29, 5A.7.1-5A.7.18 (2014).
- Peters, D. T., Cowan, C. A., Musunuru, K. Genome editing in human pluripotent stem cells. StemBook. , Harvard Stem Cell Institute. Cambridge, MA. Available from: http://www.stembook.org/node/1438 (2008-2013).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Beers, J., et al. Passaging and colony expansion of human pluripotent stem cells by enzyme-free dissociation in chemically defined culture conditions. Nat Protoc. 7, 2029-2040 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados