Method Article
Líquido cefalorraquidiano MicroRNA perfil usando Quantitative PCR em Tempo Real
Neste Artigo
Resumo
Nós descrevemos um protocolo de PCR em tempo real para o perfil de microRNAs no líquido cefalorraquidiano (LCR). Com a excepção dos protocolos de extracção de ARN, o procedimento pode ser estendido para o RNA extraído a partir de outros fluidos do corpo, as células cultivadas, ou amostras de tecido.
Resumo
MicroRNAs (miRNAs) constituem uma camada poderosa de regulação gênica, orientando RISC para atingir sítios localizados em mRNAs e, conseqüentemente, modulando sua repressão translacional. Alterações na expressão de miARN têm mostrado estar envolvidas no desenvolvimento de todas as principais doenças complexas. Além disso, resultados recentes demonstraram que os miRNAs pode ser secretada para o meio extracelular e entram na corrente sanguínea e outros fluidos corporais onde pode circular com elevada estabilidade. A função de tais miRNAs circulantes permanece difícil, mas as abordagens de alto rendimento sistemáticos, tais como matrizes de perfil miRNA, levaram à identificação de assinaturas miRNA em várias condições patológicas, incluindo doenças neurodegenerativas, e vários tipos de cancros. Neste contexto, a identificação do perfil de expressão de miRNA no líquido cefalorraquidiano, como relatado em nosso estudo recente, faz miRNAs candidatos atraentes para análise de biomarcadores. _content "> Existem várias ferramentas disponíveis para perfilar microRNAs, tais como microarrays, quantitativa PCR em tempo real (qPCR) e seqüenciamento de profundidade. Aqui, descrevemos um método sensível para o perfil de microRNAs em fluidos cerebrospinal por quantitativa PCR em tempo real. Nós utilizados os painéis humanos PCR prontos para uso Exiqon microRNA I e II V2.R, que permite a detecção de 742 microRNAs humanos únicos. Realizamos as matrizes em triplicado corridas e nós processados e analisados dados usando o software GenEx Professional 5.
Usando este protocolo, temos perfilado com sucesso microRNAs em vários tipos de linhas celulares e pilhas, CSF, plasma e tecidos embebidos em parafina fixadas em formalina.
Introdução
MicroRNAs pertencem à família de pequenos (21-23 nt de comprimento) não codificante ARN que regulam a expressão do gene de pós-transcricionalmente. microRNAs pode ser secretada no espaço extracelular, onde eles parecem ser relativamente estável. Ao determinar mudanças na expressão de miRNA pode ser um passo importante para identificação de biomarcadores, realizando perfis de miRNA e lidar com uma grande quantidade de dados pode ser intimidante.
Aqui, nós descrevemos um protocolo para determinar mudanças na expressão de miRNA no líquido cefalorraquidiano (aplicável a outros fluidos corporais) por um tempo real sensível PCR. Nós também descrevem o uso de software para análise de dados, incluindo a análise estatística e representação gráfica dos resultados. Todo o procedimento é relativamente simples e fácil e, dependendo do número de amostras a serem perfiladas e o número de máquinas de PCR em tempo real disponíveis, também relativamente rápidos. A parte experimental requer precisão na manipulaçãoRNA e pipetagem em placas de 384 poços, enquanto a seção de análise de dados usando GenEx requer alguns conhecimentos básicos em informática e estatística.
Protocolo
O protocolo a seguir descreve o procedimento padrão para isolar RNA de LCR e de perfil microRNAs usando placas de PCR prontos. Note-se que a extracção de fase orgânica é opcional, e que um ARN transportador é adicionado à amostra durante a extracção para assegurar a recuperação máxima de ARN. Consequentemente, não há necessidade de quantificar o ARN.
Em geral, a média de 6-8 placas pode ser executado no mesmo dia que o cDNA é sintetizado no dia anterior (cerca de 2 horas). A análise dos dados é executada quando todas as amostras tiverem sido perfilado e carregada no software. Dependendo do número de amostras / grupos ou a sua combinação para efeitos de comparação, a análise dos dados pode necessitar de uma a várias horas.
1. Extração de RNA
A extracção de ARN foi realizada a partir de amostras de CSF, armazenadas congeladas a -80 ° C em alíquotas até análise. Para este procedimento foi utilizado o kit de isolamento de miRvana Paris, após o instrumento de fabricanteCÇÕES para o isolamento de RNA total. Por favor, note que, embora o enriquecimento de espécies de RNA pequenos é possível com este kit, este passo não for feito eo processo de extração de RNA é encerrado após a etapa de isolamento total de RNA. Além disso, embora o kit de isolamento de miRvana Paris (como outros kits de isolamento de RNA comercialmente disponíveis) não necessita de extracção orgânico, um protocolo para este procedimento pode ser encontrada abaixo.
O protocolo de extracção de ARN consiste em duas etapas:
1.1. Extração Orgânica (não é necessário se estiver usando o kit de extração de RNA Mirvana Paris)
1.2.2. Isolamento de ARN total
1.1. Extracção orgânica
1.1.1. Preparar reagentes
- Adicionar 375 mL de 2-mercaptoetanol 2x solução de desnaturação e armazená-lo a 4 ° C.
1.1.2. Prepare o CSF
- Descongele cada alíquota de CSF no gelo antes da extração de RNA. Adicione 1 mg de RNA transportador MS2 para 0,5 ml de CSF descongelado e gentilmentemisturar. Note-se que, se a extracção orgânica é omitido, o transportador de MS2 será adicionado à amostra de isolamento de ARN antes descrito em 1.2.2.1 abaixo.
- Misturar 0,5 ml de 2x solução de desnaturação à temperatura ambiente para o CSF.
- Adicionar 1 ml de ácido-fenol: clorofórmio para o CSF mais a solução desnaturante 2x: certifique-se de retirada da fase inferior contendo o ácido-fenol: clorofórmio, o tampão não aquoso que se encontra no topo da mistura. Além disso, observe que o ácido-fenol: clorofórmio contém fenol, que é um veneno e um irritante, utilizar luvas e outros equipamentos de proteção individual ao trabalhar com este reagente.
- Vortex por 30-60 segundos para misturar. Por conveniência, os 2 ml de amostra / desnaturante solução / ácido-fenol: mistura de clorofórmio são divididos em dois tubos Eppendorf de 1,5 ml.
- Centrifugar durante 5 minutos à velocidade máxima (10000 xg), à temperatura ambiente.
- Cuidadosamente remover a fase aquosa (superior), sem perturbar a fase inferior ou a interfase e transferi-lo para umatubo fresco. Observe o volume recuperado.
1.2. Isolamento de ARN
Recomenda-se a ter um banco dedicado, um conjunto de pipetas e racks para lidar com amostras de RNA. A área de trabalho e as ferramentas são descontaminados utilizando spray de RNase Zap e limpa antes de cada experimento.
1.2.1. Preparar reagentes:
- Adicionar 21 ml de etanol a 100% a miARN solução de lavagem 1 e 40 ml de etanol a 100% para a solução de lavagem de 2/3. Por favor note que miRNA Wash Solution 1 contém tiocianato de guanidina, que é uma substância potencialmente perigosa.
1.2.2. Isolamento de ARN total
- Adicionar 1,25 volumes de etanol a temperatura ambiente de 100% para a fase aquosa da extracção orgânica e misturar completamente.
- Coloque um cartucho de filtro em um dos tubos de coleta.
- Pipeta não mais do que 700 ul da mistura de ligado / etanol para o cartucho de filtro.
- Centrifugar por 30 segundos no máximovelocidade. Aplicar a mistura superior a 700 ul em aplicações sucessivas com o mesmo filtro. Descarte o flow-through após cada centrifugação e salvar o tubo de coleta para as etapas de lavagem.
- Aplicar 700 mL miRNA solução de lavagem 1 para o cartucho do filtro e centrifugar por 15 segundos na velocidade máxima. Descarte o fluxo através do tubo de coleta e substituir o cartucho do filtro no mesmo tubo de coleta.
- Aplicar 500 mL de solução de lavagem de 2/3 e centrifugar por 15 segundos na velocidade máxima. Descarte o fluxo através do tubo de coleta e substituir o cartucho do filtro no mesmo tubo de coleta. Aplicar uma segunda 500 mL de solução de lavagem de 2/3 e centrifugar por 15 segundos na velocidade máxima. Descarte o fluxo através do tubo de coleta e substituir o cartucho do filtro no mesmo tubo de coleta. Girar o conjunto durante 1 minuto à velocidade máxima, para remover o líquido residual a partir do filtro.
- Transfira o cartucho do filtro em um tubo de coleta de fresco. Aplicar 100 & mu; L de pré-aquecido (95 ° C), água livre de nuclease para o centro do filtro, e fechar a tampa. Centrifugar durante 30 segundos a velocidade máxima, para recuperar o RNA.
- Recolher o eluato contendo RNA e armazená-lo a -80 ° C para aplicações subseqüentes.
- Quantificar 1 ml de RNA utilizando o espectrofotômetro NanoDrop 2000c. Note-se que a quantificação de RNA pode ser ignorado se um transportador de RNA é adicionado à amostra durante a extracção de RNA. Nesse caso, 8 ul de RNA são submetidos a síntese de cDNA, como detalhado abaixo.
2. miARN Perfil: Protocolo de qRT-PCR
O protocolo para a miARN perfis consiste em duas etapas:
- A síntese de cDNA da primeira cadeia
- Real-time PCR
2.1. A síntese de cDNA da primeira cadeia
2.1.1. Dilui-se ARN modelo
- Ajustar cada uma das amostras de ARN do molde para uma concentração de 5 ng / mL usando nuclease isenta de água. Se oARN não foi quantificada (ver passo 1.2.2.9) e, em seguida 8 ul de RNA são utilizados para a síntese de cDNA
2.1.2. Preparar reagentes
- Gentilmente descongelar o tampão de reação 5x e água livre de nuclease e coloque imediatamente em gelo.
- Misture em vortex. Ressuspender o RNA spike-in através da adição de 40 mL de água livre de nuclease para o tubo e misturar em vortex; deixá-lo no gelo por 15-20 min. Imediatamente antes da utilização, retire a mistura de enzimas do congelador, misture sacudindo os tubos e coloque no gelo. Gire para baixo todos os reagentes.
2.1.3. Configuração de reação de transcrição reversa
- Preparar a quantidade necessária de solução de trabalho de RT em um tubo Eppendorf de 1,5 ml ou equivalente (nas proporções indicadas na Tabela 1, com excepção para o molde de ARN), misturar em vortex (1-2 segundos a velocidade máxima), girar rapidamente para baixo ( usamos um mini centrífuga Eppendorf com velocidade fixa, 10 seg) e colocá-lo no gelo. Note-se que o RNA de pico UniSp6-In é adicionado à amostra antes da reacção de RT (ver Tabela 1).
- Dispensar a solução de trabalho RT em tubos de PCR livre de nuclease.
- Dispensar molde de ARN em cada tubo.
Nota: lembre-se de calcular 10% de excesso de volume / cada reagente para pipetagem e / ou volume morto robótico.Reagente Painel I Volume (mL) Painel I + II Volume (mL) De tampão de reacção 5x 4.4 8.8 Nuclease livre água 9,9 19,8 Mistura de enzima 2.2 4.4 UniSp6 RNA Spike-no template 1.1 2.2 RNA total de molde (5 ng / mL) 4.4 8.8 Volume Total 22 44
Tabela 1. Reverter configuração reação de transcrição.
2.1.4. Misture e girar reagentes
- Misture a reacção por agitação suave (1-2 seg) vórtex para assegurar que todos os reagentes são bem misturados. Após a mistura, spin down (mini centrífuga Eppendorf com velocidade fixa, 10 seg).
2.1.5. Incubar e inativar calor
- Programar uma máquina de PCR como se segue:
- Incubar durante 60 minutos a 42 ° C
- Heat-inactivar a transcriptase reversa durante 5 min a 95 ° C
- Imediatamente arrefecer a 4 ° C
- Armazenar a 4 ° C ou congelar
Nota: o protocolo pode ser interrompido a esta fase. O cDNA não diluída pode ser mantida a -20 ° C durante até 5 semanas (opcional armazenar a 4 ° C durante até 4 dias).
2.2.1. Preparar reagentes para PCR em tempo real
- Coloque cDNA (a partir do passo 2.1.5) em gelo.
- Água livre e PCR Master Mix nuclease Thaw no gelo. Proteger os frascos de PCR master mix de luz, cobrindo-os com papel alumínio. Imediatamente antes da utilização, misture o Mix Master PCR em vortex 1-2 sec em um mini centrífuga e girar a 1.500 xg por 1 min.
2.2.2. Combine SYBR Green master mix, água e cDNA e adicionar placas de PCR
O procedimento seguinte é recomendado para evitar a baixas concentrações de cDNA a partir de aderir à superfície do tubo:
- Antes de retirar o selo da placa, rodar rapidamente para baixo da placa (s) em uma centrífuga refrigerada a 1.500 xg durante 1 min.
- Em um tubo de 15 ml, combinam 2x Mix Master PCR e água. Painel I: 2.000 mL 2x mix master e 1.980 mL de água; Painel I + II: 4.000 mL 2x master mix e 3960 μ, L de água.
- Adicionar 20 mL de cDNA (Painel I) ou 40 mL de cDNA (painel I + II) e misturar.
- Misture suavemente em vortex 1-2 seg e spin para baixo em uma centrífuga refrigerada a 1500 xg por 1 min.
- Coloque o PCR Master Mix / cDNA mistura em uma pipeta multicanal reservatório. Pode ter de identificar um reservatório que possui o comprimento da pipeta multicanal. Isso mantém o nível de volume de alta o suficiente para permitir que alíquotas de volume iguais em toda a multicanal. Isso é fundamental para os últimos alíquotas.
- Dispensar 10 ul de mistura de PCR Master / cDNA misturar a cada poço da placa de 384 poços com uma pipeta de 16 canais.
- Selar a placa com vedação óptica.
- Rotação placa brevemente numa centrifugadora refrigerada (1500 xg durante 1 min), para misturar as soluções e remover as bolhas de ar.
Nota: o experimento pode ser interrompido neste ponto. Armazenar as reacções protegidas da luz a 4 ° C durante até 24 horas.
2.2.3. Real-Time PCR
Realizar em tempo real a amplificação por PCR e análise de curva de fusão após os parâmetros descritos na Tabela 2.
| Etapa do Processo | Configurações, Roche LC480 |
| Polymerase Ativação / desnaturação | 95 ° C, 10 min |
| Amplificação | 45 ciclos a amplificação 95 ° C, 10 seg 60 ° C, 1 min Taxa de rampa 1,6 ° C / seg Leitura óptica |
| Análise da curva de fusão | Sim |
Tabela 2. PCR em tempo real as condições de ciclo utilizando o LightCycler 480.
2.3. Análise em tempo real de dados de PCR
A análise em tempo real de dados de PCR é realizada com o Exiqon GenEx Professional 5.0, a seguir tele recomendou instruções. O manual GenEx passo-a-passo pode ser baixado em http://www.exiqon.com/ls/Documents/Scientific/Exiqon-data-analysis-guide.pdf
A análise de dados é constituído por três passos:
- Importação de dados
- Pré-processamento de dados
- A análise estatística
2.3.1. Importação de dados
- Na Roche LightCycler 480 selecionar o método de análise de 2 ª derivada e calcular os valores de Cq. Os dados são exportados como uma mesa e salvos como arquivos de texto.
- Para importar os dados, aberto GenEx e clique no botão assistente de importação Exiqon e clique em "Start". Siga as instruções para selecionar o formato, instrumento e arquivo layout da placa (s). Note que os arquivos de layout de placas (Excel arquivos) são baixados do site da Exiqon ( http://www.exiqon.com/plate-layout-files ).
- Import painéis I e II e clique em "Next".
- A tabela gerada após a importação do arquivo contém colunas predefinidas. Amostras nomes podem ser editados e colunas de classificação podem ser adicionados ou removidos nesta etapa. Clique em "Next" quando você está feito.
- Salve os dados e carregar para o Editor de Dados.
2.3.2. Pré-processamento de dados
- Conforme recomendado pelo Exiqon e GenEx, ao comparar múltiplas placas, a primeira coisa a fazer é calibrar os dados entre as placas, escolhendo calibração inter-prato do menu de pré-processamento.
- Recomenda-se a executar as matrizes de miRNA em triplicado corridas. Se um miRNA não está presente em pelo menos duas das três réplicas ele será definido como nonexpressed.
- Na próxima etapa do pré-processamento, definir um ponto de corte. A definição de um valor de corte indica que os dados com um ciclo limiar (C T) mais elevado do que este valor, são descartados. Nos presentes experimentos usando amostras de líquido cefalorraquidiano, o off corte foi fixado em 39. Therntes, todas as amostras com um C t superior a 39 nas três réplicas serão descartadas. Se um miRNA tem um C t> 39 para uma sonda (uma réplica de três), que a leitura será substituído com a média da C t para as outras duas sondas, desde que sejam ambos abaixo 39.
- Definir genes de referência. GenEx profissional tem a opção de utilizar geNorm e / ou NormFinder para identificar genes de referência. Selecione uma lista de miRNAs que têm valores semelhantes em todos Cq amostra e executar geNorm e / ou NormFinder. De acordo com esta análise, no exemplo apresentado aqui, o miR-622 e miR-1266 teve os valores mais uniformes entre as amostras e foram escolhidos como os genes de referência (Figura 1).
- Em seguida, os dados são normalizados utilizando os genes de referência e os valores obtidos correspondem ao Ac t
- No caso das reacções de síntese de cDNA foi realizada em triplicado, neste ponto os valores devem ser calculadas.
- Validar a folha de remover colunas vazias e quase vazias. Na janela de pré-processamento, selecione "Validar folha", e que se aplicam na linha de colunas vazias Remove. Na linha abaixo, escolha a porcentagem desejada de dados válidos e clique em Aplicar. Por exemplo, selecione 100%, se você quiser comparar apenas miRNAs comuns a todas as amostras.
- Manipular dados em falta.
Selecione "Dados em falta" no menu de pré-processamento e escolheu uma das opções para lidar com dados faltantes. Esta etapa é necessária. Note-se que GenEx lhe dará erro se você tentar carregar arquivos que contêm dados em falta não tratadas. - Determine quantificação relativa entre grupos de amostras (ie. Experimentais versus controle). A quantificação relativa entre os grupos é calculada usando o método ΔΔ C t. Em GenEx, clique na guia de pré-processamento e selecione quantificação relativa. Na janela, selecione o grupo de referência e clique em Aplicar. Observe que, para as representações gráficas e / ou para as análises estatísticas, expressão dados devem ser convertidos para uma escala logarítmica. No menu de pré-processamento, selecione Log2.
2.3.3. Análise Estatística
Software GenEx permite uma ampla gama de análises estatísticas, incluindo T-teste e análise de variância.
- Carregue o arquivo mdf final para GenEx e gerenciador de dados aberto. É crítico para seleccionar as amostras de correcção ou de grupos de amostras para as quais a análise estatística é realizada.
- Selecione "Estatísticas" e escolheu o ícone correspondente ao teste desejado.
- Clique em "Run" na janela do painel de controle. Exceto resultados como excel ou mdf.
2.3.4. Expressão profiling
MicroRNAs e amostras podem ser classificadas, agrupadas e visualizados em calor-mapas e dendrogramas, como se segue
- Carregue o arquivo mdf final para GenEx e gerenciador de dados aberto. Utilize esta janela para selecionar e criar grupos de amostras ou miRNAs. Clique em "Aplicar" quando você está feito.
- No canto esquerdo superior de seleção de cluster e clique no ícone heatmap
- Na janela do painel de controle clique em "Executar". O heatmap podem ser salvos em vários formatos, como TIFF ou BMP, mas também pode ser copiado e modificado conforme a necessidade.
Resultados
Os resultados deste estudo foram publicados anteriormente 1. Figura 1 mostra os resultados da análise de microRNAs de referência candidatos usando o aplicativo geNorm. Por conseguinte, dois microRNAs, miR-622 e miR-1266, foram identificados como genes de referência e foram usadas para normalizar os valores de miRNA.
Para o estudo CSF que tinha três grupos de amostras: VIH-(n = 10), COLMEIA (n = 4), e HIV + sem Encefalite (VIH +, n = 5). Os dois grupos, HIVE e HIV +, foram comparados com o controle de HIV-grupo. Após a análise estatística (seção 2.3.3) níveis de onze microRNAs expressão foi encontrada significância estatística 1. Figura 2 representa um gráfico de caixa desta onze microRNAs, miR-1203,-1224-3p, -182 *,-19b-2 *, -204,-362-5p, -484, -720, -744 *, -934, -937 e. Cada coluna mostra a distribuição dos dados entre as amostras dentro do grupo (verde para a HIVE e vermelho para HIV + sem ciaphalitis). Bigodes indicam a mediana, o 1 º (Q1) e 3 º (Q3) quartil eo máximo e nonoutliers mínimos.
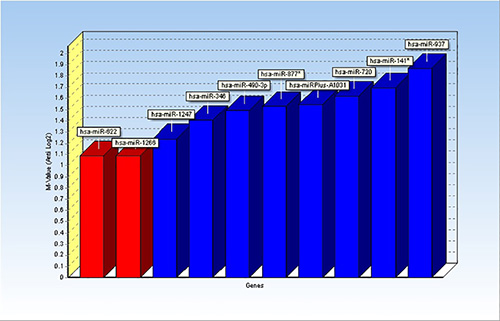
Figura 1. Bar gráfico mostrando os resultados de aplicação geNorm dentro GenEx. Dez microRNAs foram analisadas como possíveis genes de referência e resultados indicam miR-622 e miR-1266 (barras vermelhas) como os miRNAs mais estável expressas. Clique aqui para ver imagem ampliada.

Figura 2. Box plot mostrando a distribuição dos dados fou cada um dos onze miRNAs dentro dos grupos de HIVE (verde) e HIV + sem encefalite (HIV +, vermelho). Os bigodes em cada coluna indicam a mediana, 1 º (Q1, fundo da caixa) e 3 º (Q3, top da caixa) quartis, valores máximos e mínimos que são nonoutliers (linhas pretas). Clique aqui para ver imagem ampliada.
Discussão
MicroRNAs são pequenos RNAs não codificadores que regulam a expressão de genes, inibindo a tradução e / ou a promoção da degradação do mRNA 2. Devido à sua elevada estabilidade em condições livres de células, microRNAs foram detectados em muitos fluidos corporais. Além disso, o perfil de expressão de microRNAs distinta tem sido correlacionada com o estágio e / ou progressão em uma variedade de cânceres humanos 3-9.
Existem várias ferramentas disponíveis para o perfil de microRNAs, incluindo matrizes de chips clássicos ou a mais recente tecnologia de seqüenciamento de profundidade. No entanto, optou-se por utilizar uma plataforma altamente sensível qPCR 10, 11, que tem a vantagem adicional de exigir quantidade mínima de RNAs. O protocolo miRCURY LNA Universal RT microRNA PCR é otimizada para usar 20 ng de RNA total por 20 l reação de síntese de cDNA, 40 ng para a matriz de dois painel completo. Tendo a opção de utilização de pequena quantidade de ARN é muito importante quando se lida com valuabl e amostras clínicas e de difícil obtenção, como CSF ou embebidos em parafina tecidos fixados em formalina 1. Mais importante, a utilização de pequenas quantidades de ARN de minimizar a concentração de inibidores possíveis presentes na amostra. Fora do intervalo de valores de Ct para o ponto-de, provavelmente, indicando a presença de alguns inibidores na amostra. Sondas Spike-in pode ser adquirido separadamente para testar as amostras para a presença de inibidores antes de miRNA perfil. Para este teste, o tempo único verdadeiro PCR são realizadas utilizando-se concentrações crescentes de ARN (quer ng ou ul).
No presente protocolo, relatamos a extração em fase orgânica antes do isolamento do RNA. Deve-se notar que muitos dos novos kits de extração de RNA não requerem este procedimento. Por exemplo, temos perfilado com sucesso miRNAs plasma utilizando os miRCURY RNA biofluids kit de isolamento para a extracção de ARN directamente a partir de 200 ul de plasma (dados não mostrados), sem extracção com fenol / clorofórmio.
_content "> Em geral, é importante para a concepção do experimento com antecedência, a fim de determinar o número apropriado de repetições necessárias para obter resultados estatisticamente significativos. O número de repetições pode depender do número de amostras a serem analisadas e podem depender do variações dentro das amostras ou grupos de amostras. Para o nosso estudo, para o qual foi utilizado um número relativamente baixo de amostras, 20 no total, decidimos criar a reação de síntese de cDNA em triplicatas. Consulting com um biostatistician antes de configurar os experimentos podem ser uma escolha sábia e é altamente recomendado.Definindo o C t cortado valor é importante e depende do tipo de amostras, por miRNAs altamente expressos pode ser definido entre 25-35, mas para miRNAs expressos baixas, como nossas amostras de líquido cefalorraquidiano, pode ser definido superior. Um outro passo crucial quando se trata de análise dos dados é a escolha de gene (s) de referência. Para alguns dados sólidos (tais como perfis de miRNA em células cultivadas) aglobal normalização, que representa a média de C T de todas as amostras, pode ser utilizado. No entanto, esta não é uma opção para as amostras de CSF como com variações. Do mesmo modo, não foi possível utilizar como genes de referência dessas miRNAs que são geralmente considerados inalterada em fluidos corporais. Em vez disso, foram selecionados todos os miRNAs que mostraram semelhante C t em todas as amostras, incluindo réplicas, e correu a análise geNorm disponível em GenEx (Figura 1). Os resultados indicaram miR-1266 e miR-622 como o mínimo de miRNAs variavelmente expressas e eles foram usados como genes de referência. Comparação da expressão de miRNAs entre grupos pode ser determinada utilizando a ferramenta quantificação relativa em GenEx, que segue a fórmula padrão 2 - (Ct-Ctrel). Finalmente, os resultados podem ser visualizados como diagramas de mapa de calor, barras de gráficos ou outros tipos de terrenos, como o gráfico de caixa na Figura 2. Outros tipos de visualização disponíveis no GenEx incluem agrupamento hierárquico,Self-Organized Map (SOM), e Análise de Componentes Principais (PCA). Além miRNAs, o software GenEx pode ser utilizado para a análise de outros tipos de matrizes, tais como RNAs não codificadores longos (lncRNAs) de perfil. O layout da placa pode ser importado no formato Excel e os dados podem ser manipulados como descrito para a análise de miRNA. Com efeito, temos perfilado lncRNAs de rato neurónios corticais embrionárias primárias utilizando o kit de isolamento de miRvana miARN como descrito nas etapas anteriores e os dados foram analisados utilizando GenEx (resultados não publicados).
Em resumo, nós descrevemos um protocolo para o perfil de microRNAs no líquido cefalorraquidiano. Com algumas modificações relacionadas com a extracção de ARN, este procedimento pode ser adaptado ao perfil miRNAs em outros fluidos corporais e / ou outro tipo de tecidos. Note-se que no processo descrito aqui, um passo de extracção orgânico é realizado antes do isolamento do ARN. Em geral, isso não é necessário quando se utiliza kits disponíveis no mercado, como o ext Mirvana Pariskit raction. Além disso, quando se extrai o ARN a partir de fluidos corporais e uma ARN transportador é adicionado às amostras que não há necessidade de quantificar o ARN e 2-8 ul de RNA são submetidos a síntese de cDNA. A partir da nossa experiência e considerando os altos custos relacionados a este tipo de experimentos, recomendamos testar algumas amostras primeiro, e depois de consultar um estatístico para o número de amostras / réplicas para ser perfilado de modo a obter resultados estatisticamente significativos.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
O projeto descrito foi apoiada pelo Prêmio Número R01MH079751 (PI: F. Peruzzi) do Instituto Nacional de Saúde Mental. O conteúdo é de responsabilidade exclusiva de seus autores e não representam, necessariamente, a posição oficial do Instituto Nacional de Saúde Mental ou o National Institute of Health.
Materiais
| Name | Company | Catalog Number | Comments |
Table 3. List of equipment. | |||
| Finnpipette Novus Multichannel Pipetter | Thermo Scientific | HH05279 | |
| Finntip Pipette Tips | Thermo Scientific | 9400613 | |
| Thermal Cycler C1000 | Biorad | ||
| 0.2 ml Low Profile, Clear PCR tubes | Biorad | TLS0801 | |
| Flat Cap Strips | Biorad | TLS0803 | |
LightCycler 480 Real-Time PCR System | Roche | ||
LightCycler 480 Sealing Foil | Roche | 04 729 757 001 | |
| Refrigerated Centrifuge 5804R | Eppendorf | ||
| Swing-bucket Rotor, 4-place | Eppendorf | A-4-44 | |
| Bench-Top Mini Centrifuge | Fisher | 05-090-100 | |
| Bench-Top Vortex | Fisher | ||
| 1.5 ml Microcentrifuge Tubes, Sterilized | Fisher | 02-681-5 | |
| Matrix Reagent Reservoir, 25 ml | Thermo Scientific | 8093 | |
| Thermomixer Confort | Eppendorf | ||
Bench-Top Mini Centrifuge Sigma 1-15 | Sigma | ||
| Bench-Top Vortex | Velp Scientifica | F202A0173 | |
Table 4. List of reagents. | |||
| Universal c-DNA synthesis kit, 16-32 rxns | Exiqon | 203300 | |
| SYBR Green master mix, Universal RT, 25 ml | Exiqon | 203400 | |
| Ultrapure Distilled Water Dnase, Rnase free | Invitrogen | 10977-015 | |
| MicroRNA Ready to use PCR Human Panels (I+II) V2.R | Exiqon | 203608 | |
mirVana miRNA isolation Kit, 40 isolations | Ambion | AM1556 | |
| MS2 RNA carrier | Roche Applied Science | 10165948001 | |
2-mercaptoethanol 98% | Sigma Aldrich | M3148-25ml | |
| Ethanol 100% | Carlo Erba | 64-17-5 | |
| Nuclease free water | Qiagen | 1039480 | |
RNAse Zap | Ambion | AM9780 | |
Referências
- Pacifici, M., et al. Cerebrospinal fluid miRNA profile in HIV-encephalitis. J. Cell. Physiol. 10, (2012).
- Krol, J., Loedige, I., Filipowicz, W. The widespread regulation of microRNA biogenesis, function and decay. Nat. Rev. Genet.. 11, 597-610 (2010).
- Calin, G. A., Croce, C. M. MicroRNA signatures in human cancers. Nat. Rev. Cancer. 6, 857-866 (2006).
- Chen, X., et al. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Res. 18, 997-1006 (2008).
- De Smaele, E., Ferretti, E., Gulino, A. MicroRNAs as biomarkers for CNS cancer and other disorders. Brain Res. 1338, 100-111 (2010).
- Heneghan, H. M., Miller, N., Kerin, M. J. MiRNAs as biomarkers and therapeutic targets in cancer. Curr. Opin. Pharmacol. 10, 543-550 (2010).
- Kosaka, N., Iguchi, H., Ochiya, T. Circulating microRNA in body fluid: a new potential biomarker for cancer diagnosis and prognosis. Cancer Sci. 101, 2087-2092 (2010).
- Lu, J., et al. MicroRNA expression profiles classify human cancers. Nature. 435, 834-838 (2005).
- Shah, A. A., Leidinger, P., Blin, N., Meese, E. miRNA: small molecules as potential novel biomarkers in cancer. Curr. Med. Chem. 17, 4427-4432 (2010).
- Blondal, T., et al. Assessing sample and miRNA profile quality in serum and plasma or other biofluids. Methods. 59, 1-6 (2013).
- Jensen, S. G., et al. Evaluation of two commercial global miRNA expression profiling platforms for detection of less abundant miRNAs. BMC Genomics. 12, 435 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados