Method Article
Liquor microRNA Profiling mittels quantitativer Real-Time PCR
In diesem Artikel
Zusammenfassung
Wir beschreiben ein Protokoll der Echtzeit-PCR, um microRNAs in der Gehirn-Rückenmarksflüssigkeit (CSF) zu profilieren. Mit Ausnahme von RNA-Extraktionsprotokollen kann der Vorgang um RNA aus anderen Körperflüssigkeiten, Zellkulturen oder Gewebeproben extrahiert verlängert werden.
Zusammenfassung
MicroRNAs (miRNAs) sind eine starke Schicht der Genregulation Führungs RISC zu Seiten mRNAs Ziel befindet und somit durch Modulation ihrer translationale Repression. Änderungen in der miRNA-Expression wurde gezeigt, dass bei der Entwicklung von allen großen komplexen Erkrankungen beteiligt sein. Darüber hinaus zeigten neuere Erkenntnisse, dass miRNAs an die extrazelluläre Umgebung sezerniert werden und in die Blutbahn und anderen Körperflüssigkeiten, wo sie mit hoher Stabilität zirkulieren kann. Die Funktion eines solchen zirkulierenden miRNAs bleibt weitgehend ungeklärt, aber systematische Hochdurchsatz-Methoden, wie z. B. miRNA-Profiling Arrays haben zur Identifizierung von miRNA-Signaturen in verschiedenen pathologischen Bedingungen, einschließlich der neurodegenerativen Erkrankungen und einige Krebsarten führen. In diesem Zusammenhang ist die Identifizierung von miRNA-Expressionsprofils in der Cerebrospinalflüssigkeit, wie in unserer jüngsten Studie berichtet, macht miRNAs attraktive Kandidaten für Biomarker-Analyse. _content "> Es gibt verschiedene Tools für die Profilierung von microRNAs, wie Microarrays zur Verfügung, quantitative Echtzeit-PCR (qPCR) und deep sequencing. Hier beschreiben wir eine empfindliche Methode, um microRNAs in Liquor durch quantitative Echtzeit-PCR-Profil. Wir verwendet die Exiqon microRNA ready-to-use PCR menschlichen Platten I und II V2.R, die ermöglicht den Nachweis von 742 einzigartige menschliche microRNAs. Wir führten die Arrays in dreifacher Ausfertigung läuft und wir verarbeitet und analysiert Daten mit der Genex Professional 5 Software.
Über dieses Protokoll, haben wir erfolgreich in verschiedenen Arten von Zelllinien und Primärzellen, Liquor, Plasma und Formalin-fixierten und in Paraffin eingebetteten Geweben Profil microRNAs.
Einleitung
MicroRNAs gehören zu der Familie der kleinen (21-23 nt Länge) nicht-kodierenden RNAs die Genexpression posttranskriptional regulieren. Mikro-RNAs in den extrazellulären Raum, in dem sie erscheinen, relativ stabil zu sein, sezerniert werden. Während Bestimmung von Veränderungen in der miRNA-Expression kann ein wichtiger Schritt zur Identifizierung von Biomarkern sein, die Durchführung miRNA-Profile und Handhabung einer großen Menge von Daten kann einschüchternd sein.
Hier beschreiben wir ein Protokoll von Änderungen in miRNA-Expression in der Zerebrospinalflüssigkeit von einem empfindlichen Echtzeit-PCR (für andere Körperflüssigkeiten) zu bestimmen. Wir beschreiben auch die Verwendung von Software für die Datenanalyse, einschließlich der statistischen Auswertung und grafische Darstellung der Ergebnisse. Das gesamte Verfahren ist relativ einfach und unkompliziert und in Abhängigkeit von der Anzahl der Proben, die profiliert werden und die Anzahl von Echtzeit-PCR-Maschinen verfügbar sind, auch relativ schnell. Der experimentelle Teil erfordert Genauigkeit bei der HandhabungRNA und Pipettieren in 384-Well-Platten, während der Datenanalyse Abschnitt mit Genex erfordert einige Grundkenntnisse in Informatik und Statistik.
Protokoll
Das folgende Protokoll beschreibt die Standard-Verfahren, um RNA aus GFK und Profil microRNAs mit fertigen PCR-Platten isolieren. Beachten Sie, dass die organische Extraktionsphase ist optional, und daß ein Träger-RNA wird während der Extraktion gewährleistet eine maximale Rückgewinnung der RNA zu der Probe gegeben. Folglich gibt es keine Notwendigkeit, um die RNA zu quantifizieren.
Insgesamt kann ein Durchschnitt von 6-8 Platten an einem Tag ausgeführt werden, wenn die cDNA am Tag zuvor (ca. 2 h) synthetisiert. Analyse der Daten wird ausgeführt, wenn alle Proben profiliert und in die Software geladen. Abhängig von der Anzahl von Proben / Gruppen oder deren Kombination für den Vergleich kann die Datenanalyse von einem bis zu mehreren Stunden dauern.
1. RNA-Extraktion
Bei -80 ° C in Aliquoten bis zur Analyse der RNA-Extraktion von CSF-Proben durchgeführt, gefroren gelagert. Für dieses Verfahren die Mirvana Paris Isolation Kit verwendet wurde, nach Angaben des Herstellers Instrumentections für die Gesamt-RNA Isolation. Bitte beachten Sie, dass, obwohl Bereicherung für kleine RNA-Spezies mit diesem Kit möglich ist, wird dieser Schritt nicht durchgeführt und die RNA-Extraktionsverfahren ist nach dem Gesamt-RNA Isolation Schritt beendet. Darüber hinaus, obwohl die Mirvana Paris Isolation Kit (wie andere kommerziell erhältliche RNA Isolation Kits) keine organischen Extraktions nicht verlangen, ein Protokoll für dieses Verfahren finden Sie unten.
Das Protokoll für die RNA-Extraktion erfolgt in zwei Schritten:
1.1. Bio-Extraction (nicht bei Verwendung Mirvana Paris RNA-Extraktions-Kits erforderlich)
1.2.2. Gesamt-RNA Isolierung
1.1. Bio-Extraktion
1.1.1. Bereiten Reagenzien
- In 375 ul 2-Mercaptoethanol zu 2x Denaturierung Lösung, und speichern Sie sie bei 4 ° C
1.1.2. Bereiten Sie die CSF
- Auftauen jedes Aliquot CSF auf Eis vor der RNA-Extraktion. 1 ug MS2 RNA Träger zu 0,5 ml CSF aufgetaut und vorsichtigmischen. Beachten Sie, dass, wenn die organischen Extraktions weggelassen wird, wird die MS2 Träger der in 1.2.2.1 unten beschrieben Probe vor der RNA-Isolierung hinzugefügt werden.
- Mischungs 0,5 ml 2x Denaturierungslösung bei Raumtemperatur zu der CSF.
- 1 ml der Säure-Phenol: Chloroform zu der CSF plus 2x Denaturierung Lösung: achten Sie darauf, den Boden entzogen Phase, die Säure-Phenol: Chloroform, nicht die wässrigen Puffer, der oben auf der Mischung liegt. Zusätzlich ist zu beachten, dass Säure-Phenol: Chloroform enthält Phenol, das ein Gift und ein Reizmittel, Schutzhandschuhe und andere persönliche Schutzausrüstung ist bei der Arbeit mit diesem Reagenz.
- Vortex für 30-60 Sek. mischen. Zur Vereinfachung sind die 2 ml der Probe / Denaturierungslösung / Säure-Phenol: Chloroform-Mischung in zwei 1,5 ml-Eppendorf-Röhrchen aufgeteilt.
- Zentrifugieren für 5 min bei maximaler Geschwindigkeit (10.000 × g) bei Raumtemperatur.
- Die wässrige (obere) Phase vorsichtig entfernen, ohne die untere Phase oder der Interphase zu stören und es auf einfrisches Röhrchen. Beachten Sie die Lautstärke zurückgewonnen.
1.2. RNA-Isolierung
Es wird empfohlen, eine eigene Bank, Satz Pipetten und Racks für den Umgang mit RNA-Proben zu haben. Der Arbeitsbereich und Werkzeuge werden durch die Verwendung RNase Zap Spray dekontaminiert und wischt vor jedem Experiment.
1.2.1. Bereiten Reagenzien:
- In 21 ml 100% Ethanol zu miRNA Waschlösung 1 und 40 ml 100% Ethanol zum Waschlösung 2/3. Bitte beachten Sie, dass miRNA Waschlösung 1 enthält Guanidiumthiocyanat, die eine potenziell gefährliche Substanz ist.
1.2.2. Gesamt-RNA Isolierung
- In 1,25 Volumina von Raumtemperatur 100% Ethanol zu der wässrigen Phase von der organischen Extraktion und gründlich mischen.
- Legen Sie eine Filterpatrone in einen der Röhrchen.
- Pipette nicht mehr als 700 ul des Lysats / Ethanol-Mischung auf die Filterpatrone.
- Zentrifugieren für 30 s bei maximalerGeschwindigkeit. Anwenden der Mischung von mehr als 700 &mgr; l in aufeinanderfolgenden Anwendungen auf dem gleichen Filter. Entsorgen Sie die Durchfluss nach jeder Zentrifugation und speichern Sie die Röhrchen für den Waschschritten.
- Bewerben 700 ul miRNA Waschlösung 1 auf die Filterpatrone und Zentrifuge für 15 sec bei maximaler Geschwindigkeit. Verwerfen Sie den Durchfluss durch von der Röhrchen und ersetzen Sie die Filterpatrone in die gleiche Röhrchen.
- Bewerben 500 ul Waschlösung 2/3 und Zentrifuge für 15 sec bei maximaler Geschwindigkeit. Verwerfen Sie den Durchfluss durch von der Röhrchen und ersetzen Sie die Filterpatrone in die gleiche Röhrchen. Tragen Sie eine zweite 500 ul Waschlösung 2/3 und Zentrifuge für 15 sec bei maximaler Geschwindigkeit. Verwerfen Sie den Durchfluss durch von der Röhrchen und ersetzen Sie die Filterpatrone in die gleiche Röhrchen. Drehen die Anordnung für 1 min bei Maximalgeschwindigkeit um Restfluid aus dem Filter zu entfernen.
- Übertragen Sie die Filterpatrone in eine frische Röhrchen. Bewerben 100 & mu; L vorgewärmten (95 ° C) Nuklease-freies Wasser auf die Mitte des Filters, und schließen Sie den Deckel. Zentrifugieren für 30 Sekunden bei maximaler Geschwindigkeit, um die RNA zu erholen.
- Sammeln Sie das Eluat, RNA und speichern sie bei -80 ° C für spätere Anwendungen.
- Quantifizieren 1 ul RNA unter Verwendung des Nanodrop 2000c Spektrophotometer. Beachten Sie, dass RNA-Quantifizierung kann ausgelassen werden, wenn eine RNA-Träger während der RNA-Extraktion der Probe zugegeben werden. In diesem Fall 8 ul RNA der cDNA-Synthese unterzogen, wie unten beschrieben.
2. miRNA-Profil: qRT-PCR-Protokoll
Das Protokoll für die miRNA Profilierung besteht aus zwei Schritten:
- Erststrang-cDNA-Synthese
- Echtzeit-PCR-Amplifikation
2.1. Erststrang-cDNA-Synthese
2.1.1. Verdünnen Matrizen-RNA
- Einzustellen jeder der Matrizen-RNA-Proben auf eine Konzentration von 5 ng / &mgr; l unter Verwendung von Nuklease freiem Wasser. Wenn derRNA wurde nicht quantifiziert (siehe Schritt 1.2.2.9), dann 8 ul RNA für die cDNA-Synthese verwendet
2.1.2. Bereiten Reagenzien
- Tauen Sie vorsichtig die 5x Reaktionspuffer und Nuklease freiem Wasser, und sofort auf Eis legen.
- Mischen durch Vortexen. Die RNA Spike-in durch Zugabe von 40 ul Nuklease freiem Wasser auf dem Rohr und vortexen, lassen Sie es auf Eis für 15-20 min. Unmittelbar vor der Anwendung zu entfernen, die das Enzym-Mix aus der Tiefkühltruhe, mischen durch Schwenken der Röhrchen und setzen auf Eis. Spin down alle Reagenzien.
2.1.3. Reverse-Transkriptions-Reaktion Setup
- Vorbereitung der erforderlichen Menge an RT-Arbeitslösung in einem 1,5 ml Eppendorf-Röhrchen oder dgl. (in den Anteilen in Tabelle 1 angegeben, mit Ausnahme der RNA-Matrize), vortexen (1-2 Sekunden bei Höchstgeschwindigkeit), kurz Spin-down ( verwenden wir eine Mini-Eppendorf-Zentrifuge mit Festdrehzahl, 10 sec) und legen Sie es auf Eis. Beachten Sie, dass die RNA UniSp6 Spitze-In vor der RT-Reaktion zu der Probe gegeben (siehe Tabelle 1).
- Dispense RT Arbeitslösung in nucleasefreiem PCR-Röhrchen.
- Verzichten Matrizen-RNA in jedes Röhrchen.
Hinweis: denken Sie daran, 10% Überschussvolumen / jedes Reagenz für Pipettier-und / oder Roboter-Totvolumen berechnen.Reagens Panel I Volumen (ul) Panel I + II Volumen (ul) 5x Reaktionspuffer 4.4 8.8 Nuklease freiem Wasser 9.9 19,8 Enzym-Mix 2.2 4.4 UniSp6 RNA Spike-In-Vorlage 1.1 2.2 Vorlage Gesamt-RNA (5 ng / ul) 4.4 8.8 Gesamtvolumen 22 44
Tabelle 1. Reverse-Transkriptions-Reaktion-Setup.
2.1.4. Mix-und Spin-Reagenzien
- Mischen Sie die Reaktion durch schon (1-2 sec) Vortexen, um sicherzustellen, daß alle Reagenzien werden gründlich gemischt. Nach dem Mischen Spin-Down (Mini Eppendorf-Zentrifuge mit Festdrehzahl, 10 sec).
2.1.5. Inkubieren und Wärme inaktivieren
- Programmieren Sie eine PCR-Maschine wie folgt:
- Inkubation für 60 min bei 42 ° C
- Wärme-Inaktivierung der reversen Transkriptase für 5 min bei 95 ° C
- Sofort kühlen auf 4 ° C
- Lagerung bei 4 ° C oder gefrier
Hinweis: Das Protokoll kann zu diesem Zeitpunkt unterbrochen werden. Die unverdünnte cDNA kann bei -20 ° C für bis zu 5 Wochen (optional bei 4 ° C bis zu 4 Tage) gehalten werden.
2.2.1. Bereiten Reagenzien für die Real-time PCR
- Zeigen cDNA (aus Schritt 2.1.5) auf Eis.
- Tauwetter Nuklease freiem Wasser und PCR Master Mix auf Eis. Schützen Sie die PCR Master Mix Röhrchen aus Licht, indem sie mit Alufolie abdecken. Unmittelbar vor der Anwendung mischen die PCR Master Mix durch Vortexen 1-2 sec in einem Mini-Zentrifuge und drehen sich mit 1.500 xg unten für 1 min.
2.2.2. Kombinieren Sie SYBR Green Master Mix, Wasser und cDNA und in den PCR-Platten
Folgende Vorgehensweise wird empfohlen, um niedrige Konzentrationen von cDNA aus den Rohroberfläche haften zu vermeiden:
- Vor dem Entfernen der Plattendichtung, kurz drehen Sie die Platte (n) in einer Kühlzentrifuge bei 1.500 g für 1 min.
- In einem 15 ml konischen Röhrchen verbinden 2x PCR Master Mix und Wasser. Panel I: 2,000 ul 2x Master-Mix und 1980 ul Wasser; Panel I + II: 4.000 ul 2x Master-Mix und 3960 μ; L Wasser.
- In 20 ul cDNA (Panel I) oder 40 ul cDNA (Panel I + II) und mischen.
- Vorsichtig mischen durch Vortexen 1-2 sec und Spin in einer Kühlzentrifuge bei 1.500 g für 1 min.
- Setzen Sie die PCR Master Mix / cDNA-Mix in einer Mehrkanalpipette Reservoir. Sie haben ein Reservoir, das die Länge des Mehrkanal-Pipette zu identifizieren. Dies hält den Pegel der Lautstärke hoch genug, so dass für gleiche Volumen Aliquots in der gesamten Mehrkanal. Dies ist kritisch gegenüber der letzten Teilmengen.
- Dispense 10 ul PCR Master Mix / cDNA mischen, um jede Vertiefung der 384-well-Platte mit einem 16-Kanal-Pipette.
- Verschließen Sie die Platte mit optischen Abdichtung.
- Spin Platte kurz in einer Kühlzentrifuge (1500 g für 1 min), um die Lösungen zu mischen, und entfernen Sie Luftblasen.
Hinweis: Das Experiment kann an dieser Stelle angehalten werden. Bewahren Sie die Reaktionen von Licht für bis zu 24 Stunden geschützt bei 4 ° C.
2.2.3. Echtzeit-PCR
Führen Sie Echtzeit-PCR-Amplifikation und Schmelzkurvenanalyse nach den in Tabelle 2 aufgeführten Parameter.
| Prozessschritt | Einstellungen, Roche LC480 |
| Polymerase Aktivierung / Denaturierung | 95 ° C, 10 min |
| Verstärkung | 45 Zyklen bei Amplification 95 ° C, 10 sec 60 ° C, 1 Min. Ramp-Rate 1,6 ° C / sec Optische Lese |
| Schmelzkurvenanalyse | Ja |
Tabelle 2. Echtzeit-PCR-Zyklusbedingungen mit dem Roche LightCycler 480.
2.3. Echtzeit-PCR-Daten-Analyse
Echtzeit-PCR-Daten-Analyse mit dem Exiqon Genex Professional 5.0 getan, nach ter empfahl Anweisungen. Der Genex Schritt-für-Schritt-Anleitung kann heruntergeladen werden http://www.exiqon.com/ls/Documents/Scientific/Exiqon-data-analysis-guide.pdf
Datenanalyse besteht aus drei Schritten:
- Import von Daten
- Vorverarbeitung von Daten
- Die statistische Analyse
2.3.1. Import von Daten
- In der Roche LightCycler 480 wählen Sie die 2. Ableitung Analyseverfahren und die Berechnung der Cq-Werte. Die Daten werden als Tabelle exportiert und als Textdateien gespeichert.
- Um die Daten, offene Genex importieren und klicken Sie auf den Exiqon Import-Assistenten und klicken Sie auf "Start". Folgen Sie den Anweisungen, um Format, Instrumenten-und Plattenlayout-Datei (en) auswählen. Beachten Sie, dass Platte Layout-Dateien (Excel-Dateien) werden von der Exiqon Website (heruntergeladen http://www.exiqon.com/plate-layout-files ).
- Bedeutungt Panels I und II und klicken Sie auf "Weiter".
- Das nach der Datei-Import erzeugte Tabelle enthält vordefinierte Spalten. Proben Namen können bearbeitet werden und Klassifikation Spalten hinzugefügt oder in diesem Schritt entfernt werden. Klicken Sie auf "Weiter", wenn Sie fertig sind.
- Daten speichern und laden, um Daten-Editor.
2.3.2. Vorverarbeitung von Daten
- Wie von Exiqon und Genex empfohlen, beim Vergleich mehrerer Platten das erste, was zu tun ist, um die Daten zwischen den Platten durch die Wahl unter-Plattenkalibrierung von der Vorverarbeitung Menü kalibrieren.
- Es wird empfohlen, miRNA-Arrays in dreifacher Ausfertigung Läufen gefahren. Wenn ein miRNA ist nicht vorhanden, in mindestens zwei von drei Replikaten es als nonexpressed eingestellt werden.
- Im nächsten Schritt wird in der Vorverarbeitung, stellen einen Schnitt aus. Eine Schnittwert festlegen, dass Daten mit einem Schwellenwert-Zyklus (C t) höher als dieser Wert, werden verworfen. In den vorliegenden Experimenten mit Rückenmarksflüssigkeit Probe wurde der Cut-off bei 39 gesetzt. Therefore werden alle Proben mit einem C t höher als 39 in den drei Replikate verworfen. Wenn ein miRNA einen C t> 39 für eine Sonde (eine Nachbildung von drei) wird, dass das Lesen mit dem Durchschnitt der C t für die anderen zwei Sonden ausgetauscht werden, sofern sie beide unter 39.
- Definieren Referenzgenen. Genex Professional verfügt über die Option, geNorm und / oder Normfinder nutzen, um Referenzgene zu identifizieren. Wählen Sie eine Liste von miRNAs, die ähnliche Cq-Werte über alle Proben haben und führen geNorm und / oder Normfinder. Nach dieser Analyse in dem hier angegebenen Beispiel miR-622 und miR-1266 hatte die gleichmäßige Werte für die Proben und wurden als Referenzgenen gewählt (Fig. 1).
- Als nächstes werden Daten unter Verwendung der Referenzgenen normalisiert und die erhaltenen Werte entsprechen der &Dgr; t
- Wenn die cDNA-Synthese-Reaktionen wurden dreifach durchgeführt, zu diesem Zeitpunkt die Werte gemittelt werden.
- Bestätigen Blatt erneutbewegen leer und fast leeren Spalten. In der Vorlauf Fenster wählen Sie "Bestätigen Blatt" und als auf der leeren Spalten löschen Linie gelten. In der Zeile darunter, wählte den gewünschten Prozentsatz der gültigen Daten und klicken Sie auf Anwenden. Wählen Sie zB 100%, wenn Sie nur miRNAs, die für alle Proben zu vergleichen wollen.
- Griff fehlenden Daten.
Wählen Sie "Fehlende Daten" aus der Vorverarbeitung Menü und wählte eine der Optionen, um fehlende Daten zu verarbeiten. Dieser Schritt ist erforderlich. Beachten Sie, dass Genex erhalten Sie Fehler, wenn Sie versuchen, Dateien, die nicht behandelte fehlenden Daten enthalten laden. - Bestimmen relative Quantifizierung zwischen Gruppen von Proben (dh. Experiment versus Kontrolle). Die relative Quantifizierung von Gruppen unter Verwendung des ΔΔ C t-Methode berechnet. In Genex, klicken Sie auf die Registerkarte Vorverarbeitung und wählen Sie die relative Quantifizierung. In dem Fenster wählen Sie die Referenzgruppe und schlug an. Beachten Sie, dass für den grafischen Darstellungen und / oder für weitere statistische Analysen, expression Daten sollten auf einer logarithmischen Skala umgewandelt werden. In der Vorlauf-Menü Log2.
2.3.3. Statistische Analyse
Genex-Software ermöglicht eine breite Palette von statistischen Analysen, einschließlich T-Test und ANOVA.
- Lade die endgültige MDF-Datei in Genex und offene Daten-Manager. Es ist wichtig, die korrekte Muster oder Gruppen von Proben, für die statistische Analyse durchgeführt wird ausgewählt.
- Wählen Sie "Statistik" und wählte das Symbol für den gewünschten Test.
- Klicken Sie auf "Ausführen" im Fenster der Systemsteuerung. Ergebnisse speichern als Excel-oder MDF-Dateien.
2.3.4. Expression-Profiling
MicroRNAs und Proben klassifiziert werden können, geclustert und auf hitze Karten und Dendrogrammen visualisiert, wie folgt
- Lade die endgültige MDF-Datei in Genex und offene Daten-Manager. Verwenden Sie dieses Fenster, um Gruppen von Proben oder miRNAs auswählen und erstellen. Klicken Sie auf "Übernehmen", wenn Sie fertig sind.
- In der linken oberen Ecke wählen Cluster und klicken Sie auf das Symbol Heatmap
- Im Kontrollfenster klicken Sie auf "Ausführen". Die Heatmap können in verschiedenen Formaten wie TIFF oder BMP gespeichert werden, es kann auch kopiert und modifiziert werden, wie gebraucht.
Ergebnisse
Die Ergebnisse dieser Studie wurden bereits veröffentlicht worden 1. Abbildung 1 zeigt die Ergebnisse aus der Analyse der Kandidatenreferenz microRNAs mit geNorm Anwendung. Dementsprechend zwei microRNAs, miR-622 und miR-1266, wurden als Referenzgene identifiziert und wurden verwendet, um miRNA Werte normalisieren.
Für die CSF-Studie hatten wir drei Gruppen von Proben: HIV-(n = 10), HIVE (n = 4) und HIV + ohne Enzephalitis (HIV +, n = 5). Die beiden Gruppen, HIVE und HIV +, wurden mit der Kontroll HIV-Gruppe verglichen. Nach der statistischen Analyse (Abschnitt 2.3.3) Expression von elf microRNAs wurde ein statistisch signifikant. Abbildung 2 stellt eine Box-Plot dieser elf microRNAs gefunden, miR-1203-1224-3p, -182 *,-19b-2 *, -204,-362-5p, -484, -720, -744 *, -934, -937 und. Jede Spalte zeigt die Verteilung der Daten über die Proben innerhalb der Gruppe (grün für die HIVE und rot für HIV + ohne Referenzphalitis). Whiskers zeigen den Median, den 1. (Q1) und 3. (Q3) Quartil, und die maximale und minimale nonoutliers.
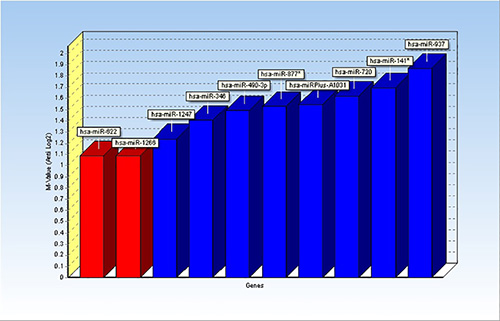
Fig. 1 ist. Grafik-Bar, welche die Ergebnisse von geNorm Anwendung innerhalb Genex. Zehn microRNAs wurden als mögliche Referenzgene analysiert und die Ergebnisse zeigen, miR-622 und miR-1266 (rote Balken) als die stabil exprimierten miRNAs. Klicken Sie hier für eine größere Ansicht.

2. Box Plot, der die Verteilung von Daten foder jeder der elf miRNAs in den Gruppen von HIVE (grün) und HIV + ohne Enzephalitis (HIV +, rot). Die Schnurrhaare in jeder Spalte zeigen den Median, 1. (Q1, Boden der Box) und 3. (Q3, oben der Box) Quartile, Maximal-und Minimalwerte, die nonoutliers (schwarze Linien) sind. Klicken Sie hier für eine größere Ansicht.
Diskussion
MicroRNAs sind kleine nicht-kodierende RNAs, die die Genexpression regulieren, durch die Hemmung der Translation und / oder die Förderung der mRNA-Abbau 2. Aufgrund ihrer hohen Stabilität in zellfreien Bedingungen haben microRNAs in vielen Körperflüssigkeiten nachgewiesen worden. Außerdem hat deutliche Expressionsprofil miRNAs mit Stufe und / oder Progression bei einer Vielzahl von menschlichen Krebsarten 3-9 korreliert.
Es gibt verschiedene Tools zur Verfügung, um microRNAs Profil, einschließlich der klassischen Chip-Arrays oder spätestens deep sequencing-Technologie. Jedoch entschieden wir uns, einen hochempfindlichen qPCR Plattform 10, 11, die den zusätzlichen Vorteil, dass eine minimale Menge von RNAs verwenden hat. Die miRCURY LNA Universal-microRNA RT-PCR-Protokoll ist darauf optimiert, verwenden 20 ng Gesamt-RNA pro 20 ul cDNA-Synthese-Reaktion, 40 ng für die volle Zwei-Panel-Arrays. Mit der Möglichkeit der Verwendung von kleinen RNA-Menge ist sehr wichtig, wenn es um valuabl e und schwer zu erhalten klinischen Proben wie CSF oder Formalin-fixierten und in Paraffin eingebetteten Gewebe ein. Wichtig ist, dass die Verwendung von geringen Mengen RNA Minimierung der Konzentration des in der Probe vorhandenen Inhibitoren möglich. Außerhalb des Bereichs Ct-Werte für die Spike-in wird wahrscheinlich die Anwesenheit von einigen Inhibitoren in der Probe. Spike-Sonden können separat erworben werden, um die Proben auf das Vorhandensein von Inhibitoren vor der miRNA-Profiling zu testen. Für diesen Test einzigen Echtzeit-PCR durchgeführt mit steigenden Konzentrationen von RNA (ng oder ob ul).
In dem vorliegenden Protokoll berichten wir über die organische Phase vor der Extraktion der RNA-Isolierung. Es ist zu beachten, dass viele neue RNA-Extraktion Kits dieses Verfahren nicht verlangen. Zum Beispiel haben wir erfolgreich Plasma miRNAs mit den profilierten miRCURY RNA Isolation Kit Biofluiden zur RNA-Extraktion direkt von 200 ul Plasma (Daten nicht gezeigt), ohne Phenol / Chloroform-Extraktion.
_content "> Im allgemeinen ist es wichtig, das Experiment im Voraus, um die richtige Anzahl von Wiederholungen erforderlich, um statistisch signifikante Ergebnisse zu erhalten, bestimmen zu entwerfen. Die Anzahl der Wiederholungen kann von der Anzahl der zu analysierenden Proben abhängen und kann auf die abhängen Variationen innerhalb der Proben oder Gruppe von Proben. Für unsere Studie, für die wir verwendet, eine relativ geringe Anzahl von Proben, insgesamt 20, entschieden wir uns in dreifacher Einrichten der cDNA-Synthese-Reaktion. Beratung mit einem Biostatistiker, bevor Sie die Experimente kann eine kluge Wahl sein und wird dringend empfohlen.Definieren der C t abgeschnitten Wert ist wichtig und hängt von der Art der Proben, für stark exprimierten miRNAs kann zwischen 25-35 eingestellt werden, aber für niedrige ausgedrückt miRNAs, wie unsere CSF Proben können höher eingestellt werden. Ein weiterer wichtiger Schritt bei Datenanalyse kommt, ist die Wahl der Referenz-Gen (en). Für einige robuste Daten (beispielsweise miRNA-Profiling in kultivierten Zellen) aglobal Normalisierung, die den Durchschnitts C t aller Proben repräsentiert, verwendet werden. Allerdings ist dies keine Option für Proben wie CSF, die Variationen zu präsentieren. Ebenso konnten wir uns nicht als Referenzgene diese miRNAs, die im Allgemeinen als unverändert in Körperflüssigkeiten zu nutzen. Stattdessen haben wir uns für alle miRNAs, die zeigten ähnliche C t in allen Proben, einschließlich Repliken, und lief die geNorm Analyse in Genex (Abbildung 1). Die Ergebnisse zeigten, miR-1266 und miR-622 als die am wenigsten variabel ausgedrückt miRNAs und sie als Referenzgene verwendet wurden. (Ct-Ctrel) - Vergleich der Expression von miRNAs zwischen Gruppen können über die relative Quantifizierung Werkzeug in Genex, die die Standardformel 2 folgt bestimmt werden. Schließlich können die Ergebnisse als Heatmap Diagramme, grafische Bars oder andere Arten von Plots, wie die Box-Plot in Abbildung 2 dargestellt werden. Andere Arten der Visualisierung in Genex gehören hierarchische Clustering,Self-Organized Map (SOM) und Hauptkomponentenanalyse (PCA). Zusätzlich zu miRNAs kann das Genex-Software für die Analyse von anderen Arten von Anordnungen, wie lange nichtkodierende RNAs (lncRNAs) Profilen verwendet werden. Die Plattenlayout kann in der Excel-Format importiert werden und die Daten können für die miRNA-Analyse beschrieben behandelt werden. In der Tat haben wir lncRNAs von primären embryonalen Maus kortikalen Neuronen mit der Mirvana miRNA Isolation Kit nach den oben beschriebenen Schritten profiliert und wir analysierten Daten mit Genex (unveröffentlichte Ergebnisse).
Zusammenfassend haben wir beschrieben, ein Protokoll zur Mikro-RNAs im Liquor Profil. Mit einigen Änderungen, um die RNA-Extraktion stehen, kann dieses Verfahren geeignet ist, miRNAs in anderen Körperflüssigkeiten und / oder eine andere Art von Geweben Profil werden. Beachten Sie, dass in dem hier beschriebenen Verfahren einen Schritt des organischen Extraktions vor der RNA-Isolierung durchgeführt. Im allgemeinen wird dies bei der Verwendung von kommerziell erhältlichen Kits erforderlich sind, wie die Mirvana Paris extraction-Kit. Zusätzlich wird bei der Extraktion von RNA aus Körperflüssigkeiten und eine Carrier-RNA zu den Proben gegeben gibt es keine Notwendigkeit, um die RNA zu quantifizieren und 2-8 &mgr; l RNA der cDNA-Synthese unterzogen. Aus unserer Erfahrung und in Anbetracht der hohen Kosten für diese Art von Experimenten verwandt, empfehlen wir testen einige Proben zuerst, und dann Rücksprache mit einem Statistiker für die Anzahl der Proben / Repliken, um statistisch signifikante Ergebnisse zu erhalten, profiliert werden.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Aus dem National Institute of Mental Health: Das beschriebene Projekt wurde von Preis Anzahl R01MH079751 (F. Peruzzi PI) unterstützt. Der Inhalt ist ausschließlich der Verantwortung der Autoren und nicht notwendigerweise die offizielle Meinung des National Institute of Mental Health oder des National Institute of Health.
Materialien
| Name | Company | Catalog Number | Comments |
Table 3. List of equipment. | |||
| Finnpipette Novus Multichannel Pipetter | Thermo Scientific | HH05279 | |
| Finntip Pipette Tips | Thermo Scientific | 9400613 | |
| Thermal Cycler C1000 | Biorad | ||
| 0.2 ml Low Profile, Clear PCR tubes | Biorad | TLS0801 | |
| Flat Cap Strips | Biorad | TLS0803 | |
LightCycler 480 Real-Time PCR System | Roche | ||
LightCycler 480 Sealing Foil | Roche | 04 729 757 001 | |
| Refrigerated Centrifuge 5804R | Eppendorf | ||
| Swing-bucket Rotor, 4-place | Eppendorf | A-4-44 | |
| Bench-Top Mini Centrifuge | Fisher | 05-090-100 | |
| Bench-Top Vortex | Fisher | ||
| 1.5 ml Microcentrifuge Tubes, Sterilized | Fisher | 02-681-5 | |
| Matrix Reagent Reservoir, 25 ml | Thermo Scientific | 8093 | |
| Thermomixer Confort | Eppendorf | ||
Bench-Top Mini Centrifuge Sigma 1-15 | Sigma | ||
| Bench-Top Vortex | Velp Scientifica | F202A0173 | |
Table 4. List of reagents. | |||
| Universal c-DNA synthesis kit, 16-32 rxns | Exiqon | 203300 | |
| SYBR Green master mix, Universal RT, 25 ml | Exiqon | 203400 | |
| Ultrapure Distilled Water Dnase, Rnase free | Invitrogen | 10977-015 | |
| MicroRNA Ready to use PCR Human Panels (I+II) V2.R | Exiqon | 203608 | |
mirVana miRNA isolation Kit, 40 isolations | Ambion | AM1556 | |
| MS2 RNA carrier | Roche Applied Science | 10165948001 | |
2-mercaptoethanol 98% | Sigma Aldrich | M3148-25ml | |
| Ethanol 100% | Carlo Erba | 64-17-5 | |
| Nuclease free water | Qiagen | 1039480 | |
RNAse Zap | Ambion | AM9780 | |
Referenzen
- Pacifici, M., et al. Cerebrospinal fluid miRNA profile in HIV-encephalitis. J. Cell. Physiol. 10, (2012).
- Krol, J., Loedige, I., Filipowicz, W. The widespread regulation of microRNA biogenesis, function and decay. Nat. Rev. Genet.. 11, 597-610 (2010).
- Calin, G. A., Croce, C. M. MicroRNA signatures in human cancers. Nat. Rev. Cancer. 6, 857-866 (2006).
- Chen, X., et al. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Res. 18, 997-1006 (2008).
- De Smaele, E., Ferretti, E., Gulino, A. MicroRNAs as biomarkers for CNS cancer and other disorders. Brain Res. 1338, 100-111 (2010).
- Heneghan, H. M., Miller, N., Kerin, M. J. MiRNAs as biomarkers and therapeutic targets in cancer. Curr. Opin. Pharmacol. 10, 543-550 (2010).
- Kosaka, N., Iguchi, H., Ochiya, T. Circulating microRNA in body fluid: a new potential biomarker for cancer diagnosis and prognosis. Cancer Sci. 101, 2087-2092 (2010).
- Lu, J., et al. MicroRNA expression profiles classify human cancers. Nature. 435, 834-838 (2005).
- Shah, A. A., Leidinger, P., Blin, N., Meese, E. miRNA: small molecules as potential novel biomarkers in cancer. Curr. Med. Chem. 17, 4427-4432 (2010).
- Blondal, T., et al. Assessing sample and miRNA profile quality in serum and plasma or other biofluids. Methods. 59, 1-6 (2013).
- Jensen, S. G., et al. Evaluation of two commercial global miRNA expression profiling platforms for detection of less abundant miRNAs. BMC Genomics. 12, 435 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten