Method Article
Liquido cerebrospinale MicroRNA profiling Utilizzando quantitativa Real Time PCR
In questo articolo
Riepilogo
Descriviamo un protocollo di PCR in tempo reale al profilo microRNA nel fluido cerebrospinale (CSF). Con l'eccezione di protocolli di estrazione di RNA, la procedura può essere estesa a RNA estratto da altri fluidi corporei, cellule coltivate, ei campioni di tessuto.
Abstract
MicroRNA (miRNA) costituiscono uno strato potente di regolazione genica guidando RISC a bersaglio siti posizionati su mRNA e, di conseguenza, modulando la loro repressione traduzionale. Cambiamenti nell'espressione miRNA hanno dimostrato di essere coinvolti nello sviluppo di tutte le principali malattie complesse. Inoltre, recenti scoperte hanno mostrato che i miRNAs possono essere secrete nell'ambiente extracellulare ed entrano nel flusso sanguigno e altri fluidi corporei dove possono circolare con elevata stabilità. La funzione di tale microRNA circolanti rimane in gran parte sfuggente, ma approcci sistematici ad alto rendimento, come miRNA array profiling, hanno portato all'identificazione di firme miRNA in diverse condizioni patologiche, tra cui le malattie neurodegenerative e alcuni tipi di tumori. In questo contesto, l'identificazione di miRNA profilo di espressione nel fluido cerebrospinale, come riportato nel nostro studio recente, rende miRNA candidati interessanti per analisi biomarker. _content "> Ci sono diversi strumenti disponibili per il profiling microRNA, come microarrays, quantitativa real-time PCR (qPCR), e sequenziamento profondo. Qui, descriviamo un metodo sensibile al profilo microRNA nel fluido cerebrospinale dal quantitativa real-time PCR. noi utilizzato i pannelli umani Exiqon microRNA pronti per l'uso PCR I e II V2.R, che consente il rilevamento di 742 unici microRNA umani. Abbiamo eseguito gli array in un parchetto in triplicato e abbiamo elaborato e analizzato i dati utilizzando il software GenEx Professional 5.
Usando questo protocollo, abbiamo profilato successo microRNA in vari tipi di linee cellulari e cellule primarie, CSF, plasma e tessuti inclusi in paraffina fissati in formalina.
Introduzione
MicroRNAs appartengono alla famiglia di piccole dimensioni (21-23 nt in lunghezza) RNA non codificanti che regolano l'espressione genica post-trascrizionale. microRNA possono essere secreti nello spazio extracellulare dove sembrano essere relativamente stabile. Mentre determinare cambiamenti nell'espressione miRNA può essere un passo importante verso l'identificazione di biomarcatori, eseguire profili miRNA e gestire una grande quantità di dati può essere intimidatorio.
Qui, descriviamo un protocollo per determinare cambiamenti nell'espressione miRNA nel liquido cerebrospinale (applicabile ad altri fluidi corporei) da un tempo reale sensibili PCR. Noi descriviamo anche l'uso di software per l'analisi dei dati, compresa l'analisi statistica e rappresentazione grafica dei risultati. L'intera procedura è relativamente semplice e lineare e, a seconda del numero di campioni da analizzare e il numero di macchine di PCR in tempo reale disponibili, anche relativamente veloce. La parte sperimentale richiede precisione nella movimentazioneRNA e pipettaggio in piastre da 384 pozzetti, mentre la sezione di analisi dei dati utilizzando GenEx richiede alcune conoscenze di base in informatica e statistica.
Protocollo
Il protocollo che segue descrive la procedura standard per isolare RNA da CSF e di profilo microRNA utilizzando piastre PCR pronti. Si noti che l'estrazione fase organica è facoltativo, e che un RNA carrier viene aggiunta al campione durante l'estrazione garantire massimo recupero di RNA. Di conseguenza, non vi è alcuna necessità di quantificare l'RNA.
Nel complesso, una media di 6-8 piastre può essere eseguito in un giorno se il cDNA viene sintetizzato il giorno prima (circa 2 ore). L'analisi dei dati viene eseguita quando tutti i campioni sono stati profilato e caricato nel software. A seconda del numero di campioni / gruppi o loro combinazione per il confronto, l'analisi dei dati può richiedere da una a diverse ore.
1. RNA Estrazione
L'estrazione di RNA è stata eseguita da campioni di CSF, congelati a -80 ° C in aliquote fino all'analisi. Per questa procedura è stato utilizzato il kit di isolamento Mirvana Parigi, seguendo strumento del produttoreZIONI per l'isolamento di RNA totale. Si prega di notare che, sebbene l'arricchimento per le piccole specie di RNA è possibile con questo kit, questo passaggio non è stato fatto e la procedura di estrazione RNA è conclusa dopo la fase di totale isolamento di RNA. Inoltre, sebbene il kit di isolamento Mirvana Parigi (come altri kit di isolamento dell'RNA disponibili in commercio) non richiede estrazione organico, un protocollo per questa procedura sono disponibili sotto.
Il protocollo per l'estrazione dell'RNA consiste di due fasi:
1.1. Estrazione biologico (non necessario se si utilizza Mirvana Parigi kit di estrazione RNA)
1.2.2. Isolamento RNA totale
1.1. Estrazione Organic
1.1.1. Preparare i reagenti
- Aggiungere 375 ml di 2-mercaptoetanolo a 2x Denaturing soluzione, e conservarlo a 4 ° C.
1.1.2. Preparare il CSF
- Scongelare ogni aliquota di CSF sul ghiaccio prima dell'estrazione dell'RNA. Aggiungere 1 mg di carrier RNA MS2 a 0,5 ml di liquor scongelato e delicatamentemescolare. Si noti che se l'estrazione organica viene omesso, il vettore MS2 sarà aggiunto al campione prima dell'isolamento dell'RNA descritto in 1.2.2.1 seguito.
- Mescolare 0,5 ml di 2x denaturazione soluzione a temperatura ambiente al CSF.
- Aggiungere 1 ml di acido-fenolo: cloroformio al CSF più il 2x denaturazione Soluzione: assicurarsi di ritirata la fase inferiore contenente acido-fenolo: cloroformio, non il tampone acquoso che si trova sulla parte superiore della miscela. Inoltre, si noti che l'acido-fenolo: cloroformio contiene fenolo, che è un veleno e un irritante, usare guanti ed altri dispositivi di protezione individuale quando si lavora con questo reagente.
- Vortex per 30-60 secondi per mescolare. Per comodità, i 2 ml di campione / denaturazione soluzione / acido-fenolo: cloroformio mix sono divisi in due provette Eppendorf da 1,5 ml.
- Centrifugare per 5 minuti a velocità massima (10.000 xg) a temperatura ambiente.
- Rimuovere con attenzione la fase acquosa (superiore) senza disturbare la fase inferiore o interfase e trasferirlo in unprovetta. Annotare il volume recuperato.
1.2. Isolamento dell'RNA
Si raccomanda di avere un banco dedicato, set di pipette e rastrelliere per la gestione di campioni di RNA. L'area di lavoro e gli strumenti sono decontaminati mediante RNasi Zap spray e salviette prima di ogni esperimento.
1.2.1. Preparare i reagenti:
- Aggiungere 21 ml di etanolo al 100% miRNA Soluzione di Lavaggio 1 e 40 ml di etanolo al 100% di soluzione di lavaggio 2/3. Si prega di notare che il miRNA soluzione di lavaggio 1 contiene guanidio thiocyanate che è una sostanza potenzialmente pericolosa.
1.2.2. Isolamento RNA totale
- Aggiungere 1,25 volumi di temperatura ambiente 100% di etanolo alla fase acquosa dall'estrazione organica e mescolare accuratamente.
- Posizionare una cartuccia filtrante in uno dei tubi di raccolta.
- Pipette non più di 700 ml di miscela lisato / etanolo sulla cartuccia del filtro.
- Centrifugare per 30 secondi alla massimavelocità. Applicare la miscela superiore 700 microlitri nelle applicazioni successive allo stesso filtro. Gettare il flow-through dopo ogni centrifugazione e salvare il tubo di raccolta per le fasi di lavaggio.
- Applicare 700 microlitri miRNA Wash Solution 1 alla cartuccia del filtro e centrifugare per 15 secondi alla massima velocità. Gettare il flusso attraverso dal tubo di raccolta e sostituire la cartuccia del filtro nello stesso tubo di raccolta.
- Applicare 500 microlitri soluzione di lavaggio 2/3 e centrifugare per 15 secondi alla massima velocità. Gettare il flusso attraverso dal tubo di raccolta e sostituire la cartuccia del filtro nello stesso tubo di raccolta. Applicare un secondo da 500 microlitri soluzione di lavaggio 2/3 e centrifugare per 15 secondi alla massima velocità. Gettare il flusso attraverso dal tubo di raccolta e sostituire la cartuccia del filtro nello stesso tubo di raccolta. Spin il gruppo per 1 min a velocità massima per rimuovere residui di liquido dal filtro.
- Trasferire la cartuccia del filtro in un tubo di raccolta fresca. Applicare 100 & mu; L preriscaldata (a 95 ° C) acqua priva di nucleasi al centro del filtro, e chiudere il tappo. Centrifugare per 30 secondi a velocità massima per recuperare il RNA.
- Raccogliere l'eluato contenente RNA e conservarlo a -80 ° C per le applicazioni successive.
- Quantificare 1 ml di RNA utilizzando lo spettrofotometro NanoDrop 2000C. Si noti che la quantificazione RNA può essere saltata se un vettore RNA viene aggiunta al campione durante l'estrazione di RNA. In tal caso 8 pl di RNA sono sottoposti a sintesi di cDNA come descritto di seguito.
2. miRNA Profilo: qRT-PCR Protocol
Il protocollo per miRNA profiling consiste di due fasi:
- First-strand sintesi del DNA
- Real-time PCR di amplificazione
2.1. First-strand sintesi del DNA
2.1.1. Diluire stampo di RNA
- Regolare ciascuno dei campioni di RNA template ad una concentrazione di 5 ng / ml con acqua nucleasi gratuito. Se l'RNA non è stato quantificato (vedere fase 1.2.2.9), poi 8 pl di RNA sono utilizzati per la sintesi di cDNA
2.1.2. Preparare i reagenti
- Scongelare delicatamente il tampone di reazione 5x e nucleasi acqua libera, e mettere immediatamente in ghiaccio.
- Mescolare nel vortex. Risospendere il RNA spike-in con l'aggiunta di acqua priva di 40 microlitri di nucleasi al tubo e mescolare nel vortex, lasciare in ghiaccio per 15-20 min. Immediatamente prima dell'uso, rimuovere il mix enzima dal freezer, mescolare sfogliando le provette e disporli sul ghiaccio. Spin giù tutti i reagenti.
2.1.3. Configurazione inversa reazione di trascrizione
- Preparare la quantità richiesta di soluzione di lavoro RT in una provetta Eppendorf da 1,5 ml o equivalente (nelle proporzioni indicate nella Tabella 1, tranne per il modello RNA), mescolare nel vortex (1-2 sec max velocità), brevemente centrifugare ( usiamo un mini Eppendorf centrifuga con velocità fissa, 10 sec) e posizionarlo sul ghiaccio. Si noti che l'RNA picco UniSp6-In viene aggiunto al campione prima della reazione RT (vedi Tabella 1).
- Dispensare soluzione di lavoro RT in provette PCR priva di nucleasi.
- Pipettare stampo di RNA in ciascun tubo.
Nota: ricordatevi di calcolare il 10% di volume in eccesso / ogni reagente per pipettaggio e / o volume morto robotica.Reagente Panel I Volume (ml) Panel I + II Volume (ml) Tampone di reazione 5x 4.4 8.8 Acqua nucleasi libero 9.9 19.8 Mix enzimatico 2.2 4.4 UniSp6 RNA Spike-in template 1.1 2.2 Template RNA totale (5 ng / ml) 4.4 8.8 Volume totale 22 44
Tabella 1. Invertire la configurazione reazione di trascrizione.
2.1.4. Mescolare e far girare i reagenti
- Mescolare la reazione delicatamente (1-2 sec) vortex per assicurare che tutti i reagenti siano ben amalgamati. Dopo la miscelazione, spin down (mini centrifuga Eppendorf con velocità fissa, 10 sec).
2.1.5. Incubare e inattivare a caldo
- Programmare una macchina PCR come segue:
- Incubare per 60 min a 42 ° C
- Calore inattivare la trascrittasi inversa per 5 minuti a 95 ° C
- Immediatamente raffreddare a 4 ° C
- Conservare a 4 ° C o congelare
Nota: il protocollo può essere interrotto in questa fase. Il cDNA diluito può essere conservato a -20 ° C per 5 settimane (negozio opzionale a 4 ° C per 4 giorni).
2.2.1. Preparare i reagenti per Real-time PCR
- Posizionare cDNA (dal punto 2.1.5) sul ghiaccio.
- Scongelare acqua libera e PCR Master mix nucleasi sul ghiaccio. Proteggere le fiale master mix per PCR dalla luce coprendoli con un foglio di alluminio. Immediatamente prima dell'uso, mescolare la miscela master PCR vortex 1-2 secondi in una mini centrifuga e centrifugare a 1500 xg per 1 min.
2.2.2. Combina SYBR Green Master Mix, acqua e cDNA e aggiungere piastre PCR
La seguente procedura è consigliata per evitare basse concentrazioni di cDNA di aderire alla superficie del tubo:
- Prima di rimuovere il sigillo piastra, brevemente centrifugare la piastra (s) in una centrifuga refrigerata a 1.500 xg per 1 min.
- In un tubo da 15 ml, unire 2x PCR miscela master e acqua. Panel I: 2,000 microlitri 2x master mix e 1980 microlitri di acqua; Panel I + II: 4,000 microlitri 2x master mix e 3960 μ, L acqua.
- Aggiungere 20 microlitri di cDNA (pannello I) o 40 microlitri di cDNA (pannello di I + II) e mescolare.
- Mescolare delicatamente vortex 1-2 sec e spin down in una centrifuga refrigerata a 1.500 xg per 1 min.
- Posizionare la PCR Master mix / cDNA mix in una pipetta multicanale serbatoio. Potrebbe essere necessario identificare un serbatoio che ha la lunghezza della pipetta multicanale. Ciò mantiene il livello di volume di abbastanza alto consentendo aliquote volume uguale in tutto il multicanale. Questo è critico verso gli ultimi aliquote.
- Pipettare 10 microlitri miscela master PCR / cDNA miscela in ciascun pozzetto della piastra a 384 pozzetti usando una pipetta a 16 canali.
- Sigillare la piastra con guarnizione ottica.
- Spin piastra brevemente in una centrifuga refrigerata (1.500 xg per 1 min), per mescolare le soluzioni e rimuovere le bolle d'aria.
Nota: l'esperimento può essere sospeso, a questo punto. Conservare le reazioni al riparo dalla luce a 4 ° C per un massimo di 24 ore.
2.2.3. Real-Time PCR
Eseguire real-time PCR amplificazione e analisi della curva di melting secondo i parametri descritti nella tabella 2.
| Fase del processo | Impostazioni, Roche LC480 |
| Polymerase Attivazione / denaturazione | 95 ° C, 10 min |
| Amplificazione | 45 cicli di amplificazione a 95 ° C, 10 sec 60 ° C, 1 min Ramp-rate 1,6 ° C / sec Lettura ottica |
| Melting analisi della curva | Sì |
Tabella 2. In tempo reale le condizioni del ciclo di PCR utilizzando la Roche LightCycler 480.
2.3. Analisi in tempo reale dei dati di PCR
In tempo reale analisi dei dati PCR è fatto con l'Exiqon GenEx Professional 5.0, a seguito di tha raccomandato istruzioni. Il manuale GenEx step-by-step può essere scaricato all'indirizzo http://www.exiqon.com/ls/Documents/Scientific/Exiqon-data-analysis-guide.pdf
L'analisi dei dati è costituito da tre fasi:
- Importazione di dati
- Pre-elaborazione dei dati
- L'analisi statistica
2.3.1. Importazione di dati
- Nel Roche LightCycler 480 selezionare il metodo di analisi nd derivato 2 e calcolare i valori Cq. I dati vengono esportati come un tavolo e salvati come file di testo.
- Per importare i dati, aprire GenEx e fare clic sul pulsante di importazione guidata Exiqon e fare clic su "Start". Seguire le istruzioni per selezionare il formato, strumento e file di layout di piastra (s). Si noti che i file di layout piastra (file excel) vengono scaricati dal sito web Exiqon ( http://www.exiqon.com/plate-layout-files ).
- Import Pannelli I e II e fare clic su "Avanti".
- La tabella generata dopo file di importazione contiene colonne predefinite. I nomi dei campioni possono essere modificati e le colonne di classificazione possono essere aggiunti o rimossi in questa fase. Fare clic su "Next" quando avete finito.
- Salvare i dati e caricare Editor dei dati.
2.3.2. Pre-elaborazione dei dati
- Come raccomandato dal Exiqon e GenEx, quando si confrontano più lastre la prima cosa da fare è quella di calibrare i dati tra le piastre scegliendo calibrazione inter-piastra dal menu pre-elaborazione.
- Si raccomanda di eseguire le matrici miRNA nei funzionamenti triplicato. Se un miRNA non è presente in almeno due delle tre repliche sarà impostato come nonexpressed.
- Nella fase successiva nella pre-elaborazione, impostare un cut-off. Definizione di un valore di cut off indica che i dati con un ciclo soglia (C t) superiore a questo valore vengono scartati. Nel presente esperimenti usando campione di fluido cerebrospinale, la off taglio è stato fissato a 39. Cirima, verranno eliminati tutti i campioni con C t superiore a 39 nelle tre repliche. Se un miRNA ha un C t> 39 per una sonda (una replica su tre), che la lettura verrà sostituito con la media del C t per le altre due sonde, a condizione che siano entrambi sotto 39.
- Definire geni di riferimento. GenEx Professional ha la possibilità di utilizzare geNorm e / o NormFinder per identificare i geni di riferimento. Selezionare una lista di miRNA che hanno valori simili Cq in tutti i campioni e corrono geNorm e / o NormFinder. Secondo questa analisi, nell'esempio fornito qui, miR-622 e miR-1266 avevano valori più uniformi per i vari campioni e sono stati scelti come geni di riferimento (Figura 1).
- Successivamente, i dati vengono normalizzati utilizzando i geni di riferimento ei valori ottenuti corrispondono alla ΔC t
- Se reazioni di sintesi di cDNA sono stati eseguiti in triplicato, a questo punto i valori devono essere mediate.
- Convalida foglio di rispostare le colonne vuote e quasi vuote. Nella finestra di pre-elaborazione selezionare "Convalida patrimoniale" e di applicare sulla Remove colonne vuote linea. Nella riga sotto, ha scelto la percentuale desiderata di dati validi e fare clic su Applica. Ad esempio, selezionare 100%, se si vuole confrontare solo miRNA comuni a tutti i campioni.
- Gestire i dati mancanti.
Selezionare "dati mancanti" dal menu Preprocessing e ha scelto una delle opzioni per gestire i dati mancanti. Questo passaggio è necessario. Si noti che GenEx vi darà errore se si tenta di caricare file che contengono dati mancanti non gestite. - Determinare la quantificazione relativa tra gruppi di campioni (per esempio. Esperimento rispetto al controllo). La quantificazione relativa tra i gruppi è calcolato utilizzando il metodo t ΔΔ C. In GenEx, fare clic sulla scheda di pre-elaborazione e selezionare quantificazione relativa. Nella finestra selezionare il gruppo di riferimento e premi applica. Si noti che per rappresentazioni grafiche e / o per ulteriori analisi statistiche, exprdati essione devono essere convertiti in una scala logaritmica. Nel menu di pre-elaborazione, selezionare log2.
2.3.3. Analisi statistica
Software GenEx consente una vasta gamma di analisi statistiche, incluse T-test e ANOVA.
- Caricare il file mdf finale in GenEx e responsabile dei dati aperti. E 'fondamentale per selezionare i campioni o gruppi corretti di campioni per cui è eseguita l'analisi statistica.
- Selezionare "Statistiche" e scegliere l'icona corrispondente alla prova desiderata.
- Fare clic su "Esegui" nella finestra del pannello di controllo. Salvare i risultati come file Excel o mdf.
2.3.4. Profili di espressione
I microRNA e campioni possono essere classificati, raggruppati e visualizzati sul calore mappe e dendrogrammi, come segue
- Caricare il file mdf finale in GenEx e responsabile dei dati aperti. Utilizzare questa finestra per selezionare e creare gruppi di campioni o miRNA. Fare clic su "Applica" quando hai finito.
- In alto a sinistra, selezionare grappolo superiore e fare clic sull'icona heatmap
- Nella finestra Pannello di controllo cliccare su "Esegui". Il heatmap può essere salvato in vari formati come tiff o bmp, ma può anche essere copiato e modificato in base alle esigenze.
Risultati
I risultati di questo studio sono stati pubblicati in precedenza 1. Figura 1 mostra i risultati dell'analisi dei microRNA riferimento candidati usando l'applicazione di geNorm. Di conseguenza, due microRNA, miR-622 e miR-1266, sono stati identificati i geni di riferimento e sono stati usati per normalizzare i valori di miRNA.
Per lo studio QCS avevamo tre gruppi di campioni: da HIV (n = 10), HIVE (n = 4), e l'HIV +, senza encefalite (HIV +, n = 5). I due gruppi, HIVE e HIV +, sono stati confrontati con il controllo di HIV-gruppo. Dopo l'analisi statistica (punto 2.3.3) livelli di espressione di undici microRNA è stato trovato statisticamente significativo 1. Figura 2 rappresenta un diagramma a riquadri di queste undici microRNA, miR-1203-1224-3p, -182 *,-19b-2 *, -204,-362-5p, -484, -720, -744 *, -934, -937 e. Ogni colonna mostra la distribuzione dei dati attraverso dei campioni all'interno del gruppo (verde per la HIVE e rosso per HIV +, senza mentophalitis). Whiskers indicano la mediana, il 1 ° (Q1) e 3 ° (Q3) quartile, e il massimo e nonoutliers minimi.
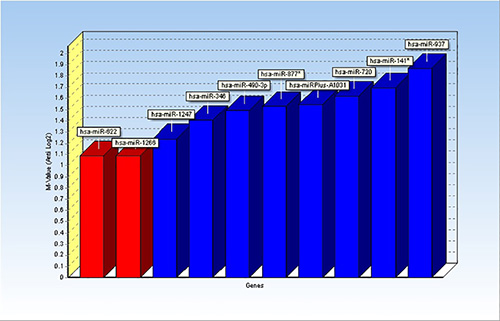
Figura 1. Barra grafica mostra i risultati di applicazione geNorm all'interno GenEx. Dieci microRNA sono stati analizzati come possibili geni di riferimento ei risultati indicano miR-622 e miR-1266 (barre rosse) come i miRNA più stabilmente espresse. Clicca qui per vedere l'immagine ingrandita.

Figura 2. Box plot che mostra la distribuzione dei dati fo di ciascuna delle undici miRNA all'interno dei gruppi di HIVE (verde) e HIV + senza encefalite (HIV +, rosso). I baffi in ogni colonna indicano la mediana, 1 ° (Q1, parte inferiore della scatola) e 3 ° (Q3, top della scatola) quartili, i valori massimi e minimi che sono nonoutliers (linee nere). Clicca qui per vedere l'immagine ingrandita.
Discussione
MicroRNA sono piccoli RNA non codificanti che regolano l'espressione genica inibendo la traduzione e / o la promozione di degradazione dell'mRNA 2. Grazie alla loro elevata stabilità in condizioni cell-free, microRNA sono stati rilevati in molti fluidi corporei. Inoltre, il profilo di espressione dei microRNA distinta è stata correlata con la fase e / o la progressione in una varietà di tumori umani 3-9.
Ci sono diversi strumenti a disposizione al profilo microRNA, compresi gli array di chip classiche o le più recenti tecnologie di sequenziamento profondo. Tuttavia, abbiamo deciso di utilizzare una piattaforma altamente sensibile qPCR 10, 11, che ha l'ulteriore vantaggio di richiedere minima quantità di RNA. Il protocollo miRCURY LNA universale RT microRNA PCR è ottimizzato per utilizzare 20 ng RNA totale per 20 microlitri di cDNA reazione di sintesi, 40 ng per l'allineamento completo di due pannelli. Avere la possibilità di utilizzare piccole quantità di RNA è molto importante quando si tratta di valuabl e di campioni clinici difficili da ottenere come CSF o inclusi in paraffina tessuti fissati in formalina 1. È importante sottolineare che, utilizzando piccole quantità di RNA minimizzare la concentrazione di possibili inibitori presenti nel campione. Fuori range valori Ct per il picco-in probabilmente indicare la presenza di alcuni inibitori nel campione. Sonde Spike-in possono essere acquistati separatamente per testare i campioni per la presenza di inibitori prima di miRNA profiling. Per questa prova, singolo tempo reale PCR vengono eseguite utilizzando concentrazioni crescenti di RNA (se ng o microlitri).
Nel presente protocollo, si segnala l'estrazione fase organica prima di isolamento dell'RNA. Va notato che molti nuovi kit di estrazione di RNA non richiedono questa procedura. Per esempio, abbiamo profilato successo miRNA plasma utilizzando i miRCURY RNA kit di isolamento biofluids per l'estrazione dell'RNA direttamente da 200 ml di plasma (dati non riportati), senza estrazione fenolo / cloroformio.
_content "> In generale, è importante progettare l'esperimento in anticipo al fine di determinare il corretto numero di repliche necessarie per ottenere risultati statisticamente significativi. Le repliche può dipendere dal numero di campioni da analizzare e può dipendere dal variazioni nei campioni o gruppo di campioni. Per il nostro studio, per il quale abbiamo utilizzato un numero relativamente basso di campioni, 20 in totale, abbiamo deciso di istituire la reazione di sintesi del DNA in triplicato. Consulting con un biostatistico prima di impostare gli esperimenti possono essere una scelta saggia ed è altamente raccomandato.Definizione del C t tagliato valore è importante e dipende dal tipo di campioni, per miRNA altamente espressi può essere impostato tra 25-35, ma per miRNA espressi basse, come i nostri campioni di LCR, può essere superiore. Un altro punto critico quando si tratta di analisi dei dati è la scelta del gene di riferimento (s). Per alcuni dati solidi (come miRNA profilatura in cellule in coltura) agnormalizzazione lobal, che rappresenta la C t media di tutti i campioni, può essere utilizzato. Tuttavia, questo non è un'opzione per campioni come CSF che presentano variazioni. Allo stesso modo, non abbiamo potuto utilizzare come geni di riferimento quei miRNA che sono generalmente considerati invariati nei fluidi corporei. Invece, abbiamo selezionato tutti i miRNA che hanno mostrato simile C t in tutti i campioni, tra cui repliche, e corremmo l'analisi geNorm disponibile in GenEx (Figura 1). I risultati hanno indicato miR-1266 e miR-622 come il meno miRNA variamente espresse e sono stati utilizzati come geni di riferimento. Confronto dell'espressione di miRNA tra gruppi può essere determinata utilizzando lo strumento quantificazione relativa in GenEx, che segue la formula standard 2 - (Ct-Ctrel). Infine, i risultati possono essere visualizzati come sugli calore diagrammi, barre grafiche o altri tipi di trame, come la trama casella in Figura 2. Altri tipi di visualizzazione disponibili in GenEx includono clustering gerarchico,Self-Organized Map (SOM), e Principal Component Analysis (PCA). Oltre alla miRNA, il software GenEx può essere utilizzato per l'analisi di altri tipi di matrici come lunghi RNA non codificanti (lncRNAs) profilatura. Il layout piastra può essere importato in formato excel e dati possono essere gestiti come descritto per l'analisi miRNA. Infatti, abbiamo profilato lncRNAs da topo primarie di neuroni corticali embrionali utilizzando il kit di isolamento Mirvana miRNA come descritto nei passaggi precedenti e abbiamo analizzato i dati utilizzando GenEx (risultati non pubblicati).
In sintesi, abbiamo descritto un protocollo al profilo microRNA nel fluido cerebrospinale. Con alcune modifiche relative a estrazione di RNA, questa procedura può essere adattato al profilo miRNAs in altri fluidi corporei e / o altri tipi di tessuti. Si noti che nella procedura qui descritta, una fase di estrazione organica viene eseguita prima dell'isolamento dell'RNA. In generale, questo non è necessario con i kit disponibili in commercio, come la ext Mirvana Parigikit raction. Inoltre, durante l'estrazione di RNA da fluidi corporei e un RNA carrier è aggiunto ai campioni non è necessario quantificare l'RNA e 2-8 microlitri RNA sono sottoposti a sintesi di cDNA. Dalla nostra esperienza e considerando gli alti costi legati a questo tipo di esperimenti, si consiglia di testare alcuni campioni prima, e poi di consulenza con un esperto di statistica per il numero di campioni / repliche da profilati in modo da ottenere risultati statisticamente significativi.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Il progetto descritto è stato supportato da Award numero R01MH079751 (PI: F. Peruzzi) del National Institute of Mental Health. Il contenuto è di esclusiva responsabilità degli autori e non rappresentano necessariamente il punto di vista ufficiali dell'Istituto Nazionale di Salute Mentale o il National Institute of Health.
Materiali
| Name | Company | Catalog Number | Comments |
Table 3. List of equipment. | |||
| Finnpipette Novus Multichannel Pipetter | Thermo Scientific | HH05279 | |
| Finntip Pipette Tips | Thermo Scientific | 9400613 | |
| Thermal Cycler C1000 | Biorad | ||
| 0.2 ml Low Profile, Clear PCR tubes | Biorad | TLS0801 | |
| Flat Cap Strips | Biorad | TLS0803 | |
LightCycler 480 Real-Time PCR System | Roche | ||
LightCycler 480 Sealing Foil | Roche | 04 729 757 001 | |
| Refrigerated Centrifuge 5804R | Eppendorf | ||
| Swing-bucket Rotor, 4-place | Eppendorf | A-4-44 | |
| Bench-Top Mini Centrifuge | Fisher | 05-090-100 | |
| Bench-Top Vortex | Fisher | ||
| 1.5 ml Microcentrifuge Tubes, Sterilized | Fisher | 02-681-5 | |
| Matrix Reagent Reservoir, 25 ml | Thermo Scientific | 8093 | |
| Thermomixer Confort | Eppendorf | ||
Bench-Top Mini Centrifuge Sigma 1-15 | Sigma | ||
| Bench-Top Vortex | Velp Scientifica | F202A0173 | |
Table 4. List of reagents. | |||
| Universal c-DNA synthesis kit, 16-32 rxns | Exiqon | 203300 | |
| SYBR Green master mix, Universal RT, 25 ml | Exiqon | 203400 | |
| Ultrapure Distilled Water Dnase, Rnase free | Invitrogen | 10977-015 | |
| MicroRNA Ready to use PCR Human Panels (I+II) V2.R | Exiqon | 203608 | |
mirVana miRNA isolation Kit, 40 isolations | Ambion | AM1556 | |
| MS2 RNA carrier | Roche Applied Science | 10165948001 | |
2-mercaptoethanol 98% | Sigma Aldrich | M3148-25ml | |
| Ethanol 100% | Carlo Erba | 64-17-5 | |
| Nuclease free water | Qiagen | 1039480 | |
RNAse Zap | Ambion | AM9780 | |
Riferimenti
- Pacifici, M., et al. Cerebrospinal fluid miRNA profile in HIV-encephalitis. J. Cell. Physiol. 10, (2012).
- Krol, J., Loedige, I., Filipowicz, W. The widespread regulation of microRNA biogenesis, function and decay. Nat. Rev. Genet.. 11, 597-610 (2010).
- Calin, G. A., Croce, C. M. MicroRNA signatures in human cancers. Nat. Rev. Cancer. 6, 857-866 (2006).
- Chen, X., et al. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Res. 18, 997-1006 (2008).
- De Smaele, E., Ferretti, E., Gulino, A. MicroRNAs as biomarkers for CNS cancer and other disorders. Brain Res. 1338, 100-111 (2010).
- Heneghan, H. M., Miller, N., Kerin, M. J. MiRNAs as biomarkers and therapeutic targets in cancer. Curr. Opin. Pharmacol. 10, 543-550 (2010).
- Kosaka, N., Iguchi, H., Ochiya, T. Circulating microRNA in body fluid: a new potential biomarker for cancer diagnosis and prognosis. Cancer Sci. 101, 2087-2092 (2010).
- Lu, J., et al. MicroRNA expression profiles classify human cancers. Nature. 435, 834-838 (2005).
- Shah, A. A., Leidinger, P., Blin, N., Meese, E. miRNA: small molecules as potential novel biomarkers in cancer. Curr. Med. Chem. 17, 4427-4432 (2010).
- Blondal, T., et al. Assessing sample and miRNA profile quality in serum and plasma or other biofluids. Methods. 59, 1-6 (2013).
- Jensen, S. G., et al. Evaluation of two commercial global miRNA expression profiling platforms for detection of less abundant miRNAs. BMC Genomics. 12, 435 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon