Method Article
Click Chemistry and Expansion Microscopy를 사용한 세포 내 사이알릴화 시각화
요약
여기에서는 대사 올리고당 엔지니어링, 클릭 화학 및 확장 현미경 검사를 결합한 간단한 프로토콜을 제안하며, 이를 통해 일상적인 현미경 장비를 사용하여 향상된 해상도로 세포 내 시알릴화된 N-당단백질의 바이오이미징을 가능하게 합니다.
초록
대사 라벨링 기술을 통해 생체 직교 리포터를 글라이칸에 통합할 수 있으며, 클릭 및 생체 직교 화학을 통해 세포 내 분자 염료의 표적 생체 접합을 가능하게 합니다. 대사 올리고당 공학(MOE)은 분자 인식과 관련된 수많은 생물학적 과정에서 글리코실화의 필수적인 역할과 암에서 유전 질환, 바이러스 및 박테리아 감염에 이르는 병리학에 미치는 영향으로 인해 상당한 관심을 끌었습니다.
MOE는 세포 표면 당접합체(glycoconjugates)의 검출로 더 잘 알려져 있지만, 생리학적 및 병리학적 맥락에서 세포 내 글라이칸 연구를 위한 매우 중요한 방법론이기도 합니다. 이러한 연구는 높은 공간 해상도의 이점을 크게 활용할 수 있습니다. 그러나 초고해상도 현미경 검사는 대부분의 실험실에서 쉽게 사용할 수 없으며 일상적인 구현에 어려움을 겪고 있습니다. 확장 현미경 검사는 형광 마커로 표지된 생물학적 표본을 물리적으로 확대하여 현미경 검사의 해상도를 향상시키는 최신 대안입니다. 샘플을 팽창 가능한 겔에 삽입하고 화학적 처리를 통해 등방성으로 팽창하게 함으로써 초고해상도 기술 없이도 향상된 정밀도와 해상도로 세포 내 구조를 시각화할 수 있습니다.
이 연구에서는 MOE와 클릭 화학의 조합을 통해 세포 내 시알릴화 글라이칸을 시각화하는 확장 현미경의 능력을 설명합니다. 구체적으로, 우리는 공동 국소화 연구를 위한 면역형광과 관련될 수 있는 시알릴화를 표적으로 하는 리포터를 고용하는 생체 직교 라벨링 및 확장 현미경 검사 절차를 제안합니다. 이 프로토콜은 sialoconjugate 생합성, 세포 내 교통 및 재활용에 대한 국소화 연구를 가능하게 합니다.
서문
형광 현미경 검사는 세포 내의 특정 분자를 라벨링하고 시각화하는 데 널리 사용되지만, 약 200-250nm보다 가까운 물체를 구별하는 능력을 제한하는 Abbe의 빛 회절 한계1에 의해 본질적으로 해상도가 제한됩니다. 이러한 제한은 빛의 파동 특성과 현미경 대물 렌즈의 개구수로 인해 발생하며, 세포 내 구조를 이미징할 때 문제가 발생합니다. 이러한 한계를 극복하면 나노미터 규모에서 특정 생물학적 과정에 대한 더 나은 통찰력을 얻을 수 있습니다.
빛의 회절 한계를 능가하기 위해 STORM(Stochastic Optical Reconstruction Microscopy) 및 STED(Stimulated Emission Depletion)와 같은 초고해상도 현미경 기술이 개발되었습니다 2,3. STORM은 형광단의 확률적 활성화에 의존하여 주어진 시간에 희박한 하위 집합만 이미지화할 수 있습니다. 이를 통해 개별 형광단의 정확한 위치 파악이 가능하여 고해상도 이미지를 재구성할 수 있습니다. 반면에 STED는 공핍 레이저를 사용하여 여기 지점 주변 주변의 형광을 선택적으로 소멸시켜 점 확산 기능을 효과적으로 좁힘으로써 해상도를 향상시킵니다.
이러한 접근 방식은 모든 형광단이 동시에 검출되는 광시야 또는 컨포칼 현미경과 대조를 이루며, 그 결과 모든 회절 패턴을 결합하고 가까운 개별 형광단을 구분하지 못해 해상도가 손실되는 이미지를 얻을 수 있습니다. 그러나 이러한 초해상도 방법에는 매우 특정한 광원, 장비, 시료 전처리 및/또는 형광단이 필요하기 때문에 비용이 많이 들고 대부분의 실험실에서 이러한 기술에 접근하기 어려우며 일상적인 실험에서 구현하기가 어렵습니다. 이러한 제약으로 인해 과학계는 쉽게 구할 수 있는 현미경 장비 및 일상적인 염색 프로토콜과 호환되는 더 높은 해상도를 달성하기 위한 대체 솔루션을 찾게 되었습니다. 2015년, 샘플을 물리적으로 확장하여 광학 현미경의 한계를 우회하는 방법은 Boyden과 동료들에 의해 확장 현미경(ExM)4이라고 불리는 개발되었습니다.
ExM은 빛의 회절 한계를 초과하지 않으면서 생물학적 시료의 나노 단위 세부 정보를 제공하는 3단계 방법입니다(그림 1). 대신, 기존의 회절 제한 현미경을 사용하여 등방성 방식으로 물리적으로 확대된 샘플을 이미지화합니다. 겔화(gelation)라고 하는 첫 번째 단계는 일반적으로 고정된 세포 또는 조직인 생물학적 샘플을 아크릴레이트 나트륨과 아크릴아미드를 기반으로 하는 팽창성 고분자 전해질 하이드로겔에 삽입하는 것으로 구성됩니다. 그런 다음 생물학적 샘플은 효소 처리를 거쳐 단백질이나 막과 같은 특정 구성 요소를 부분적으로 분해하고 조밀한 세포 구조를 분해하여 세포가 균일하게 증식할 수 있도록 합니다. 분해라고 하는 이 단계는 기계적 특성과 관련하여 샘플의 구조적 구성 요소를 균질화하여 샘플의 왜곡으로 이어질 수 있는 차등 팽창을 방지함으로써 이를 달성하는 데 도움이 됩니다. 마지막으로, 팽창이라고 하는 마지막 단계에서는 하이드로겔이 포매되고 분해된 샘플을 탈이온수에 넣어 팽창시킵니다. 이 기술은 모든 차원에서 약 4-5배의 선형 배율로 샘플이 확장되어 표준 현미경 기술을 사용하여 미세한 세포 세부 사항을 시각화할 수 있습니다. 팽창의 등방성 특성을 감안할 때, 겔 매트릭스 내에서 확대된 생물학적 샘플은 3차원의 기하학적 세부 사항과 다양한 구조 구성 요소 간의 공간적 관계를 유지합니다. 따라서 공간 정보는 팽창 시 보존되는 반면, 겔에 고정된 형광 라벨 또는 생체 분자 사이의 거리는 모든 방향에서 균일하게 증가합니다. 이를 통해 신호를 더 잘 분리할 수 있어 해상도가 향상됩니다.

그림 1: ExM 프로토콜 개요. (a) 겔화: 생물학적 샘플은 팽창성 고분자 전해질 하이드로겔 내에 고정되고 매립됩니다. (b) 소화: 샘플의 기계적 특성은 효소와 세제를 사용하여 균질화되어 팽창을 제한하고 왜곡을 생성할 수 있는 단백질과 지질막을 분해합니다. (c) 팽창: 하이드로겔이 탈이온수에 잠겨 등방성으로 팽창합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
2015년 이후 ExM은 공간 정보 보존 분야에서 상당한 기술적 발전을 이뤘습니다. 특히, 다양한 분자 앵커는 생체 분자를 팽창성 하이드로겔 5,6에 직접 공유 결합하도록 설계되었습니다. 이 프로토콜에서는 생체 분자(일반적으로 라이신 잔기 또는 단백질의 N-말단 위치)의 유리 아민 그룹과 반응하여 아크릴로일 그룹을 당단백질을 포함한 세포 단백질에 생체 접합하는 N-아크릴록시숙신이미드(NAS)를 사용했습니다. 그런 다음 이 아크릴로일 그룹은 겔화 중 가교 반응을 통해 다른 단량체와 반응하여 이러한 생체 분자를 폴리머에 직접 고정합니다.
ExM은 대사 올리고당 엔지니어링(MOE)을 포함한 광범위한 라벨링 방법과 호환됩니다. MOE는 생체 직교 화학 물질 핸들7이 장착된 대사 전구체의 유사체를 통합하여 글라이칸을 라벨링할 수 있는 강력한 도구입니다. 화학 리포터(chemical reporter)로 알려진 이러한 유사체는 독성을 일으키지 않고 대사 경로에 통합됩니다. MOE 접근법에서 리포터 단당류는 대사를 통해 활성화된 뉴클레오티드 당으로 처리된 다음 초기 당접합체로 전달됩니다. 우리의 주요 초점은 시알릴화, 특히 시알릴화된 N-당단백질의 세포 내 역학 및 이동에 대한 연구이며, 이는 세포-세포 상호 작용, 면역 조절 및 발달에 대한 역할로 인해 건강과 질병의 맥락에서 매우 중요합니다. 비정상적인 시알릴화는 암 8,9,10,11, 감염성 질환12, 유전 질환 13,14,15와 같은 질병과 관련이 있어 치료 개발 및 바이오마커 발견의 핵심 표적이 됩니다. 시알릴화를 이해하면 당생물학 및 질병 메커니즘에 대한 통찰력이 향상됩니다.
시알릴화는 인간에게 가장 풍부한 시알산인 N-아세틸뉴라민산(Neu5Ac)의 유사체 또는 Neu5Ac의 대사 전구체인 N-아세틸만노사민(ManNAc)의 유사체와 MOE를 사용하여 생체 직교 핸들 8,16을 지니고 있습니다. ManNAc는 세포질에서 Neu5Ac로 전환된 다음 핵에서 cytidine-5'-monophospho-N-neuraminic acid(CMP-Neu5Ac)로 활성화됩니다. 일단 뉴클레오티드 당으로 활성화되면, 골지체의 시알릴 전이효소는 Neu5Ac 단위를 성장하는 글라이칸 사슬의 말단 위치로 전달합니다(그림 2). 부자연스러운 ManNAc 유도체의 대사 통합에 따라, 태그가 지정된 시알릴화 글라이칸은 생체 직교 클릭 화학을 통해 리포터 핸들에 상보적인 반응기를 지닌 형광단에 공유 결합할 수 있습니다. 이를 통해 in vivo 또는 ex vivo에서 glycoconjugate를 직접 관찰할 수 있습니다.
대부분의 단당류 기자의 경우, 수동 확산을 통해 원형질막을 통과하기 위해 과아세틸화 형태가 필요합니다. 그러나 과아세틸화 및 보호되지 않은 기자 모두 시알릴화16을 효율적으로 조사하는 것으로 나타났습니다. 보호되지 않은 시알릴화 리포터는 활성 수송 메커니즘, 즉 Neu5Ac 유사체에 대한 음세포증가증과 ManNAc에 대한 아직 확인되지 않은 수송체에 의해 세포에 들어갈 수 있습니다. 보호되지 않은 당은 유사한 효과를 얻기 위해 더 높은 농도(일반적으로 100-500μM)가 필요하지만 일단 세포 내부로 들어가면 시알산 대사 경로로 직접 들어갈 수 있습니다. 대조적으로, 과아세틸화된 당은 대사적으로 활성화되기 전에 세포 내 비특이적 에스테라제에 의해 완전히 탈아세틸화되어야 합니다. 더 낮은 농도(일반적으로 10-50μM)에서 사용할 수 있지만 불완전한 탈아세틸화는 효소 활성을 방해하거나 부분적으로 아세틸화된 시알산 유사체의 통합으로 이어져 잠재적으로 다운스트림 분석을 왜곡할 수 있습니다. 또한 아세트산의 방출은 국부적으로 pH에 영향을 미쳐 잠재적으로 세포 기능에 영향을 미칠 수 있습니다. Chen과 동료들은 또한 per-O-acetylated 당이 비효소 메커니즘을 통해 단백질의 유리 시스테인 잔류물과 반응하여 off-target 혼입 및 비특이적 신호 증가로 이어진다는 것을 입증했습니다 17,18. 따라서 본 프로토콜에서는 보호되지 않은 ManNAc 보고자를 고용합니다.

그림 2: 대사 올리고당(metabolic oligosaccharide) 엔지니어링 및 시알산(sialic acids)의 라벨링. UDP-GlcNAc는 세포질에서 GNE/MNK의 UDP-GlcNAc 2-에피머라제 도메인에 의해 ManNAc로 변환됩니다. 그런 다음 ManNAc는 GNE/MNK의 ManNAc 6-키나아제 도메인에 의해 세포질에서 인산화되어 ManNAc-6-인산염을 형성합니다. N-아세틸뉴라미네이트 합성효소는 ManNAc-6-P와 포스포에놀피루베이트의 응축을 촉매하여 Neu5Ac-9-인산을 생성하고, 이후 시알산 인산가수분해효소에 의해 탈인산화되어 Neu5Ac를 생성합니다. Neu5Ac는 세포내이입(endocytosis)과 리소좀 재활용(lysosomal recycling8)을 통해 구제 경로를 통해 공급될 수도 있다. 핵으로 수송된 후 CMP-시알산 합성효소에 의해 CMP-Neu5Ac로 전환됩니다. 골지체에서 CMP-NeuAc는 성숙 당접합체에서 글라이칸의 말단 위치에 Neu5Ac 부분을 도입하는 시알릴전이효소의 기질이며, 이는 결국 세포막에서 발현되거나 분비됩니다. 생체 직교 손잡이를 가진 ManNAz 화학 리포터는 미확인 활성 수송체를 통해 세포를 관통하고 대사 경로로 들어갈 수 있습니다. 생합성 후 글라이칸에 통합된 태그가 지정된 Neu5Az 단위는 형광 프로브의 CuAAC 매개 접합을 통해 표지됩니다. 약어 : NAc = N- 아세틸; UDP-GlcNAc = 우리딘 이인산 N- 아세틸 글루코사민; ManNAc = N-아세틸만노사민; GNE = UDP-GlcNAc 2-에피머라제; MNK = ManNAc 6-키나아제; ManNAc-6-P = ManNAc-6-인산염; NANS = N-아세틸뉴라미네이트 합성효소; PEP = 포스포에놀피루베이트; Neu5Ac = N-아세틸뉴라민산; Neu5Ac-9-P = Neu5Ac-9-인산염; NANP = 시알산 인산가수분해효소; CMP = 시티딘-5'-일인산염; CMAS = CMP-시알산 합성효소; STS = 시알릴 전이효소; ManNAz = N-아지도아세틸만노사민; Neu5Az = N-아지도아세틸뉴라민산. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
가능한 생체 직교 반응 중에서 우리는 구리 촉매 아지드-알킨 순환 첨가 (CuAAC) 16 , 19 에 관심을 집중했습니다. 우리의 경험에 따르면 CuAAC는 고정 세포에서 세포 내 글라이칸을 라벨링하는 데 가장 적합한 옵션입니다. 이 반응은 잘 정립되어 있고 생물 직교 화학에서 철저하게 연구되었으며 복잡한 생물학적 환경에서 사용할 수 있도록 표준화되어 견고한 지식 기반과 최적화된 프로토콜을 제공합니다. 빠른 동역학은 실제로 높은 반응 효율과 특이성을 제공하며, 합성이 쉽고, 안정적이며, 살아있는 시스템에 없고, 네이티브 생체 분자에 대해 불활성인 아지드 및 알카인 그룹을 포함하므로 구리 독성이 문제가 되지 않는 고정 세포 응용 분야에 이상적입니다. 균주 촉진 알킨 아지드 순환 첨가 (SPAAC) 20 , 21 은 구리 독성을 피하면서 역학이 느리고 프로브가 부피가 커서 소수성 트래핑으로 인해 세포 내 응용 분야에서 백그라운드 신호가 더 높아지고 신호 대 잡음비가 감소하는 경향이 있는 반면, 역 전자 수요 Diels-Alder (IEDDA) 22 , 23는 빠르고 구리가 없지만 더 복잡한 프로브 합성을 포함하며 MOE 응용 분야에서 아직 완전히 특성화되지 않은 더 큰 리포터 그룹이 필요합니다. ExM은 고정 세포만을 필요로 하기 때문에 CuAAC는 생체 직교 라벨링을 위한 강력하고 효율적인 솔루션을 제공합니다.
이 논문은 대사 라벨링, 생물직교 클릭 화학 및 확장 현미경을 결합하여 세포 내 세포 내 시알릴화 당단백질을 시각화하기 위한 프로토콜을 제시합니다. 본 논문에 기술된 실험 절차에서는 N-아지도아세틸만노사민(ManNAz)을 화학 리포터로 사용하며, 팽창 절차 전에 작은 유기 형광단의 CuAAC 결찰을 수행합니다. 당단백질(glycoprotein)은 겔화(gelification), 분해(digestion) 및 팽창(expansion) 전에 NAS와 함께 하이드로겔에 고정됩니다. 시알산의 MOE 라벨링은 cis-Golgi 장치에 국한된 마우스 anti-GM130 1차 항체에서 볼 수 있듯이 공동 국소화 평가를 위한 면역형광 접근법과 관련될 수 있습니다. 이 프로토콜은 생리학적 상태의 세포 또는 화학적 처리를 받은 세포에 적용할 수 있습니다. 이를 설명하기 위해 클로로퀸은 세포 내 당단백질의 처리 및 이동에 영향을 미치는 리소좀 기능을 억제하는 데 사용되었습니다. 핵 염색은 세포의 국소화뿐만 아니라 ExM 공정의 품질을 나타내는 지표로 사용됩니다. 팽창 계수는 실제로 확장 전(preExM)과 확장 후(postExM)의 크기를 비교하여 측정할 수 있습니다.
프로토콜
1. 세포 파종
알림: 층류 후드 아래에서 멸균 상태에서 다음 단계를 수행하십시오. 이 방법은 본 연구에 사용된 모든 세포주(HeLa, MCF7, 1차 섬유아세포) 또는 연구에서 일반적으로 사용되는 가장 부착성 세포주 모델에 적용할 수 있습니다 20,24,25.
- 5% CO2 대기에서 37°C의 T75 플라스크에 10% 소 태아 혈청(FBS)이 보충된 DMEM 고포도당 배지에서 세포를 성장시킵니다.
- 세포가 완전히 포화도에 도달하면 세포 배양 배지를 제거하고 PBS 4mL로 세포를 세척합니다.
알림: 이 단계와 모든 후속 세척 단계에서 사용되는 PBS 용액은 사용하기 전에 0.2μm PTFE 멤브레인을 통한 여과로 멸균해야 합니다. - 0.5 g/L의 트립신이 함유된 1x 트립신-EDTA 용액 2 mL에 37 °C에서 5분 동안 세포를 배양하여 플라스크 바닥에서 세포를 방출합니다.
- 10% FBS를 함유한 DMEM 고포도당 배지 8mL를 추가하고 완전히 혼합하여 모든 세포가 플라스크 바닥에서 분리되도록 합니다.
- 세포를 15mL 코니컬 튜브에 옮기고 200 × g에서 5분 동안 원심분리합니다. 그 동안 12웰 플레이트의 각 웰에 하나의 커버슬립(직경 12mm, 두께 130-160μm)을 추가합니다.
알림: 커버슬립 두께는 후속 이미징 단계에서 사용되는 현미경의 요구 사항에 맞게 조정해야 합니다. - 원심분리 후 상층액을 제거하고 펠렛을 1mL의 세포 배양 배지에 재현탁합니다.
- 최소 두 번의 측정에서 혈구 측정으로 평균 세포/mL 수를 결정합니다.
- 세포 배양 배지에서 세포 현탁액을 mL당 300,000 세포 농도로 희석하고 각 웰에 현탁액 1mL를 시딩합니다.
- 다음 단계 전에 5% CO2 분위기 하에서 37°C에서 10% FBS를 함유하는 10% FBS를 함유하는 DMEM 고포도당 배지에서 세포가 24시간 동안 정착하도록 합니다.
2. 클로로퀸 치료(선택 사항)
참고: 2.1-2.8단계는 클로로퀸을 예로 사용하여 외부 시약(억제제, 효과기, 약물)으로 처리된 세포에 프로토콜을 사용하는 방법을 보여줍니다. 외부 시약으로 처리되지 않은 세포의 경우 이 단계를 건너뜁니다.
- 정확한 양의 클로로퀸 이인산염(CQ)을 계량하여 10mM 원액의 충분한 부피를 준비합니다.
알림: 층류 후드 아래에서 멸균 상태에서 다음 단계를 수행하십시오. - 10mM의 최종 농도를 위해 원하는 양의 탈이온수에 고체 CQ를 용해시킵니다.
- 0.2μm 필터를 통해 용액을 필터링합니다.
- 1.5mL 마이크로 원심분리 튜브에 10mM 스톡 용액 1mL를 부분 표본으로 추출하고 -20°C에서 보관합니다.
- 1.9단계에서 준비한 세포 샘플을 PBS 1mL로 세척합니다.
- 10% FBS를 함유한 9.9mL의 DMEM 고포도당 배지에 100μL의 100mM CQ를 추가하여 최종 농도 100μM의 CQ를 얻습니다.
- 5% CO2 분위기에서 37°C에서 100μM의 CQ가 보충된 배지 1mL에서 8시간 동안 세포를 배양합니다.
- PBS 1mL로 세포를 3x 세척합니다.
3. 대사 올리고당 공학
- 10% FBS를 함유하는 DMEM 고포도당 배지에서 ManNAz(또는 음성 대조군 조건의 경우 ManNAc)의 5mM 원액을 준비합니다.
참고: 이 원액은 각 웰을 1mL의 설탕 유사체 용액으로 처리할 수 있을 만큼 충분한 부피여야 합니다. 층류 후드 아래에서 멸균 상태에서 다음 단계를 수행합니다. - 0.2μm 필터를 통해 용액을 여과하고 필요할 때까지 -20°C에서 1mL 분취량에 보관합니다.
- 3.2단계에서 준비한 1mL 원액 분취액을 10% FBS를 함유한 9mL의 DMEM 고포도당 배지로 희석하여 500μM의 ManNAz 또는 ManNAc가 보충된 배지를 준비합니다.
- 1.9단계(처리되지 않은 세포 실험의 경우) 또는 2.8단계(CQ 처리된 세포 실험의 경우)에서 제조된 세포를 500μM ManNAz(대조군의 경우 500μM ManNAc)가 보충된 배지 1mL에 5%CO2 분위기 하에서 37°C에서 24시간 동안 배양합니다.
4. 정착과 투과성
알림: 이 모든 단계는 흄 후드 아래에서 수행됩니다.
- PBS 1mL로 샘플을 세척합니다.
- 500μL의 5% 파라포름알데히드를 추가하여 각 커버슬립의 세포를 고정합니다. 실온에서 15분 동안 그대로 두십시오.
- PBS 1mL로 3x 세척합니다.
- 뚜껑이 있는 충분히 큰 불투명 상자(예: 폴리스티렌 상자)를 사용하여 맞춤형 습한 챔버를 준비합니다. 상자 바닥에 젖은 압지 조각을 놓고 그 위에 파라필름을 한 겹 얹습니다.
알림: 습한 챔버는 닫혔을 때 외부 광원으로부터 샘플을 완전히 보호해야 합니다. - 파라필름에 커버슬립을 놓고 세포가 위를 향하도록 합니다.
참고: 파라필름에 샘플의 이름 또는 참조를 미리 표시하십시오. - PBS에 200μL의 0.5%(v/v) Triton X-100을 사용하여 실온에서 15분 동안 세포 투과화를 수행합니다.
- 200μL의 PBS로 3회 세척합니다.
5. 형광 라벨링
- 태그가 지정된 시알릴화 글라이칸의 CuAAC 라벨링
참고: CuAAC 반응 완충액은 사용 전에 새로 준비해야 하며, 반응물은 지정된 순서로 첨가해야 하며 아스코르브산나트륨은 마지막에 첨가해야 합니다. 산소화된 수용액에서 Ascorbate로 Cu(II)가 환원되어 형성된 Cu(I) 이온은 점차적으로 Cu(II)로 재산화되어 시간이 지남에 따라 Ascorbate 농도가 감소합니다. 결과 재현성을 유지하기 위해 버퍼는 4°C에서 며칠 이상 보관해서는 안 되며 당일에 사용하는 것이 좋습니다.- 섹션 3에서 조작된 아지드 변형 시알릴화 글라이칸을 라벨링하기 위해 5μM AlexaFluor 488 알킨(AF488Alk), 150μM CuSO4, 300μM 2-(4-((bis((1-(tert-butyl)-1H-1,2,3-triazol-4-yl)methyl)amino)methyl)-1H-1,2,3-triazol-1-yl)acetic acid(BTTAA), 0.1 M K2HPO4 및 2.5 mM 아스코르브산 나트륨으로 구성된 CuAAC 반응 완충액을 탈이온수. 각 커버슬립에 200μL를 도포할 수 있는 충분한 용액을 준비합니다.
- 각 커버슬립에 200 μL의 CuAAC 버퍼를 추가하여 반응을 시작합니다. 샘플이 방울로 균일하게 덮여 있는지 확인하고 빛으로부터 보호된 실온에서 45분 동안 반응하도록 합니다.
- 200 μL의 PBS로 3x 세척하여 반응을 중지하고 과도한 프로브와 시약을 제거합니다.
- 핵 염색
- Hoechst 33342를 사용한 핵 라벨링의 경우 PBS에서 1:2000으로 10mg/mL 원액을 희석합니다.
참고: 4',6-diamidino-2-phenylindole (DAPI)과 같은 동등한 핵 대조염색제를 사용하여 동일한 효과를 얻을 수 있습니다. - 이 용액 200 μL로 세포를 덮고 빛으로부터 보호되는 실온에서 5-10 분 동안 배양합니다.
- 200μL의 PBS로 3회 세척합니다.
- Hoechst 33342를 사용한 핵 라벨링의 경우 PBS에서 1:2000으로 10mg/mL 원액을 희석합니다.
- 면역형광 염색
- 50mL의 완충액에 대해 젤라틴 0.1g, BSA 1g, FBS 1mL 및 PBS 49mL를 함유한 소 혈청 알부민(BSA) 차단 완충액을 준비합니다.
- 200 μL의 BSA 차단 버퍼에서 4 °C에서 1 시간 동안 샘플을 배양합니다. 빛으로부터 샘플을 보호하십시오.
- 정제된 마우스 anti-GM130 항체(인식된 항원결정기: Rat GM130 aa. 869-982 | RRID: AB_398142)을 사용하여 준비된 BSA 차단 완충액에서 1:100으로 1차 항체를 희석합니다.
참고: 이 단계는 관련 1차 항체를 사용하여 다른 소기관을 표적으로 하는 면역형광 염색에 적용할 수 있으며, 이 경우 제조업체의 지침에 따라 희석 인자를 조정해야 합니다. - 항체가 포함된 용액 70μL를 커버슬립에 첨가하고 빛으로부터 보호된 4°C에서 1시간 동안 배양합니다.
- 200μL의 PBS로 3회 세척합니다.
- 형광 2차 항체(Alexa Fluor 546 anti-mouse IgG)를 BSA 차단 버퍼에 1:600으로 희석하고 각 커버슬립에 100μL를 추가합니다.
- 빛을 차단한 실온에서 1시간 동안 샘플을 배양합니다.
- 200μL의 PBS로 3회 세척합니다.
- 커버슬립을 6웰 플레이트로 옮깁니다. 샘플을 4°C의 PBS 2mL에 담아 빛으로부터 보호하여 며칠 동안 보관하십시오.
참고: 확장 전후의 비교 목적을 위한 참조로 preExM 시각화 전용으로 복제를 항상 준비하고 확장 후 라벨링 패턴이 왜곡되지 않았는지 확인하는 것이 좋습니다.
6. 샘플의 Preexpansion 이미징
참고: 다음 단계는 사용하는 바이오이미징 장비에 따라 다르며 기계의 요구 사항에 맞게 조정해야 할 수 있습니다. 지역 실험실 또는 바이오이미징 플랫폼의 규칙을 따르십시오. 여기서 6.1-6.13 단계는 레이저 스캐닝 컨포칼 현미경에서 수행됩니다.
- 현미경을 켜고 광원(레이저)이 예열되었는지 확인합니다. 바이오이미징 획득 소프트웨어를 엽니다.
- 형광단에 해당하는 레이저 여기 파장을 선택하여 채널을 생성합니다(예: 핵 염색의 경우 405nm, AF488Alk 표지된 시알릴화 글라이칸의 경우 488nm, 면역형광 표지의 경우 546nm). 그에 따라 레이저를 활성화하십시오.
- 32mm 직경의 커버슬립(130-170μm 두께)을 현미경에 맞게 조정된 홀더에 놓고 고정합니다.
- 5.3.9단계에서 준비한 샘플을 세포가 아래를 향하도록 하여 32mm 커버슬립에 놓습니다. 샘플이 마르는 것을 방지하기 위해 탈이온수 한 방울을 추가합니다.
- 적절한 대물 렌즈를 선택하며, 여기서는 1.4 개구수 또는 이에 상응하는 63x 오일 이멀젼 대물렌즈를 선택하십시오.
참고: 가장 높은 개구수가 최상의 해상도를 제공합니다. 오일 이멀젼 대물렌즈를 사용할 때 대물렌즈와 커버슬립 사이에 오일 한 방울을 추가하는 것을 잊지 마십시오. - 홀더를 현미경 스테이지에 놓습니다.
- 명시야 또는 낮은 레이저 강도 형광 모드를 사용하여 샘플에서 관심 영역을 찾습니다.
- 초점을 설정합니다.
- 소프트웨어의 세포를 라이브 형광 모드로 시각화합니다. 각 채널의 레이저 강도를 설정합니다. 광표백을 피하기 위해 낮은 레이저 출력으로 시작하십시오. 레이저 출력을 천천히 증가시키고 검출기 게인 및 오프셋을 조정하여 과다 노출 없이 형광을 명확하게 시각화하기 위해 필요에 따라 과도한 노이즈를 도입하지 않고 신호를 증폭합니다.
알림: 배경 소음을 최소화하고 신호 포화를 피하도록 매개변수를 조정해야 합니다. 음성 대조군을 포함한 일련의 실험에서 모든 샘플에 걸쳐 일관된 설정을 유지하는 것이 중요합니다. - 원하는 관측치에 도달하는 데 필요한 핀홀 크기(예: 1 Airy Unit), 스캔 속도(예: 8), 확대/축소 계수(예: 1), 밝기 값 범위(예: 16비트), 평균 수(예: 2), 모드(예: 선) 및 방법(예: 평균)과 같은 이미지 매개변수를 설정합니다.
참고: 이 값은 시스템 및 샘플에 따라 다르며 획득 전에 최적화해야 합니다. 핀홀 크기는 개구수, 배율 및 파장에 따라 달라지며, 이를 줄이면 신호 대 잡음비가 저하되는 대신 해상도가 향상될 수 있습니다. 광표백을 방지하기 위해 스캔 속도를 빠른 값(픽셀당 작은 체류 시간에 해당)으로 설정하는 것이 좋습니다. - 수집을 수행하고 나중에 참조할 수 있도록 적절한 메타데이터와 함께 원하는 형식으로 데이터를 저장합니다.
- 수집이 완료되면 한 쌍의 핀셋으로 쉽게 이동할 수 있도록 소량의 탈이온수를 추가하여 샘플을 6웰 플레이트에 다시 넣습니다.
참고: 6.12단계는 이 특정 샘플의 확장이 필요한 경우에만 수행됩니다. 그렇지 않으면 샘플을 폐기하고 다음 단계를 위해 다른 반복실험을 사용합니다.
7. 확장 현미경 프로토콜
참고: 샘플은 광표백을 방지하기 위해 빛으로부터 최대한 보호됩니다. 앵커링 및 겔화 단계는 흄 후드 아래에서 수행됩니다.
- 정박
- PBS 중 3.2mg/mL 농도의 N-아크릴로일숙신이미드(NAS)를 실온에서 1시간 동안 기계식 셰이커에 주입하여 샘플을 배양합니다. 웰당 500μL의 용액을 추가할 수 있는 충분한 양의 용액을 준비합니다(원액: DMSO의 16mg/mL NAS).
참고: NAS는 단백질의 라이신 잔류 물 및 N-말단과 반응하여 아크릴로일 그룹을 이식하여 단백질이 후속 단계에서 하이드로겔에 고정될 수 있도록 합니다. - PBS 1mL로 3x 세척합니다.
- PBS 중 3.2mg/mL 농도의 N-아크릴로일숙신이미드(NAS)를 실온에서 1시간 동안 기계식 셰이커에 주입하여 샘플을 배양합니다. 웰당 500μL의 용액을 추가할 수 있는 충분한 양의 용액을 준비합니다(원액: DMSO의 16mg/mL NAS).
- 겔화
- 1x PBS 50mL(pH 7.4)에 NaCl 5.843g, 아크릴아미드 1.25g, 비스-아크릴아미드 0.075g, 아크릴레이트나트륨 4.313g을 첨가하는 단량체 용액을 준비합니다. 각 커버슬립에 70μL를 도포할 수 있는 충분한 용액을 준비합니다. 겔화 최소 24시간 전에 1mL 분취액을 동결합니다.
주의 : 단량체는 독성이 있습니다. 적절한 보호 장비를 사용하여 조심스럽게 다루어야 하며 흄 후드 아래에서 무게를 측정해야 합니다. - 파라 필름 조각에 70 μL 방울의 단량체 용액을 놓고 1.4 μL의 10 % (v / v) N, N, N ', N'- 테트라 메틸 에틸렌 디아민 (TMEDA)을 첨가 한 다음 1.4 μL의 10 % (w / v) 과황산 암모늄 (APS)을 방울에 혼합합니다.
참고: 교차결합제 시약은 TMEDA를 먼저 첨가한 다음 APS를 첨가해야 합니다. - 재빨리 셀이 아래를 향하도록 용액에 커버슬립을 조심스럽게 놓습니다.
- 용액이 습한 챔버에서 실온에서 1시간 동안 중합되도록 합니다.
- 1x PBS 50mL(pH 7.4)에 NaCl 5.843g, 아크릴아미드 1.25g, 비스-아크릴아미드 0.075g, 아크릴레이트나트륨 4.313g을 첨가하는 단량체 용액을 준비합니다. 각 커버슬립에 70μL를 도포할 수 있는 충분한 용액을 준비합니다. 겔화 최소 24시간 전에 1mL 분취액을 동결합니다.
- 소화
- 1x TAE 완충액, 0.5%(v/v) Triton X-100, 0.8M 구아니딘 HCl 및 8U/mL proteinase K로 구성된 분해 완충액을 준비합니다.
참고: 분해 버퍼는 사용하기 전에 30°C에서 37분 동안 신선하게 준비하고 사전 배양해야 합니다. - 7.2단계에서 형성된 하이드로겔에 1mL의 분해 완충액을 추가합니다. 기계식 셰이커에서 3°C에서 37시간 동안 분해를 진행하십시오.
- 소화 버퍼를 제거하고 탈이온수 3mL로 젤 2x를 부드럽게 씻습니다.
알림: 하이드로겔은 약간 팽창하여 커버 슬립에서 분리됩니다.
- 1x TAE 완충액, 0.5%(v/v) Triton X-100, 0.8M 구아니딘 HCl 및 8U/mL proteinase K로 구성된 분해 완충액을 준비합니다.
- 확장
- 하이드로겔을 탈이온수 3mL에서 2시간 동안 팽창하도록 둡니다. 30분마다 물을 갈아주세요.
8. 샘플의 팽창 후 이미징
- 직경 32mm의 커버슬립(두께 130-170μm)을 놓고 홀더에 고정합니다.
- 하이드로겔 조각을 잘라내어 세포가 젤 바닥에 오도록 하여 커버 슬립에 놓습니다.
참고: 절제된 조각은 관찰을 위한 적절한 표면적을 제공할 수 있을 만큼 충분히 커야 하지만 젤 취급을 어렵게 하고 파손 위험을 증가시킬 수 있으므로 너무 크지 않아야 합니다(권장 크기: 5-8mm). 커버슬립의 전체 표면을 덮을 수 있을 만큼 충분한 하이드로겔 조각을 놓습니다. 이렇게 하면 조각을 제자리에 고정하고 이미징 중 드리프트를 방지하는 데 도움이 됩니다. 드리프트 문제가 있는 경우 커버슬립을 poly-D-lysine으로 사전 코팅하는 것이 유리할 수 있습니다6. - 홀더를 현미경 스테이지에 놓습니다.
- 6단계에서 설명한 대로 오일 이멀젼 대물렌즈가 있는 Laser Scanning Confocal Microscope에서 이미지 획득을 수행합니다.
9. ImageJ에서 확장 계수(EF) 계산
- ImageJ 프리웨어 또는 이와 동등한 제품에서 이미징 데이터를 엽니다.
- 다중 채널 이미지에서 작업하는 경우 Select Image(이미지 선택) | 색상 | 채널을 분할합니다.
- 핵 직경을 측정하려면 Select Image(이미지 선택) | 조정 | 임계값.... 기본 드롭다운 메뉴에서 이미지에 가장 잘 맞는 방법(예: Otsu)을 선택하고 핵의 모양이 배경에서 명확하게 잘릴 때까지 임계값을 조정합니다.
참고: 하나의 설정이 모든 이미지 또는 모든 샘플에 맞지 않을 수 있으므로 각 이미지에 대해 임계값 설정을 조정해야 합니다. - Select Analyze(분석 선택) | 측정 설정 .... 메뉴에서 Feret diameter(페렛 지름)를 선택하고 필요한 경우 소수를 조정합니다.
- 이미지 분석을 시작하려면 Select Analyze(분석 선택) | 입자를 분석하여 입자의 크기를 설정합니다. 핵의 경우 5-infinity μm² | 결과 표시 | 관리자에 추가 | 가장자리 및 오버레이에서 제외. 분석을 시작합니다.
- 분석이 시작되면 여러 창이 나타납니다. 창 결과에서 Feret 컬럼에 있는 각 핵의 최대 Feret 직경을 찾습니다. 원시 데이터를 스프레드시트에 복사하여 붙여넣고 사전 확장 및 확장 샘플의 평균 핵 크기를 계산합니다.
참고: 각 핵에 해당하는 관심 영역(ROI)이 핵의 전체 주변을 따르는지 확인하십시오. - 확장 계수(EF)를 다음과 같이 계산합니다.
EF =
참고: EF 계수는 샘플이 얼마나 확장되었는지를 결정합니다.
결과
아래는 CQ 처리(프로토콜 섹션 2) 및 면역형광 공동 국소화 염색(프로토콜 단계 5.3)을 생략하고 HeLa 세포(그림 3A) 및 MCF7 세포(그림 3B)에서 시알릴화된 당단백질을 시각화하기 위한 프로토콜의 적용입니다.

그림 3: ExM 프로토콜 이전(왼쪽)과 이후(오른쪽)의 시알릴화 라벨링 패턴 비교. (A) Hela 세포 및 (B) MCF7 세포를 500μM ManNAz로 24시간 동안 배양했습니다. 라벨링은 Alexa Fluor 488 알킨(녹색으로 표시)을 사용하여 CuAAC를 통해 수행되었습니다. 핵은 Hoechst 33342(파란색으로 표시)로 염색하였다. 눈금 막대 = 20 μm. 약어 : ExM = 확장 현미경; ManNAz = N-아지도아세틸만노사민; CuAAC = 구리 촉매 아지드-알킨 고리첨가. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
일반적으로 sialylation 라벨링은 세 가지 주요 형광 풀로 구성됩니다: 초기 glycoconjugate가 합성되는 것을 나타내는 Golgi pool, 세포막으로 내보내지거나 세포막에서 재활용되는 glycoconjugate를 나타내는 vesicular pool(일반적으로 덜 강렬함), 그리고 발현된 glycoconjugate를 보여주는 membrane pool입니다13. 대사적으로 생성된 Neu5Az로 태그된 생체 고분자 glycoconjugate만 검출되는 반면, unreacted excess reporter 또는 probe는 세척 단계에서 제거된다는 점에 유의해야 합니다. ExM 이후의 이미지는 컨포칼 현미경으로 획득되었으며 특히 골지체에서 훨씬 더 미세한 디테일과 더 적은 배경을 보여줍니다. 이 프로토콜은 시알릴화 당접합체를 발현하는 모든 유형의 부착 세포에 적용할 수 있습니다.
다양한 세포 또는 조직 유형은 ExM 고유의 신호 부분적 손실에 의해 다양한 정도로 영향을 받을 수 있으며, 이는 형광단의 부피 희석으로 인한 것입니다. 이는 또한 라디칼 중합 중 형광단의 저조한 체류 또는 차선의 앵커링 또는 소화로 인한 생체 분자의 불량으로 인해 악화될 수 있으며, 이로 인해 신호가 급격히 손실될 수 있습니다(그림 4). 이 점은 아래 논의의 주요 주제입니다. 따라서 필요한 경우 분해 단계의 매개변수를 정밀 조사 중인 세포 또는 조직 유형에 맞게 조정하는 것이 좋습니다. 소화는 관찰되는 특정 구조 또는 생체 분자에 따라 다양한 수단을 통해 최적화될 수 있습니다. 표준화된 분해 프로토콜로 시작한 다음 분해 시간, 세제 농도 및/또는 효소 농도와 같은 매개변수를 경험적으로 조정하여 원하는 결과를 얻을 수 있도록 하는 것이 좋습니다. 더 효율적인 소화는 왜곡을 방지하는 더 나은 등방성 팽창으로 이어지지만 신호 손실의 위험도 증가시킬 수 있다는 점에 유의하는 것이 중요합니다. 따라서 고품질 등방성 팽창을 달성하는 것과 신호 무결성을 유지하는 것 사이의 균형을 찾아야 합니다. 신호 유지는 특별히 설계된 분자 앵커를 사용하여 더욱 향상될 수 있습니다. 단백질 관찰에 초점을 맞춘 연구의 경우, 고온에서 SDS(Sodium Dodecyl Sulfate)만 사용하는 프로토콜은 단백질 신호를 더 잘 보존하면서 내장된 샘플을 효과적으로 소화하는 것으로 나타났습니다26,27.
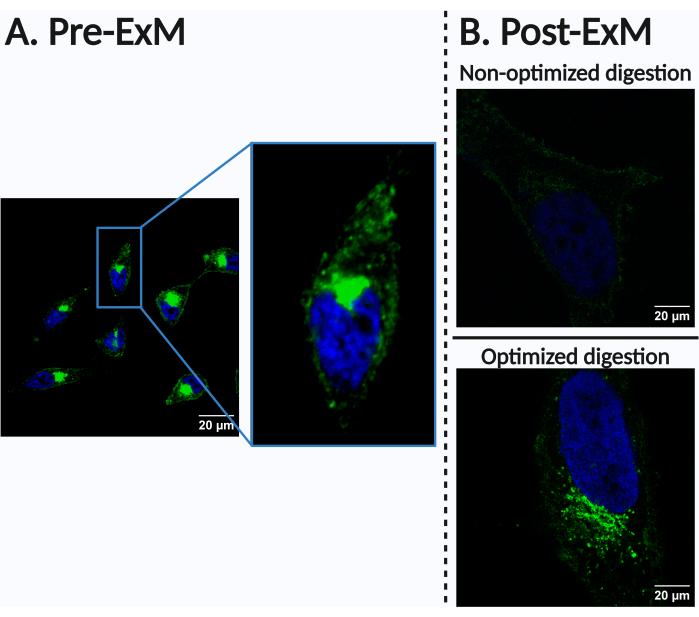
그림 4: 최적이 아닌 결과의 예. 이 그림은 특히 분해 단계에서 각 유형의 세포, 조직 및 모니터링된 생체 분자에 대한 조건 조정의 중요성을 보여줍니다. HeLa 세포를 500μM ManNAz로 24시간 배양하고 Alexa Fluor 488 알킨(녹색, 시알릴화된 N-당단백질) 및 Hoechst 33342(파란색, 핵)로 표지했습니다. (A) 라벨링 패턴 사전 확장. (B) 확장 후 라벨링 패턴. (맨 위) 조성, 농도 및/또는 길이 측면에서 제대로 최적화되지 않은 분해 매개변수는 하이드로겔에 이식된 생체 분자 보유 부족으로 인해 신호의 급격한 손실로 이어질 수 있으며, 이로 인해 충분한 감도로 바이오이미징 데이터를 수집하지 못할 수 있습니다. 이 특별한 경우, 분해 버퍼는 프로토콜 단계 7.3에서 사용된 것과 유사했습니다. 그러나 분해 시간은 90분으로, 후속 이미지에서 사용된 180분의 소화 시간에 비해 짧았습니다. (아래) 분해 매개변수를 최적화한 후 얻은 이미지(프로토콜 단계 7.3 참조). 스케일 바 = 20 μm. 약어 : ManNAz = N-azidoacetylmannosamine. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
실험의 재현성을 보장하려면 샘플을 용액에 보존하고 하이드로겔에 삽입한 후에도 빛으로부터 보호하는 것이 중요합니다. 하이드로겔은 건조되지 않도록 습한 챔버에서 유지해야 합니다. 겔화(프로토콜 단계 7.2), 분해(프로토콜 단계 7.3) 및 확장(프로토콜 단계 7.4)의 세 가지 주요 단계를 같은 날에 수행하는 것이 좋습니다. 일단 팽창되면 샘플은 탈이온수에서 4°C로 보관하고 빛으로부터 보호하면 며칠 동안 안정적이어서 바이오이미징 데이터 수집에 시간을 할애할 수 있습니다.
현미경 이미지를 분석하는 동안 확장 전(preExM)과 확장 후(postExM) 핵 Feret 직경의 평균 비율을 측정하여 팽창 계수(EF)를 평가하는 것이 좋습니다. 여기서, 이 프로토콜에서 핵은 Hoechst 33342로 염색되지만, 보다 일반적인 DAPI는 확장 현미경 검사와도 호환됩니다. Feret 직경은 핵의 경계에 위치한 두 점 사이의 최대 거리에 해당합니다. 일반적으로 성공적인 실험은 4-5의 EF 값을 제공하며(그림 5), 이는 사용된 파장 및 대물렌즈에 따라 최대 측면 해상도 ~60-70nm에 해당합니다. 핵의 평균 Feret 직경은 통계적 접근을 위해 여러 이미지에서 최소 60-100개의 세포를 측정하여 결정됩니다. 정확도를 높이기 위해 3D 재구성을 가능하게 하는 z-스택을 체계적으로 획득 및 처리하여 측정된 거리가 모든 핵의 실제 Feret 직경과 일치하는지 확인할 수 있습니다. 그러나 이 접근 방식은 사용된 현미경과 대물렌즈에 따라 획득하는 측면에서, 특히 데이터 처리 측면에서 훨씬 더 많은 시간이 소요될 수 있습니다. 이는 팽창 전과 후의 하이드로겔 크기를 비교하여 EF의 거시적 측정에 의해 교차 확인할 수 있으며, 이는 우리의 경험에 비추어 볼 때 유사한 결과로 이어집니다.

그림 5: 팽창 계수 결정. 확장 전후의 핵의 평균 크기는 ImageJ 소프트웨어에서 Feret 직경(즉, 핵 가장자리의 두 지점 사이의 가장 긴 거리)을 측정하여 결정됩니다. 평균 Feret 직경 post ExM과 pre ExM 사이의 비율을 계산하면 선형 팽창 계수가 제공됩니다. 인간 1차 섬유아세포 533T는 확장 프로토콜에 제출되었습니다. 핵은 Hoechst 33342(파란색으로 표시)로 염색하였다. 스케일 바 = 20μm. 약어: ExM = Expansion Microscopy. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이 프로토콜은 특정 세포 처리 또는 다양한 생리학적 및 병리학적 상태 간의 비교가 필요한 생물학적 질문을 해결하기 위해 고안되었습니다. 이는 항말라리아 약물인 클로로퀸(CQ)으로 처리된 인간 섬유아세포에 대한 적용으로 예시됩니다. pKa 값이 8.4 및 10.8인 이양성자 약한 염기인 CQ는 pH 트래핑을 통해 리소좀에 우선적으로 축적되어 리소좀 pH를 증가시키고 그에 따른 기능 상실을 초래합니다.
이 실험에서 ManNAz는 시알릴화 글라이칸에 통합된 다음 CuAAC를 통해 Alexa Fluor 488로 표지되고 골지체는 마우스 항-GM130 항체로 염색됩니다. 특히, 대조군 세포(그림 6A)와 CQ 처리된 세포(그림 6B) 간에 상대 강도의 뚜렷한 차이가 관찰됩니다. 대조 세포의 생리학적 조건하에서, sialoconjugate labeling의 1차 풀은 Golgi 장치 내에 국한되어 있습니다. 대조적으로, CQ 처리된 세포는 리소좀에서 시알산(sialic acid) 축적을 나타내며, 이는 재활용된 당접합체(glycoconjugate)의 포획을 입증합니다. 이는 선천성 글리코실화(glycosylation) 또는 리소좀 저장 질환(lysosomal storage disease)을 연구하는 맥락에서 특히 관련이 있다 13,14,15.

그림 6: PostExM 시알릴화된 당단백질의 국소화에 대한 클로로퀸의 효과에 대한 PostExM 시각화. 인간 1차 섬유아세포 533T는 (A) CQ로 처리하지 않았거나 (B) 24시간 동안 500μM ManNAz로 배양하기 전에 8시간 동안 100μM CQ로 처리하고 Alexa Fluor 488 알킨(녹색 채널)으로 CuAAC 표지를 처리했습니다. 골지체(적색 채널)의 공동 국소화 염색은 마우스 anti-GM130 1차 항체 및 anti-mouse Alexa Fluor 546 표지 2차 항체를 사용하여 수행되었습니다. 세포는 설명된 대로 앵커링(anchoring), 겔화(gelation), 분해(digestion) 및 팽창(expansion)을 거쳤다. 눈금 막대 = 20 μm. 약어 : ExM = 확장 현미경; CQ = 클로로퀸; ManNAz = N-아지도아세틸만노사민; CuAAC = 구리 촉매 아지드-알킨 고리첨가. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
본 CuAAC 라벨링 프로토콜은 반응 완충액에 아미노구아니딘을 포함하지 않는다. 세포 내 glycoconjugate를 시각화하는 것을 목표로 하기 때문에 세포 독성 문제를 방지하고 촉매 시스템의 흡수를 개선하기 위해 대사 통합 단계 후에 고정된 세포에서 수행됩니다. 아미노구아니딘의 사용은 일반적으로 단백질의 디하이드로아스코르베이트와 아르기닌, 히스티딘 및 라이신 잔기 사이의 부반응을 방지하기 위해 살아있는 세포의 세포 표면 표지에 권장됩니다28. 이러한 변형은 실제로 단백질 기능에 영향을 미칠 수 있으며 잠재적으로 섭동 또는 세포 독성을 유발할 수 있습니다. CuAAC 반응 완충액에 대한 또 다른 중요한 고려 사항은 BTTAA에 의한 구리 이온의 복합화가 빠르지만 즉각적이지는 않다는 것입니다. 변형된 설탕의 효율적인 라벨링을 보장하기 위해 라벨링 프로세스를 시작하기 최소 30분 전에 용액을 준비하여 초보 사용자가 결과를 재현할 수 있도록 하는 것이 좋습니다.
모든 컨포칼 현미경을 획득에 사용할 수 있습니다. 샘플이 확장됨에 따라 관찰 가능한 필드도 확장됩니다. 전체 셀을 캡처하려면 이미지 간에 10% 겹치는 영역의 타일 스캔을 수행하는 것이 좋습니다. 타일 스캔과 z-스택을 동시에 수행할 수 있지만 이 경우 더 작은 타일 스캔을 선택하는 것이 좋습니다. 이는 프로세스가 시간이 많이 걸리고 상당한 양의 원시 데이터를 생성하여 집중적인 데이터 처리로 이어지기 때문입니다. 두 방법을 모두 분리하여 확장 전 z-stack과 일치하는 매개 변수를 사용하여 관심 영역에 대해서만 z-stack을 만드는 것이 좋습니다. 팽창 젤에는 변색 방지 장착 매체가 사용되지 않기 때문에 광표백(빠른 스캔 속도, 낮은 레이저 강도)을 최소화하기 위해 그에 따라 획득 매개변수를 설정하는 것이 좋습니다.
성공적인 팽창 실험은 일반적으로 4에서 5의 팽창 계수로 이어지며, 이는 64배에서 125배 사이의 부피 증가에 해당합니다. 동일한 수의 형광단 분자가 팽창 후 훨씬 더 큰 부피에 퍼져 있기 때문에 ExM의 주요 단점은 신호 강도의 불가피한 감소입니다. 확장 후 형광 신호를 관찰할 수 있기 위해 여기 레이저의 게인 및/또는 강도는 형광단에 따라 크게 증가할 수 있습니다. 핀홀 개구부도 조절될 수 있습니다. 형광 신호에 대한 이러한 순전히 물리적 희석 효과 외에도 신호 손실의 다른 가능한 원인이 있습니다.
분자 앵커의 라벨링, 고정 및 생체 접합에 이어 겔화(프로토콜 단계 7.2)는 시료를 고분자 전해질 하이드로겔에 캡슐화하는 것을 목표로 합니다. 이러한 중합체는 아크릴아미드, 비스-아크릴아미드 및 아크릴산나트륨의 세 가지 단량체로 구성됩니다. 후자는 이온 성질로 인해 젤에 팽창 특성을 제공합니다. 사실, 이 중합체는 나트륨 이온을 물 분자로 교환하여 무게의 몇 배를 물에 흡수할 수 있습니다29. 겔의 가교는 TMEDA와 APS에 의해 촉매되는 라디칼 반응입니다. 추가 신호 감소의 원인은 이러한 라디칼 유도 중합 동안 유기 형광단의 부분적인 분해라는 점은 주목할 만합니다. 다양한 염료가 다양한 정도로 영향을 받습니다. 예를 들어, 시아닌 염료는 팽창 중에 거의 완전히 파괴되어 0.3-0.4 %30,31의 낮은 신호 유지를 나타내는 반면, Alexa Fluor 488 염료는 ExM4 이후 신호 강도의 ~57%를 유지할 수 있습니다. 다양한 유기 형광단과 형광 단백질의 신호 유지에 대한 요약은 Wen et al.31의 검토를 참조하십시오.
또한 생체 분자 보유는 프로토콜의 특정 단계에 의해 영향을 받을 수도 있습니다. 예를 들어, 하이드로겔에 고정되지 않은 단백질 단편에 위치한 글라이칸 사슬은 분해 중에 씻겨 나가 신호 강도를 더욱 감소시킬 수 있습니다(그림 4). 소화는 단백질분해효소 K 4,5,6,31을 포함하거나 포함하지 않은 트리톤 X-100과 같은 적어도 하나의 세제(32)를 사용하여 세포골격과 같은 막과 단백질 구조를 파괴한다. 분해 시간 및 분해 완충액의 조성은 특정 형광단 및 생체 분자를 가장 잘 보존하도록 조정될 수 있습니다. 따라서 분해 매개변수(프로토콜 단계 7.3)는 조성, 농도 및 지속 시간 측면에서 연구 중인 시료 유형에 맞게 최적화되어야 하며, 생체 분자 및 프로브의 등방성 팽창 및 체류를 보장하기 위해 균질화 간의 최상의 균형을 확보해야 합니다. 어떤 경우에는 분해 완충액의 유형이 겔 팽창26,27 전에 샘플의 단백질이 95 °C에서 SDS로 변성되는 Ultrastructure-ExM과 같은 더 가벼운 조건으로 완전히 수정 될 수 있습니다.
가능한 경우, 낮은 신호 포스트 ExM은 특정 프로브에 대한 필요 없이 이미징 데이터를 획득하는 동안 여기 소스의 전력 및/또는 검출기 카메라의 이득을 조정하여 간단히 해결할 수 있습니다. ExM 이후 신호 대 잡음비가 불충분한 경우, 예를 들어 형광단 자체를 고분자 매트릭스 6,14에 직접 고정할 수 있는 삼중 기능 분자를 활용하는 등 다양한 실험실에서 추가 신호 유지 및/또는 증폭 전략을 개발했습니다. 이러한 문제를 해결하기 위한 대체 솔루션으로, 겔이 충분히 투과성이 있기 때문에 팽창 후에 샘플의 염색을 수행할 수 있습니다. 그러나 이로 인해 팽창 전에 샘플을 관찰할 수 없습니다. 이 접근법은 최근 면역염색33에 대해 보고되었는데, 여기서 표본은 먼저 등방성으로 팽창한 다음 항체로 염색됩니다. 저자들은 이를 통해 형광 밝기와 에피토프 접근성을 개선하는 동시에 ExM과 초해상도 기술을 결합할 때 국소화 정확도를 방해하는 연결 오류를 줄일 수 있음을 입증했습니다.
형광 강도의 감소를 막기 위한 대안 전략이 연구되었습니다. 한 가지 예는 기존의 형광 분자 대신 폴리머 기반 형광단을 사용하는 것입니다. Liu 등은 기존 형광단보다 훨씬 밝은 형광 폴리머 도트(Pdots)34 를 개발하여 더 나은 신호 대 잡음비를 가능하게 했습니다. Pdots는 앵커와 항체로 기능화할 수 있습니다. 또 다른 예로, 클릭 확장 현미경(ClickExM)6 은 앵커가 있는 형광 스트렙타비딘을 사용하여 간접 비오틴-스트렙타비딘 전략으로 신호 유지를 향상시키는 보편적인 전략을 도입했습니다. 이를 통해 형광 프로브를 폴리머 프레임에 직접 이식할 수 있습니다. 마찬가지로, MAGNIFY(Molecule Anchorable Gel-enabled Nanoscale In-situ Fluorescence microscopY)35 는 형광 신호 유지를 증가시키기 위해 생체 분자 고정에 크게 의존합니다. 특정 분자를 겔에 결합하는 ClickExM과 달리, MAGNIFY는 메타크롤레인을 사용하여 광범위한 생체 분자를 하이드로겔 프레임에 가교결합시킵니다.
현미경에서 데이터 수집 측면에서 ExM의 또 다른 가능한 한계는 더 넓은 물리적 영역에서 확장된 샘플을 이미지화해야 한다는 점이며, 이로 인해 전체 샘플을 캡처하는 데 필요한 시간이 크게 늘어납니다. 확장 과정에서 샘플이 확대되기 때문에 고해상도 이미징을 달성하려면 동일한 생물학적 영역을 커버하기 위해 더 많은 이미지 타일을 캡처해야 하므로 획득 시간이 길어집니다. 이와 관련하여 많은 응용 분야에서 ExM은 기존 컨포칼 현미경을 대체하는 것이 아니라 포괄적이고 상세한 이미징을 위한 보완 기술로 간주될 것입니다.

그림 7: 인간 1차 섬유아세포 533T 세포의 preExM과 postExM 3D 이미징 비교. 세포를 500μM ManNAz로 24시간 동안 배양하고 CuAAC(녹색)에 의해 Alexa Fluor 488 알킨으로 표지했습니다. 골지체(Golgi apparatus)의 공동 국소화(co-localiation) 염색(빨간색)은 마우스 anti-GM130 1차 항체 및 anti-mouse Alexa Fluor 546 표지 2차 항체로 수행하고, 핵은 Hoechst 33342(파란색)로 염색하였다. (왼쪽) (A) preExM을 시각화한 셀의 Z-스택 및 (B,C) 3D 표면 렌더링. (오른쪽) 세포는 z-stack 획득 전에 (D) 설명된 대로 앵커링, 겔화, 분해 및 확장에 투여되었습니다. (E) 신호의 3D 표면 렌더링은 복잡한 세포 내 하부 구조를 감지할 때 특히 유용한 해상도 증가를 명확하게 보여줍니다. 3D 재구성 및 표면 렌더링은 신호 강도를 사용하여 생성되었습니다. 스케일 바 = 20μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
ExM은 실제로 회절 한계를 초과하지 않으면서 나노 단위 이미징을 달성하기 위한 혁신적인 기술로 부상했으며, 표준 프로토콜6에서 간과될 수 있는 생물학적 샘플의 세부 사항을 시각화할 수 있습니다. ~250nm 회절 제한 장비를 사용할 때(일반적으로 녹색 레이저가 장착된 일상적인 컨포칼 현미경의 경우와 같이) 4.5배의 팽창 계수는 일반적으로 60nm에 가까운 측면 해상도로 이어집니다. 이는 골지체(Golgi apparatus) 또는 소포(vesicle)와 같은 세포 내 구조를 연구할 때 매우 중요한 개선이며, 이는 2D 이미징뿐만 아니라 z-stack 획득에서 3D 이미지를 생성함으로써 활용될 수 있습니다(그림 7). 확장 현미경의 모든 기능을 활용하기 위해 ExM을 이 기사의 범위를 벗어나는 STED 또는 STORM과 같은 초고해상도 현미경 기술과 결합하여 달성된 해상도를 더욱 향상시킬 수 있습니다.
팽창 현미경 분야는 매우 빠른 성장과 상당한 발전을 목격하고 있어 흥미롭고 역동적인 연구 분야가 되고 있습니다. Boyden과 동료들은 2015년에 확장 현미경을 도입하여 고분자 매트릭스에서 생체 분자의 기하학을 유지하기 위해 겔 고정 가능 라벨을 사용하여 컨포칼 현미경으로 약 70nm 해상도를 달성했습니다. 초기의 관심에 이어 맞춤형 올리고뉴클레오티드-항체 접합체에 대한 필요성으로 인해 과학계는 이 방법을 위한 새로운 도구와 접근 방식을 개발하게 되었습니다. 상업적으로 이용 가능한 제제를 사용하여 단백질을 겔에 직접 고정하는 다양한 전략이 개발되어 형광 표지된 항체 또는 단백질을 사용할 수 있게 되었으며, 최근의 개발로 확장 현미경이 더욱 단순화되고 별도의 앵커링 단계 없이 다양한 생체 분자에 대한 적용이 확대되었습니다.
ExM을 통해 더 높은 해상도로 MOE 라벨링을 달성하는 것은 비용이 많이 들고 접근하기 어려운 초고해상도 현미경 없이 시알릴화 글라이칸의 공간 분포를 일상적으로 연구하는 데 특히 유용합니다. 예를 들어, 원형질막의 시알로글리칸은 이 방법으로 시각화되어 미세융모 및 기타 세포 구조에서의 분포를 밝혔습니다6. 향상된 해상도로 세포 내 글라이칸을 시각화하는 능력은 생합성, 수송 및 재활용 8,13,14,15을 연구하고 대사 질환 또는 암과 같은 병리학에서 글리칸의 역할을 해독하는 데 훨씬 더 중요합니다. ExM과 생물직교 화학을 결합하면 세포 조절 및 질병 메커니즘의 핵심인 세포 경로, 세포 기관 기능 및 상호 작용에 대한 ExM의 국소화 및 관여를 더 잘 이해할 수 있는 독특한 기회를 제공할 것입니다. 이것은 의심할 여지 없이 미래의 다양한 병리학에서 시알산이 수행하는 역할에 대한 우리의 이해를 향상시킬 것입니다.
공개
저자는 경쟁하는 재정적 이해관계나 기타 이해 상충이 없습니다.
감사의 말
이 작업을 수행하는 데 도움이 되는 기술 환경을 제공한 TisBio 시설과 PLBS 플랫폼에 감사드립니다. 이 작업은 CNRS와 Ministère de l'Enseignement Supérieur et de la Recherche의 보조금으로 지원되었습니다. 토론을 고무하고 Fibroblast 533T 세포주와 1차 항체 GM130을 제공해 주신 François Foulquier 박사, Zoé Durin, Dorothée Vicogne 여사, Céline Schulz 여사에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| (+) Sodium L-ascorbate | Sigma Aldrich | 11140 | |
| 12 well cell culture plate | Corning | 3513 | |
| Acrylamide | Sigma Aldrich | A8887 | |
| Acrylic acid N-hydroxysuccinimide ester | Sigma Aldrich | A8060 | |
| Alexa Fluor 488 alkyne | Jena Bioscience | CLK-1277-5 | |
| Alexa Fluor 546 goat anti-mouse IgG | Invitrogen | A11003 | |
| Amonium persulfate | Sigma Aldrich | 9913 | |
| Bis-Acrylamide | Sigma Aldrich | 146072 | |
| BSA | Sigma Aldrich | A7906 | |
| BTTAA | Jena Bioscience | CLK-067-100 | |
| Centrifugation tube 2 mL | EPPENDORF | 30120094 | |
| Chloroquine diphosphate salt | Sigma Aldrich | C6628 | |
| Conical tube 15 mL | Falcon | 352097 | |
| cover slips 12 mm #1 | epredia | CB00120RA120MNZ0 | |
| cover slips 32 mm #1 | epredia | CB00320RA140MNZ0 | |
| CuSO4 | Sigma Aldrich | 209198 | |
| DMEM high glucose medium | Dutscher | L0104-500 | |
| Dulbecco's Phosphate Buffered Saline (PBS) | Dutscher | L0615-500 | |
| Fetal Bovine Serum | biowest | S1810-500 | |
| Fibroblast 533T | - | - | Collected from healthy individual |
| FIJI ImageJ 2.9.0 | - | - | |
| Gelatin | Bio-RAD | 170-6537 | |
| Guanidine HCl | Sigma Aldrich | 50950 | |
| HeLa cells | ATCC | CCL-2 | |
| Hoechst 33342 | Sigma Aldrich | 14533 | |
| Imaris 10.2 | - | - | |
| K2HPO4 | Euromedex | PB0447-B | Anhydrous |
| LSM 780 Confocal Microscopy | Zeiss | - | |
| MCF7 | ATCC | HTB-22 | |
| N-acetylmannosamine (ManNAc) | BIOSYNTH | MA05269 | |
| NaCl | Carlo Erba | 479687 | |
| N-azidoacetylmannosamine (ManNAz) | BIOSYNTH | MA46002 | |
| Objectif "Plan-Apochromat" 63x/1,4 Oil DIC M27 | Zeiss | 420782-9900-799 | |
| Phosphate Buffered Saline (PBS) 10x | Euromedex | ET330 | |
| Proteinase K | Sigma Aldrich | P2308 | from Tritirachium album |
| purified mouse GM130 antibody | BD Bioscience | 610822 | 50 µg |
| Sodium acrylate | Sigma Aldrich | 408220 | |
| T75 Flask | Corning | 430641 | |
| TEMED | Sigma Aldrich | T9281 | |
| tris Acetate EDTA (TAE) 10x | Euromedex | EU0202-B | |
| Triton X-100 | Sigma Aldrich | X-100 | |
| Trypan Blue | Dutscher | 702630 | |
| Trypsine-EDTA 1x | Dutscher | L0930-100 |
참고문헌
- De Souza, N. Light microscopy at the limit. Nat Cell Biol. 11 (Suppl 1), S22(2009).
- Tam, J., Merino, D. Stochastic optical reconstruction microscopy (STORM) in comparison with stimulated emission depletion (STED) and other imaging methods. J Neurochem. 135, 643-658 (2015).
- Jeong, S., Widengren, J., Lee, J. C. Fluorescent probes for STED optical nanoscopy. Nanomaterials. 12 (1), 21(2021).
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543548(2015).
- Kang, S., et al. Expansion microscopy with a thermally adjustable expansion factor using thermoresponsive biospecimen-hydrogel hybrids. ACS Appl Mater Interfaces. 13 (24), 2896228974(2021).
- Sun, D., et al. Click-ExM enables expansion microscopy for all biomolecules. Nat Methods. 18 (1), 107113(2020).
- Dube, D., Bertozzi, C. Metabolic oligosaccharide engineering as a tool for glycobiology. Curr Opin Chem Biol. 7 (5), 616625(2003).
- Scache, J., et al. Switching azide and alkyne tags on bioorthogonal reporters in metabolic labeling of sialylated glycoconjugates: a comparative study. Sci Rep. 12 (1), 22129(2022).
- Holst, S., Wuhrer, M., Rombouts, Y. Glycosylation characteristics of colorectal cancer. Adv Cancer Res. 126, 203256(2015).
- Boyaval, F., et al. N-Glycomic signature of stage II colorectal cancer and its association with the tumor microenvironment. Mol Cell Proteomics. 20, 100057(2021).
- Ferreira, J. A., et al. Protein glycosylation in gastric and colorectal cancers: Toward cancer detection and targeted therapeutics. Cancer Lett. 387, 32-45 (2017).
- Jennings, M. P., Day, C. J., Atack, J. M. How bacteria utilize sialic acid during interactions with the host: snip, snatch, dispatch, match and attach. Microbiology. 168 (3), 001157(2022).
- Gilormini, P. A., et al. A sequential bioorthogonal dual strategy: ManNAl and SiaNAl as distinct tools to unravel sialic acid metabolic pathways. Chem Commun. 52 (11), 2318-2321 (2016).
- Vanbeselaere, J., et al. Alkynyl monosaccharide analogues as a tool for evaluating Golgi glycosylation efficiency: application to Congenital Disorders of Glycosylation (CDG). Chem Commun. 49 (96), 11293-11295 (2013).
- Gilormini, P. A., et al. Chemical glycomics enrichment: imaging the recycling of sialic acid in living cells. J Inherit Metab Dis. 4 (3), 515523(2018).
- Rigolot, V., Biot, C., Lion, C. To view your biomolecule, click inside the cell. Angew Chem. 60 (43), 2308423105(2021).
- Qin, W., et al. Artificial cysteine S-glycosylation induced by per-O-acetylated unnatural monosaccharides during metabolic Glycan Labeling. Angew Chem. 130 (7), 1835-1838 (2018).
- Qin, K., Zhang, H., Zhao, Z., Chen, X. Protein S-glyco-modification through an elimination-addition mechanism. J Am Chem Soc. 142 (20), 9382-9388 (2020).
- Rostovtsev, V. V., Green, L. G., Fokin, V. V., Sharpless, K. B. A. Stepwise Huisgen cycloaddition process: Copper(I)-catalyzed regioselective "ligation" of azides and terminal alkynes. Angew Chem. 41 (14), 25962599(2002).
- Agard, N. J., Prescher, J. A., Bertozzi, C. R. A strain-promoted [3 + 2] azide−alkyne cycloaddition for covalent modification of biomolecules in living systems. J Am Chem Soc. 126 (46), 15046-15047 (2004).
- Mbua, N. E., et al. Strain-promoted alkyne-azide cycloadditions (SPAAC) reveal new features of glycoconjugate biosynthesis. ChemBioChem. 12 (12), 19121921(2011).
- Blackman, M. L., Royzen, M., Fox, J. M. Tetrazine ligation: Fast bioconjugation based on inverse-electron-demand Diels-Alder reactivity. J Am Chem Soc. 130 (41), 1351813519(2008).
- Devaraj, N. K., Weissleder, R., Hilderbrand, S. A. Tetrazine-based cycloadditions: Application to pretargeted live cell imaging. Bioconjug Chem. 19 (12), 22972299(2008).
- Bird, R. E., et al. Bioorthogonal chemistry and its applications. Bioconjug Chem. 32 (12), 2457-2479 (2021).
- Slade, P. G., et al. Identifying the CHO secretome using mucin-type O-linked glycosylation and click-chemistry. J Proteome Res. 11 (12), 61756186(2012).
- Gambarotto, D., Hamel, V., Guichard, P. Ultrastructure expansion microscopy (U-ExM). Methods Cell Biol. 161, 5781(2021).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nat. Methods. 16, 71-74 (2019).
- Presolski, S. I., Hong, V. P., Finn, M. Copper-catalyzed azide-alkyne click chemistry for bioconjugation. Curr Protoc Chem Biol. 3 (4), 153-162 (2011).
- Santiago, F., et al. Synthesis and swelling behaviour of poly(sodium acrylate)/sepiolite superabsorbent composites and nanocomposites. Polym Int. 55 (8), 843848(2006).
- Wen, G., et al. Evaluation of direct grafting strategies via trivalent anchoring for enabling lipid membrane and cytoskeleton staining in expansion microscopy. ACS Nano. 14 (7), 78607867(2020).
- Wen, G., et al. Current progress in expansion microscopy: Chemical strategies and applications. Chem Rev. 123 (6), 32993323(2023).
- Drelich, L., et al. Toward high spatially resolved proteomics using expansion microscopy. Anal Chem. 93 (36), 12195-12203 (2021).
- Zwettler, F. U., et al. Molecular resolution imaging by post-labeling expansion single-molecule localization microscopy (Ex-SMLM). Nat Commun. 11 (1), 3388(2020).
- Liu, J., et al. Expansion microscopy with multifunctional polymer dots. Adv Mater. 33 (25), 2007854(2021).
- Klimas, A., et al. Magnify is a universal molecular anchoring strategy for expansion microscopy. Nat Biotechnol. 41 (6), 858869(2023).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유