Method Article
Click Chemistry and Expansion Microscopyによる細胞内シアル化の可視化
要約
ここでは、代謝オリゴ糖工学、クリックケミストリー、および増殖顕微鏡法を組み合わせたシンプルなプロトコールを提案し、日常的な顕微鏡装置を使用して細胞内シアル化 N-糖タンパク質のバイオイメージングを分解能を向上させました。
要約
メタボリックラベリング技術により、生体直交レポーターを糖鎖に組み込むことができ、クリックケミストリーや生体直交化学により、細胞内の分子色素の標的生体結合が可能になります。代謝オリゴ糖工学(MOE)は、分子認識を含む多数の生物学的プロセスにおけるグリコシル化の本質的な役割と、癌から遺伝性疾患、ウイルスおよび細菌感染に至るまでの病状への影響により、かなりの関心を集めています。
MOEは細胞表面の複合糖質の検出でよく知られていますが、生理学的および病理学的状況における細胞内糖鎖の研究においても非常に重要な方法論です。このような研究は、高い空間分解能から大きな恩恵を受けています。しかし、超解像顕微鏡はほとんどの研究室で容易に利用できず、日常的な導入に課題を提起しています。拡大顕微鏡は、蛍光マーカーで標識された生体試料を物理的に拡大することにより、顕微鏡の解像度を向上させる最近の代替手段です。サンプルを膨潤性ゲルに埋め込み、化学処理によって等方性に膨張させることにより、超解像技術を必要とせずに、細胞内構造をより高い精度と分解能で視覚化できます。
この研究では、MOEとクリックケミストリーを組み合わせて細胞内シアル化糖鎖を可視化する拡大顕微鏡の能力を示します。具体的には、共局在研究のための免疫蛍光法に関連する可能性のあるシアリル化を標的とするレポーターを用いた生体直交標識および拡大顕微鏡法の手順を提案します。このプロトコルにより、シアロコンジュゲート生合成、細胞内輸送、およびリサイクルのローカリゼーション研究が可能になります。
概要
蛍光顕微鏡は、細胞内の特定の分子の標識や可視化に広く使用されていますが、アッベの光の回折限界1によって本質的に分解能が制限されており、約200〜250nmより近い物体を区別する能力が制限されます。この制限は、光の波の性質と顕微鏡の対物レンズの開口数から生じ、細胞内構造をイメージングする際に課題をもたらします。これらの制限を克服することで、ナノメートルスケールでの特定の生物学的プロセスに関するより深い洞察が得られます。
光の回折限界を超えるために、STORM(Stochastic Optical Reconstruction Microscopy)やSTED(Stimulated Emission Depletion)などの超解像顕微鏡技術が開発されました2,3。STORMは、蛍光色素の確率的活性化に依存しているため、任意の時点でスパースなサブセットのみをイメージングできます。これにより、個々の蛍光色素の正確な局在化が可能になり、高解像度の画像再構成が可能になります。一方、STEDは、デプレッションレーザーを用いて励起スポットの周辺部の蛍光を選択的に消光することで分解能を向上させ、点像分布関数を効果的に狭めます。
これらのアプローチは、すべての蛍光色素が同時に検出される広視野顕微鏡や共焦点顕微鏡法とは対照的であり、すべての回折パターンを組み合わせた画像が得られ、近くにある個々の蛍光色素の区別を防ぎ、解像度の低下につながります。しかし、これらの超解像法には、非常に特殊な光源、装置、サンプル調製、および/または蛍光色素が必要であるため、これらの技術は高価で、ほとんどの研究室でアクセスが困難であり、日常的な実験での実装が困難です。これらの制約により、科学界は、容易に利用できる顕微鏡装置や日常的な染色プロトコルと互換性がある、より高い分解能を達成するための代替ソリューションを探すようになりました。2015年、Boyden氏らは、サンプルを物理的に拡張することで光学顕微鏡の限界を回避する方法、Expansion Microscopy(ExM)4を開発しました。
ExMは、光の回折限界を超えることなく、生体試料のナノスケールの詳細を提供する3段階の手法です(図1)。代わりに、従来の回折限界顕微鏡を使用して、等方性的に物理的に拡大されたサンプルを画像化します。ゲル化と呼ばれる最初のステップは、アクリル酸ナトリウムとアクリルアミドをベースにした膨潤性高分子電解質ヒドロゲルに、生体サンプル(通常は固定された細胞または組織)を埋め込むことです。その後、生体サンプルは酵素処理を受けて、タンパク質や膜などの特定の成分を部分的に分解し、高密度の細胞構造を分解して細胞が均一に増殖できるようにします。分解と呼ばれるこのステップは、機械的特性に関してサンプルの構造成分を均質化することでこれを達成し、サンプルの歪みにつながる可能性のある膨張差を防ぎます。最後に、拡張と呼ばれる最後のステップでは、ハイドロゲルを包埋して分解したサンプルを脱イオン水に入れ、膨潤させます。この技術により、サンプルはすべての次元で約4〜5倍の線形倍率で拡大し、標準的な顕微鏡技術を使用して細胞の微細な詳細を視覚化することができます。膨張の等方性を考えると、ゲルマトリックス内で拡大された生物学的サンプルは、その3次元の幾何学的詳細と、そのさまざまな構造成分間の空間的関係を保持します。したがって、空間情報は膨潤しても保持されますが、ゲルアンカーされた蛍光標識または生体分子との間の距離は、すべての方向に均一に増加します。これにより、信号の分離が向上し、分解能が向上します。

図1:ExMプロトコルの概要(a)ゲル化:生体サンプルは、膨潤性高分子電解質ハイドロゲル内に固定され、埋め込まれています。(b)消化:サンプルの機械的特性は、酵素と界面活性剤を使用して均質化され、そうでなければ膨張を制限し、歪みを引き起こすタンパク質と脂質膜を分解します。(c)膨張:ヒドロゲルを脱イオン水に浸し、等方性に膨張させます。この図の拡大版を表示するには、ここをクリックしてください。
2015年以降、ExMは空間情報の保存において大幅な技術的改善が見られました。特に、様々な分子アンカーは、膨潤性ヒドロゲル5,6に直接生体分子を共有結合的に結合するように設計されている。このプロトコールでは、生体分子の遊離アミン基(通常はリジン残基またはタンパク質のN末端位置)と反応するN-アクリルオキシスクシンイミド(NAS)を使用して、アクリロイル基を糖タンパク質を含む細胞のタンパク質に生体結合しました。このアクリロイル基は、ゲル化中に架橋反応を通じて他のモノマーと反応し、これらの生体分子を直接ポリマーに固定します。
ExMは、メタボリックオリゴ糖工学(MOE)を含む幅広い標識法に対応しています。MOEは、生体直交的なケミカルハンドル7を備えた代謝前駆体の類似体を組み込むことにより、糖鎖の標識を可能にする強力なツールです。これらの類似体は化学レポーターとして知られており、毒性を引き起こすことなく代謝経路に組み込まれます。MOEアプローチでは、レポーター単糖類が代謝的に活性化ヌクレオチド糖に加工され、その後、新生複合糖質に移行します。私たちの主な焦点は、シアリル化の研究、特に細胞間相互作用、免疫調節、および発生における役割のために健康と疾患の文脈で重要なシアリル化N-糖タンパク質の細胞内動態と輸送の研究です。異常なシアリル化は、がん8,9,10,11、感染症12、遺伝性疾患13,14,15などの疾患に関与しており、治療法の開発やバイオマーカーの発見における重要な標的となっています。シアリル化を理解することで、糖鎖生物学や疾患のメカニズムに関する洞察を深めることができます。
シアル化は、ヒトに最も多く存在するシアル酸であるN-アセチルノイラミン酸(Neu5Ac)の類似体、またはNeu5Acの代謝前駆体であるN-アセチルマンノサミン(ManNAc)の類似体を用いてMOEを用いて調べることができる8,16。ManNAcはサイトゾルでNeu5Acに変換され、核内でシチジン-5′-モノホスホ-N-ノイラミン酸(CMP-Neu5Ac)に活性化されます。ゴルジ体装置内のシアリルトランスフェラーゼは、ヌクレオチド糖に活性化されると、Neu5Ac単位を成長中の糖鎖の末端位置に移動します(図2)。不自然なManNAc誘導体が代謝的に取り込まれた後、タグ付けされたシアル化糖鎖は、生体直交的なクリックケミストリーを通じて、レポーターハンドルに相補的な反応性基を持つフルオロフォアに共有結合することができます。これにより、in vivoまたはex vivoで複合糖質を直接観察することができます。
ほとんどの単糖レポーターにとって、受動的拡散を介して原形質膜を通過するには、過アセチル化型が必要です。しかし、過アセチル化されたレポーターと保護されていないレポーターの両方が、シアル化を効率的にプローブすることが示されています16。無防備なシアル化レポーターは、Neu5Ac類似体のピノサイトーシスとManNAcのまだ同定されていないトランスポーターという能動的な輸送メカニズムによって細胞に侵入することができます。保護されていない糖は、同等の効果を得るためにより高い濃度(通常は100〜500μM)を必要としますが、細胞内に入れば、シアル酸代謝経路に直接侵入することができます。対照的に、ペルアセチル化糖は、代謝活性になる前に、細胞内の非特異的エステラーゼによって完全に脱アセチル化する必要があります。低濃度(通常は10〜50 μM)で使用できますが、不完全な脱アセチル化は酵素活性を妨げたり、部分的にアセチル化されたシアル酸類似体が組み込まれたりして、下流の分析に支障をきたす可能性があります。さらに、酢酸の放出は局所的にpHに影響を与え、細胞機能に影響を与える可能性があります。また、Chenらは、Per-O-アセチル化糖が非酵素的メカニズムを介してタンパク質中の遊離システイン残基と反応し、オフターゲット取り込みと非特異的シグナルの増加につながることを実証しました17,18。したがって、現在のプロトコルでは、保護されていないManNAcレポーターを雇用しています。

図2:代謝性オリゴ糖のエンジニアリングとシアル酸の標識。 UDP-GlcNAcは、サイトゾルのGNE/MNKのUDP-GlcNAc 2-エピメラーゼドメインによってManNAcに変換されます。その後、ManNAcは、GNE/MNKのManNAc 6-キナーゼドメインによってサイトゾル内でリン酸化され、ManNAc-6-リン酸を形成します。 N-アセチルノイラミン酸シンターゼは、ManNAc-6-Pとホスホエノールピルビン酸との縮合を触媒してNeu5Ac-9-リン酸を生成し、その後、シアル酸ホスファターゼによって脱リン酸化されてNeu5Acを生成します。Neu5Acは、エンドサイトーシスおよびリソソームリサイクル8を介してサルベージ経路によっても供給される。核に輸送された後、CMP-シアル酸合成酵素によってCMP-Neu5Acに変換されます。ゴルジ体では、CMP-NeuAcは、成熟した複合糖質上の糖鎖の末端位置にNeu5Ac部分を導入するシアリルトランスフェラーゼの基質であり、最終的に細胞膜で発現または分泌されます。生体直交ハンドルを持つManNAzケミカルレポーターは、未確認の活性トランスポーターを介して細胞に侵入し、代謝経路に入ることができます。生合成後に糖鎖に取り込まれた標識Neu5Azユニットは、CuAACを介した蛍光プローブの標識によって標識されます。略語:NAc = N-アセチル;UDP-GlcNAc =ウリジン二リン酸 N-アセチルグルコサミン;ManNAc = N-アセチルマンノサミン;GNE = UDP-GlcNAc 2-エピメラーゼ;MNK = ManNAc 6-キナーゼ;ManNAc-6-P = ManNAc-6-リン酸;NANS = N-アセチルノイラミン酸合成酵素;PEP =ホスホエノールピルビン酸;Neu5Ac = N-アセチルノイラミン酸;Neu5Ac-9-P = Neu5Ac-9-リン酸;NANP = シアル酸ホスファターゼ;CMP =シチジン-5'-一リン酸;CMAS = CMP-シアル酸シンテターゼ;ST=シアル酸転移酵素;ManNAz = N-アジドアセチルマンノサミン;Neu5Az = N-アジドアセチルノイラミン酸。 この図の拡大版を表示するには、ここをクリックしてください。
可能な生体直交反応の中で、銅触媒アジド-アルキン環化付加反応(CuAAC)16,19に関心を集中させました。私たちの経験によると、CuAACは固定細胞の細胞内糖鎖を標識するための最良の選択肢です。この反応は十分に確立されており、生体直交化学で徹底的に研究されており、複雑な生物学的環境で使用するために標準化されており、知識の強固な基盤と最適化されたプロトコルを提供しています。その高速反応速度は、確かに高い反応効率と特異性を提供し、合成が容易で安定で、生体系に存在せず、天然の生体分子に対して不活性なアジド基とアルキン基を含んでいるため、銅の毒性が問題にならない固定細胞アプリケーションに最適です。株促進アルキン-アジド環化付加(SPAAC)20,21は、銅の毒性を回避しながら、速度論が遅く、プローブがかさばるため、疎水性トラップによる細胞内アプリケーションのバックグラウンドシグナルが高くなり、信号対雑音比が低下する傾向があるのに対し、逆電子要求量ディールス・アルダー(IEDDA)22,23は高速で銅フリーですが、より複雑なプローブ合成を伴い、MOEアプリケーションではまだ完全に特性評価されていないかさばるレポーターグループが必要です。ExMは固定細胞のみを必要とするため、CuAACは生体直交標識のための堅牢で効率的なソリューションを提供します。
この論文では、代謝標識、生体直交クリックケミストリー、および拡大顕微鏡法を組み合わせて、細胞内の細胞内シアル化糖タンパク質を可視化するためのプロトコルを提示します。本論文で述べる実験手順では、化学レポーターとして N-アジドアセチルマンノサミン(ManNAz)を利用し、膨張手順に先立って低分子有機フルオロフォアのCuAACライゲーションを行います。糖タンパク質は、ゲル化、消化、および増殖の前にNASでハイドロゲルに固定されます。シアル酸のMOE標識は、 シスゴルジ体に局在するマウス抗GM130一次抗体に例示されるように、共局在評価のための免疫蛍光法と関連付けることができます。このプロトコールは、生理学的状態の細胞、または化学処理を受けた細胞に適用することができます。これを説明するために、クロロキンを使用して、細胞内の糖タンパク質の処理と輸送に影響を与えるリソソーム機能を阻害しました。核染色は、細胞の局在化だけでなく、ExMプロセスの品質を示す指標としても、ランドマークとして使用されます。膨張係数は、膨張前(preExM)と膨張後(postExM)の原子核のサイズを比較することで、実際に測定できます。
プロトコル
1. 細胞播種
注:次のステップは、層流フードの下の無菌条件下で実行します。この方法は、本研究で使用された細胞株(HeLa、MCF7、初代線維芽細胞)のいずれか、または研究で一般的に使用されるほとんどの接着性細胞株モデルに適用できる20,24,25。
- T75フラスコに10%ウシ胎児血清(FBS)を添加したDMEM高グルコース培地で、5%CO2 雰囲気下で37°Cで細胞を増殖させます。
- 細胞が完全にコンフルエントになったら、細胞培養培地を取り出し、4mLのPBSで細胞を洗浄します。
注:このステップおよびその後のすべての洗浄ステップで使用されるPBS溶液は、使用前に0.2μmPTFEメンブレンによるろ過により滅菌する必要があります。 - 0.5 g/Lのトリプシンを含む1x トリプシン-EDTA溶液2 mLで、細胞を37°Cで5分間インキュベートし、フラスコの底から細胞を放出します。
- 10% FBSを含むDMEM高グルコース培地8mLを加え、十分に混合して、すべての細胞がフラスコの底から剥離していることを確認します。
- 細胞を15mLのコニカルチューブに移し、200 × gで5分間遠心分離します。それまでの間、12ウェルプレートの各ウェルにカバーガラス(直径12 mm、厚さ130-160 μm)を1枚追加します。
注:カバーガラスの厚さは、後続のイメージングステップで使用する顕微鏡の要件に合わせて調整する必要があります。 - 遠心分離後に上清を除去し、ペレットを1 mLの細胞培養培地に再懸濁します。
- 少なくとも2つの測定値にわたる血球計算法により、平均細胞数/mLを決定します。
- 細胞懸濁液を細胞培養培地で1mLあたり300,000細胞の濃度に希釈し、懸濁液1 mLを各ウェルに播種します。
- 次のステップに進む前に、5%CO2 雰囲気下で37°Cで10%FBSを含むDMEM高グルコース培地で細胞を24時間沈殿させます。
2.クロロキン治療(オプション)
注:ステップ2.1-2.8は、クロロキンを例に、外部試薬(阻害剤、エフェクター、薬物)で処理された細胞にプロトコールを使用する方法を示しています。外部試薬で未処理の細胞については、これらのステップをスキップしてください。
- 適切な量のクロロキン二リン酸塩(CQ)を秤量し、十分な量の10 mMストック溶液を調製します。
注:次のステップは、層流フードの下の無菌条件下で実行します。 - 固体CQを所望量の脱イオン水に可溶化し、最終濃度10 mMにします。
- 0.2 μmフィルターで溶液をろ過します。
- 10 mMストック溶液1 mLを1.5 mLの微量遠心チューブに分注し、-20°Cで保存します。
- ステップ1.9で調製した細胞サンプルを1 mLのPBSで洗浄します。
- 10% FBSを含む9.9 mLのDMEM高グルコース培地に100 μLの10 mM CQを添加すると、最終濃度は100 μMのCQになります。
- 100 μMのCQを添加した培地1 mLで、5% CO2 雰囲気下で37°Cで細胞を8時間インキュベートします。
- 細胞を1mLのPBSで3回洗浄します。
3. メタボリックオリゴ糖工学
- 10% FBSを含むDMEM高グルコース培地にManNAz(またはネガティブコントロール条件の場合はManNAc)の5 mMストック溶液を調製します。
注:このストック溶液は、各ウェルを1 mLの糖アナログ溶液で処理できる十分な容量である必要があります。次のステップは、層流フードの下の無菌条件下で実行します。 - 0.2 μmフィルターで溶液をろ過し、必要になるまで-20°Cで1 mLアリコートで保存します。
- ステップ 3.2 で調製した 1 mL のストック溶液アリコートを 10% FBS を含む 9 mL の DMEM 高グルコース培地で希釈することにより、500 μM の ManNAz または ManNAc を添加した培地を調製します。
- ステップ1.9(未処理細胞での実験用)またはステップ2.8(CQ処理細胞での実験用)で調製した細胞を、500 μM ManNAz(または対照群では500 μM ManNAc)を添加した培地1 mLで、5% CO2 雰囲気下で37°Cで24時間インキュベートします。
4. 固定と透過化
注:これらの手順はすべてドラフトの下で実行されます。
- サンプルをPBS1 mLで洗浄します。
- 500 μLの5%パラホルムアルデヒドを加えて、各カバーガラスに細胞を固定します。室温で15分間休ませます。
- PBS1mLで3回洗浄します。
- 蓋付きの十分に大きな不透明なボックス(ポリスチレンボックスなど)を使用して、カスタムの湿度チャンバーを準備します。箱の底に濡れたあぶらとり紙を置き、その上にパラフィルムの層を置きます。
注:湿度の高いチャンバーは、閉じたときにサンプルを外部の光源から完全に保護する必要があります。 - カバースリップをパラフィルムに置き、セルを上に向けて置きます。
注:事前にサンプルの名前または参照をパラフィルムにラベル付けしてください。 - 0.5% (v/v) Triton X-100 200 μL を PBS 中で 15 分間室温で使用して細胞透過化を行います。
- 200 μLのPBSで3回洗浄します。
5.蛍光標識
- タグ付きシアル化糖鎖のCuAAC標識
注:CuAAC反応バッファーは使用前に新たに調製する必要があり、反応物は指定された順序で添加し、アスコルビン酸ナトリウムを最後に添加する必要があります。酸素化水溶液中で、Cu(II)をアスコルビン酸で還元して形成したCu(I)イオンは徐々にCu(II)に再酸化し、時間の経過とともにアスコルビン酸濃度が低下します。結果の再現性を維持するために、バッファーは4°Cで数日以上保存せず、できれば同じ日に使用してください。- セクション 3 で操作したアジド修飾シアル化糖鎖を標識するには、5 μM AlexaFluor 488 アルキン (AF488Alk)、150 μM CuSO4、300 μM 2-(4-((ビス((1-(tert-ブチル)-1H-1,2,3-トリアゾール-4-イル)メチル)アミノ)メチル)-1H-1,2,3-トリアゾール-1-イル)酢酸(BTTAA)、0.1 M K2HPO4、および 2.5 mM アスコルビン酸ナトリウムからなる CuAAC 反応バッファーを調製します。各カバースリップに200μLを塗布するのに十分な溶液を準備します。
- 各カバーガラスに200 μLのCuAACバッファーを添加して、反応を開始します。サンプルが液滴で均一に覆われていることを確認し、光から保護された室温で45分間反応させます。
- 200 μLのPBSで3回洗浄して反応を停止し、余分なプローブと試薬を取り除きます。
- 核染色
- Hoechst 33342による核標識では、10 mg/mLのストック溶液をPBSで1:2000に希釈します。
注:4',6-ジアミジノ-2-フェニルインドール(DAPI)などの同等の核対比染色も同じ効果で使用することができます。 - この溶液200μLで細胞を覆い、光から保護した室温で5〜10分間インキュベートします。
- 200 μLのPBSで3回洗浄します。
- Hoechst 33342による核標識では、10 mg/mLのストック溶液をPBSで1:2000に希釈します。
- 免疫蛍光染色
- 50 mLのバッファーに対して、0.1 gのゼラチン、1 gのBSA、1 mLのFBS、および49 mLのPBSを含むウシ血清アルブミン(BSA)ブロッキングバッファーを調製します。
- サンプルを200 μLのBSAブロッキングバッファーで4°Cで1時間インキュベートします。サンプルを光から保護します。
- 精製マウス抗GM130抗体(エピトープ認識:ラットGM130 aa.869-982 |RRID:AB_398142)を調製したBSAブロッキングバッファーで一次抗体を1:100に希釈します。
注:このステップは、関連する一次抗体を使用して他のオルガネラを標的とする免疫蛍光染色に適合させることができ、その場合、希釈係数はメーカーのガイダンスに従って適合させる必要があります。 - 抗体を含む溶液70 μLをカバーガラスに加え、光から保護した状態で4°Cで1時間インキュベートします。
- 200 μLのPBSで3回洗浄します。
- 蛍光二次抗体(Alexa Fluor 546 anti-mouse IgG)をBSAブロッキングバッファーで1:600に希釈し、各カバーガラスに100 μLを加えます。
- サンプルを室温で1時間インキュベートし、光から保護します。
- 200 μLのPBSで3回洗浄します。
- カバースリップを6ウェルプレートに移します。サンプルを2 mLのPBSに4°Cで数日間保存し、光から保護します。
注:拡張前と拡張後の比較目的の参照として、および拡張後にラベリングパターンが歪んでいないことを確認するために、常に視覚化専用の複製preExMを準備することをお勧めします。
6. サンプルのプレエキスパンドイメージング
注意: 次の手順は、使用するバイオイメージング機器によって異なり、マシンの要件に合わせて調整する必要がある場合があります。地域の研究所またはバイオイメージングプラットフォームのルールに従ってください。ここでのステップ6.1-6.13は、レーザー走査型共焦点顕微鏡で実行されます。
- 顕微鏡の電源を入れ、光源(レーザー)が温まっていることを確認します。バイオイメージング取得ソフトウェアを開きます。
- 蛍光色素に対応するレーザー励起波長を選択してチャンネルを作成します(例:核染色の場合は405 nm、AF488Alk標識シアル化糖鎖の場合は488 nm、免疫蛍光標識の場合は546 nm)。それに応じてレーザーをアクティブにします。
- 直径32mmのカバースリップ(厚さ130〜170μm)を顕微鏡に適合したホルダーに置き、固定します。
- ステップ5.3.9で調製したサンプルを、セルを下に向けて32 mmカバーガラスの上に置きます。脱イオン水を一滴加えて、サンプルが乾燥するのを防ぎます。
- 適切な対物レンズ、ここでは開口数1.4または同等の63倍油浸対物レンズを選択します。
注意: 最も高い開口数が最高の解像度を提供します。オイル浸対物レンズを使用する場合は、対物レンズとカバーガラスの間にオイルを一滴追加することを忘れないでください。 - ホルダーを対物レンズの上の顕微鏡ステージに置きます。
- 明視野または低レーザー強度の蛍光モードを使用して、サンプル内の関心領域を特定します。
- フォーカスを設定します。
- ソフトウェア内の細胞を ライブ蛍光 モードで可視化します。各チャンネルのレーザー強度を設定します。光退色を避けるために、低レーザー出力から始めます。レーザー出力をゆっくりと上げ、検出器のゲインとオフセットを調整して、過度のノイズを導入せずに信号を増幅し、過度の露出なしに蛍光をはっきりと視覚化します。
注意: パラメータは、バックグラウンドノイズを最小限に抑え、信号の飽和を避けるために調整する必要があります。ネガティブコントロールを含む一連の実験において、すべてのサンプルで一貫した設定を維持することが重要です。 - ピンホールサイズ(例:1エアリーユニット)、スキャン速度(例:8)、ズーム係数(例:1)、輝度値の範囲(例:16ビット)、平均数(例:2)、モード(例:ライン)、方法(例:平均)などの画像パラメータを必要に応じて設定し、目的の観測に到達します。
注:これらの値はシステムおよびサンプルに依存し、取得前に最適化する必要があります。ピンホールのサイズは、開口数、倍率、波長によって異なり、ピンホールを小さくすると、S/N比が犠牲になっても分解能が向上する可能性があります。光退色を避けるために、スキャン速度を速い値(ピクセルあたりの小さな滞留時間に対応する)に設定することをお勧めします。 - 集録を実行し、将来の参照のために適切なメタデータを使用して目的の形式でデータを保存します。
- 取得が終了したら、少量の脱イオン水を追加してサンプルを6ウェルプレートに戻し、ピンセットで移し替えを容易にします。
注:ステップ6.12は、この特定のサンプルの拡張が必要な場合にのみ実行されます。それ以外の場合は、サンプルを廃棄し、次の手順で他の反復を使用します。
7. 拡張顕微鏡のプロトコル
注:サンプルは、光退色を避けるために、光から可能な限り保護されています。アンカリングとゲル化のステップは、ヒュームフードの下で行われます。
- アンカー
- サンプルを N-アクリロイルスクシンイミド(NAS)とPBS中濃度3.2 mg/mLのインキュベートで、機械式シェーカーで室温で1時間インキュベートします。ウェルあたり500 μLの溶液を添加するのに十分な量の溶液を調製します(ストック溶液:16 mg/mL NASのDMSO溶液)。
注:NASは、タンパク質のリジン残基および N末端と反応してアクリロイル基をグラフトし、その後のステップでタンパク質がヒドロゲルに固定できるようにします。 - PBS1mLで3回洗浄します。
- サンプルを N-アクリロイルスクシンイミド(NAS)とPBS中濃度3.2 mg/mLのインキュベートで、機械式シェーカーで室温で1時間インキュベートします。ウェルあたり500 μLの溶液を添加するのに十分な量の溶液を調製します(ストック溶液:16 mg/mL NASのDMSO溶液)。
- ゲル
- 1x PBS(pH 7.4)50 mLに、5.843 gのNaCl、1.25 gのアクリルアミド、0.075 gのビスアクリルアミド、および4.313 gのアクリル酸ナトリウムを加えます。各カバーガラスに70μLを塗布するのに十分な溶液を準備します。1 mLのアリコートをゲル化の少なくとも24時間前に凍結します。
注意:モノマーは有毒です。それらは、適切な保護具を使用して慎重に取り扱われ、ドラフトの下で計量する必要があります。 - パラフィルム上に70μLのモノマー溶液を滴下し、続いて1.4μLの10%(v / v)N、N、N'、N'-テトラメチルエチレンジアミン(TMEDA)を添加し、次に1.4μLの10%(w / v)過硫酸アンモニウム(APS)を添加し、滴下に混合します。
注:架橋剤試薬は、最初にTMEDA、次にAPSという特定の順序で追加する必要があります。 - すぐに、セルを下に向けて、カバースリップを溶液の上に注意深く置きます。
- 溶液を湿ったチャンバー内で室温で1時間重合させます。
- 1x PBS(pH 7.4)50 mLに、5.843 gのNaCl、1.25 gのアクリルアミド、0.075 gのビスアクリルアミド、および4.313 gのアクリル酸ナトリウムを加えます。各カバーガラスに70μLを塗布するのに十分な溶液を準備します。1 mLのアリコートをゲル化の少なくとも24時間前に凍結します。
- 消化
- 1x TAEバッファー、0.5%(v/v)Triton X-100、0.8 MグアニジンHCl、および8 U/mLプロテイナーゼKからなる消化バッファー溶液を調製します。
注:消化バッファーは、使用前に新たに調製し、37°Cで30分間プレインキュベートする必要があります。 - ステップ7.2で形成したヒドロゲルに1 mLの消化緩衝液を加えます。機械式シェーカーで37°Cで3時間消化を進めます。
- 消化バッファーを取り出し、ゲルを2 mLの脱イオン水で3回穏やかに洗浄します。
注:ヒドロゲルはわずかに膨張し、カバースリップから外れます。
- 1x TAEバッファー、0.5%(v/v)Triton X-100、0.8 MグアニジンHCl、および8 U/mLプロテイナーゼKからなる消化バッファー溶液を調製します。
- 拡張
- ヒドロゲルを3 mLの脱イオン水で2時間膨張させます。30分ごとに水を交換してください。
8. サンプルの増殖後のイメージング
- 直径32mmのカバースリップ(厚さ130〜170μm)を置き、ホルダーに固定します。
- ヒドロゲルの一部を切り取り、セルをゲルの底に付けてカバースリップに置きます。
注:切除されたピースは、観察に十分な表面積を提供するのに十分な大きさである必要がありますが、ゲルの取り扱いが困難になり、破損のリスクが高まる可能性があるため、大きすぎないようにする必要があります(推奨サイズ:5〜8 mm)。カバーガラスの表面全体を覆うのに十分なヒドロゲル片を配置します。これにより、ピースを所定の位置に固定し、イメージング中のドリフトを防ぐことができます。ドリフトの問題がある場合は、カバーガラスをポリ-D-リジンで事前にコーティングすると有利になる可能性があります6。 - ホルダーを対物レンズの上の顕微鏡ステージに置きます。
- ステップ6で説明したように、油浸対物レンズを備えたレーザー走査型共焦点顕微鏡で画像取得を実行します。
9. ImageJでの拡張係数(EF)の計算
- ImageJフリーウェアまたは同等の画像データを開きます。
- 複数のチャネル イメージで作業している場合は、チャネルを分割し、Select Image |カラー |チャンネルを分割します。
- 原子核の直径を測定するには、Select Image |調整 |しきい値...。デフォルトのドロップダウンメニューで画像に最も適した方法(Otsuなど)を選択し、核の形状が背景からはっきりと切り取られるまでしきい値を調整します。
注:しきい値設定は、1つの設定がすべての画像またはすべてのサンプルに適合するわけではないため、画像ごとに調整する必要があります。 - フェレットの直径を決定するための測定値を設定するには、[Select Analyze |測定値の設定....メニューから[フェレット直径]を選択し、必要に応じて小数を調整します。
- 画像の分析を開始するには、[ 分析を選択] |パーティクルを解析 して、パーティクルのサイズを設定します。 原子核には、 5-Infinity μm² |結果の表示 |マネージャーに追加 |エッジとオーバーレイで除外。解析を開始します。
- 解析が開始されると、複数のウィンドウが表示されます。ウィンドウの結果で、Feret列内の各原子核の最大フェレット直径を探します。生データをコピーしてスプレッドシートに貼り付け、拡張済みサンプルと拡張済みサンプルの平均核サイズを計算します。
注:各核に対応する関心領域(ROI)が核の周辺全体に追従していることを確認してください。 - 拡張係数 (EF) は、次のように計算します。
EF =
注:EF係数は、サンプルがどの程度拡張されたかを決定します。
結果
以下に示すのは、HeLa細胞(図3A)およびMCF7細胞(図3B)のシアル化糖タンパク質を可視化するためのプロトコールの適用であり、CQ処理(プロトコールセクション2)および免疫蛍光共局在染色(プロトコールステップ5.3)を省略しています。

図3:ExMプロトコール前(左)と後(右)のシアル化標識パターンの比較。 (A)Hela細胞および(B)MCF7細胞を500 μM ManNAzで24時間インキュベートしました。標識は、CuAACを介してAlexa Fluor 488アルキン(緑色で表示)を使用して行いました。核はHoechst 33342(青色で描かれています)で染色しました。スケールバー = 20 μm. 略語: ExM = Expansion Microscopy;ManNAz = N-アジドアセチルマンノサミン;CuAAC = 銅触媒アジド-アルキン環化付加反応。 この図の拡大版を表示するには、ここをクリックしてください。
典型的には、シアリル化標識は、3つの主要な蛍光プールから成る:合成される新生複合糖質を示すゴルジプール、細胞膜に輸出される、または細胞膜からリサイクルされる複合糖質を表す小胞プール(通常は強度が低い)、および発現された複合糖質を示す膜プール13。代謝的に生成されたNeu5Azでタグ付けされた生体高分子複合糖質のみが検出され、未反応の過剰なレポーターやプローブは洗浄ステップ中に除去されることに注意することが重要です。ExM後の画像は共焦点顕微鏡で取得され、特にゴルジ装置ではるかに細かい詳細とより少ない背景を明らかにします。このプロトコールは、シアル化複合糖質を発現するあらゆるタイプの接着細胞に適用することができます。
ExMに固有のシグナルの部分的な損失により、細胞や組織の種類が異なれば、その影響も様々で、蛍光色素の体積希釈によるものです。これは、ラジカル重合中の蛍光色素の保持が不十分であったり、最適でないアンカーや消化による生体分子の保持が不十分であったりすると、シグナルの大幅な損失につながる可能性があります(図4)。この点が、以下の議論の主なトピックです。したがって、必要に応じて、精査中の細胞または組織タイプに合わせて消化ステップのパラメータを調整することをお勧めします。消化は、観察される特定の構造や生体分子に応じて、さまざまな手段で最適化できます。標準化された消化プロトコルから始めて、消化時間、界面活性剤濃度、酵素濃度などのパラメーターを経験的に調整して、望ましい結果を得ることをお勧めします。より効率的な消化は、歪みを回避してより良い等方性膨張につながることに注意することが重要ですが、これによりシグナル損失のリスクも高まる可能性があります。したがって、高品質の等方性拡張の達成とシグナルインテグリティの維持との間でバランスを見つける必要があります。シグナル保持は、特別に設計された分子アンカーを使用することでさらに強化できます。タンパク質観察に焦点を当てた研究では、高温でドデシル硫酸ナトリウム(SDS)のみを使用するプロトコルは、タンパク質シグナル26,27をよりよく保存しながら、埋め込まれたサンプルを効果的に消化することが示されています。
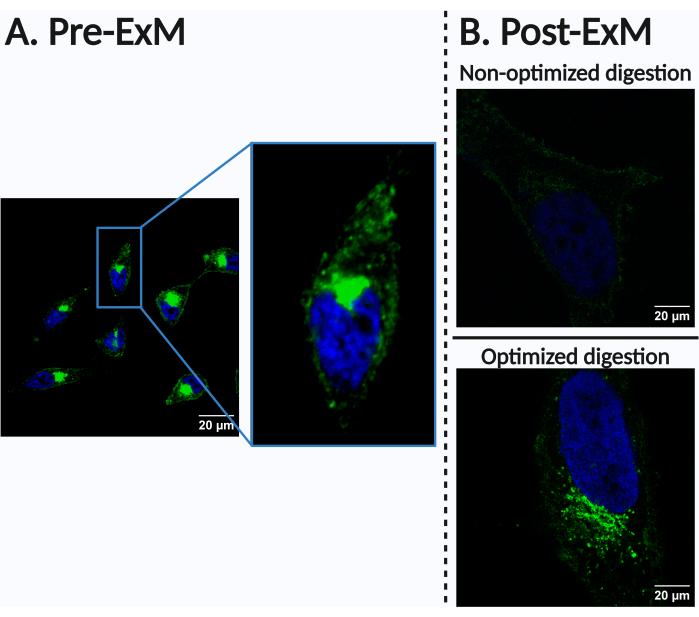
図4:最適でない結果の例。 この図は、細胞、組織、およびモニター対象の生体分子の各タイプ、特に消化ステップに対して条件を調整することの重要性を示しています。HeLa細胞を500 μM ManNAzで24時間インキュベートし、Alexa Fluor 488アルキン(緑色、シアル化 N-糖タンパク質)およびHoechst 33342(青色、核)で標識しました。(A)ラベリングパターンのプレエクスパンション。(B)拡張後のラベリングパターン。 (トップ) 組成、濃度、および/または長さの点で消化パラメータの最適化が不十分な場合、ハイドロゲル内の移植された生体分子の保持が不足しているため、シグナルが大幅に失われ、十分な感度でのバイオイメージングデータの取得が妨げられる可能性があります。この特定のケースでは、消化バッファーはプロトコルステップ7.3で使用されたものと類似していました。しかし、その後の画像で使用された180分の消化時間と比較して、消化時間は90分でした。 (ボトム) 消化パラメータの最適化後に得られた画像(プロトコールステップ7.3を参照)。スケールバー = 20 μm. 略語: ManNAz = N-アジドアセチルマンノサミン。 この図の拡大版を表示するには、ここをクリックしてください。
実験の再現性を確保するためには、サンプルを溶液中に保存し、ハイドロゲルにサンプルを埋め込んだ後でも光から保護することが重要です。ハイドロゲルは、乾燥を防ぐために湿度の高いチャンバーに保持する必要があります。ゲル化(プロトコールステップ7.2)、消化(プロトコールステップ7.3)、および増殖(プロトコールステップ7.4)の3つの主要なステップを同じ日に実行することを強くお勧めします。一度拡張すると、脱イオン水で4°Cに保たれ、光から保護されれば、サンプルは数日間安定しており、バイオイメージングデータの取得に時間をかけることができます。
顕微鏡画像の解析では、膨張前(preExM)と膨張後(postExM)の核フェレット径の平均比を測定することにより、膨張係数(EF)を評価することをお勧めします。ここでは、このプロトコルでは、核をHoechst 33342で染色しますが、従来のDAPIも拡大顕微鏡に適合します。フェレット直径は、原子核の境界に位置する2点間の最大距離に対応します。通常、成功した実験では、4〜5のEF値が得られ(図5)、利用される波長と目的に応じて、最大横方向の分解能が60〜70nmになります。核の平均フェレット直径は、統計的アプローチのためにいくつかの画像にわたって少なくとも60〜100個の細胞を測定することによって決定されます。精度を高めるために、3D再構成を可能にするzスタックを体系的に取得および処理して、測定された距離がすべての原子核の実際のフェレット直径に対応するようにすることができます。しかし、このアプローチは、使用する顕微鏡や対物レンズによる取得の点でも、特にデータ処理の点でも、かなり時間がかかる場合があります。これは、膨張前後のハイドロゲルのサイズを比較することにより、EFのマクロスケール測定によってクロスチェックすることができ、これは私たちの経験では、同様の結果につながります。

図5:拡張係数の決定。 膨張前後の原子核の平均サイズは、ImageJソフトウェアでフェレット直径(つまり、原子核の端にある2点間の最長距離)を測定することによって決定されます。ExM後とExM以前の平均フェレ直径の比率を計算すると、線形膨張係数が得られます。ヒト初代線維芽細胞533Tを拡大プロトコルに提出した。核はHoechst 33342(青色で描かれています)で染色しました。スケールバー = 20 μm. 略語: ExM = Expansion Microscopy. この図の拡大版を表示するには、ここをクリックしてください。
このプロトコルは、特定の細胞治療やさまざまな生理学的および病理学的条件の比較を必要とする生物学的な問題に対処するように設計されています。これは、抗マラリア薬クロロキン(CQ)で治療されたヒト線維芽細胞への適用で例示されています。CQは、pKa値が8.4および10.8の二プロティック弱塩基であり、pHトラップを介してリソソームに優先的に蓄積し、その結果、リソソームpHが上昇し、その後の機能が失われます。
この実験では、ManNAzをシアル化糖鎖に組み入れ、CuAACを介してAlexa Fluor 488で標識し、ゴルジ体をマウス抗GM130抗体で染色します。特に、コントロール細胞(図6A)とCQ処理細胞(図6B)との間には、相対強度に明確な違いが観察されます。制御細胞の生理学的条件下では、シアロコンジュゲート標識の一次プールはゴルジ装置内に局在しています。対照的に、CQ処理された細胞はリソソーム中にシアル酸の蓄積を示し、リサイクルされた複合糖質の捕捉を証明しています。これは、グリコシル化またはリソソーム蓄積症の先天性疾患を研究する文脈で特に関連性があります13,14,15。

図6:ポストExMのシアル化糖タンパク質の局在に対するクロロキンの影響のPostExMの視覚化。 ヒト初代線維芽細胞533Tは、(A)CQで処理しないか、または(B)100μM CQで8時間処理した後、500μM ManNAzで24時間インキュベートし、Alexa Fluor 488アルキン(緑チャンネル)でCuAAC標識しました。ゴルジ装置(赤チャネル)の共局在染色を、マウス抗GM130一次抗体および抗マウスAlexa Fluor 546標識二次抗体を用いて行った。細胞は、記載されているように、アンカリング、ゲル化、消化、および増殖に供した。スケールバー = 20 μm. 略語: ExM = Expansion Microscopy;CQ = クロロキン;ManNAz = N-アジドアセチルマンノサミン;CuAAC = 銅触媒アジド-アルキン環化付加反応。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
現在のCuAAC標識プロトコルは、反応緩衝液にアミノグアニジンを含んでいません。細胞内複合糖質の可視化を目的としているため、代謝取り込みステップ後に固定された細胞に対して行われ、細胞毒性の問題を回避し、触媒系の取り込みを改善します。アミノグアニジンの使用は、通常、生細胞の細胞表面標識に推奨され、デヒドロアスコルビン酸とタンパク質のアルギニン、ヒスチジン、およびリジン残基との間の副反応を防ぐためである28。これらの修飾は、実際にタンパク質の機能に影響を及ぼし、摂動や細胞毒性を引き起こす可能性があります。CuAAC反応バッファーのもう一つの重要な考慮事項は、BTTAAによる銅イオンの錯体化は迅速ですが、即時ではないということです。修飾糖の効率的な標識を確実にするために、標識プロセスを開始する少なくとも30分前に溶液を調製し、初心者ユーザーによる結果の再現性を確保することをお勧めします。
取得には、任意の共焦点顕微鏡を使用できます。サンプルが拡大すると、観測可能なフィールドも拡大します。セル全体をキャプチャするには、画像間のオーバーラップが 10% の領域のタイル スキャンを実行することをお勧めします。タイルスキャンとZスタックを同時に実行することは可能ですが、その場合は小さなタイルスキャンを選択することをお勧めします。これは、このプロセスに時間がかかり、大量の生データが生成されるため、集中的なデータ処理につながるためです。両方の方法を分離し、関心のある領域のみの z スタックを作成し、展開前の z スタックと一致するパラメーターを使用することをお勧めします。拡張ゲルには退色防止用封入剤を使用しないため、光退色を最小限に抑えるために、取得パラメータを適切に設定することをお勧めします(スキャン速度が速く、レーザー強度が小さい)。
拡張実験が成功すると、通常、膨張係数は4〜5になり、これは64倍から125倍の体積増加に相当します。膨張後、同じ数の蛍光色素分子がはるかに大きな体積に広がるため、ExMの主な欠点は、シグナル強度の低下が避けられないことです。膨張後の蛍光シグナルを観察できるようにするために、蛍光色素分子に応じて励起レーザーのゲインや強度を大幅に増やすことができます。ピンホールの開口部も変調されている可能性があります。蛍光シグナルに対するこの純粋に物理的な希釈効果に加えて、シグナル損失の他の考えられる原因があります。
分子アンカーの標識、固定、および生体結合に続いて、ゲル化(プロトコルステップ7.2)は、サンプルを高分子電解質ヒドロゲルにカプセル化することを目的としています。このようなポリマーは、アクリルアミド、ビスアクリルアミド、アクリル酸ナトリウムの3つのモノマーで構成されています。後者は、そのイオン性のためにゲルにその膨張特性を与える。実際、このポリマーは、ナトリウムイオンを水分子29と交換することにより、その重量の数倍を水に吸収することができる。ゲルの架橋は、TMEDAおよびAPSによって触媒されるラジカル反応です。注目すべきは、さらなるシグナル減少の原因として、このラジカル誘起重合中の有機蛍光色素の部分的な分解であることです。さまざまな染料がさまざまな程度で影響を受けます。例えば、シアニン色素は伸長中にほぼ完全に破壊され、0.3-0.4 %30,31のシグナル保持不良を示しますが、Alexa Fluor 488色素はExM4後のシグナル強度の~57%の保持を可能にします。さまざまな有機蛍光色素および蛍光タンパク質のシグナル保持の要約については、Wen et al.31によるレビューを参照してください。
さらに、生体分子の保持は、プロトコールの特定のステップによっても影響を受ける可能性があります。例えば、ハイドロゲルに固定されていないタンパク質断片上に位置する糖鎖は、消化中に洗い流され、シグナル強度がさらに低下する可能性があります(図4)。消化は、プロテイナーゼK 4,5,6,31の有無にかかわらず、トリトンX−100のような少なくとも1つの界面活性剤32を使用して、細胞骨格のような膜およびタンパク質構造を破壊する。消化時間と消化バッファーの組成は、特定の蛍光色素や生体分子を最適に保存するように調整できます。したがって、分解パラメータ(プロトコールステップ7.3)は、組成、濃度、および持続時間の点で研究中のサンプルのタイプに合わせて最適化し、生体分子とプローブの等方性拡張と保持を確保するための均質化の最適なバランスを得る必要があります。場合によっては、サンプルのタンパク質がゲル膨張26,27の前の95°CでSDSで変性されるUltrastructure-ExMのように、消化バッファーのタイプをより穏やかな条件に完全に変更することができます。
可能であれば、特定のプローブを必要とせずに、イメージングデータの取得中に励起ソースの電力および/または検出器カメラのゲインを調整することで、ExM後の信号の低下を簡単に対処できます。ExM後にS/N比が不十分な場合、例えば、フルオロフォア自体をポリマーマトリックス6,14に直接固定することを可能にする三官能性分子を利用するなど、さまざまな研究室によって追加のシグナル保持および/または増幅戦略が開発されている。これらの課題に対処するための代替ソリューションとして、ゲルが十分に透過性であるため、サンプルの染色は拡張後に行うことができます。ただし、これにより、膨張前のサンプルの観察が妨げられます。このアプローチは、最近、免疫染色33で報告されており、検体を最初に等方的に拡大し、次に抗体で染色します。その結果、蛍光の輝度とエピトープのアクセシビリティが向上すると同時に、ExMと超解像技術を組み合わせる際の局在精度を阻害する連鎖誤差が減少する可能性があることを実証しました。
蛍光強度の低下を阻止するための代替戦略が検討されています。一例として、従来の蛍光分子の代わりにポリマーベースの蛍光色素を使用することが挙げられます。Liuらは、従来の蛍光色素よりもはるかに明るい蛍光ポリマードット(Pdots)34 を開発し、より優れたS/N比を可能にしました。Pdotは、アンカーと抗体で官能基化することができます。別の例では、Click Expansion Microscopy(ClickExM)6 は、アンカーを持つ蛍光ストレプトアビジンを使用して、間接的なビオチン-ストレプトアビジン戦略によりシグナル保持を強化するユニバーサル戦略を導入しました。これにより、蛍光プローブをポリマーフレームに直接グラフトすることができます。同様に、Molecule Anchorable Gel-enabled Nanoscale In-situ Fluorescence microscopY (MAGNIFY)35 は、蛍光シグナルの保持を高めるために生体分子のアンカリングに大きく依存しています。特定の分子をゲルに結合させるClickExMとは対照的に、MAGNIFYはメタクロレインを使用して、さまざまな生体分子をハイドロゲルのフレームに架橋します。
顕微鏡でのデータ収集に関してExMが許容するもう1つの制限は、拡大したサンプルをより広い物理領域でイメージングする必要があるため、サンプル全体の取得に必要な時間が大幅に増加することです。膨張プロセスによりサンプルが拡大するため、高解像度のイメージングを実現するには、同じ生物学的領域をカバーするためにより多くの画像タイルをキャプチャする必要があり、取得時間が長くなります。この点で、多くのアプリケーションにおいて、ExMは従来の共焦点顕微鏡法の代替物とは見なされず、包括的で詳細なイメージングのための補完的な技術と見なされます。

図7:ヒト初代線維芽細胞533T細胞のpreExMとpostExMの3Dイメージングの比較。細胞を500 μM ManNAzで24時間インキュベートし、CuAAC(緑色)によるAlexa Fluor 488アルキンで標識しました。マウス抗GM130一次抗体および抗マウスAlexa Fluor 546標識二次抗体を用いてゴルジ装置(赤色)の共局在染色を行い、Hoechst 33342(青色)で核を染色した。(左)(A)Zスタックと(B,C)preExMで可視化された細胞の3D表面レンダリング。(右)細胞は、zスタック取得に先立って、(D)に記載されているように、アンカリング、ゲル化、消化、および増殖に供した。(E)信号の3D表面レンダリングは、複雑な細胞内下部構造を検出する際に特に役立つ分解能の向上を明確に示しています。3D再構成とサーフェスレンダリングは、信号強度を使用して作成されました。スケールバー = 20 μm. この図の拡大版を表示するには、ここをクリックしてください。
ExMは、回折限界を実際に超えることなくナノスケールイメージングを達成するための革新的な技術として登場し、標準プロトコル6では見落とされる可能性のある生体サンプルの詳細を可視化することを可能にしました。回折が ~250 nm に制限された装置を使用する場合 (緑色レーザーを装備した日常的な共焦点顕微鏡の場合と同様)、4.5 倍の膨張係数により、通常、横方向の分解能は 60 nm 近くになります。これは、ゴルジ装置や小胞などの細胞内構造を研究する際に非常に大きな改善であり、2Dイメージングだけでなく、zスタック取得から3D画像を生成することもできます(図7)。拡張顕微鏡の能力をフルに活用するために、ExMをSTEDやSTORMなどの超解像顕微鏡技術と組み合わせることで、達成される分解能をさらに向上させることができますが、これはこの記事の範囲外です。
拡大顕微鏡の分野は、非常に急速な成長と大幅な進歩を目の当たりにしており、エキサイティングでダイナミックな研究分野となっています。Boyden氏らは2015年にExpansion Microscopyを導入し、ゲルアンカー可能なラベルを使用してポリマーマトリックス中の生体分子の形状を保持することにより、共焦点顕微鏡で約70 nmの分解能を達成しました。当初の関心に続いて、カスタムメイドのオリゴヌクレオチド-抗体コンジュゲートの必要性が、科学界をこの方法の新しいツールとアプローチの開発に駆り立てました。市販の薬剤を使用してタンパク質をゲルに直接アンカリングするためのさまざまな戦略が開発され、蛍光標識された抗体またはタンパク質の使用が可能になり、最近の開発により、拡張顕微鏡法がさらに簡素化され、個別のアンカリングステップを必要とせずにさまざまな生体分子への適用が拡大されました。
ExMにより高分解能でMOE標識を達成することは、高価でアクセスが困難な超解像顕微鏡を必要とせずに、シアル化糖鎖の空間分布を日常的に研究するのに特に有用です。例えば、この方法では、原形質膜上のシアログライカンを可視化し、微絨毛やその他の細胞構造におけるその分布を明らかにしています6。細胞内糖鎖の生合成、輸送、リサイクル8,13,14,15を研究し、代謝性疾患やがんなどの病態における糖鎖の役割を解読するためには、細胞内糖鎖を分解能を向上させて可視化する能力がさらに重要になります。ExMと生体直交化学を組み合わせることで、細胞の経路、オルガネラ機能、細胞調節や疾患メカニズムの鍵となる相互作用への局在と関与をより深く理解するユニークな機会が得られます。これにより、将来、さまざまな病状でシアル酸が果たす役割についての理解が確実に向上します。
開示事項
著者には、競合する金銭的利益やその他の利益相反はありません。
謝辞
この作業の達成に役立つ技術環境を提供してくれたTisBioの施設とPLBSプラットフォームに感謝します。この研究は、CNRSとMinistère de l'Enseignement Supérieur et de la Rechercheからの助成金によって支援されました。François Foulquier博士、Zoé Durin博士、Dorothée Vicogne夫人、Céline Schulz夫人には、議論を刺激し、Fibroblast 533T細胞株と一次抗体GM130を提供してくださったことに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| (+) Sodium L-ascorbate | Sigma Aldrich | 11140 | |
| 12 well cell culture plate | Corning | 3513 | |
| Acrylamide | Sigma Aldrich | A8887 | |
| Acrylic acid N-hydroxysuccinimide ester | Sigma Aldrich | A8060 | |
| Alexa Fluor 488 alkyne | Jena Bioscience | CLK-1277-5 | |
| Alexa Fluor 546 goat anti-mouse IgG | Invitrogen | A11003 | |
| Amonium persulfate | Sigma Aldrich | 9913 | |
| Bis-Acrylamide | Sigma Aldrich | 146072 | |
| BSA | Sigma Aldrich | A7906 | |
| BTTAA | Jena Bioscience | CLK-067-100 | |
| Centrifugation tube 2 mL | EPPENDORF | 30120094 | |
| Chloroquine diphosphate salt | Sigma Aldrich | C6628 | |
| Conical tube 15 mL | Falcon | 352097 | |
| cover slips 12 mm #1 | epredia | CB00120RA120MNZ0 | |
| cover slips 32 mm #1 | epredia | CB00320RA140MNZ0 | |
| CuSO4 | Sigma Aldrich | 209198 | |
| DMEM high glucose medium | Dutscher | L0104-500 | |
| Dulbecco's Phosphate Buffered Saline (PBS) | Dutscher | L0615-500 | |
| Fetal Bovine Serum | biowest | S1810-500 | |
| Fibroblast 533T | - | - | Collected from healthy individual |
| FIJI ImageJ 2.9.0 | - | - | |
| Gelatin | Bio-RAD | 170-6537 | |
| Guanidine HCl | Sigma Aldrich | 50950 | |
| HeLa cells | ATCC | CCL-2 | |
| Hoechst 33342 | Sigma Aldrich | 14533 | |
| Imaris 10.2 | - | - | |
| K2HPO4 | Euromedex | PB0447-B | Anhydrous |
| LSM 780 Confocal Microscopy | Zeiss | - | |
| MCF7 | ATCC | HTB-22 | |
| N-acetylmannosamine (ManNAc) | BIOSYNTH | MA05269 | |
| NaCl | Carlo Erba | 479687 | |
| N-azidoacetylmannosamine (ManNAz) | BIOSYNTH | MA46002 | |
| Objectif "Plan-Apochromat" 63x/1,4 Oil DIC M27 | Zeiss | 420782-9900-799 | |
| Phosphate Buffered Saline (PBS) 10x | Euromedex | ET330 | |
| Proteinase K | Sigma Aldrich | P2308 | from Tritirachium album |
| purified mouse GM130 antibody | BD Bioscience | 610822 | 50 µg |
| Sodium acrylate | Sigma Aldrich | 408220 | |
| T75 Flask | Corning | 430641 | |
| TEMED | Sigma Aldrich | T9281 | |
| tris Acetate EDTA (TAE) 10x | Euromedex | EU0202-B | |
| Triton X-100 | Sigma Aldrich | X-100 | |
| Trypan Blue | Dutscher | 702630 | |
| Trypsine-EDTA 1x | Dutscher | L0930-100 |
参考文献
- De Souza, N. Light microscopy at the limit. Nat Cell Biol. 11 (Suppl 1), S22(2009).
- Tam, J., Merino, D. Stochastic optical reconstruction microscopy (STORM) in comparison with stimulated emission depletion (STED) and other imaging methods. J Neurochem. 135, 643-658 (2015).
- Jeong, S., Widengren, J., Lee, J. C. Fluorescent probes for STED optical nanoscopy. Nanomaterials. 12 (1), 21(2021).
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543548(2015).
- Kang, S., et al. Expansion microscopy with a thermally adjustable expansion factor using thermoresponsive biospecimen-hydrogel hybrids. ACS Appl Mater Interfaces. 13 (24), 2896228974(2021).
- Sun, D., et al. Click-ExM enables expansion microscopy for all biomolecules. Nat Methods. 18 (1), 107113(2020).
- Dube, D., Bertozzi, C. Metabolic oligosaccharide engineering as a tool for glycobiology. Curr Opin Chem Biol. 7 (5), 616625(2003).
- Scache, J., et al. Switching azide and alkyne tags on bioorthogonal reporters in metabolic labeling of sialylated glycoconjugates: a comparative study. Sci Rep. 12 (1), 22129(2022).
- Holst, S., Wuhrer, M., Rombouts, Y. Glycosylation characteristics of colorectal cancer. Adv Cancer Res. 126, 203256(2015).
- Boyaval, F., et al. N-Glycomic signature of stage II colorectal cancer and its association with the tumor microenvironment. Mol Cell Proteomics. 20, 100057(2021).
- Ferreira, J. A., et al. Protein glycosylation in gastric and colorectal cancers: Toward cancer detection and targeted therapeutics. Cancer Lett. 387, 32-45 (2017).
- Jennings, M. P., Day, C. J., Atack, J. M. How bacteria utilize sialic acid during interactions with the host: snip, snatch, dispatch, match and attach. Microbiology. 168 (3), 001157(2022).
- Gilormini, P. A., et al. A sequential bioorthogonal dual strategy: ManNAl and SiaNAl as distinct tools to unravel sialic acid metabolic pathways. Chem Commun. 52 (11), 2318-2321 (2016).
- Vanbeselaere, J., et al. Alkynyl monosaccharide analogues as a tool for evaluating Golgi glycosylation efficiency: application to Congenital Disorders of Glycosylation (CDG). Chem Commun. 49 (96), 11293-11295 (2013).
- Gilormini, P. A., et al. Chemical glycomics enrichment: imaging the recycling of sialic acid in living cells. J Inherit Metab Dis. 4 (3), 515523(2018).
- Rigolot, V., Biot, C., Lion, C. To view your biomolecule, click inside the cell. Angew Chem. 60 (43), 2308423105(2021).
- Qin, W., et al. Artificial cysteine S-glycosylation induced by per-O-acetylated unnatural monosaccharides during metabolic Glycan Labeling. Angew Chem. 130 (7), 1835-1838 (2018).
- Qin, K., Zhang, H., Zhao, Z., Chen, X. Protein S-glyco-modification through an elimination-addition mechanism. J Am Chem Soc. 142 (20), 9382-9388 (2020).
- Rostovtsev, V. V., Green, L. G., Fokin, V. V., Sharpless, K. B. A. Stepwise Huisgen cycloaddition process: Copper(I)-catalyzed regioselective "ligation" of azides and terminal alkynes. Angew Chem. 41 (14), 25962599(2002).
- Agard, N. J., Prescher, J. A., Bertozzi, C. R. A strain-promoted [3 + 2] azide−alkyne cycloaddition for covalent modification of biomolecules in living systems. J Am Chem Soc. 126 (46), 15046-15047 (2004).
- Mbua, N. E., et al. Strain-promoted alkyne-azide cycloadditions (SPAAC) reveal new features of glycoconjugate biosynthesis. ChemBioChem. 12 (12), 19121921(2011).
- Blackman, M. L., Royzen, M., Fox, J. M. Tetrazine ligation: Fast bioconjugation based on inverse-electron-demand Diels-Alder reactivity. J Am Chem Soc. 130 (41), 1351813519(2008).
- Devaraj, N. K., Weissleder, R., Hilderbrand, S. A. Tetrazine-based cycloadditions: Application to pretargeted live cell imaging. Bioconjug Chem. 19 (12), 22972299(2008).
- Bird, R. E., et al. Bioorthogonal chemistry and its applications. Bioconjug Chem. 32 (12), 2457-2479 (2021).
- Slade, P. G., et al. Identifying the CHO secretome using mucin-type O-linked glycosylation and click-chemistry. J Proteome Res. 11 (12), 61756186(2012).
- Gambarotto, D., Hamel, V., Guichard, P. Ultrastructure expansion microscopy (U-ExM). Methods Cell Biol. 161, 5781(2021).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nat. Methods. 16, 71-74 (2019).
- Presolski, S. I., Hong, V. P., Finn, M. Copper-catalyzed azide-alkyne click chemistry for bioconjugation. Curr Protoc Chem Biol. 3 (4), 153-162 (2011).
- Santiago, F., et al. Synthesis and swelling behaviour of poly(sodium acrylate)/sepiolite superabsorbent composites and nanocomposites. Polym Int. 55 (8), 843848(2006).
- Wen, G., et al. Evaluation of direct grafting strategies via trivalent anchoring for enabling lipid membrane and cytoskeleton staining in expansion microscopy. ACS Nano. 14 (7), 78607867(2020).
- Wen, G., et al. Current progress in expansion microscopy: Chemical strategies and applications. Chem Rev. 123 (6), 32993323(2023).
- Drelich, L., et al. Toward high spatially resolved proteomics using expansion microscopy. Anal Chem. 93 (36), 12195-12203 (2021).
- Zwettler, F. U., et al. Molecular resolution imaging by post-labeling expansion single-molecule localization microscopy (Ex-SMLM). Nat Commun. 11 (1), 3388(2020).
- Liu, J., et al. Expansion microscopy with multifunctional polymer dots. Adv Mater. 33 (25), 2007854(2021).
- Klimas, A., et al. Magnify is a universal molecular anchoring strategy for expansion microscopy. Nat Biotechnol. 41 (6), 858869(2023).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved