Method Article
Visualisierung der intrazellulären Sialylierung mit Click-Chemie und Expansionsmikroskopie
In diesem Artikel
Zusammenfassung
Hier schlagen wir ein einfaches Protokoll vor, das metabolisches Oligosaccharid-Engineering, Click-Chemie und Expansionsmikroskopie kombiniert und die Biobildgebung von intrazellulären sialylierten N-Glykoproteinen mit verbesserter Auflösung unter Verwendung von Routinemikroskopiegeräten ermöglicht.
Zusammenfassung
Metabolische Markierungstechniken ermöglichen den Einbau von bioorthogonalen Reportern in Glykane und ermöglichen so die gezielte Biokonjugation von molekularen Farbstoffen innerhalb von Zellen durch Click- und bioorthogonale Chemie. Das metabolische Oligosaccharid-Engineering (MOE) hat aufgrund der wesentlichen Rolle der Glykosylierung in zahlreichen biologischen Prozessen, die die molekulare Erkennung betreffen, und ihrer Auswirkungen auf Pathologien, die von Krebs über genetische Störungen bis hin zu viralen und bakteriellen Infektionen reichen, großes Interesse geweckt.
Obwohl MOE besser für den Nachweis von Glykokonjugaten auf der Zelloberfläche bekannt ist, ist es auch eine sehr wichtige Methode für die Untersuchung intrazellulärer Glykane in physiologischen und pathologischen Kontexten. Solche Studien profitieren stark von der hohen räumlichen Auflösung. Die hochauflösende Mikroskopie ist jedoch in den meisten Laboren nicht ohne weiteres verfügbar und stellt die tägliche Umsetzung vor Herausforderungen. Die Expansionsmikroskopie ist eine neue Alternative, die die Auflösung der Mikroskopie erhöht, indem biologische Proben, die mit Fluoreszenzmarkern markiert sind, physikalisch vergrößert werden. Durch die Einbettung der Probe in ein quellfähiges Gel und die isotrope Ausdehnung durch chemische Behandlung können subzelluläre Strukturen mit verbesserter Präzision und Auflösung visualisiert werden, ohne dass Superauflösungstechniken erforderlich sind.
In dieser Arbeit veranschaulichen wir die Fähigkeit der Expansionsmikroskopie, intrazelluläre sialylierte Glykane durch den kombinierten Einsatz von MOE und Click-Chemie sichtbar zu machen. Konkret schlagen wir ein Verfahren für die bioorthogonale Markierung und Expansionsmikroskopie vor, das einen Reporter verwendet, der auf die Sialylierung abzielt und für Co-Lokalisationsstudien mit Immunfluoreszenz assoziiert sein kann. Dieses Protokoll ermöglicht Lokalisierungsstudien der Sialokonjugat-Biosynthese, des intrazellulären Transports und des Recyclings.
Einleitung
Die Fluoreszenzmikroskopie wird zwar häufig zur Markierung und Visualisierung spezifischer Moleküle in Zellen verwendet, ist aber von Natur aus in ihrer Auflösung durch Abbes Beugungsgrenze des Lichts1 begrenzt, die die Fähigkeit einschränkt, zwischen Objekten zu unterscheiden, die näher als etwa 200-250 nm sind. Diese Einschränkung ergibt sich aus der Wellennatur des Lichts und der numerischen Apertur der Objektivlinse des Mikroskops, was eine Herausforderung bei der Abbildung subzellulärer Strukturen darstellt. Die Überwindung dieser Einschränkungen ermöglicht bessere Einblicke in bestimmte biologische Prozesse auf nanometrischer Skala.
Um die Beugungsgrenze des Lichts zu überwinden, wurden hochauflösende Mikroskopietechniken wie STORM (Stochastic Optical Reconstruction Microscopy) und STED (Stimulated Emission Depletion) entwickelt 2,3. STORM beruht auf der stochastischen Aktivierung von Fluorophoren, so dass zu jedem Zeitpunkt nur eine dünn besetzte Teilmenge abgebildet werden kann. Dies ermöglicht die präzise Lokalisierung einzelner Fluorophore, was die Rekonstruktion eines hochauflösenden Bildes ermöglicht. STED hingegen verbessert die Auflösung, indem ein Verarmungslaser verwendet wird, um die Fluoreszenz um die Peripherie des Anregungsflecks selektiv zu löschen, wodurch die Punktspreizungsfunktion effektiv verengt wird.
Diese Ansätze stehen im Gegensatz zur Weitfeld- oder Konfokalmikroskopie, bei der alle Fluorophore gleichzeitig detektiert werden, was zu einem Bild führt, das alle Beugungsmuster kombiniert und die Unterscheidung zwischen einzelnen Fluorophoren in der Nähe verhindert, was zu einem Auflösungsverlust führt. Diese superauflösenden Methoden erfordern jedoch sehr spezifische Lichtquellen, Geräte, Probenvorbereitung und/oder Fluorophore, was diese Technologien kostspielig, in den meisten Labors schwer zugänglich und in Routineexperimenten schwierig zu implementieren macht. Diese Einschränkungen haben die wissenschaftliche Gemeinschaft dazu veranlasst, nach alternativen Lösungen zu suchen, um eine höhere Auflösung zu erreichen, die mit leicht verfügbaren Mikroskopiegeräten und routinemäßigen Färbeprotokollen kompatibel sind. Im Jahr 2015 entwickelten Boyden und seine Kollegen eine Methode, um die Einschränkungen der optischen Mikroskopie zu umgehen, indem die Probe physikalisch erweitert wird, die als Expansion Microscopy (ExM)4 bezeichnet wird.
ExM ist eine dreistufige Methode, die nanoskalige Details biologischer Proben liefert, ohne die Beugungsgrenze des Lichts zu überschreiten (Abbildung 1). Stattdessen werden herkömmliche beugungsbegrenzte Mikroskope verwendet, um Proben abzubilden, die physikalisch isotrop vergrößert wurden. Der erste Schritt, Gelierung genannt, besteht darin, die biologische Probe, typischerweise fixierte Zellen oder Gewebe, in ein quellfähiges Polyelektrolyt-Hydrogel auf Basis von Natriumacrylat und Acrylamid einzubetten. Die biologische Probe wird dann enzymatisch behandelt, um bestimmte Bestandteile wie Proteine oder Membranen teilweise abzubauen und dichte Zellstrukturen aufzubrechen, um sicherzustellen, dass sich die Zellen gleichmäßig ausdehnen können. Dieser Schritt, der als Aufschluss bezeichnet wird, trägt dazu bei, indem die Strukturkomponenten der Probe in Bezug auf die mechanischen Eigenschaften homogenisiert werden, wodurch eine unterschiedliche Ausdehnung verhindert wird, die zu einer Verformung der Probe führen könnte. Im letzten Schritt, der sogenannten Expansion, wird die in das Hydrogel eingebettete und verdaute Probe in deionisiertes Wasser gelegt, wodurch sie aufquillt. Diese Technologie führt dazu, dass sich die Probe in jeder Dimension um einen linearen Vergrößerungsfaktor von etwa 4-5x ausdehnt, was die Visualisierung feiner zellulärer Details mit Standard-Mikroskopietechniken ermöglicht. Aufgrund der isotropen Natur der Expansion behält die innerhalb der Gelmatrix vergrößerte biologische Probe ihre dreidimensionalen geometrischen Details und die räumlichen Beziehungen zwischen ihren verschiedenen Strukturkomponenten bei. Räumliche Informationen bleiben also beim Aufquellen erhalten, während der Abstand zwischen den gelverankerten Fluoreszenzmarkierungen oder Biomolekülen gleichmäßig in alle Richtungen zunimmt. Dies ermöglicht eine bessere Trennung der Signale, was zu einer höheren Auflösung führt.

Abbildung 1: Überblick über das ExM-Protokoll. (a) Gelierung: Die biologische Probe wird in einem quellfähigen Polyelektrolyt-Hydrogel verankert und eingebettet. (b) Aufschluss: Die mechanischen Eigenschaften der Probe werden durch den Einsatz von Enzymen und Detergenzien homogenisiert, wodurch Proteine und Lipidmembranen abgebaut werden, die sonst die Ausdehnung einschränken und Verzerrungen verursachen würden. (c) Expansion: Das Hydrogel wird in deionisiertes Wasser getaucht, wodurch es sich isotrop ausdehnt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Seit 2015 hat ExM erhebliche technologische Verbesserungen bei der Bewahrung von räumlichen Informationen erfahren. Insbesondere wurden verschiedene molekulare Anker entwickelt, um Biomoleküle kovalent direkt an das quellfähige Hydrogel 5,6 zu binden. In diesem Protokoll haben wir N-Acryloxysuccinimid (NAS) verwendet, das mit den freien Amingruppen von Biomolekülen (typischerweise Lysinreste oder N-terminale Positionen von Proteinen) reagiert, um eine Acryloylgruppe an die Proteine der Zelle, einschließlich Glykoproteine, zu biokonjugieren. Diese Acryloylgruppe reagiert dann während der Gelierung durch Vernetzungsreaktionen mit anderen Monomeren und verankert diese Biomoleküle direkt am Polymer.
ExM ist mit einer Vielzahl von Markierungsmethoden kompatibel, einschließlich des Metabolic Oligosaccharide Engineering (MOE). MOE ist ein leistungsfähiges Werkzeug, das die Markierung von Glykanen ermöglicht, indem Analoga von Stoffwechselvorläufern einbezogen werden, die mit einem bioorthogonalen chemischen Griffausgestattet sind 7. Diese Analoga, die als chemische Reporter bekannt sind, integrieren sich in Stoffwechselwege, ohne Toxizität zu verursachen. Beim MOE-Ansatz werden Reportermonosaccharide metabolisch zu aktivierten Nukleotidzuckern verarbeitet und dann auf naszierende Glykokonjugate übertragen. Unser Hauptaugenmerk liegt auf der Erforschung der Sialylierung, insbesondere der intrazellulären Dynamik und des Transports von sialylierten N-Glykoproteinen, die aufgrund ihrer Rolle bei Zell-Zell-Interaktionen, Immunregulation und Entwicklung im Zusammenhang mit Gesundheit und Krankheit von entscheidender Bedeutung ist. Die abnormale Sialylierung wird mit Krankheiten wie Krebs 8,9,10,11, Infektionskrankheiten 12 und genetischen Störungen 13,14,15 in Verbindung gebracht, was sie zu einem wichtigen Ziel für die therapeutische Entwicklung und die Entdeckung von Biomarkern macht. Das Verständnis der Sialylierung verbessert die Einblicke in die Glykobiologie und Krankheitsmechanismen.
Die Sialylierung kann unter Verwendung von MOE mit Analoga der N-Acetylneuraminsäure (Neu5Ac), der am häufigsten vorkommenden Sialinsäure beim Menschen, oder mit Analoga von N-Acetylmannosamin (ManNAc), einem metabolischen Vorläufer von Neu5Ac, untersucht werden, der einen bioorthogonalen Griff trägt 8,16. ManNAc wird im Zytosol in Neu5Ac umgewandelt und dann im Zellkern zu Cytidin-5′-monophospho-N-neuraminsäure (CMP-Neu5Ac) aktiviert. Nach der Aktivierung zu einem Nukleotidzucker übertragen Sialyltransferasen im Golgi-Apparat Neu5Ac-Einheiten an die Endpositionen wachsender Glykanketten (Abbildung 2). Nach dem metabolischen Einbau von unnatürlichen ManNAc-Derivaten können die markierten sialylierten Glykane durch bioorthogonale Click-Chemie kovalent an ein Fluorophor gebunden werden, das eine reaktive Gruppe komplementär zum Reportergriff trägt. Dies ermöglicht die direkte Beobachtung von Glykokonjugaten in vivo oder ex vivo.
Für die meisten Monosaccharid-Reporter ist eine peracetylierte Form erforderlich, um die Plasmamembran durch passive Diffusion zu durchqueren. Es wurde jedoch gezeigt, dass sowohl peracetylierte als auch ungeschützte Reporter die Sialylierung effizient untersuchenkönnen 16. Ungeschützte Sialylierungsreporter sind in der Lage, durch aktive Transportmechanismen in Zellen einzudringen, nämlich Pinozytose für Neu5Ac-Analoga und ein noch nicht identifizierter Transporter für ManNAc. Während ungeschützte Zucker eine höhere Konzentration (typischerweise 100-500 μM) benötigen, um vergleichbare Wirkungen zu erzielen, können sie, sobald sie sich in der Zelle befinden, direkt in den Sialinsäure-Stoffwechselweg gelangen. Im Gegensatz dazu müssen peracetylierte Zucker durch intrazelluläre unspezifische Esterasen vollständig deacetyliert werden, bevor sie metabolisch aktiv werden. Obwohl sie bei niedrigeren Konzentrationen (typischerweise 10-50 μM) verwendet werden können, kann eine unvollständige Deacetylierung die Enzymaktivität beeinträchtigen oder zum Einbau von teilweise acetylierten Sialinsäure-Analoga führen, was die nachgelagerte Analyse möglicherweise verzerrt. Darüber hinaus kann die Freisetzung von Essigsäure den pH-Wert lokal beeinflussen, was sich möglicherweise auf die Zellfunktion auswirkt. Chen und Kollegen haben auch gezeigt, dass per-O-acetylierte Zucker über einen nicht-enzymatischen Mechanismus mit freien Cysteinresten in Proteinen reagieren, was zu einem Off-Target-Einbau und einem erhöhten unspezifischen Signal führt 17,18. In dem vorliegenden Protokoll beschäftigen wir daher ungeschützte ManNAc-Reporter.

Abbildung 2: Metabolisches Oligosaccharid-Engineering und Markierung von Sialinsäuren. UDP-GlcNAc wird durch die UDP-GlcNAc 2-Epimerase-Domäne von GNE/MNK im Zytosol in ManNAc umgewandelt. ManNAc wird dann im Zytosol durch die ManNAc 6-Kinase-Domäne von GNE/MNK zu ManNAc-6-phosphat phosphoryliert. Die N-Acetylneuraminat-Synthase katalysiert die Kondensation von ManNAc-6-P mit Phosphoenolpyruvat zur Herstellung von Neu5Ac-9-phosphat, das anschließend durch Sialinsäurephosphatase dephosphoryliert wird, um Neu5Ac zu erhalten. Neu5Ac kann auch über den Salvage-Weg über Endozytose und lysosomales Recycling zugeführt werden8. Nach dem Transport zum Zellkern wird es durch CMP-Sialinsäure-Synthetase in CMP-Neu5Ac umgewandelt. In der Golgi-Apparatur ist CMP-NeuAc das Substrat von Sialyltransferasen, die an den Endpositionen von Glykanen auf reifenden Glykokonjugaten eine Neu5A-Einheit einführen, die schließlich an der Zellmembran exprimiert oder sezerniert werden. ManNAz-Chemikalienreporter, die einen bioorthogonalen Griff tragen, können durch einen nicht identifizierten aktiven Transporter in die Zelle eindringen und in den Stoffwechselweg gelangen. Die markierten Neu5Az-Einheiten, die nach der Biosynthese in Glykane eingebaut werden, werden dann durch CuAAC-vermittelte Konjugation einer Fluoreszenzsonde markiert. Abkürzungen: NAc = N-Acetyl; UDP-GlcNAc = Uridindiphosphat N-Acetylglucosamin; ManNAc = N-Acetylmannosamin; GNE = UDP-GlcNAc 2-Epimerase; MNK = ManNAc 6-Kinase; ManNAc-6-P = ManNAc-6-phosphat; NANS = N-Acetylneuraminat-Synthase; PEP = Phosphoenolpyruvat; Neu5Ac = N-Acetylneuraminsäure; Neu5Ac-9-P = Neu5Ac-9-phosphat; NANP = Sialinsäure-Phosphatase; CMP = Cytidin-5′-Monophosphat; CMAS = CMP-Sialinsäure-Synthetase; STs = Sialyltransferasen; ManNAz = N-Azidoacetylmannosamine; Neu5Az = N-Azidoacetylneuraminsäure. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Unter den möglichen bioorthogonalen Reaktionen konzentrierten wir uns auf die kupferkatalysierte Azid-Alkin-Cycloaddition (CuAAC)16,19. Nach unseren Erfahrungen ist CuAAC die beste Option für die Markierung intrazellulärer Glykane in fixierten Zellen. Diese Reaktion ist gut etabliert, in der bioorthogonalen Chemie gründlich untersucht und wurde für den Einsatz in komplexen biologischen Umgebungen standardisiert, was eine solide Wissensgrundlage und optimierte Protokolle bietet. Seine schnelle Kinetik bietet in der Tat eine hohe Reaktionseffizienz und Spezifität, und es handelt sich um Azid- und Alkingruppen, die leicht zu synthetisieren, stabil sind, in lebenden Systemen nicht vorkommen und gegenüber nativen Biomolekülen inert sind, was es ideal für Anwendungen mit festen Zellen macht, bei denen Kupfertoxizität kein Problem darstellt. Die stammgeförderte Alkin-Azid-Cycloaddition (SPAAC)20,21 vermeidet zwar Kupfertoxizität, hat aber eine langsamere Kinetik und voluminösere Sonden, was aufgrund des hydrophoben Fallens tendenziell zu einem höheren Hintergrundsignal und einem verringerten Signal-Rausch-Verhältnis für intrazelluläre Anwendungen führt, während der inverse Elektronenbedarf Diels-Alder (IEDDA)22,23, obwohl sie schnell und kupferfrei ist, erfordert eine komplexere Sondensynthese und erfordert sperrigere Reportergruppen, die in MOE-Anwendungen noch nicht vollständig charakterisiert wurden. Da für ExM ausschließlich fixierte Zellen benötigt werden, bietet CuAAC eine robuste und effiziente Lösung für die bioorthogonale Markierung.
In dieser Arbeit wird ein Protokoll für die Visualisierung intrazellulärer sialylierter Glykoproteine in Zellen vorgestellt, das metabolische Markierung, bioorthogonale Klickchemie und Expansionsmikroskopie kombiniert. In den in dieser Arbeit beschriebenen experimentellen Verfahren verwenden wir N-Azidoacetylmannosamin (ManNAz) als chemischen Reporter, und die CuAAC-Ligation von kleinen organischen Fluorophoren wird vor dem Expansionsverfahren durchgeführt. Glykoproteine werden vor der Gelierung, Verdauung und Expansion mit NAS am Hydrogel verankert. Die MOE-Markierung von Sialinsäuren kann mit Immunfluoreszenzansätzen zur Kolokalisationsbewertung assoziiert werden, wie hier am Beispiel eines Maus-Anti-GM130-Primärantikörpers, der im cis-Golgi-Apparat lokalisiert ist. Dieses Protokoll kann auf Zellen in ihrem physiologischen Zustand oder auf Zellen angewendet werden, die einer chemischen Behandlung unterzogen wurden. Um dies zu veranschaulichen, wurde Chloroquin verwendet, um die lysosomale Funktion zu hemmen, die die Verarbeitung und den Transport von Glykoproteinen innerhalb der Zellen beeinflusst. Die Kernfärbung wird als Meilenstein verwendet, nicht nur zur Lokalisierung von Zellen, sondern auch als Indikator für die Qualität des ExM-Prozesses. Der Expansionsfaktor kann in der Tat gemessen werden, indem man die Größe des Kerns vor der Expansion (preExM) und nach der Expansion (postExM) vergleicht.
Protokoll
1. Aussaat von Zellen
HINWEIS: Führen Sie die nächsten Schritte unter sterilen Bedingungen unter einer Laminar-Flow-Haube durch. Diese Methode kann auf jede der in der vorliegenden Arbeit verwendeten Zelllinien (HeLa, MCF7, primäre Fibroblasten) oder auf die meisten adhärenten Zelllinienmodelle angewendet werden, die üblicherweise in der Forschung verwendet werden 20,24,25.
- Züchten Sie Zellen in DMEM-Medium mit hohem Glukosegehalt, das mit 10 % fötalem Rinderserum (FBS) in einem T75-Kolben bei 37 °C und einer 5 % CO2 -Atmosphäre ergänzt wird.
- Sobald die Zellen die volle Konfluenz erreicht haben, entfernen Sie das Zellkulturmedium und waschen Sie die Zellen mit 4 ml PBS.
HINWEIS: PBS-Lösungen, die in diesem Schritt und allen nachfolgenden Waschschritten verwendet werden, müssen vor der Verwendung durch Filtration durch eine 0,2 μm PTFE-Membran sterilisiert werden. - Die Zellen werden 5 Minuten lang bei 37 °C in 2 ml 1x Trypsin-EDTA-Lösung mit 0,5 g/l Trypsin inkubiert, um sie vom Boden des Kolbens freizusetzen.
- Fügen Sie 8 ml DMEM-Medium mit hohem Glukosegehalt mit 10 % FBS hinzu und mischen Sie gründlich, um sicherzustellen, dass sich jede einzelne Zelle vom Boden des Kolbens löst.
- Die Zellen werden in ein konisches 15-ml-Röhrchen überführt und 5 Minuten lang bei 200 × g zentrifugiert. In der Zwischenzeit fügen Sie in jeder Vertiefung einer 12-Well-Platte ein Deckglas (Durchmesser 12 mm, Dicke 130-160 μm) hinzu.
HINWEIS: Die Deckglasdicke sollte an die Anforderungen des Mikroskops angepasst werden, das in den nachfolgenden Bildgebungsschritten verwendet wird. - Entfernen Sie den Überstand nach der Zentrifugation und resuspendieren Sie das Pellet in 1 ml Zellkulturmedium.
- Bestimmen Sie die durchschnittliche Anzahl von Zellen/ml durch Hämozytometrie über mindestens zwei Messungen.
- Verdünnen Sie die Zellsuspension auf eine Konzentration von 300.000 Zellen pro ml in Zellkulturmedium und säen Sie 1 ml der Suspension in jede Vertiefung.
- Lassen Sie die Zellen 24 Stunden lang in einem DMEM-Medium mit hohem Glukosegehalt von 10 % FBS bei 37 °C unter einer 5 % CO2 -Atmosphäre ruhen, bevor Sie mit den nächsten Schritten beginnen.
2. Chloroquin-Behandlung (optional)
HINWEIS: Die Schritte 2.1-2.8 veranschaulichen am Beispiel von Chloroquin, wie das Protokoll auf Zellen angewendet wird, die mit einem externen Reagenz (Inhibitor, Effektor, Medikament) behandelt werden. Überspringen Sie diese Schritte für Zellen, die nicht mit externen Reagenzien behandelt wurden.
- Wiegen Sie die richtige Menge Chloroquindiphosphatsalz (CQ), um ein ausreichendes Volumen einer 10 mM Stammlösung herzustellen.
HINWEIS: Führen Sie die nächsten Schritte unter sterilen Bedingungen unter einer Laminar-Flow-Haube durch. - Solubilisieren Sie den festen CQ in der gewünschten Menge deionisiertem Wasser für eine Endkonzentration von 10 mM.
- Die Lösung durch einen 0,2 μm Filter filtrieren.
- Aliquotieren Sie 1 mL der 10 mM Stammlösung in 1,5 mL Mikrozentrifugenröhrchen und lagern Sie diese bei -20 °C.
- Die in Schritt 1.9 vorbereitete Zellprobe wird mit 1 ml PBS gewaschen.
- Fügen Sie 100 μl 10 mM CQ in 9,9 mL DMEM-Medium mit hohem Glukosegehalt mit 10 % FBS hinzu, um eine Endkonzentration von 100 μM CQ zu erreichen.
- Inkubieren Sie die Zellen 8 h lang in 1 ml eines Mediums, das mit 100 μM CQ bei 37 °C unter einer 5 % CO2 -Atmosphäre ergänzt wurde.
- Waschen Sie die Zellen 3x mit 1 mL PBS.
3. Metabolisches Oligosaccharid-Engineering
- Bereiten Sie eine 5 mM Stammlösung von ManNAz (oder ManNAc für die Negativkontrollbedingung) in DMEM-Medium mit hohem Glukosegehalt mit 10 % FBS vor.
HINWEIS: Diese Stammlösung sollte ein ausreichendes Volumen haben, so dass jede Vertiefung mit 1 ml Zuckeranaloglösung behandelt werden kann. Führen Sie die nächsten Schritte unter sterilen Bedingungen unter einer Laminar-Flow-Haube durch. - Die Lösung wird durch einen 0,2-μm-Filter filtriert und bis zur Verwendung in 1 ml-Aliquoten bei -20 °C gelagert.
- Ein mit 500 μM ManNAz oder ManNAc ergänztes Medium wird hergestellt, indem eine in Schritt 3.2 hergestellte 1-ml-Stammlösung aliquot mit 9 ml DMEM-Medium mit hohem Glukosegehalt von 10 % FBS verdünnt wird.
- Die in Schritt 1.9 (für Versuche an unbehandelten Zellen) oder in Schritt 2.8 (für Versuche an CQ-behandelten Zellen) hergestellten Zellen werden in 1 ml des Mediums, zugegeben mit 500 μM ManNAz (oder 500 μM ManNAc für die Kontrollgruppe), für 24 h bei 37 °C unter einer Atmosphäre von 5 % CO2 inkubiert.
4. Fixierung und Permeabilisierung
HINWEIS: Alle diese Schritte werden unter einem Abzug durchgeführt.
- Waschen Sie die Proben mit 1 mL PBS.
- Fixieren Sie die Zellen auf jedem Deckglas, indem Sie 500 μl 5% Paraformaldehyd hinzufügen. 15 min bei Raumtemperatur ruhen lassen.
- 3x mit 1 mL PBS waschen.
- Bereiten Sie eine benutzerdefinierte Feuchtkammer vor, indem Sie eine ausreichend große, undurchsichtige Schachtel (z. B. eine Styroporbox) mit Deckel verwenden. Lege ein feuchtes Blatt Löschpapier auf den Boden der Schachtel und lege dann eine Schicht Parafilm darauf.
HINWEIS: Die Feuchtkammer sollte die Probe im geschlossenen Zustand vollständig vor äußeren Lichtquellen schützen. - Legen Sie die Deckgläser mit den Zellen nach oben auf den Parafilm.
HINWEIS: Beschriften Sie den Parafilm im Voraus mit dem Namen oder der Referenz der Proben. - Führen Sie die Zellpermeabilisierung mit 200 μl 0,5 % (v/v) Triton X-100 in PBS für 15 min bei Raumtemperatur durch.
- 3x mit 200 μL PBS waschen.
5. Fluoreszenz-Markierung
- CuAAC-Markierung von markierten sialylierten Glykanen
HINWEIS: Der CuAAC-Reaktionspuffer muss vor der Verwendung frisch zubereitet werden, und die Reaktanten sollten in der angegebenen Reihenfolge hinzugefügt werden, wobei Natriumascorbat zuletzt zugegeben wird. In sauerstoffhaltigen wässrigen Lösungen oxidieren Cu(I)-Ionen, die durch die Reduktion von Cu(II) mit Ascorbat gebildet werden, allmählich wieder zu Cu(II), was im Laufe der Zeit zu einer Abnahme der Ascorbatkonzentration führt. Um die Reproduzierbarkeit des Ergebnisses zu erhalten, sollte der Puffer nicht länger als einige Tage bei 4 °C gelagert werden und wird vorzugsweise am selben Tag verwendet.- Zur Markierung der in Abschnitt 3 hergestellten azidmodifizierten sialylierten Glykane wird ein CuAAC-Reaktionspuffer hergestellt, der aus 5 μM AlexaFluor 488-Alkin (AF488Alk), 150 μM CuSO4, 300 μM 2-(4-((bis(1-(tert-butyl)-1H-1,2,3-triazol-4-yl)methyl)amino)methyl)-1H-1,2,3-triazol-1-yl)essigsäure (BTTAA), 0,1 M K2HPO4 und 2,5 mM Natriumascorbat in deionisiertem Wasser besteht. Bereiten Sie genügend Lösung vor, um 200 μl auf jedes Deckglas aufzutragen.
- Geben Sie 200 μl CuAAC-Puffer auf jedes Deckglas, um die Reaktion zu starten. Stellen Sie sicher, dass die Probe homogen mit dem Tropfen bedeckt ist, und lassen Sie sie 45 Minuten lang bei Raumtemperatur lichtgeschützt reagieren.
- 3x mit 200 μl PBS waschen, um die Reaktion zu stoppen und überschüssige Sonde und Reagenzien zu entfernen.
- Färbung von Zellkernen
- Für die nukleare Markierung mit Hoechst 33342 wird eine 10 mg/ml-Stammlösung bei 1:2000 in PBS verdünnt.
ANMERKUNG: Äquivalente nukleare Gegenfärbungen wie 4',6-Diamidino-2-phenylindol (DAPI) können mit dem gleichen Effekt verwendet werden. - Decken Sie die Zellen mit 200 μl dieser Lösung ab und inkubieren Sie sie 5-10 Minuten lang bei Raumtemperatur, geschützt vor Licht.
- 3x mit 200 μL PBS waschen.
- Für die nukleare Markierung mit Hoechst 33342 wird eine 10 mg/ml-Stammlösung bei 1:2000 in PBS verdünnt.
- Immunfluoreszenz-Färbung
- Bereiten Sie einen Rinderserumalbumin (BSA)-Blockpuffer vor, der 0,1 g Gelatine, 1 g BSA, 1 ml FBS und 49 ml PBS für 50 ml Puffer enthält.
- Die Proben werden 1 h lang bei 4 °C in 200 μl BSA-Blockierungspuffer inkubiert. Schützen Sie die Probe vor Licht.
- Für die Immunfluoreszenzfärbung des Golgi-Apparats unter Verwendung eines gereinigten Maus-Anti-GM130-Antikörpers (Epitop erkannt: Ratte GM130 aa. 869-982 | RRID: AB_398142), den Primärantikörper bei 1:100 in dem vorbereiteten BSA-Blockierungspuffer verdünnen.
HINWEIS: Dieser Schritt kann an die Immunfluoreszenzfärbung angepasst werden, die auf andere Organellen abzielt, indem ein relevanter Primärantikörper verwendet wird, wobei in diesem Fall der Verdünnungsfaktor gemäß den Empfehlungen des Herstellers angepasst werden sollte. - 70 μl der Lösung, die den Antikörper enthält, auf die Deckgläser geben und 1 h bei 4 °C lichtgeschützt inkubieren.
- 3x mit 200 μL PBS waschen.
- Verdünnen Sie den fluoreszierenden Sekundärantikörper (Alexa Fluor 546 Anti-Maus-IgG) bei 1:600 in BSA-Blockierungspuffer und fügen Sie 100 μl auf jedes Deckglas hinzu.
- Inkubieren Sie die Proben 1 h lang bei Raumtemperatur und vor Licht geschützt.
- 3x mit 200 μL PBS waschen.
- Übertragen Sie die Deckgläser auf eine 6-Well-Platte. Lagern Sie die Proben in 2 mL PBS bei 4 °C für einige Tage lichtgeschützt.
HINWEIS: Es wird empfohlen, ein Replikat immer ausschließlich für die Visualisierung von preExM als Referenz für Vergleichszwecke vor und nach der Erweiterung vorzubereiten und sicherzustellen, dass das Beschriftungsmuster nach der Erweiterung nicht verzerrt wurde.
6. Präexpansionsbildgebung von Proben
HINWEIS: Die folgenden Schritte hängen von den verwendeten Bioimaging-Geräten ab und müssen möglicherweise an die Anforderungen des Geräts angepasst werden. Bitte befolgen Sie die Regeln des örtlichen Labors oder der Bioimaging-Plattform. Die Schritte 6.1-6.13 werden hier an einem konfokalen Laser-Scanning-Mikroskop durchgeführt.
- Schalten Sie das Mikroskop ein und stellen Sie sicher, dass die Lichtquellen (Laser) erwärmt sind. Öffnen Sie die Bioimaging-Erfassungssoftware.
- Wählen Sie die Laseranregungswellenlängen aus, die den Fluorophoren entsprechen, um die Kanäle zu erstellen (z. B. 405 nm für die Kernfärbung, 488 nm für AF488Alk-markierte sialylierte Glykane und 546 nm für die Immunfluoreszenzmarkierung). Aktivieren Sie die Laser entsprechend.
- Legen Sie ein Deckglas mit einem Durchmesser von 32 mm (130-170 μm Dicke) in eine für das Mikroskop geeignete Halterung und befestigen Sie sie.
- Die in Schritt 5.3.9 vorbereitete Probe wird mit den Küvetten nach unten auf das 32-mm-Deckglas gelegt. Fügen Sie einen Tropfen entionisiertes Wasser hinzu, um ein Austrocknen der Probe zu verhindern.
- Wählen Sie ein passendes Objektiv aus, hier ein 63-fach Öl-Immersionsobjektiv mit einer numerischen Apertur von 1,4 oder gleichwertig.
HINWEIS: Die höchste numerische Apertur bietet die beste Auflösung. Wenn Sie ein Ölimmersionsobjektiv verwenden, denken Sie daran, einen Tropfen Öl zwischen das Objektiv und das Deckglas zu geben. - Platzieren Sie die Halterung auf dem Mikroskoptisch über dem Objektiv.
- Verwenden Sie den Hellfeld- oder Laserintensitäts-Fluoreszenzmodus, um einen interessanten Bereich in der Probe zu lokalisieren.
- Stellen Sie den Fokus ein.
- Visualisieren Sie die Zellen in der Software im Live-Fluoreszenz-Modus . Stellen Sie die Laserintensität für jeden Kanal ein. Beginnen Sie mit einer geringen Laserleistung, um Photobleaching zu vermeiden. Erhöhen Sie langsam die Laserleistung und passen Sie die Verstärkung und den Offset des Detektors an, um das Signal zu verstärken, ohne übermäßiges Rauschen einzuführen, um die Fluoreszenz klar und ohne Überbelichtung sichtbar zu machen.
HINWEIS: Die Parameter sollten angepasst werden, um sowohl Hintergrundgeräusche zu minimieren als auch Signalsättigung zu vermeiden. Es ist von entscheidender Bedeutung, in einer Reihe von Experimenten, einschließlich Negativkontrollen, konsistente Einstellungen für alle Proben aufrechtzuerhalten. - Stellen Sie Bildparameter wie Lochblendengröße (z. B. 1 Airy Unit), Scangeschwindigkeit (z. B. 8), Zoomfaktor (z. B. 1), Bereich der Helligkeitswerte (z. B. 16 Bit) und Mittelungszahl (z. B. 2), Modus (z. B. Linie) und Methode (z. B. Mittelwert) nach Bedarf ein, um die gewünschte Beobachtung zu erreichen.
HINWEIS: Diese Werte sind system- und probenabhängig und sollten vor der Erfassung optimiert werden. Die Größe der Lochblende hängt von der numerischen Apertur, der Vergrößerung und der Wellenlänge ab: Eine Verringerung kann die Auflösung auf Kosten des Signal-Rausch-Verhältnisses verbessern. Es wird empfohlen, die Scangeschwindigkeit auf schnelle Werte einzustellen (entsprechend einer kleinen Verweilzeit pro Pixel), um Photobleaching zu vermeiden. - Führen Sie die Erfassung durch und speichern Sie die Daten im gewünschten Format mit den richtigen Metadaten zum späteren Nachschlagen.
- Sobald die Erfassung abgeschlossen ist, geben Sie die Probe wieder in die 6-Well-Platte, indem Sie eine kleine Menge deionisiertes Wasser hinzufügen, um den Transfer mit einer Pinzette zu erleichtern.
HINWEIS: Schritt 6.12 wird nur durchgeführt, wenn eine Erweiterung dieser speziellen Probe erforderlich ist. Andernfalls verwerfen Sie die Stichprobe, und verwenden Sie andere Replikate für die folgenden Schritte.
7. Protokoll der Expansionsmikroskopie
HINWEIS: Die Proben werden so weit wie möglich vor Licht geschützt, um Photobleichen zu vermeiden. Die Verankerungs- und Gelierungsschritte werden unter einem Abzug durchgeführt.
- Verankerung
- Die Proben werden mit N-Acryloylsuccinimid (NAS) in einer Konzentration von 3,2 mg/ml PBS auf einem mechanischen Schüttler für 1 h bei Raumtemperatur inkubiert. Bereiten Sie eine ausreichende Menge Lösung vor, um 500 μl Lösung pro Vertiefung hinzuzufügen (Stammlösung: 16 mg/ml NAS in DMSO).
HINWEIS: NAS reagiert mit den Lysinresten und N-Termini von Proteinen, um eine Acryloylgruppe zu transplantieren, so dass sich die Proteine in nachfolgenden Schritten am Hydrogel verankern können. - 3x mit 1 mL PBS waschen.
- Die Proben werden mit N-Acryloylsuccinimid (NAS) in einer Konzentration von 3,2 mg/ml PBS auf einem mechanischen Schüttler für 1 h bei Raumtemperatur inkubiert. Bereiten Sie eine ausreichende Menge Lösung vor, um 500 μl Lösung pro Vertiefung hinzuzufügen (Stammlösung: 16 mg/ml NAS in DMSO).
- Erstarren
- Bereiten Sie eine Monomerlösung her, die wie folgt zusammengesetzt ist: In 50 mL 1x PBS (pH 7,4) fügen Sie 5,843 g NaCl, 1,25 g Acrylamid, 0,075 g Bis-Acrylamid und 4,313 g Natriumacrylat hinzu. Bereiten Sie genügend Lösung vor, um 70 μl auf jedes Deckglas aufzutragen. Frieren Sie 1 ml Aliquote mindestens 24 h vor der Gelierung ein.
ACHTUNG: Die Monomere sind giftig; Sie müssen vorsichtig mit geeigneter Schutzausrüstung behandelt und unter einem Abzug gewogen werden. - Auf ein Stück Parafilm wird ein 70 μl Tropfen Monomerlösung gegeben, gefolgt von der Zugabe von 1,4 μl 10 % (v/v) N,N,N',N'-Tetramethylethylendiamin (TMEDA), dann 1,4 μl 10 % (w/v) Ammoniumpersulfat (APS), die in den Tropfen gemischt werden.
HINWEIS: Die Vernetzer-Reagenzien müssen in dieser speziellen Reihenfolge hinzugefügt werden, zuerst TMEDA, dann APS. - Legen Sie das Deckglas schnell vorsichtig mit den Zellen nach unten auf die Lösung.
- Lassen Sie die Lösung 1 h bei Raumtemperatur in einer feuchten Kammer polymerisieren.
- Bereiten Sie eine Monomerlösung her, die wie folgt zusammengesetzt ist: In 50 mL 1x PBS (pH 7,4) fügen Sie 5,843 g NaCl, 1,25 g Acrylamid, 0,075 g Bis-Acrylamid und 4,313 g Natriumacrylat hinzu. Bereiten Sie genügend Lösung vor, um 70 μl auf jedes Deckglas aufzutragen. Frieren Sie 1 ml Aliquote mindestens 24 h vor der Gelierung ein.
- Verdauung
- Bereiten Sie eine Aufschlusspufferlösung vor, die aus 1x TAE-Puffer, 0,5 % (v/v) Triton X-100, 0,8 M Guanidin-HCl und 8 U/mL Proteinase K besteht.
HINWEIS: Der Aufschlusspuffer muss frisch zubereitet und vor der Verwendung 30 Minuten lang bei 37 °C vorinkubiert werden. - 1 ml Aufschlusspuffer zu dem in Schritt 7.2 gebildeten Hydrogel geben. Der Aufschluss wird 3 Stunden lang bei 37 °C auf einem mechanischen Schüttler durchgeführt.
- Entfernen Sie den Aufschlusspuffer und waschen Sie das Gel vorsichtig 3x mit 2 mL deionisiertem Wasser.
HINWEIS: Das Hydrogel dehnt sich leicht aus und löst sich vom Deckglas.
- Bereiten Sie eine Aufschlusspufferlösung vor, die aus 1x TAE-Puffer, 0,5 % (v/v) Triton X-100, 0,8 M Guanidin-HCl und 8 U/mL Proteinase K besteht.
- Erweiterung
- Lassen Sie das Hydrogel 2 h lang in 3 mL deionisiertem Wasser expandieren. Wechseln Sie das Wasser alle 30 Minuten.
8. Post-Expansions-Imaging von Proben
- Legen Sie ein Deckglas mit einem Durchmesser von 32 mm (130-170 μm Dicke) auf und befestigen Sie es auf einer Halterung.
- Schneiden Sie ein Stück des Hydrogels aus und legen Sie es auf das Deckglas, wobei sich die Zellen am unteren Rand des Gels befinden.
HINWEIS: Die herausgeschnittenen Stücke sollten groß genug sein, um eine ausreichende Oberfläche für die Beobachtung zu bieten, aber nicht zu groß, da dies die Handhabung des Gels erschweren und das Bruchrisiko erhöhen könnte (empfohlene Größe: 5 bis 8 mm). Platzieren Sie genügend Hydrogelstücke, um die gesamte Oberfläche des Deckglases zu bedecken. Dies hilft, die Teile an Ort und Stelle zu sichern und ein Abdriften während der Bildgebung zu verhindern. Bei Abdriftproblemen kann sich eine Vorbeschichtung der Deckgläser mit Poly-D-Lysin als vorteilhaft erweisen6. - Platzieren Sie die Halterung auf dem Mikroskoptisch über dem Objektiv.
- Führen Sie die Bildaufnahme an einem konfokalen Laser-Scanning-Mikroskop mit einem Ölimmersionsobjektiv durch, wie in Schritt 6 beschrieben.
9. Berechnung des Expansionsfaktors (EF) in ImageJ
- Öffnen Sie die Imaging-Daten in der Freeware ImageJ oder einer gleichwertigen Version.
- Wenn Sie an einem Bild mit mehreren Kanälen arbeiten, teilen Sie die Kanäle und arbeiten Sie nur mit dem Nukleuskanal, indem Sie zu Bild auswählen | Farbe | Kanäle teilen.
- Um den Kerndurchmesser zu messen, markieren Sie das Signal, das dem Kern entspricht, indem Sie einen Schwellenwert bestimmen, indem Sie auf Bild auswählen | Anpassen | Schwelle.... Wählen Sie im Standard-Dropdown-Menü eine Methode aus, die am besten zum Bild passt (z. B. Otsu), und passen Sie den Schwellenwert an, bis die Form des Kerns deutlich aus dem Hintergrund herausgeschnitten wird.
HINWEIS: Die Schwellenwerteinstellung muss für jedes Bild angepasst werden, da eine Einstellung möglicherweise nicht für alle Bilder oder jedes Sample geeignet ist. - Stellen Sie die Messung so ein, dass der Feret-Durchmesser bestimmt wird, indem Sie auf Analysieren auswählen | Maße einstellen.... Wählen Sie im Menü Feret-Durchmesser und passen Sie bei Bedarf die Dezimalzahl an.
- Um mit der Analyse des Bildes zu beginnen, klicken Sie auf Analysieren auswählen | Partikel analysieren , um die Größe der Partikel festzulegen; Wählen Sie für den Zellkern 5-unendlich μm² | Ergebnisse anzeigen | Zum Manager hinzufügen | An Kanten ausschließen & Überlagern. Starten Sie die Analyse.
- Wenn die Analyse beginnt, werden mehrere Fenster angezeigt. Suchen Sie in den Fensterergebnissen nach dem maximalen Feret-Durchmesser jedes Kerns in der Spalte Feret. Kopieren Sie die Rohdaten und fügen Sie sie in eine Tabelle ein und berechnen Sie die durchschnittliche Zellkerngröße für vorexpandierte und expandierte Proben.
HINWEIS: Stellen Sie sicher, dass die Regions of Interest (ROIs), die jedem Kern entsprechen, der gesamten Peripherie des Kerns folgen. - Berechnen Sie den Expansionsfaktor (EF) wie folgt:
EF =
HINWEIS: Der EF-Faktor bestimmt, wie stark die Stichprobe erweitert wurde.
Ergebnisse
Im Folgenden ist die Anwendung des Protokolls zur Visualisierung von sialylierten Glykoproteinen in HeLa-Zellen (Abbildung 3A) und MCF7-Zellen (Abbildung 3B) dargestellt, wobei die CQ-Behandlung (Protokollabschnitt 2) und die Immunfluoreszenz-Colokalisationsfärbung (Protokollschritt 5.3) weggelassen werden.

Abbildung 3: Vergleich des Sialylierungsmarkierungsmusters vor (links) und nach (rechts) dem ExM-Protokoll. (A) Hela-Zellen und (B) MCF7-Zellen wurden 24 h lang mit 500 μM ManNAz inkubiert. Die Markierung erfolgte durch CuAAC mit Alexa Fluor 488 Alkin (grün dargestellt). Die Zellkerne wurden mit Hoechst 33342 (blau dargestellt) angefärbt. Maßstabsbalken = 20 μm. Abkürzungen: ExM = Expansion Microscopy; ManNAz = N-Azidoacetylmannosamine; CuAAC = Kupfer-katalysierte Azid-Alkin-Cycloaddition. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Typischerweise besteht die Sialylierungsmarkierung aus drei Hauptfluoreszenzpools: einem Golgi-Pool, der die Synthese naszierender Glykokonjugate anzeigt, einem vesikulären Pool (normalerweise weniger intensiv), der Glykokonjugate darstellt, die in die Zellmembran exportiert oder von ihr recycelt werden, und einem Membranpool, der Glykokonjugate zeigt, die exprimiert wurden13. Es ist wichtig zu beachten, dass nur Biopolymer-Glykokonjugate, die mit dem metabolisch erzeugten Neu5Az markiert sind, nachgewiesen werden, während nicht umgesetzte überschüssige Reporter oder Sonden während der Waschschritte eliminiert werden. Die Bilder nach ExM wurden mit einem konfokalen Mikroskop aufgenommen und zeigen viel feinere Details und weniger Hintergrund, insbesondere im Golgi-Apparat. Das Protokoll kann auf jede Art von adhärenten Zellen angewendet werden, die sialylierte Glykokonjugate exprimieren.
Verschiedene Zell- oder Gewebetypen können in unterschiedlichem Maße durch den teilweisen Verlust des ExM-intrinsischen Signals beeinflusst werden, der auf die volumetrische Verdünnung von Fluorophoren zurückzuführen ist. Dies kann sich auch durch eine schlechte Retention von Fluorophoren während der radikalischen Polymerisation oder von Biomolekülen aufgrund einer suboptimalen Verankerung oder Verdauung verschlimmern, was zu einem drastischen Signalverlust führen kann (Abbildung 4). Dieser Punkt ist das Hauptthema der folgenden Diskussion. Es wird daher empfohlen, die Parameter der Verdauungsschritte bei Bedarf an den untersuchten Zell- oder Gewebetyp anzupassen. Die Verdauung kann auf verschiedene Weise optimiert werden, abhängig von den spezifischen Strukturen oder Biomolekülen, die beobachtet werden. Wir empfehlen, mit einem standardisierten Aufschlussprotokoll zu beginnen und dann Parameter wie Aufschlusszeit, Waschmittelkonzentration und/oder Enzymkonzentration empirisch anzupassen, um die gewünschten Ergebnisse zu erzielen. Es ist wichtig zu beachten, dass ein effizienterer Aufschluss zu einer besseren isotropen Expansion führt, wodurch Verzerrungen vermieden werden, was aber auch das Risiko von Signalverlusten erhöhen kann. Daher muss ein Gleichgewicht zwischen dem Erreichen einer qualitativ hochwertigen isotropen Expansion und der Beibehaltung der Signalintegrität gefunden werden. Die Signalspeicherung kann durch die Verwendung speziell entwickelter molekularer Anker weiter verbessert werden. In Studien, die sich auf die Proteinbeobachtung konzentrierten, wurde gezeigt, dass Protokolle, die nur Natriumdodecylsulfat (SDS) bei erhöhten Temperaturen verwenden, die eingebettete Probe effektiv verdauen und gleichzeitig die Proteinsignale besser erhalten26,27.
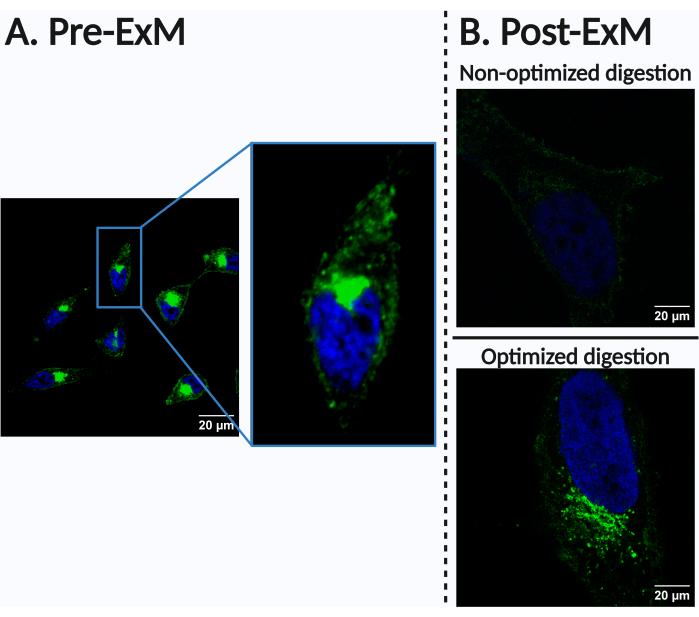
Abbildung 4: Beispiel für ein suboptimales Ergebnis. Diese Abbildung zeigt, wie wichtig es ist, die Bedingungen an jeden Zelltyp, jedes Gewebe und jedes überwachte Biomolekül anzupassen, insbesondere für den Verdauungsschritt. HeLa-Zellen wurden 24 h mit 500 μM ManNAz inkubiert und mit Alexa Fluor 488 Alkin (grün, sialylierte N-Glykoproteine) und Hoechst 33342 (blau, Zellkerne) markiert. (A) Beschriftungsmuster vor der Expansion. (B) Beschriftungsmuster nach der Erweiterung. (Nach oben) Schlecht optimierte Verdauungsparameter in Bezug auf Zusammensetzung, Konzentration und/oder Länge können zu drastischen Signalverlusten führen, da das transplantierte Biomolekül im Hydrogel nicht retentiert ist, was eine bioimagingische Datenerfassung mit ausreichender Empfindlichkeit verhindert. In diesem speziellen Fall war der Verdauungspuffer ähnlich wie in Protokollschritt 7.3 verwendet. Die Aufschlusszeit betrug jedoch 90 Minuten, verglichen mit der Aufschlusszeit von 180 Minuten, die im nachfolgenden Bild verwendet wurde. (Unten) Das Bild wurde nach Optimierung der Aufschlussparameter erhalten (siehe Protokollschritt 7.3). Maßstabsbalken = 20 μm. Abkürzung: ManNAz = N-Azidoacetylmannosamine. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Um die Reproduzierbarkeit des Experiments zu gewährleisten, ist es entscheidend, die Proben in Lösung zu konservieren und sie vor Licht zu schützen, auch nachdem sie in das Hydrogel eingebettet wurden. Das Hydrogel muss in einer feuchten Kammer aufbewahrt werden, um ein Austrocknen zu verhindern. Es wird dringend empfohlen, die drei Hauptschritte – Gelierung (Protokollschritt 7.2), Aufschluss (Protokollschritt 7.3) und Expansion (Protokollschritt 7.4) – am selben Tag durchzuführen. Nach der Expansion ist die Probe einige Tage lang stabil, wenn sie bei 4 °C in entionisiertem Wasser aufbewahrt und vor Licht geschützt wird, so dass Zeit für die Aufnahme von Bioimaging-Daten bleibt.
Bei der Analyse von Mikroskopiebildern wird empfohlen, den Expansionsfaktor (EF) zu bewerten, indem das durchschnittliche Verhältnis des Feret-Durchmessers des Kerns vor (preExM) und nach der Expansion (postExM) gemessen wird. Hier werden in diesem Protokoll die Zellkerne mit Hoechst 33342 gefärbt, aber das konventionellere DAPI ist auch mit der Expansionsmikroskopie kompatibel. Der Feret-Durchmesser entspricht dem maximalen Abstand zwischen zwei Punkten, die sich an der Grenze des Kerns befinden. Typischerweise liefern erfolgreiche Experimente einen EF-Wert von 4 bis 5 (Abbildung 5), was einer maximalen lateralen Auflösung von ~60-70 nm entspricht, abhängig von der verwendeten Wellenlänge und dem Objektiv. Der durchschnittliche Feret-Durchmesser von Zellkernen wird bestimmt, indem mindestens 60 bis 100 Zellen in mehreren Bildern für einen statistischen Ansatz gemessen werden. Für eine höhere Genauigkeit können Z-Stacks, die eine 3D-Rekonstruktion ermöglichen, systematisch erfasst und verarbeitet werden, um sicherzustellen, dass der gemessene Abstand dem tatsächlichen Feret-Durchmesser für alle Kerne entspricht. Dieser Ansatz kann jedoch deutlich zeitaufwändiger sein, sowohl in Bezug auf die Aufnahme je nach verwendetem Mikroskop und Objektiv als auch insbesondere in Bezug auf die Datenaufbereitung. Dies kann durch eine makroskalige Messung der EF überprüft werden, indem die Größe des Hydrogels vor und nach der Expansion verglichen wird, was nach unserer Erfahrung zu ähnlichen Ergebnissen führt.

Abbildung 5: Bestimmung des Ausdehnungsfaktors. Die durchschnittliche Größe von Kernen vor und nach der Expansion wird durch die Messung ihres Feret-Durchmessers (d.h. des längsten Abstands zwischen zwei Punkten am Rand des Kerns) in der ImageJ-Software bestimmt. Die Berechnung des Verhältnisses zwischen dem durchschnittlichen Feret-Durchmesser nach ExM und vor ExM liefert den linearen Ausdehnungsfaktor. Die humanen primären Fibroblasten 533T wurden dem Expansionsprotokoll unterzogen. Die Zellkerne wurden mit Hoechst 33342 (blau dargestellt) angefärbt. Maßstabsbalken = 20 μm. Abkürzung: ExM = Expansion Microscopy. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Dieses Protokoll wurde entwickelt, um biologische Fragen zu beantworten, die spezifische Zellbehandlungen oder Vergleiche zwischen verschiedenen physiologischen und pathologischen Zuständen erfordern. Ein Beispiel dafür ist hier die Anwendung an humanen Fibroblasten, die mit dem Malariamedikament Chloroquin (CQ) behandelt wurden. CQ, eine diprotische schwache Base mit pKa-Werten von 8,4 und 10,8, reichert sich bevorzugt in Lysosomen über pH-Trapping an, was zu einem erhöhten lysosomalen pH-Wert und einem anschließenden Funktionsverlust führt.
In diesem Experiment wird ManNAz in sialylierte Glykane eingebaut, die dann mit Alexa Fluor 488 über CuAAC markiert werden, während die Golgi-Apparatur mit dem Maus-Anti-GM130-Antikörper gefärbt wird. Bemerkenswert ist, dass deutliche Unterschiede in der relativen Intensität zwischen Kontrollzellen (Abbildung 6A) und CQ-behandelten Zellen (Abbildung 6B) beobachtet werden. Unter physiologischen Bedingungen in Kontrollzellen ist der primäre Pool der Sialokonjugatmarkierung innerhalb des Golgi-Apparats lokalisiert. Im Gegensatz dazu weisen CQ-behandelte Zellen eine Sialinsäure-Akkumulation in Lysosomen auf, was auf das Einfangen von recycelten Glykokonjugaten hinweist. Dies ist besonders relevant im Zusammenhang mit der Untersuchung angeborener Störungen der Glykosylierung oder lysosomaler Speicherkrankheiten 13,14,15.

Abbildung 6: PostExM-Visualisierung der Wirkung von Chloroquin auf die Lokalisation von sialylierten Glykoproteinen postExM. Humane primäre Fibroblasten 533T wurden entweder (A) nicht mit CQ behandelt oder (B) mit 100 μM CQ für 8 h vor der Inkubation mit 500 μM ManNAz für 24 h und CuAAC-Markierung mit Alexa Fluor 488 Alkin (grüner Kanal). Die Co-Lokalisationsfärbung des Golgi-Apparats (roter Kanal) wurde mit einem Maus-Anti-GM130-Primärantikörper und einem Anti-Maus-Alexa Fluor 546-markierten Sekundärantikörper durchgeführt. Die Zellen wurden wie beschrieben einer Verankerung, Gelierung, Verdauung und Expansion unterzogen. Maßstabsbalken = 20 μm. Abkürzungen: ExM = Expansion Microscopy; CQ = Chloroquin; ManNAz = N-Azidoacetylmannosamine; CuAAC = Kupfer-katalysierte Azid-Alkin-Cycloaddition. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Das derzeitige CuAAC-Markierungsprotokoll enthält kein Aminoguanidin im Reaktionspuffer. Da es darauf abzielt, intrazelluläre Glykokonjugate zu visualisieren, wird es an Zellen durchgeführt, die nach dem metabolischen Einbauschritt fixiert sind, um Probleme mit der Zytotoxizität zu vermeiden und die Aufnahme des katalytischen Systems zu verbessern. Die Verwendung von Aminoguanidin wird typischerweise für die Markierung der Zelloberfläche lebender Zellen empfohlen, um Nebenreaktionen zwischen Dehydroascorbat und Arginin-, Histidin- und Lysinresten von Proteinen zu verhindern28. Diese Modifikationen könnten in der Tat die Proteinfunktion beeinträchtigen und möglicherweise Störungen oder Zytotoxizität verursachen. Ein weiterer wichtiger Aspekt für den CuAAC-Reaktionspuffer ist, dass die Komplexierung von Kupferionen durch BTTAA schnell, aber nicht sofort erfolgt. Um eine effiziente Markierung der modifizierten Zucker zu gewährleisten, empfehlen wir, die Lösung mindestens 30 Minuten vor Beginn des Markierungsprozesses vorzubereiten, um die Reproduzierbarkeit der Ergebnisse für unerfahrene Anwender zu gewährleisten.
Jedes konfokale Mikroskop kann für die Aufnahme verwendet werden. Wenn sich die Stichprobe erweitert, nimmt auch das beobachtbare Feld zu. Um eine ganze Zelle zu erfassen, empfiehlt es sich, einen Kachelscan des Bereichs mit einer Überlappung von 10 % zwischen den Bildern durchzuführen. Obwohl es möglich ist, einen Kachelscan und einen Z-Stack gleichzeitig durchzuführen, empfehlen wir, sich in diesem Fall für einen kleineren Kachelscan zu entscheiden. Dies liegt daran, dass der Prozess zeitaufwändig ist und eine beträchtliche Menge an Rohdaten erzeugt, was zu einer intensiven Datenverarbeitung führt. Es ist ratsam, beide Methoden zu trennen und einen Z-Stapel nur für die Bereiche von Interesse zu erstellen, mit Parametern, die mit dem Z-Stapel vor der Erweiterung übereinstimmen. Da bei expandierten Gelen kein Antifading-Eindeckmedium verwendet wird, empfehlen wir, die Aufnahmeparameter entsprechend einzustellen, um das Photobleaching zu minimieren (schnelle Scangeschwindigkeit, niedrige Laserintensitäten).
Erfolgreiche Expansionsexperimente führen typischerweise zu einem Expansionsfaktor von 4 bis 5, was einer volumetrischen Erhöhung zwischen 64x und 125x entspricht. Da die gleiche Anzahl an Fluorophormolekülen nach der Expansion über ein viel größeres Volumen verteilt wird, ist ein großer Nachteil von ExM eine unvermeidliche Verringerung der Signalintensität. Um das Fluoreszenzsignal nach der Expansion beobachten zu können, kann die Verstärkung und/oder die Intensität des Anregungslasers je nach Fluorophor stark erhöht werden. Die Lochblende kann ebenfalls moduliert werden. Neben diesem rein physikalischen Verdünnungseffekt auf das Fluoreszenzsignal gibt es noch weitere mögliche Ursachen für Signalverluste.
Nach der Markierung, Fixierung und Biokonjugation der molekularen Anker zielt die Gelierung (Protokollschritt 7.2) darauf ab, die Probe in ein Polyelektrolyt-Hydrogel einzukapseln. Solche Polymere bestehen aus drei Monomeren: Acrylamid, Bis-Acrylamid und Natriumacrylat. Letzteres verleiht dem Gel aufgrund seiner ionischen Natur seine expandierende Eigenschaft; Tatsächlich kann dieses Polymer ein Vielfaches seines Gewichts an Wasser absorbieren, indem es Natriumionen gegen Wassermoleküle austauscht29. Die Vernetzung des Gels ist eine radikalische Reaktion, die durch TMEDA und APS katalysiert wird. Bemerkenswert ist, dass eine Ursache für die weitere Signalreduktion der partielle Abbau von organischen Fluorophoren während dieser radikalinduzierten Polymerisation ist. Verschiedene Farbstoffe sind in unterschiedlichem Maße betroffen; Zum Beispiel werden Cyaninfarbstoffe während der Expansion fast vollständig zerstört und weisen eine schlechte Signalretention von 0,3-0,4 % auf30,31, während Alexa Fluor 488-Farbstoffe eine Beibehaltung von ~57% der Signalintensität nach ExM4 ermöglichen. Für eine Zusammenfassung der Signalretention verschiedener organischer Fluorophore und fluoreszierender Proteine siehe die Übersichtsarbeit von Wen et al.31.
Darüber hinaus kann die Biomolekülretention auch durch bestimmte Schritte des Protokolls beeinflusst werden. Zum Beispiel können Glykanketten, die sich auf Proteinfragmenten befinden, die nicht an das Hydrogel verankert sind, während der Verdauung ausgewaschen werden, was die Signalintensität weiter verringert (Abbildung 4). Bei der Verdauung wird mindestens ein Detergens32, wie Triton X-100, mit oder ohne Proteinase K 4,5,6,31 verwendet, um Membranen und Proteinstrukturen wie das Zytoskelett zu stören. Die Aufschlusszeit und die Zusammensetzung des Aufschlusspuffers können angepasst werden, um bestimmte Fluorophore und Biomoleküle bestmöglich zu erhalten. Die Aufschlussparameter (Protokollschritt 7.3) sollten daher für die Art der untersuchten Probe in Bezug auf Zusammensetzung, Konzentration und Dauer optimiert werden, um das beste Gleichgewicht zwischen Homogenisierung, um eine isotrope Expansion und Retention von Biomolekülen und Sonden zu gewährleisten, zu erreichen. In bestimmten Fällen kann die Art des Verdauungspuffers für mildere Bedingungen vollständig modifiziert werden, wie z. B. bei Ultrastructure-ExM, wo die Proteine der Probe in SDS bei 95 °C vor der Gelexpansion denaturiert werden26,27.
Wenn möglich, kann der untere Signalnachschub ExM einfach durch Anpassen der Leistung der Anregungsquelle und/oder der Verstärkung der Detektorkamera während der Aufnahme der Bilddaten angesprochen werden, ohne dass spezielle Sonden erforderlich sind. In Fällen, in denen das Signal-Rausch-Verhältnis nach der ExM unzureichend wäre, wurden von verschiedenen Laboratorien zusätzliche Signalerhaltungs- und/oder Verstärkungsstrategien entwickelt, beispielsweise unter Verwendung trifunktionaler Moleküle, die eine direkte Verankerung der Fluorophore an der Polymermatrixermöglichen 6,14. Als alternative Lösung zur Bewältigung dieser Herausforderungen kann die Färbung der Probe nach der Expansion erfolgen, da das Gel ausreichend durchlässig ist. Dies verhindert jedoch die Beobachtung der Probe vor der Expansion. Dieser Ansatz wurde kürzlich für die Immunfärbung33 berichtet, bei der Proben zunächst isotrop expandiert und dann mit Antikörpern gefärbt werden. Die Autoren zeigten, dass dies zu einer verbesserten Fluoreszenzhelligkeit und Epitopzugänglichkeit führen kann, während gleichzeitig Verknüpfungsfehler reduziert werden, die die Lokalisierungsgenauigkeit bei der Kombination von ExM- und Superauflösungstechniken beeinträchtigen.
Alternative Strategien, um die Verringerung der Fluoreszenzintensität zu vereiteln, wurden erforscht. Ein Beispiel ist die Verwendung von polymerbasierten Fluorophoren anstelle von herkömmlichen fluoreszierenden Molekülen. Liu et al. entwickelten fluoreszierende Polymerpunkte (Pdots)34 , die viel heller sind als herkömmliche Fluorophore und ein besseres Signal-Rausch-Verhältnis ermöglichen. Pdots können mit einem Anker und einem Antikörper funktionalisiert werden. In einem anderen Beispiel führte die Click-Expansionsmikroskopie (ClickExM)6 eine universelle Strategie zur Verbesserung der Signalspeicherung mit einer indirekten Biotin-Streptavidin-Strategie ein, bei der ein fluoreszierendes Streptavidin verwendet wird, das einen Anker trägt. Dies ermöglicht das direkte Pfropfen der Fluoreszenzsonden auf den Polymerrahmen. In ähnlicher Weise stützt sich das Molecule Anchorable Gel-enabled Nanoscale In-situ Fluorescence MicroscopY (MAGNIFY)35 stark auf die Verankerung von Biomolekülen, um die Fluoreszenzsignalretention zu erhöhen. Im Gegensatz zu ClickExM, bei dem ein bestimmtes Molekül an das Gel gebunden wird, verwendet MAGNIFY Methacrolein, um eine breite Palette von Biomolekülen mit dem Rahmen des Hydrogels zu vernetzen.
Eine weitere mögliche Einschränkung von ExM in Bezug auf die Datenerfassung am Mikroskop ist die Notwendigkeit, eine expandierte Probe über einen größeren physikalischen Bereich abzubilden, was den Zeitaufwand für die Erfassung der gesamten Probe erheblich erhöht. Da der Expansionsprozess die Probe vergrößert, erfordert das Erreichen einer hochauflösenden Bildgebung die Aufnahme von mehr Bildkacheln, um dieselbe biologische Region abzudecken, was zu längeren Aufnahmezeiten führt. In dieser Hinsicht würde ExM für eine Reihe von Anwendungen nicht als Ersatz für die klassische konfokale Mikroskopie angesehen werden, sondern als ergänzende Technik für eine umfassende und detaillierte Bildgebung.

Abbildung 7: Vergleich der 3D-Bildgebung von preExM und postExM von humanen primären Fibroblasten 533T-Zellen. Die Zellen wurden 24 h lang mit 500 μM ManNAz inkubiert und mit Alexa Fluor 488 Alkin mittels CuAAC (grüne Farbe) markiert. Die Co-Lokalisationsfärbung des Golgi-Apparats (rote Farbe) wurde mit einem Maus-Anti-GM130-Primärantikörper und einem Anti-Maus-Alexa Fluor 546-markierten Sekundärantikörper durchgeführt, und die Zellkerne wurden mit Hoechst 33342 (blaue Farbe) gefärbt. (Links) (A) Z-Stapel und (B,C) 3D-Oberflächendarstellung von Zellen, visualisiert vor ExM. (Rechts) Die Zellen wurden vor der Z-Stack-Akquisition der Verankerung, Gelierung, dem Verdau und der Expansion unterzogen, wie beschrieben (D). (E) Die 3D-Oberflächendarstellung der Signale verdeutlicht die Auflösungssteigerung, die besonders bei der Detektion komplizierter intrazellulärer Substrukturen wertvoll ist. Die 3D-Rekonstruktion und das Oberflächen-Rendering wurden unter Verwendung der Signalintensität erstellt. Maßstabsbalken = 20 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
ExM hat sich zu einer innovativen Technik entwickelt, mit der die Bildgebung im Nanomaßstab erreicht werden kann, ohne die Beugungsgrenze zu überschreiten, und die Visualisierung von Details aus biologischen Proben ermöglicht, die sonst in Standardprotokollen übersehen würden6. Bei Verwendung eines mit ~250 nm beugungsbegrenzten Geräts (wie es bei konfokalen Routinemikroskopen mit grünen Lasern üblich ist) führt ein Expansionsfaktor von 4,5x in der Regel zu einer lateralen Auflösung nahe 60 nm. Dies ist eine sehr signifikante Verbesserung bei der Untersuchung subzellulärer Strukturen wie dem Golgi-Apparat oder Vesikel, die in der 2D-Bildgebung, aber auch durch die Erzeugung von 3D-Bildern aus der Z-Stack-Erfassung genutzt werden können (Abbildung 7). Um die volle Leistungsfähigkeit der Expansionsmikroskopie zu nutzen, kann die erreichte Auflösung durch die Kombination von ExM mit hochauflösenden Mikroskopietechniken wie STED oder STORM weiter verbessert werden, was den Rahmen dieses Artikels sprengen würde.
Das Gebiet der Expansionsmikroskopie erlebt ein sehr schnelles Wachstum und bedeutende Fortschritte, was es zu einem spannenden und dynamischen Forschungsgebiet macht. Boyden und seine Mitarbeiter führten die Expansionsmikroskopie im Jahr 2015 ein und erreichten mit konfokalen Mikroskopen eine Auflösung von etwa 70 nm, indem gelverankerbare Markierungen verwendet wurden, um die Geometrie der Biomoleküle in der Polymermatrix beizubehalten. Nach dem anfänglichen Interesse veranlasste der Bedarf an maßgeschneiderten Oligonukleotid-Antikörper-Konjugaten die wissenschaftliche Gemeinschaft, neue Werkzeuge und Ansätze für diese Methode zu entwickeln. Es wurden verschiedene Strategien entwickelt, um Proteine mit kommerziell erhältlichen Wirkstoffen direkt am Gel zu verankern, die die Verwendung von fluoreszenzmarkierten Antikörpern oder Proteinen ermöglichen, und neuere Entwicklungen haben die Expansionsmikroskopie weiter vereinfacht und ihre Anwendung auf verschiedene Biomoleküle ausgeweitet, ohne dass separate Verankerungsschritte erforderlich sind.
Das Erreichen einer MOE-Markierung mit höherer Auflösung durch ExM ist besonders nützlich, um die räumliche Verteilung von sialylierten Glykanen routinemäßig zu untersuchen, ohne dass hochauflösende Mikroskope erforderlich sind, die teuer und schwer zugänglich sind. Zum Beispiel wurden mit dieser Methode Sialglykane auf der Plasmamembran sichtbar gemacht, was ihre Verteilung in Mikrovilli und anderen zellulären Strukturen aufzeigt6. Die Fähigkeit, intrazelluläre Glykane mit verbesserter Auflösung zu visualisieren, ist umso wichtiger, um ihre Biosynthese, ihren Transport undihr Recycling zu untersuchen 8,13,14,15 und ihre Rolle bei Pathologien wie Stoffwechselerkrankungen oder Krebs zu entschlüsseln. Die Kombination von ExM und bioorthogonaler Chemie bietet eine einzigartige Gelegenheit, ihre Lokalisierung und Beteiligung an zellulären Signalwegen, Organellenfunktionen und Wechselwirkungen, die für die Zellregulation und Krankheitsmechanismen von entscheidender Bedeutung sind, besser zu verstehen. Dies wird zweifellos unser Verständnis der Rolle von Sialinsäuren bei verschiedenen Pathologien in der Zukunft verbessern.
Offenlegungen
Die Autoren haben keine konkurrierenden finanziellen Interessen oder andere Interessenkonflikte.
Danksagungen
Wir danken den TisBio-Einrichtungen und der PLBS-Plattform für die Bereitstellung des technischen Umfelds, das für die Durchführung dieser Arbeit förderlich ist. Diese Arbeit wurde durch Zuschüsse des CNRS und des Ministère de l'Enseignement Supérieur et de la Recherche unterstützt. Wir danken Dr. François Foulquier, Dr. Zoé Durin, Frau Dorothée Vicogne und Frau Céline Schulz für die anregenden Gespräche und die Bereitstellung der Fibroblast 533T-Zelllinie und des primären Antikörpers GM130.
Materialien
| Name | Company | Catalog Number | Comments |
| (+) Sodium L-ascorbate | Sigma Aldrich | 11140 | |
| 12 well cell culture plate | Corning | 3513 | |
| Acrylamide | Sigma Aldrich | A8887 | |
| Acrylic acid N-hydroxysuccinimide ester | Sigma Aldrich | A8060 | |
| Alexa Fluor 488 alkyne | Jena Bioscience | CLK-1277-5 | |
| Alexa Fluor 546 goat anti-mouse IgG | Invitrogen | A11003 | |
| Amonium persulfate | Sigma Aldrich | 9913 | |
| Bis-Acrylamide | Sigma Aldrich | 146072 | |
| BSA | Sigma Aldrich | A7906 | |
| BTTAA | Jena Bioscience | CLK-067-100 | |
| Centrifugation tube 2 mL | EPPENDORF | 30120094 | |
| Chloroquine diphosphate salt | Sigma Aldrich | C6628 | |
| Conical tube 15 mL | Falcon | 352097 | |
| cover slips 12 mm #1 | epredia | CB00120RA120MNZ0 | |
| cover slips 32 mm #1 | epredia | CB00320RA140MNZ0 | |
| CuSO4 | Sigma Aldrich | 209198 | |
| DMEM high glucose medium | Dutscher | L0104-500 | |
| Dulbecco's Phosphate Buffered Saline (PBS) | Dutscher | L0615-500 | |
| Fetal Bovine Serum | biowest | S1810-500 | |
| Fibroblast 533T | - | - | Collected from healthy individual |
| FIJI ImageJ 2.9.0 | - | - | |
| Gelatin | Bio-RAD | 170-6537 | |
| Guanidine HCl | Sigma Aldrich | 50950 | |
| HeLa cells | ATCC | CCL-2 | |
| Hoechst 33342 | Sigma Aldrich | 14533 | |
| Imaris 10.2 | - | - | |
| K2HPO4 | Euromedex | PB0447-B | Anhydrous |
| LSM 780 Confocal Microscopy | Zeiss | - | |
| MCF7 | ATCC | HTB-22 | |
| N-acetylmannosamine (ManNAc) | BIOSYNTH | MA05269 | |
| NaCl | Carlo Erba | 479687 | |
| N-azidoacetylmannosamine (ManNAz) | BIOSYNTH | MA46002 | |
| Objectif "Plan-Apochromat" 63x/1,4 Oil DIC M27 | Zeiss | 420782-9900-799 | |
| Phosphate Buffered Saline (PBS) 10x | Euromedex | ET330 | |
| Proteinase K | Sigma Aldrich | P2308 | from Tritirachium album |
| purified mouse GM130 antibody | BD Bioscience | 610822 | 50 µg |
| Sodium acrylate | Sigma Aldrich | 408220 | |
| T75 Flask | Corning | 430641 | |
| TEMED | Sigma Aldrich | T9281 | |
| tris Acetate EDTA (TAE) 10x | Euromedex | EU0202-B | |
| Triton X-100 | Sigma Aldrich | X-100 | |
| Trypan Blue | Dutscher | 702630 | |
| Trypsine-EDTA 1x | Dutscher | L0930-100 |
Referenzen
- De Souza, N. Light microscopy at the limit. Nat Cell Biol. 11 (Suppl 1), S22(2009).
- Tam, J., Merino, D. Stochastic optical reconstruction microscopy (STORM) in comparison with stimulated emission depletion (STED) and other imaging methods. J Neurochem. 135, 643-658 (2015).
- Jeong, S., Widengren, J., Lee, J. C. Fluorescent probes for STED optical nanoscopy. Nanomaterials. 12 (1), 21(2021).
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543548(2015).
- Kang, S., et al. Expansion microscopy with a thermally adjustable expansion factor using thermoresponsive biospecimen-hydrogel hybrids. ACS Appl Mater Interfaces. 13 (24), 2896228974(2021).
- Sun, D., et al. Click-ExM enables expansion microscopy for all biomolecules. Nat Methods. 18 (1), 107113(2020).
- Dube, D., Bertozzi, C. Metabolic oligosaccharide engineering as a tool for glycobiology. Curr Opin Chem Biol. 7 (5), 616625(2003).
- Scache, J., et al. Switching azide and alkyne tags on bioorthogonal reporters in metabolic labeling of sialylated glycoconjugates: a comparative study. Sci Rep. 12 (1), 22129(2022).
- Holst, S., Wuhrer, M., Rombouts, Y. Glycosylation characteristics of colorectal cancer. Adv Cancer Res. 126, 203256(2015).
- Boyaval, F., et al. N-Glycomic signature of stage II colorectal cancer and its association with the tumor microenvironment. Mol Cell Proteomics. 20, 100057(2021).
- Ferreira, J. A., et al. Protein glycosylation in gastric and colorectal cancers: Toward cancer detection and targeted therapeutics. Cancer Lett. 387, 32-45 (2017).
- Jennings, M. P., Day, C. J., Atack, J. M. How bacteria utilize sialic acid during interactions with the host: snip, snatch, dispatch, match and attach. Microbiology. 168 (3), 001157(2022).
- Gilormini, P. A., et al. A sequential bioorthogonal dual strategy: ManNAl and SiaNAl as distinct tools to unravel sialic acid metabolic pathways. Chem Commun. 52 (11), 2318-2321 (2016).
- Vanbeselaere, J., et al. Alkynyl monosaccharide analogues as a tool for evaluating Golgi glycosylation efficiency: application to Congenital Disorders of Glycosylation (CDG). Chem Commun. 49 (96), 11293-11295 (2013).
- Gilormini, P. A., et al. Chemical glycomics enrichment: imaging the recycling of sialic acid in living cells. J Inherit Metab Dis. 4 (3), 515523(2018).
- Rigolot, V., Biot, C., Lion, C. To view your biomolecule, click inside the cell. Angew Chem. 60 (43), 2308423105(2021).
- Qin, W., et al. Artificial cysteine S-glycosylation induced by per-O-acetylated unnatural monosaccharides during metabolic Glycan Labeling. Angew Chem. 130 (7), 1835-1838 (2018).
- Qin, K., Zhang, H., Zhao, Z., Chen, X. Protein S-glyco-modification through an elimination-addition mechanism. J Am Chem Soc. 142 (20), 9382-9388 (2020).
- Rostovtsev, V. V., Green, L. G., Fokin, V. V., Sharpless, K. B. A. Stepwise Huisgen cycloaddition process: Copper(I)-catalyzed regioselective "ligation" of azides and terminal alkynes. Angew Chem. 41 (14), 25962599(2002).
- Agard, N. J., Prescher, J. A., Bertozzi, C. R. A strain-promoted [3 + 2] azide−alkyne cycloaddition for covalent modification of biomolecules in living systems. J Am Chem Soc. 126 (46), 15046-15047 (2004).
- Mbua, N. E., et al. Strain-promoted alkyne-azide cycloadditions (SPAAC) reveal new features of glycoconjugate biosynthesis. ChemBioChem. 12 (12), 19121921(2011).
- Blackman, M. L., Royzen, M., Fox, J. M. Tetrazine ligation: Fast bioconjugation based on inverse-electron-demand Diels-Alder reactivity. J Am Chem Soc. 130 (41), 1351813519(2008).
- Devaraj, N. K., Weissleder, R., Hilderbrand, S. A. Tetrazine-based cycloadditions: Application to pretargeted live cell imaging. Bioconjug Chem. 19 (12), 22972299(2008).
- Bird, R. E., et al. Bioorthogonal chemistry and its applications. Bioconjug Chem. 32 (12), 2457-2479 (2021).
- Slade, P. G., et al. Identifying the CHO secretome using mucin-type O-linked glycosylation and click-chemistry. J Proteome Res. 11 (12), 61756186(2012).
- Gambarotto, D., Hamel, V., Guichard, P. Ultrastructure expansion microscopy (U-ExM). Methods Cell Biol. 161, 5781(2021).
- Gambarotto, D., et al. Imaging cellular ultrastructures using expansion microscopy (U-ExM). Nat. Methods. 16, 71-74 (2019).
- Presolski, S. I., Hong, V. P., Finn, M. Copper-catalyzed azide-alkyne click chemistry for bioconjugation. Curr Protoc Chem Biol. 3 (4), 153-162 (2011).
- Santiago, F., et al. Synthesis and swelling behaviour of poly(sodium acrylate)/sepiolite superabsorbent composites and nanocomposites. Polym Int. 55 (8), 843848(2006).
- Wen, G., et al. Evaluation of direct grafting strategies via trivalent anchoring for enabling lipid membrane and cytoskeleton staining in expansion microscopy. ACS Nano. 14 (7), 78607867(2020).
- Wen, G., et al. Current progress in expansion microscopy: Chemical strategies and applications. Chem Rev. 123 (6), 32993323(2023).
- Drelich, L., et al. Toward high spatially resolved proteomics using expansion microscopy. Anal Chem. 93 (36), 12195-12203 (2021).
- Zwettler, F. U., et al. Molecular resolution imaging by post-labeling expansion single-molecule localization microscopy (Ex-SMLM). Nat Commun. 11 (1), 3388(2020).
- Liu, J., et al. Expansion microscopy with multifunctional polymer dots. Adv Mater. 33 (25), 2007854(2021).
- Klimas, A., et al. Magnify is a universal molecular anchoring strategy for expansion microscopy. Nat Biotechnol. 41 (6), 858869(2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten