Method Article
フィーダーフリー条件下で培養したヒト多能性幹細胞におけるCRISPR-Cas9媒介遺伝子欠失
要約
提示された方法は、高効率のレンチウイルス媒介遺伝子送達システムを使用して、L2HGDH遺伝子を標的とするsgRNAを安定的に発現するヒト胚性幹細胞(hESC)株H9におけるCRISPR媒介遺伝子ノックアウトの生成を説明するものです。
要約
ゲノム編集のためのCRISPR-Cas9システムは、幹細胞を含む哺乳類細胞の遺伝子機能研究に革命をもたらしました。しかし、この手法を特に多能性幹細胞に実用化するには、時間や労力がかかる、編集効率が低いなどの課題があります。ここでは、L2HGDH遺伝子のsgRNAを安定的に発現するヒト胚性幹細胞(hESC)系統において、高効率で安定したレンチウイルス媒介遺伝子送達システムを用いて、CRISPR媒介遺伝子ノックアウトを作製する方法について述べる。L2HGDH遺伝子のエクソン1を標的とするsgRNAを化学合成し、高効率な単一ベクター系でCas9とsgRNAの構成的発現を組み合わせたlentiCRISPR v2-puroベクターにクローニングすることで、hESC感染に対するレンチウイルス力価の高値とピューロマイシンによる安定選択を実現しました。ピューロマイシン選択細胞をさらに増殖させ、限定希釈法を用いて単一細胞クローンを得た。単一クローンを増殖させ、ウェスタンブロット分析を用いてL2HGDH発現を100%減少させることで確認されたように、L2HGDH遺伝子のホモ接合型ノックアウトクローンをいくつか取得しました。さらに、MSBSP-PCRを用いて、CRISPR変異部位を、選択したホモ接合クローンにおけるCas9のPAM認識配列の上流にマッピングした。正確な挿入/欠失を解析するためにサンガーシーケンシングを実施し、クローンの機能特性評価を行いました。この方法は、以前に報告された非ウイルス性遺伝子導入法と比較して、ホモ接合性欠失の割合が有意に高かった。このレポートはL2HGDH遺伝子に焦点を当てていますが、この堅牢で費用対効果の高いアプローチは、遺伝子機能研究のために多能性幹細胞の他の遺伝子のホモ接合ノックアウトを作成するために使用できます。
概要
ヒト胚性幹細胞(hESC)と人工多能性幹細胞(iPSC)は、体内のあらゆる細胞に分化する可能性を秘めた幹細胞です。これらの細胞は、ヒトの発生を研究するだけでなく、さまざまな疾患の根底にあるメカニズムを理解するための貴重なツールとして機能し、再生医療、疾患モデリング、および創薬に大きな期待を寄せています。このような研究には、特定の遺伝子が生物の発生、機能、および調節にどのように寄与しているかを調査することが含まれます1,2。
遺伝子の機能を解読するために、遺伝子ノックアウトや過剰発現などの遺伝子操作やゲノム編集など、さまざまな手法やアプローチが用いられています。その中でも、CRISPR-Cas9技術は、遺伝子ノックアウトおよび遺伝子編集研究のための最も効率的なアプローチとして浮上しています1,2,3。CRISPR-Cas9システムは、特定の目的のDNA配列を同定して結合するために特別に設計されたシングルガイドRNA(sgRNA)分子を利用して機能します。分子ガイドとして機能するsgRNAは、Cas9酵素を修飾が必要なゲノム内の正確な位置に導きます。結合すると、Cas9は指定された部位でDNAの二本鎖切断を開始します。DNAの切断に続いて、細胞の固有の修復メカニズムが活性化されます。これらには、非相同末端結合(NHEJ)と相同組換え修復(HDR)の2つの主要な修復経路が含まれます。NHEJはしばしば、切断部位に挿入または欠失(インデル)を引き起こし、遺伝子の破壊または不活性化につながります。逆に、HDRは、切断部位に新しいDNA配列を挿入することを可能にし、標的とした遺伝子改変の導入を容易にする4。
多能性幹細胞における遺伝子欠失の重要性を考慮して、hESC/iPSCにおけるCRISPR-Cas9を介した遺伝子ノックアウトに関するいくつかのプロトコルが発表されています。しかし、これらのプロトコルの多くは、非常に時間がかかり、労働集約的である、非ウイルス性遺伝子送達法の使用による効率が低いなど、大きな制限に直面しています5。これらの課題は、hESC/iPS細胞ではさらに顕著であり、これらの細胞は他の細胞タイプと比較して編集効率が低いことが知られています5。これらの制限の一部は、Cas9およびsgRNAを含むプラスミド導入の効率を高めることで対処できます。これは、レンチウイルスベクターシステムを使用して成功裏に達成でき、遺伝子編集の結果を大幅に改善できます。レンチウイルスのパッケージングプロトコルは確立されており、わかりやすいため、経験の浅い研究者でも研究室で簡単に採用できます。レンチウイルスは、hESCやiPS細胞など、さまざまな細胞種で高い感染効率を示します。したがって、Cas9-sgRNA発現にレンチウイルスシステムを利用することは、遺伝子機能研究のためのhESC/iPS細胞の日常的な遺伝子編集実験に最適です。
ここでは、従来のプロトコルよりも比較的短い時間で、hESCの高効率CRISPR-Cas9ベースの遺伝子欠失のためのシンプルでわかりやすい方法を提供します(図1)。Cas9とsgRNAを構成的に発現するレンチウイルスベクターが使用されてきましたが、Cas9の発現を制御するために、薬物誘導性のCas9発現に簡単に置き換えることができます。
プロトコル
本研究で使用した遺伝子配列、試薬、および機器の詳細は、 資料表に記載されています。
1. シングルガイドRNA(sgRNA)の設計、クローニング、レンチウイルスベクターの作製
注: L2HGDH 遺伝子のエクソン1を標的とする2つの異なるsgRNA配列は、どちらもQiuら6から適応され、それぞれ配列1および2のAGGおよびTGGのPAM部位が使用される。両方のsgRNAの長さは20 bpで、末端を修飾して、前の報告6で説明したように、制限酵素Bsmb1を標的ベクター(LentiCRISPRv2)にクローニングするためのリンカー配列を追加しました。リンカー配列は、クローニングを目的としたsgRNAの設計中に追加されました。
- アニール化学合成されたsgRNA6およびクローンを、以前に報告された7と同様に最適化されたプロトコルを使用してBsmb1シングルカットレンチウイルスベクター、LentiCRISPRv2に作製します。
- 標準化および最適化されたプロトコール7を使用して、レンチウイルスのパッケージングと濃縮を進めます。
- レンチウイルスの作製には、DMEM、10% FBS、1x ペニシリン/ストレプトマイシン(P/S)を使用してHEK293T細胞(1 × 105 細胞/cm2)を播種し、湿度の高い条件下で5%CO2 の雰囲気下、37°Cの組織培養インキュベーターで一晩インキュベートします。
- 90%がコンフルエントな場合、エントリーベクター(エンプティバックボーンまたはsgRNA発現プラスミド)およびパッケージングプラスミドとHEK293T細胞を共トランスフェクションし、前述のように低コストのカチオン性ポリマーPEIを使用します7。
- トランスフェクション後48時間および72時間でレンチウイルス上清(LVS)粒子を含む馴化培地を回収し、ショ糖クッションを使用して超遠心分離を進め、チューブを1,25,000 x g で4°Cで2時間回転させます。
- LVS粒子濃度をPBS中の元の容量の少なくとも200倍まで進め、分量し、使用するまで-80°Cで保存します。
- レンチウイルス粒子の力価を測定するには、製造元の指示に従ってqPCR レンチウイルス力価キットを使用します( 材料の表を参照)。
2. レンチウイルス感染と単一細胞クローン増殖
- LVS粒子による感染の場合、500 μLの培地に完全なマトリゲル(1:50)コーティングされたP24ウェルプレートに1 × 105 細胞/0.5 mLのhESC培養培地(基礎培地+ P / S + 10 μMのRock Inhibitor)を1個10個入れたhESC(H9)細胞懸濁液をシードし、細胞が接着するまで一晩インキュベートします。LVSに感染しないが、ピューロマイシンで処理された追加のウェルを播種して、非感染コントロールとして機能します。
注:細胞カウントは、手動血球計算盤または自動セルカウンターを使用して実行できます。 - 翌日、感染の多重度(MOI)が10の細胞に8μg/mLのポリブレンを感染させ、37°Cで8時間インキュベートした後、培地を新鮮なhESC培地+ Rock InhibitorなしのP/Sで交換し、細胞が90%コンフルエントになるまで培養を続けます。
- 細胞が90%の密度(通常は感染後48〜72時間)に達したときに、培地に0.8 μg/mL濃度のピューロマイシンを補充してピューロマイシンの選択を開始し、非感染細胞(対照群)ですべての細胞が死ぬまで選択を続けます。
- 選択が完了したら(通常は4〜6日)、安定細胞を分割し(1:4)、凍結保存とさらなる分析のために増殖します。
- L2HGDH-sgRNA-1を発現する細胞を用いて、シングルセル選択とクローン増殖を行う6.
- この目的のために、500細胞/10 mLの完全増殖培地に相当する細胞懸濁液を調製し、この懸濁液100 μLを96ウェルプレートの各ウェルに播種します。
- 細胞を3日間放置してから観察します。
- シングルクローンを生成するウェルをマークし、コロニーがさらに拡大、凍結保存、および分析するのに十分なサイズ(通常は2週間)に達するまで、1日おきに培地を交換します。
3. gDNA抽出、MS-BSP PCR、サンガーシーケンシング
- 製造元の指示に従って、ゲノムDNA単離キットを使用して細胞からgDNAを単離します( 材料の表を参照)。
- Mutation Sites Based Specific Primers(MS-BSP)解析8を使用して、PAM認識配列の上流の変異部位のマッピングを進めます。
- このため、偏りのない右プライマーL2H-UMSBSP-R1は、エクソン1の外側の領域を増幅して任意のターゲットを増幅するように設計されています。
- sgRNAと同一の配列を持つバイアスレフトプライマーL2H-BMSBSP-F1を設計し、PAM認識配列に近いターゲット配列を増幅します。
注:PCRの非常に高いストリンジェントな条件下では、生成物は変異していないクローンのゲル上で観察されます。PAM認識配列の上流付近に変異を示すCRISPRノックアウトクローンでは、生成物は観察されません。 - 一塩基対変異をマッピングするために、PCRは L2HGDH の全エクソン1にまたがる468 bp配列を増幅し、サンガーシーケンシングに続いてclustalw8を用いたマルチシーケンスアライメント解析を行います。
4. hESC分化と胚様体(EB)形成アッセイ
- デュアルSMAD阻害法9,10,11を使用して以前に説明したように確立されたプロトコルに従って、神経外胚葉の運命に向けたhESC(H9)のコントロールおよび異なるCRISPRクローンの指向性分化を進めます。
- 90%のコンフルエント度で、細胞をLDN193189(200 nM)およびSB431542(10 μM)で最初の24時間100% KSR培地で処理し、その後XAV939(2 μM)をさらに2日間添加します。
- 3日後、N2培地(DMEM/F12に1x N2サプリメント、1x P/S、1x P/S、1%(v/v)10 mM MEM、および0.1%(v/v)2-メルカプトエタノール(75%、50%、25%)を添加したノックアウトDMEM)の割合を減らします。)を 8 日間かけて 100% N2 培地に注入します。
- 12日目の開始時に、PAX6を神経外胚葉マーカーとして使用して免疫染色のために細胞を固定します。
- 中胚葉および内胚葉の運命決定研究では、以前の出版物12,13で説明されている低分子CHIR99021ベースのアプローチを採用します。
- このためには、細胞が70%のコンフルエント度に達したら、Definitive Endoderm(DE)培地で3 μMのCHIR99021で24時間処理した後、Brachuaryを中胚葉特異的マーカーとして使用して免疫染色を固定します。
- 内胚葉期では、FOXA2を使用して免疫染色のために固定する前に、CHIR99021を添加せずにDE培地のみでさらに24時間細胞を培養します。
- EB形成アッセイでは、顕微鏡写真を撮る前に、マトリゲルを使用せずに細胞を懸濁条件下で24時間培養せずに、低接着細胞表面に細胞を播種します。
5. ウェスタンブロット解析
- PBSを使用して細胞を2回洗浄し、1% SDSおよび1xプロテアーゼおよびホスファターゼ阻害剤カクテルを含む1x RIPAバッファーを使用して溶解します。
- ライセートを16,000 x g で4°Cで10分間遠心分離し、その後上清を回収します。
- 製造元の指示に従って、BCAタンパク質アッセイキットを使用して全細胞タンパク質を定量します。4×ローディング色素サンプルバッファーを使用して、サンプルを2 μg/μLに調整します。
- タンパク質サンプルを70°Cで10分間変性させ、各サンプルを等量ロードし、4%-12%グラジエントSDS-PAGEゲル7 を使用して分離し、続いて100Vの定電圧で4°Cで1時間PVDFメンブレンに移します。
- 5%脱脂乳を使用してメンブレンをブロックし、一次抗体希釈液中で4°Cで一晩、回転しながらインキュベートします。
- 次に、PBSTバッファーを使用してメンブレンを5回洗浄し、HRP標識した適切な二次抗体で室温で1時間インキュベートします。
- メンブレンをPBST 5xで再度洗浄し、化学発光基質とインキュベートし、X線フィルムを使用して現像します。
6. 免疫染色
- P4ウェルプレートに細胞を播種し、固定前に少なくとも24時間インキュベートして、細胞が表面に適切に付着できるようにします。
- 細胞をPBSで3回洗浄して、死んだ細胞と培地成分を除去し、続いて4%PFAを使用して室温で15分間固定します。
- 0.3% triton X-100を使用して細胞を透過処理し、続いてPBS中の2% BSAを室温で1時間使用して非特異的結合をブロッキングします。
- サンプルを一次抗体(OCT4、NANOG、SOX2、KI67、PAX6、Brachuary、FOXA2)と1% BSAで希釈し、4°Cで一晩インキュベートします。
- 細胞をPBSで3回洗浄し、適切な二次抗体(ヤギ抗マウス488、ヤギ抗ウサギ488、ヤギ抗マウス546、ヤギ抗ウサギ546、ヤギ抗ウサギ546)と1%BSAで希釈し、室温で1時間インキュベートします。
- 最後に、サンプルをPBS 3xで洗浄し、DAPIで対比染色し、蛍光顕微鏡を使用して画像化します。
結果
lentiCRISPRv2ピューロにおけるL2HGDH sgRNAのクローニング
lentiCRISPRv2ピューロベクターを市販で入手し( 「Table of Materials」を参照)、BsmB1で消化した結果、1.8 Kbのスタッファーフラグメントが放出されました。 図2Aに示すように、ベクターの完全な消化が観察されました。各コンストラクトについて、リバースsgRNA配列をプライマーとして、およびベクター配列内からのフォワードプライマー(U6-459F)を用いて、6つのクローンをインサートの有無についてスクリーニングした。このアプローチを使用すると、 図2Bに示すように、インサートを有するクローンのみが288 bpのPCR産物を生じ、sgRNAの逆配列を含まない陰性クローンはPCR産物を得られませんでした。
H9感染、安定選択、ウェスタンブロット
hESCは、 L2HGDH 遺伝子のエクソン1を標的とする2つの異なるsgRNAを発現するレンチウイルス粒子に感染し、ピューロマイシンで選択されました。ウェスタンブロット解析は、安定して共発現するsgRNA細胞とCas9細胞の不均一集団から実施され、使用された2つのsgRNAのうちどちらが遺伝子欠失の誘導においてより効率的であるかを立証しました。 図3Aに示すように、 L2HGDH-sgRNA-1は、不均一な細胞集団におけるL2HGDHの発現を減少させるのに、他の配列よりも効率的でした。
クローン選択、増殖、タンパク質解析
L2HGDH-sgRNA-1を発現する細胞について、シングルセル選択とクローン増殖を行った。単一のクローンを産出したウェルをマークし、コロニーがさらに拡大し、凍結保存し、分析するのに十分なサイズ(通常は2週間)に達するまで成長させました(図3B)。ウェスタンブロット分析を使用して合計22個のクローンを解析したところ、L2HGDHに対して5つのホモ接合型ノックアウト(23%)が得られました。この結果は、レンチビラール媒介遺伝子導入を用いた遺伝子ノックアウトにより、1つのカセットからsgRNAとCas9の両方が安定的に共発現する高い効率性を示しています。図4は、5つのホモ接合型ノックアウトクローンとコントロール細胞のウェスタンブロット解析を示しており、選択したクローンでは、コントロールと比較して発現が100%減少していることが示されています。
DNAレベルでの解析
Cas9のPAM認識配列の上流にあるCRISPR変異部位をマッピングするために、コントロール細胞と5つの異なるクローンのゲノムDNAを用いて、Mutation Sites Based Specific Primers Polymerase Chain Reaction(MS-BSP PCR)を行いました。 図5に示すように、2つのクローン(A5およびB4)はPAM認識配列のすぐ近くに変異を持っていますが、他の3つのクローン(A1、A9、およびB12)は、変異がPAM部位にあまり近くなく、 L2HGDH 遺伝子のエクソン1内の20bp sgRNA配列のさらに上流にある可能性があることを示しました。
DNAの正確な突然変異を見つけるために、コントロール細胞(ベクターのみに感染したH9)からの精製PCR産物、および3つの異なるCRISPRクローン、A5、A1、およびB4をサンガーシーケンシングにかけ、PAMサイト8の上流にマルチプルアライメントツールclustalwを使用して突然変異部位をマッピングした。その結果、PAM部位の上流のA5クローンに欠失変異が明らかになりました。他の2つのクローン、A1およびB4は、 図6で強調表示されているように、PAM部位の上流に挿入変異を示しました。
クローンの機能解析
hESCでCRISPRノックアウト細胞株を作製したら、次のステップは、その多能性と分化能を確認することです。そのために、A1、A5、B4の3種類のクローンについて異なる多能性マーカーを試験し、その結果を対照細胞(ベクターのみの感染H9)細胞と比較しました。その結果、OCT4、NANOG、およびSOX2マーカーの免疫染色によって決定されたノックアウト細胞株の多能性電位に変化は見られませんでした。次に、コントロール細胞株とノックアウト細胞株を細胞増殖マーカーKI67についても染色しましたが、これもコントロールと比較してノックアウト細胞株に変化が見られませんでした。これらの結果は、CRISPRノックアウトがこれらの細胞の自己再生特性に影響を与えなかったことを示しています。また、EBやコロニー形成も影響を受けませんでした(図7)。当研究室で確立した in vitro 法を用いて、PAX6(神経外胚葉マーカー)、Brachyury(中胚葉マーカー)、FOXA2(内胚葉マーカー)の免疫染色により、すべてのクローンを3つの生殖細胞に分化させることに成功しました。ノックアウト細胞株の分化能に変化は対照細胞と比較して観察されず、ノックアウト細胞が分化能を保持していることが示されました(図8)。

図1:5つのステップでスタディデザインを示す概略図。 ステップ1:sgRNAの設計とBsmb1消化V2 CRISPRベクターへのクローニング。ステップ2:ポリフェクションを用いたHEK293T細胞への同時トランスフェクションによるsgRNAとパッケージングプラスミドを運ぶエントリーベクターを用いたレンチウイルス粒子の作製と、超遠心分離によるウイルス濃縮。ステップ3:MOIが10〜20のレンチウイルス粒子を使用したhESCの感染と、ピューロマイシンを使用した安定した細胞選択、続いてsgRNAの効率を確認するための不均一な細胞集団の分析。ステップ4:不均一集団で最大のノックダウンを示した細胞からの限定希釈法による単一細胞クローニングおよび単一クローンの選択。ステップ5:単一クローンの拡張とDNAレベルでの解析、タンパク質、およびクローンの幹細胞性と分化の可能性の機能解析。 この図の拡大版を表示するには、ここをクリックしてください。

図2:lentiCRISPR V2.puroベクターにおけるsgRNAのクローニング。 (A)制限酵素BsmB1によるlentiCRISPR V2.puroベクターの消化。矢印は、ベクターの消化時に放出される1.8 Kbのスタッファーフラグメントを示しています。(B)L2HGDH sgRNAコンストラクト1および2の異なるクローンのコロニーPCR分析。 この図の拡大版を表示するには、ここをクリックしてください。

図3:シングルセル選択を用いたウェスタンブロットとクローン増殖。 (A)安定して共発現するsgRNA(L2H-1およびL2H-2)およびCas9およびCas9の不均一集団の2つの異なる生物学的複製(S1およびS2)からのウェスタンブロット分析、およびコントロール(C)H9 hESC。(B) L2HGDH-sgRNA-1を安定的に発現するH9細胞の単一細胞選択とクローン増殖。矢印は、単一のクローンのみを示す96ウェルプレートのウェルを示しています。スケールバー = 100 μM. この図の拡大版を表示するには、ここをクリックしてください。

図4:H9の異なるクローンからのウェスタンブロット分析 (A-C) 22クローンの全細胞ライセートからのウェスタンブロット分析では、L2HGDHに対して5つのホモ接合ノックアウト(23%)が得られました。(D)ウェスタンブロットを用いた異なる生物学的複製実験によるL2HGDHノックアウトの確認。この図の拡大版を表示するには、ここをクリックしてください。
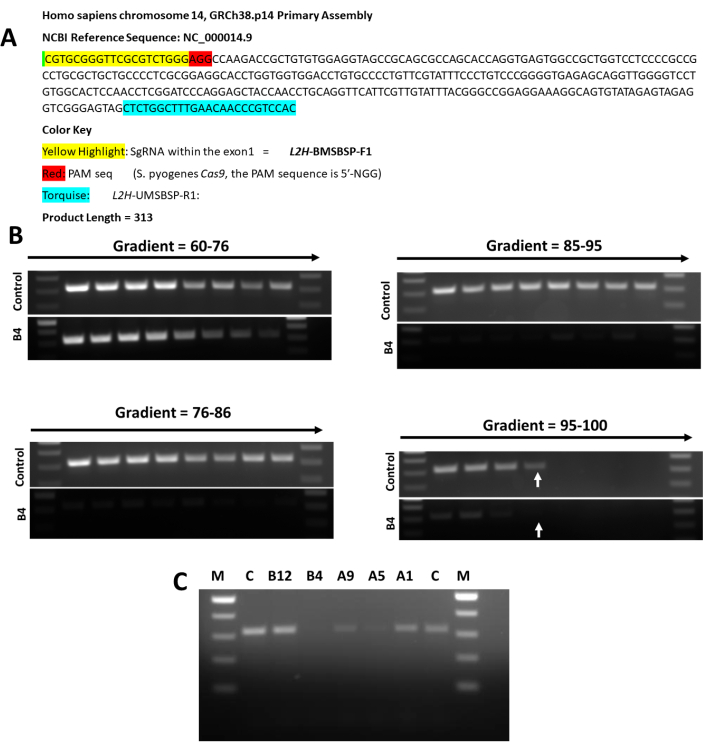
図5:さまざまなH9クローンのMSBSP PCR解析。 (A)MSBSP PCR解析に用いた L2HGDH エクソン1の配列解析。(B)コントロールとH9 V2 L2HGDH KO Clone B4を用いたMSBSP PCR解析のためのアニーリング温度の最適化。96.8 では、コントロールにはバンドが表示されますが、B4 には矢印で示されているようにバンドは表示されません。(C)すべてのホモ接合KOクローンの解析により、エクソン1内の変異部位のマッピングが以下のものになりました。A1 = In/Del が PAM サイトの近くにない。A5 = In/Del PAM サイトに近い。A9 = In/Del PAM サイトにかなり近い。B4 = In/Del PAM サイトに非常に近い B12 = In/Del PAM サイトの近くにありません。この図の拡大版を表示するには、ここをクリックしてください。

図6:サンガーシーケンシングされたPCR産物と参照 L2HGDH エクソン1配列とのアラインメント。 A1、A5、B4由来のサンガーシーケンシングPCR産物、およびコントロール細胞のマルチアライメントを、NCBIデータベースからの L2HGDH の第1エクソン内の参照配列とともに行いました。 この図の拡大版を表示するには、ここをクリックしてください。
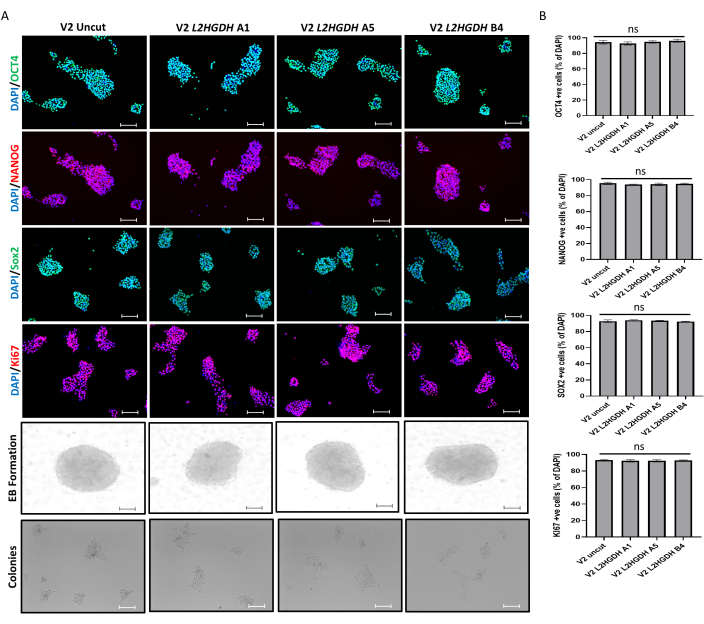
図7:CRISPRノックアウト細胞株の機能特性評価 (A)細胞を免疫染色し、多能性マーカー(OCT4、NANOG、SOX2)、自己複製(KI67)、EB、およびコントロール感染H9 hESCに対するコロニー形成について調べた。スケールバー:100μM.(B)陽性(+ve)発現細胞の割合をDAPI染色と比較して計算した。 この図の拡大版を表示するには、ここをクリックしてください。

図8:CRISPRノックアウト細胞株の分化可能性。 PAX6(神経外胚葉)、Brachyury(中胚葉)、およびFOXA2(内胚葉)のマーカーを使用した、対照感染H9細胞に対するCRISPRノックアウト細胞株の分化能に関する機能的特性評価。スケールバー:100 μMこの 図の拡大版を表示するには、ここをクリックしてください。
| プロトコルステップ | 所要時間 |
| LentiCRISPRv2におけるsgRNAのクローニング | 4日間 |
| レンチウイルスの産生と力価測定 | 5日間 |
| hESC感染、安定した細胞選択、および増殖 | 2週間 |
| シングルセルクローンの選択と増殖 | 2週間 |
| クローンの機能解析 | 2週間 |
表1:プロトコルのステップと必要な推定時間のタイムライン。
ディスカッション
この研究では、CRISPR-Cas9技術を通じて、hESCの遺伝子欠失を高効率かつ費用対効果の高い方法で実現する方法を標準化しました。この方法は、hESCの感染からシングルセルクローンの選択と増殖まで、3〜4週間以内にhESCのL2HGDH遺伝子のホモ接合性欠失を達成することに成功しました(表1)。CRISPR-Cas9を介した遺伝子操作は、ほとんどの細胞で一過性トランスフェクションによって達成できますが、幹細胞ではトランスフェクション効率が低く、細胞毒性が高いため、これは困難になります。さまざまな研究で、回収されたクローンの最大10%が欠失を示していると報告されています。例えば、Maliらは、AAVS1遺伝子座3を標的とした後、ヒト誘導多能性幹細胞におけるゲノム編集効率が2%から4%であると報告した。別の研究では、ナイーブなhESCにおいて、多能性と神経分化に関与する長いノンコーディングRNAであるTSUNAを標的とする編集効率が1.3%〜8.4%であると報告されました14。Ocanaらによるヒト多能性幹細胞のCFTR遺伝子を標的とした後、最大10%の効率が報告されました.15したがって、効率的な遺伝子編集を達成するためにはCas9-gRNAの効率的な導入方法が必要であり、これは特に幹細胞に当てはまります。これらの細胞は、典型的な体細胞や不死化細胞株5と比較して、一過性送達方法に対する感度と耐性のレベルが高いためです。
本研究では、hESCへのsgRNAのレンチウイルス媒介導入を使用することにより、ホモ接合型CRISPRノックアウト細胞株を得るための非常に効率的な方法が報告されています。レンチウイルスベクターを使用してCRISPR-Cas9をhESCに導入することは、化学的または機械的手段による非ウイルスベクタートランスフェクションを使用するよりもはるかに効率的で、遺伝子欠失の達成に時間がかからないことが証明されました(データは示されていません)。これにより、クローン選択と個々のクローンの増殖後のゲノム編集効率が有意に向上しました(~23%)。したがって、レンチウイルスを介したsgRNAおよびCas9のhESCへの送達、およびレンチウイルスの産生のための費用対効果の高いアプローチを組み合わせることにより、本方法は高効率で時間をかけずに済むだけでなく、費用対効果も高い。
ここでは、アプローチを成功させるための推奨事項をいくつか紹介します: (1) Cas9 と sgRNA の両方を発現する単一のレンチウイルスベクターシステムを使用する。(2)Cas9およびsgRNAを発現する同じプラスミド上にピューロマイシンなどの抗生物質選択マーカーが存在すると、細胞のほぼ100%がCas9-sgRNA発現に対して陽性であることが保証されます。これにより、遺伝子編集イベントが成功する可能性が大幅に高まります。(3)ヒトES細胞の感染に有用であるためには、レンチウイルスベクターを濃縮してウイルスを高力価で生成することが不可欠です。使用されるさまざまな技術の中で、最も経済的なアプローチは、大量のLVSを使用し、それらを元の容量の100〜500倍濃縮して、107〜108 IU / mLの範囲の力価を達成することです。(4)プラスミドを用いたHEK293T細胞トランスフェクションの費用対効果が高く効率的な方法として、ウイルスパッケージングのためのレンチウイルスベクタートランスフェクションにPEI(ポリエチレンイミン)を使用することは、一般的に使用されるトランスフェクション試薬リポフェクタミン7と比較して、同等に効率的であり、したがって費用対効果が高い。(5)平均レンチウイルス力価は5×107 IU / mLの範囲である必要があります。(6)これらの細胞の目的の遺伝子のホモ接合性欠失を達成するためには、単一細胞選択によるクローン増殖が必要です。(7)これまでの研究では、ヒト多能性幹細胞におけるCRISPR-Cas9を介した遺伝子編集により、オフターゲット変異が非常に少ないことが示されています16,17、オフターゲット部位を確認することは依然として賢明です。これは、特に臨床的に重要な実験において、オフターゲット効果を最小限に抑えるための効率的なgRNA設計ツールと、編集された細胞株18のゲノムワイドシーケンシングによって達成できる。(8)最後に、選択したクローンの正常な核型をGバンディングや高分解能解析などの方法を用いて検証し、大規模なゲノム変化がないことを確認する必要があります。小核アッセイは、小核形成の頻度を評価するためにも実施でき、これはゲノムの不安定性を示している可能性があります。
この方法の主な制限の1つは、レンチウイルスベクターシステムの使用であり、これによりCas9とsgRNAの安定したゲノム統合が実現し、編集後にそれらの構成的発現が引き起こされますが、これは望ましくありません。将来的には、Cas9遺伝子の発現を駆動する薬物誘導性プロモーター(例えば、Cas9の発現を制御するためのドキシサイクリン処理によるTet-onシステムなど)に構成的プロモーターを置き換えることにより、この方法を改善することができる。
開示事項
著者は、利益相反がないことを宣言します。
謝辞
この研究は、アラブ首長国連邦大学(UAEU)からの助成金 #12M105、助成金 #12R167(Zayed Center for Health Sciences)、21R105(Zayed Bin Sultan Charitable and Humanitarian Foundation(ZCHF))、およびアブダビの先端技術研究評議会(ATRC)の技術プログラム管理の柱であるASPIREから、ASPIREプレシジョンメディシン研究所アブダビ(ASPIREPMRIAD)の助成金番号VRI-20-10を通じて支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 2-MERCAPTOETHANOL | Invitrogen | 31350010 | |
| 38.5 mL, Sterile + Certified Free Open-Top Thinwall Ultra-Clear Tubes | Beckman Coulter | C14292 | |
| Accutase | Stem cell technologies | 7920 | |
| bFGF Recombinant human | Invitrogen | PHG0261 | |
| Brachyury Rabbit mAb | Abclonal | A5078 | |
| BsmBI-v2 | NEB | R0739S | |
| chir99021 | Tocris | 4423/10 | |
| Corning Matrigel Basement Membrane Matrix, LDEV-free | Corning | 354234 | |
| Cyclopamine | Stem cell technologies | 72074 | |
| DMEM media | Invitrogen | 11995073 | |
| DMEM NUTRIENT MIX F12 | Invitrogen | 11320033 | |
| DPBS w/o: Ca and Mg | PAN Biotech | P04-36500 | |
| Fetal bovie serum | Invitrogen | 10270106 | |
| FoxA2/HNF3β | CST | 8186 | |
| GAPDH (14C10) Rabbit mAb Antibody | CST | 2118S | |
| Gentle Cell Dissociation Reagent | Stem cell technologies | 7174 | |
| HyClone Non Essential Amino Acids (NEAA) 100x Solution | GE healthcare | SH30238.01 | |
| Ki-67 (D3B5) Rabbit mAb | CST | 9129 | |
| KnockOut Serum Replacement | Invitrogen | 10828028 | |
| L GLUTAMINE, 100x | Invitrogen | 2924190090 | |
| L2H-BMSBSP-F1 | Macrogen | CGTGCGGGTTCGCGTCTGGG | |
| L2HGDH Polyclonal antibody | Proteintech | 15707-1-AP | |
| L2HGDH-SgRNA1-F | Macrogen | CACCGCGTGCGG GTTCGCGTCTGGG | |
| L2HGDH-SgRNA1-R | Macrogen | AAACCCCAGACGC GAACCCGCACGC | |
| L2HGDH-SgRNA2-F | Macrogen | CACCGCCCGCGG GCTTTTCGCCGG | |
| L2HGDH-SgRNA2-R | Macrogen | AAACCCGGCGAA AAGCCCGCGGGC | |
| L2H-SeqF1 | Macrogen | GCTAAAGAGCGC GGGTCCTCGG | |
| L2H-SeqR1 | Macrogen | GTGGACGGGTTG TTCAAAGCCAGAG | |
| L2H-UMSBSP-R1 | Macrogen | GTGGACGGGTTG TTCAAAGCCAGAG | |
| LentiCRISPRv2 | Addgene | 52961 | |
| mTesR1 complete media | Stem cell technologies | 85850 | |
| Nanog Antibody | CST | 3580 | |
| NEUROBASAL MEDIUM 1x CTS | Invitrogen | A1371201 | |
| Neuropan 2 Supplement 100x | PAN Biotech | P07-11050 | |
| Neuropan 27 Supplement 50x | PAN Biotech | P07-07200 | |
| Oct-4 Antibody | CST | 2750 | |
| Pax6 (D3A9V) XP Rabbit mAb | CST | 60433 | |
| PENICILLIN STREPTOMYCIN SOL | Invitrogen | 15140122 | |
| pMD2.G | Addgene | 12259 | |
| Polybrene infection reagent | Sigma | TR1003- G | |
| Polyethylenimine, branched | Sigma | 408727 | |
| psPAX2.0 | Addgene | 12260 | |
| Puromycin | Invitrogen | A1113802 | |
| qPCR Lentivirus Titer Kit | Abm | LV900 | |
| Rock inhibitor Y-27632 dihydrochloride | Tocris | 1254 | |
| SB 431542 | Tocris | 1614/10 | |
| Sox2 Antibody | CST | 2748 | |
| Sucrose | Sigma | 57-50-1 | |
| TRYPSIN .05% EDTA | Invitrogen | 25300062 | |
| U6-459F | Macrogen | GAGGGCCTATT TCCCATGATTC | |
| Wizard Genomic DNA Purification Kit | Promega | A1120 | |
| XAV 939 | Tocris | 3748/10 |
参考文献
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jiang, F., Doudna, J. A. CRISPR-Cas9 structures and mechanisms. Annu Rev Biophys. 46 (1), 505-529 (2017).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Zhang, Z., et al. CRISPR/Cas9 genome-editing system in human stem cells: Current status and future prospects. Mol Ther Nucleic Acids. 9, 230-241 (2017).
- Qiu, Z., et al. MYC regulation of D2HGDH and L2HGDH influences the epigenome and epitranscriptome. Cell Chem Biol. 27 (5), 538-550.e537 (2020).
- Sheikh, M. A., Ansari, S. A. Lentiviral mediated delivery of shRNAs to hESCs and NPCs using low-cost cationic polymer polyethylenimine (PEI). J Vis Exp. (183), e63953 (2022).
- Guo, J., et al. A simple and cost-effective method for screening of CRISPR/Cas9-induced homozygous/biallelic mutants. Plant Methods. 14 (1), 40 (2018).
- Ardah, M. T., Parween, S., Varghese, D. S., Emerald, B. S., Ansari, S. A. Saturated fatty acid alters embryonic cortical neurogenesis through modulation of gene expression in neural stem cells. J Nutr Biochem. 62, 230-246 (2018).
- Parween, S., et al. Higher O-GlcNAc levels are associated with defects in progenitor proliferation and premature neuronal differentiation. Front Cell Neurosci. 11, 415 (2017).
- Parween, S., et al. Nutrient-sensitive protein O-GlcNAcylation modulates the transcriptome through epigenetic mechanisms during embryonic neurogenesis. Life Sci Alliance. 5 (8), e202201385 (2022).
- Varghese, D. S., et al. Developmental modeling of hepatogenesis using obese iPSCs-hepatocyte differentiation uncovers pathological features. Cell Death Dis. 13 (7), 670 (2022).
- Varghese, D. S., Alawathugoda, T. T., Ansari, S. A. Fine-tuning of hepatocyte differentiation from human embryonic stem cells: Growth factor. Stem Cells Int. 2019 (5), 5968236 (2019).
- Jacobs, E. Z., et al. CRISPR/Cas9-mediated genome editing in naïve human embryonic stem cells. Sci Rep. 7 (1), 16650 (2017).
- Cuevas-Ocaña, S., et al. A cell-based optimized approach for rapid and efficient gene editing of human pluripotent stem cells. Int J Mol Sci. 24 (3), 12345 (2023).
- Veres, A., et al. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN-targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell. 15 (1), 27-30 (2014).
- Smith, C., Gore, A., Yan, W., Abalde-Atristain, L., Li, Z., He, C. Whole-genome sequencing analysis reveals high specificity of CRISPR/Cas9 and TALEN-based genome editing in human iPSCs. Cell Stem Cell. 15 (1), 12-13 (2014).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target effects in CRISPR/Cas9-mediated genome engineering. Mol Ther Nucleic Acids. 4 (7), e264 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved