Method Article
Deleción génica mediada por CRISPR-Cas9 en células madre pluripotentes humanas cultivadas en condiciones libres de alimentadores
En este artículo
Resumen
El método presentado describe la generación de un knockout génico mediado por CRISPR en la línea H9 de células madre embrionarias humanas (hESC), que expresa de manera estable sgRNAs dirigidos al gen L2HGDH utilizando un sistema de entrega de genes mediado por lentivirales altamente eficiente.
Resumen
El sistema CRISPR-Cas9 para la edición del genoma ha revolucionado los estudios de la función génica en células de mamíferos, incluidas las células madre. Sin embargo, la aplicación práctica de esta técnica, particularmente en células madre pluripotentes, presenta ciertos desafíos, como ser intensivo en tiempo y mano de obra y tener una baja eficiencia de edición. Aquí, describimos la generación de un knockout génico mediado por CRISPR en una línea de células madre embrionarias humanas (hESC) que expresa de manera estable sgRNAs para el gen L2HGDH, utilizando un sistema de entrega de genes mediado por lentivirales altamente eficiente y estable. Los sgRNAs dirigidos al exón 1 del gen L2HGDH se sintetizaron químicamente y se clonaron en el vector lentiCRISPR v2-puro, que combina la expresión constitutiva de sgRNAs con Cas9 en un sistema de vector único altamente eficiente para lograr títulos lentivirales más altos para la infección por hESC y una selección estable utilizando puromicina. Las células seleccionadas con puromicina se expandieron aún más y se obtuvieron clones de células individuales utilizando el método de dilución limitada. Los clones individuales se expandieron y se obtuvieron varios clones homocigotos knockout para el gen L2HGDH, como lo confirmó una reducción del 100% en la expresión de L2HGDH mediante el análisis de Western blot. Además, mediante MSBSP-PCR, se mapeó el sitio de mutación de CRISPR aguas arriba de la secuencia de reconocimiento de PAM de Cas9 en los clones homocigotos seleccionados. Se realizó la secuenciación Sanger para analizar las inserciones/deleciones exactas, y se realizó la caracterización funcional de los clones. Este método produjo un porcentaje significativamente mayor de deleciones homocigóticas en comparación con los métodos de administración de genes no virales informados anteriormente. Aunque este informe se centra en el gen L2HGDH, este enfoque sólido y rentable se puede utilizar para crear knockouts homocigotos para otros genes en células madre pluripotentes para estudios de función génica.
Introducción
Las células madre embrionarias humanas (hESCs) y las células madre pluripotentes inducidas (iPSCs) son células madre con el potencial de diferenciarse en todos los tipos de células del cuerpo. Estas células sirven como herramientas valiosas para estudiar el desarrollo humano, así como para comprender los mecanismos subyacentes de diversas enfermedades, lo que ofrece una gran promesa para la medicina regenerativa, el modelado de enfermedades y el descubrimiento de fármacos. Dichos estudios implican investigar cómo genes específicos contribuyen al desarrollo, funcionamiento y regulación de los organismos 1,2.
Se emplean varias técnicas y enfoques para descifrar la función de los genes, incluida la manipulación genética, como la eliminación o sobreexpresión de genes, y la edición del genoma. Entre ellas, la tecnología CRISPR-Cas9 se ha convertido en el enfoque más eficiente para los estudios de knockout y edición de genes 1,2,3. El sistema CRISPR-Cas9 funciona utilizando una sola molécula de ARN guía (sgRNA) diseñada específicamente para identificar y unirse a una secuencia de ADN particular de interés. Actuando como una guía molecular, el sgRNA dirige la enzima Cas9 a la ubicación precisa en el genoma que requiere modificación. Una vez unido, Cas9 inicia una ruptura de doble cadena en el ADN en el sitio designado. Tras la escisión del ADN, se activan los mecanismos de reparación inherentes a la célula. Estos incluyen dos vías de reparación principales: la unión de extremos no homólogos (NHEJ) y la reparación dirigida por homología (HDR). La NHEJ a menudo da lugar a inserciones o deleciones (indels) en el sitio de ruptura, lo que conduce a la interrupción o inactivación de genes. Por el contrario, el HDR permite la inserción de nuevas secuencias de ADN en el lugar de la ruptura, lo que facilita la introducción de alteraciones genéticas específicas4.
Dada la importancia de las deleciones génicas en las células madre pluripotentes, se han publicado varios protocolos sobre los knockouts génicos mediados por CRISPR-Cas9 en hESCs/iPSCs. Sin embargo, muchos de estos protocolos se enfrentan a limitaciones significativas, como ser extremadamente lentos, laboriosos y tener una baja eficiencia debido al uso de métodos de administración de genes no virales5. Estos desafíos son aún más pronunciados en las hESCs/iPSCs, ya que se sabe que estas células tienen una menor eficiencia de edición en comparación con otros tipos de células5. Algunas de estas limitaciones se pueden abordar aumentando la eficiencia de la entrega de plásmidos que contienen Cas9 y sgRNA. Esto se puede lograr con éxito utilizando un sistema de vectores lentivirales, que puede mejorar significativamente los resultados de la edición de genes. Los protocolos de empaquetado de lentivirus están bien establecidos y son sencillos, lo que permite una fácil adopción en los laboratorios, incluso por parte de investigadores con experiencia limitada. Los lentivirus exhiben una alta eficiencia de infección en varios tipos de células, incluidas las hESC y las iPSC. Por lo tanto, el uso de un sistema lentiviral para la expresión de Cas9-sgRNA es ideal para experimentos rutinarios de edición de genes en hESCs/iPSCs para estudios de función génica.
Aquí, proporcionamos un método simple y directo para deleciones de genes basadas en CRISPR-Cas9 altamente eficientes en hESC en una duración de tiempo comparativamente más corta que los protocolos convencionales (Figura 1). Aunque se ha utilizado un vector lentiviral con expresión constitutiva de Cas9 y sgRNA, podría reemplazarse fácilmente con la expresión de Cas9 inducible por fármacos para una expresión de Cas9 controlable.
Protocolo
Los detalles de las secuencias de genes, los reactivos y el equipo utilizados en este estudio se enumeran en la Tabla de Materiales.
1. Diseño de ARN guía única (sgRNA), clonación y producción de vectores lentivirales
NOTA: Se utilizan dos secuencias diferentes de sgRNA dirigidas al exón 1 del gen L2HGDH , ambas adaptadas de Qiu et al.6con sitios PAM de AGG y TGG para las secuencias 1 y 2, respectivamente. Ambos sgRNAs tenían una longitud de 20 pb, y los extremos se modificaron para añadir secuencias enlazadoras para que la enzima de restricción Bsmb1 se clonara en el vector objetivo (LentiCRISPRv2) como se describe en el informe anterior6. Se añadieron secuencias de enlace durante el diseño de sgRNAs con fines de clonación.
- Los sgRNAs6 sintetizados químicamente por el recocido annal y su clonación en el vector lentiviral de corte único Bsmb1, LentiCRISPRv2, utilizando un protocolo optimizado como se informó anteriormente7.
- Proceda con el empaquetamiento y concentración lentiviral utilizando un protocolo estandarizado y optimizado7.
- Para la producción de lentivirus, se siembran células HEK293T (1 × 105 células/cm2) utilizando DMEM, FBS al 10% y 1x penicilina/estreptomicina (P/S) e incubar durante la noche en una incubadora de cultivo de tejidos a 37 °C en una atmósfera de 5% de CO2 en condiciones húmedas.
- Cuando el 90% confluye, co-transfecta células HEK293T con vector de entrada (columna vertebral vacía o plásmidos que expresan sgRNA) y empaqueta plásmidos utilizando PEI de polímero catiónico de bajo costo como se describió anteriormente7.
- Recoja los medios acondicionados que contienen partículas de sobrenadante lentiviral (LVS) a las 48 h y 72 h después de la transfección y proceda a la ultracentrifugación con cojín de sacarosa y centrifuga los tubos a 1,25,000 x g durante 2 h a 4 °C.
- Proceda con la concentración de partículas del VI al menos 200 veces el volumen original en PBS, alícuota, y almacene a -80 °C hasta su uso.
- Determine el título de las partículas lentivirales utilizando el kit de titulación de lentivirus qPCR siguiendo las instrucciones del fabricante (consulte la tabla de materiales).
2. Infecciones lentivirales y propagación clonal unicelular
- En el caso de infecciones con partículas de LVS, la suspensión celular de semilla hESC (H9) con 1 × 105 células/0,5 mL de medio de cultivo de hESC (medio basal + P/S + 10 μM de inhibidor de roca) en placas de pocillos P24 recubiertas de Matrigel completo (1:50) en 500 μL de medio e incubar durante la noche para permitir que las células se adhieran. Siembre pocillos adicionales que no se infectarían con LVS, pero se tratarían con puromicina para que sirvieran como control no infectado.
NOTA: El recuento de células se puede realizar utilizando un hemocitómetro manual o un contador de células automatizado. - Al día siguiente, infecte las células con una multiplicidad de infección (MOI) de 10 junto con 8 μg/mL de polibreno e incube a 37 °C durante 8 h, seguido de un reemplazo del medio con medio hESC fresco + P/S sin inhibidor de rocas y continúe cultivando hasta que las células sean 90% confluentes.
- Comience la selección de puromicina suplementando el medio con una concentración de 0,8 μg/mL de puromicina cuando las células alcancen el 90% de confluencia, lo que suele ser 48-72 h después de la infección, y continúe la selección hasta que todas las células mueran en las células no infectadas (grupo de control).
- Una vez completada la selección (generalmente de 4 a 6 días), divida las células estables (1:4) y amplíe para la criopreservación y el análisis posterior.
- Realizar la selección de una sola célula y la expansión clonal utilizando células que expresan L2HGDH-sgRNA-16.
- Para ello, prepare una suspensión celular equivalente a 500 células/10 mL de medio de crecimiento completo y siembre 100 μL de esta suspensión en cada pocillo de una placa de 96 pocillos.
- Deje las células intactas durante 3 días y luego observe.
- Marque los pocillos que producen clones individuales y cambie el medio cada dos días hasta que se logre un tamaño suficiente para que las colonias (generalmente 2 semanas) se expandan más, se crioconserven y se analicen.
3. Extracción de ADNg, PCR MS-BSP y secuenciación de Sanger
- Aísle el ADNg de las células utilizando el kit de aislamiento de ADN genómico siguiendo las instrucciones del fabricante (consulte la tabla de materiales).
- Proceda con el mapeo de los sitios de mutación aguas arriba de las secuencias de reconocimiento de PAM utilizando el análisis de cebadores específicos basados en sitios de mutación (MS-BSP)8.
- Para esto, un cebador derecho imparcial L2H-UMSBSP-R1 está diseñado para amplificar la región fuera del Exon1 para amplificar cualquier objetivo.
- Diseñe un cebador izquierdo sesgado L2H-BMSBSP-F1 con una secuencia idéntica al sgRNA para amplificar la secuencia objetivo cerca de las secuencias de reconocimiento de PAM.
NOTA: En condiciones muy estrictas de PCR, el producto se observará en el gel en clones no mutados. No se observaría ningún producto en los clones knockout de CRISPR que exhibieran mutaciones cerca de las secuencias de reconocimiento de PAM. - Con el fin de mapear las mutaciones de un solo par de bases, la PCR amplifica una secuencia de 468 pb que abarca todo el exón 1 de L2HGDH y se somete a una secuenciación de Sanger seguida de un análisis de alineación de secuencias múltiples utilizando clustalw8.
4. Ensayo de diferenciación de hESC y formación de cuerpos embrioides (EB)
- Proceder a la diferenciación dirigida del control y de los diferentes clones CRISPR de hESCs (H9) hacia el destino del neuro-ectodermo siguiendo los protocolos establecidos descritos anteriormente utilizando el método de inhibición dual smad 9,10,11.
- Cuando tengan una confluencia del 90 %, trate las células con LDN193189 (200 nM) y SB431542 (10 μM) durante 24 h iniciales en medios 100% KSR seguidos de la adición de XAV939 (2 μM) durante 2 días adicionales.
- Después de 3 días, reduzca el porcentaje de medios KSR (Knockout DMEM suplementado con 15% (v/v) KSR, 1% (v/v) L-glutamina, 1% (v/v) P/S, 1% (v/v) 10 mM MEM y 0,1% (v/v) 2-mercaptoetanol (75%, 50%, 25%) combinándolo con medios N2 (DMEM/F12 suplementado con 1x suplemento de N2, 1x P/S, ) al 100% de N2 durante un período de 8 días.
- Al comienzo del día 12, fije las células para la inmunotinción utilizando PAX6 como marcador de neuroectodermo.
- Para los estudios de determinación del destino mesodermo y endodérmico, se empleó un enfoque basado en CHIR99021 moléculas pequeñas descrito en publicaciones anteriores12,13.
- Para ello, cuando las células alcancen el 70% de confluencia, tratar con 3 μM de CHIR99021 en medio Definitive Endoderm (DE) durante 24 h seguido de la fijación para inmunotinción utilizando Brachuary como marcador específico del mesodermo.
- Para la etapa endodérmica, cultive las células durante 24 h adicionales en medio DE solo sin la adición de CHIR99021 antes de fijar para la inmunotinción con FOXA2.
- Para el ensayo de formación de EB, siembre las células en superficies celulares de baja adhesión sin usar Matrigel para cultivar células en condiciones de suspensión durante 24 h antes de tomar micrografías.
5. Análisis de Western blot
- Lavar las células dos veces con PBS y lisar con 1x tampón RIPA que contiene 1% de SDS y 1x cóctel de inhibidores de proteasa y fosfatasa.
- Aclarar los lisados por centrifugación a 16.000 x g durante 10 min a 4 °C, seguido de la recogida de los sobrenadantes.
- Cuantifique la proteína celular total utilizando el kit de ensayo de proteínas BCA siguiendo las instrucciones del fabricante. Ajuste las muestras a 2 μg/μL utilizando un tampón de muestra de colorante de carga de 4×.
- Desnaturalice las muestras de proteínas a 70 °C durante 10 min, cargue cantidades iguales de cada muestra y resuelva utilizando geles SDS-PAGE7 con un gradiente de 4%-12% seguido de una transferencia a la membrana de PVDF a un voltaje constante de 100 V durante 1 h a 4 °C.
- Bloquee las membranas con leche descremada al 5% e incube en diluciones de anticuerpos primarios a 4 °C durante la noche con rotación.
- A continuación, lave las membranas 5 veces con tampón PBST e incube en anticuerpos secundarios apropiados conjugados con HRP durante 1 h a temperatura ambiente.
- Lavar las membranas de nuevo con PBST 5x, incubar con sustrato quimioluminiscente y revelar con películas de rayos X.
6. Inmunotinción
- Siembre las células en placas de pocillos P4 e incube durante al menos 24 horas antes de la fijación para permitir la unión adecuada de las células a las superficies.
- Lave las celdas 3 veces con PBS para eliminar las celdas muertas, así como los componentes del medio, seguido de la fijación con PFA al 4% durante 15 minutos a temperatura ambiente.
- Permeabilizar las células mediante el uso de tritón X-100 al 0,3% seguido de un bloqueo para la unión inespecífica mediante el uso de BSA al 2% en PBS durante 1 h a temperatura ambiente.
- Incubar las muestras con anticuerpos primarios (OCT4, NANOG, SOX2, KI67, PAX6, Brachuary, FOXA2) diluidos en BSA al 1% durante la noche a 4 °C.
- Lavar las células 3 veces con PBS e incubar con anticuerpos secundarios apropiados (Cabra anti-ratón 488, Cabra anti-conejo 488, Cabra anti-ratón 546, Cabra anti-conejo 546) diluidos en BSA al 1% durante 1 h a temperatura ambiente.
- Por último, lavar las muestras con PBS 3x y contrateñirlas con DAPI y obtener imágenes con un microscopio de fluorescencia.
Resultados
Clonación de sgRNAs L2HGDH en lentiCRISPRv2 puro
El vector puro lentiCRISPRv2 se obtuvo comercialmente (ver Tabla de Materiales) y se digirió con BsmB1, lo que resultó en la liberación de un fragmento de relleno de 1,8 Kb. Como se muestra en la Figura 2A, se observó una digestión completa del vector. Para cada constructo, se examinaron seis clones para detectar la presencia o ausencia de inserto utilizando una secuencia inversa de sgRNA como cebador y un cebador directo (U6-459F) desde dentro de la secuencia vectorial. Con este enfoque, solo los clones que tenían el inserto produjeron un producto de PCR de 288 pb, y los clones negativos que no contenían la secuencia inversa de sgRNA resultaron en ningún producto de PCR, como se muestra en la Figura 2B.
Infecciones por H9, selecciones estables y Western blot
Las hESCs se infectaron con partículas lentivirales que expresan dos sgRNAs diferentes dirigidos al exón 1 del gen L2HGDH y se seleccionaron con puromicina. El análisis de Western blot se realizó a partir de poblaciones heterogéneas de células de sgRNA y Cas9 que coexpresan de forma estable para establecer cuál de los dos sgRNA utilizados era más eficiente para inducir la deleción génica. Como se muestra en la Figura 3A, L2HGDH-sgRNA-1 fue más eficiente que la otra secuencia para reducir la expresión de L2HGDH en poblaciones celulares heterogéneas.
Selección clonal, expansión y análisis de proteínas
La selección unicelular y la expansión clonal se realizaron para las células que expresan L2HGDH-sgRNA-1. Los pocillos que produjeron clones individuales se marcaron y crecieron hasta que se alcanzó un tamaño suficiente para que las colonias (generalmente 2 semanas) se expandieran más, criopreservaran y analizaran (Figura 3B). Se analizaron un total de 22 clones mediante el análisis de Western blot, que arrojó cinco knockouts homocigotos (23%) para L2HGDH. Este resultado muestra una alta eficiencia de la eliminación de genes mediante la entrega de genes mediada por lentivirales para la coexpresión estable de sgRNA y Cas9 a partir de un solo casete. La Figura 4 muestra un análisis de Western blot de los cinco clones knockout homocigotos junto con las células de control, mostrando una expresión reducida del 100% en comparación con el control en los clones seleccionados.
Análisis a nivel de ADN
Con el fin de mapear el sitio de mutación de CRISPR aguas arriba de la secuencia de reconocimiento de PAM de Cas9, se realizó una reacción en cadena de la polimerasa basada en cebadores específicos basados en sitios de mutación (MS-BSP PCR) utilizando ADN genómico de células de control y de cinco clones diferentes. Como se muestra en la Figura 5, dos clones (A5 y B4) tienen mutaciones inmediatamente cercanas a la secuencia de reconocimiento de PAM, mientras que los otros tres clones (A1, A9 y B12) mostraron que la mutación podría no estar muy cerca del sitio PAM y está más arriba de la secuencia de sgRNA de 20 pb dentro del exón 1 del gen L2HGDH .
Con el fin de encontrar las mutaciones exactas en el ADN, los productos de PCR purificados de células de control (solo vector infectado H9), así como tres clones diferentes de CRISPR, A5, A1 y B4, se sometieron a secuenciación de Sanger, y los sitios de mutación se mapearon utilizando la herramienta de alineaciones múltiples clustalw aguas arriba del sitio PAM8. Los resultados revelaron una mutación de deleción en el clon A5 aguas arriba del sitio PAM. Los otros dos clones, A1 y B4, mostraron una mutación de inserción aguas arriba del sitio PAM, como se destaca en la Figura 6.
Análisis funcional de clones
Tras la generación de líneas celulares knockout CRISPR en hESCs, el siguiente paso es confirmar su pluripotencia y potencial de diferenciación. Para ello, probamos diferentes marcadores de pluripotencia para tres clones diferentes, A1, A5 y B4, y comparamos los resultados con células de control (infectadas solo por vectores H9). Los resultados no mostraron cambios en el potencial de pluripotencia de las líneas celulares knockout determinado por inmunotinción para los marcadores OCT4, NANOG y SOX2. A continuación, las líneas celulares de control y knockout también se tiñeron para el marcador de proliferación celular, KI67, que tampoco mostró cambios en las líneas celulares knockout en comparación con el control. Estos resultados muestran que el knockout de CRISPR no afectó a las propiedades de autorrenovación de estas células. Además, la EB y la formación de colonias tampoco se vieron afectadas (Figura 7). Utilizando métodos in vitro establecidos en nuestro laboratorio, todos los clones se diferenciaron con éxito en tres células de la capa germinal, como se confirmó mediante inmunotinción para PAX6 (marcador de neuroectodermo), Brachyury (marcador de mesodermo) y FOXA2 (marcador de endodermo). No se observaron cambios en el potencial de diferenciación de las líneas celulares knockout en comparación con las células de control, lo que demostró que las células knockout conservaron su potencial de diferenciación (Figura 8).

Figura 1: Diagrama esquemático que muestra el diseño del estudio en 5 pasos. Paso 1: Diseño y clonación de sgRNAs en el vector CRISPR V2 digerido por Bsmb1. Paso 2: Generación de partículas lentivirales utilizando vectores de entrada que transportan sgRNA junto con plásmidos de empaquetamiento mediante co-transfección HEK293T células mediante polifecciones y concentración viral mediante ultracentrifugación. Paso 3: Infección de hESCs usando partículas lentivirales a MOI de 10-20 y selección celular estable usando puromicina seguida de análisis de poblaciones celulares heterogéneas para ver la eficiencia de los sgRNAs. Paso 4: Clonación de células individuales mediante el método de dilución limitada de las células que mostraron el máximo derribo en poblaciones heterogéneas y selección de clones individuales. Paso 5: Expansión de clones individuales y análisis a nivel de ADN, proteínas y análisis funcional de clones para determinar su tallo y potencial de diferenciación. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Clonación de sgRNAs en el vector lentiCRISPR V2.puro. (A) Digestión del vector lentiCRISPR V2.puro con la enzima de restricción, BsmB1. La flecha indica un fragmento de relleno de 1,8 Kb liberado tras la digestión del vector. (B) Análisis de PCR de colonias para diferentes clones de las construcciones 1 y 2 de sgRNA de L2HGDH. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Western blots y propagación clonal mediante selección de una sola célula. (A) Análisis de Western blot a partir de dos réplicas biológicas diferentes (S1 y S2) de poblaciones heterogéneas de sgRNA co-expresivo estable (L2H-1 y L2H-2) y Cas9 junto con hESCs de control (C) H9. (B) Selección de una sola célula y expansión clonal de células H9 que expresan de manera estable L2HGDH-sgRNA-1. Las flechas indican pocillos de placas de 96 pocillos que muestran solo clones individuales. Barra de escala = 100 μM. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Análisis de Western blot de diferentes clones de H9. (A-C) El análisis de Western blot de lisados totales de células de 22 clones arrojó cinco knockouts homocigotos (23%) para L2HGDH. (D) Confirmación de knockouts de L2HGDH a partir de un experimento de réplica biológica diferente utilizando Western blot. Haga clic aquí para ver una versión más grande de esta figura.
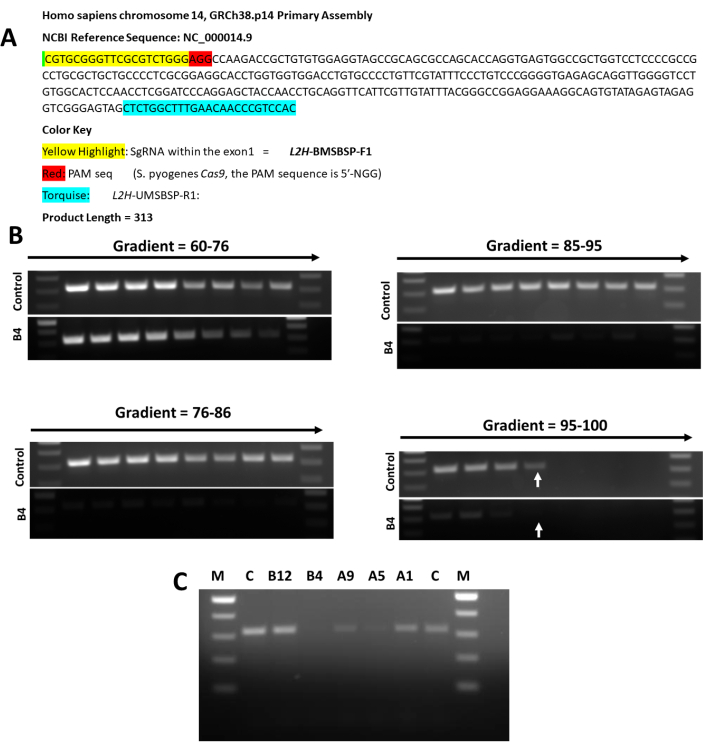
Figura 5: Análisis de PCR MSBSP de diferentes clones de H9. (A) Análisis de secuencia del exón 1 de L2HGDH utilizado para el análisis de PCR MSBSP. (B) Optimización de la temperatura de recocido para el análisis de PCR MSBSP utilizando control y H9 V2 L2HGDH KO Clon B4. En 96.8, el control muestra una banda, pero B4 no muestra una banda, como se muestra con flechas. (C) El análisis de todos los clones homocigotos de KO dio como resultado el siguiente mapeo del sitio de mutación dentro del exón 1. A1 = En/Supr no cerca del sitio PAM; A5 = En/Del Cerca del sitio PAM; A9 = En/Del Bastante cerca del sitio PAM; B4 = In/Supr: Muy cerca del sitio PAM B12 = In/Supr, no cerca del sitio PAM. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Alineación de los productos de PCR secuenciados de Sanger con la secuencia del exón 1 de referencia L2HGDH . Se realizaron múltiples alineaciones de productos de PCR secuenciados por Sanger de A1, A5 y B4, y celdas de control, junto con una secuencia de referencia dentro del primer exón de L2HGDH de la base de datos del NCBI. Haga clic aquí para ver una versión más grande de esta figura.
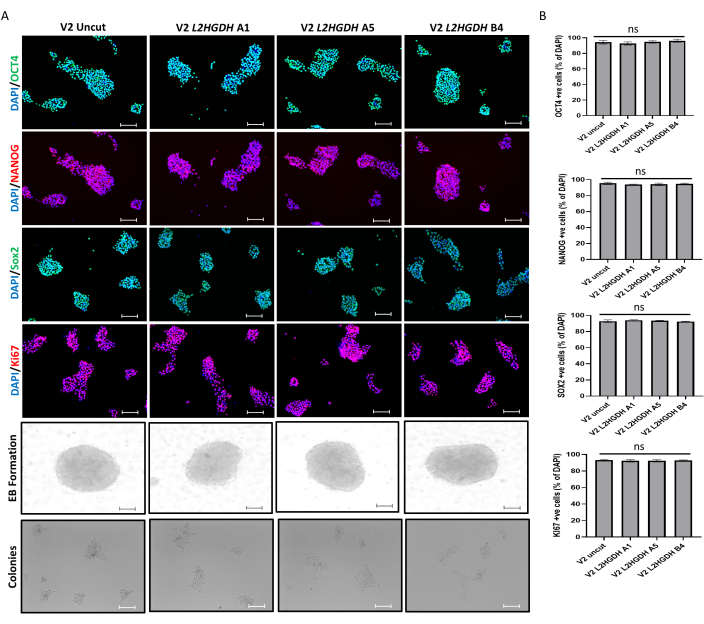
Figura 7: Caracterización funcional de líneas celulares knockout de CRISPR. (A) Las células fueron inmunoteñidas para marcadores de pluripotencia (OCT4, NANOG, SOX2), autorrenovación (KI67), EBs y formación de colonias con respecto a las hESCs H9 infectadas de control. Barras de escala: 100 μM. (B) Se calculó el porcentaje de células que expresan positivamente (+ve) en relación con la tinción con DAPI. Haga clic aquí para ver una versión más grande de esta figura.

Figura 8: Potencial de diferenciación de líneas celulares knockout CRISPR. Caracterización funcional de líneas celulares knockout CRISPR en términos de su potencial de diferenciación utilizando marcadores para PAX6 (neuroectodermo), Brachyury (mesodermo) y FOXA2 (endodermo) con respecto a las células H9 infectadas control. Barras de escala: 100 μM. Haga clic aquí para ver una versión más grande de esta figura.
| Paso de protocolo | Tiempo requerido |
| Clonación de sgRNAs en LentiCRISPRv2 | 4 días |
| Producción de lentivirus y determinación de títulos | 5 días |
| Infecciones por hESCs, selección de células estables y expansión | 2 semanas |
| Selección y expansión clonal de una sola célula | 2 semanas |
| Análisis funcional de clones | 2 semanas |
Tabla 1: Cronograma de los pasos del protocolo y tiempo estimado requerido.
Discusión
Este estudio ha estandarizado un método que permite deleciones génicas altamente eficientes y rentables en hESCs a través de la tecnología CRISPR-Cas9. Este método logró con éxito la deleción homocigota del gen L2HGDH en hESCs en 3-4 semanas, desde la infección por hESC hasta la selección y propagación clonal de una sola célula (Tabla 1). Aunque las manipulaciones genéticas mediadas por CRISPR-Cas9 se pueden lograr mediante transfecciones transitorias en la mayoría de las células, esto se convierte en un desafío en las células madre debido a la poca eficiencia de transfección y la alta toxicidad celular. Diferentes estudios han reportado hasta un 10% de los clones recuperados que muestran deleciones. Por ejemplo, Mali et al. informaron de una eficiencia de edición del genoma del 2% al 4% en células madre pluripotentes inducidas humanas después de dirigirse al locus3 de AAVS1. Otro estudio informó de eficiencias de edición del 1,3%-8,4% en hESCs ingenuas para dirigirse a TUNA, un ARN largo no codificante implicado en la pluripotencia y la diferenciación neuronal14. Ocaña et al. informaron de una eficiencia de hasta el 10% después de dirigirse al gen CFTR en células madre pluripotentes humanas.15 Por lo tanto, se requiere un método de administración eficiente de Cas9-gRNA para lograr una edición génica eficiente, y esto es especialmente cierto para las células madre, ya que estas células exhiben un mayor nivel de sensibilidad y resistencia a los métodos de administración transitorios en comparación con las células somáticas típicas y las líneas celulares inmortalizadas5.
En el presente estudio, se informa de una forma muy eficiente de obtener líneas celulares homocigóticas de CRISPR knockout mediante el uso de la administración mediada por lentivirales de sgRNAs en hESCs. El uso de vectores lentivirales para administrar CRISPR-Cas9 en las hESC demostró ser mucho más eficiente y requirió menos tiempo para lograr la eliminación de genes que el uso de transfecciones de vectores no virales por medios químicos o mecánicos (datos no mostrados). Esto resultó en una eficiencia de edición del genoma significativamente mayor (~23%) después de la selección clonal y la propagación de clones individuales. Por lo tanto, al combinar la administración mediada por lentivirus de sgRNAs y Cas9 en hESCs, así como un enfoque rentable para la producción de lentivirus, el método actual no solo es altamente eficiente y requiere menos tiempo, sino que también es rentable.
Estas son algunas de las recomendaciones para un enfoque exitoso: (1) Usar un solo sistema de vectores lentivirales que exprese Cas9 y sgRNA. (2) La presencia de un marcador de selección de antibióticos, como la puromicina, en el mismo plásmido que expresa Cas9 y sgRNAs asegura que casi el 100% de las células son positivas para la expresión de Cas9-sgRNA. Esto aumenta significativamente la probabilidad de eventos exitosos de edición de genes. (3) Con el fin de ser útil para las infecciones en hESCs, es indispensable concentrar los vectores lentivirales para producir altos títulos de virus. Entre las diversas técnicas utilizadas, el enfoque más económico es utilizar grandes volúmenes de VI y concentrarlos entre 100 y 500 veces el volumen original para lograr títulos en el rango de 107 a 108 UI/mL. (4) Para el método rentable y eficiente de HEK293T transfecciones celulares con los plásmidos, el uso de PEI (polietileneimina) para la transfección de vectores lentivirales para el empaquetamiento de virus es igualmente eficiente y, por lo tanto, rentable en comparación con el reactivo de transfección lipofectamina de uso común7. (5) Los títulos lentivirales promedio deben estar en el rango de 5 × 107 UI/mL. (6) La propagación clonal por selección de una sola célula es necesaria para lograr deleciones homocigóticas del gen de interés en estas células. (7) Aunque estudios previos han indicado que la edición génica mediada por CRISPR-Cas9 en células madre pluripotentes humanas da lugar a muy pocas mutaciones fuera del objetivo16,17, sigue siendo prudente comprobar los sitios fuera del objetivo. Esto se puede lograr a través de herramientas eficientes de diseño de ARNg para minimizar los efectos fuera del objetivo y la secuenciación de todo el genoma de líneas celulares editadas18, especialmente para experimentos de importancia clínica. (8) Por último, el cariotipo normal de los clones seleccionados debe verificarse utilizando métodos como la banda G y el análisis de alta resolución para garantizar que no se produzcan cambios genómicos a gran escala. También se puede realizar un ensayo de micronúcleos para evaluar la frecuencia de formación de micronúcleos, lo que puede indicar inestabilidad genómica.
Una de las principales limitaciones de este método es el uso de un sistema de vectores lentivirales, lo que condujo a la integración genómica estable de Cas9 y sgRNAs, lo que llevó a su expresión constitutiva después de la edición, lo cual no es deseable. En el futuro, este método puede mejorarse sustituyendo los promotores constitutivos por un promotor inducible por fármaco que impulse la expresión del gen Cas9, como el sistema Tet-on con tratamiento con doxiciclina para la expresión controlada de Cas9.
Divulgaciones
Los autores declaran que no existe conflicto de intereses.
Agradecimientos
Este trabajo contó con el apoyo de becas de investigación de la Universidad de los Emiratos Árabes Unidos (UAEU) - subvención #12M105, subvención #12R167 (Centro Zayed de Ciencias de la Salud), 21R105 (Fundación Benéfica y Humanitaria Zayed Bin Sultan (ZCHF)), y ASPIRE, el pilar de gestión del programa de tecnología del Consejo de Investigación de Tecnología Avanzada (ATRC) de Abu Dhabi, a través del número de subvención VRI-20-10 del Instituto de Investigación de Medicina de Precisión ASPIRE de Abu Dhabi (ASPIREPMRIAD).
Materiales
| Name | Company | Catalog Number | Comments |
| 2-MERCAPTOETHANOL | Invitrogen | 31350010 | |
| 38.5 mL, Sterile + Certified Free Open-Top Thinwall Ultra-Clear Tubes | Beckman Coulter | C14292 | |
| Accutase | Stem cell technologies | 7920 | |
| bFGF Recombinant human | Invitrogen | PHG0261 | |
| Brachyury Rabbit mAb | Abclonal | A5078 | |
| BsmBI-v2 | NEB | R0739S | |
| chir99021 | Tocris | 4423/10 | |
| Corning Matrigel Basement Membrane Matrix, LDEV-free | Corning | 354234 | |
| Cyclopamine | Stem cell technologies | 72074 | |
| DMEM media | Invitrogen | 11995073 | |
| DMEM NUTRIENT MIX F12 | Invitrogen | 11320033 | |
| DPBS w/o: Ca and Mg | PAN Biotech | P04-36500 | |
| Fetal bovie serum | Invitrogen | 10270106 | |
| FoxA2/HNF3β | CST | 8186 | |
| GAPDH (14C10) Rabbit mAb Antibody | CST | 2118S | |
| Gentle Cell Dissociation Reagent | Stem cell technologies | 7174 | |
| HyClone Non Essential Amino Acids (NEAA) 100x Solution | GE healthcare | SH30238.01 | |
| Ki-67 (D3B5) Rabbit mAb | CST | 9129 | |
| KnockOut Serum Replacement | Invitrogen | 10828028 | |
| L GLUTAMINE, 100x | Invitrogen | 2924190090 | |
| L2H-BMSBSP-F1 | Macrogen | CGTGCGGGTTCGCGTCTGGG | |
| L2HGDH Polyclonal antibody | Proteintech | 15707-1-AP | |
| L2HGDH-SgRNA1-F | Macrogen | CACCGCGTGCGG GTTCGCGTCTGGG | |
| L2HGDH-SgRNA1-R | Macrogen | AAACCCCAGACGC GAACCCGCACGC | |
| L2HGDH-SgRNA2-F | Macrogen | CACCGCCCGCGG GCTTTTCGCCGG | |
| L2HGDH-SgRNA2-R | Macrogen | AAACCCGGCGAA AAGCCCGCGGGC | |
| L2H-SeqF1 | Macrogen | GCTAAAGAGCGC GGGTCCTCGG | |
| L2H-SeqR1 | Macrogen | GTGGACGGGTTG TTCAAAGCCAGAG | |
| L2H-UMSBSP-R1 | Macrogen | GTGGACGGGTTG TTCAAAGCCAGAG | |
| LentiCRISPRv2 | Addgene | 52961 | |
| mTesR1 complete media | Stem cell technologies | 85850 | |
| Nanog Antibody | CST | 3580 | |
| NEUROBASAL MEDIUM 1x CTS | Invitrogen | A1371201 | |
| Neuropan 2 Supplement 100x | PAN Biotech | P07-11050 | |
| Neuropan 27 Supplement 50x | PAN Biotech | P07-07200 | |
| Oct-4 Antibody | CST | 2750 | |
| Pax6 (D3A9V) XP Rabbit mAb | CST | 60433 | |
| PENICILLIN STREPTOMYCIN SOL | Invitrogen | 15140122 | |
| pMD2.G | Addgene | 12259 | |
| Polybrene infection reagent | Sigma | TR1003- G | |
| Polyethylenimine, branched | Sigma | 408727 | |
| psPAX2.0 | Addgene | 12260 | |
| Puromycin | Invitrogen | A1113802 | |
| qPCR Lentivirus Titer Kit | Abm | LV900 | |
| Rock inhibitor Y-27632 dihydrochloride | Tocris | 1254 | |
| SB 431542 | Tocris | 1614/10 | |
| Sox2 Antibody | CST | 2748 | |
| Sucrose | Sigma | 57-50-1 | |
| TRYPSIN .05% EDTA | Invitrogen | 25300062 | |
| U6-459F | Macrogen | GAGGGCCTATT TCCCATGATTC | |
| Wizard Genomic DNA Purification Kit | Promega | A1120 | |
| XAV 939 | Tocris | 3748/10 |
Referencias
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jiang, F., Doudna, J. A. CRISPR-Cas9 structures and mechanisms. Annu Rev Biophys. 46 (1), 505-529 (2017).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Zhang, Z., et al. CRISPR/Cas9 genome-editing system in human stem cells: Current status and future prospects. Mol Ther Nucleic Acids. 9, 230-241 (2017).
- Qiu, Z., et al. MYC regulation of D2HGDH and L2HGDH influences the epigenome and epitranscriptome. Cell Chem Biol. 27 (5), 538-550.e537 (2020).
- Sheikh, M. A., Ansari, S. A. Lentiviral mediated delivery of shRNAs to hESCs and NPCs using low-cost cationic polymer polyethylenimine (PEI). J Vis Exp. (183), e63953 (2022).
- Guo, J., et al. A simple and cost-effective method for screening of CRISPR/Cas9-induced homozygous/biallelic mutants. Plant Methods. 14 (1), 40 (2018).
- Ardah, M. T., Parween, S., Varghese, D. S., Emerald, B. S., Ansari, S. A. Saturated fatty acid alters embryonic cortical neurogenesis through modulation of gene expression in neural stem cells. J Nutr Biochem. 62, 230-246 (2018).
- Parween, S., et al. Higher O-GlcNAc levels are associated with defects in progenitor proliferation and premature neuronal differentiation. Front Cell Neurosci. 11, 415 (2017).
- Parween, S., et al. Nutrient-sensitive protein O-GlcNAcylation modulates the transcriptome through epigenetic mechanisms during embryonic neurogenesis. Life Sci Alliance. 5 (8), e202201385 (2022).
- Varghese, D. S., et al. Developmental modeling of hepatogenesis using obese iPSCs-hepatocyte differentiation uncovers pathological features. Cell Death Dis. 13 (7), 670 (2022).
- Varghese, D. S., Alawathugoda, T. T., Ansari, S. A. Fine-tuning of hepatocyte differentiation from human embryonic stem cells: Growth factor. Stem Cells Int. 2019 (5), 5968236 (2019).
- Jacobs, E. Z., et al. CRISPR/Cas9-mediated genome editing in naïve human embryonic stem cells. Sci Rep. 7 (1), 16650 (2017).
- Cuevas-Ocaña, S., et al. A cell-based optimized approach for rapid and efficient gene editing of human pluripotent stem cells. Int J Mol Sci. 24 (3), 12345 (2023).
- Veres, A., et al. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN-targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell. 15 (1), 27-30 (2014).
- Smith, C., Gore, A., Yan, W., Abalde-Atristain, L., Li, Z., He, C. Whole-genome sequencing analysis reveals high specificity of CRISPR/Cas9 and TALEN-based genome editing in human iPSCs. Cell Stem Cell. 15 (1), 12-13 (2014).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target effects in CRISPR/Cas9-mediated genome engineering. Mol Ther Nucleic Acids. 4 (7), e264 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados