Method Article
حذف الجينات بوساطة CRISPR-Cas9 في الخلايا الجذعية البشرية متعددة القدرات المزروعة في ظروف خالية من المغذيات
In This Article
Summary
تصف الطريقة المقدمة توليد ضربة قاضية جينية بوساطة كريسبر في خط الخلايا الجذعية الجنينية البشرية (hESC) H9 ، والذي يعبر بثبات عن sgRNAs التي تستهدف جين L2HGDH باستخدام نظام توصيل الجينات بوساطة الفيروسية عالية الكفاءة.
Abstract
أحدث نظام CRISPR-Cas9 لتحرير الجينوم ثورة في دراسات وظائف الجينات في خلايا الثدييات ، بما في ذلك الخلايا الجذعية. ومع ذلك ، فإن التطبيق العملي لهذه التقنية ، لا سيما في الخلايا الجذعية متعددة القدرات ، يمثل تحديات معينة ، مثل كونها كثيفة الوقت والعمالة وكفاءة التحرير المنخفضة. هنا ، نصف توليد ضربة قاضية جينية بوساطة كريسبر في خط الخلايا الجذعية الجنينية البشرية (hESC) الذي يعبر بثبات عن sgRNAs لجين L2HGDH ، باستخدام نظام توصيل جيني بوساطة فيروسات عدسية عالية الكفاءة ومستقرة. تم تصنيع sgRNAs التي تستهدف exon 1 من جين L2HGDH كيميائيا واستنساخها في ناقل lentiCRISPR v2-puro ، والذي يجمع بين التعبير التأسيسي ل sgRNAs مع Cas9 في نظام ناقل أحادي عالي الكفاءة لتحقيق عيار أعلى للفيروسات العدسية لعدوى hESC والاختيار المستقر باستخدام بورومايسين. تم توسيع الخلايا المختارة من بورومايسين بشكل أكبر ، وتم الحصول على استنساخ أحادي الخلية باستخدام طريقة التخفيف المحدود. تم توسيع المستنسخة المفردة ، وتم الحصول على العديد من المستنسخة بالضربة القاضية متماثلة اللواقح لجين L2HGDH ، كما تم تأكيده من خلال انخفاض بنسبة 100٪ في تعبير L2HGDH باستخدام تحليل اللطخة الغربية. علاوة على ذلك ، باستخدام MSBSP-PCR ، تم تعيين موقع طفرة CRISPR في المنبع من تسلسل التعرف على PAM ل Cas9 في المستنسخة متماثلة اللواقح المختارة. تم إجراء تسلسل Sanger لتحليل عمليات الإدراج / الحذف الدقيقة ، وتم إجراء التوصيف الوظيفي للنسخ. أنتجت هذه الطريقة نسبة أعلى بكثير من عمليات الحذف متماثلة اللواقح مقارنة بطرق توصيل الجينات غير الفيروسية التي تم الإبلاغ عنها سابقا. على الرغم من أن هذا التقرير يركز على جين L2HGDH ، إلا أنه يمكن استخدام هذا النهج القوي والفعال من حيث التكلفة لإنشاء ضربة قاضية متماثلة اللواقح للجينات الأخرى في الخلايا الجذعية متعددة القدرات لدراسات وظائف الجينات.
Introduction
الخلايا الجذعية الجنينية البشرية (hESCs) والخلايا الجذعية المستحثة متعددة القدرات (iPSCs) هي خلايا جذعية لديها القدرة على التمايز إلى جميع أنواع الخلايا في الجسم. تعمل هذه الخلايا كأدوات قيمة لدراسة التنمية البشرية ، وكذلك لفهم الآليات الأساسية للأمراض المختلفة ، مما يوفر وعدا هائلا للطب التجديدي ونمذجة الأمراض واكتشاف الأدوية. تتضمن هذه الدراسات التحقيق في كيفية مساهمة جينات معينة في تطوير الكائنات الحية وعملها وتنظيمها1،2.
يتم استخدام تقنيات وأساليب مختلفة لفك تشفير وظيفة الجينات ، بما في ذلك التلاعب الجيني ، مثل الضربة القاضية للجينات أو الإفراط في التعبير ، وتحرير الجينوم. من بين هؤلاء ، برزت تقنية CRISPR-Cas9 كأكثر الطرق فعالية لدراسات خروج المغلوب الجيني وتحرير الجينات1،2،3. يعمل نظام CRISPR-Cas9 من خلال استخدام جزيء RNA واحد (sgRNA) مصمم خصيصا لتحديد تسلسل DNA معين ذي أهمية والارتباط به. يعمل sgRNA كدليل جزيئي ، ويوجه إنزيم Cas9 إلى الموقع الدقيق في الجينوم الذي يتطلب التعديل. بمجرد ربطه ، يبدأ Cas9 في كسر مزدوج الشريطة في الحمض النووي في الموقع المحدد. بعد انقسام الحمض النووي ، يتم تنشيط آليات الإصلاح المتأصلة في الخلية. وتشمل هذه مسارين رئيسيين للإصلاح: الانضمام النهائي غير المتماثل (NHEJ) والإصلاح الموجه بالتماثل (HDR). غالبا ما يؤدي NHEJ إلى عمليات إدخال أو حذف (indels) في موقع الكسر ، مما يؤدي إلى تعطيل الجينات أو تعطيلها. على العكس من ذلك ، يتيح HDR إدخال تسلسلات DNA جديدة في موقع الكسر ، مما يسهل إدخال التعديلات الجينيةالمستهدفة 4.
نظرا لأهمية حذف الجينات في الخلايا الجذعية متعددة القدرات ، تم نشر العديد من البروتوكولات على الضربة القاضية الجينية بوساطة CRISPR-Cas9 في hESCs / iPSCs. ومع ذلك ، تواجه العديد من هذه البروتوكولات قيودا كبيرة ، مثل كونها تستغرق وقتا طويلا للغاية ، وكثيفة العمالة ، وذات كفاءة منخفضة بسبب استخدام طرق توصيل الجينات غير الفيروسية5. هذه التحديات أكثر وضوحا في hESCs / iPSCs ، حيث من المعروف أن هذه الخلايا تتمتع بكفاءة تحرير أقل مقارنة بأنواع الخلايا الأخرى5. يمكن معالجة بعض هذه القيود عن طريق زيادة كفاءة توصيل البلازميد الذي يحتوي على Cas9 و sgRNAs. يمكن تحقيق ذلك بنجاح باستخدام نظام ناقل الفيروسات ، والذي يمكن أن يحسن بشكل كبير نتائج تحرير الجينات. بروتوكولات تغليف الفيروسات العدسية راسخة ومباشرة ، مما يسمح باعتمادها السهل في المختبرات ، حتى من قبل الباحثين ذوي الخبرة المحدودة. تظهر فيروسات العدسات كفاءة عدوى عالية عبر أنواع مختلفة من الخلايا ، بما في ذلك hESCs و iPSCs. لذلك ، يعد استخدام نظام العدسي الفيروسي لتعبير Cas9-sgRNA مثاليا لتجارب التحرير الجيني الروتينية في hESCs / iPSCs لدراسات وظائف الجينات.
هنا ، نقدم طريقة بسيطة ومباشرة لعمليات حذف الجينات المستندة إلى CRISPR-Cas9 عالية الكفاءة في hESCs في فترة زمنية أقصر نسبيا من البروتوكولات التقليدية (الشكل 1). على الرغم من استخدام ناقل فيروسي عدسي مع تعبير تأسيسي ل Cas9 و sgRNA ، إلا أنه يمكن استبداله بسهولة بتعبير Cas9 المحفز عن الدواء لتعبير Cas9 الذي يمكن التحكم فيه.
Protocol
يتم سرد تفاصيل التسلسلات الجينية والكواشف والمعدات المستخدمة في هذه الدراسة في جدول المواد.
1. تصميم الحمض النووي الريبي الفردي (sgRNA) والاستنساخ وإنتاج نواقل الفيروسات العدسية
ملاحظة: يتم استخدام تسلسلين مختلفين من sgRNA يستهدفان exon 1 من L2HGDH الجين ، وكلاهما مقتبس من Qiu et al.6مع مواقع PAM ل AGG و TGG للتسلسلين 1 و 2 ، على التوالي. كان طول كل من sgRNAs 20 نقطة أساس ، وتم تعديل النهايات لإضافة تسلسلات رابط لإنزيم التقييد Bsmb1 ليتم استنساخه في المتجه المستهدف (LentiCRISPRv2) كما هو موضح في التقرير السابق6. تمت إضافة تسلسلات الرابط أثناء تصميم sgRNAs لأغراض الاستنساخ.
- صلب sgRNAs المركبة كيميائيا6 واستنساخ في ناقل الفيروس العسسي أحادي القطع Bsmb1 ، LentiCRISPRv2 باستخدام بروتوكول محسن كما تم الإبلاغ عنهسابقا 7.
- استمر في تعبئة الفيروسات العدسية وتركيزها باستخدام بروتوكول موحد ومحسن7.
- لإنتاج الفيروسات العدسية، HEK293T البذور الخلايا (1 × 105 خلايا/سم2) باستخدام DMEM، و 10٪ FBS و 1x البنسلين/الستربتومايسين (P / S) وتحتضن طوال الليل في حاضنة زراعة الأنسجة عند 37 درجة مئوية في جو من 5٪ ثاني أكسيد الكربون2 في ظروف رطبة.
- عندما تكون 90٪ ملتقية ، فإن الخلايا HEK293T ذات ناقل الدخول (العمود الفقري الفارغ أو sgRNA الذي يعبر عن البلازميدات) وتعبئة البلازميدات باستخدام البوليمر الكاتيوني منخفض التكلفة PEI كما هو موضحسابقا 7.
- اجمع الوسائط المكيفة التي تحتوي على جزيئات الطافي الفيروسي (LVS) في 48 ساعة و 72 ساعة بعد التعدي والمضي قدما في الطرد المركزي الفائق باستخدام وسادة السكروز وقم بتدوير الأنابيب عند 1،25،000 × جم لمدة ساعتين عند 4 درجات مئوية.
- استمر في تركيز جسيمات LVS إلى ما لا يقل عن 200 ضعف الحجم الأصلي في PBS ، aliquot ، وقم بتخزينه عند -80 درجة مئوية حتى الاستخدام.
- حدد عيار جزيئات الفيروسات العدسية باستخدام مجموعة أدوات قياس المعايرة من الفيروسات العدسية qPCR باتباع تعليمات الشركة المصنعة (انظر جدول المواد).
2. الالتهابات الفيروسية وانتشار نسيلة خلية واحدة
- بالنسبة للعدوى بجزيئات LVS ، تحتوي معلق خلية البذور hESC (H9) على 1 × 105 خلايا / 0.5 مل من وسائط ثقافة hESC (الوسائط القاعدية + P / S + 10 ميكرومتر من مثبط الصخور) على ألواح بئر P24 مطلية بالكامل ب Matrigel (1:50) في 500 ميكرولتر من الوسائط وتحتضن طوال الليل للسماح للخلايا بالالتصاق. بذر الآبار الإضافية التي لن تصاب ب LVS ولكنها تعالج بالبورومايسين لتكون بمثابة مكافحة غير مصابة.
ملاحظة: يمكن إجراء عد الخلايا باستخدام مقياس كثافة الدم اليدوي أو عداد الخلايا الآلي. - في اليوم التالي ، قم بإصابة الخلايا عند تعدد العدوى (MOI) من 10 جنبا إلى جنب مع 8 ميكروغرام / مل من البوليبرين واحتضانها عند 37 درجة مئوية لمدة 8 ساعات متبوعة باستبدال الوسائط بوسائط hESC جديدة + P / S بدون مثبط الصخور واستمر في الزراعة حتى تتلاقى الخلايا بنسبة 90٪.
- ابدأ في اختيار البيرومايسين عن طريق استكمال الوسائط بتركيز 0.8 ميكروغرام / مل من بورومايسين عندما تصل الخلايا إلى 90٪ من التقاء ، والذي عادة ما يكون 48-72 ساعة بعد الإصابة ، واستمر في الانتقاء حتى تموت جميع الخلايا في الخلايا غير المصابة (المجموعة الضابطة).
- بعد اكتمال الاختيار (عادة 4-6 أيام) ، قم بتقسيم الخلايا المستقرة (1: 4) وقم بالتوسع للحفظ بالتبريد وإجراء مزيد من التحليل.
- قم بإجراء اختيار خلية واحدة وتمدد نسيلي باستخدام الخلايا التي تعبر عن L2HGDH-sgRNA-16.
- لهذا الغرض ، قم بإعداد معلق خلوي يعادل 500 خلية / 10 مل من وسائط النمو الكاملة وبذرة 100 ميكرولتر من هذا المعلق في كل بئر من صفيحة 96 بئرا.
- اترك الخلايا دون إزعاج لمدة 3 أيام ثم راقب.
- ضع علامة على الآبار التي تنتج نسخا مفردة وقم بتغيير الوسائط كل يومين حتى يتم تحقيق حجم كاف للمستعمرات (عادة أسبوعين) للتوسع أكثر ، والحفظ بالتبريد ، والتحليل.
3. استخراج gDNA ، MS-BSP PCR ، وتسلسل Sanger
- عزل gDNA من الخلايا باستخدام مجموعة عزل الحمض النووي الجيني باتباع تعليمات الشركة المصنعة (انظر جدول المواد).
- تابع رسم خرائط مواقع الطفرات في المنبع من تسلسلات التعرف على PAM باستخدام تحليل البادئات المحددة القائمة على مواقع الطفرات (MS-BSP)8.
- لهذا الغرض ، تم تصميم التمهيدي الأيمن غير المتحيز L2H-UMSBSP-R1 لتضخيم المنطقة خارج Exon1 لتضخيم أي أهداف.
- صمم برايمر أيسر متحيز L2H-BMSBSP-F1 بتسلسل مماثل ل sgRNA لتضخيم التسلسل المستهدف بالقرب من تسلسلات التعرف على PAM.
ملاحظة: في الظروف الصارمة جدا لتفاعل البوليميراز المتسلسل ، سيتم ملاحظة المنتج على الجل في نسخ غير متحولة. لن يتم ملاحظة أي منتج في استنساخ CRISPR-knockout الذي يظهر طفرات قريبة من المنبع من تسلسلات التعرف على PAM. - من أجل تعيين طفرات زوج قاعدة واحدة ، يقوم تفاعل البوليميراز المتسلسل بتضخيم تسلسل 468 نقطة أساس يمتد على الإكسون 1 بأكمله من L2HGDH ويخضع لتسلسل سانجر متبوعا بتحليل محاذاة تسلسل متعدد باستخدام clustalw8.
4. مقايسة تكوين hESC وتكوين الجسم الجنيني (EB)
- تابع التمايز الموجه للتحكم واستنساخ CRISPR المختلفة من hESCs (H9) نحو مصير الأديم الظاهر العصبي باتباع البروتوكولات المعمول بها كما هو موضح سابقا باستخدام طريقة تثبيط smad المزدوج9،10،11.
- عندما تكون عند التقاء 90٪ ، عالج الخلايا ب LDN193189 (200 نانومتر) و SB431542 (10 ميكرومتر) لمدة 24 ساعة أولية في وسائط KSR بنسبة 100٪ متبوعة بإضافة XAV939 (2 ميكرومتر) لمدة يومين إضافيين.
- بعد 3 أيام ، قلل النسبة المئوية لوسائط KSR (Knockout DMEM مكمل ب 15٪ (v / v) KSR ، 1٪ (v / v) L-glutamine ، 1٪ (v / v) P / S ، 1٪ (v / v) 10 mM MEM ، و 0.1٪ (v / v) 2-mercaptoethanol (75٪ ، 50٪ ، 25٪) عن طريق الجمع مع وسائط N2 (DMEM / F12 مكمل بملحق 1x N2 ، 1x P / S ، ) إلى وسائط N2 بنسبة 100٪ على مدى 8 أيام.
- في بداية اليوم 12 ، قم بإصلاح الخلايا للتلوين المناعي باستخدام PAX6 كعلامة للأديم الظاهر العصبي.
- بالنسبة لدراسات تحديد مصير الأديم المتوسط والأديم الباطن ، استخدم نهجا صغيرا قائما على CHIR99021 الجزيء الموصوف في المنشورات السابقة12،13.
- لهذا ، عندما تصل الخلايا إلى 70٪ من التقاء ، عالج ب 3 ميكرومتر من CHIR99021 في وسائط الأديم الباطن النهائي (DE) لمدة 24 ساعة متبوعا بتثبيت التلوين المناعي باستخدام Brachuary كعلامة خاصة بالأديم المتوسط.
- بالنسبة لمرحلة الأديم الباطن ، قم بزراعة الخلايا لمدة 24 ساعة إضافية في وسائط DE وحدها دون إضافة CHIR99021 قبل التثبيت للتلوين المناعي باستخدام FOXA2.
- لمقايسة تكوين EB ، قم بزرع الخلايا على أسطح الخلايا منخفضة الارتباط دون استخدام Matrigel لزراعة الخلايا في ظروف التعليق لمدة 24 ساعة قبل أخذ الصور المجهرية.
5. تحليل اللطخة الغربية
- اغسل الخلايا مرتين باستخدام PBS و lyse باستخدام 1x RIPA buffer يحتوي على 1٪ SDS و 1x كوكتيل مثبط البروتياز والفوسفاتيز.
- قم بتنظيف المحللات عن طريق الطرد المركزي عند 16,000 × جم لمدة 10 دقائق عند 4 درجات مئوية متبوعا بجمع المواد الطافية.
- حدد كمية بروتين الخلية الكلي باستخدام مجموعة فحص بروتين BCA باتباع تعليمات الشركة المصنعة. اضبط العينات على 2 ميكروغرام / ميكرولتر باستخدام 4× تحميل عينة الصبغة.
- قم بتغيير طبيعة عينات البروتين عند 70 درجة مئوية لمدة 10 دقائق ، وقم بتحميل كميات متساوية من كل عينة ، وحلها باستخدام 4٪ -12٪ من المواد الهلامية SDS-PAGEالمتدرجة 7 متبوعة بالنقل إلى غشاء PVDF بجهد ثابت يبلغ 100 فولت لمدة ساعة واحدة عند 4 درجات مئوية.
- سد الأغشية باستخدام 5٪ حليب خالي الدسم واحتضان في تخفيفات الأجسام المضادة الأولية عند 4 درجات مئوية طوال الليل مع الدوران.
- بعد ذلك ، اغسل الأغشية 5x باستخدام المخزن المؤقت PBST واحتضانه في الأجسام المضادة الثانوية المناسبة المترافقة ب HRP لمدة ساعة واحدة في درجة حرارة الغرفة.
- اغسل الأغشية مرة أخرى باستخدام PBST 5x ، واحتضن بركيزة كيميائية ، وطورها باستخدام أفلام الأشعة السينية.
6. تلغيم المناعة
- قم بزرع الخلايا على ألواح البئر P4 واحتضانها لمدة 24 ساعة على الأقل قبل التثبيت للسماح بالارتباط المناسب للخلايا بالأسطح.
- اغسل الخلايا 3 مرات باستخدام PBS لإزالة أي خلايا ميتة بالإضافة إلى مكونات الوسائط ، متبوعا بالتثبيت باستخدام 4٪ PFA لمدة 15 دقيقة في درجة حرارة الغرفة.
- قم باختراق الخلايا باستخدام 0.3٪ triton X-100 متبوعا بحجب الارتباط غير المحدد باستخدام 2٪ BSA في PBS لمدة ساعة واحدة في درجة حرارة الغرفة.
- احتضان العينات بالأجسام المضادة الأولية (OCT4 ، NANOG ، SOX2 ، KI67 ، PAX6 ، Brachuary ، FOXA2) المخففة في 1٪ BSA بين عشية وضحاها عند 4 درجات مئوية.
- اغسل الخلايا 3x باستخدام PBS واحتضانها بالأجسام المضادة الثانوية المناسبة (الماعز المضاد للفأر 488 ، الماعز المضاد للأرانب 488 ، الماعز المضاد للفأر 546 ، الماعز المضاد للأرانب 546) المخفف في 1٪ BSA لمدة ساعة واحدة في درجة حرارة الغرفة.
- أخيرا ، اغسل العينات باستخدام PBS 3x وقم بمواضدتها باستخدام DAPI والصورة باستخدام مجهر فلوري.
النتائج
استنساخ L2HGDH sgRNAs في lentiCRISPRv2 puro
تم الحصول على ناقل lentiCRISPRv2 puro تجاريا (انظر جدول المواد) وهضمه باستخدام BsmB1 ، مما أدى إلى إطلاق جزء حشو 1.8 كيلو بايت. كما هو موضح في الشكل 2 أ ، لوحظ هضم كامل للناقل. لكل بناء ، تم فحص ستة نسخ لوجود أو عدم وجود إدراج باستخدام تسلسل sgRNA العكسي كأساس وبرايمر أمامي (U6-459F) من داخل تسلسل المتجه. باستخدام هذا النهج ، فقط تلك المستنسخة التي تحتوي على الإدخال أسفرت عن منتج PCR يبلغ 288 نقطة أساس ، والنتائج المستنسخة السلبية التي لا تحتوي على التسلسل العكسي ل sgRNA لم ينتج عنها منتج PCR ، كما هو موضح في الشكل 2 ب.
عدوى H9 ، والاختيارات المستقرة ، واللطخة الغربية
أصيبت hESCs بجزيئات فيروسات تعبر عن اثنين من sgRNAs المختلفة التي تستهدف exon 1 من جين L2HGDH وتم اختيارها باستخدام البيورومايسين. تم إجراء تحليل اللطخة الغربية من مجموعات غير متجانسة من خلايا sgRNA و Cas9 التي تشارك في التعبير بثبات لتحديد أي من sgRNAs المستخدمة كان أكثر كفاءة في تحفيز حذف الجينات. كما هو موضح في الشكل 3 أ ، كان L2HGDH-sgRNA-1 أكثر كفاءة من التسلسل الآخر في تقليل التعبير عن L2HGDH في مجموعات الخلايا غير المتجانسة.
اختيار النسيل والتوسع وتحليل البروتين
تم إجراء اختيار الخلية المفردة والتوسع النسيلي للخلايا التي تعبر عن L2HGDH-sgRNA-1. تم تمييز الآبار التي أنتجت استنساخا مفردة ونمت حتى تم تحقيق حجم كاف للمستعمرات (عادة أسبوعين) للتوسع بشكل أكبر وحفظها بالتبريد وتحليلها (الشكل 3 ب). تم تحليل ما مجموعه 22 نسخة باستخدام تحليل اللطخة الغربية ، والتي أسفرت عن خمس ضربات قاضية متماثلة اللواقح (23٪) ل L2HGDH. تظهر هذه النتيجة كفاءة عالية للضربة القاضية الجينية باستخدام توصيل الجينات بوساطة الفيروسات العدسية للتعبير المشترك المستقر لكل من sgRNA و Cas9 من كاسيت واحد. يوضح الشكل 4 تحليل اللطخة الغربية لاستنساخ الضربة القاضية الخمسة متماثلة اللواقح جنبا إلى جنب مع خلايا التحكم ، مما يدل على انخفاض التعبير بنسبة 100٪ مقارنة بالتحكم في المستنسخة المحددة.
التحليل على مستوى الحمض النووي
من أجل تعيين موقع طفرة CRISPR في المنبع من تسلسل التعرف على PAM ل Cas9 ، تم إجراء تفاعل سلسلة البوليميراز البادئاتي المستندة إلى مواقع الطفرات (MS-BSP PCR) باستخدام الحمض النووي الجيني من خلايا التحكم ومن خمسة نسخ مختلفة. كما هو موضح في الشكل 5 ، هناك نسختان مستنسخة (A5 و B4) لهما طفرات قريبة مباشرة من تسلسل التعرف على PAM ، بينما أظهرت المستنسخة الثلاثة الأخرى (A1 و A9 و B12) أن الطفرة قد لا تكون قريبة جدا من موقع PAM وهي في المنبع من تسلسل sgRNA 20bp داخل exon 1 من L2HGDH الجين.
من أجل العثور على الطفرات الدقيقة في الحمض النووي ، تم تعريض منتجات تفاعل البوليميراز المتسلسل المنقى من خلايا التحكم (ناقل H9 المصاب فقط) بالإضافة إلى ثلاثة نسخ مختلفة من CRISPR ، A5 و A1 و B4 ، لتسلسل Sanger ، وتم تعيين مواقع الطفرات باستخدام أداة المحاذاة المتعددة clustalw في المنبع من موقع PAM8. كشفت النتائج عن طفرة حذف في استنساخ A5 في المنبع من موقع PAM. أظهر الاثنان الآخران ، A1 و B4 ، طفرة إدخالية في اتجاه المنبع من موقع PAM ، كما هو موضح في الشكل 6.
التحليل الوظيفي للنسخ
عند توليد خطوط خلايا CRISPR بالضربة القاضية في hESCs ، فإن الخطوة التالية هي تأكيد إمكاناتها متعددة القدرات والتمايز. لهذا الغرض ، اختبرنا علامات مختلفة من تعدد القدرات لثلاثة نسخ مختلفة ، A1 و A5 و B4 ، وقارنا النتائج بخلايا التحكم (H9 المصابة بالنواقل فقط). أظهرت النتائج عدم وجود تغيير في إمكانات تعدد القدرات لخطوط الخلايا بالضربة القاضية كما هو محدد عن طريق التلوين المناعي لعلامات OCT4 و NANOG و SOX2. بعد ذلك ، تم تلطيخ خطوط خلايا التحكم والضربة القاضية أيضا لعلامة تكاثر الخلايا ، KI67 ، والتي لم تظهر أيضا أي تغيير في خطوط الخلايا بالضربة القاضية مقارنة بعنصر التحكم. تظهر هذه النتائج أن ضربة المغلوب CRISPR لم تؤثر على خصائص التجديد الذاتي لهذه الخلايا. بالإضافة إلى ذلك ، لم يتأثر تكوين المستعمرة والمستعمرة أيضا (الشكل 7). باستخدام طرق المختبر التي تم إنشاؤها في مختبرنا ، تم تمييز جميع المستنسخة بنجاح إلى ثلاث خلايا طبقة جرثومية كما تم تأكيدها من خلال التلوين المناعي ل PAX6 (علامة الأديم الظاهري العصبي) ، و Brachyury (علامة الأديم المتوسط) ، و FOXA2 (علامة الأديم الباطن). لم يلاحظ أي تغيير في إمكانات التمايز لخطوط الخلايا بالضربة القاضية مقارنة بخلايا التحكم ، مما أظهر أن خلايا الضربة القاضية احتفظت بإمكاناتها التمايزية (الشكل 8).

الشكل 1: رسم تخطيطي يوضح تصميم الدراسة في 5 خطوات. الخطوة 1: تصميم واستنساخ sgRNAs في ناقل V2 CRISPR المهضوم Bsmb1. الخطوة 2: توليد جزيئات الفيروسات العدسية باستخدام ناقل الدخول الذي يحمل sgRNA جنبا إلى جنب مع البلازميدات التعبئة عن طريق نقل الخلايا HEK293T باستخدام polyfections والتركيز الفيروسي باستخدام الطرد المركزي الفائق. الخطوة 3: إصابة hESCs باستخدام جزيئات الفيروسات العدسية في وزارة الداخلية من 10-20 واختيار الخلايا المستقرة باستخدام بورومايسين متبوعا بتحليل مجموعات الخلايا غير المتجانسة لمعرفة كفاءة sgRNAs. الخطوة 4: استنساخ خلية واحدة عن طريق الحد من طريقة التخفيف من الخلايا التي أظهرت أقصى ضربة قاضية في المجموعات غير المتجانسة واختيار المستنسخة المفردة. الخطوة 5: توسيع المستنسخة المفردة والتحليل على مستوى الحمض النووي والبروتين والتحليل الوظيفي للنسخ من أجل الجذع وإمكانات التمايز. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 2: استنساخ sgRNAs في متجه lentiCRISPR V2.puro. (أ) هضم ناقل lentiCRISPR V2.puro مع إنزيم التقييد ، BsmB1. يشير السهم إلى جزء حشو 1.8 كيلو بايت تم إطلاقه عند هضم المتجه. (ب) تحليل مستعمرة PCR لانسخ مختلفة من L2HGDH sgRNA يبنيات 1 و 2. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 3: البقع الغربية والانتشار النسيلي باستخدام اختيار خلية واحدة. (أ) تحليل اللطخة الغربية من مكررتين بيولوجيتين مختلفتين (S1 و S2) للمجموعات غير المتجانسة من sgRNA التي تعبر بشكل ثابت عن بعضها البعض (L2H-1 و L2H-2) و Cas9 جنبا إلى جنب مع التحكم (C) H9 hESCs. (ب) اختيار الخلية المفردة والتوسع النسيلي لخلايا H9 التي تعبر بثبات عن L2HGDH-sgRNA-1. تشير الأسهم إلى آبار من 96 لوحة بئر تظهر نسخا مفردة فقط. شريط المقياس = 100 ميكرومتر. الرجاء النقر هنا لعرض نسخة أكبر من هذا الشكل.

الشكل 4: تحليل اللطخة الغربية من نسخ مختلفة من H9. (AC) أسفر تحليل اللطخة الغربية من إجمالي محللات الخلايا المكونة من 22 نسخة عن خمس ضربات قاضية متماثلة اللواقح (23٪) ل L2HGDH. (د) تأكيد الضربات القاضية L2HGDH من تجربة تكرار بيولوجية مختلفة باستخدام اللطخة الغربية. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
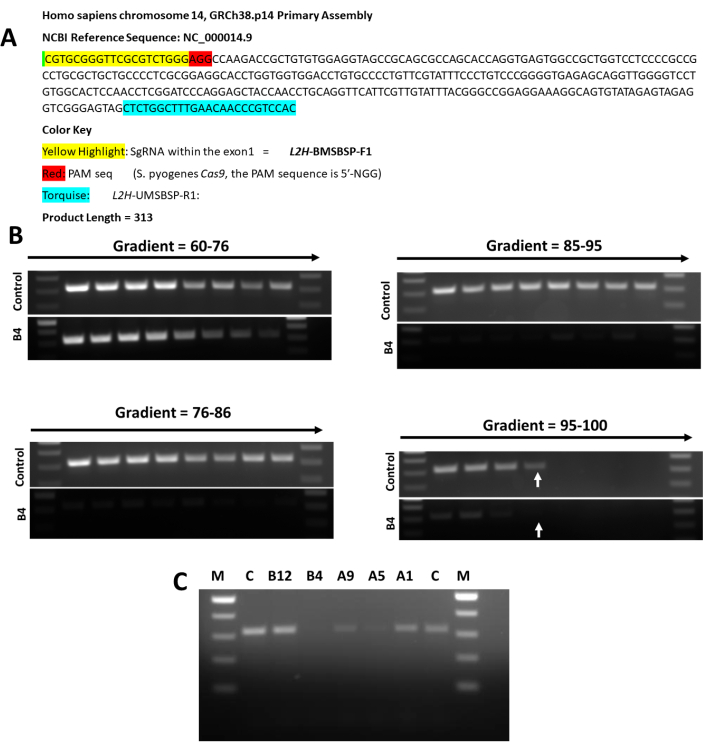
الشكل 5: تحليل MSBSP PCR لاستنساخ H9 المختلفة. (أ) تحليل تسلسل L2HGDH exon 1 المستخدم لتحليل MSBSP PCR. (ب) تحسين درجة حرارة التلدين لتحليل MSBSP PCR باستخدام التحكم و H9 V2 L2HGDH KO Clone B4. في 96.8 ، يعرض عنصر التحكم نطاقا ، لكن B4 لا يظهر نطاقا ، كما هو موضح في الأسهم. (ج) أدى تحليل جميع استنساخ KO متماثل اللواقح إلى رسم الخرائط أدناه لموقع الطفرة داخل exon 1. A1 = In/Del غير قريب من موقع PAM ؛ A5 = داخل / ديل بالقرب من موقع PAM ؛ A9 = في / ديل قريب إلى حد ما من موقع PAM ؛ B4 = In / Del قريب جدا من موقع PAM B12 = In / Del غير قريب من موقع PAM. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 6: محاذاة منتجات PCR المتسلسلة من Sanger مع تسلسل L2HGDH exon 1 المرجعي. تم إجراء محاذاة متعددة لمنتجات PCR المتسلسلة من Sanger من A1 و A5 و B4 ، وخلايا التحكم جنبا إلى جنب مع تسلسل مرجعي داخل أول exon ل L2HGDH من قاعدة بيانات NCBI. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
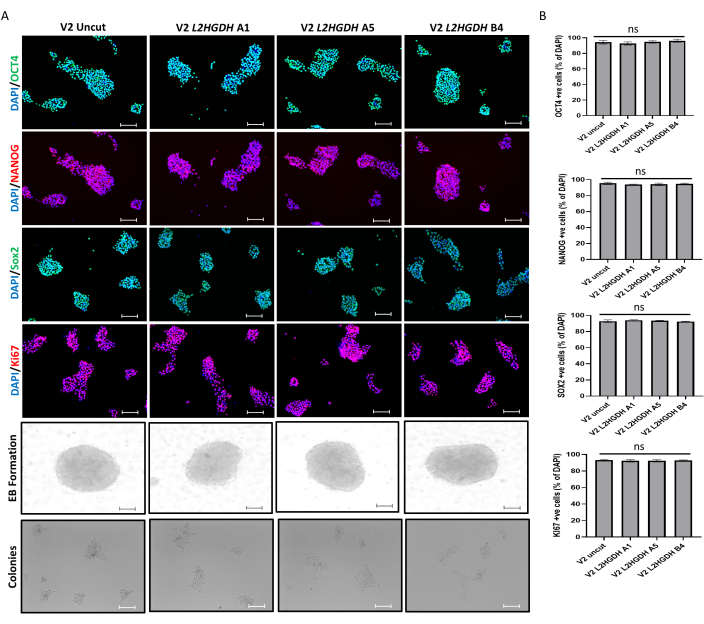
الشكل 7: التوصيف الوظيفي لخطوط خلايا الضربة القاضية CRISPR. (أ) تم تلطيخ الخلايا بالمناعة لعلامات تعدد القدرات (OCT4 ، NANOVG ، SOX2) ، والتجديد الذاتي (KI67) ، و EBs ، وتكوين المستعمرة فيما يتعلق بالتحكم H9 hESCs المصابة. أشرطة المقياس: 100 ميكرومتر. (ب) تم حساب النسبة المئوية للخلايا الشفيرة بشكل إيجابي (+ ve) بالنسبة إلى تلطيخ DAPI. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 8: إمكانات التمايز لخطوط خلايا CRISPR بالضربة القاضية. التوصيف الوظيفي لخطوط خلايا CRISPR بالضربة القاضية من حيث إمكانات التمايز باستخدام علامات PAX6 (الأديم الظاهر العصبي) ، العضدية (الأديم المتوسط) ، و FOXA2 (الأديم الباطن) فيما يتعلق بخلايا H9 المصابة بالسيطرة. أشرطة المقياس: 100 ميكرومتر. الرجاء النقر هنا لعرض نسخة أكبر من هذا الشكل.
| خطوة البروتوكول | الوقت المطلوب |
| استنساخ sgRNAs في LentiCRISPRv2 | 4 أيام |
| إنتاج فيروس العدسات وتحديد العيار | 5 أيام |
| عدوى hESC ، واختيارات الخلايا المستقرة ، والتوسع | 2 أسابيع |
| اختيار نسيلي أحادي الخلية وتوسعها | 2 أسابيع |
| التحليل الوظيفي للنسخ | 2 أسابيع |
الجدول 1: الجدول الزمني لخطوات البروتوكول والوقت المقدر المطلوب.
Discussion
قامت هذه الدراسة بتوحيد طريقة تتيح حذف الجينات عالية الكفاءة والفعالية من حيث التكلفة في hESCs من خلال تقنية CRISPR-Cas9. نجحت هذه الطريقة في الحذف المتماثل اللواقح لجين L2HGDH في hESCs في غضون 3-4 أسابيع ، بدءا من عدوى hESC إلى الانتقاء النسيلي أحادي الخلية وانتشاره (الجدول 1). على الرغم من أن التلاعب الجيني بوساطة CRISPR-Cas9 يمكن تحقيقه عن طريق التعداء العابرة في معظم الخلايا ، إلا أن هذا يصبح صعبا في الخلايا الجذعية بسبب ضعف كفاءة التعداء وارتفاع سمية الخلايا. أفادت دراسات مختلفة أن ما يصل إلى 10٪ من المستنسخة المستردة تظهر عمليات الحذف. على سبيل المثال ، مالي وآخرون أبلغوا عن كفاءة تحرير الجينوم بنسبة 2٪ إلى 4٪ في الخلايا الجذعية متعددة القدرات المستحثة البشرية بعد استهداف الموضع3 AAVS1. أفادت دراسة أخرى بكفاءة تحرير تتراوح بين 1.3٪ -8.4٪ في hESCs الساذجة لاستهداف TUNA ، وهو RNA طويل غير مشفر يشارك في تعدد القدرات والتمايز العصبي14. تم الإبلاغ عن كفاءة تصل إلى 10٪ بعد استهداف جين CFTR في الخلايا الجذعية البشرية متعددة القدرات بواسطة Ocana et al.15وبالتالي ، فإن طريقة التوصيل الفعالة ل Cas9-gRNA مطلوبة لتحقيق تحرير جيني فعال ، وهذا ينطبق بشكل خاص على الخلايا الجذعية حيث تظهر هذه الخلايا مستوى متزايدا من الحساسية والمقاومة لطرق التوصيل العابرة مقارنة بالخلايا الجسدية النموذجية وخطوط الخلايا الخالدة5.
في هذه الدراسة ، تم الإبلاغ عن طريقة عالية الكفاءة للحصول على خطوط خلايا خروج المغلوب CRISPR متماثلة اللواقح باستخدام توصيل sgRNAs بوساطة الفيروسات إلى hESCs. أثبت استخدام نواقل الفيروسات العدسية لإيصال CRISPR-Cas9 إلى hESCs أنه أكثر كفاءة وأقل استهلاكا للوقت في تحقيق حذف الجينات من استخدام عمليات نقل النواقل غير الفيروسية من خلال الوسائل الكيميائية أو الميكانيكية (البيانات غير معروضة). أدى ذلك إلى كفاءة تحرير الجينوم أعلى بكثير (~ 23٪) بعد الاختيار النسيلي وانتشار المستنسخة الفردية. لذلك ، من خلال الجمع بين التوصيل بوساطة الفيروسات من sgRNAs و Cas9 في hESCs بالإضافة إلى نهج فعال من حيث التكلفة لإنتاج فيروس العدسات ، فإن الطريقة الحالية ليست فقط عالية الكفاءة وأقل استهلاكا للوقت ولكنها أيضا فعالة من حيث التكلفة.
فيما يلي بعض التوصيات لنهج ناجح: (1) استخدام نظام ناقل فيروسي واحد يعبر عن كل من Cas9 و sgRNAs. (2) إن وجود علامة اختيار المضادات الحيوية ، مثل بورومايسين ، على نفس البلازميد الذي يعبر عن Cas9 و sgRNAs يضمن أن ما يقرب من 100٪ من الخلايا إيجابية لتعبير Cas9-sgRNA. هذا يزيد بشكل كبير من احتمالية نجاح أحداث تحرير الجينات. (3) لكي تكون مفيدة للعدوى في hESCs ، لا غنى عن تركيز نواقل الفيروسات العسقرية لإنتاج عيارات عالية من الفيروسات. من بين التقنيات المختلفة المستخدمة ، فإن الطريقة الأكثر اقتصادا هي استخدام كميات كبيرة من LVS وتركيزها بمقدار 100-500 ضعف الحجم الأصلي لتحقيق عيار في حدود 107 إلى 108 وحدة دولية / مل. (4) بالنسبة للطريقة الفعالة من حيث التكلفة والفعالة المتمثلة في HEK293T عمليات نقل الخلايا مع البلازميدات ، فإن استخدام PEI (البولي إيثيلينيمين) لتعداء ناقلات الفيروسات العسقرية لتغليف الفيروسات يكون بنفس القدر من الكفاءة ، وبالتالي فهو فعال من حيث التكلفة مقارنة بكاشف التعدي الشائع الاستخدام ليبوفيكتامين7. (5) يجب أن يتراوح متوسط عيار الفيروسات العدسية بين 5 × 107 وحدة دولية / مل. (6) مطلوب الانتشار النسيلي عن طريق اختيار خلية واحدة لتحقيق عمليات حذف متماثلة الزيجوت للجين محل الاهتمام في هذه الخلايا. (7) على الرغم من أن الدراسات السابقة أشارت إلى أن تحرير الجينات بوساطة CRISPR-Cas9 في الخلايا الجذعية البشرية متعددة القدرات ينتج عنه عدد قليل جدا من الطفرات غير المستهدفة16،17 ، إلا أنه لا يزال من الحكمة التحقق من المواقع غير المستهدفة. يمكن تحقيق ذلك من خلال أدوات تصميم gRNA الفعالة لتقليل التأثيرات غير المستهدفة والتسلسل على مستوى الجينوم لخطوط الخلايا المعدلة18 ، خاصة بالنسبة للتجارب ذات الأهمية السريرية. (8) أخيرا ، يجب التحقق من النمط النووي الطبيعي للنسخ المختارة باستخدام طرق مثل G-banding والتحليل عالي الدقة لضمان عدم وجود تغييرات جينومية واسعة النطاق. يمكن أيضا إجراء فحص النواة الدقيقة لتقييم تواتر تكوين النوى الدقيقة ، مما قد يشير إلى عدم الاستقرار الجيني.
يتمثل أحد القيود الرئيسية لهذه الطريقة في استخدام نظام ناقل الفيروسات العدسية ، مما أدى إلى التكامل الجيني المستقر ل Cas9 و sgRNAs ، مما أدى إلى التعبير التأسيسي بعد التحرير ، وهو أمر غير مرغوب فيه. في المستقبل ، يمكن تحسين هذه الطريقة عن طريق استبدال المحفزات التأسيسية بمحفز محفز للأدوية يقود التعبير عن جين Cas9 ، مثل نظام Tet-on بمعالجة الدوكسيسيكلين للتعبير المتحكم فيه عن Cas9.
Disclosures
ويعلن أصحاب البلاغ أنه لا يوجد تضارب في المصالح.
Acknowledgements
وقد تم دعم هذا العمل من خلال منح بحثية من جامعة الإمارات العربية المتحدة - #12M105 المنح ومنح #12R167 (مركز زايد للعلوم الصحية) و21R105 (مؤسسة زايد بن سلطان للأعمال الخيرية والإنسانية) و"أسباير"، وهي ركيزة إدارة البرامج التكنولوجية في مجلس أبحاث التكنولوجيا المتقدمة في أبوظبي، من خلال منحة الجائزة رقم VRI-20-10 من معهد أسباير لأبحاث الطب الدقيق في أبوظبي.
Materials
| Name | Company | Catalog Number | Comments |
| 2-MERCAPTOETHANOL | Invitrogen | 31350010 | |
| 38.5 mL, Sterile + Certified Free Open-Top Thinwall Ultra-Clear Tubes | Beckman Coulter | C14292 | |
| Accutase | Stem cell technologies | 7920 | |
| bFGF Recombinant human | Invitrogen | PHG0261 | |
| Brachyury Rabbit mAb | Abclonal | A5078 | |
| BsmBI-v2 | NEB | R0739S | |
| chir99021 | Tocris | 4423/10 | |
| Corning Matrigel Basement Membrane Matrix, LDEV-free | Corning | 354234 | |
| Cyclopamine | Stem cell technologies | 72074 | |
| DMEM media | Invitrogen | 11995073 | |
| DMEM NUTRIENT MIX F12 | Invitrogen | 11320033 | |
| DPBS w/o: Ca and Mg | PAN Biotech | P04-36500 | |
| Fetal bovie serum | Invitrogen | 10270106 | |
| FoxA2/HNF3β | CST | 8186 | |
| GAPDH (14C10) Rabbit mAb Antibody | CST | 2118S | |
| Gentle Cell Dissociation Reagent | Stem cell technologies | 7174 | |
| HyClone Non Essential Amino Acids (NEAA) 100x Solution | GE healthcare | SH30238.01 | |
| Ki-67 (D3B5) Rabbit mAb | CST | 9129 | |
| KnockOut Serum Replacement | Invitrogen | 10828028 | |
| L GLUTAMINE, 100x | Invitrogen | 2924190090 | |
| L2H-BMSBSP-F1 | Macrogen | CGTGCGGGTTCGCGTCTGGG | |
| L2HGDH Polyclonal antibody | Proteintech | 15707-1-AP | |
| L2HGDH-SgRNA1-F | Macrogen | CACCGCGTGCGG GTTCGCGTCTGGG | |
| L2HGDH-SgRNA1-R | Macrogen | AAACCCCAGACGC GAACCCGCACGC | |
| L2HGDH-SgRNA2-F | Macrogen | CACCGCCCGCGG GCTTTTCGCCGG | |
| L2HGDH-SgRNA2-R | Macrogen | AAACCCGGCGAA AAGCCCGCGGGC | |
| L2H-SeqF1 | Macrogen | GCTAAAGAGCGC GGGTCCTCGG | |
| L2H-SeqR1 | Macrogen | GTGGACGGGTTG TTCAAAGCCAGAG | |
| L2H-UMSBSP-R1 | Macrogen | GTGGACGGGTTG TTCAAAGCCAGAG | |
| LentiCRISPRv2 | Addgene | 52961 | |
| mTesR1 complete media | Stem cell technologies | 85850 | |
| Nanog Antibody | CST | 3580 | |
| NEUROBASAL MEDIUM 1x CTS | Invitrogen | A1371201 | |
| Neuropan 2 Supplement 100x | PAN Biotech | P07-11050 | |
| Neuropan 27 Supplement 50x | PAN Biotech | P07-07200 | |
| Oct-4 Antibody | CST | 2750 | |
| Pax6 (D3A9V) XP Rabbit mAb | CST | 60433 | |
| PENICILLIN STREPTOMYCIN SOL | Invitrogen | 15140122 | |
| pMD2.G | Addgene | 12259 | |
| Polybrene infection reagent | Sigma | TR1003- G | |
| Polyethylenimine, branched | Sigma | 408727 | |
| psPAX2.0 | Addgene | 12260 | |
| Puromycin | Invitrogen | A1113802 | |
| qPCR Lentivirus Titer Kit | Abm | LV900 | |
| Rock inhibitor Y-27632 dihydrochloride | Tocris | 1254 | |
| SB 431542 | Tocris | 1614/10 | |
| Sox2 Antibody | CST | 2748 | |
| Sucrose | Sigma | 57-50-1 | |
| TRYPSIN .05% EDTA | Invitrogen | 25300062 | |
| U6-459F | Macrogen | GAGGGCCTATT TCCCATGATTC | |
| Wizard Genomic DNA Purification Kit | Promega | A1120 | |
| XAV 939 | Tocris | 3748/10 |
References
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jiang, F., Doudna, J. A. CRISPR-Cas9 structures and mechanisms. Annu Rev Biophys. 46 (1), 505-529 (2017).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Zhang, Z., et al. CRISPR/Cas9 genome-editing system in human stem cells: Current status and future prospects. Mol Ther Nucleic Acids. 9, 230-241 (2017).
- Qiu, Z., et al. MYC regulation of D2HGDH and L2HGDH influences the epigenome and epitranscriptome. Cell Chem Biol. 27 (5), 538-550.e537 (2020).
- Sheikh, M. A., Ansari, S. A. Lentiviral mediated delivery of shRNAs to hESCs and NPCs using low-cost cationic polymer polyethylenimine (PEI). J Vis Exp. (183), e63953 (2022).
- Guo, J., et al. A simple and cost-effective method for screening of CRISPR/Cas9-induced homozygous/biallelic mutants. Plant Methods. 14 (1), 40 (2018).
- Ardah, M. T., Parween, S., Varghese, D. S., Emerald, B. S., Ansari, S. A. Saturated fatty acid alters embryonic cortical neurogenesis through modulation of gene expression in neural stem cells. J Nutr Biochem. 62, 230-246 (2018).
- Parween, S., et al. Higher O-GlcNAc levels are associated with defects in progenitor proliferation and premature neuronal differentiation. Front Cell Neurosci. 11, 415 (2017).
- Parween, S., et al. Nutrient-sensitive protein O-GlcNAcylation modulates the transcriptome through epigenetic mechanisms during embryonic neurogenesis. Life Sci Alliance. 5 (8), e202201385 (2022).
- Varghese, D. S., et al. Developmental modeling of hepatogenesis using obese iPSCs-hepatocyte differentiation uncovers pathological features. Cell Death Dis. 13 (7), 670 (2022).
- Varghese, D. S., Alawathugoda, T. T., Ansari, S. A. Fine-tuning of hepatocyte differentiation from human embryonic stem cells: Growth factor. Stem Cells Int. 2019 (5), 5968236 (2019).
- Jacobs, E. Z., et al. CRISPR/Cas9-mediated genome editing in naïve human embryonic stem cells. Sci Rep. 7 (1), 16650 (2017).
- Cuevas-Ocaña, S., et al. A cell-based optimized approach for rapid and efficient gene editing of human pluripotent stem cells. Int J Mol Sci. 24 (3), 12345 (2023).
- Veres, A., et al. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN-targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell. 15 (1), 27-30 (2014).
- Smith, C., Gore, A., Yan, W., Abalde-Atristain, L., Li, Z., He, C. Whole-genome sequencing analysis reveals high specificity of CRISPR/Cas9 and TALEN-based genome editing in human iPSCs. Cell Stem Cell. 15 (1), 12-13 (2014).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target effects in CRISPR/Cas9-mediated genome engineering. Mol Ther Nucleic Acids. 4 (7), e264 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved