Method Article
CRISPR-Cas9-vermittelte Gendeletion in humanen pluripotenten Stammzellen, die unter feederfreien Bedingungen kultiviert wurden
In diesem Artikel
Zusammenfassung
Die vorgestellte Methode beschreibt die Erzeugung eines CRISPR-vermittelten Gen-Knockouts in der humanen embryonalen Stammzelllinie (hESC) H9, die sgRNAs, die auf das L2HGDH-Gen abzielen, unter Verwendung eines hocheffizienten lentiviralen Gentransfersystems stabil exprimiert.
Zusammenfassung
Das CRISPR-Cas9-System für die Genom-Editierung hat die Genfunktionsstudien in Säugetierzellen, einschließlich Stammzellen, revolutioniert. Die praktische Anwendung dieser Technik, insbesondere bei pluripotenten Stammzellen, bringt jedoch einige Herausforderungen mit sich, wie z. B. die zeit- und arbeitsintensive und geringe Editierungseffizienz. Hier beschreiben wir die Erzeugung eines CRISPR-vermittelten Gen-Knockouts in einer humanen embryonalen Stammzelllinie (hESC), die sgRNAs für das L2HGDH-Gen stabil exprimiert, unter Verwendung eines hocheffizienten und stabilen lentiviralen Gentransfersystems. Die sgRNAs, die auf das Exon 1 des L2HGDH-Gens abzielen, wurden chemisch synthetisiert und in den lentiCRISPR v2-puro-Vektor kloniert, der die konstitutive Expression von sgRNAs mit Cas9 in einem hocheffizienten Einzelvektorsystem kombiniert, um höhere lentivirale Titer für hES-Infektionen und eine stabile Selektion mit Puromycin zu erreichen. Puromycin-selektierte Zellen wurden weiter expandiert, und Einzelzellklone wurden unter Verwendung der Methode der begrenzten Verdünnung erhalten. Die einzelnen Klone wurden expandiert, und es wurden mehrere homozygote Knockout-Klone für das L2HGDH-Gen erhalten, was durch eine 100%ige Reduktion der L2HGDH-Expression mittels Western-Blot-Analyse bestätigt wurde. Darüber hinaus wurde mittels MSBSP-PCR die CRISPR-Mutationsstelle stromaufwärts der PAM-Erkennungssequenz von Cas9 in den ausgewählten homozygoten Klonen kartiert. Es wurde eine Sanger-Sequenzierung durchgeführt, um die genauen Insertionen/Deletionen zu analysieren, und es wurde eine funktionelle Charakterisierung der Klone durchgeführt. Diese Methode erzeugte einen signifikant höheren Prozentsatz an homozygoten Deletionen im Vergleich zu zuvor berichteten nicht-viralen Gentransfermethoden. Obwohl sich dieser Bericht auf das L2HGDH-Gen konzentriert, kann dieser robuste und kostengünstige Ansatz verwendet werden, um homozygote Knockouts für andere Gene in pluripotenten Stammzellen für Genfunktionsstudien zu erzeugen.
Einleitung
Humane embryonale Stammzellen (hESCs) und induzierte pluripotente Stammzellen (iPSCs) sind Stammzellen mit dem Potenzial, sich in alle Zelltypen des Körpers zu differenzieren. Diese Zellen dienen als wertvolle Werkzeuge für die Erforschung der menschlichen Entwicklung sowie für das Verständnis der zugrunde liegenden Mechanismen verschiedener Krankheiten und sind somit ein enormes Versprechen für die regenerative Medizin, die Modellierung von Krankheiten und die Wirkstoffforschung. In solchen Studien wird untersucht, wie bestimmte Gene zur Entwicklung, Funktion und Regulation von Organismen beitragen 1,2.
Um die Funktion von Genen zu entschlüsseln, werden verschiedene Techniken und Ansätze eingesetzt, darunter genetische Manipulationen wie Gen-Knockout oder -Überexpression und Genom-Editing. Unter diesen hat sich die CRISPR-Cas9-Technologie als der effizienteste Ansatz für Gen-Knockout- und Gen-Editing-Studien erwiesen 1,2,3. Das CRISPR-Cas9-System funktioniert mit der Verwendung eines einzigen Guide-RNA (sgRNA)-Moleküls, das speziell entwickelt wurde, um eine bestimmte DNA-Sequenz von Interesse zu identifizieren und an sie zu binden. Die sgRNA fungiert als molekularer Leitfaden und leitet das Cas9-Enzym genau an die Stelle im Genom, die modifiziert werden muss. Einmal gebunden, initiiert Cas9 einen doppelsträngigen Bruch in der DNA an der angegebenen Stelle. Nach der Spaltung der DNA werden die zelleigenen Reparaturmechanismen aktiviert. Dazu gehören zwei Hauptreparaturwege: die nicht-homologe Endverbindung (NHEJ) und die homologiegerichtete Reparatur (HDR). NHEJ führt häufig zu Insertionen oder Deletionen (Indels) an der Bruchstelle, was zu einer Störung oder Inaktivierung des Gens führt. Umgekehrt ermöglicht HDR das Einfügen neuer DNA-Sequenzen an der Bruchstelle, was die Einführung gezielter genetischer Veränderungen erleichtert4.
Angesichts der Bedeutung von Gendeletionen in pluripotenten Stammzellen wurden mehrere Protokolle zu CRISPR-Cas9-vermittelten Gen-Knockouts in hESCs/iPSCs veröffentlicht. Viele dieser Protokolle sind jedoch mit erheblichen Einschränkungen konfrontiert, z. B. sind sie extrem zeitaufwändig, arbeitsintensiv und haben aufgrund der Verwendung nicht-viraler Genverabreichungsmethoden eine geringe Effizienz5. Diese Herausforderungen sind bei hESCs/iPSCs noch ausgeprägter, da diese Zellen bekanntermaßen im Vergleich zu anderen Zelltypen eine geringere Editierungseffizienz aufweisen5. Einige dieser Einschränkungen können durch eine Erhöhung der Effizienz der Plasmidabgabe, die Cas9 und sgRNAs enthält, behoben werden. Dies kann erfolgreich mit einem lentiviralen Vektorsystem erreicht werden, das die Ergebnisse der Geneditierung erheblich verbessern kann. Die Verpackungsprotokolle für Lentiviren sind gut etabliert und unkompliziert und ermöglichen eine einfache Anwendung in Laboren, selbst von Forschern mit begrenzter Erfahrung. Lentiviren weisen eine hohe Infektionseffizienz bei verschiedenen Zelltypen auf, einschließlich hES-Zellen und iPS-Zellen. Daher ist die Verwendung eines lentiviralen Systems für die Cas9-sgRNA-Expression ideal für routinemäßige Gen-Editing-Experimente in hESCs/iPSCs für Genfunktionsstudien.
Hier stellen wir eine einfache und unkomplizierte Methode für hocheffiziente CRISPR-Cas9-basierte Gendeletionen in hES-Zellen in einer vergleichsweise kürzeren Zeitdauer als herkömmliche Protokolle zur Verfügung (Abbildung 1). Obwohl ein lentiviraler Vektor mit konstitutiver Expression von Cas9 und sgRNA verwendet wurde, könnte er leicht durch eine medikamenteninduzierbare Cas9-Expression für eine kontrollierbare Cas9-Expression ersetzt werden.
Protokoll
Die Einzelheiten zu den in dieser Studie verwendeten Gensequenzen, Reagenzien und Geräten sind in der Materialtabelle aufgeführt.
1. Design der Single Guide RNA (sgRNA), Klonierung und Produktion lentiviraler Vektoren
HINWEIS: Es werden zwei verschiedene sgRNA-Sequenzen verwendet, die auf das Exon 1 des L2HGDH-Gens abzielen und beide von Qiu et al.6mit PAM-Stellen von AGG und TGG für die Sequenzen 1 bzw. 2 adaptiert wurden. Beide sgRNAs waren 20 bp lang, und die Enden wurden modifiziert, um Linkersequenzen für das Restriktionsenzym Bsmb1 hinzuzufügen, das im Zielvektor (LentiCRISPRv2) kloniert werden soll, wie im vorherigen Bericht6 beschrieben. Linkersequenzen wurden während des Designs von sgRNAs für Klonierungszwecke hinzugefügt.
- Anneal synthetisierte chemisch synthetisierte sgRNAs6 und klonierte in den Bsmb1-Single-Cut-lentiviralen Vektor, LentiCRISPRv2, unter Verwendung eines optimierten Protokolls, wie zuvor berichtet7.
- Fahren Sie mit der lentiviralen Verpackung und Konzentration unter Verwendung eines standardisierten und optimierten Protokollsfort 7.
- Für die Lentivirus-Produktion HEK293T Zellen (1 × 105 Zellen/cm2) mit DMEM, 10 % FBS und 1x Penicillin/Streptomycin (P/S) säen und über Nacht in einem Gewebekultur-Inkubator bei 37 °C bei einer Atmosphäre von 5 % CO2 unter feuchten Bedingungen inkubieren.
- Wenn 90 % konfluent sind, co-transfizieren HEK293T Zellen mit Eintrittsvektor (leeres Rückgrat oder sgRNA-exprimierende Plasmide) und Verpackungsplasmiden unter Verwendung des kostengünstigen kationischen Polymers PEI, wie zuvor beschrieben7.
- Sammeln Sie die konditionierten Medien, die Partikel des lentiviralen Überstands (LVS) enthalten, 48 h und 72 h nach der Transfektion, fahren Sie mit der Ultrazentrifugation unter Verwendung eines Saccharosekissens fort und drehen Sie die Röhrchen 2 h lang bei 4 °C bei 1,25,000 x g .
- Fahren Sie mit der LVS-Partikelkonzentration auf mindestens das 200-fache des ursprünglichen Volumens in PBS fort, aliquotieren Sie es und lagern Sie es bis zur Verwendung bei -80 °C.
- Bestimmen Sie den Titer von lentiviralen Partikeln mit dem qPCR Lentivirus Titer Kit gemäß den Anweisungen des Herstellers (siehe Materialtabelle).
2. Lentivirale Infektionen und einzellige klonale Vermehrung
- Bei Infektionen mit LVS-Partikeln wird eine hESC (H9)-Zellsuspension mit 1 × 105 Zellen/0,5 ml hESC-Kulturmedium (Basalmedium + P/S + 10 μM Rock-Inhibitor) auf vollständigen, mit Matrigel (1:50) beschichteten P24-Well-Platten in 500 μl Medium geseedet und über Nacht inkubiert, damit sich die Zellen anheften können. Säen Sie zusätzliche Vertiefungen aus, die nicht mit LVS infiziert, aber mit Puromycin behandelt werden, um als nicht infizierte Kontrolle zu dienen.
HINWEIS: Die Zellzählung kann mit einem manuellen Hämozytometer oder einem automatisierten Zellzähler durchgeführt werden. - Am nächsten Tag infizieren Sie die Zellen bei einer Multiplizität der Infektion (MOI) von 10 zusammen mit 8 μg/ml Polybren und inkubieren Sie 8 h lang bei 37 °C, gefolgt von einem Medienaustausch mit frischem hESC-Medium + P/S ohne Rock-Inhibitor und setzen Sie die Kultivierung fort, bis die Zellen zu 90 % konfluiert sind.
- Beginnen Sie die Puromycin-Selektion, indem Sie das Medium mit einer Konzentration von 0,8 μg/ml Puromycin ergänzen, wenn die Zellen eine Konfluenz von 90 % erreichen, was normalerweise 48-72 Stunden nach der Infektion der Fall ist, und setzen Sie die Selektion fort, bis alle Zellen in den nicht infizierten Zellen (Kontrollgruppe) absterben.
- Nachdem die Selektion abgeschlossen ist (in der Regel 4-6 Tage), teilen Sie stabile Zellen (1:4) und expandieren Sie sie für die Kryokonservierung und weitere Analyse.
- Führen Sie eine Einzelzellselektion und klonale Expansion mit Zellen durch, die L2HGDH-sgRNA-1exprimieren 6.
- Zu diesem Zweck wird eine Zellsuspension hergestellt, die 500 Zellen/10 ml vollständiges Wachstumsmedium entspricht, und 100 μl dieser Suspension in jede Vertiefung einer 96-Well-Platte aussäen.
- Lassen Sie die Zellen 3 Tage lang ungestört und beobachten Sie dann.
- Markieren Sie die Vertiefungen, die einzelne Klone liefern, und wechseln Sie das Medium jeden zweiten Tag, bis eine ausreichende Größe erreicht ist, damit die Kolonien (normalerweise 2 Wochen) weiter expandieren, kryokonservieren und analysieren können.
3. gDNA-Extraktion, MS-BSP PCR und Sanger-Sequenzierung
- Isolieren Sie gDNA aus Zellen mit dem genomischen DNA-Isolationskit gemäß den Anweisungen des Herstellers (siehe Materialtabelle).
- Fahren Sie mit der Kartierung der Mutationsstellen vor den PAM-Erkennungssequenzen mit Hilfe der MS-BSP-Analyse (Mutation Sites Based Specific Primers) fort8.
- Zu diesem Zweck wurde ein unvoreingenommener rechter Primer L2H-UMSBSP-R1 entwickelt, um den Bereich außerhalb des Exon1 zu amplifizieren, um beliebige Ziele zu amplifizieren.
- Entwerfen Sie einen verzerrten linken Primer L2H-BMSBSP-F1 mit einer identischen Sequenz wie sgRNA, um die Zielsequenz nahe an PAM-Erkennungssequenzen zu amplifizieren.
HINWEIS: Bei sehr hohen strengen PCR-Bedingungen wird das Produkt bei nicht mutierten Klonen auf dem Gel beobachtet. In CRISPR-Knockout-Klonen, die Mutationen nahe dem Upstream der PAM-Erkennungssequenzen aufwiesen, wurde kein Produkt beobachtet. - Um Mutationen einzelner Basenpaare zu kartieren, wird eine PCR-Amplifikation einer 468 bp-Sequenz, die das gesamte Exon 1 von L2HGDH umfasst, einer Sanger-Sequenzsequenzierung unterzogen, gefolgt von einer Multiple-Sequenz-Alignment-Analyse mit clustalw8.
4. Assay zur hES-Differenzierung und zur Bildung des Embryoidkörpers (EB)
- Fahren Sie mit der gerichteten Differenzierung der Kontroll- und verschiedenen CRISPR-Klone von hESCs (H9) in Richtung Neuroektodermschicksal fort, indem Sie etablierten Protokollen folgen, wie zuvor unter Verwendung der dualen smad-Hemmmethodebeschrieben 9,10,11.
- Bei einer Konfluenz von 90 % werden die Zellen zunächst 24 Stunden lang mit LDN193189 (200 nM) und SB431542 (10 μM) in 100 % KSR-Medien behandelt, gefolgt von der Zugabe von XAV939 (2 μM) für weitere 2 Tage.
- Reduzieren Sie nach 3 Tagen den Anteil an KSR-Medien (Knockout-DMEM, ergänzt mit 15 % (v/v) KSR, 1 % (v/v) L-Glutamin, 1 % (v/v) P/S, 1 % (v/v) 10 mM MEM und 0,1 % (v/v) 2-Mercaptoethanol (75 %, 50 %, 25 %) durch Kombination mit N2-Medien (DMEM/F12 ergänzt mit 1x N2-Supplement, 1x P/S, ) über einen Zeitraum von 8 Tagen auf 100 % N2-Medien übertragen.
- Zu Beginn von Tag 12 fixieren Sie die Zellen für die Immunfärbung mit PAX6 als Neuroektodermmarker.
- Für Studien zur Bestimmung des Mesoderms und des endodermalen Schicksals ist ein auf CHIR99021 kleiner Moleküle basierender Ansatz zu verwenden, der in früheren Veröffentlichungenbeschrieben wurde 12,13.
- Wenn die Zellen eine Konfluenz von 70 % erreichen, wird sie 24 h lang mit 3 μM CHIR99021 in definitiven Endodermmedien (DE) behandelt, gefolgt von einer Fixierung zur Immunfärbung mit Brachuary als Mesoderm-spezifischem Marker.
- Für das endodermale Stadium werden die Zellen für weitere 24 Stunden ohne Zusatz von CHIR99021 in DE-Medien kultiviert, bevor sie für die Immunfärbung mit FOXA2 fixiert werden.
- Für den EB-Formationsassay säen Sie die Zellen auf Zelloberflächen mit geringer Adhäsion ohne Matrigel aus, um die Zellen 24 Stunden lang unter Suspensionsbedingungen zu kultivieren, bevor Sie Mikroskopaufnahmen machen.
5. Western-Blot-Analyse
- Waschen Sie die Zellen zweimal mit PBS und lysieren Sie sie mit 1x RIPA-Puffer mit 1% SDS und 1x Protease- und Phosphatasehemmer-Cocktail.
- Die Lysate werden durch Zentrifugation bei 16.000 x g für 10 min bei 4 °C gereinigt, gefolgt von der Sammlung der Überstände.
- Quantifizieren Sie das Gesamtzellprotein mit dem BCA-Protein-Assay-Kit gemäß den Anweisungen des Herstellers. Stellen Sie die Proben auf 2 μg/μl mit 4 × Füllstoffpuffer ein.
- Denaturieren Sie die Proteinproben 10 Minuten lang bei 70 °C, laden Sie gleiche Mengen jeder Probe und lösen Sie sie mit SDS-PAGE-Gelen mit einem Gradienten von 4 %bis 12 % auf, gefolgt von der Übertragung auf die PVDF-Membran bei einer konstanten Spannung von 100 V für 1 h bei 4 °C.
- Blockieren Sie die Membranen mit 5 % fettfreier Milch und inkubieren Sie in primären Antikörperverdünnungen bei 4 °C über Nacht mit Rotation.
- Anschließend werden die Membranen 5x mit PBST-Puffer gewaschen und 1 h bei Raumtemperatur in HRP-konjugierten Sekundärantikörpern inkubiert.
- Die Membranen erneut mit PBST 5x waschen, mit chemilumineszierendem Substrat inkubieren und mit Röntgenfilmen entwickeln.
6. Immunfärbung
- Säen Sie die Zellen auf P4-Well-Platten und inkubieren Sie sie mindestens 24 Stunden lang, bevor Sie fixiert werden, um eine ordnungsgemäße Anheftung der Zellen an Oberflächen zu ermöglichen.
- Waschen Sie die Zellen 3x mit PBS, um abgestorbene Zellen sowie Medienbestandteile zu entfernen, gefolgt von einer Fixierung mit 4% PFA für 15 Minuten bei Raumtemperatur.
- Permeabilisieren Sie die Zellen mit 0,3 % Triton X-100, gefolgt von einer Blockierung für unspezifische Bindung durch Verwendung von 2 % BSA in PBS für 1 h bei Raumtemperatur.
- Die Proben werden mit Primärantikörpern (OCT4, NANOG, SOX2, KI67, PAX6, Brachuary, FOXA2) inkubiert, die über Nacht bei 4 °C in 1 % BSA verdünnt werden.
- Waschen Sie die Zellen 3x mit PBS und inkubieren Sie sie mit geeigneten Sekundärantikörpern (Ziege gegen Maus 488, Ziege gegen Kaninchen 488, Ziege gegen Maus 546, Ziege gegen Kaninchen 546), verdünnt in 1% BSA für 1 h bei Raumtemperatur.
- Zum Schluss werden die Proben mit PBS 3x gewaschen und mit DAPI gegengefärbt und mit einem Fluoreszenzmikroskop abgebildet.
Ergebnisse
Klonierung von L2HGDH sgRNAs in lentiCRISPRv2 puro
Der lentiCRISPRv2 puro-Vektor wurde kommerziell gewonnen (siehe Materialtabelle) und mit BsmB1 verdaut, was zur Freisetzung eines 1,8 Kb großen Stufferfragments führte. Wie in Abbildung 2A gezeigt, wurde ein vollständiger Aufschluss des Vektors beobachtet. Für jedes Konstrukt wurden sechs Klone auf das Vorhandensein oder Fehlen von Inserts untersucht, wobei eine reverse sgRNA-Sequenz als Primer und ein forward-Primer (U6-459F) aus der Vektorsequenz verwendet wurden. Bei diesem Ansatz ergaben nur die Klone mit dem Insert ein PCR-Produkt von 288 bp, und negative Klone, die nicht die umgekehrte Sequenz von sgRNA enthielten, führten zu keinem PCR-Produkt, wie in Abbildung 2B gezeigt.
H9-Infektionen, stabile Selektionen und Western Blot
hES-Zellen wurden mit lentiviralen Partikeln infiziert, die zwei verschiedene sgRNAs exprimierten, die auf das Exon 1 des L2HGDH-Gens abzielen, und mit Puromycin selektiert. Die Western-Blot-Analyse wurde an heterogenen Populationen von stabil co-exprimierenden sgRNA- und Cas9-Zellen durchgeführt, um festzustellen, welche der beiden verwendeten sgRNAs bei der Induktion der Gendeletion effizienter war. Wie in Abbildung 3A gezeigt, war L2HGDH-sgRNA-1 effizienter als die andere Sequenz bei der Verringerung der Expression von L2HGDH in heterogenen Zellpopulationen.
Klonale Selektion, Expansion und Proteinanalyse
Einzelzellselektion und klonale Expansion wurden für Zellen durchgeführt, die L2HGDH-sgRNA-1 exprimieren. Die Vertiefungen, die einzelne Klone lieferten, wurden markiert und gezüchtet, bis eine ausreichende Größe erreicht war, damit die Kolonien (in der Regel 2 Wochen) weiter expandieren, kryokonservieren und analysieren konnten (Abbildung 3B). Insgesamt wurden 22 Klone mittels Western-Blot-Analyse analysiert, die fünf homozygote Knockouts (23%) für L2HGDH ergab. Dieses Ergebnis zeigt eine hohe Effizienz des Gen-Knockouts unter Verwendung von lentiviralem Gentransfer für eine stabile Co-Expression von sgRNA und Cas9 aus einer einzigen Kassette. Abbildung 4 zeigt eine Western-Blot-Analyse der fünf homozygoten Knockout-Klone zusammen mit Kontrollzellen, die eine um 100% reduzierte Expression im Vergleich zur Kontrolle in den ausgewählten Klonen zeigt.
Analyse auf DNA-Ebene
Um die CRISPR-Mutationsstelle stromaufwärts der PAM-Erkennungssequenz von Cas9 zu kartieren, wurde die Mutation Sites Based Specific Primers Polymerase Chain Reaction (MS-BSP PCR) unter Verwendung genomischer DNA von Kontrollzellen und von fünf verschiedenen Klonen durchgeführt. Wie in Abbildung 5 gezeigt, weisen zwei Klone (A5 und B4) Mutationen in unmittelbarer Nähe der PAM-Erkennungssequenz auf, während die anderen drei Klone (A1, A9 und B12) zeigten, dass die Mutation möglicherweise nicht sehr nahe an der PAM-Stelle liegt und weiter stromaufwärts der 20bp-sgRNA-Sequenz innerhalb des Exons 1 des L2HGDH-Gens liegt.
Um die genauen Mutationen in der DNA zu finden, wurden gereinigte PCR-Produkte aus Kontrollzellen (Vektor infizierte nur H9) sowie drei verschiedene CRISPR-Klone, A5, A1 und B4, einer Sanger-Sequenzierung unterzogen und Mutationsstellen mit dem Multiple-Alignments-Tool clustalw stromaufwärts der PAM-Stelle8 kartiert. Die Ergebnisse zeigten eine Deletionsmutation im A5-Klon stromaufwärts der PAM-Stelle. Die beiden anderen Klone, A1 und B4, zeigten eine Insertionsmutation stromaufwärts der PAM-Stelle, wie in Abbildung 6 dargestellt.
Funktionale Analyse von Klonen
Nach der Generierung von CRISPR-Knockout-Zelllinien in hES-Zellen besteht der nächste Schritt darin, deren Pluripotenz und Differenzierungspotenzial zu bestätigen. Zu diesem Zweck testeten wir verschiedene Pluripotenzmarker für drei verschiedene Klone, A1, A5 und B4, und verglichen die Ergebnisse mit Kontrollzellen (nur vektorinfizierte H9-Zellen). Die Ergebnisse zeigten keine Veränderung des Pluripotenzpotenzials von Knockout-Zelllinien, das durch Immunfärbung für OCT4-, NANOG- und SOX2-Marker bestimmt wurde. Als nächstes wurden die Kontroll- und Knockout-Zelllinien auch für den Zellproliferationsmarker KI67 gefärbt, der ebenfalls keine Veränderung der Knockout-Zelllinien im Vergleich zur Kontrolle zeigte. Diese Ergebnisse zeigen, dass der CRISPR-Knockout die Selbsterneuerungseigenschaften dieser Zellen nicht beeinflusste. Darüber hinaus wurden auch die EB- und Koloniebildung nicht beeinflusst (Abbildung 7). Mit Hilfe von in vitro Methoden, die in unserem Labor etabliert wurden, wurden alle Klone erfolgreich in drei Keimblattzellen differenziert, was durch Immunfärbung für PAX6 (Neuroektodermmarker), Brachyury (Mesodermmarker) und FOXA2 (Endodermmarker) bestätigt wurde. Es wurde keine Veränderung des Differenzierungspotenzials von Knockout-Zelllinien im Vergleich zu Kontrollzellen beobachtet, was zeigte, dass die Knockout-Zellen ihr Differenzierungspotenzial beibehielten (Abbildung 8).

Abbildung 1: Schematische Darstellung des Studiendesigns in 5 Schritten. Schritt 1: Design und Klonierung von sgRNAs in einen Bsmb1-verdauten V2-CRISPR-Vektor. Schritt 2: Erzeugung von lentiviralen Partikeln unter Verwendung von Eintrittsvektoren, die sgRNA tragen, zusammen mit Verpackungsplasmiden durch Co-Transfektion HEK293T Zellen mittels Polyfektionen und Viruskonzentration mittels Ultrazentrifugation. Schritt 3: Infektion von hES-Zellen mit lentiviralen Partikeln bei einem MOI von 10-20 und stabile Zellselektion mit Puromycin, gefolgt von der Analyse heterogener Zellpopulationen, um die Effizienz von sgRNAs zu sehen. Schritt 4: Klonierung einzelner Zellen durch Begrenzung der Verdünnungsmethode aus den Zellen, die in heterogenen Populationen den maximalen Knockdown zeigten, und Selektion einzelner Klone. Schritt 5: Erweiterung einzelner Klone und Analyse auf DNA-Ebene, Protein- und Funktionsanalyse von Klonen auf Stamm- und Differenzierungspotenzial. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Klonierung von sgRNAs im lentiCRISPR V2.puro-Vektor. (A) Verdau des lentiCRISPR V2.puro-Vektors mit dem Restriktionsenzym BsmB1. Der Pfeil zeigt ein 1,8 KB großes Stufferfragment, das beim Aufschluss des Vektors freigesetzt wird. (B) Kolonie-PCR-Analyse für verschiedene Klone der L2HGDH sgRNA-Konstrukte 1 und 2. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Western Blots und klonale Vermehrung mittels Einzelzellselektion. (A) Western-Blot-Analyse von zwei verschiedenen biologischen Replikaten (S1 und S2) heterogener Populationen von stabil co-exprimierender sgRNA (L2H-1 und L2H-2) und Cas9 zusammen mit Kontroll-(C) H9-hES-Zellen. (B) Einzelzellselektion und klonale Expansion von H9-Zellen, die L2HGDH-sgRNA-1 stabil exprimieren. Pfeile zeigen Vertiefungen mit 96-Well-Platten an, die nur einzelne Klone zeigen. Maßstabsleiste = 100 μM. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Western-Blot-Analyse von verschiedenen Klonen von H9. (A-C) Die Western-Blot-Analyse von Gesamtzelllysaten von 22 Klonen ergab fünf homozygote Knockouts (23%) für L2HGDH. (D) Bestätigung von L2HGDH-Knockouts aus einem anderen biologischen Replikatexperiment mittels Western Blot. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
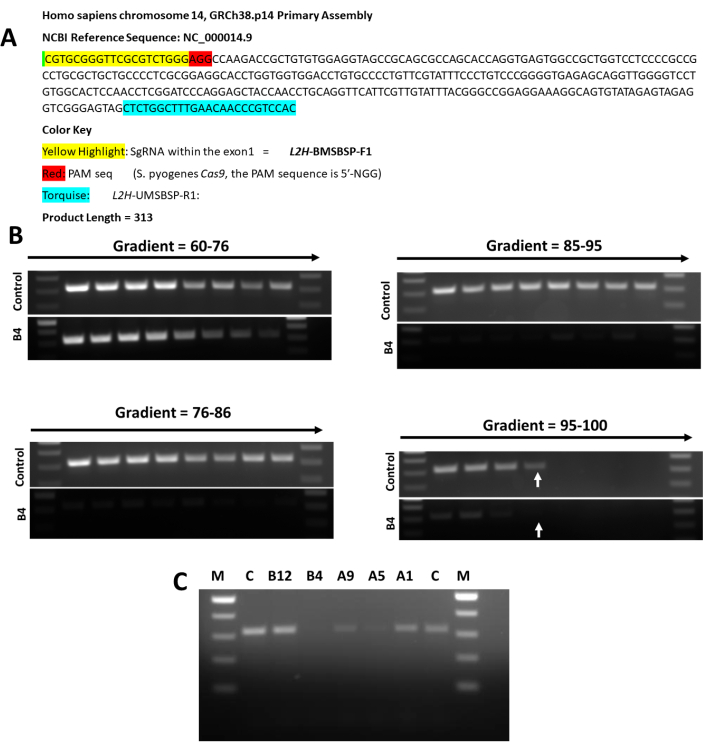
Abbildung 5: MSBSP PCR-Analyse verschiedener H9-Klone. (A) Sequenzanalyse des L2HGDH-Exons 1, das für die MSBSP-PCR-Analyse verwendet wird. (B) Optimierung der Annealing-Temperatur für die MSBSP PCR-Analyse mittels Kontrolle und H9 V2 L2HGDH KO Klon B4. Bei 96,8 zeigt das Steuerelement ein Band an, aber B4 zeigt kein Band an, wie durch Pfeile dargestellt. (C) Die Analyse aller homozygoten KO-Klone führte zu der folgenden Kartierung der Mutationsstelle innerhalb von Exon 1. A1 = In/Del nicht in der Nähe des PAM-Standorts; A5 = In/Del In der Nähe des PAM-Standorts; A9 = In/Del Ziemlich nah am PAM-Standort; B4 = In/Del Sehr nah am PAM-Standort B12 = In/Del nicht in der Nähe des PAM-Standorts. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Ausrichtung von Sanger-sequenzierten PCR-Produkten mit der Referenzsequenz des L2HGDH-Exons 1. Mehrere Alignments von Sanger-sequenzierten PCR-Produkten aus A1, A5 und B4 und Kontrollzellen wurden zusammen mit einer Referenzsequenz innerhalb des ersten Exons von L2HGDH aus der NCBI-Datenbank durchgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
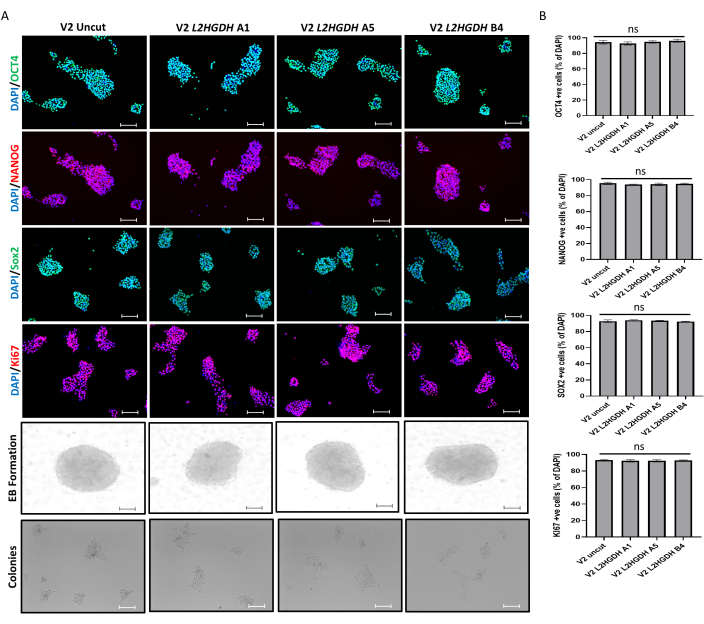
Abbildung 7: Funktionelle Charakterisierung von CRISPR-Knockout-Zelllinien. (A) Die Zellen wurden im Vergleich zu den kontrollierten H9-hES-Zellen auf Pluripotenzmarker (OCT4, NANOG, SOX2), Selbsterneuerung (KI67), EBs und Koloniebildung immungefärbt. Maßstab: 100 μM. (B) Der Prozentsatz der positiv (+ve) exprimierenden Zellen wurde relativ zur DAPI-Färbung berechnet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 8: Differenzierungspotenzial von CRISPR-Knockout-Zelllinien. Funktionelle Charakterisierung von CRISPR-Knockout-Zelllinien hinsichtlich ihres Differenzierungspotenzials anhand von Markern für PAX6 (Neuroektoderm), Brachyury (Mesoderm) und FOXA2 (Endoderm) in Bezug auf die kontrollinfizierten H9-Zellen. Maßstabsbalken: 100 μM. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Schritt des Protokolls | Zeitaufwand |
| Klonierung von sgRNAs in LentiCRISPRv2 | 4 Tage |
| Lentivirus-Produktion und Titerbestimmung | 5 Tage |
| hES-Infektionen, stabile Zellselektion und Expansion | 2 Wochen |
| Klonale Selektion und Expansion einzelner Zellen | 2 Wochen |
| Funktionale Analyse von Klonen | 2 Wochen |
Tabelle 1: Zeitplan für die Protokollschritte und geschätzter Zeitaufwand.
Diskussion
In dieser Studie wurde eine Methode standardisiert, die durch die CRISPR-Cas9-Technologie hocheffiziente und kostengünstige Gendeletionen in hES-Zellen ermöglicht. Mit dieser Methode wurde innerhalb von 3-4 Wochen eine homozygote Deletion des L2HGDH-Gens in hES-Zellen erreicht, beginnend mit der hES-Infektion bis hin zur einzelzelligen klonalen Selektion und Vermehrung (Tabelle 1). Obwohl CRISPR-Cas9-vermittelte Genmanipulationen in den meisten Zellen durch transiente Transfektionen erreicht werden können, wird dies in Stammzellen aufgrund der schlechten Transfektionseffizienz und der hohen Zelltoxizität schwierig. Verschiedene Studien haben berichtet, dass bis zu 10% der wiedergefundenen Klone Deletionen aufwiesen. Zum Beispiel berichteten Mali et al. über eine Genom-Editing-Effizienz von 2 % bis 4 % in humanen induzierten pluripotenten Stammzellen, nachdem sie auf AAVS1-Locus3 abzielten. Eine andere Studie berichtete über eine Editierungseffizienz von 1,3 % bis 8,4 % in naiven hES-Zellen, um auf TUNA abzuzielen, eine lange nicht-kodierende RNA, die an der Pluripotenz und neuronalen Differenzierung beteiligt ist14. Ocana et al. berichteten über eine Effizienz von bis zu 10 %, nachdem das CFTR-Gen in humanen pluripotenten Stammzellen angegriffen wurde.15Daher ist eine effiziente Verabreichungsmethode von Cas9-gRNA erforderlich, um eine effiziente Geneditierung zu erreichen, und dies gilt insbesondere für Stammzellen, da diese Zellen im Vergleich zu typischen somatischen Zellen und immortalisierten Zelllinien ein erhöhtes Maß an Empfindlichkeit und Resistenz gegenüber transienten Verabreichungsmethoden aufweisen5.
In der vorliegenden Studie wird über einen hocheffizienten Weg zur Gewinnung homozygoter CRISPR-Knockout-Zelllinien berichtet, indem die lentivirale Verabreichung von sgRNAs in hESCs verwendet wird. Die Verwendung lentiviraler Vektoren zur Verabreichung von CRISPR-Cas9 in die hES-Zellen erwies sich als viel effizienter und weniger zeitaufwändig bei der Gendeletion als die Verwendung nicht-viraler Vektortransfektionen auf chemischem oder mechanischem Wege (Daten nicht gezeigt). Dies führte zu einer signifikant höheren Effizienz der Genomeditierung (~23%) nach klonaler Selektion und Vermehrung einzelner Klone. Durch die Kombination der lentiviralen vermittelten Verabreichung von sgRNAs und Cas9 in hESCs sowie eines kostengünstigen Ansatzes für die Produktion von Lentiviren ist die vorliegende Methode daher nicht nur hocheffizient und weniger zeitaufwändig, sondern auch kostengünstig.
Hier sind einige der Empfehlungen für einen erfolgreichen Ansatz: (1) Verwendung eines einzelnen lentiviralen Vektorsystems, das sowohl Cas9 als auch sgRNAs exprimiert. (2) Das Vorhandensein eines Antibiotika-Selektionsmarkers, wie z. B. Puromycin, auf demselben Plasmid, das Cas9 und sgRNAs exprimiert, stellt sicher, dass fast 100 % der Zellen positiv für die Cas9-sgRNA-Expression sind. Dies erhöht die Wahrscheinlichkeit erfolgreicher Gen-Editing-Ereignisse erheblich. (3) Um für Infektionen in hES-Zellen nützlich zu sein, ist es unerlässlich, die lentiviralen Vektoren zu konzentrieren, um hohe Titer von Viren zu erhalten. Unter den verschiedenen verwendeten Techniken besteht der wirtschaftlichste Ansatz darin, große LVS-Volumina zu verwenden und sie um das 100-500-fache des ursprünglichen Volumens zu konzentrieren, um Titer im Bereich von 107 bis 108 IE/ml zu erreichen. (4) Für das kostengünstige und effiziente Verfahren zur HEK293T von Zelltransfektionen mit den Plasmiden ist die Verwendung von PEI (Polyethylenimin) für die lentivirale Vektortransfektion für die Virusverpackung im Vergleich zu dem üblicherweise verwendeten Transfektionsreagenz Lipofectamin7 ebenso effizient und daher kostengünstig. (5) Die durchschnittlichen lentiviralen Titer müssen im Bereich von 5 × 107 I.E./ml liegen. (6) Eine klonale Vermehrung durch Einzelzellselektion ist erforderlich, um homozygote Deletionen des interessierenden Gens in diesen Zellen zu erreichen. (7) Obwohl frühere Studien darauf hingewiesen haben, dass die durch CRISPR-Cas9 vermittelte Geneditierung in humanen pluripotenten Stammzellen zu sehr wenigen Off-Target-Mutationen führt16,17, ist es dennoch ratsam, nach Off-Target-Stellen zu suchen. Dies kann durch effiziente gRNA-Designwerkzeuge zur Minimierung von Off-Target-Effekten und genomweite Sequenzierung von editierten Zelllinienerreicht werden 18, insbesondere für Experimente von klinischer Bedeutung. (8) Schließlich sollte der normale Karyotyp ausgewählter Klone mit Methoden wie G-Banding und hochauflösender Analyse überprüft werden, um sicherzustellen, dass es keine großflächigen genomischen Veränderungen gibt. Ein Mikronukleus-Assay kann auch durchgeführt werden, um die Häufigkeit der Mikrokernbildung zu beurteilen, was auf eine genomische Instabilität hinweisen kann.
Eine der größten Einschränkungen dieser Methode ist die Verwendung eines lentiviralen Vektorsystems, das zu einer stabilen genomischen Integration von Cas9 und sgRNAs führte, was zu einer konstitutiven Expression nach dem Editieren führte, was unerwünscht ist. In Zukunft kann diese Methode verbessert werden, indem konstitutive Promotoren durch einen medikamenteninduzierbaren Promotor ersetzt werden, der die Expression des Cas9-Gens antreibt, wie z. B. das Tet-on-System mit Doxycyclin-Behandlung zur kontrollierten Expression von Cas9.
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Diese Arbeit wurde durch Forschungsstipendien der Universität der Vereinigten Arabischen Emirate (VAEU) unterstützt - Grant #12M105, Grant #12R167 (Zayed Center for Health Sciences), 21R105 (Zayed Bin Sultan Charitable and Humanitarian Foundation (ZCHF)) und ASPIRE, die Säule des Technologieprogrammmanagements des Advanced Technology Research Council (ATRC) von Abu Dhabi, über das ASPIRE Precision Medicine Research Institute Abu Dhabi (ASPIREPMRIAD) mit der Fördernummer VRI-20-10.
Materialien
| Name | Company | Catalog Number | Comments |
| 2-MERCAPTOETHANOL | Invitrogen | 31350010 | |
| 38.5 mL, Sterile + Certified Free Open-Top Thinwall Ultra-Clear Tubes | Beckman Coulter | C14292 | |
| Accutase | Stem cell technologies | 7920 | |
| bFGF Recombinant human | Invitrogen | PHG0261 | |
| Brachyury Rabbit mAb | Abclonal | A5078 | |
| BsmBI-v2 | NEB | R0739S | |
| chir99021 | Tocris | 4423/10 | |
| Corning Matrigel Basement Membrane Matrix, LDEV-free | Corning | 354234 | |
| Cyclopamine | Stem cell technologies | 72074 | |
| DMEM media | Invitrogen | 11995073 | |
| DMEM NUTRIENT MIX F12 | Invitrogen | 11320033 | |
| DPBS w/o: Ca and Mg | PAN Biotech | P04-36500 | |
| Fetal bovie serum | Invitrogen | 10270106 | |
| FoxA2/HNF3β | CST | 8186 | |
| GAPDH (14C10) Rabbit mAb Antibody | CST | 2118S | |
| Gentle Cell Dissociation Reagent | Stem cell technologies | 7174 | |
| HyClone Non Essential Amino Acids (NEAA) 100x Solution | GE healthcare | SH30238.01 | |
| Ki-67 (D3B5) Rabbit mAb | CST | 9129 | |
| KnockOut Serum Replacement | Invitrogen | 10828028 | |
| L GLUTAMINE, 100x | Invitrogen | 2924190090 | |
| L2H-BMSBSP-F1 | Macrogen | CGTGCGGGTTCGCGTCTGGG | |
| L2HGDH Polyclonal antibody | Proteintech | 15707-1-AP | |
| L2HGDH-SgRNA1-F | Macrogen | CACCGCGTGCGG GTTCGCGTCTGGG | |
| L2HGDH-SgRNA1-R | Macrogen | AAACCCCAGACGC GAACCCGCACGC | |
| L2HGDH-SgRNA2-F | Macrogen | CACCGCCCGCGG GCTTTTCGCCGG | |
| L2HGDH-SgRNA2-R | Macrogen | AAACCCGGCGAA AAGCCCGCGGGC | |
| L2H-SeqF1 | Macrogen | GCTAAAGAGCGC GGGTCCTCGG | |
| L2H-SeqR1 | Macrogen | GTGGACGGGTTG TTCAAAGCCAGAG | |
| L2H-UMSBSP-R1 | Macrogen | GTGGACGGGTTG TTCAAAGCCAGAG | |
| LentiCRISPRv2 | Addgene | 52961 | |
| mTesR1 complete media | Stem cell technologies | 85850 | |
| Nanog Antibody | CST | 3580 | |
| NEUROBASAL MEDIUM 1x CTS | Invitrogen | A1371201 | |
| Neuropan 2 Supplement 100x | PAN Biotech | P07-11050 | |
| Neuropan 27 Supplement 50x | PAN Biotech | P07-07200 | |
| Oct-4 Antibody | CST | 2750 | |
| Pax6 (D3A9V) XP Rabbit mAb | CST | 60433 | |
| PENICILLIN STREPTOMYCIN SOL | Invitrogen | 15140122 | |
| pMD2.G | Addgene | 12259 | |
| Polybrene infection reagent | Sigma | TR1003- G | |
| Polyethylenimine, branched | Sigma | 408727 | |
| psPAX2.0 | Addgene | 12260 | |
| Puromycin | Invitrogen | A1113802 | |
| qPCR Lentivirus Titer Kit | Abm | LV900 | |
| Rock inhibitor Y-27632 dihydrochloride | Tocris | 1254 | |
| SB 431542 | Tocris | 1614/10 | |
| Sox2 Antibody | CST | 2748 | |
| Sucrose | Sigma | 57-50-1 | |
| TRYPSIN .05% EDTA | Invitrogen | 25300062 | |
| U6-459F | Macrogen | GAGGGCCTATT TCCCATGATTC | |
| Wizard Genomic DNA Purification Kit | Promega | A1120 | |
| XAV 939 | Tocris | 3748/10 |
Referenzen
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jiang, F., Doudna, J. A. CRISPR-Cas9 structures and mechanisms. Annu Rev Biophys. 46 (1), 505-529 (2017).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Zhang, Z., et al. CRISPR/Cas9 genome-editing system in human stem cells: Current status and future prospects. Mol Ther Nucleic Acids. 9, 230-241 (2017).
- Qiu, Z., et al. MYC regulation of D2HGDH and L2HGDH influences the epigenome and epitranscriptome. Cell Chem Biol. 27 (5), 538-550.e537 (2020).
- Sheikh, M. A., Ansari, S. A. Lentiviral mediated delivery of shRNAs to hESCs and NPCs using low-cost cationic polymer polyethylenimine (PEI). J Vis Exp. (183), e63953 (2022).
- Guo, J., et al. A simple and cost-effective method for screening of CRISPR/Cas9-induced homozygous/biallelic mutants. Plant Methods. 14 (1), 40 (2018).
- Ardah, M. T., Parween, S., Varghese, D. S., Emerald, B. S., Ansari, S. A. Saturated fatty acid alters embryonic cortical neurogenesis through modulation of gene expression in neural stem cells. J Nutr Biochem. 62, 230-246 (2018).
- Parween, S., et al. Higher O-GlcNAc levels are associated with defects in progenitor proliferation and premature neuronal differentiation. Front Cell Neurosci. 11, 415 (2017).
- Parween, S., et al. Nutrient-sensitive protein O-GlcNAcylation modulates the transcriptome through epigenetic mechanisms during embryonic neurogenesis. Life Sci Alliance. 5 (8), e202201385 (2022).
- Varghese, D. S., et al. Developmental modeling of hepatogenesis using obese iPSCs-hepatocyte differentiation uncovers pathological features. Cell Death Dis. 13 (7), 670 (2022).
- Varghese, D. S., Alawathugoda, T. T., Ansari, S. A. Fine-tuning of hepatocyte differentiation from human embryonic stem cells: Growth factor. Stem Cells Int. 2019 (5), 5968236 (2019).
- Jacobs, E. Z., et al. CRISPR/Cas9-mediated genome editing in naïve human embryonic stem cells. Sci Rep. 7 (1), 16650 (2017).
- Cuevas-Ocaña, S., et al. A cell-based optimized approach for rapid and efficient gene editing of human pluripotent stem cells. Int J Mol Sci. 24 (3), 12345 (2023).
- Veres, A., et al. Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN-targeted human stem cell clones detected by whole-genome sequencing. Cell Stem Cell. 15 (1), 27-30 (2014).
- Smith, C., Gore, A., Yan, W., Abalde-Atristain, L., Li, Z., He, C. Whole-genome sequencing analysis reveals high specificity of CRISPR/Cas9 and TALEN-based genome editing in human iPSCs. Cell Stem Cell. 15 (1), 12-13 (2014).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target effects in CRISPR/Cas9-mediated genome engineering. Mol Ther Nucleic Acids. 4 (7), e264 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten